Method Article
Doppia strategia di interferenza CRISPR per il targeting delle interazioni letali sintetiche tra RNA non codificanti nelle cellule tumorali
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo studio presenta un doppio sistema CRISPRi che ha come bersaglio lunghi RNA non codificanti in cellule di melanoma. Consente il knockdown genico combinatorio e lo screening letale sintetico, identificando le interazioni lncRNA specifiche del cancro per potenziali strategie terapeutiche.
Abstract
Gli RNA lunghi non codificanti (lncRNA) rappresentano una classe vasta e funzionalmente diversificata di molecole di RNA, con oltre 100.000 previste nel genoma umano. Sebbene gli lncRNA siano meno conservati tra le specie rispetto ai geni codificanti proteine, svolgono un ruolo critico nella regolazione genica, nelle interazioni della cromatina e nella progressione del cancro. Il loro coinvolgimento nel cancro li rende promettenti bersagli terapeutici. L'interferenza CRISPR (CRISPRi), che utilizza Cas9 cataliticamente inattivo fuso con un repressore trascrizionale come KRAB-MeCP2, offre un metodo preciso per colpire gli lncRNA nucleari e valutarne le funzioni. Questo studio introduce un doppio sistema CRISPRi che utilizza le tecnologie CRISPRi ortogonali di Staphylococcus aureus e Streptococcus pyogenes basate su dCas9-KRAB-MeCP2, ottimizzate per il targeting combinatorio di lncRNA in cellule di melanoma umano. Il protocollo facilita il knockdown genico combinatorio o lo screening letale sintetico di coppie di lncRNA, fornendo un nuovo strumento per la ricerca sul cancro. Esplorando la letalità sintetica tra gli lncRNA, questo approccio può aiutare a identificare le interazioni tra lncRNA critiche per la sopravvivenza delle cellule tumorali, offrendo nuove strategie terapeutiche. Viene dimostrata la funzionalità del doppio sistema, evidenziando il suo potenziale nell'identificazione di interazioni critiche tra lncRNA specifiche per il cancro.
Introduzione
Sebbene meno del 3% del genoma umano codifichi proteine, circa l'80% del genoma è trascritto 1,2. Tra le unità trascrizionali non codificanti, decine di migliaia sono classificate come RNA lunghi non codificanti (lncRNA) superiori a 200 nucleotidi e si stima che il numero totale di lncRNA umani superi i 100.000 3,4. A differenza dei geni codificanti, gli lncRNA sono meno conservati tra le specie. Dato che gli esseri umani condividono il 99% del loro genoma con primati come gli scimpanzé, si ipotizza che gli lncRNA abbiano un'influenza molto maggiore sull'evoluzione fenotipica 5,6. Questi risultati indicano importanti funzioni cellulari degli lncRNA. Sebbene la regolazione degli lncRNA e le loro interazioni con le proteine leganti l'RNA e altri RNA rimangano non completamente comprese, e molti lncRNA debbano ancora essere completamente annotati, è chiaro che gli lncRNA mostrano modelli di espressione specifici per cellule e tessuti in salute e malattia, come il cancro 7,8,9,10. Sono implicati in diverse funzioni, tra cui la regolazione della trascrizione genica, il coinvolgimento nelle interazioni della cromatina11, l'elaborazione dell'RNA12, la stabilizzazione dell'RNA13 e la regolazione della traduzione14.
Nel cancro, lncRNA altamente diversi e specifici per tipo di cellula influenzano lo sviluppo e le metastasi tumorali regolando l'espressione genica, evidenziando il loro potenziale come preziosi bersagli terapeutici15. Oltre al rilevamento di lncRNA come biomarcatori in campioni tumorali16, il targeting di lncRNA specifici per tumore per interrompere le loro funzioni a valle ha un potenziale significativo sia nelle applicazioni cliniche che nella ricerca di base. Gli approcci basati sull'RNA per chiarire i ruoli degli lncRNA includono oligonucleotidi antisenso (ASO), RNA a forcina corta e piccoli RNA interferenti (siRNA)17,18. Mentre il siRNA è comunemente utilizzato per gli screening di silenziamento genico, il knockdown basato su siRNA è limitato al citoplasma19. Tuttavia, lncRNA opera frequentemente all'interno del nucleo.
In alternativa, l'interferenza CRISPRi (Clustered Regularly Interspaced Short Palindromic Repeats) può essere utilizzata per inibire gli lncRNA nei tumori umani20. Inoltre, gli screening CRISPRi per l'intero genoma possono essere facilmente programmati e mirano a un'ampia gamma di geni codificanti e non codificanti per esaminarne l'impatto funzionale21,22. In CRISPRi un Cas9 catalitico deficiente (dCas9) è fuso in un dominio repressore trascrizionale, come il dominio23 della scatola associata a Krüppel (KRAB). La repressione genica da parte di dCas9-KRAB è guidata da un RNA guida (gRNA) verso la regione di interesse. CRISPRi controlla i geni a livello di DNA, portando a una maggiore efficienza e ai fenotipi desiderati con perdita di funzione in contrasto con l'interferenza dell'RNA, che è attiva a livello post-trascrizionale24. In risposta alla limitata efficacia di KRAB nel silenziamento del bersaglio, è stata introdotta una fusione di KRAB e MeCP2 con dCas9 come strategia di repressione più efficace25.
Sebbene il silenziamento di un singolo gene possa influenzare la vitalità del cancro, le interazioni letali sintetiche doppie o multiple possono salvare le cellule tumorali dalla morte cellulare26. La letalità sintetica coinvolge due o più geni, ognuno dei quali può compensare la funzione dell'altro. Per superare i problemi con gli screening knockdown di un singolo gene, le doppie strategie CRISPR mirate a coppie di geni codificanti proteine letali offrono un approccio promettente27.
Qui, presentiamo un protocollo per l'uso combinatorio del targeting ortogonale basato su CRISPRi di lncRNA o altre coppie di RNA non codificanti utilizzando Staphylococcus aureus e Streptococcus pyogenes dCas9 fusi a KRAB-MeCP2 in cellule di melanoma umano (vedi Figura 1). Il protocollo può essere utilizzato per il knockdown combinato classico di CRISPR o come screening basato su CRISPRi di coppie letali sintetiche nel cancro. L'uso di due diverse specie di dCas9, SpCas9 e SaCas9, nell'approccio CRISPRi consente di mirare in modo indipendente a loci genomici distinti con una reattività crociata minima, migliorando la specificità e la flessibilità e garantendo al contempo un'elevata selettività sul bersaglio. Vengono utilizzati diversi motivi adiacenti protospaziatori (PAM): NGG per SpCas9 e NNGRRT per SaCas9. Il doppio sistema dCas9 affronta sfide come la limitata compatibilità con gli sgRNA, consentendo una modulazione precisa e simultanea e riducendo la competizione del complesso sgRNA-RNP quando si utilizza un singolo tipo di dCas9. Questa innovazione migliora la robustezza e la versatilità dello screening della doppia libreria di sgRNA. In conclusione, forniamo un doppio sistema CRISPRi completamente funzionale in cellule di melanoma come modello di cancro.
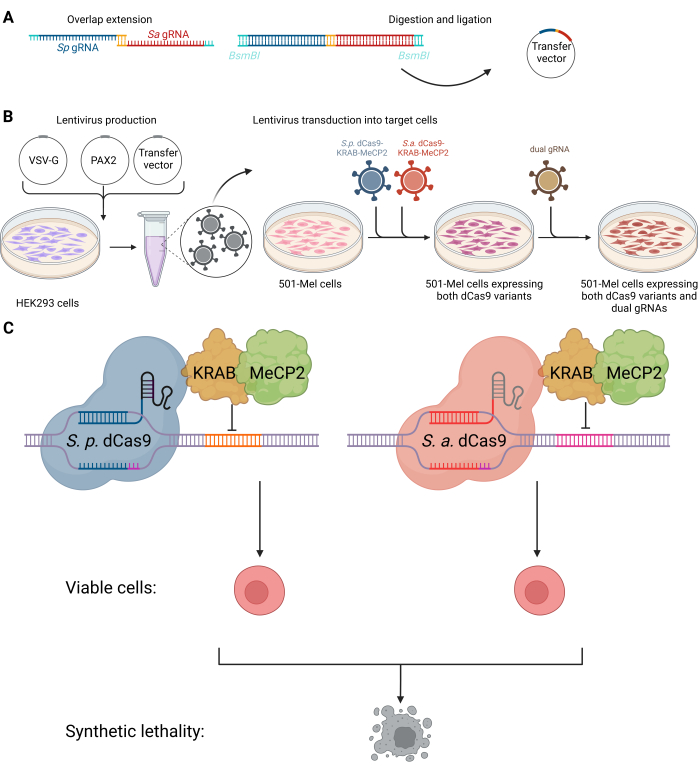
Figura 1: Schema del doppio sistema CRISPRi per indirizzare le interazioni letali sintetiche di RNA non codificanti. (A) Procedura di clonaggio del vettore gRNA duale. (B) Le cellule HEK293 sono state trasfettate con il plasmide dell'involucro VSV-G, il plasmide PAX2 di imballaggio e il vettore di trasferimento per produrre lentivirus per la successiva trasduzione in cellule 501-mel. I lentivirus contenenti le informazioni genetiche per SpdCas9-KRAB-MeCP2 (blu) e SadCas9-KRAB-MeCP2 (rosso) sono stati integrati contemporaneamente in cellule 501-mel. Dopo la selezione degli antibiotici, è stata eseguita una seconda trasduzione lentivirale per integrare i doppi gRNA desiderati (verde oliva). (C) Le cellule 501-mel che esprimono le varianti dCas9 interagiscono con i loro corrispondenti gRNA per silenziare l'espressione genica bersaglio, con conseguente morte delle cellule tumorali. Creato con BioRender.com. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
La linea cellulare di melanoma umano 501-mel (RRID: CVCL_4633) è stata gentilmente fornita dall'Aifantis Lab (New York University). La linea cellulare Lenti-X 293T HEK è stata acquistata da Takara Bio. Queste linee cellulari sono state coltivate nel glucosio medio di Eagle modificato (DMEM) di Dulbecco, integrato con il 10% di siero fetale bovino (FBS) a 37 °C in un'atmosfera di CO2 al 5% in condizioni sterili. Le cellule sono state fatte passare fino a raggiungere l'80%-90% di confluenza. Le linee cellulari sono state regolarmente testate per la contaminazione da micoplasma.
1. Progettazione di gRNA utilizzando una piattaforma di progettazione di gRNA online
- Progettare un gRNA specifico per SpCas9 e un gRNA specifico per SaCas9 per ogni lncRNA bersaglio per valutare quale variante produce risultati di knockdown superiori.
- Prendi di mira gli lncRNA desiderati, qui RP11-120D5.1 (RP11) e XLOC_030781 (XLOC), utilizzando il gRNA più efficiente per ogni S. pyogenes (gRNA-sp) Cas9 e S. aureus (gRNA-sa) Cas9.
NOTA: I gRNA di S. pyogenes possono essere estratti da Petroulia et al.28. - Segui le raccomandazioni specifiche del sito per dare priorità all'attività on-target più elevata, espressa da punteggi on-target elevati, riducendo al minimo le corrispondenze off-target come suggerito dagli strumenti di progettazione del gRNA.
- Scegli il genoma GRCh38 (hg, Homo sapiens). Selezionare il sito PAM per gli specifici SpCas9 (NGG) e SaCas9 (NNGRRT).
- Usa il metodo di punteggio fuori bersaglio di Hsu et al.29. Posizionare i gRNA in una finestra compresa tra -150 bp e +50 bp attorno al sito di inizio trascrizionale per ottenere prestazioni ottimali.
- Scegliere i gRNA con il punteggio più alto on-target e off-target (vedere la Tabella supplementare 1).
- Combina un gRNA di controllo non mirato o criptato, qui contro Rosa26, con ciascun gRNA bersaglio per creare un doppio vettore di gRNA, mirato a un lncRNA. La combinazione di gRNA bersaglio in un plasmide consente la modifica sito-specifica di entrambi i siti bersaglio.
NOTA: Il doppio vettore di gRNA che ospita il gRNA di controllo e il gRNA bersaglio consente il knockdown di un lncRNA bersaglio, mentre i doppi vettori di gRNA contenenti gRNA mirati ai siti consentono il knockdown simultaneo di lncRNA mirati.
2. Clonazione del gRNA
- Eseguire una PCR di estensione sovrapposta utilizzando due frammenti di DNA contenenti la sequenza di gRNA. Utilizzare il primer diretto U6 e il primer inverso H1 (vedi Tabella supplementare 2) per l'amplificazione come descritto da Najm et al.27.
- Impostare una reazione PCR da 50 μl con 25 μl di 2 master mix PCR ad alta fedeltà con tampone HF e 1 μl di ciascun frammento di DNA contenente il gRNA di S. pyogenes o S. aureus . Eseguire la reazione nelle seguenti condizioni cicliche: denaturazione iniziale a 98 °C per 30 s, seguita da 15 cicli di denaturazione a 98 °C per 5 s, ricottura a 55 °C per 10 s e allungamento a 72 °C per 15 s.
- Aggiungere 2,5 μl di innesco diretto U6 e 2,5 μl di innesco inverso H1 dopo aver completato i 15 cicli iniziali e sottoporre la reazione ad altri 20 cicli nelle stesse condizioni, con un'estensione finale per 5 minuti a 72 °C.
- Purificare i prodotti PCR utilizzando un kit di purificazione PCR secondo le istruzioni del produttore.
- Digerire 1 μg del plasmide modificato utilizzando 1 μL di enzima di restrizione BsmB1 e 1 μL di tampone 10x per 30 minuti a 37 °C in una reazione di 10 μL.
- Clonare i prodotti della PCR nel plasmide dual-gRNA-Zeo-GFP digerito (vedere la Figura 1 supplementare e il File 1 supplementare) utilizzando un protocollo di reazione di assemblaggio del DNA HiFi come descritto da altri30.
- Trasformare le cellule di E. coli chimicamente competenti mescolando 50 μL di coltura batterica con 4 μL di prodotto di assemblaggio secondo le istruzioni del produttore.
3. Produzione di lentivirus
ATTENZIONE: Maneggiare i lentivirus attivi in tutti i passaggi seguendo le linee guida di sicurezza appropriate. Eseguire sempre lavori con virus in una cabina di sicurezza di Classe 2 per evitare pericoli derivanti dall'inevitabile formazione di aerosol. Eseguire tutti i lavori al di fuori dell'armadio di sicurezza, come la centrifugazione, in contenitori a tenuta di aerosol e rotori approvati per l'uso con organismi del gruppo di rischio 2. Indossare indumenti protettivi adeguati, tra cui camice da laboratorio, guanti monouso e occhiali di sicurezza, in ogni momento nell'area di lavoro. Le superfici di lavoro devono essere trattate con una soluzione disinfettante e anche gli altri rifiuti devono essere trattati con una soluzione disinfettante, che consente loro di rimanere nell'armadio sigillato sotto la luce UV per almeno 240 minuti. Le pipette sierologiche e i puntali delle pipette utilizzati devono essere sciacquati in una soluzione disinfettante prima di essere smaltiti in flaconi autoclavabili. Successivamente, i rifiuti devono essere smaltiti nel contenitore dei rifiuti S2. I rifiuti S2 saranno inattivati attraverso l'autoclave. Le linee cellulari trasdotte possono essere manipolate in condizioni S1 non prima di 2 giorni dopo la trasduzione e solo dopo almeno due cambi completi del terreno di coltura con un terreno privo di virus.
- Contare le cellule HEK Lenti-X 293T mescolando 10 μL di cellule con 10 μL di soluzione di blu di tripano in un emocitometro. Piastra 4 x 106 celle Lenti-X 293T HEK per piastra da 10 cm per ottenere una confluenza del ~80% il giorno successivo.
- Lavora in un laboratorio di Classe 2 da questo passo in poi. Miscelare 500 μl di un mezzo di trasfezione adatto con il vettore di trasferimento SpdCas9-KRAB-MeCP2 da 11,25 μg (vedere il file supplementare 2), il plasmide dell'involucro VSV-G da 5,5 μg e il vettore pPAX2 da 16,5 μg. Incubare per 5 minuti a temperatura ambiente. Per la seconda variante dCas9, utilizzare il vettore di trasferimento SadCas9-KRAB-MeCP2 (vedi File supplementare 3).
- In una provetta separata, miscelare 500 μL di terreno sierico ridotto con 36 μL di reagente PEI (stock: 1 mg/mL). Incubare per 5 minuti a temperatura ambiente.
- Unire le miscele e incubare per 15 minuti a temperatura ambiente. Aggiungere la miscela con cautela goccia a goccia al terreno cellulare (6 mL) e incubare per una notte a 37 °C e 5% di CO2. Assicurarsi che le cellule rimangano attaccate per evitare una ridotta resa lentivirale.
- Gettare il terreno in un flacone di scarto autoclavabile utilizzando una pipetta sierologica dopo 12-15 ore. D'ora in poi, sciacquare i materiali di consumo, comprese le pipette sierologiche e i puntali per pipette, con la soluzione disinfettante prima di smaltirli nei rifiuti dell'autoclave. Aggiungere 5 mL di terreno DMEM fresco alle cellule trasfettate utilizzando una nuova pipetta.
- Raccogliere i lentivirus trasferendo il surnatante utilizzando una pipetta sierologica in una provetta canonica da 50 mL 48 ore dopo la trasfezione. Conservate il tubo canonico in frigorifero. Aggiungere 5 mL di terreno fresco alle cellule e incubare le cellule per altre 24 ore.
- Ripetere la raccolta dei lentivirus trasferendo il surnatante nella stessa provetta canonica da 50 mL contenente il primo raccolto. Aggiungere nuovamente 5 mL di terreno fresco, incubare per 24 ore e trasferire successivamente il surnatante nella provetta canonica da 50 mL. Conservare i surnatanti combinati del primo, secondo e terzo raccolto nella provetta canonica da 50 mL a 4 °C fino alla filtrazione e alla centrifugazione.
- Centrifugare il surnatante combinato per 5 minuti a 500 x g e 4 °C in contenitori a tenuta di aerosol e rotori approvati per l'uso con organismi del gruppo di rischio 2 per pellettare cellule staccate e detriti. Filtrare il surnatante attraverso filtri per siringa sterili da 0,2 μm per colture cellulari.
- Concentrare il surnatante filtrato a 1.000 x g e 4 °C utilizzando un'unità di filtro centrifugo con un appropriato taglio del peso molecolare da 100 kDa a circa 500 μL. Conservare le aliquote del virus da 50-100 μl a -80 °C fino a nuovo utilizzo.
4. Trasduzione del lentivirus e selezione di cellule trasfettate stabili
ATTENZIONE: Eseguire sempre lavori con virus in un armadio di sicurezza di Classe 2.
- Piastra di 501-mel in una piastra a 6 pozzetti a 2 x 105 cellule/pozzetto con un volume di coltura finale di 2 mL, 1 giorno prima della trasduzione.
- Sostituire il terreno di coltura con 2 mL di terreno DMEM preriscaldato integrato con polibrene a una concentrazione finale di 6 μg/mL.
- D'ora in poi, è richiesto il lavoro nel laboratorio di Classe 2. Aggiungere contemporaneamente alle cellule 50 μL di lentivirus contenenti SpdCas9-KRAB-MeCP2 e SadCas9-KRAB-MeCP2. Sciacquare i materiali di consumo, comprese le pipette sierologiche e i puntali per pipette, con la soluzione disinfettante prima di smaltirli nei rifiuti dell'autoclave. Agitare delicatamente la piastra e incubare a 37 °C in un'atmosfera di CO2 al 5%.
- Gettare il terreno in un flacone autoclavabile utilizzando una pipetta sierologica e aggiungere 2 mL di terreno fresco contenente blasticidina (10 μg/mL) e puromicina (2 μg/mL) 16 ore dopo la trasduzione.
- Continuare la selezione sostituendo il terreno con un terreno fresco da 2 mL contenente blasticidina (10 μg/mL) e puromicina (2 μg/mL) dopo 2 giorni. Gestire le cellule trasdotte in condizioni S1 dopo 7 giorni dalla trasduzione.
NOTA: La selezione degli antibiotici deve essere eseguita fino alla morte di tutte le cellule di controllo non trasdotte. Possono essere utilizzate strategie di selezione alternative a seconda della cassetta di resistenza nel vettore di trasferimento. Se necessario, ottimizzare le concentrazioni di antibiotici utilizzando una curva di uccisione. Per ottenere popolazioni cellulari che esprimono fusioni di dCas9 a livelli sufficienti, può essere necessaria una selezione elaborata di cloni di singole cellule attraverso citometria a flusso o diluizione e piastratura seriale, seguita da quantificazione proteica.
5. Quantificazione delle proteine e analisi Western blot
- Contare le cellule 501-mel trasfettate stabili mescolando 10 μL di cellule con 10 μL di soluzione di Trypan Blue in un emocitometro. Centrifugare 1 x 106 celle per 5 minuti a 500 x g e 4 °C. Scartare il surnatante e risospendere il pellet cellulare in 1 mL di PBS ghiacciato.
- Ripetere la centrifugazione e risospendere il pellet cellulare in 50 μL di tampone RIPA contenente inibitori della proteasi. Incubare su ghiaccio per 15 minuti e rimuovere i detriti cellulari mediante centrifugazione a 13.000 x g per 10 minuti a 4 °C. Conservare il surnatante a -20 °C o utilizzarlo immediatamente per la quantificazione delle proteine come descritto da altri31.
- Mescolare 10 μg di proteine con 4 tamponi LDS, 50 mM DTT e riempire fino a 20 μl con acqua distillata. Separare le proteine su un gel di separazione al 10% utilizzando SDS-PAGE a base di Tris/glicina a 135 V per 1 ora come descritto in precedenza32. Trasferire le proteine su una membrana in PVDF a 90 V per 90 minuti con un trasferimento in vasca umida.
- Incubare la membrana in tampone bloccante (5% di latte scremato in TBS-T) per 1 ora a 180 giri/min a temperatura ambiente.
- Aggiungere l'anticorpo monoclonale di topo anti-GAPDH 1:10.000 nel tampone di blocco come controllo interno. Aggiungere l'anticorpo primario anti-Cas9 (S. pyogenes) o anti-Cas9 (S. aureus) in diluizione 1:1.000 nel tampone bloccante e incubare per una notte a 4 °C agitando delicatamente.
- Lavare la membrana 3 volte con tampone TBST per 10 minuti ciascuna. Incubare con anticorpo HRP secondario anti-topo (1:10.000) per 1 ora a temperatura ambiente.
- Lavare 3 volte con tampone TBST per 10 minuti ciascuno. Rileva i segnali utilizzando il substrato ECL e un sistema di imaging.
6. Saggio di vitalità della repressione di LncRNA doppio
ATTENZIONE: Eseguire sempre lavori con virus in un armadio di sicurezza di Classe 2.
- Seminare 0,1 x 105 cellule contenenti SadCas9-KRAB e SpdCas9-KRAB trasfettati stabili 7 giorni dopo la trasduzione in una piastra a 96 pozzetti in 100 μL di terreno DMEM preriscaldato 1 giorno prima della trasduzione.
- Il giorno della trasduzione, sostituire il terreno di coltura con 100 μL di terreno DMEM preriscaldato integrato con polibrene a una concentrazione finale di 6 μg/mL. D'ora in poi, è richiesto il lavoro nel laboratorio di Classe 2. Aggiungere il volume appropriato di lentivirus alle cellule.
NOTA: Il volume dei lentivirus contenenti il vettore a doppio gRNA deve essere esaminato utilizzando la GFP per generare una curva lineare titolando i lentivirus. - Sostituire il terreno con un terreno fresco contenente 500 μg/mL di zeocina, 5 μg/mL di blasticidina e 1 μg/mL di puromicina 16 ore dopo la trasduzione.
- Eseguire il rilevamento della luminescenza 5 giorni dopo la trasduzione utilizzando un test di vitalità cellulare secondo le istruzioni del produttore.
Risultati
Le cassette di espressione di SadCas9-KRAB-MeCP2 e SpdCas9-KRAB-MeCP2 sono state integrate simultaneamente in cellule 501-mel utilizzando la trasduzione con quantità uguali di lentivirus. Poiché questo protocollo CRISPRi si basa sull'espressione delle varianti dCas9-KRAB-MeCP2, l'analisi Western blot è ideale per confermare la loro presenza a livello proteico. Dopo l'arricchimento di cellule trasfettate positivamente, sono stati raccolti campioni per l'analisi Western blot per confermare la presenza degli ortologhi dCas9-KRAB-MeCP2. Dopo il successo della trasduzione, ci si aspetta una singola banda corrispondente alla proteina di fusione SpdCas9-KRAB-MeCP2 (202 kDa) e alla proteina di fusione SadCas9-KRAB-MeCP2 (170 kDa) ed è stata confermata, come mostrato in Figura 2A, utilizzando anticorpi specifici per i rispettivi ortologhi Cas9. Tuttavia, se il Western blot non è fattibile, la qPCR può essere utilizzata per verificare le proteine di fusione a livello di mRNA.
Una volta generate le cellule 501-mel che esprimono stabilmente i repressori funzionali, la seconda trasduzione lentivirale è stata eseguita utilizzando un vettore a doppio gRNA. In precedenza era stato dimostrato che RP11 e XLOC erano sovraregolati in colture a breve termine di melanoma metastatico e il targeting individuale di CRISPRi ha dimostrato il loro impatto sulla proliferazione e sulla sopravvivenza cellulare28. Pertanto, abbiamo selezionato questa coppia di lncRNA per la valutazione come una combinazione di RNA non codificante letale sintetico proof-of-concept, anticipando un fenotipo di crescita cumulativo e facilmente osservabile nelle cellule modificate.
Per studiare gli effetti del doppio CRISPRi nelle cellule tumorali utilizzando saggi di vitalità, entrambi gli lncRNA mirati devono essere efficacemente repressi. La qPCR è il metodo più adatto per confermare questa repressione misurando i loro livelli di trascrizione. Abbiamo valutato la funzionalità del doppio protocollo CRISPRi sviluppato determinando i livelli di RNA ed esaminando la vitalità cellulare quando si prende di mira la coppia RP11 e XLOC potenzialmente letale. Pertanto, sono stati introdotti diversi gRNA che hanno come bersaglio RP11 e XLOC per ridurre i livelli di RNA bersaglio. La repressione dell'RNA è stata studiata utilizzando gRNA target-specifici per SadCas9-KRAB-MeCP2 o SpdCas9-KRAB-MeCP2, testati singolarmente e in combinazione insieme a un gRNA di controllo mirato a Rosa26 (vedi Figura 2B). Sebbene i gRNA che hanno come bersaglio XLOC o RP11 abbiano soppresso efficacemente i rispettivi bersagli, il knockdown simultaneo di XLOC e RP11 è stato ottenuto solo con la coppia XLOC-sa/RP11-sp.
Poiché sia i livelli di RNA XLOC che RP11 sono stati ridotti con successo nella linea cellulare 501-mel con la coppia di gRNA XLOC-sa/RP11-sp selezionata, è stato eseguito un doppio test di vitalità della repressione dell'lncRNA (vedi Figura 2C). Dopo 5 giorni, la vitalità cellulare era dell'81% e dell'89% del controllo di Rosa26 quando i gRNA bersaglio sono stati utilizzati singolarmente. La combinazione di RP11-sp e XLOC-sa ha portato a un'ulteriore riduzione significativa rispetto al solo RP11-sp, con una vitalità cellulare al 59% del controllo Rosa26.
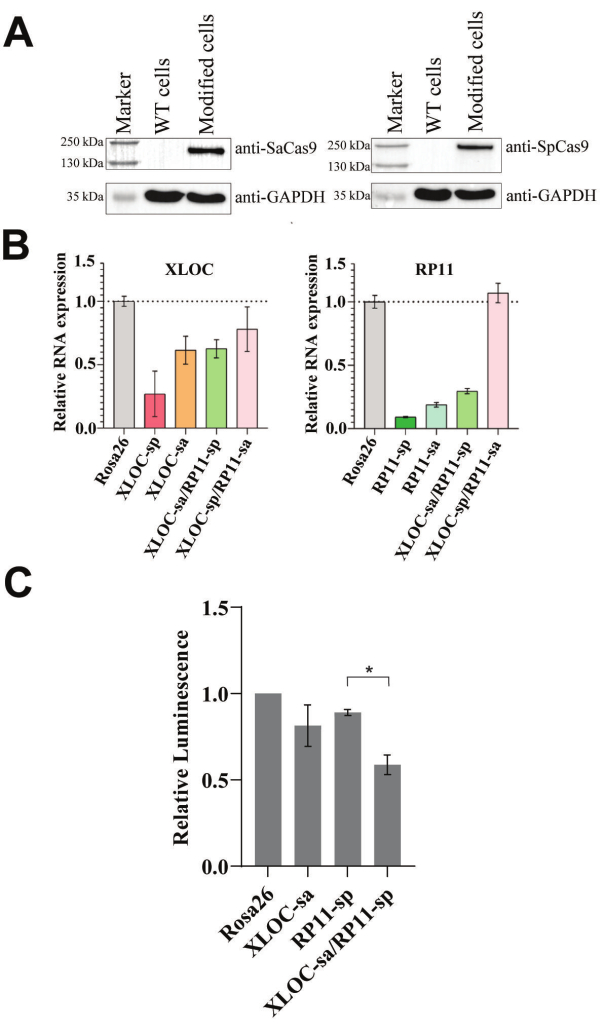
Figura 2: Funzionalità del doppio sistema di repressione dell'lncRNA. (A) L'analisi Western blot è stata eseguita per rilevare le varianti dCas9-KRAB-MeCP2 in cellule 501-mel modificate utilizzando anticorpi anti-Cas9. Come controllo sono state utilizzate cellule 501-mel wild-type (WT) senza trasduzione (vedi Figura supplementare 3, Figura supplementare 4, Figura supplementare 5, Figura supplementare 6 per il raw blot). (B) analisi basata sulla qPCR dei livelli di RP11 e XLOC RNA relativi alle cellule di controllo Rosa26 dopo la trasduzione di doppi gRNA in cellule 501-mel modificate. GAPDH è stato usato come gene delle pulizie. I dati sono espressi come media ± SD. (C) Il saggio di vitalità della repressione a doppio lncRNA è stato eseguito dopo l'integrazione di doppi gRNA in cellule 501-mel modificate in tre trasduzioni indipendenti. I valori di luminescenza sono stati normalizzati al controllo Rosa26. I risultati di vitalità sono espressi come media (n = 3) ± SEM. È stato eseguito un t-test non appaiato. L'asterisco (*) indica una differenza significativa a p < 0,05. Clicca qui per visualizzare una versione più grande di questa figura.
Figura supplementare 1: Mappa plasmidica. La mappa è stata creata con Benchling (https://benchling.com/). I plasmidi utilizzati nel protocollo sono stati modificati da Addgene #96921 e #110824. Clicca qui per scaricare questo file.
Figura supplementare 2: Figura 2 supplementare: Analisi del western blot di
Espressione dCas9-KRAB. (A) Espressione di SadCas9-KRAB in cellule 501-mel 6 giorni dopo la trasfezione. (B) La trasduzione sequenziale di SadCas9-KRAB e SpdCas9-KRAB in cellule 501-mel ha portato a una riduzione dell'espressione di SadCas9-KRAB 3 settimane dopo la trasfezione. (C) La trasduzione simultanea di SadCas9-KRAB e SpdCas9-KRAB in cellule 501-mel ha determinato una riduzione dell'espressione di SadCas9-KRAB 3 settimane dopo la trasfezione. (D) L'espressione di SpdCas9-KRAB può essere osservata dopo la trasduzione simultanea di SadCas9-KRAB e SpdCas9-KRAB. LV. Il volume di lentivirus utilizzati in μL. Fare clic qui per scaricare questo file.
Figura 3 supplementare: Dati grezzi SaCas9-KRAB del Western blot. Clicca qui per scaricare questo file.
Figura 4 supplementare: Dati grezzi Western blot SpCas9-KRAB. Clicca qui per scaricare questo file.
Figura supplementare 5: Dati grezzi GAPDH_SpCas9-KRAB. Fare clic qui per scaricare questo file.
Figura 6 supplementare: Dati grezzi GAPDH_SaCas9-KRAB. Fare clic qui per scaricare questo file.
Tabella supplementare 1: Sequenze di RNA guida utilizzate. Clicca qui per scaricare questo file.
Tabella supplementare 2: Sequenze di DNA utilizzate. Clicca qui per scaricare questo file.
File supplementare 1: File FASTA di dual-grna-zeo-gfp. Clicca qui per scaricare questo file.
File supplementare 2: file FASTA di dSpCas9-krab-mecp2. Clicca qui per scaricare questo file.
File supplementare 3: file FASTA di dSaCas9-krab-mecp2. Clicca qui per scaricare questo file.
Discussione
In questo studio, abbiamo implementato una doppia strategia di targeting di lncRNA in cellule di melanoma utilizzando CRISPRi basata su dCas9-KRAB-MeCP2 di due specie ortogonali. Utilizzando questo sistema, abbiamo represso con successo una coppia letale sintetica di lncRNA, RP11 e XLOC, portando ad un aumento della morte cellulare, a differenza di quando venivano repressi individualmente. Tuttavia, i sistemi CRISPRi comportano un rischio di repressione genica fuori bersaglio, influenzando potenzialmente l'interpretazione dei risultati. Sebbene la convalida sperimentale non sia stata condotta, abbiamo minimizzato i potenziali effetti off-target durante la progettazione del gRNA in silico.
La variabilità nell'efficienza di trasduzione può influire sulla riproducibilità. Pertanto, abbiamo ottimizzato la trasduzione applicando quantità uguali di lentivirus, determinate attraverso una calibrazione della curva standard utilizzando concentrazioni variabili di lentivirus. Tuttavia, l'aumento delle dimensioni del carico del vettore di nuova concezione può imporre limitazioni all'efficienza dell'imballaggio lentivirale, riducendo potenzialmente l'efficienza di trasduzione, in particolare nelle linee cellulari difficili da trasdurre. Di conseguenza, il raggiungimento di una trasduzione sufficiente e di una robusta espressione di dCas9 può richiedere in alcuni casi titoli virali elevati. Inoltre, un knockdown insufficiente può essere aggirato monitorando frequentemente i livelli della proteina dCas9-KRAB-MeCP2 e l'espressione dell'RNA di lncRNA mirati. In alternativa, sono disponibili versioni dCas9 con un solo sistema repressore per ovviare a questa limitazione, con il costo di una minore efficienza25.
Solo la combinazione XLOC-sa-RP11-sp ha dimostrato un'attività di successo, mentre la configurazione inversa XLOC-sp-RP11-sa è risultata inefficace. Una spiegazione plausibile per questa differenza nell'efficacia del knockdown è l'interazione tra la specificità della PAM e l'accessibilità della cromatina33. Dato che SaCas9 e SpCas9 riconoscono sequenze PAM distinte, il sito bersaglio per SpCas9 nell'orientamento XLOC-sp-RP11-sa potrebbe non avere un PAM ottimale, riducendo così la sua affinità di legame e la successiva attività. Inoltre, la disposizione spaziale delle due varianti della proteina di fusione dCas9 potrebbe influenzare l'architettura e l'accessibilità della cromatina34,35, limitando potenzialmente il reclutamento del macchinario trascrizionale o dei fattori regolatori associati nella configurazione XLOC-sp-RP11-sa. Questi risultati suggeriscono che il posizionamento relativo di SaCas9 e SpCas9 all'interno di un dato contesto genomico può avere un impatto critico sulla loro efficacia funzionale, un aspetto che giustifica ulteriori indagini per strategie di modulazione ottimizzate basate su CRISPR.
Il protocollo sviluppato è adattabile per il targeting o lo screening di altre coppie di RNA non codificanti letali sintetiche modificando il doppio vettore di gRNA. Una volta che una linea cellulare di interesse esprime stabilmente entrambe le varianti di dCas9, diventa possibile una varietà di approcci. Mentre RNAi, ASO e shRNA possono essere impiegati per il silenziamento genico, CRISPRi offre una metodologia di screening più semplice ed è concepibile anche la sua combinazione con piccoli composti chimici o inibitori. Sostituendo il vettore a doppio gRNA con una libreria a doppio gRNA, questo protocollo consente lo screening sistematico di coppie letali sintetiche o altre dipendenze genetiche nelle cellule tumorali, migliorando così la precisione del targeting delle cellule tumorali.
Sebbene abbiamo ottenuto l'integrazione delle varianti dCas9-KRAB-MeCP2 attraverso la trasduzione lentivirale, è possibile utilizzare anche metodi alternativi di trasfezione stabile per incorporare gli enzimi desiderati nelle cellule 501-mel. Ciò è particolarmente rilevante, poiché, nella nostra esperienza, il silenziamento di costrutti di grandi dimensioni come dCas9-KRAB-MeCP2 può verificarsi nel tempo36,37. In alternativa, dCas9-KRAB ha dimostrato di essere un'alternativa più piccola23. Nella nostra esperienza, la trasduzione con le proteine di fusione dCas9-KRAB più piccole derivate da ortologhi Cas9 ha portato a una ridotta espressione proteica durante la coltivazione prolungata (vedi Figura 2 supplementare). Pertanto, l'espressione costante delle proteine deve essere monitorata regolarmente in momenti ricorrenti. Inoltre, la trasduzione di due grandi proteine di fusione insieme a marcatori di selezione e cassette di espressione a doppio gRNA può indurre stress cellulare o può raggiungere capacità di impacchettamento lentivirale.
Raggiungere un'efficienza di knockdown del 100% nel doppio CRISPRi può essere difficile a causa di una progettazione non ottimale del gRNA o di un'espressione insufficiente di dCas9. Per risolvere questo problema, è necessario testare più gRNA per bersaglio utilizzando strumenti di progettazione avanzati per identificare le sequenze più efficaci. Inoltre, l'ottimizzazione dell'espressione di dCas9-KRAB-MeCP2 attraverso una migliore progettazione vettoriale e la caratterizzazione di linee cellulari clonali può migliorare l'efficienza del knockdown.
Strategie alternative per l'integrazione del sistema CRISPRi includono l'uso di vettori trasposasi o l'integrazione diretta da CRISPR per ottenere un inserimento stabile in siti genomici sicuri, noti per mantenere un'espressione stabile nel tempo38. Tuttavia, molti tipi di cellule, comprese le cellule di melanoma, sono difficili da trasfettare39, rendendo la trasduzione lentivirale il metodo preferito. L'elettroporazione di enzimi a base di trasposisi o Cas può offrire un mezzo alternativo per integrare i costrutti dCas9-KRAB-MeCP2 nelle cellule di melanoma40. L'implementazione di un sistema di knockout inducibile sarebbe vantaggiosa per attivare l'espressione di dCas9-KRAB-MeCP2 secondo necessità piuttosto che l'espressione costitutiva per minimizzare lo stress cellulare.
Infine, l'approccio sviluppato non è limitato alle cellule di melanoma, ma può essere esteso a qualsiasi tipo di cellula in cui si sta studiando la letalità sintetica. Questo approccio è esplicitamente destinato ad essere esteso alla modalità di screening della libreria di gRNA. Tuttavia, a causa dell'aumentata complessità combinatoria, ad esempio con più di tre gRNA per coppia di geni, compresi i controlli e una copertura di almeno 300 volte, solo una frazione sub-trascrittomica di lncRNA espressi può essere bersagliata praticamente. Pertanto, gli utenti devono preselezionare attentamente i candidati considerando fattori come i livelli di espressione degli lncRNA e la loro rilevanza nota in specifici contesti cellulari prima di procedere con lo screening della libreria. Tuttavia, la versatilità combinatoria di questo approccio lo rende uno strumento prezioso per studiare le interazioni letali sintetiche e altre dipendenze genetiche degli lncRNA in vari tipi di cancro e potenzialmente in altre malattie. Ciò amplia il toolkit attualmente disponibile per lo studio di queste interazioni di rete nelle cellule.
Divulgazioni
Jochen Imig è attualmente finanziato da Pfizer Inc. CGCIII è sponsorizzato da Pfizer Inc., Merck KGaA e AstraZeneca PLC. Gli sponsor non hanno avuto alcun ruolo nella progettazione, esecuzione, interpretazione o scrittura dello studio. Tutti gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Ringraziamo Stefan Raunser (Max Planck Institute of Molecular Physiology, Dortmund) per l'accesso allo spazio di laboratorio di Biosicurezza di Livello 2. Un ringraziamento speciale a Eric Wang per i suoi contributi intellettuali e a tutti i membri passati e presenti del laboratorio Imig per le loro preziose discussioni. Jochen Imig è attualmente finanziato da Pfizer Inc. presso CGC III.
Materiali
| Name | Company | Catalog Number | Comments |
| Amicon Ultra Centrifugal Filter, 100 kDa MWCO | Millipore | UFC910024 | |
| anti-Cas9 (S. aureus) (6H4) mouse monoclonal antibody | Cell Signaling Technology | 48989 | |
| anti-Cas9 (S. pyogenes) (7A9-3A3) monoclonal antibody | Cell Signaling Technology | 14697 | |
| anti-GAPDH mouse monoclonal antibody | Sigma Aldrich | G8795 | |
| Anti-Mouse IgG (whole molecule)–Peroxidase antibody produced in rabbit | Sigma Aldrich | A9044 | |
| Bio-Rad ChemiDoc MP Imaging System | Bio-Rad | ||
| Blasticidine S hydrochloride | Sigma Aldrich | 15205-25MG | |
| CellTiter-Glo Luminescent Cell Viability Assay | Promega | G7570 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge | Eppendorf | 5415R | |
| CFX Connect Real-Time PCR Detection System | Bio-Rad | ||
| Clarity Western ECL Substrate, 500 mL | Bio-Rad | 1705061 | |
| CO2 Incubator Model CB 170 | Binder | ||
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Esp3I (BsmB1) restriction enzyme | Thermo Scientific | ER0451 | |
| Falcon 10 mL Serological Pipet | Corning | 356551 | |
| Falcon 25 mL Serological Pipet | Corning | 357525 | |
| Falcon 5 mL Serological Pipet | Corning | 356543 | |
| Fetal Bovine Serum | Sigma Aldrich | F9665-500ML | |
| Gibco Dulbecco’s modified Eagle’s medium (DMEM), high glucose, pyruvate | Gibco | 41966029 | |
| Human melanoma cell line 501-mel | was kindly provided by the Aifantis Lab (New York University) | RRID: CVCL_4633 | |
| Immobilon -P PVDF Membrane | Millipore | IPVH00010 | |
| Lenti-X 293T HEK cell line | Takara Bio | 632180 | |
| Mini Trans-Blot Cell system | Bio-Rad | ||
| Mini-PROTEAN Tetra Handcast System | Bio-Rad | ||
| NEBuilder HiFi DNA Assembly Reaction | New England Biolabs | E2621 | |
| Non-fat milk powder | Biomol | 54650 | |
| NuPAGE LDS Sample Buffer (4x) | Invitrogen | NP0008 | |
| One Shot Stbl Chemically Competent E. coli | Invitrogen | C737303 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985062 | |
| PCR Tubes 0.5 ml (Flat Cap) | VWR International | 732-3207 | |
| Phosphate-Buffered Saline (PBS) | Corning | 45000-446 | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | Thermo Scientific | F531L | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Polybrene | Sigma Aldrich | TR-1003-G | |
| Polyethylenimine, branched | Sigma Aldrich | 408727 | |
| Puromycin dihydrochloride | Santa Cruz Biotechnology | sc-108071A | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| SafeSeal Microcentrifuge Tube 1.5 mL | Sarstedt | 72,706 | |
| Sodium chloride, 5 M Aqua Solution, RNase Free | Alfa Aesar | J60434.AE | |
| Sodium Dodecyl Sulfate (SDS) | Sigma Aldrich | L3771 | |
| Syringe Filter PES 33mm 0.2 μM | Fisher Scientific | 15206869 | |
| TC Dish 100, Standard | Sarstedt | 8,33,902 | |
| TC Plate 6 Well, Standard, F | Sarstedt | 83,39,20,005 | |
| Tris base | Roche | 10708976001 | |
| TWEEN 20 | Sigma Aldrich | P9416-50ML |
Riferimenti
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Szcześniak, M. W., Wanowska, E., Mukherjee, N., Ohler, U., Makałowska, I. Towards a deeper annotation of human lncRNAs. Biochim Biophys Acta Gene Regul Mech. 1863 (4), 194385(2020).
- Fang, S., et al. NONCODEV5: a comprehensive annotation database for long non-coding RNAs. Nucl Acids Res. 46 (D1), D308-D314 (2018).
- Lin, J., et al. Human-specific lncRNAs contributed critically to human evolution by distinctly regulating gene expression. eLife. 12, RP89001.2(2023).
- Sarropoulos, I., Marin, R., Cardoso-Moreira, M., Kaessmann, H. Developmental dynamics of lncRNAs across mammalian organs and species. Nature. 571 (7766), 510-514 (2019).
- Melé, M., et al. Human genomics. The human transcriptome across tissues and individuals. Science. 348 (6235), 660-665 (2015).
- Cabili, M. N., et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev. 25 (18), 1915-1927 (2011).
- Liu, S. J., Dang, H. X., Lim, D. A., Feng, F. Y., Maher, C. A. Long noncoding RNAs in cancer metastasis. Nat Rev Cancer. 21 (7), 446-460 (2021).
- Ahmad, M., Weiswald, L. B., Poulain, L., Denoyelle, C., Meryet-Figuiere, M. Involvement of lncRNAs in cancer cells migration, invasion and metastasis: cytoskeleton and ECM crosstalk. J Exp Clin Cancer Res. 42 (1), 173(2023).
- Jain, A. K., et al. LncPRESS1 Is a p53-Regulated LncRNA that Safeguards Pluripotency by Disrupting SIRT6-Mediated De-acetylation of Histone H3K56. Mol Cell. 64 (5), 967-981 (2016).
- Bergmann, J. H., et al. Regulation of the ESC transcriptome by nuclear long noncoding RNAs. Genome Res. 25 (9), 1336-1346 (2015).
- Kretz, M., et al. Control of somatic tissue differentiation by the long non-coding RNA TINCR. Nature. 493 (7431), 231-235 (2013).
- Yoon, J. H., et al. LincRNA-p21 suppresses target mRNA translation. Mol Cell. 47 (4), 648-655 (2012).
- Huarte, M. The emerging role of lncRNAs in cancer. Nat Med. 21 (11), 1253-1261 (2015).
- Badowski, C., He, B., Garmire, L. X. Blood-derived lncRNAs as biomarkers for cancer diagnosis: the Good, the Bad and the Beauty. NPJ Precis Oncol. 6 (1), 40(2022).
- Zong, X., et al. Knockdown of nuclear-retained long noncoding RNAs using modified DNA antisense oligonucleotides. Meth Mol Biol. 1262, 321-331 (2015).
- Gagnon, K. T., Li, L., Chu, Y., Janowski, B. A., Corey, D. R. RNAi factors are present and active in human cell nuclei. Cell Rep. 6 (1), 211-221 (2014).
- Zeng, Y., Cullen, B. R. RNA interference in human cells is restricted to the cytoplasm. RNA. 8 (7), 855-860 (2002).
- Yang, J., et al. CRISPR/Cas9-mediated noncoding RNA editing in human cancers. RNA Biol. 15 (1), 35-43 (2018).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355 (6320), aah7111(2017).
- Tsung, K., et al. CRISPRi screen of long non-coding RNAs identifies LINC03045 regulating glioblastoma invasion. PLoS Genet. 20 (6), e1011314(2024).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Boettcher, M., McManus, M. T. Choosing the right tool for the job: RNAi, TALEN, or CRISPR. Mol Cell. 58 (4), 575-585 (2015).
- Yeo, N. C., et al. An enhanced CRISPR repressor for targeted mammalian gene regulation. Nat Meth. 15 (8), 611-616 (2018).
- Parrish, P. C. R., et al. Discovery of synthetic lethal and tumor suppressor paralog pairs in the human genome. Cell Rep. 36 (9), 109597(2021).
- Najm, F. J., et al. Orthologous CRISPR-Cas9 enzymes for combinatorial genetic screens. Nat Biotechnol. 36 (2), 179-189 (2018).
- Petroulia, S., et al. CRISPR-inhibition screen for lncRNAs linked to melanoma growth and metastasis. bioRxiv. , (2024).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- New England Biolabs. NEBuilder HiFi DNA Assembly Reaction (E2621) v2. , (2020).
- Cortés-Ríos, J., et al. Protein quantification by bicinchoninic acid (BCA) assay follows complex kinetics and can be performed at short incubation times. Anal Biochem. 608, 113904(2020).
- Junior, N. Polyacrylamide Gel Electrophoresis (SDS-PAGE) v1. , (2019).
- Chen, X., Liu, J., Janssen, J. M., Gonçalves, M. A. F. V. The chromatin structure differentially impacts high-specificity CRISPR-Cas9 Nuclease strategies. Mol Ther Nucl Acids. 8, 558-563 (2017).
- Schep, R., et al. Impact of chromatin context on Cas9-induced DNA double-strand break repair pathway balance. Mol Cell. 81 (10), 2216-2230.e10 (2021).
- Daer, R. M., Cutts, J. P., Brafman, D. A., Haynes, K. A. The impact of chromatin dynamics on Cas9-Mediated genome editing in human cells. ACS Syn Biol. 6 (3), 428-438 (2017).
- Chavez, M., Rane, D. A., Chen, X., Qi, L. S. Stable expression of large transgenes via the knock-in of an integrase-deficient lentivirus. Nat Biomed Eng. 7 (5), 661-671 (2023).
- Ellis, J. Silencing and variegation of gammaretrovirus and lentivirus vectors. Human Gene Ther. 16 (11), 1241-1246 (2005).
- Shrestha, D., et al. Genomics and epigenetics guided identification of tissue-specific genomic safe harbors. Genome Biol. 23 (1), 199(2022).
- Chu, Z., et al. Enhanced gene transfection and induction of apoptosis in melanoma cells by branched poly(β-amino ester)s with uniformly distributed branching units. J Control Release. 367, 197-208 (2024).
- Han, S. Y., et al. Nucleofection is a highly effective gene transfer techfnique for human melanoma cell lines. Exp Dermatol. 17 (5), 405-411 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon