Method Article
암세포에서 비암호화 RNA 간의 합성 치사 상호작용을 표적으로 하기 위한 이중 CRISPR-간섭 전략
요약
이 연구는 흑색종 세포에서 긴 non-coding RNA를 표적으로 하는 이중 CRISPRi 시스템을 제시합니다. 이를 통해 조합 유전자 녹다운(combinatorial gene knockdown) 및 합성 치사 스크리닝(synthetic lethal screening)을 가능하게 하여 잠재적인 치료 전략을 위한 암 특이적 lncRNA 상호 작용을 식별할 수 있습니다.
초록
긴 비암호화 RNA(lncRNA)는 인간 게놈에서 100,000개 이상이 예측되는 방대하고 기능적으로 다양한 종류의 RNA 분자를 나타냅니다. lncRNA는 단백질 코딩 유전자에 비해 종 간에 덜 보존되어 있지만 유전자 조절, 염색질 상호 작용 및 암 진행에 중요한 역할을 합니다. 암에 대한 그들의 관여는 그들을 유망한 치료 표적으로 만듭니다. KRAB-MeCP2와 같은 전사 억제제와 융합된 촉매 비활성 Cas9를 활용하는 CRISPR 간섭(CRISPRi)은 핵 lncRNA를 표적으로 하고 그 기능을 평가하기 위한 정확한 방법을 제공합니다. 본 연구는 인간 흑색종 세포에서 lncRNA의 combinatorial targeting에 최적화된 dCas9-KRAB-MeCP2를 기반으로 한 황 색포도상구균 및 연쇄상구균 화농성 구균의 직교 CRISPRi 기술을 사용하는 이중 CRISPRi 시스템을 소개합니다. 이 프로토콜은 lncRNA 쌍의 조합 유전자 녹다운(combinatorial gene knockdown) 또는 합성 치사 스크리닝을 용이하게 하여 암 연구를 위한 새로운 도구를 제공합니다. lncRNA 간의 합성 치사율을 탐구함으로써 이 접근 방식은 암세포 생존에 중요한 lncRNA 상호 작용을 식별하는 데 도움이 될 수 있으며 새로운 치료 전략을 제공할 수 있습니다. 이중 시스템의 기능이 입증되어 중요한 암 특이적 lncRNA 상호 작용을 식별할 수 있는 잠재력을 강조합니다.
서문
인간 게놈의 3% 미만이 단백질을 암호화하지만 게놈의 약 80%가 전사됩니다 1,2. 비암호화 전사 단위 중 수만 개는 200개의 뉴클레오티드를 초과하는 긴 비암호화 RNA(lncRNA)로 분류되며, 인간 lncRNA의 총 수는 100,000개를 초과하는 것으로 추정됩니다 3,4. 코딩 유전자와 대조적으로, lncRNA는 종 간에 덜 보존됩니다. 인간이 침팬지와 같은 영장류와 게놈의 99%를 공유한다는 점을 감안할 때, lncRNA는 표현형 진화에 훨씬 더 큰 영향을 미치기 위해 가설을 세웠습니다 5,6. 이러한 발견은 lncRNA의 중요한 세포 기능을 나타냅니다. lncRNAs의 규칙 및 RNA 결합 단백질 및 다른 RNAs와의 그들의 상호 작용은 불완전하게 이해된 채로 남아 있고, 많은 lncRNAs는 아직 완전히 주석이 달리지 않더라도, lncRNAs가 암 7,8,9,10와 같은 건강 그리고 질병에 있는 세포와 조직 특정한 표정 본을 전시한ᄂ다는 것은 분명합니다. 이들은 유전자 전사 조절(gene transcription regulation), 염색질 상호작용(chromatin interaction)11, RNA 처리(RNA processing)12, RNA 안정화(RNA stabilization)13, 번역 조절(regulation of translation)14 등 다양한 기능에 관여하고 있습니다.
암에서 매우 다양한 세포 유형 특이적 lncRNA는 유전자 발현을 조절하여 종양 발달 및 전이에 영향을 미치며, 가치 있는 치료 표적으로서의 잠재력을 강조합니다15. 종양 샘플에서 바이오마커로 lncRNA를 검출하는 것 외에도16, 종양 특이적 lncRNA를 표적으로 하여 다운스트림 기능을 방해하는 것은 임상 응용 분야와 기초 연구 모두에서 상당한 잠재력을 가지고 있습니다. lncRNA의 역할을 규명하기 위한 RNA 기반 접근 방식에는 안티센스 올리고뉴클레오티드(ASO), 짧은 헤어핀 RNA 및 작은 간섭 RNA(siRNA)가 포함됩니다17,18. siRNA는 일반적으로 유전자 침묵 스크리닝에 사용되지만, siRNA 기반 knockdown은 세포질19로 제한됩니다. 그러나 lncRNA는 핵 내에서 자주 작동합니다.
대안적으로, CRISPRi(Clustered Regularly Interspaced Short Palindromic Repeats interference)를 사용하여 인간 암에서 lncRNA를 억제할 수 있습니다20. 또한, 게놈 와이드 CRISPRi 스크리닝은 쉽게 프로그래밍할 수 있으며 광범위한 코딩 및 비코딩 유전자를 표적으로 하여 기능적 영향을 조사할 수 있습니다21,22. CRISPRi에서 촉매 결핍 Cas9 (dCas9)는 KRAB(Krüppel-associated box) 도메인과 같은 전사 억제 도메인(transcriptional repressor domain)에 융합됩니다23. dCas9-KRAB에 의한 유전자 억제는 가이드 RNA(gRNA)에 의해 관심 영역으로 유도됩니다. CRISPRi는 DNA 수준에서 유전자를 제어하여 전사 후 수준24에서 활성화되는 RNA 간섭과 대조적으로 더 높은 효율성과 원하는 기능 상실 표현형을 유도합니다. 표적 침묵에 대한 KRAB의 제한된 효능에 대응하여, 보다 효과적인 억제 전략으로 KRAB 및 MeCP2와 dCas9의 융합이 도입되었습니다25.
단일 유전자 침묵이 암 생존력에 영향을 미칠 수 있지만, 합성 이중 또는 다중 치사적 상호 작용은 세포 사멸로부터 암세포를 구할 수 있습니다26. 합성 치사율은 두 개 이상의 유전자를 포함하며, 각 유전자는 다른 유전자의 기능을 보상할 수 있습니다. 단일 유전자 녹다운(knockdown) 스크리닝의 문제점을 극복하기 위해, 치사량 단백질 코딩 유전자 쌍을 표적으로 하는 이중 CRISPR 전략은 유망한 접근법을 제공합니다27.
여기에서는 인간 흑색종 세포에서 KRAB-MeCP2에 융합된 황색포도상구균 및 연쇄상구균 화농성 dCas9를 사용하여 lncRNA 또는 기타 비암호화 RNA 쌍의 직교 CRISPRi 기반 표적화의 조합 사용을 위한 프로토콜을 제시합니다( 그림 1 참조). 이 프로토콜은 고전적인 CRISPR 조합 녹다운(knockdown) 또는 암에서 합성 치사쌍의 CRISPRi 기반 스크리닝으로 활용될 수 있습니다. CRISPRi 접근법에서 두 가지 다른 dCas9 종인 SpCas9 및 SaCas9를 사용하면 최소한의 교차 반응으로 고유한 게놈 유전자 자리를 독립적으로 표적화할 수 있으므로 높은 표적 선택성을 보장하면서 특이성과 유연성을 향상시킬 수 있습니다. 다양한 프로토 스페이서 인접 모티프 (PAM)가 사용됩니다 : SpCas9의 경우 NGG, SaCas9의 경우 NNGRRT가 사용됩니다. 이중 dCas9 시스템은 단일 유형의 dCas9을 사용할 때 정밀한 동시 조절을 가능하게 하고 sgRNA-RNP 복잡한 경쟁을 줄여 제한된 sgRNA 호환성과 같은 문제를 해결합니다. 이 혁신은 이중 sgRNA 라이브러리 스크리닝의 견고성과 다양성을 향상시킵니다. 결론적으로, 우리는 암 모델로서 흑색종 세포에서 완전한 기능을 갖춘 이중 CRISPRi 시스템을 제공합니다.
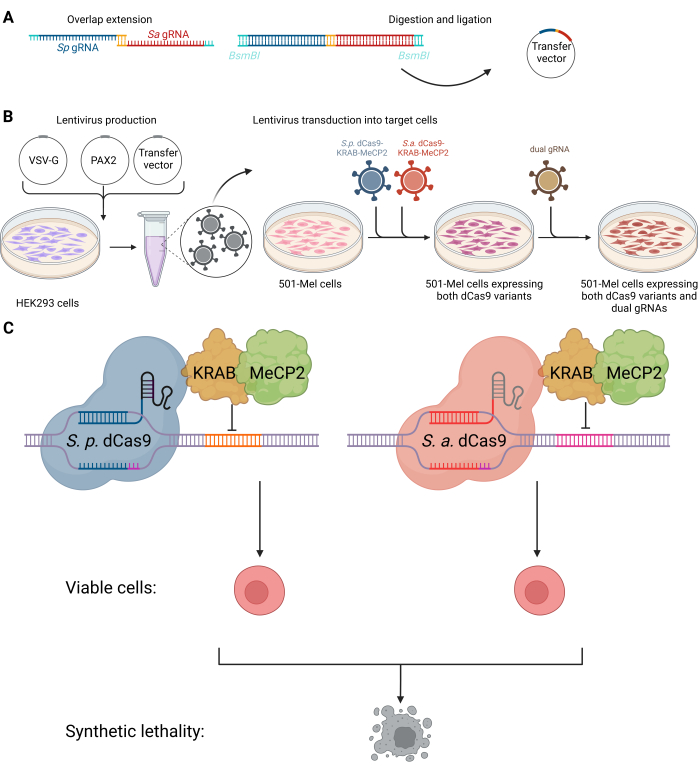
그림 1: 비암호화 RNA의 합성 치사 상호작용을 표적으로 하는 이중 CRISPRi 시스템의 계획. (A) 이중 gRNA 벡터의 클로닝 절차. (B) HEK293 세포에 envelope plasmid VSV-G, 패키징 plasmid PAX2 및 transfer vector를 transfection하여 501-mel 세포로의 후속 transduction을 위한 렌티바이러스를 생성했습니다. SpdCas9-KRAB-MeCP2(파란색) 및 SadCas9-KRAB-MeCP2(빨간색)에 대한 유전 정보를 포함하는 렌티바이러스를 501-mel 세포에 동시에 통합했습니다. 항생제 선별 후, 원하는 이중 gRNA(올리브 그린)를 통합하기 위해 두 번째 렌티바이러스 형질도입을 수행했습니다. (C) dCas9 변이체를 발현하는 501-mel 세포는 해당 gRNA와 상호 작용하여 표적 유전자 발현을 침묵시켜 암세포 사멸을 초래합니다. BioRender.com 로 만들어졌습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
인간 흑색종 세포주 501-mel (RRID: CVCL_4633)은 Aifantis Lab(New York University)에서 친절하게 제공했습니다. Lenti-X 293T HEK 세포주는 Takara Bio에서 구입했습니다. 이러한 세포주는 무균 조건 하에서 5%CO2 대기에서 37°C에서 10% 소 태아 혈청(FBS)이 보충된 Dulbecco의 변형된 DMEM(Modified Eagle's medium) 고포도당에서 배양되었습니다. 세포는 80%-90% 농도에 도달할 때까지 계대탐색을 진행하였다. 세포주는 마이코플라스마 오염에 대해 정기적으로 테스트되었습니다.
1. 온라인 gRNA 디자인 플랫폼을 사용한 gRNA 디자인
- 각 타겟 lncRNA에 대해 SpCas9에 특이적인 하나의 gRNA와 SaCas9에 특이적인 하나의 gRNA를 설계하여 어떤 변이가 우수한 knockdown 결과를 산출하는지 평가합니다.
- 각 S. pyogenes (gRNA-sp) Cas9 및 S. aureus (gRNA-sa) Cas9에 대해 가장 효율적인 gRNA를 사용하여 원하는 lncRNA(여기서는 RP11-120D5.1 (RP11) 및 XLOC_030781 (XLOC)를 표적으로 합니다.
참고: S. pyogenes gRNA는 Petroulia et al.28에서 추출할 수 있습니다. - 사이트별 권장 사항에 따라 높은 on-target 점수로 표현되는 가장 높은 on-target activity의 우선순위를 정하는 동시에 gRNA design tool에서 제안한 off-target match를 최소화합니다.
- 게놈 GRCh38 (hg, Homo sapiens)을 선택합니다. 특정 SpCas9(NGG) 및 SaCas9(NNGRRT)에 대한 PAM 사이트를 선택합니다.
- Hsu et al.29의 오프타겟 스코어링 방법을 사용합니다. 최적의 성능을 위해 전사 시작 부위 주변에 -150 bp에서 +50 bp의 범위 내에 gRNA를 배치합니다.
- on-target 및 off-target 점수가 가장 높은 gRNA를 선택합니다( 보충 표 1 참조).
- Non-targeting 또는 scrambled control gRNA를 Rosa26에 대해 각 타겟 gRNA와 결합하여 하나의 lncRNA를 타겟하는 이중 gRNA 벡터를 생성합니다. 하나의 플라스미드에 타겟 gRNA를 결합하면 두 타겟 부위를 모두 부위에 특이적으로 변형할 수 있습니다.
참고: control gRNA와 target gRNA를 포함하는 이중 gRNA 벡터는 하나의 target lncRNA를 knockdown할 수 있으며, site를 타겟하는 gRNA를 포함하는 이중 gRNA 벡터는 targeted lncRNAs를 동시에 knockdown할 수 있습니다.
2. gRNA 클로닝
- gRNA 염기서열을 포함하는 두 개의 DNA 단편을 사용하여 중첩 확장 PCR을 수행합니다. Najm et al.27에 기술된 바와 같이 증폭을 위해 U6 순방향 프라이머 및 H1 역방향 프라이머(보충 표 2 참조)를 사용합니다.
- HF 완충액이 있는 25μL의 2x High-fidelity PCR 마스터 믹스와 S. pyogenes 또는 S. aureus gRNA를 포함하는 각 DNA 단편 1μL로 50μL PCR 반응을 설정합니다. 다음과 같은 사이클링 조건에서 반응을 실행합니다: 98°C에서 30초 동안 초기 변성, 98°C에서 5초 동안 변성 15회 주기, 55°C에서 10초 동안 어닐링, 72°C에서 15초 동안 연신율.
- 처음 15개의 주기를 완료한 후에 U6 앞으로 뇌관의 2.5 μL 그리고 H1 반전 뇌관의 2.5 μL를 추가하고, 72 °C에 5 분 동안 마지막 연장과 더불어 동일한 조건 하에서 추가 20 주기에 반응을 복종시키십시오.
- 제조업체의 지침에 따라 PCR 정제 키트를 사용하여 PCR 산물을 정제합니다.
- 10 μL 반응에서 37 °C에서 30분 동안 1 μL의 BsmB1 제한 효소와 1 μL의 10x 완충액을 사용하여 1 μg의 변형된 플라스미드를 분해합니다.
- PCR 산물을 다른 사람에 의해 기술된 바와 같이 HiFi DNA 조립 반응 프로토콜을 사용하여 분해된 dual-gRNA-Zeo-GFP 플라스미드( 보충 그림 1 및 보충 파일 1 참조)로 복제합니다30.
- 제조업체의 지침에 따라 50μL의 박테리아 배양액과 4μL의 조립 제품을 혼합하여 화학적으로 적합한 E. coli 세포를 변형시킵니다.
3. 렌티바이러스 생산
주의: 활성 렌티바이러스는 모든 단계에서 적절한 안전 지침을 준수하면서 취급하십시오. 불가피한 에어로졸 형성으로 인한 위험을 방지하기 위해 항상 클래스 2 안전 캐비닛에서 바이러스에 대한 작업을 수행하십시오. 원심분리와 같은 안전 캐비닛 외부의 모든 작업은 위험 그룹 2 유기체와 함께 사용하도록 승인된 에어로졸 밀폐 용기 및 로터에서 수행하십시오. 작업 영역에서 항상 실험실 가운, 일회용 장갑, 보안경을 포함한 적절한 보호복을 착용하십시오. 작업 표면은 소독 용액으로 처리해야 하며 다른 폐기물도 소독 용액으로 처리하여 최소 240분 동안 자외선 아래에서 밀봉된 캐비닛에 남아 있어야 합니다. 사용된 혈청학적 피펫과 피펫 팁은 오토클레이브 병에 폐기하기 전에 소독 용액으로 헹궈야 합니다. 그 후, 폐기물은 S2 폐기물 용기에 처리해야 합니다. S2 폐기물은 오토클레이빙을 통해 비활성화됩니다. 형질도입된 세포주는 형질도입 후 2일 이내에 S1 조건에서 처리할 수 있으며, 바이러스가 없는 배지로 배양 배지를 최소 2회 이상 완전히 변화시킨 후에만 처리할 수 있습니다.
- 혈구계에서 10μL의 세포와 10μL의 Trypan blue 용액을 혼합하여 Lenti-X 293T HEK 세포를 계수합니다. 플레이트 4 x 1010cm 플레이트당 6개의 Lenti-X 293T HEK 셀은 다음날 ~80% 포화도를 달성합니다.
- 이 단계부터 클래스 2 실험실에서 작업하십시오. 500μL의 적절한 transfection medium을 11.25μg 전달 벡터 SpdCas9-KRAB-MeCP2( 보충 파일 2 참조), 5.5μg envelope plasmid VSV-G 및 16.5μg pPAX2 벡터와 혼합합니다. 실온에서 5분 동안 배양합니다. 두 번째 dCas9 변형의 경우 전송 벡터 SadCas9-KRAB-MeCP2를 사용합니다( 보충 파일 3 참조).
- 별도의 튜브에서 500μL의 환원 혈청 배지와 36μL의 PEI 시약(재고: 1mg/mL)을 혼합합니다. 실온에서 5분 동안 배양합니다.
- 혼합물을 결합하고 실온에서 15분 동안 배양합니다. 혼합물을 세포 배지(6mL)에 조심스럽게 적가하고 37°C 및 5% CO2에서 밤새 배양합니다. 렌티바이러스 수율 감소를 방지하기 위해 세포가 부착된 상태로 유지되도록 합니다.
- 12-15시간 후에 혈청학적 피펫을 사용하여 배지를 오토클레이브 가능한 폐기물 병에 폐기합니다. 이제부터는 혈청학적 피펫 및 피펫 팁을 포함한 소모품을 오토클레이브 폐기물에 폐기하기 전에 소독 용액으로 헹굽니다. 새 피펫을 사용하여 transfection된 세포에 새로운 DMEM 배지 5mL를 추가합니다.
- transfection 48시간 후 혈청학적 피펫을 사용하여 상등액을 50mL canonical tube로 옮겨 렌티바이러스를 채취합니다. 표준 튜브를 냉장고에 보관하십시오. 세포에 5mL의 새로운 배지를 추가하고 24시간 동안 세포를 추가로 배양합니다.
- 첫 번째 수확물을 담고 있는 동일한 50 mL canonical tube에 상등액을 옮겨 렌티바이러스 수집을 반복합니다. 다시 5mL의 새로운 배지를 추가하고 24시간 동안 배양한 후 상등액을 50mL 표준 튜브로 옮깁니다. 첫 번째, 두 번째 및 세 번째 수확물의 결합된 상등액을 50mL 표준 튜브에 넣고 여과 및 원심분리할 때까지 4°C에서 보관합니다.
- 분리된 세포와 파편을 펠릿화하기 위해 위험 그룹 2 유기체와 함께 사용하도록 승인된 에어로졸 밀폐 용기 및 로터에서 500 x g 및 4°C에서 5분 동안 복합 상층액을 원심분리합니다. 0.2μm 멸균 세포 배양 등급 주사기 필터를 통해 상층액을 여과합니다.
- 100kDa에서 약 500μL의 적절한 분자량 컷오프를 가진 원심 필터 장치를 사용하여 여과된 상층액을 1,000 x g 및 4°C에서 농축합니다. 50-100 μL의 바이러스 부분 표본을 -80 °C에서 계속 사용할 때까지 보관합니다.
4. 렌티바이러스 형질도입 및 안정된 형질주입 세포의 선별
주의: 바이러스에 대한 작업은 항상 클래스 2 안전 캐비닛에서 수행하십시오.
- 형질도입 1일 전에 2 x 105 cells/well의 6-well 플레이트에 2 ml의 최종 배양 부피로 501-mel 세포를 플레이트화합니다.
- 배양 배지를 최종 농도 6μg/mL에서 폴리브레렌이 보충된 예열된 DMEM 배지 2mL로 교체합니다.
- 이제부터 클래스 2 실험실에서 작업해야 합니다. SpdCas9-KRAB-MeCP2 및 SadCas9-KRAB-MeCP2를 함유한 렌티바이러스 50μL를 세포에 동시에 첨가합니다. 혈청학적 피펫 및 피펫 팁을 포함한 소모품을 오토클레이브 폐기물에 폐기하기 전에 소독 용액으로 헹굽니다. 플레이트를 부드럽게 흔들고 37°C의 5% CO2 대기에서 배양합니다.
- 혈청학적 피펫을 사용하여 배지를 오토클레이브 가능한 병에 버리고 형질 주입 16시간 후에 블라스티시딘(10μg/mL) 및 퓨로마이신(2μg/mL)이 포함된 새 배지 2mL를 추가합니다.
- 2일 후에 배지를 blasticidin(10μg/mL) 및 퓨로마이신(2μg/mL)이 함유된 새로운 2mL 배지로 교체하여 선택을 계속합니다. 형질도입 후 7일 후 S1 조건에서 형질도입된 세포를 처리합니다.
참고: 항생제 선택은 모든 비형질도입 대조 세포가 죽을 때까지 수행해야 합니다. 대체 선택 전략은 전달 벡터의 저항 카세트에 따라 사용될 수 있습니다. 필요한 경우 살상 곡선을 사용하여 항생제 농도를 최적화합니다. 충분한 수준에서 dCas9 융합체를 발현하는 세포 집단을 얻기 위해서는 유세포 분석 또는 연속 희석 및 도금을 통한 정교한 단일 세포 클론 선택과 단백질 정량화가 필요할 수 있습니다.
5. 단백질 정량화 및 웨스턴 블롯 분석
- 혈구계에서 10μL의 세포와 10μL의 Trypan Blue 용액을 혼합하여 안정적으로 transfection된 501-mel 세포를 계수합니다. 1 x 106 개의 셀을 500 x g 및 4°C에서 5분 동안 원심분리기합니다. 상층액을 버리고 세포 펠릿을 얼음처럼 차가운 PBS 1mL에 재현탁합니다.
- 원심분리를 반복하고 단백질 분해 효소 억제제가 포함된 50μL의 RIPA 완충액에 세포 펠릿을 재현탁합니다. 얼음 위에서 15분 동안 배양하고 4°C에서 10분 동안 13,000 x g 의 원심분리로 세포 파편을 제거합니다. 상등액을 -20 °C에 보관하거나 다른 사람들이 설명한 대로 단백질 정량을 위해 즉시 사용하십시오31.
- 10 μg 단백질을 4 x LDS 완충액, 50 mM DTT와 혼합하고 최대 20 μL를 증류수로 채웁니다. 이전에 기술된 것과 같이 1 h 동안 135 V에 Tris/glycine 근거한 SDS-PAGE를 이용하여 10% 별거 젤에 단백질을 분리하십시오32. 습식 탱크 이송을 통해 90V 속도의 PVDF 멤브레인으로 90분 동안 단백질을 전달합니다.
- 실온에서 180rpm으로 1시간 동안 차단 버퍼(TBS-T의 5% 무지방 우유)에 멤브레인을 배양합니다.
- 차단 버퍼에 1:10,000 항 GAPDH 마우스 단클론 항체를 내부 대조군으로 추가합니다. 1차 항체인 anti-Cas9 (S. pyogenes) 또는 anti-Cas9 (S. aureus)를 차단 완충액에 1:1,000 희석으로 첨가하고 4°C에서 부드럽게 흔들어 밤새 배양합니다.
- TBST 버퍼로 멤브레인을 각각 3분 동안 10회 세척합니다. 2차 항-마우스 HRP 항체(1:10,000)를 실온에서 1시간 동안 배양합니다.
- TBST 버퍼로 각각 3분 동안 10번 씻습니다. ECL 기판 및 이미징 시스템을 사용하여 신호를 검출할 수 있습니다.
6. 이중 LncRNA 억제 생존력 분석
주의: 바이러스에 대한 작업은 항상 클래스 2 안전 캐비닛에서 수행하십시오.
- 형질도입 1일 전 100μL의 예열된 DMEM 배지의 96웰 플레이트에서 형질주입 7일 후 안정적인 형질주입된 SadCas9-KRAB 및 SpdCas9-KRAB를 함유하는0.1 x 10 5개 세포를 시드합니다.
- 형질도입 당일, 배양 배지를 최종 농도 6μg/mL의 폴리브레렌이 보충된 100μL의 예열된 DMEM 배지로 교체합니다. 이제부터 클래스 2 실험실에서 작업해야 합니다. 적절한 양의 렌티바이러스를 세포에 추가합니다.
참고: 이중 gRNA 벡터를 포함하는 렌티바이러스의 부피는 렌티바이러스를 적정하여 선형 곡선을 생성하기 위해 GFP를 사용하여 검사해야 합니다. - 형질 주입 16시간 후 배지를 500μg/mL Zeocin, 5μg/mL blasticidin 및 1μg/mL puromycin을 함유한 새 배지로 교체합니다.
- 형질도입 5일 후 제조업체의 지침에 따라 세포 생존율 분석을 사용하여 발광 검출을 수행합니다.
결과
SadCas9-KRAB-MeCP2 및 SpdCas9-KRAB-MeCP2의 발현 카세트를 동일한 양의 렌티바이러스와 함께 형질도입을 사용하여 501-mel 세포에 동시에 통합했습니다. 이 CRISPRi 프로토콜은 dCas9-KRAB-MeCP2 변이체의 발현에 의존하기 때문에 웨스턴 블롯 분석은 단백질 수준에서 이의 존재를 확인하는 데 이상적입니다. 양성으로 transfection된 세포를 농축한 후, dCas9-KRAB-MeCP2 ortholog의 존재를 확인하기 위해 웨스턴 블롯 분석을 위해 샘플을 수집했습니다. 성공적인 형질도입 시 SpdCas9-KRAB-MeCP2 융합 단백질(202 kDa) 및 SadCas9-KRAB-MeCP2 융합 단백질(170 kDa)에 해당하는 단일 밴드가 예상되며, 그림 2A와 같이 각 Cas9 ortholog에 특이적인 항체를 사용하여 확인되었습니다. 그러나 웨스턴 블롯이 실현 불가능한 경우 qPCR을 사용하여 mRNA 수준에서 융합 단백질을 확인할 수 있습니다.
기능적 억제인자를 안정적으로 발현하는 501-mel 세포가 생성되면 이중 gRNA 벡터를 사용하여 두 번째 렌티바이러스 형질도입을 수행했습니다. RP11 및 XLOC는 이전에 전이성 흑색종의 단기 배양에서 상향 조절되는 것으로 나타났으며, 개별 CRISPRi 표적화는 증식 및 세포 생존에 미치는 영향을 입증했습니다28. 따라서, 우리는 변형된 세포에서 누적되고 쉽게 관찰할 수 있는 성장 표현형을 예상하여 개념 증명 합성 치사 비암호화 RNA 조합으로 평가를 위해 이 lncRNA 쌍을 선택했습니다.
생존율 분석을 사용하여 암세포에서 이중 CRISPRi의 효과를 조사하려면 두 targeted lncRNA를 모두 효과적으로 억제해야 합니다. qPCR은 전사체 수준을 측정하여 이러한 억제를 확인하는 데 가장 적합한 방법입니다. 우리는 RNA 수준을 결정하고 잠재적으로 치명적일 수 있는 RP11 및 XLOC 쌍을 표적으로 할 때 세포 생존력을 조사하여 개발된 이중 CRISPRi 프로토콜의 기능을 평가했습니다. 따라서 타겟 RNA 수치를 낮추기 위해 RP11 및 XLOC를 타겟하는 다른 gRNA가 도입되었습니다. RNA의 억제는 SadCas9-KRAB-MeCP2 또는 SpdCas9-KRAB-MeCP2에 대한 타겟 특이적 gRNA를 사용하여 조사되었으며, Rosa26을 타겟하는 control gRNA와 함께 개별적으로 또는 조합하여 테스트되었습니다( 그림 2B 참조). XLOC 또는 RP11을 개별적으로 타겟하는 gRNA는 각각의 타겟을 효과적으로 억제했지만, XLOC-sa/RP11-sp 쌍을 사용해야만 XLOC과 RP11을 동시에 knockdown할 수 있었습니다.
선택된 XLOC-sa/RP11-sp gRNA 쌍을 사용하여 501-mel 세포주에서 XLOC 및 RP11 RNA 수준이 모두 성공적으로 감소함에 따라 이중 lncRNA 억제 생존력 분석이 수행되었습니다( 그림 2C 참조). 5일 후, 타겟 gRNA를 개별적으로 사용했을 때 세포 생존율은 Rosa26 control의 81%와 89%였습니다. RP11-sp와 XLOC-sa를 병용하면 RP11-sp 단독에 비해 훨씬 더 감소했으며, 세포 생존율은 Rosa26 대조군의 59%에 달했습니다.
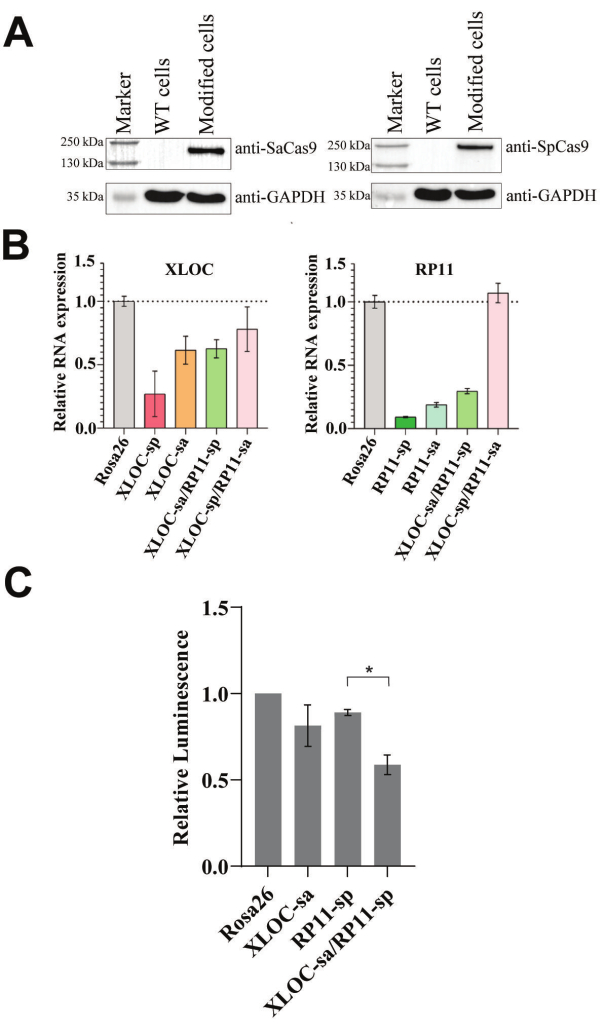
그림 2: 이중 lncRNA 억제 시스템의 기능. (A) 항-Cas9 항체를 사용하여 변형된 501-mel 세포에서 dCas9-KRAB-MeCP2 변이체를 검출하기 위해 웨스턴 블롯 분석을 수행했습니다. 형질도입이 없는 501-mel 야생형(WT) 세포를 대조군으로 사용하였다(원시 블롯에 대해서는 보충 그림 3, 보충 그림 4, 보충 그림 5, 보충 그림 6 참조). (B) 이중 gRNA를 변형된 501-mel 세포로 형질도입한 후 Rosa26 대조 세포와 관련된 RP11 및 XLOC RNA 수치의 qPCR 기반 분석. GAPDH는 하우스키핑 유전자로 사용되었습니다. 데이터는 평균 ± SD로 표현됩니다.(C) 이중 lncRNA 억제 생존율 분석은 이중 gRNA를 3개의 독립적인 transduction에서 변형된 501-mel 세포에 통합한 후 수행되었습니다. 발광 값은 Rosa26 컨트롤로 정규화되었습니다. 생존도 결과는 SEM± 평균(n = 3)으로 표현됩니다. 쌍을 이루지 않은 t-검정을 수행했습니다. 별표(*)는 p < 0.05에서 유의한 차이를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 그림 1: 플라스미드 맵. 이 맵은 Benchling(https://benchling.com/)으로 제작되었습니다. 프로토콜에서 사용된 플라스미드는 Addgene #96921 및 #110824에서 변형되었습니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 2: 보충 그림 2: 의 웨스턴 블롯 분석
dCas9-KRAB 표현식. (A) transfection 6일 후 501-mel 세포에서 SadCas9-KRAB 발현. (B) 501-mel 세포에서 SadCas9-KRAB 및 SpdCas9-KRAB의 순차적 형질도입은 형질주입 3주 후 SadCas9-KRAB 발현을 감소시켰습니다. (C) 501-mel 세포에서 SadCas9-KRAB과 SpdCas9-KRAB을 동시에 transduction한 결과, transfection 3주 후 SadCas9-KRAB 발현이 감소했다. (D) SadCas9-KRAB 및 SpdCas9-KRAB의 동시 형질도입 후 SpdCas9-KRAB 발현이 관찰될 수 있습니다. LV. μL에 사용되는 렌티바이러스의 부피. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 3: 웨스턴 블롯 SaCas9-KRAB 원시 데이터. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 4: 웨스턴 블롯 SpCas9-KRAB 원시 데이터. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 5: GAPDH_SpCas9-KRAB 원시 데이터. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 6: GAPDH_SaCas9-KRAB 원시 데이터. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 표 1: 활용된 가이드 RNA 염기서열. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 표 2: 활용된 DNA 염기서열. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 1: dual-grna-zeo-gfp의 FASTA 파일. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 2: dSpCas9-krab-mecp2의 FASTA 파일. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 3: dSaCas9-krab-mecp2의 FASTA 파일. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
본 연구에서는 두 개의 직교 종의 dCas9-KRAB-MeCP2를 기반으로 CRISPRi를 사용하여 흑색종 세포에서 이중 lncRNA 표적화 전략을 구현했습니다. 이 시스템을 활용하여 lncRNA의 합성 치사 쌍인 RP11 및 XLOC를 성공적으로 억제하여 개별적으로 억제할 때와 달리 세포 사멸을 증가시켰습니다. 그러나 CRISPRi 시스템은 off-target 유전자 억제의 위험이 있어 결과 해석에 영향을 미칠 수 있습니다. 실험적 검증은 수행되지 않았지만 in silico에서 gRNA 설계 중 잠재적인 off-target 효과를 최소화했습니다.
transduction efficiency의 가변성은 재현성에 영향을 미칠 수 있습니다. 따라서 다양한 렌티바이러스 농도를 사용하는 표준 곡선 보정을 통해 결정된 동일한 양의 렌티바이러스를 적용하여 transduction을 최적화했습니다. 그러나 새롭게 설계된 벡터의 화물 크기 증가는 렌티바이러스 패키징 효율성에 제한을 가할 수 있으며, 특히 transduction이 어려운 세포주에서 transduction efficiency를 감소시킬 수 있습니다. 결과적으로, 충분한 transduction과 강력한 dCas9 발현을 달성하려면 특정 사례에서 높은 바이러스 역가가 필요할 수 있습니다. 또한 dCas9-KRAB-MeCP2 단백질 수준과 표적 lncRNA의 RNA 발현을 자주 모니터링하여 불충분한 knockdown을 피할 수 있습니다. 또는 이러한 제한을 극복하기 위해 하나의 억제 시스템만 사용할 수 있는 dCas9 버전이 있지만 효율성 감소비용 25.
XLOC-sa-RP11-sp 조합만이 성공적인 활성을 보인 반면, 역 XLOC-sp-RP11-sa 구성은 효과가 없었습니다. 이러한 knockdown 효능의 차이에 대한 그럴듯한 설명은 PAM 특이성과 염색질 접근성33 사이의 상호 작용입니다. SaCas9 및 SpCas9가 별개의 PAM 서열을 인식한다는 점을 감안할 때, XLOC-sp-RP11-sa 방향에서 SpCas9의 타겟 부위에는 최적의 PAM이 부족할 수 있으며, 이로 인해 결합 친화도 및 후속 활성이 감소할 수 있습니다. 또한, 두 dCas9 융합 단백질 변이체의 공간적 배열은 염색질 구조 및 접근성34,35에 영향을 미칠 수 있으며, XLOC-sp-RP11-sa 구성에서 전사 기계 또는 관련 조절 인자의 모집을 잠재적으로 제한할 수 있습니다. 이러한 결과는 주어진 게놈 맥락 내에서 SaCas9 및 SpCas9의 상대적 위치가 기능적 효능에 결정적인 영향을 미칠 수 있음을 시사하며, 이는 최적화된 CRISPR 기반 조절 전략에 대한 추가 조사가 필요한 측면입니다.
개발된 프로토콜은 이중 gRNA 벡터를 변형하여 다른 합성 치사적 비암호화 RNA 쌍을 표적화하거나 스크리닝하는 데 적용할 수 있습니다. 관심 세포주가 두 dCas9 변이체를 모두 안정적으로 발현하면 다양한 접근 방식이 실현 가능해집니다. RNAi, ASO 및 shRNA는 유전자 침묵에 사용될 수 있지만 CRISPRi는 보다 간단한 스크리닝 방법론을 제공하며 작은 화합물 또는 억제제와의 조합도 생각할 수 있습니다. 이 프로토콜은 이중 gRNA 벡터를 이중 gRNA 라이브러리로 대체함으로써 암세포에서 합성 치사쌍 또는 기타 유전적 의존성을 체계적으로 스크리닝할 수 있도록 하여 암세포 표적화의 정밀도를 향상시킵니다.
렌티바이러스 형질도입을 통해 dCas9-KRAB-MeCP2 변이체의 통합을 달성했지만, 원하는 효소를 501-mel 세포에 통합하기 위해 대안적인 안정적인 형질주입 방법을 사용할 수도 있습니다. 우리의 경험에 따르면 dCas9-KRAB-MeCP2와 같은 대형 구조체의 침묵은 시간이 지남에 따라 발생할 수 있기 때문에 이것은 특히 관련이 있습니다36,37. 대안적으로, dCas9-KRAB은 더 작은 대안인 것으로 나타났다23. 우리의 경험에 비추어 볼 때, Cas9 ortholog에서 유래한 더 작은 dCas9-KRAB 융합 단백질을 사용한 형질도입은 장기간의 배양 동안 단백질 발현을 감소시켰습니다(보충 그림 2 참조). 그러므로, 일관된 단백질 발현은 반복되는 시점에서 정기적으로 모니터링되어야 합니다. 또한, selection marker 및 이중 gRNA 발현 카세트와 함께 두 개의 큰 융합 단백질을 transduction하면 세포 스트레스를 유발하거나 lentiviral 패키징 용량에 도달할 수 있습니다.
듀얼 CRISPRi에서 100% knockdown 효율을 달성하는 것은 최적화되지 않은 gRNA 설계 또는 불충분한 dCas9 발현으로 인해 어려울 수 있습니다. 이 문제를 해결하기 위해 가장 효과적인 염기서열을 식별하기 위해 고급 설계 도구를 사용하여 타겟당 여러 gRNA를 테스트해야 합니다. 또한 클론 세포주의 향상된 벡터 설계 및 특성화를 통해 dCas9-KRAB-MeCP2 발현을 최적화하면 knockdown 효율을 향상시킬 수 있습니다.
CRISPRi 시스템을 통합하기 위한 대안 전략에는 시간이 지남에 따라 안정적인 발현을 유지하는 것으로 알려진 세이프 하버 게놈 부위에 안정적으로 삽입하기 위해 transposase 벡터 또는 CRISPR 유도 통합의 사용이 포함됩니다38. 그러나 흑색종 세포를 포함한 많은 세포 유형은39 transfection이 어렵기 때문에 렌티바이러스 transduction이 선호되는 방법입니다. 전이효소 또는 Cas-기반 효소의 전기천공법은 dCas9-KRAB-MeCP2 구축물을 흑색종 세포에 통합하기 위한 대안적인 수단을 제공할 수 있다(40). 유도 가능한 knockout 시스템을 구현하면 세포 스트레스를 최소화하기 위해 구성적 발현보다 필요에 따라 dCas9-KRAB-MeCP2 발현을 활성화하는 데 유리할 것입니다.
마지막으로, 개발된 접근법은 흑색종 세포에 국한되지 않고 합성 치사율이 조사되고 있는 모든 세포 유형으로 확장될 수 있습니다. 이 접근법은 gRNA library screening modality로 확장하기 위한 것입니다. 그러나 대조군 및 최소 300 x coverage를 포함하여 유전자 쌍당 3개 이상의 gRNA가 있는 경우와 같은 조합 복잡성 증가로 인해 발현된 lncRNA의 sub-transcriptomic fraction만 실질적으로 표적화할 수 있습니다. 따라서 사용자는 라이브러리 스크리닝을 진행하기 전에 lncRNA 발현 수준 및 특정 세포 맥락에서 알려진 관련성과 같은 요인을 고려하여 후보 물질을 신중하게 사전 선택해야 합니다. 그럼에도 불구하고, 이 접근법의 조합 다양성은 다양한 암 유형 및 잠재적으로 다른 질병에 걸쳐 lncRNA의 합성 치사 상호 작용 및 기타 유전적 종속성을 조사하는 데 유용한 도구입니다. 이는 세포에서 이러한 네트워크 상호 작용을 연구하기 위해 현재 사용 가능한 툴킷을 확장합니다.
공개
Jochen Imig는 현재 Pfizer Inc.의 자금 지원을 받고 있으며, CGCIII는 Pfizer Inc., Merck KGaA 및 AstraZeneca PLC의 후원을 받고 있습니다. 의뢰자는 연구의 설계, 실행, 해석 또는 작성에 아무런 역할도 하지 않았습니다. 모든 저자는 공개할 이해 상충이 없습니다.
감사의 말
Stefan Raunser(도르트문트 막스 플랑크 분자 생리학 연구소)가 생물안전 레벨 2 실험실 공간에 접근할 수 있게 해준 것에 감사드립니다. 지적인 기여를 해준 Eric Wang과 귀중한 토론을 해준 Imig 연구소의 모든 전현직 구성원에게 특별한 감사를 드립니다. Jochen Imig는 현재 CGC III에서 Pfizer Inc.의 자금 지원을 받는 CGCIII입니다.
자료
| Name | Company | Catalog Number | Comments |
| Amicon Ultra Centrifugal Filter, 100 kDa MWCO | Millipore | UFC910024 | |
| anti-Cas9 (S. aureus) (6H4) mouse monoclonal antibody | Cell Signaling Technology | 48989 | |
| anti-Cas9 (S. pyogenes) (7A9-3A3) monoclonal antibody | Cell Signaling Technology | 14697 | |
| anti-GAPDH mouse monoclonal antibody | Sigma Aldrich | G8795 | |
| Anti-Mouse IgG (whole molecule)–Peroxidase antibody produced in rabbit | Sigma Aldrich | A9044 | |
| Bio-Rad ChemiDoc MP Imaging System | Bio-Rad | ||
| Blasticidine S hydrochloride | Sigma Aldrich | 15205-25MG | |
| CellTiter-Glo Luminescent Cell Viability Assay | Promega | G7570 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge | Eppendorf | 5415R | |
| CFX Connect Real-Time PCR Detection System | Bio-Rad | ||
| Clarity Western ECL Substrate, 500 mL | Bio-Rad | 1705061 | |
| CO2 Incubator Model CB 170 | Binder | ||
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Esp3I (BsmB1) restriction enzyme | Thermo Scientific | ER0451 | |
| Falcon 10 mL Serological Pipet | Corning | 356551 | |
| Falcon 25 mL Serological Pipet | Corning | 357525 | |
| Falcon 5 mL Serological Pipet | Corning | 356543 | |
| Fetal Bovine Serum | Sigma Aldrich | F9665-500ML | |
| Gibco Dulbecco’s modified Eagle’s medium (DMEM), high glucose, pyruvate | Gibco | 41966029 | |
| Human melanoma cell line 501-mel | was kindly provided by the Aifantis Lab (New York University) | RRID: CVCL_4633 | |
| Immobilon -P PVDF Membrane | Millipore | IPVH00010 | |
| Lenti-X 293T HEK cell line | Takara Bio | 632180 | |
| Mini Trans-Blot Cell system | Bio-Rad | ||
| Mini-PROTEAN Tetra Handcast System | Bio-Rad | ||
| NEBuilder HiFi DNA Assembly Reaction | New England Biolabs | E2621 | |
| Non-fat milk powder | Biomol | 54650 | |
| NuPAGE LDS Sample Buffer (4x) | Invitrogen | NP0008 | |
| One Shot Stbl Chemically Competent E. coli | Invitrogen | C737303 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985062 | |
| PCR Tubes 0.5 ml (Flat Cap) | VWR International | 732-3207 | |
| Phosphate-Buffered Saline (PBS) | Corning | 45000-446 | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | Thermo Scientific | F531L | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Polybrene | Sigma Aldrich | TR-1003-G | |
| Polyethylenimine, branched | Sigma Aldrich | 408727 | |
| Puromycin dihydrochloride | Santa Cruz Biotechnology | sc-108071A | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| SafeSeal Microcentrifuge Tube 1.5 mL | Sarstedt | 72,706 | |
| Sodium chloride, 5 M Aqua Solution, RNase Free | Alfa Aesar | J60434.AE | |
| Sodium Dodecyl Sulfate (SDS) | Sigma Aldrich | L3771 | |
| Syringe Filter PES 33mm 0.2 μM | Fisher Scientific | 15206869 | |
| TC Dish 100, Standard | Sarstedt | 8,33,902 | |
| TC Plate 6 Well, Standard, F | Sarstedt | 83,39,20,005 | |
| Tris base | Roche | 10708976001 | |
| TWEEN 20 | Sigma Aldrich | P9416-50ML |
참고문헌
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Szcześniak, M. W., Wanowska, E., Mukherjee, N., Ohler, U., Makałowska, I. Towards a deeper annotation of human lncRNAs. Biochim Biophys Acta Gene Regul Mech. 1863 (4), 194385(2020).
- Fang, S., et al. NONCODEV5: a comprehensive annotation database for long non-coding RNAs. Nucl Acids Res. 46 (D1), D308-D314 (2018).
- Lin, J., et al. Human-specific lncRNAs contributed critically to human evolution by distinctly regulating gene expression. eLife. 12, RP89001.2(2023).
- Sarropoulos, I., Marin, R., Cardoso-Moreira, M., Kaessmann, H. Developmental dynamics of lncRNAs across mammalian organs and species. Nature. 571 (7766), 510-514 (2019).
- Melé, M., et al. Human genomics. The human transcriptome across tissues and individuals. Science. 348 (6235), 660-665 (2015).
- Cabili, M. N., et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev. 25 (18), 1915-1927 (2011).
- Liu, S. J., Dang, H. X., Lim, D. A., Feng, F. Y., Maher, C. A. Long noncoding RNAs in cancer metastasis. Nat Rev Cancer. 21 (7), 446-460 (2021).
- Ahmad, M., Weiswald, L. B., Poulain, L., Denoyelle, C., Meryet-Figuiere, M. Involvement of lncRNAs in cancer cells migration, invasion and metastasis: cytoskeleton and ECM crosstalk. J Exp Clin Cancer Res. 42 (1), 173(2023).
- Jain, A. K., et al. LncPRESS1 Is a p53-Regulated LncRNA that Safeguards Pluripotency by Disrupting SIRT6-Mediated De-acetylation of Histone H3K56. Mol Cell. 64 (5), 967-981 (2016).
- Bergmann, J. H., et al. Regulation of the ESC transcriptome by nuclear long noncoding RNAs. Genome Res. 25 (9), 1336-1346 (2015).
- Kretz, M., et al. Control of somatic tissue differentiation by the long non-coding RNA TINCR. Nature. 493 (7431), 231-235 (2013).
- Yoon, J. H., et al. LincRNA-p21 suppresses target mRNA translation. Mol Cell. 47 (4), 648-655 (2012).
- Huarte, M. The emerging role of lncRNAs in cancer. Nat Med. 21 (11), 1253-1261 (2015).
- Badowski, C., He, B., Garmire, L. X. Blood-derived lncRNAs as biomarkers for cancer diagnosis: the Good, the Bad and the Beauty. NPJ Precis Oncol. 6 (1), 40(2022).
- Zong, X., et al. Knockdown of nuclear-retained long noncoding RNAs using modified DNA antisense oligonucleotides. Meth Mol Biol. 1262, 321-331 (2015).
- Gagnon, K. T., Li, L., Chu, Y., Janowski, B. A., Corey, D. R. RNAi factors are present and active in human cell nuclei. Cell Rep. 6 (1), 211-221 (2014).
- Zeng, Y., Cullen, B. R. RNA interference in human cells is restricted to the cytoplasm. RNA. 8 (7), 855-860 (2002).
- Yang, J., et al. CRISPR/Cas9-mediated noncoding RNA editing in human cancers. RNA Biol. 15 (1), 35-43 (2018).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355 (6320), aah7111(2017).
- Tsung, K., et al. CRISPRi screen of long non-coding RNAs identifies LINC03045 regulating glioblastoma invasion. PLoS Genet. 20 (6), e1011314(2024).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Boettcher, M., McManus, M. T. Choosing the right tool for the job: RNAi, TALEN, or CRISPR. Mol Cell. 58 (4), 575-585 (2015).
- Yeo, N. C., et al. An enhanced CRISPR repressor for targeted mammalian gene regulation. Nat Meth. 15 (8), 611-616 (2018).
- Parrish, P. C. R., et al. Discovery of synthetic lethal and tumor suppressor paralog pairs in the human genome. Cell Rep. 36 (9), 109597(2021).
- Najm, F. J., et al. Orthologous CRISPR-Cas9 enzymes for combinatorial genetic screens. Nat Biotechnol. 36 (2), 179-189 (2018).
- Petroulia, S., et al. CRISPR-inhibition screen for lncRNAs linked to melanoma growth and metastasis. bioRxiv. , (2024).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- New England Biolabs. NEBuilder HiFi DNA Assembly Reaction (E2621) v2. , (2020).
- Cortés-Ríos, J., et al. Protein quantification by bicinchoninic acid (BCA) assay follows complex kinetics and can be performed at short incubation times. Anal Biochem. 608, 113904(2020).
- Junior, N. Polyacrylamide Gel Electrophoresis (SDS-PAGE) v1. , (2019).
- Chen, X., Liu, J., Janssen, J. M., Gonçalves, M. A. F. V. The chromatin structure differentially impacts high-specificity CRISPR-Cas9 Nuclease strategies. Mol Ther Nucl Acids. 8, 558-563 (2017).
- Schep, R., et al. Impact of chromatin context on Cas9-induced DNA double-strand break repair pathway balance. Mol Cell. 81 (10), 2216-2230.e10 (2021).
- Daer, R. M., Cutts, J. P., Brafman, D. A., Haynes, K. A. The impact of chromatin dynamics on Cas9-Mediated genome editing in human cells. ACS Syn Biol. 6 (3), 428-438 (2017).
- Chavez, M., Rane, D. A., Chen, X., Qi, L. S. Stable expression of large transgenes via the knock-in of an integrase-deficient lentivirus. Nat Biomed Eng. 7 (5), 661-671 (2023).
- Ellis, J. Silencing and variegation of gammaretrovirus and lentivirus vectors. Human Gene Ther. 16 (11), 1241-1246 (2005).
- Shrestha, D., et al. Genomics and epigenetics guided identification of tissue-specific genomic safe harbors. Genome Biol. 23 (1), 199(2022).
- Chu, Z., et al. Enhanced gene transfection and induction of apoptosis in melanoma cells by branched poly(β-amino ester)s with uniformly distributed branching units. J Control Release. 367, 197-208 (2024).
- Han, S. Y., et al. Nucleofection is a highly effective gene transfer techfnique for human melanoma cell lines. Exp Dermatol. 17 (5), 405-411 (2008).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유