Method Article
Stratégie d’interférence CRISPR double pour cibler les interactions létales synthétiques entre des ARN non codants dans des cellules cancéreuses
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Cette étude présente un double système CRISPRi ciblant les ARN longs non codants dans les cellules de mélanome. Il permet l’inactivation combinatoire de gènes et le criblage létal synthétique, en identifiant les interactions d’ARNlnc spécifiques au cancer pour des stratégies thérapeutiques potentielles.
Résumé
Les ARN longs non codants (ARNlnc) représentent une classe vaste et fonctionnellement diversifiée de molécules d’ARN, avec plus de 100 000 prédictions dans le génome humain. Bien que les ARNlnc soient moins conservés d’une espèce à l’autre que les gènes codant pour des protéines, ils jouent un rôle essentiel dans la régulation des gènes, les interactions avec la chromatine et la progression du cancer. Leur implication dans le cancer en fait des cibles thérapeutiques prometteuses. L’interférence CRISPR (CRISPRi), utilisant un Cas9 catalytiquement inactif fusionné avec un répresseur transcriptionnel tel que KRAB-MeCP2, offre une méthode précise pour cibler les ARNlnc nucléaires et évaluer leurs fonctions. Cette étude introduit un système CRISPRi double utilisant les technologies CRISPRi orthogonales de Staphylococcus aureus et Streptococcus pyogenes basé sur dCas9-KRAB-MeCP2, optimisé pour le ciblage combinatoire des ARNlnc dans les cellules de mélanome humain. Le protocole facilite l’inactivation combinatoire de gènes ou le criblage létal synthétique de paires d’ARNlnc, fournissant ainsi un nouvel outil pour la recherche sur le cancer. En explorant la létalité synthétique entre les ARNlnc, cette approche peut aider à identifier les interactions entre les ARNlnc essentielles à la survie des cellules cancéreuses, offrant ainsi de nouvelles stratégies thérapeutiques. La fonctionnalité du système dual est démontrée, mettant en évidence son potentiel dans l’identification des interactions critiques de lncRNA spécifiques au cancer.
Introduction
Bien que moins de 3 % du génome humain code pour des protéines, environ 80 % du génome est transcrit 1,2. Parmi les unités transcriptionnelles non codantes, des dizaines de milliers sont classées comme de longs ARN non codants (ARNlnc) dépassant 200 nucléotides, et le nombre total d’ARNlnc humains est estimé à plus de 100 000 3,4. Contrairement aux gènes codants, les ARNlnc sont moins conservés d’une espèce à l’autre. Étant donné que les humains partagent 99 % de leur génome avec des primates comme les chimpanzés, on suppose que les ARNlnc ont une influence beaucoup plus grande sur l’évolution phénotypique 5,6. Ces résultats indiquent des fonctions cellulaires importantes des ARNlnc. Bien que la régulation des ARNlnc et leurs interactions avec les protéines de liaison à l’ARN et d’autres ARN restent incomplètement comprises, et que de nombreux ARNlnc n’aient pas encore été entièrement annotés, il est clair que les ARNlnc présentent des modèles d’expression spécifiques aux cellules et aux tissus dans la santé et les maladies, telles que le cancer 7,8,9,10. Ils sont impliqués dans diverses fonctions, notamment la régulation de la transcription des gènes, l’implication dans les interactions de la chromatine11, le traitement de l’ARN12, la stabilisation de l’ARN13 et la régulation de la traduction14.
Dans le cancer, des ARNlnc très divers et spécifiques à des types cellulaires influencent le développement tumoral et les métastases en régulant l’expression des gènes, mettant en évidence leur potentiel en tant que cibles thérapeutiques précieuses15. Au-delà de la détection des ARNlnc en tant que biomarqueurs dans des échantillons tumoraux16, le ciblage des ARNlnc spécifiques aux tumeurs pour perturber leurs fonctions en aval présente un potentiel important à la fois dans les applications cliniques et la recherche fondamentale. Les approches basées sur l’ARN pour élucider les rôles des ARNlnc comprennent les oligonucléotides antisens (ASOs), les ARN en épingle à cheveux courts et les petits ARN interférents (siRNAs)17,18. Alors que le siRNA est couramment utilisé pour les criblages de silençage génique, l’inactivation basée sur le siRNA est limitée au cytoplasme19. Cependant, lncRNA opère fréquemment à l’intérieur du noyau.
Alternativement, l’interférence CRISPRi (Clustered Regularly Interspaced Short Palindromic Repeats) peut être utilisée pour inhiber les ARNlnc dans les cancers humains20. De plus, les cribles CRISPRi à l’échelle du génome peuvent être facilement programmés et cibler un large éventail de gènes codants et non codants pour examiner leur impact fonctionnel21,22. Dans CRISPRi, un Cas9 déficient catalytique (dCas9) est fusionné à un domaine répresseur transcriptionnel, tel que le domaine23 de la boîte associée à Krüppel (KRAB). La répression génique par dCas9-KRAB est guidée par un ARN guide (ARNg) vers la région d’intérêt. CRISPRi contrôle les gènes au niveau de l’ADN, ce qui conduit à une plus grande efficacité et à des phénotypes de perte de fonction souhaités, contrairement à l’interférence de l’ARN, qui est active au niveau post-transcriptionnel24. En réponse à l’efficacité limitée de KRAB dans le silençage cible, une fusion de KRAB et de MeCP2 avec dCas9 a été introduite comme stratégie de répression plus efficace25.
Bien que le silençage d’un seul gène puisse affecter la viabilité du cancer, des interactions létales synthétiques doubles ou multiples peuvent sauver les cellules cancéreuses de la mort cellulaire26. La létalité synthétique implique deux gènes ou plus, chacun pouvant compenser la fonction de l’autre. Pour surmonter les problèmes liés aux cribles d’inactivation d’un seul gène, les stratégies CRISPR doubles ciblant des paires de gènes codant pour des protéines létales offrent une approche prometteuse27.
Ici, nous présentons un protocole pour l’utilisation combinatoire du ciblage orthogonal basé sur CRISPRi de lncRNA ou d’autres paires d’ARN non codants en utilisant Staphylococcus aureus et Streptococcus pyogenes dCas9 fusionnés à KRAB-MeCP2 dans des cellules de mélanome humain (voir Figure 1). Le protocole peut être utilisé pour l’inactivation combinatoire classique de CRISPR ou comme criblage basé sur CRISPRi de paires létales synthétiques dans le cancer. L’utilisation de deux espèces dCas9 différentes, SpCas9 et SaCas9, dans l’approche CRISPRi permet de cibler indépendamment des loci génomiques distincts avec une réactivité croisée minimale, améliorant la spécificité et la flexibilité tout en assurant une grande sélectivité sur la cible. Différents motifs adjacents protospacer (PAMs) sont utilisés : NGG pour SpCas9 et NNGRRT pour SaCas9. Le système dCas9 double résout des problèmes tels que la compatibilité limitée des ARNsg en permettant une modulation précise et simultanée et en réduisant la concurrence du complexe ARNg-RNP lors de l’utilisation d’un seul type de dCas9. Cette innovation améliore la robustesse et la polyvalence du criblage de banques d’ARNsg doubles. En conclusion, nous fournissons un système CRISPRi double entièrement fonctionnel dans les cellules de mélanome en tant que modèle de cancer.
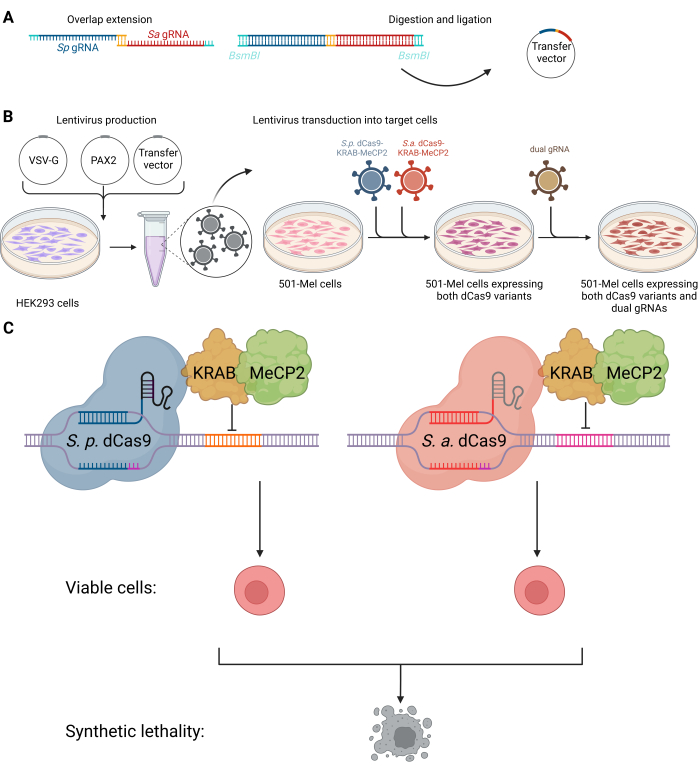
Figure 1 : Schéma du système CRISPRi double pour cibler les interactions létales synthétiques des ARN non codants. (A) Procédure de clonage du vecteur ARNg double. (B) Les cellules HEK293 ont été transfectées avec le plasmide d’enveloppe VSV-G, le plasmide d’emballage PAX2 et le vecteur de transfert pour produire des lentivirus en vue d’une transduction ultérieure en cellules 501-mel. Des lentivirus contenant l’information génétique de SpdCas9-KRAB-MeCP2 (bleu) et SadCas9-KRAB-MeCP2 (rouge) ont été intégrés simultanément dans des cellules 501-mel. Suite à la sélection d’antibiotiques, une seconde transduction lentivirale a été réalisée pour intégrer les ARNg doubles souhaités (vert olive). (C) Les cellules 501-mel exprimant les variants dCas9 interagissent avec leurs ARNg correspondants pour réduire au silence l’expression des gènes cibles, entraînant la mort des cellules cancéreuses. Créé avec BioRender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
La lignée cellulaire de mélanome humain 501-mel (RRID : CVCL_4633) a été aimablement fournie par le laboratoire Aifantis (Université de New York). La lignée cellulaire Lenti-X 293T HEK a été achetée auprès de Takara Bio. Ces lignées cellulaires ont été cultivées dans un milieu modifié de Dulbecco (DMEM) à haute teneur en glucose, complété par 10 % de sérum de veau fœtal (FBS) à 37 °C dans une atmosphère à 5 % de CO2 dans des conditions stériles. Les cellules ont été traversées jusqu’à ce qu’elles atteignent une confluence de 80 à 90 %. Les lignées cellulaires ont été régulièrement testées pour détecter la contamination par les mycoplasmes.
1. Concevoir des ARNg à l’aide d’une plateforme de conception d’ARNg en ligne
- Concevez un ARNg spécifique à SpCas9 et un ARNg spécifique à SaCas9 pour chaque ARNlnc cible afin d’évaluer quelle variante donne des résultats d’inactivation supérieurs.
- Ciblez les ARNlnc souhaités, ici RP11-120D5.1 (RP11) et XLOC_030781 (XLOC), en utilisant l’ARNg le plus efficace pour chaque S. pyogenes (gRNA-sp) Cas9 et S. aureus (gRNA-sa) Cas9.
REMARQUE : Les ARNg de S. pyogenes peuvent être extraits de Petroulia et al.28. - Suivez les recommandations spécifiques au site pour donner la priorité à l’activité ciblée la plus élevée, exprimée par des scores cibles élevés, tout en minimisant les correspondances hors cible, comme suggéré par les outils de conception d’ARNg.
- Choisissez le génome GRCh38 (hg, Homo sapiens). Sélectionnez le site PAM pour les applications spécifiques SpCas9 (NGG) et SaCas9 (NNGRRT).
- Utiliser la méthode de notation hors cible de Hsu et al.29. Positionnez les ARNg dans une fenêtre de -150 pb à +50 pb autour du site de départ transcriptionnel pour des performances optimales.
- Choisissez les ARNg ayant le score le plus élevé sur la cible et hors cible (voir le tableau supplémentaire 1).
- Combinez un ARNg témoin non ciblé ou brouillé, ici contre Rosa26, avec chaque ARNg cible pour créer un vecteur d’ARNg double, ciblant un ARNlnc. La combinaison d’ARNg cibles dans un plasmide permet une modification spécifique au site des deux sites cibles.
REMARQUE : Le vecteur d’ARNg double hébergeant l’ARNg de contrôle et l’ARNg cible permet l’inactivation d’un ARNlnc cible, tandis que les vecteurs d’ARNg doubles contenant des ARNg ciblant des sites permettent l’inactivation simultanée d’ARNlnc ciblés.
2. Clonage de l’ARNg
- Effectuez une PCR d’extension de chevauchement à l’aide de deux fragments d’ADN contenant la séquence d’ARNg. Utilisez l’amorce directe U6 et l’amorce inverse H1 (voir le tableau supplémentaire 2) pour l’amplification, comme décrit par Najm et al.27.
- Mettez en place une réaction PCR de 50 μL avec 25 μL de 2 mélanges maîtres PCR haute fidélité avec tampon HF et 1 μL de chaque fragment d’ADN contenant l’ARNg de S. pyogenes ou de S. aureus . Exécutez la réaction dans les conditions de cycle suivantes : dénaturation initiale à 98 °C pendant 30 s, suivie de 15 cycles de dénaturation à 98 °C pendant 5 s, de recuit à 55 °C pendant 10 s et d’allongement à 72 °C pendant 15 s.
- Ajouter 2,5 μL d’amorce directe U6 et 2,5 μL d’amorce inverse H1 après avoir terminé les 15 cycles initiaux, et soumettre la réaction à 20 cycles supplémentaires dans les mêmes conditions, avec une extension finale pendant 5 min à 72 °C.
- Purifier les produits PCR à l’aide d’un kit de purification PCR selon les instructions du fabricant.
- Digérer 1 μg du plasmide modifié à l’aide de 1 μL d’enzyme de restriction BsmB1 et 1 μL de tampon 10x pendant 30 min à 37 °C dans une réaction de 10 μL.
- Cloner les produits de PCR dans le plasmide digéré à double ARNg-Zeo-GFP (voir la figure supplémentaire 1 et le fichier supplémentaire 1) à l’aide d’un protocole de réaction d’assemblage d’ADN HiFi tel que décrit par d’autres30.
- Transformez des cellules E. coli chimiquement compétentes en mélangeant 50 μL de culture bactérienne avec 4 μL de produit d’assemblage selon les instructions du fabricant.
3. Production de lentivirus
ATTENTION : Manipulez les lentivirus actifs à toutes les étapes en suivant les consignes de sécurité appropriées. Effectuez toujours le travail avec des virus dans une enceinte de sécurité de classe 2 pour éviter les dangers liés à la formation inévitable d’aérosols. Effectuer tous les travaux à l’extérieur de l’enceinte de sécurité, comme la centrifugation, dans des contenants étanches aux aérosols et des rotors approuvés pour une utilisation avec des organismes du groupe de risque 2. Portez des vêtements de protection appropriés, y compris une blouse de laboratoire, des gants jetables et des lunettes de sécurité, en tout temps dans la zone de travail. Les surfaces de travail doivent être traitées avec une solution désinfectante, et les autres déchets doivent également être traités avec une solution désinfectante, ce qui leur permet de rester dans l’armoire scellée sous la lumière UV pendant au moins 240 min. Les pipettes sérologiques et les pointes de pipette utilisées doivent être rincées dans une solution désinfectante avant d’être éliminées dans des flacons autoclavables. Ensuite, les déchets doivent être éliminés dans le conteneur à déchets S2. Les déchets S2 seront inactivés par autoclavage. Les lignées cellulaires transduites peuvent être manipulées dans des conditions S1 au plus tôt 2 jours après la transduction et seulement après au moins deux changements complets du milieu de culture avec un milieu exempt de virus.
- Comptez les cellules Lenti-X 293T HEK en mélangeant 10 μL de cellules avec 10 μL de solution de bleu de trypan dans un hémocytomètre. Plaque 4 x 106 cellules Lenti-X 293T HEK par plaque de 10 cm pour atteindre ~80 % de confluence le lendemain.
- Travailler dans un laboratoire de classe 2 à partir de cette étape. Mélanger 500 μL d’un milieu de transfection approprié avec un vecteur de transfert de 11,25 μg SpdCas9-KRAB-MeCP2 (voir le fichier supplémentaire 2), un plasmide d’enveloppe de 5,5 μg VSV-G et un vecteur pPAX2 de 16,5 μg. Incuber 5 min à température ambiante. Pour la deuxième variante dCas9, utilisez le vecteur de transfert SadCas9-KRAB-MeCP2 (voir le fichier supplémentaire 3).
- Dans un tube séparé, mélanger 500 μL de milieu sérique réduit avec 36 μL de réactif PEI (stock : 1 mg/mL). Incuber 5 min à température ambiante.
- Mélanger les mélanges et incuber 15 min à température ambiante. Ajouter le mélange délicatement dans le milieu cellulaire (6 ml) et incuber toute la nuit à 37 °C et 5 % de CO2. Assurez-vous que les cellules restent attachées pour éviter une réduction du rendement lentiviral.
- Verser le milieu dans un flacon de déchets autoclavable à l’aide d’une pipette sérologique après 12 à 15 h. Désormais, rincez les consommables, y compris les pipettes sérologiques et les pointes de pipette, avec la solution désinfectante avant de les jeter dans les déchets de l’autoclave. Ajouter 5 mL de milieu DMEM frais dans les cellules transfectées à l’aide d’une pipette neuve.
- Récolter les lentivirus en transférant le surnageant à l’aide d’une pipette sérologique dans un tube canonique de 50 mL 48 h après la transfection. Conservez le tube canonique au réfrigérateur. Ajoutez 5 ml de milieu frais aux cellules et incubez les cellules pendant 24 heures supplémentaires.
- Répéter la collecte des lentivirus en transférant le surnageant dans le même tube canonique de 50 mL contenant la première récolte. Encore une fois, ajoutez 5 ml de milieu frais, incubez pendant 24 h et transférez ensuite le surnageant dans le tube canonique de 50 ml. Stocker les surnageants combinés des première, deuxième et troisième récoltes dans le tube canonique de 50 mL à 4 °C jusqu’à la filtration et la centrifugation.
- Centrifuger le surnageant combiné pendant 5 min à 500 x g et 4 °C dans des récipients étanches aux aérosols et des rotors approuvés pour une utilisation avec des organismes du groupe de risque 2 pour granuler les cellules détachées et les débris. Filtrez le surnageant à l’aide de filtres à seringue stériles de 0,2 μm de culture cellulaire.
- Concentrer le surnageant filtré à 1 000 x g et 4 °C à l’aide d’une unité de filtration centrifuge avec une coupure de poids moléculaire appropriée de 100 kDa à environ 500 μL. Conserver les aliquotes virales de 50 à 100 μL à -80 °C jusqu’à ce qu’elles soient utilisées à nouveau.
4. Transduction du lentivirus et sélection de cellules transfectées stables
ATTENTION : Effectuez toujours des travaux avec des virus dans une enceinte de sécurité de classe 2.
- Plaquez les cellules 501-mel dans une plaque à 6 puits à 2 x 105 cellules/puits avec un volume de culture final de 2 mL, 1 jour avant la transduction.
- Remplacer le milieu de culture par 2 mL de milieu DMEM préchauffé complété par du polybrène à une concentration finale de 6 μg/mL.
- Désormais, le travail en laboratoire de classe 2 est requis. Ajouter simultanément 50 μL de lentivirus contenant SpdCas9-KRAB-MeCP2 et SadCas9-KRAB-MeCP2 aux cellules. Rincez les consommables, y compris les pipettes sérologiques et les pointes de pipette, avec la solution désinfectante avant de les jeter dans les déchets de l’autoclave. Agitez doucement la plaque et incubez à 37 °C dans une atmosphère à 5 % de CO2 .
- Verser le milieu dans un flacon autoclavable à l’aide d’une pipette sérologique et ajouter 2 mL de milieu frais contenant de la blasticidine (10 μg/mL) et de la puromycine (2 μg/mL) 16 h après la transduction.
- Poursuivez la sélection en remplaçant le milieu par un milieu frais de 2 mL contenant de la blasticidine (10 μg/mL) et de la puromycine (2 μg/mL) après 2 jours. Manipulez les cellules transduites dans des conditions S1 après 7 jours après la transduction.
REMARQUE : La sélection de l’antibiotique doit être effectuée jusqu’à ce que toutes les cellules témoins non transduites soient mortes. D’autres stratégies de sélection peuvent être utilisées en fonction de la cassette de résistance dans le vecteur de transfert. Si nécessaire, optimisez les concentrations d’antibiotiques à l’aide d’une courbe de destruction. Une sélection élaborée de clones unicellulaires par cytométrie en flux ou dilution et placage en série, suivie d’une quantification des protéines, peut être nécessaire pour obtenir des populations cellulaires exprimant des fusions dCas9 à des niveaux suffisants.
5. Quantification des protéines et analyse par transfert Western
- Comptez les cellules 501-mel transfectées stables en mélangeant 10 μL de cellules avec 10 μL de solution de Trypan Blue dans un hémocytomètre. Centrifuger 1 x 106 cellules pendant 5 min à 500 x g et 4 °C. Jeter le surnageant et remettre en suspension la pastille de cellule dans 1 mL de PBS glacé.
- Répéter la centrifugation et remettre en suspension la pastille cellulaire dans 50 μL de tampon RIPA contenant des inhibiteurs de protéase. Incuber sur glace pendant 15 min et retirer les débris cellulaires par centrifugation à 13 000 x g pendant 10 min à 4 °C. Stocker le surnageant à -20 °C ou l’utiliser immédiatement pour la quantification des protéines, comme décrit par d’autres31.
- Mélangez 10 μg de protéines avec 4 tampons LDS, 50 mM de DTT et remplissez jusqu’à 20 μL d’eau distillée. Séparez les protéines sur un gel de séparation à 10 % en utilisant le SDS-PAGE à base de Tris/glycine à 135 V pendant 1 h comme décrit précédemment32. Transfert des protéines sur une membrane PVDF à 90 V pendant 90 min avec un transfert en réservoir humide.
- Incuber la membrane dans un tampon bloquant (5 % de lait écrémé dans le TBS-T) pendant 1 h à 180 tr/min à température ambiante.
- Ajoutez 1:10 000 anticorps monoclonal de souris anti-GAPDH dans le tampon de blocage en tant que contrôle interne. Ajouter l’anticorps primaire anti-Cas9 (S. pyogenes) ou anti-Cas9 (S. aureus) sous forme de dilution 1:1 000 dans le tampon de blocage et incuber pendant une nuit à 4 °C en agitant doucement.
- Lavez la membrane 3x avec un tampon TBST pendant 10 min chacune. Incuber avec l’anticorps secondaire anti-souris HRP (1:10 000) pendant 1 h à température ambiante.
- Lavez 3 fois avec un tampon TBST pendant 10 min chacun. Détectez les signaux à l’aide d’un substrat ECL et d’un système d’imagerie.
6. Essai de viabilité de la répression de l’ARNlnc double
ATTENTION : Effectuez toujours des travaux avec des virus dans une enceinte de sécurité de classe 2.
- Grainez 0,1 x 105 cellules contenant des cellules stables transfectées SadCas9-KRAB et SpdCas9-KRAB 7 jours après la transduction dans une plaque à 96 puits dans 100 μL de milieu DMEM préchauffé 1 jour avant la transduction.
- Le jour de la transduction, remplacer le milieu de culture par 100 μL de milieu DMEM préchauffé complété par du polybrène à une concentration finale de 6 μg/mL. Désormais, le travail en laboratoire de classe 2 est requis. Ajoutez le volume approprié de lentivirus aux cellules.
REMARQUE : Le volume des lentivirus contenant le vecteur ARNg double doit être examiné à l’aide de la GFP pour générer une courbe linéaire en titrant les lentivirus. - Remplacer le milieu par un milieu frais contenant 500 μg/mL de Zeocin, 5 μg/mL de blasticidine et 1 μg/mL de puromycine 16 h après la transduction.
- Effectuez la détection de luminescence 5 jours après la transduction à l’aide d’un test de viabilité cellulaire selon les instructions du fabricant.
Résultats
Les cassettes d’expression de SadCas9-KRAB-MeCP2 et SpdCas9-KRAB-MeCP2 ont été intégrées simultanément dans des cellules 501-mel en utilisant la transduction avec des quantités égales de lentivirus. Étant donné que ce protocole CRISPRi repose sur l’expression de variants dCas9-KRAB-MeCP2, l’analyse par Western blot est idéale pour confirmer leur présence au niveau protéique. Après l’enrichissement des cellules transfectées positivement, des échantillons ont été prélevés pour l’analyse par transfert Western afin de confirmer la présence des orthologues dCas9-KRAB-MeCP2. Une fois la transduction réussie, une seule bande correspondant à la protéine de fusion SpdCas9-KRAB-MeCP2 (202 kDa) et à la protéine de fusion SadCas9-KRAB-MeCP2 (170 kDa) est attendue et a été confirmée, comme le montre la figure 2A, à l’aide d’anticorps spécifiques aux orthologues Cas9 respectifs. Cependant, si le transfert Western n’est pas possible, la qPCR peut être utilisée pour vérifier les protéines de fusion au niveau de l’ARNm.
Une fois que des cellules 501-mel exprimant de manière stable les répresseurs fonctionnels ont été générées, une deuxième transduction lentivirale a été effectuée à l’aide d’un vecteur ARNg double. Il a déjà été démontré que RP11 et XLOC étaient régulés à la hausse dans des cultures à court terme de mélanome métastatique, et le ciblage individuel de CRISPRi a démontré leur impact sur la prolifération et la survie cellulaire28. Ainsi, nous avons sélectionné cette paire d’ARNlnc pour l’évaluation en tant que combinaison d’ARN létal non codant synthétique de preuve de concept, anticipant un phénotype de croissance cumulatif et facilement observable dans les cellules modifiées.
Pour étudier les effets du double CRISPRi dans les cellules cancéreuses à l’aide de tests de viabilité, les deux ARNlnc ciblés doivent être efficacement réprimés. La qPCR est la méthode la plus appropriée pour confirmer ce refoulement en mesurant leurs niveaux de transcrits. Nous avons évalué la fonctionnalité du protocole dual CRISPRi développé en déterminant les niveaux d’ARN et en examinant la viabilité cellulaire lors du ciblage de la paire potentiellement mortelle RP11 et XLOC. Par conséquent, différents ARNg ciblant RP11 et XLOC ont été introduits pour réduire les niveaux d’ARN cibles. La répression de l’ARN a été étudiée à l’aide d’ARNg spécifiques à la cible pour SadCas9-KRAB-MeCP2 ou SpdCas9-KRAB-MeCP2, testés individuellement et en combinaison avec un ARNg témoin ciblant Rosa26 (voir Figure 2B). Bien que les ARNg ciblant XLOC ou RP11 aient supprimé efficacement leurs cibles respectives, l’inactivation simultanée de XLOC et de RP11 n’a été réalisée qu’avec la paire XLOC-sa/RP11-sp.
Comme les niveaux d’ARN XLOC et RP11 ont été réduits avec succès dans la lignée cellulaire 501-mel avec la paire d’ARNg XLOC-sa/RP11-sp sélectionnée, un test de viabilité de répression de lncRNA double a été réalisé (voir Figure 2C). Après 5 jours, la viabilité cellulaire était de 81 % et celle du témoin Rosa26 de 89 % lorsque les ARNg cibles ont été utilisés individuellement. La combinaison de RP11-sp et de XLOC-sa a entraîné une réduction significative supplémentaire par rapport à RP11-sp seul, avec une viabilité cellulaire à 59 % du témoin Rosa26.
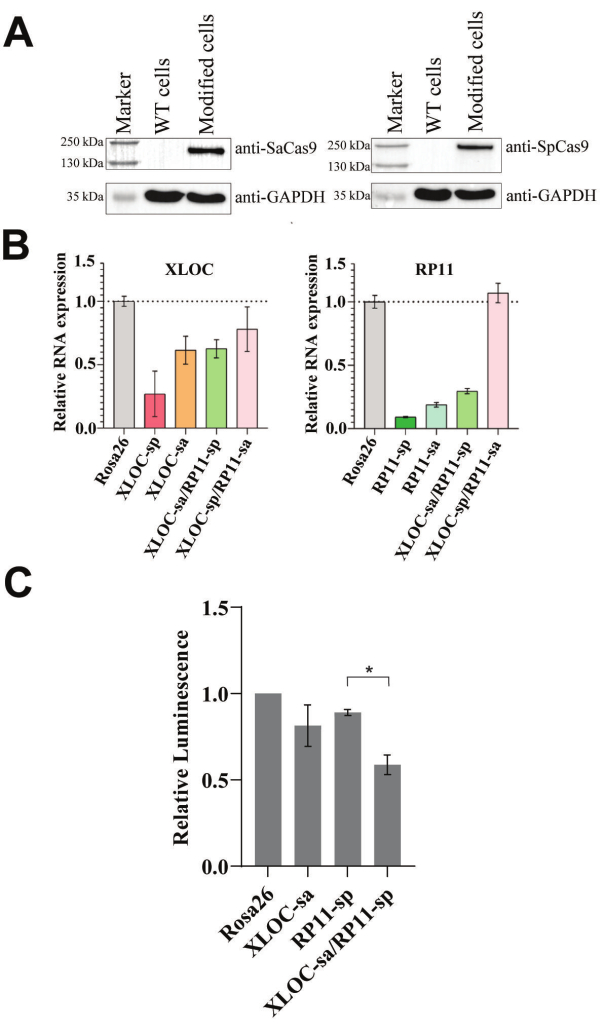
Figure 2 : Fonctionnalité du système de répression à ARNlnc double. (A) Une analyse par transfert Western a été effectuée pour détecter les variants dCas9-KRAB-MeCP2 dans des cellules 501-mel modifiées à l’aide d’anticorps anti-Cas9. Des cellules de type sauvage (WT) 501-mel sans transduction ont été utilisées comme contrôle (voir la figure supplémentaire 3, la figure supplémentaire 4, la figure supplémentaire 5 et la figure supplémentaire 6 pour le transfert brut). (B) l’analyse par qPCR des niveaux d’ARN RP11 et XLOC par rapport aux cellules témoins de Rosa26 après la transduction des ARNg doubles en cellules 501-mel modifiées. GAPDH a été utilisé comme gène d’entretien ménager. Les données sont exprimées en moyenne ± écart-type. (C) Le test de viabilité de la répression des ARNlnc doubles a été réalisé après l’intégration d’ARNg doubles dans des cellules 501-mel modifiées en trois transductions indépendantes. Les valeurs de luminescence ont été normalisées par rapport au contrôle Rosa26. Les résultats de viabilité sont exprimés sous la forme de la moyenne (n = 3) ± SEM. Un test t non apparié a été effectué. L’astérisque (*) indique une différence significative à p < 0,05. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire 1 : Carte plasmidique. La carte a été créée avec Benchling (https://benchling.com/). Les plasmides utilisés dans le protocole ont été modifiés à partir d’Addgene #96921 et #110824. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 2 : Figure supplémentaire 2 : Analyse par transfert Western
Expression dCas9-KRAB. (A) Expression de SadCas9-KRAB dans les cellules 501-mel 6 jours après la transfection. (B) La transduction séquentielle de SadCas9-KRAB et SpdCas9-KRAB dans les cellules 501-mel a conduit à une réduction de l’expression de SadCas9-KRAB 3 semaines après la transfection. (C) La transduction simultanée de SadCas9-KRAB et SpdCas9-KRAB dans des cellules 501-mel a entraîné une réduction de l’expression de SadCas9-KRAB 3 semaines après la transfection. (D) L’expression de SpdCas9-KRAB a pu être observée après la transduction simultanée de SadCas9-KRAB et SpdCas9-KRAB. LV. Le volume de lentivirus utilisés dans μL. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 3 : Données brutes du transfert Western SaCas9-KRAB. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 4 : Données brutes SpCas9-KRAB par transfert Western. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 5 : Données brutes GAPDH_SpCas9-KRAB. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 6 : Données brutes GAPDH_SaCas9-KRAB. Veuillez cliquer ici pour télécharger ce fichier.
Tableau supplémentaire 1 : Séquences d’ARN guide utilisées. Veuillez cliquer ici pour télécharger ce fichier.
Tableau supplémentaire 2 : Séquences d’ADN utilisées. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 1 : Fichier FASTA de dual-grna-zeo-gfp. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 2 : Fichier FASTA de dSpCas9-krab-mecp2. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 3 : Fichier FASTA de dSaCas9-krab-mecp2. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Dans cette étude, nous avons mis en œuvre une stratégie de ciblage double de lncRNA dans des cellules de mélanome à l’aide de CRISPRi basé sur dCas9-KRAB-MeCP2 de deux espèces orthogonales. En utilisant ce système, nous avons réussi à réprimer une paire létale synthétique d’ARNlnc, RP11 et XLOC, entraînant une augmentation de la mort cellulaire, contrairement à ce qui était le cas lorsqu’ils étaient réprimés individuellement. Cependant, les systèmes CRISPRi comportent un risque de répression génique hors cible, ce qui peut affecter l’interprétation des résultats. Bien qu’aucune validation expérimentale n’ait été effectuée, nous avons minimisé les effets potentiels hors cible lors de la conception de l’ARNg in silico.
La variabilité de l’efficacité de la transduction peut avoir un impact sur la reproductibilité. Par conséquent, nous avons optimisé la transduction en appliquant des quantités égales de lentivirus, déterminées par un étalonnage de courbe standard à l’aide de concentrations variables de lentivirus. Cependant, l’augmentation de la taille de la cargaison du vecteur nouvellement conçu peut imposer des limites à l’efficacité de l’emballage lentiviral, ce qui pourrait réduire l’efficacité de la transduction, en particulier dans les lignées cellulaires difficiles à transduire. Par conséquent, l’obtention d’une transduction suffisante et d’une expression robuste de dCas9 peut nécessiter des titres viraux élevés dans certains cas. De plus, une inactivation insuffisante peut être contournée en surveillant fréquemment les niveaux de protéine dCas9-KRAB-MeCP2 et l’expression de l’ARN des ARNlnc ciblés. Alternativement, il existe des versions dCas9 disponibles avec un seul système de répresseur pour surmonter cette limitation, mais avec un coût d’efficacité réduitede 25.
Seule la combinaison XLOC-sa-RP11-sp a démontré une activité réussie, tandis que la configuration inverse XLOC-sp-RP11-sa a été inefficace. Une explication plausible de cette différence d’efficacité est l’interaction entre la spécificité du PAM et l’accessibilité de la chromatine33. Étant donné que SaCas9 et SpCas9 reconnaissent des séquences PAM distinctes, le site cible de SpCas9 dans l’orientation XLOC-sp-RP11-sa peut manquer d’un PAM optimal, réduisant ainsi son affinité de liaison et son activité ultérieure. De plus, la disposition spatiale des deux variantes de la protéine de fusion dCas9 pourrait influencer l’architecture et l’accessibilité de la chromatine34,35, limitant potentiellement le recrutement de la machinerie transcriptionnelle ou des facteurs régulateurs associés dans la configuration XLOC-sp-RP11-sa. Ces résultats suggèrent que le positionnement relatif de SaCas9 et SpCas9 dans un contexte génomique donné peut avoir un impact critique sur leur efficacité fonctionnelle, un aspect qui justifie une étude plus approfondie pour des stratégies de modulation optimisées basées sur CRISPR.
Le protocole développé est adaptable pour cibler ou cribler d’autres paires d’ARN non codants létaux synthétiques en modifiant le vecteur ARNg double. Une fois qu’une lignée cellulaire d’intérêt exprime de manière stable les deux variantes de dCas9, diverses approches deviennent possibles. Alors que l’ARNi, l’ASO et les shARN peuvent être utilisés pour le silençage génique, CRISPRi offre une méthodologie de criblage plus simple, et sa combinaison avec de petits composés chimiques ou inhibiteurs est également envisageable. En remplaçant le vecteur ARNg double par une banque d’ARNg double, ce protocole permet le criblage systématique de paires létales synthétiques ou d’autres dépendances génétiques dans les cellules cancéreuses, améliorant ainsi la précision du ciblage des cellules cancéreuses.
Bien que nous ayons réussi à intégrer les variants dCas9-KRAB-MeCP2 par transduction lentivirale, d’autres méthodes de transfection stables peuvent également être utilisées pour incorporer les enzymes souhaitées dans les cellules 501-mel. Ceci est particulièrement pertinent, car, d’après notre expérience, le silençage de grandes constructions telles que dCas9-KRAB-MeCP2 peut se produire au fil du temps36,37. Alternativement, il s’est avéré que dCas9-KRAB était une alternative plus petite23. D’après notre expérience, la transduction avec les protéines de fusion dCas9-KRAB plus petites dérivées des orthologues Cas9 a conduit à une réduction de l’expression des protéines lors d’une culture prolongée (voir Figure supplémentaire 2). Par conséquent, l’expression constante des protéines doit être régulièrement surveillée à des moments récurrents. De plus, la transduction de deux grandes protéines de fusion avec des marqueurs de sélection et des cassettes d’expression d’ARNg doubles peut induire un stress cellulaire ou atteindre des capacités d’emballage lentiviral.
Atteindre une efficacité de 100 % dans un double CRISPRi peut être difficile en raison d’une conception sous-optimale de l’ARNg ou d’une expression insuffisante de dCas9. Pour résoudre ce problème, plusieurs ARNg par cible doivent être testés à l’aide d’outils de conception avancés afin d’identifier les séquences les plus efficaces. De plus, l’optimisation de l’expression de dCas9-KRAB-MeCP2 grâce à une meilleure conception de vecteurs et à la caractérisation de lignées cellulaires clonales peut améliorer l’efficacité de l’inhibition.
D’autres stratégies d’intégration du système CRISPRi comprennent l’utilisation de vecteurs transposases ou l’intégration dirigée par CRISPR pour obtenir une insertion stable dans les sites génomiques de la sphère de sécurité, qui sont connus pour maintenir une expression stable dans le temps38. Cependant, de nombreux types de cellules, y compris les cellules de mélanome, sont difficiles à transfecter39, ce qui fait de la transduction lentivirale la méthode préférée. L’électroporation d’enzymes à base de transposase ou de cas peut offrir un moyen alternatif d’intégrer les constructions dCas9-KRAB-MeCP2 dans les cellules de mélanome40. La mise en œuvre d’un système d’inactivation par knock-out serait avantageuse pour activer l’expression de dCas9-KRAB-MeCP2 selon les besoins plutôt que l’expression constitutive pour minimiser le stress cellulaire.
Enfin, l’approche développée ne se limite pas aux cellules de mélanome, mais peut être étendue à tout type de cellule où la létalité synthétique est étudiée. Cette approche est explicitement destinée à être étendue à la modalité de criblage de banques d’ARNg. Cependant, en raison de la complexité combinatoire accrue, par exemple, avec plus de trois ARNg par paire de gènes, y compris les contrôles et une couverture d’au moins 300 fois, seule une fraction sous-transcriptomique des ARNlnc exprimés peut être ciblée de manière pratique. Par conséquent, les utilisateurs doivent présélectionner soigneusement les candidats en tenant compte de facteurs tels que les niveaux d’expression de lncRNA et leur pertinence connue dans des contextes cellulaires spécifiques avant de procéder au criblage de la bibliothèque. Néanmoins, la polyvalence combinatoire de cette approche en fait un outil précieux pour étudier les interactions létales synthétiques et d’autres dépendances génétiques des ARNlnc dans divers types de cancer et potentiellement d’autres maladies. Cela élargit la boîte à outils actuellement disponible pour étudier ces interactions de réseau dans les cellules.
Déclarations de divulgation
Jochen Imig est actuellement financé par Pfizer Inc. CGCIII est parrainé par Pfizer Inc., Merck KGaA et AstraZeneca PLC. Les sponsors n’ont joué aucun rôle dans la conception, l’exécution, l’interprétation ou la rédaction de l’étude. Tous les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous remercions Stefan Raunser (Institut Max Planck de physiologie moléculaire, Dortmund) pour l’accès à l’espace de laboratoire de niveau 2 de biosécurité. Un merci spécial à Eric Wang pour ses contributions intellectuelles et à tous les membres passés et présents du laboratoire Imig pour leurs précieuses discussions. Jochen Imig est actuellement financé par Pfizer Inc. à la CGC III.
matériels
| Name | Company | Catalog Number | Comments |
| Amicon Ultra Centrifugal Filter, 100 kDa MWCO | Millipore | UFC910024 | |
| anti-Cas9 (S. aureus) (6H4) mouse monoclonal antibody | Cell Signaling Technology | 48989 | |
| anti-Cas9 (S. pyogenes) (7A9-3A3) monoclonal antibody | Cell Signaling Technology | 14697 | |
| anti-GAPDH mouse monoclonal antibody | Sigma Aldrich | G8795 | |
| Anti-Mouse IgG (whole molecule)–Peroxidase antibody produced in rabbit | Sigma Aldrich | A9044 | |
| Bio-Rad ChemiDoc MP Imaging System | Bio-Rad | ||
| Blasticidine S hydrochloride | Sigma Aldrich | 15205-25MG | |
| CellTiter-Glo Luminescent Cell Viability Assay | Promega | G7570 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge | Eppendorf | 5415R | |
| CFX Connect Real-Time PCR Detection System | Bio-Rad | ||
| Clarity Western ECL Substrate, 500 mL | Bio-Rad | 1705061 | |
| CO2 Incubator Model CB 170 | Binder | ||
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Esp3I (BsmB1) restriction enzyme | Thermo Scientific | ER0451 | |
| Falcon 10 mL Serological Pipet | Corning | 356551 | |
| Falcon 25 mL Serological Pipet | Corning | 357525 | |
| Falcon 5 mL Serological Pipet | Corning | 356543 | |
| Fetal Bovine Serum | Sigma Aldrich | F9665-500ML | |
| Gibco Dulbecco’s modified Eagle’s medium (DMEM), high glucose, pyruvate | Gibco | 41966029 | |
| Human melanoma cell line 501-mel | was kindly provided by the Aifantis Lab (New York University) | RRID: CVCL_4633 | |
| Immobilon -P PVDF Membrane | Millipore | IPVH00010 | |
| Lenti-X 293T HEK cell line | Takara Bio | 632180 | |
| Mini Trans-Blot Cell system | Bio-Rad | ||
| Mini-PROTEAN Tetra Handcast System | Bio-Rad | ||
| NEBuilder HiFi DNA Assembly Reaction | New England Biolabs | E2621 | |
| Non-fat milk powder | Biomol | 54650 | |
| NuPAGE LDS Sample Buffer (4x) | Invitrogen | NP0008 | |
| One Shot Stbl Chemically Competent E. coli | Invitrogen | C737303 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985062 | |
| PCR Tubes 0.5 ml (Flat Cap) | VWR International | 732-3207 | |
| Phosphate-Buffered Saline (PBS) | Corning | 45000-446 | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | Thermo Scientific | F531L | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Polybrene | Sigma Aldrich | TR-1003-G | |
| Polyethylenimine, branched | Sigma Aldrich | 408727 | |
| Puromycin dihydrochloride | Santa Cruz Biotechnology | sc-108071A | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| SafeSeal Microcentrifuge Tube 1.5 mL | Sarstedt | 72,706 | |
| Sodium chloride, 5 M Aqua Solution, RNase Free | Alfa Aesar | J60434.AE | |
| Sodium Dodecyl Sulfate (SDS) | Sigma Aldrich | L3771 | |
| Syringe Filter PES 33mm 0.2 μM | Fisher Scientific | 15206869 | |
| TC Dish 100, Standard | Sarstedt | 8,33,902 | |
| TC Plate 6 Well, Standard, F | Sarstedt | 83,39,20,005 | |
| Tris base | Roche | 10708976001 | |
| TWEEN 20 | Sigma Aldrich | P9416-50ML |
Références
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Szcześniak, M. W., Wanowska, E., Mukherjee, N., Ohler, U., Makałowska, I. Towards a deeper annotation of human lncRNAs. Biochim Biophys Acta Gene Regul Mech. 1863 (4), 194385(2020).
- Fang, S., et al. NONCODEV5: a comprehensive annotation database for long non-coding RNAs. Nucl Acids Res. 46 (D1), D308-D314 (2018).
- Lin, J., et al. Human-specific lncRNAs contributed critically to human evolution by distinctly regulating gene expression. eLife. 12, RP89001.2(2023).
- Sarropoulos, I., Marin, R., Cardoso-Moreira, M., Kaessmann, H. Developmental dynamics of lncRNAs across mammalian organs and species. Nature. 571 (7766), 510-514 (2019).
- Melé, M., et al. Human genomics. The human transcriptome across tissues and individuals. Science. 348 (6235), 660-665 (2015).
- Cabili, M. N., et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev. 25 (18), 1915-1927 (2011).
- Liu, S. J., Dang, H. X., Lim, D. A., Feng, F. Y., Maher, C. A. Long noncoding RNAs in cancer metastasis. Nat Rev Cancer. 21 (7), 446-460 (2021).
- Ahmad, M., Weiswald, L. B., Poulain, L., Denoyelle, C., Meryet-Figuiere, M. Involvement of lncRNAs in cancer cells migration, invasion and metastasis: cytoskeleton and ECM crosstalk. J Exp Clin Cancer Res. 42 (1), 173(2023).
- Jain, A. K., et al. LncPRESS1 Is a p53-Regulated LncRNA that Safeguards Pluripotency by Disrupting SIRT6-Mediated De-acetylation of Histone H3K56. Mol Cell. 64 (5), 967-981 (2016).
- Bergmann, J. H., et al. Regulation of the ESC transcriptome by nuclear long noncoding RNAs. Genome Res. 25 (9), 1336-1346 (2015).
- Kretz, M., et al. Control of somatic tissue differentiation by the long non-coding RNA TINCR. Nature. 493 (7431), 231-235 (2013).
- Yoon, J. H., et al. LincRNA-p21 suppresses target mRNA translation. Mol Cell. 47 (4), 648-655 (2012).
- Huarte, M. The emerging role of lncRNAs in cancer. Nat Med. 21 (11), 1253-1261 (2015).
- Badowski, C., He, B., Garmire, L. X. Blood-derived lncRNAs as biomarkers for cancer diagnosis: the Good, the Bad and the Beauty. NPJ Precis Oncol. 6 (1), 40(2022).
- Zong, X., et al. Knockdown of nuclear-retained long noncoding RNAs using modified DNA antisense oligonucleotides. Meth Mol Biol. 1262, 321-331 (2015).
- Gagnon, K. T., Li, L., Chu, Y., Janowski, B. A., Corey, D. R. RNAi factors are present and active in human cell nuclei. Cell Rep. 6 (1), 211-221 (2014).
- Zeng, Y., Cullen, B. R. RNA interference in human cells is restricted to the cytoplasm. RNA. 8 (7), 855-860 (2002).
- Yang, J., et al. CRISPR/Cas9-mediated noncoding RNA editing in human cancers. RNA Biol. 15 (1), 35-43 (2018).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355 (6320), aah7111(2017).
- Tsung, K., et al. CRISPRi screen of long non-coding RNAs identifies LINC03045 regulating glioblastoma invasion. PLoS Genet. 20 (6), e1011314(2024).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Boettcher, M., McManus, M. T. Choosing the right tool for the job: RNAi, TALEN, or CRISPR. Mol Cell. 58 (4), 575-585 (2015).
- Yeo, N. C., et al. An enhanced CRISPR repressor for targeted mammalian gene regulation. Nat Meth. 15 (8), 611-616 (2018).
- Parrish, P. C. R., et al. Discovery of synthetic lethal and tumor suppressor paralog pairs in the human genome. Cell Rep. 36 (9), 109597(2021).
- Najm, F. J., et al. Orthologous CRISPR-Cas9 enzymes for combinatorial genetic screens. Nat Biotechnol. 36 (2), 179-189 (2018).
- Petroulia, S., et al. CRISPR-inhibition screen for lncRNAs linked to melanoma growth and metastasis. bioRxiv. , (2024).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- New England Biolabs. NEBuilder HiFi DNA Assembly Reaction (E2621) v2. , (2020).
- Cortés-Ríos, J., et al. Protein quantification by bicinchoninic acid (BCA) assay follows complex kinetics and can be performed at short incubation times. Anal Biochem. 608, 113904(2020).
- Junior, N. Polyacrylamide Gel Electrophoresis (SDS-PAGE) v1. , (2019).
- Chen, X., Liu, J., Janssen, J. M., Gonçalves, M. A. F. V. The chromatin structure differentially impacts high-specificity CRISPR-Cas9 Nuclease strategies. Mol Ther Nucl Acids. 8, 558-563 (2017).
- Schep, R., et al. Impact of chromatin context on Cas9-induced DNA double-strand break repair pathway balance. Mol Cell. 81 (10), 2216-2230.e10 (2021).
- Daer, R. M., Cutts, J. P., Brafman, D. A., Haynes, K. A. The impact of chromatin dynamics on Cas9-Mediated genome editing in human cells. ACS Syn Biol. 6 (3), 428-438 (2017).
- Chavez, M., Rane, D. A., Chen, X., Qi, L. S. Stable expression of large transgenes via the knock-in of an integrase-deficient lentivirus. Nat Biomed Eng. 7 (5), 661-671 (2023).
- Ellis, J. Silencing and variegation of gammaretrovirus and lentivirus vectors. Human Gene Ther. 16 (11), 1241-1246 (2005).
- Shrestha, D., et al. Genomics and epigenetics guided identification of tissue-specific genomic safe harbors. Genome Biol. 23 (1), 199(2022).
- Chu, Z., et al. Enhanced gene transfection and induction of apoptosis in melanoma cells by branched poly(β-amino ester)s with uniformly distributed branching units. J Control Release. 367, 197-208 (2024).
- Han, S. Y., et al. Nucleofection is a highly effective gene transfer techfnique for human melanoma cell lines. Exp Dermatol. 17 (5), 405-411 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon