Method Article
がん細胞のノンコーディングRNA間の合成致死的相互作用を標的とするためのデュアルCRISPR干渉戦略
要約
この研究では、黒色腫細胞の長いノンコーディングRNAを標的とするデュアルCRISPRiシステムを示しています。これは、組み合わせ遺伝子ノックダウンと合成致死スクリーニングを可能にし、潜在的な治療戦略のためのがん特異的なlncRNA相互作用を特定します。
要約
長鎖ノンコーディングRNA(lncRNA)は、広大で機能的に多様なクラスのRNA分子を表しており、ヒトゲノムでは100,000以上が予測されています。lncRNAは、タンパク質をコードする遺伝子に比べて種間保存性が低いものの、遺伝子の調節、クロマチン相互作用、がんの進行に重要な役割を果たしています。彼らのがんへの関与は、彼らを有望な治療標的にします。CRISPR干渉(CRISPRi)は、触媒的に不活性なCas9をKRAB-MeCP2などの転写抑制因子と融合させ、核lncRNAを標的化し、その機能を評価するための正確な方法を提供します。この研究では、dCas9-KRAB-MeCP2に基づく黄色ブドウ球菌および化膿連鎖球菌の相補的なCRISPRi技術を使用したデュアルCRISPRiシステムを紹介し、ヒト黒色腫細胞のlncRNAのコンビナトリアルターゲティングに最適化します。このプロトコルは、lncRNAペアのコンビナトリアル遺伝子ノックダウンまたは合成致死スクリーニングを容易にし、がん研究のための新しいツールを提供します。このアプローチは、lncRNA間の合成致死性を探索することにより、がん細胞の生存に重要なlncRNA相互作用を特定するのに役立ち、新しい治療戦略を提供します。デュアルシステムの機能が実証され、重要ながん特異的なlncRNA相互作用を同定する可能性が浮き彫りになりました。
概要
ヒトゲノムの3%未満がタンパク質をコードしていますが、ゲノムの約80%が転写されています1,2。ノンコーディング転写単位のうち、数万個が200ヌクレオチドを超える長鎖ノンコーディングRNA(lncRNA)に分類され、ヒトlncRNAの総数は10万個を超えると推定されています3,4。遺伝子をコードするのとは対照的に、lncRNAは種間であまり保存されていません。ヒトがゲノムの99%をチンパンジーのような霊長類と共有していることを考えると、lncRNAは表現型の進化にはるかに大きな影響を与えると仮定されています5,6。これらの知見は、lncRNAの重要な細胞機能を示しています。lncRNAの制御およびRNA結合タンパク質や他のRNAとの相互作用は未だ完全には理解されておらず、多くのlncRNAにはまだ完全にはアノテーションされていませんが、lncRNAががんなどの健康や疾患において細胞および組織特異的な発現パターンを示すことは明らかです7,8,9,10。それらは、遺伝子転写制御、クロマチン相互作用への関与11、RNAプロセシング12、RNA安定化13、翻訳の制御14など、多様な機能に関与している。
がんでは、非常に多様な細胞型特異的なlncRNAが遺伝子発現を調節することで腫瘍の発生と転移に影響を与え、貴重な治療標的としての可能性が浮き彫りになっています15。腫瘍サンプル中のバイオマーカーとしてのlncRNAの検出だけでなく、腫瘍特異的なlncRNAを標的にしてその下流機能を破壊することは、臨床応用と基礎研究の両方で大きな可能性を秘めています。lncRNAの役割を解明するためのRNAベースのアプローチには、アンチセンスオリゴヌクレオチド(ASO)、ショートヘアピンRNA、および低分子干渉RNA(siRNA)が含まれます17,18。siRNAは一般的に遺伝子サイレンシングスクリーニングに利用されますが、siRNAベースのノックダウンは細胞質に限定されています19。しかし、lncRNAはしばしば核内で動作します。
あるいは、Clustered Regularly Interspaced Short Palindromic Repeats interference(CRISPRi)を使用して、ヒトの癌におけるlncRNAを阻害することができる20。さらに、ゲノムワイドなCRISPRiスクリーニングは、容易にプログラムでき、広範囲のコード遺伝子とノンコーディング遺伝子を標的として、それらの機能的影響を調べることができる21,22。CRISPRiでは、触媒欠損Cas9(dCas9)がKrüppel関連ボックス(KRAB)ドメイン23などの転写抑制ドメインに融合しています。dCas9-KRABによる遺伝子抑制は、ガイドRNA(gRNA)によって目的領域に誘導されます。CRISPRiはDNAレベルで遺伝子を制御し、転写後レベルで活動するRNA干渉とは対照的に、より高い効率と望ましい機能喪失表現型をもたらす24。標的サイレンシングにおけるKRABの有効性が限られていることに対応して、KRABとMeCP2とdCas9との融合がより効果的な抑制戦略として導入されました25。
単一遺伝子サイレンシングはがんの生存率に影響を与える可能性があるが、合成の二重または複数の致死的相互作用は、がん細胞を細胞死から救うことができる26。合成致死性には2つ以上の遺伝子が関与し、それぞれが他の遺伝子の機能を補うことができます。単一遺伝子ノックダウンスクリーニングの問題を克服するために、致死的なタンパク質をコードする遺伝子ペアを標的とするデュアルCRISPR戦略は、有望なアプローチを提供します27。
ここでは、ヒトメラノーマ細胞のKRAB-MeCP2に融合した 黄色ブドウ球菌 および 化膿連鎖 球菌dCas9を使用して、lncRNAまたはその他のノンコーディングRNAペアの直交CRISPRiベースのターゲティングのコンビナトリアル使用のためのプロトコルを示します( 図1を参照)。このプロトコルは、組み合わせ型の古典的CRISPRノックダウンや、がんにおける合成致死ペアのCRISPRiベースのスクリーニングに利用できます。CRISPRiアプローチでは、 SpCas9と SaCas9の2つの異なるdCas9種を使用することで、交差反応性を最小限に抑えながら異なるゲノム遺伝子座を独立して標的化することができ、高いオンターゲット選択性を確保しながら特異性と柔軟性を高めることができます。 SPCas9にはNGG、 SaCas9にはNNGRRTなど、さまざまなプロトスペーサー隣接モチーフ(PAM)が利用されます。デュアルdCas9システムは、単一のタイプのdCas9を使用する場合、正確な同時調節を可能にし、sgRNA-RNP複合体の競合を減らすことで、sgRNAの適合性が限られているなどの課題に対処します。この革新により、デュアルsgRNAライブラリースクリーニングの堅牢性と汎用性が向上します。結論として、私たちは、がんモデルとして黒色腫細胞に完全に機能するデュアルCRISPRiシステムを提供します。
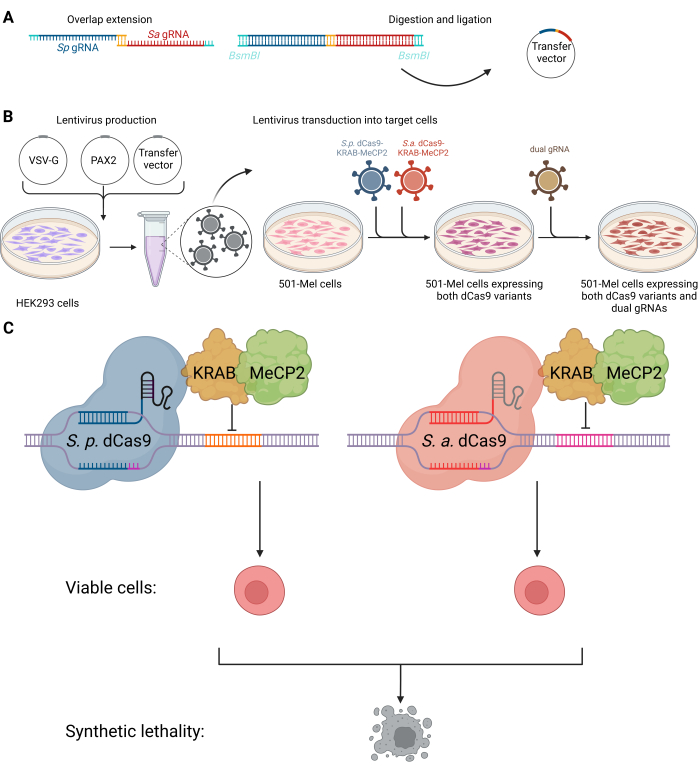
図1:ノンコーディングRNAの合成致死的相互作用を標的とするデュアルCRISPRiシステムのスキーム。 (A)デュアルgRNAベクターのクローニング手順。(B)HEK293細胞にエンベローププラスミドVSV-G、パッケージングプラスミドPAX2、およびトランスファーベクターをトランスフェクションしてレンチウイルスを作製し、その後501-mel細胞に形質導入しました。 SpdCas9-KRAB-MeCP2(青)と SadCas9-KRAB-MeCP2(赤)の遺伝情報を含むレンチウイルスを501-mel細胞に同時に組み込みました。抗生物質の選択に続いて、目的のデュアルgRNA(オリーブグリーン)を統合するために、2回目のレンチウイルス形質導入を行いました。(C)dCas9変異体を発現する501-mel細胞は、対応するgRNAと相互作用して標的遺伝子発現をサイレンシングし、がん細胞死をもたらします。BioRender.com で作成。 この図の拡大版を表示するには、ここをクリックしてください。
プロトコル
ヒト黒色腫細胞株501-mel(RRID:CVCL_4633)は、Aifantis Lab(ニューヨーク大学)のご厚意により提供されました。Lenti-X 293T HEK細胞株は、Takara Bioから購入しました。これらの細胞株は、無菌条件下で5%CO2 雰囲気中、37°Cで10%ウシ胎児血清(FBS)を添加したダルベッコ改変イーグル培地(DMEM)高グルコースで培養しました。細胞は、80%〜90%のコンフルエントに達するまで継代した。細胞株は、マイコプラズマ汚染について定期的に試験されました。
1. オンラインgRNAデザインプラットフォームを用いたgRNAデザイン
- 各標的lncRNAについて 、SpCas9に特異的な1つのgRNAと SaCas9に特異的な1つのgRNAを設計し、どちらのバリアントが優れたノックダウン結果をもたらすかを評価します。
- 所望のlncRNA、ここではRP11-120D5.1(RP11)およびXLOC_030781(XLOC)を標的とし、各 化膿性ブドウ球菌 (gRNA-sp)Cas9および 黄色ブドウ球菌 (gRNA-sa)Cas9に最も効率的なgRNAを使用する。
注: 化膿性ブドウ球菌gRNA は、Petrouliaらから抽出することができる28。 - 施設固有の推奨事項に従って、高いオンターゲットスコアで表される最も高いオンターゲット活性を優先すると同時に、gRNA デザインツールで提案されているようにオフターゲットの一致を最小限に抑えます。
- ゲノムGRCh38(hg、 ホモサピエンス)を選択します。特定の SpCas9 (NGG) と SaCas9 (NNGRRT) の PAM サイトを選択します。
- Hsu et al.29 のオフターゲット採点法を使用します。最適なパフォーマンスを得るためには、転写開始部位の周囲に-150 bpから+50 bpのウィンドウ内にgRNAを配置します。
- オンターゲットスコアとオフターゲットスコアが最も高いgRNAを選択します( 補足表1を参照)。
- ノンターゲティングまたはスクランブルされたコントロールgRNA(ここではRosa26に対して)を各ターゲットgRNAと組み合わせて、1つのlncRNAを標的とするデュアルgRNAベクターを作成します。1つのプラスミドに標的gRNAを組み合わせることで、両方の標的部位の部位特異的な修飾が可能になります。
注:制御gRNAと標的gRNAを保有するデュアルgRNAベクターは、1つの標的lncRNAのノックダウンを可能にし、部位を標的とするgRNAを含むデュアルgRNAベクターは、標的lncRNAの同時ノックダウンを可能にします。
2. gRNAクローニング
- gRNA配列を含む2つのDNA断片を用いてオーバーラップ延長PCRを行います。Najm et al.27に記載されているように、増幅にはU6フォワードプライマーとH1リバースプライマー(補足表2を参照)を使用してください。
- 25 μL の 2x High-fidelity PCR Master mix with HF buffer と、 化膿性ブドウ球菌 または 黄色ブドウ球菌 gRNA のいずれかを含む DNA フラグメント 1 μL を用いて、50 μL PCR 反応をセットアップします。反応を以下のサイクル条件で実行します:初期変性を98°Cで30秒間、続いて98°Cで5秒間15サイクルの変性、55°Cで10秒間アニール、72°Cで15秒間の伸長を行います。
- 最初の15サイクルを完了した後、2.5 μLのU6フォワードプライマーと2.5 μLのH1リバースプライマーを添加し、同じ条件下でさらに20サイクルの反応を行い、最終的に72°Cで5分間延長します。
- メーカーの指示に従って、PCR精製キットを使用してPCR産物を精製します。
- 1 μLのBsmB1制限酵素と10xバッファー1 μLを使用して、10 μLの反応で37°Cで30分間、1 μgの修飾プラスミドを消化します。
- PCR産物を消化したdual-gRNA-Zeo-GFPプラスミド( 補足図1 および 補足ファイル1を参照)にクローニングし、他の30で説明されているHiFi DNAアセンブリ反応プロトコルを使用する。
- 製造元の指示に従って、50 μLの細菌培養物と4 μLの組立製品を混合することにより、化学的にコンピテントな 大腸菌 細胞を形質転換します。
3.レンチウイルスの生産
注意: 活性レンチウイルスは、適切な安全ガイドラインに従いながら、すべてのステップで取り扱ってください。避けられないエアロゾル形成による危険を防ぐために、常にクラス2の安全キャビネットでウイルスを使用した作業を行ってください。遠心分離など、安全キャビネットの外側でのすべての作業は、リスクグループ2の生物での使用が承認されたエアロゾル密閉容器およびローターで行ってください。作業エリアでは、白衣、使い捨て手袋、安全ゴーグルなどの適切な防護服を常に着用してください。作業面は消毒液で処理する必要があり、他の廃棄物も消毒液で処理する必要があり、UV光の下で密閉されたキャビネットに少なくとも240分間留まることができます。使用済みの血清ピペットとピペットチップは、オートクレーブ可能なボトルで廃棄する前に消毒液ですすぐ必要があります。その後、廃棄物はS2廃棄物コンテナに廃棄する必要があります。S2廃棄物はオートクレーブによって不活化されます。形質導入細胞株は、形質導入後2日以内に、ウイルスを含まない培地で培地を少なくとも2回完全に交換した後にのみ、S1条件下で取り扱うことができます。
- 血球計算盤で10 μLの細胞と10 μLのトリパンブルー溶液を混合して、Lenti-X 293T HEK細胞をカウントします。プレート 4 x 106 Lenti-X 293T HEK 細胞/10 cm プレートで、翌日に ~80% のコンフルエンシーを達成します。
- このステップ以降は、クラス2の研究室で働きます。適切なトランスフェクション培地500 μLを、11.25 μgのトランスファーベクターSpdCas9-KRAB-MeCP2( 補足ファイル2を参照)、5.5 μgのエンベローププラスミドVSV-G、および16.5 μgのpPAX2ベクターと混合します。室温で5分間インキュベートします。2番目のdCas9バリアントには、トランスファー ベクターSadCas9-KRAB-MeCP2を使用します( 補足ファイル3を参照)。
- 別のチューブで、500 μL の低血清培地と 36 μL の PEI 試薬 (ストック: 1 mg/mL) を混合します。室温で5分間インキュベートします。
- 混合物を混合し、室温で15分間インキュベートします。混合物を細胞培地(6 mL)に慎重に滴下し、37°Cおよび5%CO2で一晩インキュベートします。レンチウイルスの収量が減少しないように、細胞が付着したままであることを確認してください。
- 12〜15時間後に、血清学的ピペットを使用して培地をオートクレーブ可能な廃棄物ボトルに廃棄します。今後は、血清ピペットやピペットチップなどの消耗品を消毒液ですすいでから、オートクレーブ廃棄物に廃棄してください。新しいピペットを使用して、トランスフェクションした細胞に5 mLの新鮮なDMEM培地を加えます。
- トランスフェクションの48時間後に、血清学的ピペットを使用して上清を50 mLの標準チューブに移すことにより、レンチウイルスを回収します。カノニカルチューブを冷蔵庫に保管します。5 mLの新鮮な培地を細胞に加え、細胞をさらに24時間インキュベートします。
- 上清を最初の収穫物を含む同じ50mLのカノニカルチューブに移すことにより、レンチウイルスの収集を繰り返します。再度、5 mLの新鮮な培地を加え、24時間インキュベートし、その後、上清を50 mLの標準チューブに移します。1回目、2回目、3回目の採取で組み合わせた上清を、ろ過および遠心分離まで、4°Cの50 mL標準チューブで保存します。
- リスクグループ2生物での使用が承認されたエアロゾル密閉容器およびローターで、混合した上清を500 x g および4°Cで5分間遠心分離し、剥離した細胞および破片をペレット化します。0.2 μmの滅菌細胞培養グレードのシリンジフィルターで上清をろ過します。
- ろ過した上清を、適切な分子量カットオフが100 kDaの遠心フィルターユニットを使用して、1,000 x g および4°Cで約500 μLに濃縮します。50〜100 μLのウイルスアリコートを-80°Cでさらに使用するまで保存します。
4. レンチウイルス形質導入と安定トランスフェクション細胞の選択
注意: ウイルスを使用した作業は、常にクラス2の安全キャビネットで行ってください。
- 形質導入の1日前に、501メル細胞を2 x 105 細胞/ウェルの6ウェルプレートに、最終培養量2 mLでプレート化します。
- 培地を、最終濃度6 μg/mLのポリブレンを添加した予温したDMEM培地2 mLと交換します。
- これからは、クラス2の研究室での作業が必要です。 SpdCas9-KRAB-MeCP2および SadCas9-KRAB-MeCP2を含むレンチウイルス50 μLを細胞に同時に加えます。血清ピペットやピペットチップなどの消耗品は、オートクレーブ廃棄物に廃棄する前に消毒液ですすいでください。プレートを静かに振とうし、5% CO2 雰囲気中で37°Cでインキュベートします。
- 血清学的ピペットを使用して培地をオートクレーブ可能なボトルに廃棄し、形質導入後16時間後にブラストシジン(10 μg/mL)およびピューロマイシン(2 μg/mL)を含む新鮮な培地2 mLを加えます。
- 2日後に、ブラストシジン(10 μg/mL)とピューロマイシン(2 μg/mL)を含む新鮮な2 mL培地と交換して、選択を続けます。形質導入後7日後にS1条件下で形質導入細胞を取り扱います。
注:抗生物質の選択は、形質導入されていないすべての対照細胞が死滅するまで実行する必要があります。転送ベクトルの抵抗カセットに応じて、別の選択戦略を使用できます。必要に応じて、殺傷曲線を使用して抗生物質濃度を最適化します。フローサイトメトリーまたは段階希釈およびプレーティングによる精巧な単一細胞クローン選択と、それに続くタンパク質定量により、dCas9融合を十分なレベルで発現する細胞集団を得る必要がある場合があります。
5. タンパク質定量とウェスタンブロット解析
- 血球計算盤で10 μLの細胞と10 μLのTrypan Blue溶液を混合することにより、トランスフェクションされた安定な501-mel細胞をカウントします。1 x 106 個の細胞を500 x g および4°Cで5分間遠心分離します。 上清を捨て、細胞ペレットを氷冷PBS1 mLに再懸濁します。
- 遠心分離を繰り返し、細胞ペレットをプロテアーゼ阻害剤を含む50 μLのRIPAバッファーに再懸濁します。氷上で15分間インキュベートし、13,000 x g で4°Cで10分間遠心分離して細胞破片を除去します。 上清を-20°Cで保存するか、他の人が説明しているようにタンパク質の定量にすぐに使用してください31。
- 10 μgのタンパク質を4 x LDSバッファー、50 mM DTTと混合し、蒸留水で最大20 μLを満たします。前述の32のように、Tris/グリシンベースのSDS-PAGEを135Vで1時間使用して、10%分離ゲル上でタンパク質を分離する。湿式タンクトランスファーにより、タンパク質を90Vで90分間PVDFメンブレンに転写します。
- メンブレンをブロッキングバッファー(TBS-T中の5%無脂肪乳)で室温180rpmで1時間インキュベートします。
- 1:10,000の抗GAPDHマウスモノクローナル抗体をブロッキングバッファーに内部コントロールとして追加します。一次抗体である抗Cas9(化膿性ブドウ球菌)または抗Cas9(黄色ブドウ球菌)をブロッキングバッファーに1:1,000希釈分として加え、穏やかに振とうしながら4°Cで一晩インキュベートします。
- メンブレンをTBSTバッファーで3回、各10分間洗浄します。二次抗マウスHRP抗体(1:10,000)と室温で1時間インキュベートします。
- TBSTバッファーで3回、各10分間洗浄します。ECL Substrateとイメージングシステムを使用して信号を検出します。
6. デュアルLncRNA抑制生存率アッセイ
注意: ウイルスを使用した作業は、常にクラス2の安全キャビネットで行ってください。
- 形質導入の1日前に、100 μLの予熱済みDMEM培地中の96ウェルプレートに、形質導入の7日後に安定トランスフェクションしたSadCas9-KRABおよびSpdCas9-KRABを含む5細胞をシードします。
- 形質導入の日に、培地を、最終濃度6 μg/mLのポリブレンを添加した予熱したDMEM培地100 μLと交換します。これからは、クラス2の研究室での作業が必要です。細胞に適切な量のレンチウイルスを追加します。
注:デュアルgRNAベクターを含むレンチウイルスの量は、GFPを使用して調べ、レンチウイルスを滴定することにより線形曲線を生成する必要があります。 - 形質導入後16時間後に、500 μg/mL Zeocin、5 μg/mL blasticidin、および 1 μg/mL ピューロマイシンを含む新しい培地と交換してください。
- 形質導入の5日後に、メーカーの指示に従って細胞生存率アッセイを用いて発光検出を行います。
結果
SadCas9-KRAB-MeCP2およびSpdCas9-KRAB-MeCP2の発現カセットを、等量のレンチウイルスによる形質導入を使用して同時に501-mel細胞に組み込みました。このCRISPRiプロトコールはdCas9-KRAB-MeCP2バリアントの発現に依存しているため、ウェスタンブロット分析はタンパク質レベルでの存在を確認するのに理想的です。陽性トランスフェクション細胞の濃縮後、ウェスタンブロット分析のためにサンプルを採取し、dCas9-KRAB-MeCP2オルソログの存在を確認しました。形質導入が成功すると、SpdCas9-KRAB-MeCP2融合タンパク質(202 kDa)およびSadCas9-KRAB-MeCP2融合タンパク質(170 kDa)に対応する単一バンドが期待され、図2Aに示すように、それぞれのCas9オルソログに特異的な抗体を使用して確認されました。しかし、ウェスタンブロットが不可能な場合は、qPCRを使用してmRNAレベルで融合タンパク質を検証することができます。
機能リプレッサーを安定的に発現する501-mel細胞が作製されたら、デュアルgRNAベクターを用いて2回目のレンチウイルス形質導入を行った。RP11とXLOCは、転移性黒色腫の短期培養でアップレギュレーションされることが以前に示されており、個々のCRISPRiターゲティングは、増殖と細胞生存への影響を実証しました28。したがって、このlncRNAペアを概念実証の合成致死ノンコーディングRNAの組み合わせとして評価するために選択し、改変された細胞における累積的で容易に観察可能な増殖表現型を予想しました。
生存率アッセイを用いてがん細胞におけるデュアルCRISPRiの効果を調べるには、両方の標的lncRNAを効果的に抑制する必要があります。qPCRは、転写産物のレベルを測定することにより、この抑制を確認するのに最も適した方法です。私たちは、RNAレベルを決定し、潜在的に致死的なRP11とXLOCのペアを標的とした場合の細胞生存率を調べることにより、開発されたデュアルCRISPRiプロトコルの機能を評価しました。そのため、RP11とXLOCを標的とする異なるgRNAを導入し、標的RNAのレベルを低下させました。RNAの抑制は、 SadCas9-KRAB-MeCP2または SpdCas9-KRAB-MeCP2のいずれかの標的特異的gRNAを使用して調査し、Rosa26を標的とする対照gRNAと個別に、または組み合わせて試験しました26( 図2Bを参照)。XLOCまたはRP11をそれぞれ標的とするgRNAは、それぞれの標的を効果的に抑制しましたが、XLOCとRP11の両方の同時ノックダウンは、XLOC-sa/RP11-spペアでのみ達成されました。
XLOC-sa/RP11-sp gRNAペアを選択した501-mel細胞株において、XLOCおよびRP11 RNAレベルの両方を首尾よく低減したため、デュアルlncRNA抑制生存率アッセイを実施しました( 図2Cを参照)。5日後、細胞生存率は、標的gRNAを個別に使用した場合、Rosa26コントロールの81%と89%でした。RP11-spとXLOC-saの併用は、RP11-sp単独と比較してさらに有意な低下をもたらし、細胞生存率はRosa26コントロールの59%であった。
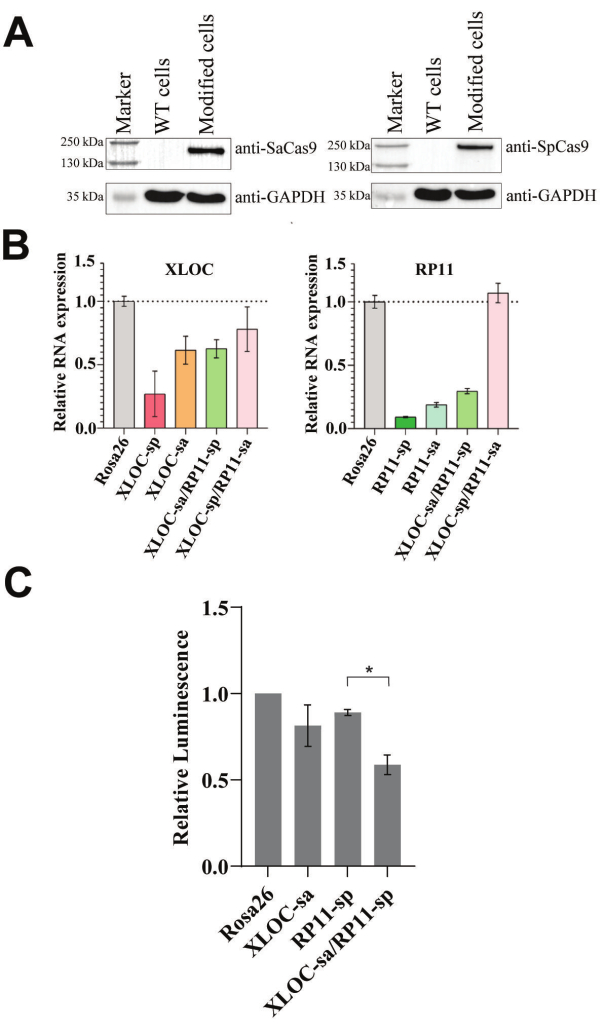
図2:デュアルlncRNA抑制システムの機能 (A)ウェスタンブロット分析を行い、抗Cas9抗体を用いて改変された501-mel細胞のdCas9-KRAB-MeCP2変異体を検出しました。形質導入を伴わない501-melの野生型(WT)細胞をコントロールとして使用した(生ブロットについては、補足図3、補足図4、補足図5、補足図6を参照)。(B)修飾された501-mel細胞へのデュアルgRNAの形質導入後のRosa26コントロール細胞と比較したRP11およびXLOC RNAレベルのqPCRベースの分析。GAPDHはハウスキーピング遺伝子として使用されました。(C)デュアルlncRNA抑制生存率アッセイは±、デュアルgRNAを3つの独立した形質導入で修飾された501-mel細胞に統合した後に実施しました。発光値はRosa26コントロールに正規化しました。生存率はSEM±平均(n = 3)で表されます。対応のないt検定が実施されました。アスタリスク(*)は、p < 0.05 での有意差を示します。 この図の拡大版を表示するには、ここをクリックしてください。
補足図1:プラスミドマップ。 マップは Benchling (https://benchling.com/) を使用して作成されました。プロトコールで利用されたプラスミドは、Addgene #96921および#110824から改変されました。 このファイルをダウンロードするには、ここをクリックしてください。
補足図2:補足図2:ウェスタンブロット解析
dCas9-KRAB式。 (A)トランスフェクションの6日後における501-mel細胞におけるSadCas9-KRABの発現。(B)501-mel細胞におけるSadCas9-KRABおよびSpdCas9-KRABの逐次形質導入は、トランスフェクション後3週間でSadCas9-KRAB発現の低下をもたらした。(C)501-mel細胞におけるSadCas9-KRABとSpdCas9-KRABの同時形質導入は、トランスフェクション後3週間でSadCas9-KRABの発現を低下させる結果となった。(D)SadCas9-KRABとSpdCas9-KRABの同時形質導入後にSpdCas9-KRABの発現が観察された。LV. レンチウイルスの使用量 (μL) この ファイルをダウンロードするには、ここをクリックしてください。
補足図3:ウェスタンブロットSaCas9-KRABの生データ。 このファイルをダウンロードするには、ここをクリックしてください。
補足図4:ウェスタンブロットSpCas9-KRABの生データ。このファイルをダウンロードするには、ここをクリックしてください。
補足図5:GAPDH_SpCas9-KRABの生データ。このファイルをダウンロードするには、ここをクリックしてください。
補足図6:GAPDH_SaCas9-KRABの生データ。このファイルをダウンロードするには、ここをクリックしてください。
補足表1:利用されたガイドRNA配列。このファイルをダウンロードするには、ここをクリックしてください。
補足表2:利用されたDNA配列。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル1:dual-grna-zeo-gfpのFASTAファイル。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル2:dSpCas9-krab-mecp2のFASTAファイル。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル3:dSaCas9-krab-mecp2のFASTAファイル。このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
この研究では、2つの直交種由来のdCas9-KRAB-MeCP2に基づくCRISPRiを使用して、メラノーマ細胞にデュアルlncRNAターゲティング戦略を実装しました。このシステムを利用して、合成致死ペアのlncRNAであるRP11とXLOCの抑制に成功し、個別に抑制した場合とは異なり、細胞死の増加をもたらしました。しかし、CRISPRiシステムにはオフターゲット遺伝子の抑制のリスクがあり、結果の解釈に影響を与える可能性があります。実験的検証は行われませんでしたが、gRNA設計 in silico中に潜在的なオフターゲット効果を最小限に抑えることができました。
形質導入効率のばらつきは、再現性に影響を与える可能性があります。そのため、さまざまなレンチウイルス濃度を使用した標準曲線キャリブレーションによって決定された同量のレンチウイルスを適用することにより、形質導入を最適化しました。しかし、新たに設計されたベクターのカーゴサイズの増加は、レンチウイルスのパッケージング効率に制限を課す可能性があり、特に形質導入が困難な細胞株では、形質導入効率が低下する可能性があります。その結果、十分な形質導入と堅固なdCas9発現を達成するためには、場合によっては高いウイルス力価が必要になることがあります。さらに、dCas9-KRAB-MeCP2タンパク質レベルと標的lncRNAのRNA発現を頻繁にモニタリングすることで、不十分なノックダウンを回避することができます。あるいは、この制限を克服するために、リプレッサーシステムが1つだけ利用可能なdCas9バージョンがありますが、効率が低下するというコストがかかります25。
XLOC-sa-RP11-spの組み合わせのみが成功した活動を示し、逆XLOC-sp-RP11-sa構成は効果がありませんでした。ノックダウン効果のこの違いについてのもっともらしい説明は、PAM特異性とクロマチンアクセシビリティとの間の相互作用である33。SaCas9およびSpCas9が別個のPAM配列を認識することを考えると、XLOC-sp-RP11-sa配向のSpCas9の標的部位は最適なPAMを欠いている可能性があり、その結果、その結合親和性およびその後の活性が低下する可能性がある。さらに、2つのdCas9融合タンパク質変異体の空間的配置は、クロマチンの構造およびアクセシビリティ34,35に影響を及ぼし、XLOC-sp-RP11-sa配置における転写機構または関連する調節因子の動員を制限する可能性がある。これらの知見は、特定のゲノムコンテキスト内でのSaCas9とSpCas9の相対的な位置付けが、それらの機能的有効性に重大な影響を与える可能性があることを示唆しており、この側面は、最適化されたCRISPRベースの調節戦略のためのさらなる研究が必要である。
開発されたプロトコルは、デュアルgRNAベクターを修飾することにより、他の合成致死ノンコーディングRNAペアを標的化またはスクリーニングすることに適応できます。目的の細胞株が両方のdCas9変異体を安定して発現すると、さまざまなアプローチが可能になります。RNAi、ASO、shRNAは遺伝子サイレンシングに用いることができますが、CRISPRiはより簡単なスクリーニング方法を提供し、低分子化合物や阻害剤との組み合わせも考えられます。このプロトコルでは、デュアルgRNAベクターをデュアルgRNAライブラリーに置き換えることで、がん細胞の合成致死ペアやその他の遺伝的依存関係の体系的なスクリーニングが可能になり、がん細胞のターゲティングの精度が向上します。
レンチウイルス形質導入によりdCas9-KRAB-MeCP2変異体の統合を達成しましたが、目的の酵素を501-mel細胞に組み込むために、別の安定トランスフェクション法を使用することもできます。我々の経験では、dCas9-KRAB-MeCP2のような大きな構造物のサイレンシングは、時間の経過とともに起こり得る36,37。あるいは、dCas9-KRABはより小さな代替であることが示されました23。私たちの経験では、Cas9オルソログに由来するより小さなdCas9-KRAB融合タンパク質による形質導入は、長期培養中のタンパク質発現の低下につながりました(補足図2を参照)。したがって、一貫したタンパク質発現は、定期的な時点で定期的にモニタリングする必要があります。さらに、2つの大きな融合タンパク質を選択マーカーおよびデュアルgRNA発現カセットとともに形質導入すると、細胞ストレスが誘発されたり、レンチウイルスのパッケージング容量に達する可能性があります。
デュアルCRISPRiで100%のノックダウン効率を達成することは、gRNA設計が最適ではなかったり、dCas9の発現が不十分であったりするため、困難な場合があります。これに対処するには、ターゲットごとに複数のgRNAを高度なデザインツールを使用してテストし、最も効果的な配列を特定する必要があります。さらに、ベクター設計の改善とクローン細胞株の特性評価を通じてdCas9-KRAB-MeCP2の発現を最適化することで、ノックダウン効率を高めることができます。
CRISPRiシステムを統合するための代替戦略には、トランスポゼースベクターの使用またはCRISPR指向の統合を使用して、長期にわたって安定した発現を維持することで知られるセーフハーバーゲノム部位への安定した挿入を達成することが含まれる38。しかし、メラノーマ細胞を含む多くの細胞種は、39のトランスフェクションが困難であるため、レンチウイルス形質導入が好ましい方法となっています。トランスポゼースまたはCasベースの酵素のエレクトロポレーションは、dCas9-KRAB-MeCP2コンストラクトをメラノーマ細胞40に統合するための代替手段を提供し得る。誘導性ノックアウトシステムを実装することは、細胞ストレスを最小限に抑えるための構成的発現よりも、必要に応じてdCas9-KRAB-MeCP2発現を活性化するのに有利です。
最後に、開発されたアプローチはメラノーマ細胞に限定されず、合成致死性が調査されている任意の細胞タイプに拡張できます。このアプローチは、gRNAライブラリーのスクリーニングモダリティに向けてスケールアップすることを明示的に意図しています。しかし、遺伝子ペアあたり3つ以上のgRNAがコントロールを含み、少なくとも300倍の範囲をカバーするなど、組み合わせの複雑さが増しているため、発現したlncRNAのサブトランスクリプトーム画分のみを実質的に標的にすることができます。したがって、ユーザーは、ライブラリスクリーニングを進める前に、lncRNAの発現レベルや特定の細胞状況での既知の関連性などの要素を考慮して、候補を事前に選択する必要があります。それにもかかわらず、このアプローチの組み合わせの多様性は、さまざまながんの種類や他の疾患にわたるlncRNAの合成致死相互作用やその他の遺伝的依存関係を調査するための貴重なツールとなっています。これにより、細胞内のこれらのネットワーク相互作用を研究するための現在利用可能なツールキットが拡張されます。
開示事項
Jochen Imigは現在、Pfizer Inc.がCGCIIIに資金提供しており、CGCIIIはPfizer Inc.、Merck KGaA、およびAstraZeneca PLCが後援しています。スポンサーは、研究のデザイン、実行、解釈、または執筆に何の役割も果たしていませんでした。すべての著者には、開示すべき利益相反はありません。
謝辞
Stefan Raunser氏(マックスプランク分子生理学研究所、ドルトムント)は、バイオセーフティレベル2のラボスペースへのアクセスに感謝します。Eric Wang氏の知的貢献と、貴重な議論をしてくださったImigラボの過去および現在のメンバー全員に感謝します。Jochen Imigは現在、CGC IIIでPfizer Inc.から資金提供を受けているCGCIIIです。
資料
| Name | Company | Catalog Number | Comments |
| Amicon Ultra Centrifugal Filter, 100 kDa MWCO | Millipore | UFC910024 | |
| anti-Cas9 (S. aureus) (6H4) mouse monoclonal antibody | Cell Signaling Technology | 48989 | |
| anti-Cas9 (S. pyogenes) (7A9-3A3) monoclonal antibody | Cell Signaling Technology | 14697 | |
| anti-GAPDH mouse monoclonal antibody | Sigma Aldrich | G8795 | |
| Anti-Mouse IgG (whole molecule)–Peroxidase antibody produced in rabbit | Sigma Aldrich | A9044 | |
| Bio-Rad ChemiDoc MP Imaging System | Bio-Rad | ||
| Blasticidine S hydrochloride | Sigma Aldrich | 15205-25MG | |
| CellTiter-Glo Luminescent Cell Viability Assay | Promega | G7570 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge | Eppendorf | 5415R | |
| CFX Connect Real-Time PCR Detection System | Bio-Rad | ||
| Clarity Western ECL Substrate, 500 mL | Bio-Rad | 1705061 | |
| CO2 Incubator Model CB 170 | Binder | ||
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Esp3I (BsmB1) restriction enzyme | Thermo Scientific | ER0451 | |
| Falcon 10 mL Serological Pipet | Corning | 356551 | |
| Falcon 25 mL Serological Pipet | Corning | 357525 | |
| Falcon 5 mL Serological Pipet | Corning | 356543 | |
| Fetal Bovine Serum | Sigma Aldrich | F9665-500ML | |
| Gibco Dulbecco’s modified Eagle’s medium (DMEM), high glucose, pyruvate | Gibco | 41966029 | |
| Human melanoma cell line 501-mel | was kindly provided by the Aifantis Lab (New York University) | RRID: CVCL_4633 | |
| Immobilon -P PVDF Membrane | Millipore | IPVH00010 | |
| Lenti-X 293T HEK cell line | Takara Bio | 632180 | |
| Mini Trans-Blot Cell system | Bio-Rad | ||
| Mini-PROTEAN Tetra Handcast System | Bio-Rad | ||
| NEBuilder HiFi DNA Assembly Reaction | New England Biolabs | E2621 | |
| Non-fat milk powder | Biomol | 54650 | |
| NuPAGE LDS Sample Buffer (4x) | Invitrogen | NP0008 | |
| One Shot Stbl Chemically Competent E. coli | Invitrogen | C737303 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985062 | |
| PCR Tubes 0.5 ml (Flat Cap) | VWR International | 732-3207 | |
| Phosphate-Buffered Saline (PBS) | Corning | 45000-446 | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | Thermo Scientific | F531L | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Polybrene | Sigma Aldrich | TR-1003-G | |
| Polyethylenimine, branched | Sigma Aldrich | 408727 | |
| Puromycin dihydrochloride | Santa Cruz Biotechnology | sc-108071A | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| SafeSeal Microcentrifuge Tube 1.5 mL | Sarstedt | 72,706 | |
| Sodium chloride, 5 M Aqua Solution, RNase Free | Alfa Aesar | J60434.AE | |
| Sodium Dodecyl Sulfate (SDS) | Sigma Aldrich | L3771 | |
| Syringe Filter PES 33mm 0.2 μM | Fisher Scientific | 15206869 | |
| TC Dish 100, Standard | Sarstedt | 8,33,902 | |
| TC Plate 6 Well, Standard, F | Sarstedt | 83,39,20,005 | |
| Tris base | Roche | 10708976001 | |
| TWEEN 20 | Sigma Aldrich | P9416-50ML |
参考文献
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Szcześniak, M. W., Wanowska, E., Mukherjee, N., Ohler, U., Makałowska, I. Towards a deeper annotation of human lncRNAs. Biochim Biophys Acta Gene Regul Mech. 1863 (4), 194385(2020).
- Fang, S., et al. NONCODEV5: a comprehensive annotation database for long non-coding RNAs. Nucl Acids Res. 46 (D1), D308-D314 (2018).
- Lin, J., et al. Human-specific lncRNAs contributed critically to human evolution by distinctly regulating gene expression. eLife. 12, RP89001.2(2023).
- Sarropoulos, I., Marin, R., Cardoso-Moreira, M., Kaessmann, H. Developmental dynamics of lncRNAs across mammalian organs and species. Nature. 571 (7766), 510-514 (2019).
- Melé, M., et al. Human genomics. The human transcriptome across tissues and individuals. Science. 348 (6235), 660-665 (2015).
- Cabili, M. N., et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev. 25 (18), 1915-1927 (2011).
- Liu, S. J., Dang, H. X., Lim, D. A., Feng, F. Y., Maher, C. A. Long noncoding RNAs in cancer metastasis. Nat Rev Cancer. 21 (7), 446-460 (2021).
- Ahmad, M., Weiswald, L. B., Poulain, L., Denoyelle, C., Meryet-Figuiere, M. Involvement of lncRNAs in cancer cells migration, invasion and metastasis: cytoskeleton and ECM crosstalk. J Exp Clin Cancer Res. 42 (1), 173(2023).
- Jain, A. K., et al. LncPRESS1 Is a p53-Regulated LncRNA that Safeguards Pluripotency by Disrupting SIRT6-Mediated De-acetylation of Histone H3K56. Mol Cell. 64 (5), 967-981 (2016).
- Bergmann, J. H., et al. Regulation of the ESC transcriptome by nuclear long noncoding RNAs. Genome Res. 25 (9), 1336-1346 (2015).
- Kretz, M., et al. Control of somatic tissue differentiation by the long non-coding RNA TINCR. Nature. 493 (7431), 231-235 (2013).
- Yoon, J. H., et al. LincRNA-p21 suppresses target mRNA translation. Mol Cell. 47 (4), 648-655 (2012).
- Huarte, M. The emerging role of lncRNAs in cancer. Nat Med. 21 (11), 1253-1261 (2015).
- Badowski, C., He, B., Garmire, L. X. Blood-derived lncRNAs as biomarkers for cancer diagnosis: the Good, the Bad and the Beauty. NPJ Precis Oncol. 6 (1), 40(2022).
- Zong, X., et al. Knockdown of nuclear-retained long noncoding RNAs using modified DNA antisense oligonucleotides. Meth Mol Biol. 1262, 321-331 (2015).
- Gagnon, K. T., Li, L., Chu, Y., Janowski, B. A., Corey, D. R. RNAi factors are present and active in human cell nuclei. Cell Rep. 6 (1), 211-221 (2014).
- Zeng, Y., Cullen, B. R. RNA interference in human cells is restricted to the cytoplasm. RNA. 8 (7), 855-860 (2002).
- Yang, J., et al. CRISPR/Cas9-mediated noncoding RNA editing in human cancers. RNA Biol. 15 (1), 35-43 (2018).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355 (6320), aah7111(2017).
- Tsung, K., et al. CRISPRi screen of long non-coding RNAs identifies LINC03045 regulating glioblastoma invasion. PLoS Genet. 20 (6), e1011314(2024).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Boettcher, M., McManus, M. T. Choosing the right tool for the job: RNAi, TALEN, or CRISPR. Mol Cell. 58 (4), 575-585 (2015).
- Yeo, N. C., et al. An enhanced CRISPR repressor for targeted mammalian gene regulation. Nat Meth. 15 (8), 611-616 (2018).
- Parrish, P. C. R., et al. Discovery of synthetic lethal and tumor suppressor paralog pairs in the human genome. Cell Rep. 36 (9), 109597(2021).
- Najm, F. J., et al. Orthologous CRISPR-Cas9 enzymes for combinatorial genetic screens. Nat Biotechnol. 36 (2), 179-189 (2018).
- Petroulia, S., et al. CRISPR-inhibition screen for lncRNAs linked to melanoma growth and metastasis. bioRxiv. , (2024).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- New England Biolabs. NEBuilder HiFi DNA Assembly Reaction (E2621) v2. , (2020).
- Cortés-Ríos, J., et al. Protein quantification by bicinchoninic acid (BCA) assay follows complex kinetics and can be performed at short incubation times. Anal Biochem. 608, 113904(2020).
- Junior, N. Polyacrylamide Gel Electrophoresis (SDS-PAGE) v1. , (2019).
- Chen, X., Liu, J., Janssen, J. M., Gonçalves, M. A. F. V. The chromatin structure differentially impacts high-specificity CRISPR-Cas9 Nuclease strategies. Mol Ther Nucl Acids. 8, 558-563 (2017).
- Schep, R., et al. Impact of chromatin context on Cas9-induced DNA double-strand break repair pathway balance. Mol Cell. 81 (10), 2216-2230.e10 (2021).
- Daer, R. M., Cutts, J. P., Brafman, D. A., Haynes, K. A. The impact of chromatin dynamics on Cas9-Mediated genome editing in human cells. ACS Syn Biol. 6 (3), 428-438 (2017).
- Chavez, M., Rane, D. A., Chen, X., Qi, L. S. Stable expression of large transgenes via the knock-in of an integrase-deficient lentivirus. Nat Biomed Eng. 7 (5), 661-671 (2023).
- Ellis, J. Silencing and variegation of gammaretrovirus and lentivirus vectors. Human Gene Ther. 16 (11), 1241-1246 (2005).
- Shrestha, D., et al. Genomics and epigenetics guided identification of tissue-specific genomic safe harbors. Genome Biol. 23 (1), 199(2022).
- Chu, Z., et al. Enhanced gene transfection and induction of apoptosis in melanoma cells by branched poly(β-amino ester)s with uniformly distributed branching units. J Control Release. 367, 197-208 (2024).
- Han, S. Y., et al. Nucleofection is a highly effective gene transfer techfnique for human melanoma cell lines. Exp Dermatol. 17 (5), 405-411 (2008).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved