Method Article
Kanser Hücrelerinde Kodlamayan RNA'lar Arasındaki Sentetik Ölümcül Etkileşimleri Hedeflemek için Çift CRISPR-Girişim Stratejisi
Bu Makalede
Özet
Bu çalışma, melanom hücrelerinde uzun kodlamayan RNA'ları hedefleyen ikili bir CRISPRi sistemi sunmaktadır. Potansiyel terapötik stratejiler için kansere özgü lncRNA etkileşimlerini belirleyerek kombinatoryal gen yıkımı ve sentetik ölümcül taramayı mümkün kılar.
Özet
Uzun kodlamayan RNA'lar (lncRNA'lar), insan genomunda 100.000'den fazla tahmin edilen, geniş ve işlevsel olarak çeşitli bir RNA molekülü sınıfını temsil eder. lncRNA'lar, protein kodlayan genlere kıyasla türler arasında daha az korunmuş olsalar da, gen regülasyonunda, kromatin etkileşimlerinde ve kanserin ilerlemesinde kritik roller oynarlar. Kansere katılımları onları umut verici terapötik hedefler haline getirir. KRAB-MeCP2 gibi bir transkripsiyonel baskılayıcı ile kaynaşmış katalitik olarak aktif olmayan Cas9 kullanan CRISPR girişimi (CRISPRi), nükleer lncRNA'ları hedeflemek ve işlevlerini değerlendirmek için kesin bir yöntem sunar. Bu çalışma, insan melanom hücrelerinde lncRNA'ların kombinatoryal hedeflemesi için optimize edilmiş, dCas9-KRAB-MeCP2'ye dayalı Staphylococcus aureus ve Streptococcus pyogenes'ten ortogonal CRISPRi teknolojilerini kullanan ikili bir CRISPRi sistemini tanıtmaktadır. Protokol, lncRNA çiftlerinin kombinatoryal gen yıkımını veya sentetik ölümcül taramasını kolaylaştırarak kanser araştırmaları için yeni bir araç sağlar. Bu yaklaşım, lncRNA'lar arasındaki sentetik ölümcüllüğü keşfederek, kanser hücresinin hayatta kalması için kritik olan lncRNA etkileşimlerini tanımlamaya yardımcı olabilir ve yeni terapötik stratejiler sunar. İkili sistemin işlevselliği gösterilmekte ve kritik kansere özgü lncRNA etkileşimlerini belirleme potansiyeli vurgulanmaktadır.
Giriş
İnsan genomunun %3'ünden daha azı proteinleri kodlamasına rağmen, genomun yaklaşık %80'i kopyalanır 1,2. Kodlamayan transkripsiyonel birimler arasında on binlercesi, 200 nükleotidi aşan uzun kodlamayan RNA'lar (lncRNA'lar) olarak sınıflandırılır ve toplam insan lncRNA sayısının 100.000'i aştığı tahmin edilmektedir 3,4. Kodlayan genlerin aksine, lncRNA'lar türler arasında daha az korunur. İnsanların genomlarının %99'unu şempanzeler gibi primatlarla paylaştığı göz önüne alındığında, lncRNA'ların fenotipik evrim üzerinde çok daha büyük bir etkiye sahip olduğu varsayılmaktadır 5,6. Bu bulgular lncRNA'ların önemli hücresel fonksiyonlarını göstermektedir. lncRNA'ların düzenlenmesi ve bunların RNA bağlayıcı proteinler ve diğer RNA'lar ile etkileşimleri tam olarak anlaşılmamış olmasına ve birçok lncRNA'nın henüz tam olarak açıklanmamış olmasına rağmen, lncRNA'ların sağlık ve hastalıkta hücreye ve dokuya özgü ekspresyon kalıpları sergilediği açıktır, kansergibi 7,8,9,10. Gen transkripsiyon regülasyonu, kromatin etkileşimlerine katılım11, RNA işleme12, RNA stabilizasyonu13 ve translasyonundüzenlenmesi 14 dahil olmak üzere çeşitli işlevlerde rol oynarlar.
Kanserde, çok çeşitli hücre tipine özgü lncRNA'lar, gen ekspresyonunu düzenleyerek tümör gelişimini ve metastazı etkiler ve değerli terapötik hedefler olarak potansiyellerini vurgular15. lncRNA'ların tümör örneklerinde biyobelirteç olarak saptanmasının ötesinde,16, tümöre özgü lncRNA'ların aşağı akış işlevlerini bozmak için hedeflenmesi hem klinik uygulamalarda hem de temel araştırmalarda önemli bir potansiyele sahiptir. lncRNA'ların rollerini aydınlatmak için RNA tabanlı yaklaşımlar arasında antisens oligonükleotidler (ASO'lar), kısa firkete RNA'ları ve küçük enterferans yapan RNA'lar (siRNA'lar) bulunur17,18. siRNA, gen susturma ekranları için yaygın olarak kullanılırken, siRNA bazlı yıkım sitoplazma19 ile sınırlıdır. Bununla birlikte, lncRNA sıklıkla çekirdek içinde çalışır.
Alternatif olarak, insan kanserlerinde lncRNA'ları inhibe etmek için Kümelenmiş Düzenli Aralıklı Kısa Palindromik Tekrarlar girişimi (CRISPRi) kullanılabilir20. Ayrıca, genom çapında CRISPRi ekranları kolayca programlanabilir ve işlevsel etkilerini incelemek için çok çeşitli kodlayan ve kodlamayan genleri hedefleyebilir21,22. CRISPRi'de katalitik eksik bir Cas9 (dCas9), Krüppel ile ilişkili kutu (KRAB) alanı23 gibi bir transkripsiyonel baskılayıcı alana kaynaştırılır. dCas9-KRAB tarafından gen baskılanması, ilgilenilen bölgeye bir kılavuz RNA (gRNA) tarafından yönlendirilir. CRISPRi, DNA seviyelerinde genleri kontrol eder, bu da transkripsiyon sonrası seviye24'te aktif olan RNA girişiminin aksine daha yüksek verimlilik ve istenen fonksiyon kaybı fenotiplerine yol açar. KRAB'ın hedef susturmadaki sınırlı etkinliğine yanıt olarak, daha etkili bir baskılama stratejisi olarak KRAB ve MeCP2'nin dCas9 ile füzyonu tanıtıldı25.
Tek gen susturma kanser canlılığını etkileyebilse de, sentetik ikili veya çoklu öldürücü etkileşimler kanser hücrelerini hücre ölümünden kurtarabilir26. Sentetik ölümcüllük, her biri diğerinin işlevini telafi edebilen iki veya daha fazla geni içerir. Tek gen yıkım ekranlarıyla ilgili sorunların üstesinden gelmek için, ölümcül protein kodlayan gen çiftlerini hedefleyen ikili CRISPR stratejileri umut verici bir yaklaşım sunmaktadır27.
Burada, insan melanom hücrelerinde KRAB-MeCP2'ye kaynaşmış Staphylococcus aureus ve Streptococcus pyogenes dCas9 kullanılarak lncRNA veya diğer kodlamayan RNA çiftlerinin ortogonal CRISPRi tabanlı hedeflemesinin kombinatoryal kullanımı için bir protokol sunuyoruz (bkz. Şekil 1). Protokol, kombinatoryal klasik CRISPR yıkımı için veya kanserde sentetik ölümcül çiftlerin CRISPRi tabanlı taraması için kullanılabilir. CRISPRi yaklaşımında iki farklı dCas9 türünün, SpCas9 ve SaCas9'un kullanılması, minimum çapraz reaktivite ile farklı genomik lokusların bağımsız olarak hedeflenmesine izin verir, özgüllüğü ve esnekliği artırırken yüksek hedef seçiciliği sağlar. Farklı protospacer bitişik motifler (PAM'ler) kullanılır: SpCas9 için NGG ve SaCas9 için NNGRRT. Çift dCas9 sistemi, tek bir dCas9 tipi kullanıldığında hassas, eşzamanlı modülasyon sağlayarak ve sgRNA-RNP karmaşık rekabetini azaltarak sınırlı sgRNA uyumluluğu gibi zorlukları ele alır. Bu yenilik, çift sgRNA kütüphane taramasının sağlamlığını ve çok yönlülüğünü artırır. Sonuç olarak, bir kanser modeli olarak melanom hücrelerinde tamamen işlevsel bir ikili CRISPRi sistemi sağlıyoruz.
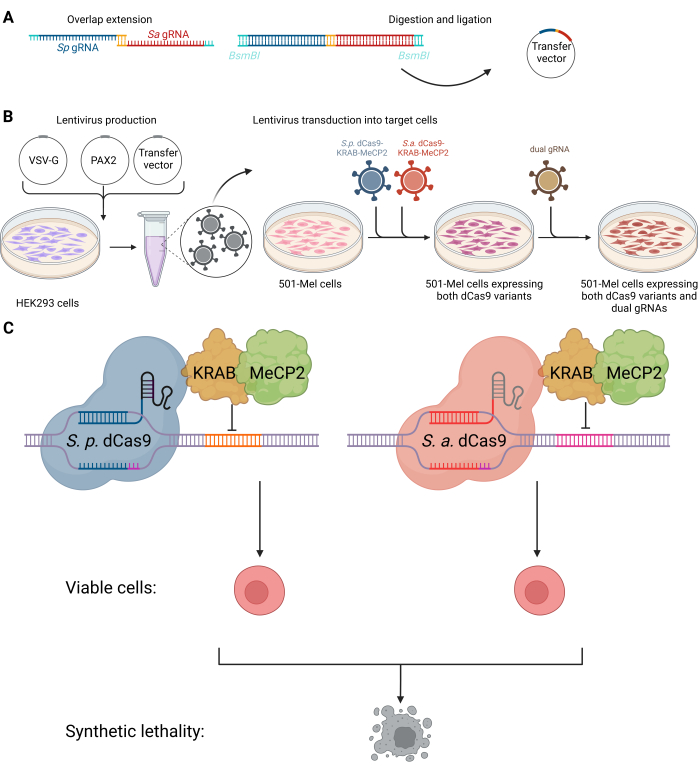
Şekil 1: Kodlamayan RNA'ların sentetik ölümcül etkileşimlerini hedeflemek için ikili CRISPRi sisteminin şeması. (A) Çift gRNA vektörünün klonlama prosedürü. (B) HEK293 hücreleri, zarf plazmidi VSV-G, paketleme plazmidi PAX2 ve transfer vektörü ile transfekte edildi ve daha sonra 501-mel hücrelerine transdüksiyon için lentivirüsler üretildi. SpdCas9-KRAB-MeCP2 (mavi) ve SadCas9-KRAB-MeCP2 (kırmızı) için genetik bilgi içeren lentivirüsler aynı anda 501-mel hücrelerine entegre edildi. Antibiyotik seçimini takiben, istenen ikili gRNA'ları (zeytin yeşili) entegre etmek için ikinci bir lentiviral transdüksiyon gerçekleştirildi. (C) dCas9 varyantlarını eksprese eden 501-mel hücreleri, hedef gen ekspresyonunu susturmak için karşılık gelen gRNA'ları ile etkileşime girerek kanser hücresi ölümüne neden olur. BioRender.com ile oluşturuldu. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protokol
İnsan melanom hücre hattı 501-mel (RRID: CVCL_4633), Aifantis Laboratuvarı (New York Üniversitesi) tarafından nazikçe sağlandı. Lenti-X 293T HEK hücre hattı Takara Bio'dan satın alındı. Bu hücre hatları, steril koşullar altında% 5 CO2 atmosferinde% 37 ° C'de% 10 fetal sığır serumu (FBS) ile desteklenmiş% 10 fetal sığır serumu (FBS) ile desteklenmiş Dulbecco'nun modifiye edilmiş Eagle's medium (DMEM) yüksek glikozunda kültürlendi. Hücreler %80-90 birleşim noktasına ulaşana kadar geçirildi. Hücre hatları mikoplazma kontaminasyonu için düzenli olarak test edildi.
1. Çevrimiçi bir gRNA tasarım platformu kullanarak gRNA'ların tasarlanması
- Hangi varyantın üstün nakavt sonuçları verdiğini değerlendirmek için her hedef lncRNA için SpCas9'a özgü bir gRNA ve SaCas9'a özgü bir gRNA tasarlayın.
- İstenen lncRNA'ları, burada RP11-120D5.1 (RP11) ve XLOC_030781 (XLOC), her biri için en verimli gRNA'yı kullanarak hedefleyin S. pyogenes (gRNA-sp) Cas9 ve S. aureus (gRNA-sa) Cas9.
NOT: S. pyogenes gRNA'ları Petroulia ve ark.28'den ekstrakte edilebilir. - gRNA tasarım araçlarının önerdiği şekilde hedef dışı eşleşmeleri en aza indirirken, yüksek hedef puanlarla ifade edilen en yüksek hedef etkinliğine öncelik vermek için sahaya özel önerileri izleyin.
- GRCh38 (hg, Homo sapiens) genomunu seçin. Belirli SpCas9 (NGG) ve SaCas9 (NNGRRT) için PAM sitesini seçin.
- Hsu ve ark.29'un hedef dışı puanlama yöntemini kullanın. Optimum performans için gRNA'ları transkripsiyonel başlangıç bölgesinin etrafında -150 bp ila +50 bp'lik bir pencere içinde konumlandırın.
- En yüksek hedef ve hedef dışı puana sahip gRNA'ları seçin (Ek Tablo 1'e bakınız).
- Bir lncRNA'yı hedefleyen ikili bir gRNA vektörü oluşturmak için burada Rosa26'ya karşı hedeflemeyen veya karıştırılmış bir kontrol gRNA'sını her hedef gRNA ile birleştirin. Hedef gRNA'ların bir plazmitte kombinasyonu, her iki hedef bölgenin bölgeye özgü modifikasyonunu sağlar.
NOT: Kontrol gRNA'sını ve hedef gRNA'yı barındıran ikili gRNA vektörü, bir hedef lncRNA'nın yıkılmasını sağlarken, bölgeleri hedefleyen gRNA'ları içeren çift gRNA vektörleri, hedeflenen lncRNA'ların aynı anda yıkılmasına izin verir.
2. gRNA klonlama
- gRNA dizisini içeren iki DNA parçası kullanarak bir örtüşme uzatma PCR gerçekleştirin. Najm ve ark.6 tarafından açıklandığı gibi amplifikasyon için U1 ileri astar ve H27 ters astarı (Ek Tablo 2'ye bakınız) kullanın.
- HF tamponlu 25 μL 2x Yüksek kaliteli PCR ana karışımı ve S. pyogenes veya S. aureus gRNA içeren her DNA fragmanından 1 μL ile 50 μL'lik bir PCR reaksiyonu ayarlayın. Reaksiyonu aşağıdaki döngü koşulları altında çalıştırın: 30 saniye boyunca 98 ° C'de ilk denatürasyon, ardından 5 saniye boyunca 98 ° C'de 15 döngü denatürasyon, 55 s boyunca 10 ° C'de tavlama ve 72 s boyunca 15 ° C'de uzama.
- İlk 15 döngüyü tamamladıktan sonra 2,5 μL U6 ileri astar ve 2,5 μL H1 ters astar ekleyin ve reaksiyonu aynı koşullar altında 20 döngüye daha tabi tutun ve 72 ° C'de 5 dakikalık bir son uzatma ile reaksiyonu uygulayın.
- PCR ürünlerini, üreticinin talimatlarına göre bir PCR saflaştırma kiti kullanarak saflaştırın.
- 10 μL'lik bir reaksiyonda 37 ° C'de 30 dakika boyunca 1 μL BsmB1 kısıtlama enzimi ve 1 μL 10x tampon kullanarak 1 μg modifiye plazmiti sindirin.
- PCR ürünlerini, diğerleri tarafından açıklandığı gibi bir HiFi DNA montaj reaksiyon protokolü kullanarak sindirilmiş çift gRNA-Zeo-GFP plazmidine klonlayın (Ek Şekil 1 ve Ek Dosya 1'e bakın)30.
- Üreticinin talimatlarına göre 50 μL bakteri kültürünü 4 μL montaj ürünü ile karıştırarak kimyasal olarak yetkin E. coli hücrelerini dönüştürün.
3. Lentivirus üretimi
DİKKAT: Uygun güvenlik yönergelerini takip ederken aktif lentivirüsleri tüm adımlarda kullanın. Kaçınılmaz aerosol oluşumundan kaynaklanan tehlikeleri önlemek için her zaman Sınıf 2 güvenlik kabininde virüslerle çalışın. Santrifüjleme gibi güvenlik kabini dışındaki tüm çalışmaları, Risk Grubu 2 organizmalarla kullanım için onaylanmış aerosol geçirmez kaplarda ve rotorlarda gerçekleştirin. Çalışma alanında her zaman laboratuvar önlüğü, tek kullanımlık eldivenler ve koruyucu gözlükler dahil olmak üzere uygun koruyucu giysiler giyin. Çalışma yüzeyleri bir dezenfektan solüsyonu ile muamele edilmeli ve diğer atıklar da bir dezenfektan solüsyonu ile muamele edilmeli ve en az 240 dakika UV ışığı altında kapalı kabinde kalmalarına izin verilmelidir. Kullanılan serolojik pipetler ve pipet uçları, otoklavlanabilir şişelere atılmadan önce dezenfektan bir solüsyonda durulanmalıdır. Daha sonra atıklar S2 çöp konteynerine atılmalıdır. S2 atığı otoklavlama yoluyla etkisiz hale getirilecektir. Transdüksiyonlu hücre hatları, transdüksiyondan en geç 2 gün sonra ve ancak kültür ortamının virüssüz bir ortamla en az iki tam değişikliğinden sonra S1 koşulları altında ele alınabilir.
- Bir hemositometrede 10 μL hücreyi 10 μL Tripan mavisi çözeltisi ile karıştırarak Lenti-X 293T HEK hücrelerini sayın. Plaka: Ertesi gün ~% 80 birleşme elde etmek için 10 cm plaka başına 4 x 106 Lenti-X 293T HEK hücresi.
- Bu adımdan itibaren Sınıf 2 laboratuvarda çalışır. 500 μL uygun bir transfeksiyon ortamını 11.25 μg transfer vektörü SpdCas9-KRAB-MeCP2 (bkz. Ek Dosya 2), 5.5 μg zarf plazmidi VSV-G ve 16.5 μg pPAX2 vektörü ile karıştırın. Oda sıcaklığında 5 dakika inkübe edin. İkinci dCas9 varyantı için, SadCas9-KRAB-MeCP2 transfer vektörünü kullanın (bkz. Ek Dosya 3).
- Ayrı bir tüpte, 500 μL indirgenmiş serum ortamını 36 μL PEI reaktifi ile karıştırın (stok: 1 mg / mL). Oda sıcaklığında 5 dakika inkübe edin.
- Karışımları birleştirin ve oda sıcaklığında 15 dakika inkübe edin. Karışımı dikkatlice hücre ortamına (6 mL) damla damla ekleyin ve gece boyunca 37 °C ve% 5 CO2'de inkübe edin. Lentiviral verimin azalmasını önlemek için hücrelerin bağlı kaldığından emin olun.
- Ortamı 12-15 saat sonra serolojik bir pipet kullanarak otoklavlanabilir bir atık şişesine atın. Şu andan itibaren, serolojik pipetler ve pipet uçları da dahil olmak üzere sarf malzemelerini otoklav atığına atmadan önce dezenfektan solüsyonla durulayın. Yeni bir pipet kullanarak transfekte edilmiş hücrelere 5 mL taze DMEM ortamı ekleyin.
- Transfeksiyondan 48 saat sonra serolojik bir pipet kullanarak süpernatanı 50 mL'lik bir kanonik tüpe aktararak lentivirüsleri hasat edin. Kanonik tüpü buzdolabında saklayın. Hücrelere 5 mL taze ortam ekleyin ve hücreleri 24 saat daha inkübe edin.
- Süpernatanı ilk hasadı içeren aynı 50 mL kanonik tüpe aktararak lentivirüslerin toplanmasını tekrarlayın. Yine, 5 mL taze ortam ekleyin, 24 saat inkübe edin ve süpernatanı daha sonra 50 mL kanonik tüpe aktarın. Birinci, ikinci ve üçüncü hasadın birleşik süpernatanlarını filtrasyon ve santrifüjlemeye kadar 50 mL kanonik tüpte 4 ° C'de saklayın.
- Kombine süpernatanı, ayrılmış hücreleri ve kalıntıları peletlemek için Risk Grubu 2 organizmalarla kullanım için onaylanmış aerosol geçirmez kaplarda ve rotorlarda 500 x g ve 4 °C'de 5 dakika santrifüjleyin. Süpernatanı 0.2 μm steril hücre kültürü dereceli şırınga filtrelerinden süzün.
- Filtrelenmiş süpernatanı, 100 kDa ila yaklaşık 500 μL arasında uygun bir moleküler ağırlık kesimine sahip bir santrifüj filtre ünitesi kullanarak 1.000 x g ve 4 ° C'de konsantre edin. 50-100 μL'lik virüs alikotlarını daha fazla kullanıma kadar -80 ° C'de saklayın.
4. Lentivirus transdüksiyonu ve stabil transfekte edilmiş hücrelerin seçimi
DİKKAT: Virüslerle her zaman Sınıf 2 güvenlik kabininde çalışın.
- Transdüksiyondan 1 gün önce, 2 mL'lik bir son kültür hacmi ile 2 x 105 hücre / kuyuda 6 oyuklu bir plakada plaka 501-mel hücreleri.
- Kültür ortamını, 6 μg / mL'lik bir nihai konsantrasyonda polibren ile desteklenmiş 2 mL önceden ısıtılmış DMEM ortamı ile değiştirin.
- Şu andan itibaren, Sınıf 2 laboratuvarında çalışmak gereklidir. Hücrelereaynı anda Sp dCas9-KRAB-MeCP2 ve SadCas9-KRAB-MeCP2 içeren 50 μL lentivirüs ekleyin. Serolojik pipetler ve pipet uçları da dahil olmak üzere sarf malzemelerini otoklav atığına atmadan önce dezenfektan solüsyonla durulayın. Plakayı hafifçe çalkalayın ve 37 °C'de% 5 CO2 atmosferinde inkübe edin.
- Ortamı serolojik bir pipet kullanarak otoklavlanabilir bir şişeye atın ve transdüksiyondan 16 saat sonra blasticidin (10 μg / mL) ve puromisin (2 μg / mL) içeren 2 mL taze ortam ekleyin.
- Ortamı 2 gün sonra blasticidin (10 μg/mL) ve puromisin (2 μg/mL) içeren taze 2 mL besiyeri ile değiştirerek seçime devam edin. Transdüksiyondan 7 gün sonra transdüksiyon hücreleri S1 koşulları altında ele alın.
NOT: Antibiyotik seçimi, transdüksiyona uğramamış tüm kontrol hücreleri ölene kadar yapılmalıdır. Transfer vektöründeki direnç kasetine bağlı olarak alternatif seçim stratejileri kullanılabilir. Gerekirse, bir öldürme eğrisi kullanarak antibiyotik konsantrasyonlarını optimize edin. Yeterli seviyelerde dCas9 füzyonlarını eksprese eden hücre popülasyonlarını elde etmek için akış sitometrisi veya seri seyreltme ve kaplama yoluyla ayrıntılı tek hücreli klon seçimi ve ardından protein miktar tayini gerekebilir.
5. Protein miktar tayini ve Western blot analizi
- Bir hemositometrede 10 μL hücreyi 10 μL Tripan Mavisi çözeltisi ile karıştırarak stabil transfekte edilmiş 501-mel hücrelerini sayın. Hücrelerin 1 x 106'sını 500 x g ve 4 ° C'de 5 dakika santrifüjleyin. Süpernatanı atın ve hücre peletini 1 mL buz gibi soğuk PBS'de yeniden süspanse edin.
- Santrifüjlemeyi tekrarlayın ve hücre peletini proteaz inhibitörleri içeren 50 μL RIPA tamponunda yeniden süspanse edin. Buz üzerinde 15 dakika inkübe edin ve 4 ° C'de 10 dakika boyunca 13.000 x g'da santrifüjleme ile hücre kalıntılarını temizleyin. Süpernatanı -20 °C'de saklayın veya başkaları tarafından tarif edildiği gibi protein miktar tayini için hemen kullanın31.
- 10 μg proteini 4 x LDS tamponu, 50 mM DTT ile karıştırın ve 20 μL'ye kadar damıtılmış su ile doldurun. Daha önce tarif edildiği gibi 1 saat boyunca 135 V'ta Tris/glisin bazlı SDS-PAGE kullanarak proteinleri %10'luk bir ayırma jeli üzerinde ayırın32. Proteinleri, ıslak tank transferi ile 90 dakika boyunca 90 V'ta bir PVDF membranına aktarın.
- Membranı oda sıcaklığında 180 rpm'de 1 saat boyunca bloke edici tamponda (TBS-T'de %5 yağsız süt) inkübe edin.
- Dahili kontrol olarak engelleme tamponuna 1: 10.000 anti-GAPDH fare monoklonal antikoru ekleyin. Birincil antikor anti-Cas9'u (S. pyogenes) veya anti-Cas9'u (S. aureus) bloke edici tamponda 1: 1.000 seyreltme olarak ekleyin ve hafifçe çalkalama ile gece boyunca 4 ° C'de inkübe edin.
- Membranı 3x TBST tamponu ile her biri 10 dakika yıkayın. İkincil anti-fare HRP antikoru (1: 10.000) ile oda sıcaklığında 1 saat inkübe edin.
- Her biri 10 dakika boyunca TBST tamponu ile 3 kez yıkayın. ECL Substrate ve bir görüntüleme sistemi kullanarak sinyalleri tespit edin.
6. Çift LncRNA baskılama canlılık deneyi
DİKKAT: Virüslerle her zaman Sınıf 2 güvenlik kabininde çalışın.
- Transdüksiyondan 1 gün önce 100 μL önceden ısıtılmış DMEM ortamında 96 oyuklu bir plakada transdüksiyondan 7 gün sonra, stabil transfekte edilmiş SadCas9-KRAB ve SpdCas9-KRAB içeren tohum 0.1 x 105.
- Transdüksiyon gününde, kültür ortamını, 6 μg / mL'lik bir nihai konsantrasyonda polibren ile desteklenmiş 100 μL önceden ısıtılmış DMEM ortamı ile değiştirin. Şu andan itibaren, Sınıf 2 laboratuvarında çalışmak gereklidir. Hücrelere uygun hacimde lentivirüs ekleyin.
NOT: Çift gRNA vektörünü içeren lentivirüslerin hacmi, lentivirüsleri titre ederek doğrusal bir eğri oluşturmak için GFP kullanılarak incelenmelidir. - Ortamı, transdüksiyondan 16 saat sonra 500 μg / mL Zeosin, 5 μg / mL blastisidin ve 1 μg / mL puromisin içeren taze ortamla değiştirin.
- Üreticinin talimatlarına göre bir hücre canlılığı testi kullanarak transdüksiyondan 5 gün sonra lüminesans tespiti gerçekleştirin.
Sonuçlar
SadCas9-KRAB-MeCP2 ve SpdCas9-KRAB-MeCP2'nin ekspresyon kasetleri, eşit miktarda lentivirüs ile transdüksiyon kullanılarak aynı anda 501-mel hücrelerine entegre edildi. Bu CRISPRi protokolü, dCas9-KRAB-MeCP2 varyantlarının ekspresyonuna dayandığından, Western blot analizi, protein seviyesindeki varlıklarını doğrulamak için idealdir. Pozitif transfekte edilmiş hücrelerin zenginleştirilmesini takiben, dCas9-KRAB-MeCP2 ortologlarının varlığını doğrulamak için Western blot analizi için örnekler toplandı. Başarılı transdüksiyondan sonra, SpdCas9-KRAB-MeCP2 füzyon proteinine (202 kDa) ve SadCas9-KRAB-MeCP2 füzyon proteinine (170 kDa) karşılık gelen tek bir bant beklenir ve Şekil 2A'da gösterildiği gibi, ilgili Cas9 ortologlarına özgü antikorlar kullanılarak doğrulanmıştır. Bununla birlikte, Western blot mümkün değilse, mRNA seviyesinde füzyon proteinlerini doğrulamak için qPCR kullanılabilir.
Fonksiyonel baskılayıcıları stabil bir şekilde eksprese eden 501-mel hücreleri oluşturulduktan sonra, bir çift gRNA vektörü kullanılarak ikinci lentiviral transdüksiyon gerçekleştirildi. RP11 ve XLOC'un daha önce kısa süreli metastatik melanom kültürlerinde yukarı regüle olduğu gösterilmişti ve bireysel CRISPRi hedeflemesi, proliferasyon ve hücre sağkalımı üzerindeki etkilerini gösterdi28. Bu nedenle, bu lncRNA çiftini, modifiye edilmiş hücrelerde kümülatif, kolayca gözlemlenebilir bir büyüme fenotipi öngörerek, kavram kanıtı sentetik öldürücü kodlamayan bir RNA kombinasyonu olarak değerlendirme için seçtik.
Canlılık deneyleri kullanarak kanser hücrelerinde ikili CRISPRi'nin etkilerini araştırmak için, hedeflenen her iki lncRNA'nın da etkili bir şekilde baskılanması gerekir. qPCR, transkript seviyelerini ölçerek bu baskıyı doğrulamak için en uygun yöntemdir. Geliştirilen ikili CRISPRi protokolünün işlevselliğini, RNA seviyelerini belirleyerek ve potansiyel olarak ölümcül RP11 ve XLOC çiftini hedeflerken hücre canlılığını inceleyerek değerlendirdik. Bu nedenle, hedef RNA seviyelerini düşürmek için RP11 ve XLOC'u hedefleyen farklı gRNA'lar tanıtıldı. RNA'nın baskılanması, SadCas9-KRAB-MeCP2 veya SpdCas9-KRAB-MeCP2 için hedefe özgü gRNA'lar kullanılarak araştırıldı, Rosa26'yı hedefleyen bir kontrol gRNA'sı ile birlikte ayrı ayrı ve kombinasyon halinde test edildi (bkz. Şekil 2B). XLOC veya RP11'i hedefleyen gRNA'lar ayrı ayrı kendi hedeflerini etkili bir şekilde bastırsa da, hem XLOC hem de RP11'in aynı anda yıkılması yalnızca XLOC-sa/RP11-sp çifti ile sağlandı.
Seçilen XLOC-sa/RP11-sp gRNA çifti ile 501-mel hücre hattında hem XLOC hem de RP11 RNA seviyeleri başarılı bir şekilde azaltıldığından, bir ikili lncRNA baskılama canlılık testi gerçekleştirildi (bkz. Şekil 2C). 5 gün sonra, hedef gRNA'lar ayrı ayrı kullanıldığında hücre canlılığı Rosa26 kontrolünün %81 ve %89'u idi. RP11-sp ve XLOC-sa'nın birleştirilmesi, tek başına RP11-sp'ye kıyasla daha önemli bir azalma ile sonuçlandı ve Rosa26 kontrolünün% 59'unda hücre canlılığı ile sonuçlandı.
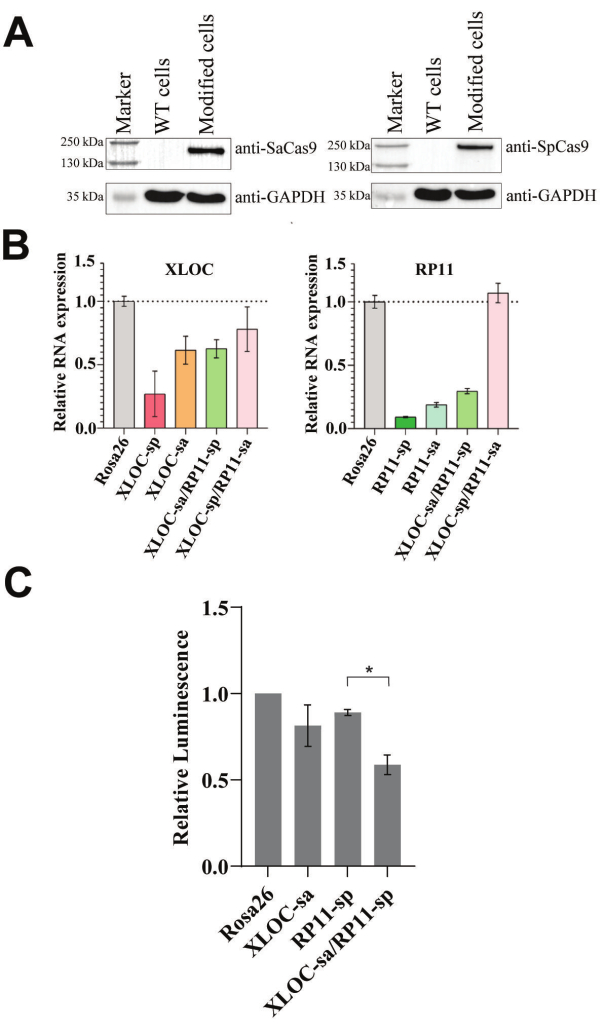
Şekil 2: Çift lncRNA baskılama sisteminin işlevselliği. (A) Anti-Cas9 antikorları kullanılarak modifiye edilmiş 501-mel hücrelerinde dCas9-KRAB-MeCP2 varyantlarını tespit etmek için Western blot analizi yapıldı. Transdüksiyonu olmayan 501-mel vahşi tip (WT) hücreler kontrol olarak kullanıldı (ham leke için Ek Şekil 3, Ek Şekil 4, Ek Şekil 5, Ek Şekil 6). (B) Çift gRNA'ların modifiye 501-mel hücrelerine transdüksiyonundan sonra Rosa26 kontrol hücrelerine göre RP11 ve XLOC RNA seviyelerinin qPCR tabanlı analizi. Temizlik geni olarak GAPDH kullanıldı. Veriler ortalama ± SD olarak ifade edilir. (C) İkili lncRNA baskılama canlılık testi, çift gRNA'ların üç bağımsız transdüksiyonda modifiye edilmiş 501-mel hücrelerine entegre edilmesinden sonra gerçekleştirildi. Lüminesans değerleri Rosa26 kontrolüne göre normalize edildi. Canlılık sonuçları, SEM ± ortalama (n = 3) olarak ifade edilir. Eşlenmemiş bir t-testi yapıldı. Yıldız işareti (*) p < 0.05'te anlamlı bir fark olduğunu gösterir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Ek Şekil 1: Plazmid haritası. Harita Benchling (https://benchling.com/) ile oluşturuldu. Protokolde kullanılan plazmitler Addgene #96921 ve #110824'den modifiye edildi. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Şekil 2: Ek Şekil 2: Western blot analizi
dCas9-KRAB ifadesi. (A) Transfeksiyondan 6 gün sonra 501-mel hücrelerinde SadCas9-KRAB ekspresyonu. (B) SadCas9-KRAB ve SpdCas9-KRAB'ın 501-mel hücrelerinde sıralı transdüksiyonu, transfeksiyondan 3 hafta sonra SadCas9-KRAB ekspresyonunun azalmasına yol açtı. (C) SadCas9-KRAB ve SpdCas9-KRAB'ın 501-mel hücrelerinde eşzamanlı transdüksiyonu, transfeksiyondan 3 hafta sonra SadCas9-KRAB ekspresyonunun azalmasına neden oldu. (D) SpdCas9-KRAB ekspresyonu, SadCas9-KRAB ve SpdCas9-KRAB'ın eş zamanlı transdüksiyonundan sonra gözlemlenebilir. LV. μL'de kullanılan lentivirüslerin hacmi. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Şekil 3: Western blot SaCas9-KRAB ham verileri. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Şekil 4: Western blot SpCas9-KRAB ham verileri. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Şekil 5: GAPDH_SpCas9-KRAB ham verileri. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Şekil 6: GAPDH_SaCas9-KRAB ham verileri. Bu Dosyayı indirmek için lütfen buraya tıklayın.
Ek Tablo 1: Kullanılan kılavuz RNA dizileri. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Tablo 2: Kullanılan DNA dizileri. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Dosya 1: dual-grna-zeo-gfp'nin FASTA dosyası. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Dosya 2: dSpCas9-krab-mecp2'nin FASTA dosyası. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Dosya 3: dSaCas9-krab-mecp2'nin FASTA dosyası. Bu dosyayı indirmek için lütfen buraya tıklayın.
Tartışmalar
Bu çalışmada, iki ortogonal türden dCas9-KRAB-MeCP2'ye dayalı CRISPRi kullanarak melanom hücrelerinde ikili bir lncRNA hedefleme stratejisi uyguladık. Bu sistemi kullanarak, sentetik öldürücü bir lncRNA çifti olan RP11 ve XLOC'u başarılı bir şekilde bastırdık ve bu da tek tek bastırıldıklarından farklı olarak hücre ölümünün artmasına neden oldu. Bununla birlikte, CRISPRi sistemleri, potansiyel olarak sonuç yorumlamasını etkileyen, hedef dışı gen baskısı riski taşır. Deneysel doğrulama yapılmamasına rağmen, in silico gRNA tasarımı sırasında potansiyel hedef dışı etkileri en aza indirdik.
İletim verimliliğindeki değişkenlik tekrarlanabilirliği etkileyebilir. Bu nedenle, değişen lentivirüs konsantrasyonları kullanılarak standart bir eğri kalibrasyonu ile belirlenen eşit miktarda lentivirüs uygulayarak iletimi optimize ettik. Bununla birlikte, yeni tasarlanan vektörün artan kargo boyutu, lentiviral paketleme verimliliğine sınırlamalar getirebilir ve özellikle iletilmesi zor hücre hatlarında iletim verimliliğini potansiyel olarak azaltabilir. Sonuç olarak, yeterli transdüksiyon ve sağlam dCas9 ekspresyonu elde etmek, bazı durumlarda yüksek viral titreler gerektirebilir. Ek olarak, dCas9-KRAB-MeCP2 protein seviyeleri ve hedeflenen lncRNA'ların RNA ekspresyonu sık sık izlenerek yetersiz yıkım önlenebilir. Alternatif olarak, bu sınırlamanın üstesinden gelmek için yalnızca bir baskılayıcı sisteme sahip dCas9 versiyonları mevcuttur, ancak düşük verimlilikmaliyeti 25 vardır.
Sadece XLOC-sa-RP11-sp kombinasyonu başarılı aktivite gösterirken, ters XLOC-sp-RP11-sa konfigürasyonu etkisizdi. Knockdown etkinliğindeki bu fark için makul bir açıklama, PAM özgüllüğü ve kromatin erişilebilirliği arasındaki etkileşimdir33. SaCas9 ve SpCas9'un farklı PAM dizilerini tanıdığı göz önüne alındığında, XLOC-sp-RP11-sa oryantasyonundaki SpCas9 için hedef bölge optimal bir PAM'den yoksun olabilir, böylece bağlanma afinitesini ve sonraki aktivitesini azaltır. Ayrıca, iki dCas9 füzyon proteini varyantının uzamsal düzenlemesi, kromatin mimarisini ve erişilebilirliği 34,35 etkileyebilir ve potansiyel olarak XLOC-sp-RP11-sa konfigürasyonunda transkripsiyonel mekanizmanın veya ilişkili düzenleyici faktörlerin işe alınmasını kısıtlayabilir. Bu bulgular, SaCas9 ve SpCas9'un belirli bir genomik bağlam içinde göreceli olarak konumlandırılmasının, optimize edilmiş CRISPR tabanlı modülasyon stratejileri için daha fazla araştırmayı garanti eden bir özellik olan fonksiyonel etkinliklerini kritik bir şekilde etkileyebileceğini göstermektedir.
Geliştirilen protokol, ikili gRNA vektörünü modifiye ederek diğer sentetik öldürücü kodlamayan RNA çiftlerini hedeflemek veya taramak için uyarlanabilir. Bir hücre ilgi hattı, her iki dCas9 varyantını da kararlı bir şekilde ifade ettiğinde, çeşitli yaklaşımlar uygulanabilir hale gelir. RNAi, ASO'lar ve shRNA'lar gen susturma için kullanılabilirken, CRISPRi daha basit bir tarama metodolojisi sunar ve küçük kimyasal bileşikler veya inhibitörlerle kombinasyonu da düşünülebilir. Bu protokol, ikili gRNA vektörünü bir çift gRNA kütüphanesi ile değiştirerek, kanser hücrelerinde sentetik öldürücü çiftlerin veya diğer genetik bağımlılıkların sistematik olarak taranmasına izin verir, böylece kanser hücresi hedeflemesinin hassasiyetini artırır.
Her ne kadar dCas9-KRAB-MeCP2 varyantlarının lentiviral transdüksiyon yoluyla entegrasyonunu sağlamış olsak da, istenen enzimleri 501-mel hücrelerine dahil etmek için alternatif stabil transfeksiyon yöntemleri de kullanılabilir. Bu özellikle önemlidir, çünkü deneyimlerimize göre, dCas9-KRAB-MeCP2 gibi büyük yapıların susturulması zamanla meydana gelebilir36,37. Alternatif olarak, dCas9-KRAB'ın daha küçük bir alternatif olduğu gösterilmiştir23. Deneyimlerimize göre, Cas9 ortologlarından türetilen daha küçük dCas9-KRAB füzyon proteinleri ile transdüksiyon, uzun süreli kültivasyon sırasında protein ekspresyonunun azalmasına yol açmıştır (bkz. Ek Şekil 2). Bu nedenle, tutarlı protein ekspresyonu, tekrarlayan zaman noktalarında düzenli olarak izlenmelidir. Ek olarak, iki büyük füzyon proteininin seçim belirteçleri ve çift gRNA ekspresyon kasetleri ile birlikte transdüksiyonu, hücresel stresi indükleyebilir veya lentiviral paketleme kapasitelerine ulaşabilir.
Çift CRISPRi'de %100 knockdown verimliliği elde etmek, yetersiz gRNA tasarımı veya yetersiz dCas9 ekspresyonu nedeniyle zor olabilir. Bunu ele almak için, en etkili dizileri belirlemek için hedef başına birden fazla gRNA, gelişmiş tasarım araçları kullanılarak test edilmelidir. Ek olarak, geliştirilmiş vektör tasarımı ve klonal hücre hatlarının karakterizasyonu yoluyla dCas9-KRAB-MeCP2 ekspresyonunun optimize edilmesi, knockdown verimliliğini artırabilir.
CRISPRi sistemini entegre etmek için alternatif stratejiler arasında, zaman içinde kararlı ekspresyonu sürdürmesiyle bilinen güvenli liman genomik bölgelerine kararlı bir şekilde yerleştirilmesini sağlamak için transpozaz vektörlerinin veya CRISPR'ye yönelik entegrasyonun kullanılması yer alır38. Bununla birlikte, melanom hücreleri de dahil olmak üzere birçok hücre tipinin39'u transfekte etmesi zordur ve bu da lentiviral transdüksiyonu tercih edilen yöntem haline getirir. Transpozaz veya Cas bazlı enzimlerin elektroporasyonu, dCas9-KRAB-MeCP2 yapılarını melanom hücrelerine40 entegre etmek için alternatif bir yol sunabilir. İndüklenebilir bir nakavt sisteminin uygulanması, hücresel stresi en aza indirmek için kurucu ekspresyon yerine dCas9-KRAB-MeCP2 ekspresyonunu gerektiği gibi aktive etmek için avantajlı olacaktır.
Son olarak, geliştirilen yaklaşım melanom hücreleriyle sınırlı değildir, ancak sentetik ölümcüllüğün araştırıldığı herhangi bir hücre tipine genişletilebilir. Bu yaklaşımın açıkça gRNA kütüphanesi tarama modalitesine doğru ölçeklendirilmesi amaçlanmıştır. Bununla birlikte, artan kombinatoryal karmaşıklık nedeniyle, örneğin, kontroller ve en az 300 x kapsama dahil olmak üzere gen çifti başına üçten fazla gRNA ile, eksprese edilen lncRNA'ların yalnızca bir alt transkriptomik fraksiyonu pratik olarak hedeflenebilir. Bu nedenle, kullanıcılar, kitaplık taramasına devam etmeden önce lncRNA ekspresyon seviyeleri ve bunların belirli hücresel bağlamlardaki bilinen alaka düzeyi gibi faktörleri göz önünde bulundurarak adayları dikkatli bir şekilde önceden seçmelidir. Bununla birlikte, bu yaklaşımın kombinatoryal çok yönlülüğü, onu çeşitli kanser türleri ve potansiyel olarak diğer hastalıklar arasında lncRNA'ların sentetik ölümcül etkileşimlerini ve diğer genetik bağımlılıklarını araştırmak için değerli bir araç haline getirir. Bu, hücrelerdeki bu ağ etkileşimlerini incelemek için şu anda mevcut olan araç setini genişletir.
Açıklamalar
Jochen Imig şu anda Pfizer Inc. tarafından finanse edilen CGCIII'dir. CGCIII, Pfizer Inc., Merck KGaA ve AstraZeneca PLC tarafından desteklenmektedir. Sponsorların çalışmanın tasarımında, yürütülmesinde, yorumlanmasında veya yazılmasında hiçbir rolü yoktu. Tüm yazarların açıklanacak herhangi bir çıkar çatışması yoktur.
Teşekkürler
Stefan Raunser'e (Max Planck Moleküler Fizyoloji Enstitüsü, Dortmund) Biyogüvenlik Seviye 2 laboratuvar alanına erişim için teşekkür ederiz. Entelektüel katkıları için Eric Wang'a ve değerli tartışmaları için Imig laboratuvarının tüm geçmiş ve şimdiki üyelerine özel teşekkürler. Jochen Imig şu anda CGC III'te Pfizer Inc. tarafından finanse edilen CGCIII'dir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Amicon Ultra Centrifugal Filter, 100 kDa MWCO | Millipore | UFC910024 | |
| anti-Cas9 (S. aureus) (6H4) mouse monoclonal antibody | Cell Signaling Technology | 48989 | |
| anti-Cas9 (S. pyogenes) (7A9-3A3) monoclonal antibody | Cell Signaling Technology | 14697 | |
| anti-GAPDH mouse monoclonal antibody | Sigma Aldrich | G8795 | |
| Anti-Mouse IgG (whole molecule)–Peroxidase antibody produced in rabbit | Sigma Aldrich | A9044 | |
| Bio-Rad ChemiDoc MP Imaging System | Bio-Rad | ||
| Blasticidine S hydrochloride | Sigma Aldrich | 15205-25MG | |
| CellTiter-Glo Luminescent Cell Viability Assay | Promega | G7570 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge | Eppendorf | 5415R | |
| CFX Connect Real-Time PCR Detection System | Bio-Rad | ||
| Clarity Western ECL Substrate, 500 mL | Bio-Rad | 1705061 | |
| CO2 Incubator Model CB 170 | Binder | ||
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Esp3I (BsmB1) restriction enzyme | Thermo Scientific | ER0451 | |
| Falcon 10 mL Serological Pipet | Corning | 356551 | |
| Falcon 25 mL Serological Pipet | Corning | 357525 | |
| Falcon 5 mL Serological Pipet | Corning | 356543 | |
| Fetal Bovine Serum | Sigma Aldrich | F9665-500ML | |
| Gibco Dulbecco’s modified Eagle’s medium (DMEM), high glucose, pyruvate | Gibco | 41966029 | |
| Human melanoma cell line 501-mel | was kindly provided by the Aifantis Lab (New York University) | RRID: CVCL_4633 | |
| Immobilon -P PVDF Membrane | Millipore | IPVH00010 | |
| Lenti-X 293T HEK cell line | Takara Bio | 632180 | |
| Mini Trans-Blot Cell system | Bio-Rad | ||
| Mini-PROTEAN Tetra Handcast System | Bio-Rad | ||
| NEBuilder HiFi DNA Assembly Reaction | New England Biolabs | E2621 | |
| Non-fat milk powder | Biomol | 54650 | |
| NuPAGE LDS Sample Buffer (4x) | Invitrogen | NP0008 | |
| One Shot Stbl Chemically Competent E. coli | Invitrogen | C737303 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985062 | |
| PCR Tubes 0.5 ml (Flat Cap) | VWR International | 732-3207 | |
| Phosphate-Buffered Saline (PBS) | Corning | 45000-446 | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | Thermo Scientific | F531L | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Polybrene | Sigma Aldrich | TR-1003-G | |
| Polyethylenimine, branched | Sigma Aldrich | 408727 | |
| Puromycin dihydrochloride | Santa Cruz Biotechnology | sc-108071A | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| SafeSeal Microcentrifuge Tube 1.5 mL | Sarstedt | 72,706 | |
| Sodium chloride, 5 M Aqua Solution, RNase Free | Alfa Aesar | J60434.AE | |
| Sodium Dodecyl Sulfate (SDS) | Sigma Aldrich | L3771 | |
| Syringe Filter PES 33mm 0.2 μM | Fisher Scientific | 15206869 | |
| TC Dish 100, Standard | Sarstedt | 8,33,902 | |
| TC Plate 6 Well, Standard, F | Sarstedt | 83,39,20,005 | |
| Tris base | Roche | 10708976001 | |
| TWEEN 20 | Sigma Aldrich | P9416-50ML |
Referanslar
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Szcześniak, M. W., Wanowska, E., Mukherjee, N., Ohler, U., Makałowska, I. Towards a deeper annotation of human lncRNAs. Biochim Biophys Acta Gene Regul Mech. 1863 (4), 194385(2020).
- Fang, S., et al. NONCODEV5: a comprehensive annotation database for long non-coding RNAs. Nucl Acids Res. 46 (D1), D308-D314 (2018).
- Lin, J., et al. Human-specific lncRNAs contributed critically to human evolution by distinctly regulating gene expression. eLife. 12, RP89001.2(2023).
- Sarropoulos, I., Marin, R., Cardoso-Moreira, M., Kaessmann, H. Developmental dynamics of lncRNAs across mammalian organs and species. Nature. 571 (7766), 510-514 (2019).
- Melé, M., et al. Human genomics. The human transcriptome across tissues and individuals. Science. 348 (6235), 660-665 (2015).
- Cabili, M. N., et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev. 25 (18), 1915-1927 (2011).
- Liu, S. J., Dang, H. X., Lim, D. A., Feng, F. Y., Maher, C. A. Long noncoding RNAs in cancer metastasis. Nat Rev Cancer. 21 (7), 446-460 (2021).
- Ahmad, M., Weiswald, L. B., Poulain, L., Denoyelle, C., Meryet-Figuiere, M. Involvement of lncRNAs in cancer cells migration, invasion and metastasis: cytoskeleton and ECM crosstalk. J Exp Clin Cancer Res. 42 (1), 173(2023).
- Jain, A. K., et al. LncPRESS1 Is a p53-Regulated LncRNA that Safeguards Pluripotency by Disrupting SIRT6-Mediated De-acetylation of Histone H3K56. Mol Cell. 64 (5), 967-981 (2016).
- Bergmann, J. H., et al. Regulation of the ESC transcriptome by nuclear long noncoding RNAs. Genome Res. 25 (9), 1336-1346 (2015).
- Kretz, M., et al. Control of somatic tissue differentiation by the long non-coding RNA TINCR. Nature. 493 (7431), 231-235 (2013).
- Yoon, J. H., et al. LincRNA-p21 suppresses target mRNA translation. Mol Cell. 47 (4), 648-655 (2012).
- Huarte, M. The emerging role of lncRNAs in cancer. Nat Med. 21 (11), 1253-1261 (2015).
- Badowski, C., He, B., Garmire, L. X. Blood-derived lncRNAs as biomarkers for cancer diagnosis: the Good, the Bad and the Beauty. NPJ Precis Oncol. 6 (1), 40(2022).
- Zong, X., et al. Knockdown of nuclear-retained long noncoding RNAs using modified DNA antisense oligonucleotides. Meth Mol Biol. 1262, 321-331 (2015).
- Gagnon, K. T., Li, L., Chu, Y., Janowski, B. A., Corey, D. R. RNAi factors are present and active in human cell nuclei. Cell Rep. 6 (1), 211-221 (2014).
- Zeng, Y., Cullen, B. R. RNA interference in human cells is restricted to the cytoplasm. RNA. 8 (7), 855-860 (2002).
- Yang, J., et al. CRISPR/Cas9-mediated noncoding RNA editing in human cancers. RNA Biol. 15 (1), 35-43 (2018).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355 (6320), aah7111(2017).
- Tsung, K., et al. CRISPRi screen of long non-coding RNAs identifies LINC03045 regulating glioblastoma invasion. PLoS Genet. 20 (6), e1011314(2024).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Boettcher, M., McManus, M. T. Choosing the right tool for the job: RNAi, TALEN, or CRISPR. Mol Cell. 58 (4), 575-585 (2015).
- Yeo, N. C., et al. An enhanced CRISPR repressor for targeted mammalian gene regulation. Nat Meth. 15 (8), 611-616 (2018).
- Parrish, P. C. R., et al. Discovery of synthetic lethal and tumor suppressor paralog pairs in the human genome. Cell Rep. 36 (9), 109597(2021).
- Najm, F. J., et al. Orthologous CRISPR-Cas9 enzymes for combinatorial genetic screens. Nat Biotechnol. 36 (2), 179-189 (2018).
- Petroulia, S., et al. CRISPR-inhibition screen for lncRNAs linked to melanoma growth and metastasis. bioRxiv. , (2024).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- New England Biolabs. NEBuilder HiFi DNA Assembly Reaction (E2621) v2. , (2020).
- Cortés-Ríos, J., et al. Protein quantification by bicinchoninic acid (BCA) assay follows complex kinetics and can be performed at short incubation times. Anal Biochem. 608, 113904(2020).
- Junior, N. Polyacrylamide Gel Electrophoresis (SDS-PAGE) v1. , (2019).
- Chen, X., Liu, J., Janssen, J. M., Gonçalves, M. A. F. V. The chromatin structure differentially impacts high-specificity CRISPR-Cas9 Nuclease strategies. Mol Ther Nucl Acids. 8, 558-563 (2017).
- Schep, R., et al. Impact of chromatin context on Cas9-induced DNA double-strand break repair pathway balance. Mol Cell. 81 (10), 2216-2230.e10 (2021).
- Daer, R. M., Cutts, J. P., Brafman, D. A., Haynes, K. A. The impact of chromatin dynamics on Cas9-Mediated genome editing in human cells. ACS Syn Biol. 6 (3), 428-438 (2017).
- Chavez, M., Rane, D. A., Chen, X., Qi, L. S. Stable expression of large transgenes via the knock-in of an integrase-deficient lentivirus. Nat Biomed Eng. 7 (5), 661-671 (2023).
- Ellis, J. Silencing and variegation of gammaretrovirus and lentivirus vectors. Human Gene Ther. 16 (11), 1241-1246 (2005).
- Shrestha, D., et al. Genomics and epigenetics guided identification of tissue-specific genomic safe harbors. Genome Biol. 23 (1), 199(2022).
- Chu, Z., et al. Enhanced gene transfection and induction of apoptosis in melanoma cells by branched poly(β-amino ester)s with uniformly distributed branching units. J Control Release. 367, 197-208 (2024).
- Han, S. Y., et al. Nucleofection is a highly effective gene transfer techfnique for human melanoma cell lines. Exp Dermatol. 17 (5), 405-411 (2008).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır