Method Article
Duale CRISPR-Interferenzstrategie zur gezielten gezielten Behandlung synthetischer letaler Wechselwirkungen zwischen nicht-kodierenden RNAs in Krebszellen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
In dieser Studie wird ein duales CRISPRi-System vorgestellt, das auf lange nicht-kodierende RNAs in Melanomzellen abzielt. Es ermöglicht den kombinatorischen Gen-Knockdown und das synthetische Letal-Screening, wodurch krebsspezifische lncRNA-Wechselwirkungen für potenzielle therapeutische Strategien identifiziert werden.
Zusammenfassung
Lange nicht-kodierende RNAs (lncRNAs) stellen eine große und funktionell vielfältige Klasse von RNA-Molekülen dar, von denen über 100.000 im menschlichen Genom vorhergesagt werden. Obwohl lncRNAs im Vergleich zu proteinkodierenden Genen über Spezies hinweg weniger konserviert sind, spielen sie eine entscheidende Rolle bei der Genregulation, den Chromatininteraktionen und dem Fortschreiten von Krebs. Ihre Beteiligung an Krebs macht sie zu vielversprechenden therapeutischen Angriffspunkten. Die CRISPR-Interferenz (CRISPRi), bei der katalytisch inaktives Cas9 verwendet wird, das mit einem transkriptionellen Repressor wie KRAB-MeCP2 fusioniert ist, bietet eine präzise Methode, um nukleäre lncRNAs ins Visier zu nehmen und ihre Funktionen zu bewerten. In dieser Studie wird ein duales CRISPRi-System vorgestellt, das orthogonale CRISPRi-Technologien aus Staphylococcus aureus und Streptococcus pyogenes auf Basis von dCas9-KRAB-MeCP2 verwendet und für das kombinatorische Targeting von lncRNAs in humanen Melanomzellen optimiert ist. Das Protokoll ermöglicht den kombinatorischen Gen-Knockdown oder das synthetische letale Screening von lncRNA-Paaren und stellt damit ein neuartiges Werkzeug für die Krebsforschung dar. Durch die Erforschung der synthetischen Letalität zwischen lncRNAs kann dieser Ansatz dazu beitragen, lncRNA-Wechselwirkungen zu identifizieren, die für das Überleben von Krebszellen entscheidend sind, und neue therapeutische Strategien bieten. Die Funktionalität des dualen Systems wird demonstriert und sein Potenzial bei der Identifizierung kritischer krebsspezifischer lncRNA-Wechselwirkungen unterstrichen.
Einleitung
Obwohl weniger als 3 % des menschlichen Genoms Proteine kodieren, werden etwa 80 % des Genoms transkribiert 1,2. Unter den nicht-kodierenden Transkriptionseinheiten werden Zehntausende als lange nicht-kodierende RNAs (lncRNAs) mit mehr als 200 Nukleotiden klassifiziert, und die Gesamtzahl der humanen lncRNAs wird auf über 100.000 geschätzt 3,4. Im Gegensatz zu kodierenden Genen sind lncRNAs bei allen Arten weniger konserviert. Angesichts der Tatsache, dass der Mensch 99% seines Genoms mit Primaten wie Schimpansen teilt, wird angenommen, dass lncRNAs einen viel größeren Einfluss auf die phänotypische Evolution haben 5,6. Diese Ergebnisse deuten auf wichtige zelluläre Funktionen von lncRNAs hin. Obwohl die Regulation von lncRNAs und ihre Wechselwirkungen mit RNA-bindenden Proteinen und anderen RNAs noch unvollständig verstanden sind und viele lncRNAs noch nicht vollständig annotiert sind, ist es klar, dass lncRNAs zell- und gewebespezifische Expressionsmuster bei Gesundheit und Krankheit aufweisen, wie z. B. Krebs 7,8,9,10. Sie sind an verschiedenen Funktionen beteiligt, darunter die Regulation der Gentranskription, die Beteiligung an Chromatininteraktionen11, die RNA-Verarbeitung12, die RNA-Stabilisierung13 und die Regulation der Translation14.
Bei Krebs beeinflussen sehr unterschiedliche zelltypspezifische lncRNAs die Tumorentwicklung und Metastasierung, indem sie die Genexpression regulieren, was ihr Potenzial als wertvolle therapeutische Ziele unterstreicht15. Neben dem Nachweis von lncRNAs als Biomarker in Tumorproben16 birgt die gezielte Störung ihrer nachgeschalteten Funktionen durch tumorspezifische lncRNAs sowohl in klinischen Anwendungen als auch in der Grundlagenforschung erhebliches Potenzial. RNA-basierte Ansätze zur Aufklärung der Rolle von lncRNAs umfassen Antisense-Oligonukleotide (ASOs), kurze Hairpin-RNAs und kleine interferierende RNAs (siRNAs)17,18. Während siRNA häufig für Gen-Silencing-Screens verwendet wird, ist der siRNA-basierte Knockdown auf das Zytoplasmabeschränkt 19. lncRNA arbeitet jedoch häufig im Zellkern.
Alternativ kann die Clustered Regularly Interspaced Short Palindromic Repeats Interference (CRISPRi) verwendet werden, um lncRNAs bei menschlichen Krebserkrankungenzu hemmen 20. Darüber hinaus können genomweite CRISPRi-Screens einfach programmiert werden und zielen auf ein breites Spektrum kodierender und nicht-kodierender Gene ab, um ihre funktionellen Auswirkungen zu untersuchen21,22. In CRISPRi wird ein katalytisch defizientes Cas9 (dCas9) mit einer transkriptionellen Repressordomäne, wie z.B. der Krüppel-associated box (KRAB) Domäne23, fusioniert. Die Genrepression durch dCas9-KRAB wird durch eine Leit-RNA (gRNA) in die interessierende Region geleitet. CRISPRi kontrolliert Gene auf DNA-Ebene, was zu einer höheren Effizienz und den gewünschten Loss-off-Funktionsphänotypen führt, im Gegensatz zur RNA-Interferenz, die auf der posttranskriptionellen Ebene aktiv ist24. Als Reaktion auf die begrenzte Wirksamkeit von KRAB beim Target-Silencing wurde eine Fusion von KRAB und MeCP2 mit dCas9 als effektivere Unterdrückungsstrategie eingeführt25.
Obwohl das Silencing einzelner Gene die Lebensfähigkeit von Krebs beeinträchtigen kann, können synthetische duale oder multiple letale Wechselwirkungen Krebszellen vor dem Zelltod retten26. Bei der synthetischen Letalität sind zwei oder mehr Gene beteiligt, von denen jedes die Funktion des anderen kompensieren kann. Um Probleme mit Einzelgen-Knockdown-Screenings zu überwinden, bieten duale CRISPR-Strategien, die auf letale proteinkodierende Genpaare abzielen, einen vielversprechenden Ansatz27.
In dieser Arbeit stellen wir ein Protokoll für die kombinatorische Verwendung von orthogonalem CRISPRi-basiertem Targeting von lncRNA oder anderen nicht-kodierenden RNA-Paaren unter Verwendung von Staphylococcus aureus und Streptococcus pyogenes dCas9 vor, die mit KRAB-MeCP2 in humanen Melanomzellen fusioniert sind (siehe Abbildung 1). Das Protokoll kann für den kombinatorischen klassischen CRISPR-Knockdown oder als CRISPRi-basiertes Screening von synthetischen Letalpaaren bei Krebs verwendet werden. Die Verwendung von zwei verschiedenen dCas9-Spezies, SpCas9 und SaCas9, im CRISPRi-Ansatz ermöglicht ein unabhängiges Targeting unterschiedlicher genomischer Loci mit minimaler Kreuzreaktivität, was die Spezifität und Flexibilität verbessert und gleichzeitig eine hohe On-Target-Selektivität gewährleistet. Es werden verschiedene Protospacer-benachbarte Motive (PAMs) verwendet: NGG für SpCas9 und NNGRRT für SaCas9. Das duale dCas9-System adressiert Herausforderungen wie die eingeschränkte sgRNA-Kompatibilität, indem es eine präzise, gleichzeitige Modulation ermöglicht und die Konkurrenz zwischen sgRNA-RNP-Komplexen reduziert, wenn ein einzelner Typ von dCas9 verwendet wird. Diese Innovation verbessert die Robustheit und Vielseitigkeit des Dual-sgRNA-Bibliotheksscreenings. Zusammenfassend lässt sich sagen, dass wir ein voll funktionsfähiges duales CRISPRi-System in Melanomzellen als Krebsmodell zur Verfügung stellen.
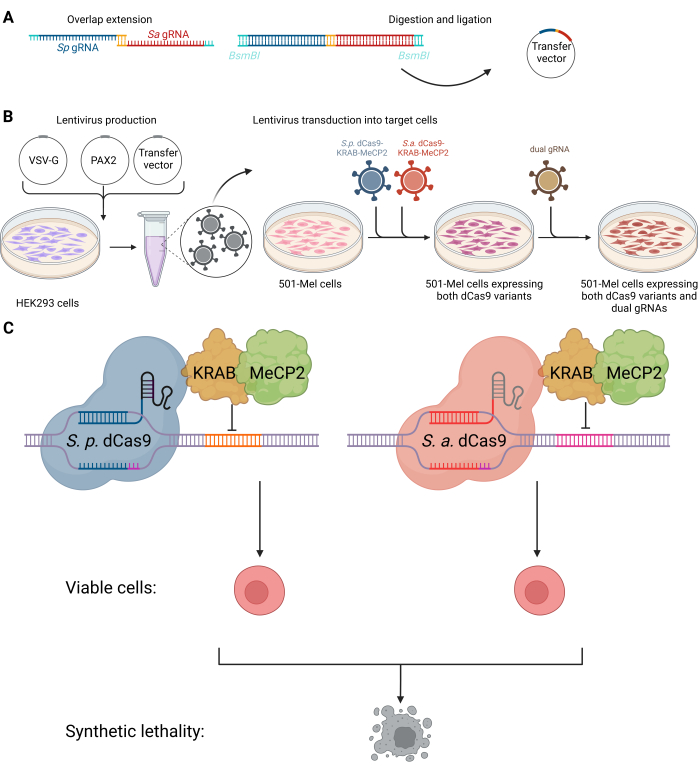
Abbildung 1: Schema des dualen CRISPRi-Systems zur gezielten Behandlung synthetischer letaler Wechselwirkungen von nicht-kodierenden RNAs. (A) Klonierungsverfahren des dualen gRNA-Vektors. (B) HEK293-Zellen wurden mit dem Hüllplasmid VSV-G, dem Verpackungsplasmid PAX2 und dem Transfervektor transfiziert, um Lentiviren für die anschließende Transduktion in 501-mel-Zellen zu produzieren. Lentiviren, die die genetische Information für SpdCas9-KRAB-MeCP2 (blau) und SadCas9-KRAB-MeCP2 (rot) enthalten, wurden gleichzeitig in 501-mel-Zellen eingebaut. Nach der Antibiotikaauswahl wurde eine zweite lentivirale Transduktion durchgeführt, um die gewünschten dualen gRNAs (olivgrün) zu integrieren. (C) 501-mel-Zellen, die die dCas9-Varianten exprimieren, interagieren mit ihren entsprechenden gRNAs, um die Expression des Zielgens zum Schweigen zu bringen, was zum Tod von Krebszellen führt. Erstellt mit BioRender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
Die humane Melanom-Zelllinie 501-mel (RRID: CVCL_4633) wurde freundlicherweise vom Aifantis Lab (New York University) zur Verfügung gestellt. Die Lenti-X 293T HEK-Zelllinie wurde von Takara Bio gekauft. Diese Zelllinien wurden in Dulbecco's modifiziertem Eagle's Medium (DMEM) hoher Glukose kultiviert, ergänzt mit 10 % fötalem Rinderserum (FBS) bei 37 °C in einer 5 % CO2 -Atmosphäre unter sterilen Bedingungen. Die Zellen wurden so lange durchgelassen, bis sie eine Konfluenz von 80 % bis 90 % erreichten. Die Zelllinien wurden regelmäßig auf Mykoplasmenkontamination untersucht.
1. Entwerfen von gRNAs mit einer Online-gRNA-Designplattform
- Entwerfen Sie eine gRNA, die spezifisch für SpCas9 ist, und eine gRNA, die spezifisch für SaCas9 für jede Ziel-lncRNA ist, um zu bewerten, welche Variante überlegene Knockdown-Ergebnisse liefert.
- Richten Sie sich an die gewünschten lncRNAs, hier RP11-120D5.1 (RP11) und XLOC_030781 (XLOC), wobei jeweils die effizienteste gRNA für S. pyogenes (gRNA-sp) Cas9 und S. aureus (gRNA-sa) Cas9 verwendet wird.
HINWEIS: Die S. pyogenes gRNAs können aus Petroulia et al.28 extrahiert werden. - Befolgen Sie standortspezifische Empfehlungen, um die höchste On-Target-Aktivität zu priorisieren, die sich in hohen On-Target-Werten ausdrückt, und gleichzeitig Off-Target-Übereinstimmungen zu minimieren, wie von gRNA-Designtools vorgeschlagen.
- Wählen Sie das Genom GRCh38 (hg, Homo sapiens). Wählen Sie den PAM-Standort für die spezifischen SpCas9 (NGG) und SaCas9 (NNGRRT) aus.
- Verwenden Sie die Off-Target-Scoring-Methode von Hsu et al.29. Positionieren Sie die gRNAs in einem Zeitfenster von -150 bp bis +50 bp um die transkriptionelle Startstelle, um eine optimale Leistung zu erzielen.
- Wählen Sie gRNAs mit dem höchsten On-Target- und Off-Target-Score (siehe Ergänzende Tabelle 1).
- Kombinieren Sie eine nicht zielgerichtete oder verschlüsselte Kontroll-gRNA, hier gegen Rosa26, mit jeder Ziel-gRNA, um einen dualen gRNA-Vektor zu erzeugen, der auf eine lncRNA abzielt. Die Kombination von Ziel-gRNAs in einem Plasmid ermöglicht eine ortsspezifische Modifikation beider Zielstellen.
HINWEIS: Der duale gRNA-Vektor, der Kontroll-gRNA und Ziel-gRNA enthält, ermöglicht den Knockdown einer Ziel-lncRNA, während duale gRNA-Vektoren, die gRNAs enthalten, die auf Stellen abzielen, den gleichzeitigen Knockdown von gezielten lncRNAs ermöglichen.
2. gRNA-Klonierung
- Führen Sie eine Überlappungsverlängerungs-PCR mit zwei DNA-Fragmenten durch, die die gRNA-Sequenz enthalten. Verwenden Sie den U6-Forward-Primer und den H1-Reverse-Primer (siehe Ergänzende Tabelle 2) zur Amplifikation, wie von Najm et al.27 beschrieben.
- Richten Sie eine 50-μl-PCR-Reaktion mit 25 μl 2x High-Fidelity-PCR-Mastermix mit HF-Puffer und 1 μl jedes DNA-Fragments ein, das entweder die S. pyogenes oder S. aureus gRNA enthält. Die Reaktion wird unter den folgenden Zyklusbedingungen durchgeführt: anfängliche Denaturierung bei 98 °C für 30 s, gefolgt von 15 Zyklen Denaturierung bei 98 °C für 5 s, Glühen bei 55 °C für 10 s und Dehnung bei 72 °C für 15 s.
- Nach Abschluss der ersten 15 Zyklen werden 2,5 μl U6-Forward-Primer und 2,5 μl H1-Reverse-Primer hinzugefügt und die Reaktion unter gleichen Bedingungen weiteren 20 Zyklen unterzogen, wobei die letzte Verlängerung um 5 Minuten bei 72 °C erfolgt.
- Reinigen Sie die PCR-Produkte mit einem PCR-Aufreinigungskit gemäß den Anweisungen des Herstellers.
- 1 μg des modifizierten Plasmids wird mit 1 μl BsmB1-Restriktionsenzym und 1 μl 10x Puffer für 30 min bei 37 ΰC in einer 10 μL-Reaktion verdaut.
- Klonieren Sie die PCR-Produkte in das verdaute dual-gRNA-Zeo-GFP-Plasmid (siehe Ergänzende Abbildung 1 und Ergänzende Datei 1) unter Verwendung eines HiFi-DNA-Assemblierungsreaktionsprotokolls, wie von anderenbeschrieben 30.
- Transformieren Sie chemisch kompetente E. coli-Zellen , indem Sie 50 μl Bakterienkultur mit 4 μl Montageprodukt gemäß den Anweisungen des Herstellers mischen.
3. Produktion von Lentiviren
VORSICHT: Gehen Sie bei allen Schritten mit aktiven Lentiviren um und befolgen Sie dabei die entsprechenden Sicherheitsrichtlinien. Arbeiten mit Viren führen Sie immer in einer Sicherheitswerkbank der Klasse 2 durch, um Gefahren durch unvermeidbare Aerosolbildung zu vermeiden. Führen Sie alle Arbeiten außerhalb der Sicherheitswerkbank, wie z. B. Zentrifugieren, in aerosoldichten Behältern und Rotoren durch, die für die Verwendung mit Organismen der Risikogruppe 2 zugelassen sind. Tragen Sie im Arbeitsbereich jederzeit geeignete Schutzkleidung, einschließlich eines Laborkittels, Einweghandschuhen und einer Schutzbrille. Die Arbeitsflächen müssen mit einer Desinfektionslösung behandelt werden, andere Abfälle müssen ebenfalls mit einer Desinfektionslösung behandelt werden, so dass sie mindestens 240 Minuten unter UV-Licht im geschlossenen Schrank verbleiben können. Gebrauchte serologische Pipetten und Pipettenspitzen müssen vor der Entsorgung in autoklavierbaren Flaschen in einer Desinfektionslösung gespült werden. Anschließend sollte der Abfall im Abfallbehälter S2 entsorgt werden. Der S2-Abfall wird durch Autoklavieren inaktiviert. Transduzierte Zelllinien können unter S1-Bedingungen frühestens 2 Tage nach der Transduktion und erst nach mindestens zwei vollständigen Wechseln des Kulturmediums mit einem virusfreien Medium gehandhabt werden.
- Zählen Sie Lenti-X 293T HEK-Zellen, indem Sie 10 μl Zellen mit 10 μl Trypanblau-Lösung in einem Hämozytometer mischen. Platte: 4 x 106 Lenti-X 293T HEK-Zellen pro 10 cm Platte, um am nächsten Tag eine Konfluenz von ~80% zu erreichen.
- Arbeiten Sie ab diesem Schritt in einem Labor der Klasse 2. Mischen Sie 500 μl eines geeigneten Transfektionsmediums mit dem 11,25 μg Transfervektor SpdCas9-KRAB-MeCP2 (siehe Zusatzdatei 2), dem 5,5 μg Hüllplasmid VSV-G und dem 16,5 μg pPAX2-Vektor. 5 Minuten bei Raumtemperatur inkubieren. Für die zweite dCas9-Variante verwenden Sie den Übertragungsvektor SadCas9-KRAB-MeCP2 (siehe Zusatzdatei 3).
- Mischen Sie in einem separaten Röhrchen 500 μl reduziertes Serummedium mit 36 μl PEI-Reagenz (Bestand: 1 mg/ml). 5 Minuten bei Raumtemperatur inkubieren.
- Mischen Sie die Mischungen und inkubieren Sie sie 15 Minuten lang bei Raumtemperatur. Die Mischung vorsichtig tropfenweise in das Zellmedium (6 ml) geben und über Nacht bei 37 °C und 5 % CO2 inkubieren. Stellen Sie sicher, dass die Zellen haften, um eine verringerte lentivirale Ausbeute zu vermeiden.
- Entsorgen Sie das Medium nach 12-15 Stunden mit einer serologischen Pipette in eine autoklavierbare Abfallflasche. Spülen Sie von nun an Verbrauchsmaterialien, einschließlich serologischer Pipetten und Pipettenspitzen, mit der Desinfektionslösung, bevor Sie sie im Autoklavenabfall entsorgen. Geben Sie 5 ml frisches DMEM-Medium mit einer neuen Pipette in die transfizierten Zellen.
- Ernten Sie Lentiviren, indem Sie den Überstand mit einer serologischen Pipette 48 h nach der Transfektion in ein kanonisches 50-ml-Röhrchen übertragen. Bewahren Sie die kanonische Tube im Kühlschrank auf. Geben Sie 5 ml frisches Medium in die Zellen und inkubieren Sie die Zellen für weitere 24 Stunden.
- Wiederholen Sie die Entnahme von Lentiviren, indem Sie den Überstand in dasselbe kanonische 50-ml-Röhrchen übertragen, das die erste Ernte enthält. Füge erneut 5 ml frisches Medium hinzu, inkubiere 24 Stunden lang und übertrage den Überstand anschließend in das kanonische 50-ml-Röhrchen. Lagern Sie kombinierte Überstände der ersten, zweiten und dritten Ernte im kanonischen 50-ml-Röhrchen bei 4 °C bis zur Filtration und Zentrifugation.
- Der kombinierte Überstand wird 5 Minuten lang bei 500 x g und 4 °C in aerosoldichten Behältern und Rotoren zentrifugiert, die für die Verwendung mit Organismen der Risikogruppe 2 zugelassen sind, um abgelöste Zellen und Ablagerungen zu pelletieren. Filtrieren Sie den Überstand durch 0,2 μm sterile Spritzenvorsatzfilter in Zellkulturqualität.
- Der filtrierte Überstand wird unter Verwendung einer Zentrifugalfiltereinheit mit einem geeigneten Molekulargewichtsgrenzwert von 100 kDa auf ca. 500 μl bei 1.000 μg und 4 ΰC konzentriert. Virusaliquote von 50-100 μl werden bis zur weiteren Verwendung bei -80 μC gelagert.
4. Lentivirus-Transduktion und Selektion stabiler transfizierter Zellen
ACHTUNG: Arbeiten mit Viren immer in einer Sicherheitswerkbank der Klasse 2 durchführen.
- 501-mel-Zellen in einer 6-Well-Platte bei 2 x 105 Zellen/Well mit einem endgültigen Kulturvolumen von 2 ml, 1 Tag vor der Transduktion plattieren.
- Ersetzen Sie das Nährmedium durch 2 mL vorgewärmtes DMEM-Medium, das mit Polybren in einer Endkonzentration von 6 μg/ml versetzt wurde.
- Von nun an ist die Arbeit im Labor der Klasse 2 erforderlich. Geben Sie gleichzeitig 50 μl Lentiviren, die SpdCas9-KRAB-MeCP2 und SadCas9-KRAB-MeCP2 enthalten, in die Zellen. Spülen Sie Verbrauchsmaterialien, einschließlich serologischer Pipetten und Pipettenspitzen, mit der Desinfektionslösung, bevor Sie sie im Autoklavenabfall entsorgen. Die Platte leicht schütteln und bei 37 °C in einer Atmosphäre von 5 % CO2 inkubieren.
- Entsorgen Sie das Medium mit einer serologischen Pipette in eine autoklavierbare Flasche und fügen Sie 16 Stunden nach der Transduktion 2 ml frisches Medium hinzu, das Blasticidin (10 μg/ml) und Puromycin (2 μg/ml) enthält.
- Setzen Sie die Auswahl fort, indem Sie das Medium nach 2 Tagen durch frisches 2-ml-Medium ersetzen, das Blasticidin (10 μg/ml) und Puromycin (2 μg/ml) enthält. Behandeln Sie transduzierte Zellen unter S1-Bedingungen nach 7 Tagen nach der Transduktion.
HINWEIS: Die Antibiotikaauswahl muss so lange durchgeführt werden, bis alle nicht transduzierten Kontrollzellen abgestorben sind. Abhängig von der Widerstandskassette im Transfervektor können alternative Selektionsstrategien verwendet werden. Optimieren Sie bei Bedarf die Antibiotikakonzentrationen anhand einer Abtötungskurve. Eine aufwendige Einzelzell-Klonauswahl durch Durchflusszytometrie oder serielle Verdünnung und Plating, gefolgt von einer Proteinquantifizierung, kann erforderlich sein, um Zellpopulationen zu erhalten, die dCas9-Fusionen in ausreichendem Maße exprimieren.
5. Proteinquantifizierung und Western-Blot-Analyse
- Zählen Sie stabile transfizierte 501-Mel-Zellen, indem Sie 10 μl Zellen mit 10 μl Trypanblau-Lösung in einem Hämozytometer mischen. 1 x 106 der Zellen für 5 min bei 500 x g und 4 °C zentrifugieren. Entsorgen Sie den Überstand und resuspendieren Sie das Zellpellet in 1 ml eiskaltem PBS.
- Wiederholen Sie die Zentrifugation und resuspendieren Sie das Zellpellet in 50 μl RIPA-Puffer, der Proteaseinhibitoren enthält. 15 min auf Eis inkubieren und Zelltrümmer durch Zentrifugation bei 13.000 x g für 10 min bei 4 °C entfernen. Der Überstand ist bei -20 °C zu lagern oder sofort zur Proteinquantifizierung zu verwenden, wie von anderen beschrieben31.
- 10 μg Protein mit 4 x LDS-Puffer, 50 mM DTT mischen und bis zu 20 μL mit destilliertem Wasser auffüllen. Trennen Sie die Proteine auf einem 10%igen Trenngel unter Verwendung von Tris/Glycin-basiertem SDS-PAGE bei 135 V für 1 h, wie zuvor beschrieben32. Übertragen Sie Proteine auf eine PVDF-Membran bei 90 V für 90 min mit einem Nasstanktransfer.
- Inkubieren Sie die Membran in Blockierungspuffer (5 % fettfreie Milch in TBS-T) für 1 h bei 180 U/min bei Raumtemperatur.
- Geben Sie 1:10.000 monoklonale Anti-GAPDH-Antikörper für die Maus als interne Kontrolle in den Blockierungspuffer. Den Primärantikörper anti-Cas9 (S. pyogenes) oder anti-Cas9 (S. aureus) in einer Verdünnung von 1:1.000 in den Blockierungspuffer geben und über Nacht bei 4 °C unter leichtem Schütteln inkubieren.
- Waschen Sie die Membran 3x mit TBST-Puffer für jeweils 10 min. Inkubieren Sie mit einem sekundären Anti-Maus-HRP-Antikörper (1:10.000) für 1 h bei Raumtemperatur.
- 3x mit TBST-Puffer für je 10 min waschen. Erkennen Sie Signale mit einem ECL-Substrat und einem Bildgebungssystem.
6. Dualer LncRNA-Repressions-Viabilitätsassay
ACHTUNG: Arbeiten mit Viren immer in einer Sicherheitswerkbank der Klasse 2 durchführen.
- Seed 0,1 x 105 Zellen mit stabilem transfiziertem SadCas9-KRAB und SpdCas9-KRAB 7 Tage nach der Transduktion in einer 96-Well-Platte in 100 μl vorgewärmtem Medium 1 Tag vor der Transduktion.
- Am Tag der Transduktion wird das Nährmedium durch 100 μl vorgewärmtes DMEM-Medium ersetzt, das mit Polybren in einer Endkonzentration von 6 μg/ml versetzt wurde. Von nun an ist die Arbeit im Labor der Klasse 2 erforderlich. Geben Sie das entsprechende Volumen an Lentiviren in die Zellen.
HINWEIS: Das Volumen von Lentiviren, die den dualen gRNA-Vektor enthalten, muss mit GFP untersucht werden, um durch Titration von Lentiviren eine lineare Kurve zu erzeugen. - Ersetzen Sie das Medium 16 h nach der Transduktion durch ein frisches Medium mit 500 μg/ml Zeocin, 5 μg/ml Blasticidin und 1 μg/ml Puromycin.
- Führen Sie die Lumineszenzdetektion 5 Tage nach der Transduktion mit einem Zellviabilitätsassay gemäß den Anweisungen des Herstellers durch.
Ergebnisse
Die Expressionskassetten von SadCas9-KRAB-MeCP2 und SpdCas9-KRAB-MeCP2 wurden gleichzeitig mittels Transduktion mit gleichen Mengen an Lentiviren in 501-mel-Zellen integriert. Da dieses CRISPRi-Protokoll auf der Expression von dCas9-KRAB-MeCP2-Varianten beruht, ist die Western-Blot-Analyse ideal, um deren Vorhandensein auf Proteinebene zu bestätigen. Nach der Anreicherung der positiv transfizierten Zellen wurden Proben für die Western-Blot-Analyse entnommen, um das Vorhandensein der dCas9-KRAB-MeCP2-Orthologe zu bestätigen. Nach erfolgreicher Transduktion wird eine einzelne Bande erwartet, die dem SpdCas9-KRAB-MeCP2-Fusionsprotein (202 kDa) und dem SadCas9-KRAB-MeCP2-Fusionsprotein (170 kDa) entspricht, und wurde, wie in Abbildung 2A gezeigt, unter Verwendung von Antikörpern bestätigt, die für die jeweiligen Cas9-Orthologe spezifisch sind. Wenn ein Western Blot jedoch nicht möglich ist, kann die qPCR verwendet werden, um die Fusionsproteine auf mRNA-Ebene zu verifizieren.
Nachdem 501-Mel-Zellen erzeugt worden waren, die die funktionellen Repressoren stabil exprimierten, wurde eine zweite lentivirale Transduktion unter Verwendung eines dualen gRNA-Vektors durchgeführt. Es wurde bereits gezeigt, dass RP11 und XLOC in Kurzzeitkulturen von metastasiertem Melanom hochreguliert sind, und individuelles CRISPRi-Targeting zeigte ihren Einfluss auf die Proliferation und das Zellüberleben28. Daher wählten wir dieses lncRNA-Paar für die Evaluierung als Proof-of-Concept-Kombination aus synthetischer letaler nicht-kodierender RNA aus, wobei wir einen kumulativen, leicht beobachtbaren Wachstumsphänotyp in den modifizierten Zellen erwarteten.
Um die Auswirkungen von dualem CRISPRi auf Krebszellen mit Hilfe von Viabilitätsassays zu untersuchen, müssen beide zielgerichteten lncRNAs effektiv unterdrückt werden. Die qPCR ist die am besten geeignete Methode, um diese Repression durch Messung der Transkriptspiegel zu bestätigen. Wir haben die Funktionalität des entwickelten dualen CRISPRi-Protokolls evaluiert, indem wir die RNA-Spiegel bestimmt und die Zellviabilität untersucht haben, wenn das potenziell letale RP11- und XLOC-Paar angegriffen wird. Daher wurden verschiedene gRNAs, die auf RP11 und XLOC abzielen, eingeführt, um die Ziel-RNA-Spiegel zu reduzieren. Die Repression von RNA wurde unter Verwendung von zielspezifischen gRNAs für SadCas9-KRAB-MeCP2 oder SpdCas9-KRAB-MeCP2 untersucht, die einzeln und in Kombination mit einer Kontroll-gRNA getestet wurden, die auf Rosa26 abzielt (siehe Abbildung 2B). Obwohl gRNAs, die individuell auf XLOC oder RP11 abzielen, ihre jeweiligen Ziele effektiv unterdrückten, wurde ein gleichzeitiger Knockdown von XLOC und RP11 nur mit dem XLOC-sa/RP11-sp-Paar erreicht.
Da sowohl die XLOC- als auch die RP11-RNA-Spiegel in der 501-Mel-Zelllinie mit dem ausgewählten XLOC-sa/RP11-sp-gRNA-Paar erfolgreich reduziert werden konnten, wurde ein dualer lncRNA-Repressions-Viabilitätstest durchgeführt (siehe Abbildung 2C). Nach 5 Tagen betrug die Zellviabilität 81 % und 89 % der Rosa26-Kontrolle, wenn die Ziel-gRNAs einzeln verwendet wurden. Die Kombination von RP11-sp und XLOC-sa führte zu einer weiteren signifikanten Reduktion im Vergleich zu RP11-sp allein, wobei die Zellviabilität bei 59% der Rosa26-Kontrolle lag.
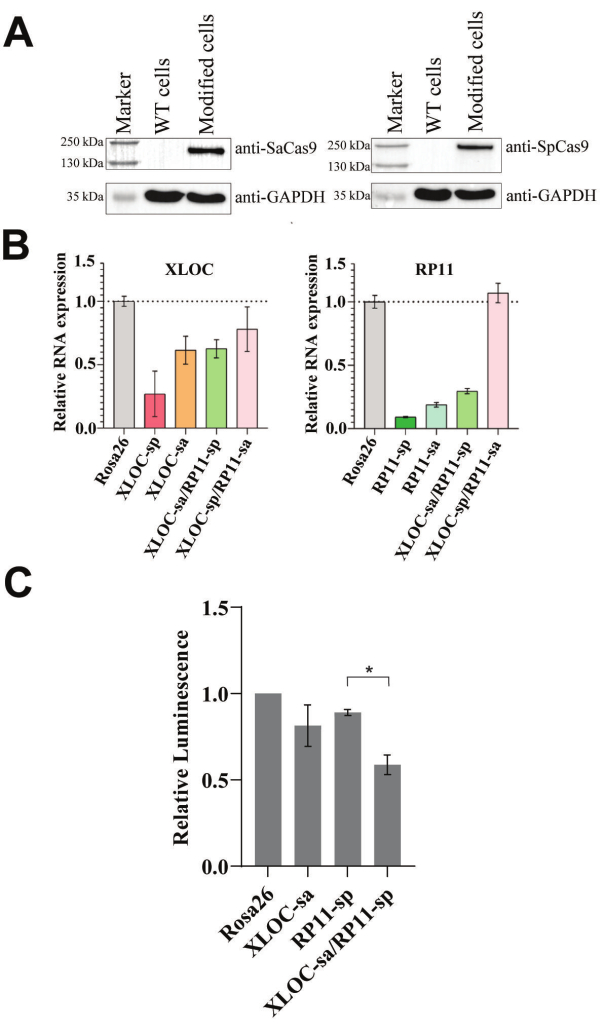
Abbildung 2: Funktionsweise des dualen lncRNA-Repressionssystems. (A) Die Western-Blot-Analyse wurde durchgeführt, um die dCas9-KRAB-MeCP2-Varianten in modifizierten 501-mel-Zellen mit anti-Cas9-Antikörpern nachzuweisen. Als Kontrolle wurden 501-Mel-Wildtyp-Zellen (WT) ohne Transduktion verwendet (siehe Ergänzende Abbildung 3, Ergänzende Abbildung 4, Ergänzende Abbildung 5, Ergänzende Abbildung 6 für den Roh-Blot). (B) qPCR-basierte Analyse von RP11- und XLOC-RNA-Spiegeln im Vergleich zu Rosa26-Kontrollzellen nach der Transduktion von dualen gRNAs in modifizierte 501-mel-Zellen. GAPDH wurde als Haushalts-Gen verwendet. Die Daten sind als Mittelwert ± SD ausgedrückt. (C) Der duale lncRNA-Repressions-Viabilitätsassay wurde nach Integration von dualen gRNAs in modifizierte 501-mel-Zellen in drei unabhängigen Transduktionen durchgeführt. Die Lumineszenzwerte wurden auf die Rosa26-Kontrolle normiert. Die Viabilitätsergebnisse werden als Mittelwert (n = 3) ± SEM ausgedrückt. Es wurde ein ungepaarter t-Test durchgeführt. Ein Sternchen (*) zeigt eine signifikante Differenz bei p < 0,05 an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1: Plasmidkarte. Die Karte wurde mit Benchling (https://benchling.com/) erstellt. Die im Protokoll verwendeten Plasmide wurden aus Addgene #96921 und #110824 modifiziert. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 2: Ergänzende Abbildung 2: Western-Blot-Analyse von
dCas9-KRAB-Ausdruck. (A) SadCas9-KRAB-Expression in 501-Mel-Zellen 6 Tage nach der Transfektion. (B) Die sequentielle Transduktion von SadCas9-KRAB und SpdCas9-KRAB in 501-mel-Zellen führte 3 Wochen nach der Transfektion zu einer verminderten SadCas9-KRAB-Expression. (C) Die gleichzeitige Transduktion von SadCas9-KRAB und SpdCas9-KRAB in 501-Mel-Zellen führte 3 Wochen nach der Transfektion zu einer verminderten SadCas9-KRAB-Expression. (D) Die SpdCas9-KRAB-Expression konnte nach gleichzeitiger Transduktion von SadCas9-KRAB und SpdCas9-KRAB beobachtet werden. LV. Die Menge der verwendeten Lentiviren in μL. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 3: Western-Blot-SaCas9-KRAB-Rohdaten. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 4: Western-Blot-SpCas9-KRAB-Rohdaten. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 5: GAPDH_SpCas9-KRAB-Rohdaten. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 6: GAPDH_SaCas9-KRAB-Rohdaten. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle 1: Verwendete Guide-RNA-Sequenzen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle 2: Verwendete DNA-Sequenzen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 1: FASTA-Datei von dual-grna-zeo-gfp. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 2: FASTA-Datei von dSpCas9-krab-mecp2. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 3: FASTA-Datei von dSaCas9-krab-mecp2. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
In dieser Studie haben wir eine duale lncRNA-Targeting-Strategie in Melanomzellen mit CRISPRi implementiert, die auf dCas9-KRAB-MeCP2 von zwei orthogonalen Spezies basiert. Mit diesem System konnten wir erfolgreich ein synthetisches letales Paar von lncRNAs, RP11 und XLOC, repressieren, was zu einem erhöhten Zelltod führte, im Gegensatz zu der Unterdrückung einzeln. CRISPRi-Systeme bergen jedoch das Risiko einer Off-Target-Genrepression, die sich möglicherweise auf die Interpretation der Ergebnisse auswirkt. Obwohl keine experimentelle Validierung durchgeführt wurde, minimierten wir mögliche Off-Target-Effekte während des gRNA-Designs in silico.
Die Variabilität der Transduktionseffizienz kann sich auf die Reproduzierbarkeit auswirken. Daher optimierten wir die Transduktion durch die Anwendung gleicher Mengen an Lentiviren, die durch eine Standardkurvenkalibrierung mit unterschiedlichen Lentivirus-Konzentrationen bestimmt wurden. Die erhöhte Ladungsgröße des neu entwickelten Vektors kann jedoch die Effizienz der lentiviralen Verpackung einschränken, was die Transduktionseffizienz möglicherweise verringert, insbesondere bei schwer zu transduzierenden Zelllinien. Infolgedessen kann das Erreichen einer ausreichenden Transduktion und einer robusten dCas9-Expression in bestimmten Fällen hohe Virustiter erfordern. Darüber hinaus kann ein unzureichender Knockdown durch häufige Überwachung der dCas9-KRAB-MeCP2-Proteinspiegel und der RNA-Expression von Ziel-lncRNAs umgangen werden. Alternativ gibt es dCas9-Versionen mit nur einem Repressorsystem, um diese Einschränkung zu überwinden, jedoch auf Kosten einer verringerten Effizienz25.
Nur die Kombination XLOC-sa-RP11-sp zeigte eine erfolgreiche Aktivität, während die umgekehrte XLOC-sp-RP11-sa-Konfiguration ineffektiv war. Eine plausible Erklärung für diesen Unterschied in der Knockdown-Wirksamkeit ist das Zusammenspiel zwischen PAM-Spezifität und Chromatinzugänglichkeit33. Angesichts der Tatsache, dass SaCas9 und SpCas9 unterschiedliche PAM-Sequenzen erkennen, könnte die Zielstelle für SpCas9 in der XLOC-sp-RP11-sa-Orientierung kein optimales PAM aufweisen, wodurch ihre Bindungsaffinität und nachfolgende Aktivität reduziert wird. Darüber hinaus könnte die räumliche Anordnung der beiden dCas9-Fusionsproteinvarianten die Chromatinarchitektur und Zugänglichkeitbeeinflussen 34,35, was möglicherweise die Rekrutierung der Transkriptionsmaschinerie oder damit verbundener regulatorischer Faktoren in der XLOC-sp-RP11-sa-Konfiguration einschränkt. Diese Ergebnisse deuten darauf hin, dass die relative Positionierung von SaCas9 und SpCas9 in einem gegebenen genomischen Kontext ihre funktionelle Wirksamkeit entscheidend beeinflussen kann, ein Aspekt, der weitere Untersuchungen für optimierte CRISPR-basierte Modulationsstrategien rechtfertigt.
Das entwickelte Protokoll ist anpassbar für das Targeting oder Screening anderer synthetischer letaler nicht-kodierender RNA-Paare durch Modifikation des dualen gRNA-Vektors. Sobald eine Zelllinie von Interesse beide dCas9-Varianten stabil exprimiert, wird eine Vielzahl von Ansätzen möglich. Während RNAi, ASOs und shRNAs für das Gen-Silencing eingesetzt werden können, bietet CRISPRi eine einfachere Screening-Methode, und seine Kombination mit kleinen chemischen Verbindungen oder Inhibitoren ist ebenfalls denkbar. Durch die Substitution des dualen gRNA-Vektors durch eine duale gRNA-Bibliothek ermöglicht dieses Protokoll das systematische Screening von synthetischen letalen Paaren oder anderen genetischen Abhängigkeiten in Krebszellen, wodurch die Präzision des Targetings von Krebszellen verbessert wird.
Obwohl wir die Integration der dCas9-KRAB-MeCP2-Varianten durch lentivirale Transduktion erreicht haben, können auch alternative stabile Transfektionsmethoden verwendet werden, um die gewünschten Enzyme in 501-mel-Zellen einzubauen. Dies ist besonders relevant, da nach unserer Erfahrung das Stummschalten großer Konstrukte wie dCas9-KRAB-MeCP2 im Laufe der Zeit auftreten kann36,37. Alternativ wurde gezeigt, dass dCas9-KRAB eine kleinere Alternativeist 23. Unserer Erfahrung nach führte die Transduktion mit den kleineren dCas9-KRAB-Fusionsproteinen, die von Cas9-Orthologen abgeleitet wurden, zu einer verminderten Proteinexpression während einer längeren Kultivierung (siehe Ergänzende Abbildung 2). Daher sollte die konsistente Proteinexpression regelmäßig zu wiederkehrenden Zeitpunkten überwacht werden. Darüber hinaus kann die Transduzierung von zwei großen Fusionsproteinen zusammen mit Selektionsmarkern und dualen gRNA-Expressionskassetten zellulären Stress induzieren oder lentivirale Verpackungskapazitäten erreichen.
Das Erreichen einer 100%igen Knockdown-Effizienz bei dualem CRISPRi kann aufgrund des suboptimalen gRNA-Designs oder der unzureichenden dCas9-Expression eine Herausforderung darstellen. Um dies zu erreichen, sollten mehrere gRNAs pro Ziel mit fortschrittlichen Designwerkzeugen getestet werden, um die effektivsten Sequenzen zu identifizieren. Darüber hinaus kann die Optimierung der dCas9-KRAB-MeCP2-Expression durch verbessertes Vektordesign und Charakterisierung klonaler Zelllinien die Knockdown-Effizienz verbessern.
Alternative Strategien zur Integration des CRISPRi-Systems umfassen die Verwendung von Transposase-Vektoren oder die CRISPR-gerichtete Integration, um eine stabile Insertion in genomische Safe-Harbor-Stellen zu erreichen, die dafür bekannt sind, ihre Expression über die Zeit stabil zu halten38. Bei vielen Zelltypen, einschließlich Melanomzellen, ist es jedoch schwierig,39 zu transfizieren, so dass die lentivirale Transduktion die bevorzugte Methode ist. Die Elektroporation von Transposase- oder Cas-basierten Enzymen könnte eine alternative Möglichkeit bieten, die dCas9-KRAB-MeCP2-Konstrukte in Melanomzellenzu integrieren 40. Die Implementierung eines induzierbaren Knockout-Systems wäre vorteilhaft, um die dCas9-KRAB-MeCP2-Expression nach Bedarf zu aktivieren, anstatt die konstitutive Expression zu aktivieren, um den zellulären Stress zu minimieren.
Schließlich ist der entwickelte Ansatz nicht auf Melanomzellen beschränkt, sondern kann auf jeden Zelltyp ausgeweitet werden, in dem die synthetische Letalität untersucht wird. Dieser Ansatz soll explizit in Richtung der gRNA-Bibliotheks-Screening-Modalität skaliert werden. Aufgrund der erhöhten kombinatorischen Komplexität, z.B. mit mehr als drei gRNAs pro Genpaar, einschließlich Kontrollen und mindestens 300-facher Abdeckung, kann jedoch nur eine subtranskriptomische Fraktion der exprimierten lncRNAs praktisch anvisiert werden. Daher müssen die Nutzer eine sorgfältige Vorauswahl der Kandidaten treffen, indem sie Faktoren wie die lncRNA-Expressionsniveaus und ihre bekannte Relevanz in bestimmten zellulären Kontexten berücksichtigen, bevor sie mit dem Bibliotheksscreening fortfahren. Nichtsdestotrotz macht die kombinatorische Vielseitigkeit dieses Ansatzes ihn zu einem wertvollen Werkzeug für die Untersuchung synthetischer letaler Wechselwirkungen und anderer genetischer Abhängigkeiten von lncRNAs bei verschiedenen Krebsarten und möglicherweise anderen Krankheiten. Damit wird das derzeit verfügbare Toolkit zur Untersuchung dieser Netzwerkinteraktionen in Zellen erweitert.
Offenlegungen
Jochen Imig ist derzeit CGCIII finanziert von Pfizer Inc. CGCIII wird von Pfizer Inc., Merck KGaA und AstraZeneca PLC gesponsert. Die Sponsoren spielten keine Rolle bei der Konzeption, Durchführung, Interpretation oder dem Schreiben der Studie. Alle Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Wir danken Stefan Raunser (Max-Planck-Institut für molekulare Physiologie, Dortmund) für den Zugang zu Laborräumen der Biosicherheitsstufe 2. Besonderer Dank geht an Eric Wang für seine intellektuellen Beiträge und an alle ehemaligen und aktuellen Mitglieder des Imig-Labors für ihre wertvollen Diskussionen. Jochen Imig ist derzeit CGCIII finanziert von Pfizer Inc. am CGC III.
Materialien
| Name | Company | Catalog Number | Comments |
| Amicon Ultra Centrifugal Filter, 100 kDa MWCO | Millipore | UFC910024 | |
| anti-Cas9 (S. aureus) (6H4) mouse monoclonal antibody | Cell Signaling Technology | 48989 | |
| anti-Cas9 (S. pyogenes) (7A9-3A3) monoclonal antibody | Cell Signaling Technology | 14697 | |
| anti-GAPDH mouse monoclonal antibody | Sigma Aldrich | G8795 | |
| Anti-Mouse IgG (whole molecule)–Peroxidase antibody produced in rabbit | Sigma Aldrich | A9044 | |
| Bio-Rad ChemiDoc MP Imaging System | Bio-Rad | ||
| Blasticidine S hydrochloride | Sigma Aldrich | 15205-25MG | |
| CellTiter-Glo Luminescent Cell Viability Assay | Promega | G7570 | |
| Centrifuge | Eppendorf | 5804R | |
| Centrifuge | Eppendorf | 5415R | |
| CFX Connect Real-Time PCR Detection System | Bio-Rad | ||
| Clarity Western ECL Substrate, 500 mL | Bio-Rad | 1705061 | |
| CO2 Incubator Model CB 170 | Binder | ||
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | |
| Esp3I (BsmB1) restriction enzyme | Thermo Scientific | ER0451 | |
| Falcon 10 mL Serological Pipet | Corning | 356551 | |
| Falcon 25 mL Serological Pipet | Corning | 357525 | |
| Falcon 5 mL Serological Pipet | Corning | 356543 | |
| Fetal Bovine Serum | Sigma Aldrich | F9665-500ML | |
| Gibco Dulbecco’s modified Eagle’s medium (DMEM), high glucose, pyruvate | Gibco | 41966029 | |
| Human melanoma cell line 501-mel | was kindly provided by the Aifantis Lab (New York University) | RRID: CVCL_4633 | |
| Immobilon -P PVDF Membrane | Millipore | IPVH00010 | |
| Lenti-X 293T HEK cell line | Takara Bio | 632180 | |
| Mini Trans-Blot Cell system | Bio-Rad | ||
| Mini-PROTEAN Tetra Handcast System | Bio-Rad | ||
| NEBuilder HiFi DNA Assembly Reaction | New England Biolabs | E2621 | |
| Non-fat milk powder | Biomol | 54650 | |
| NuPAGE LDS Sample Buffer (4x) | Invitrogen | NP0008 | |
| One Shot Stbl Chemically Competent E. coli | Invitrogen | C737303 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985062 | |
| PCR Tubes 0.5 ml (Flat Cap) | VWR International | 732-3207 | |
| Phosphate-Buffered Saline (PBS) | Corning | 45000-446 | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | Thermo Scientific | F531L | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Polybrene | Sigma Aldrich | TR-1003-G | |
| Polyethylenimine, branched | Sigma Aldrich | 408727 | |
| Puromycin dihydrochloride | Santa Cruz Biotechnology | sc-108071A | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| SafeSeal Microcentrifuge Tube 1.5 mL | Sarstedt | 72,706 | |
| Sodium chloride, 5 M Aqua Solution, RNase Free | Alfa Aesar | J60434.AE | |
| Sodium Dodecyl Sulfate (SDS) | Sigma Aldrich | L3771 | |
| Syringe Filter PES 33mm 0.2 μM | Fisher Scientific | 15206869 | |
| TC Dish 100, Standard | Sarstedt | 8,33,902 | |
| TC Plate 6 Well, Standard, F | Sarstedt | 83,39,20,005 | |
| Tris base | Roche | 10708976001 | |
| TWEEN 20 | Sigma Aldrich | P9416-50ML |
Referenzen
- Lander, E. S., et al. Initial sequencing and analysis of the human genome. Nature. 409 (6822), 860-921 (2001).
- ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Szcześniak, M. W., Wanowska, E., Mukherjee, N., Ohler, U., Makałowska, I. Towards a deeper annotation of human lncRNAs. Biochim Biophys Acta Gene Regul Mech. 1863 (4), 194385(2020).
- Fang, S., et al. NONCODEV5: a comprehensive annotation database for long non-coding RNAs. Nucl Acids Res. 46 (D1), D308-D314 (2018).
- Lin, J., et al. Human-specific lncRNAs contributed critically to human evolution by distinctly regulating gene expression. eLife. 12, RP89001.2(2023).
- Sarropoulos, I., Marin, R., Cardoso-Moreira, M., Kaessmann, H. Developmental dynamics of lncRNAs across mammalian organs and species. Nature. 571 (7766), 510-514 (2019).
- Melé, M., et al. Human genomics. The human transcriptome across tissues and individuals. Science. 348 (6235), 660-665 (2015).
- Cabili, M. N., et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev. 25 (18), 1915-1927 (2011).
- Liu, S. J., Dang, H. X., Lim, D. A., Feng, F. Y., Maher, C. A. Long noncoding RNAs in cancer metastasis. Nat Rev Cancer. 21 (7), 446-460 (2021).
- Ahmad, M., Weiswald, L. B., Poulain, L., Denoyelle, C., Meryet-Figuiere, M. Involvement of lncRNAs in cancer cells migration, invasion and metastasis: cytoskeleton and ECM crosstalk. J Exp Clin Cancer Res. 42 (1), 173(2023).
- Jain, A. K., et al. LncPRESS1 Is a p53-Regulated LncRNA that Safeguards Pluripotency by Disrupting SIRT6-Mediated De-acetylation of Histone H3K56. Mol Cell. 64 (5), 967-981 (2016).
- Bergmann, J. H., et al. Regulation of the ESC transcriptome by nuclear long noncoding RNAs. Genome Res. 25 (9), 1336-1346 (2015).
- Kretz, M., et al. Control of somatic tissue differentiation by the long non-coding RNA TINCR. Nature. 493 (7431), 231-235 (2013).
- Yoon, J. H., et al. LincRNA-p21 suppresses target mRNA translation. Mol Cell. 47 (4), 648-655 (2012).
- Huarte, M. The emerging role of lncRNAs in cancer. Nat Med. 21 (11), 1253-1261 (2015).
- Badowski, C., He, B., Garmire, L. X. Blood-derived lncRNAs as biomarkers for cancer diagnosis: the Good, the Bad and the Beauty. NPJ Precis Oncol. 6 (1), 40(2022).
- Zong, X., et al. Knockdown of nuclear-retained long noncoding RNAs using modified DNA antisense oligonucleotides. Meth Mol Biol. 1262, 321-331 (2015).
- Gagnon, K. T., Li, L., Chu, Y., Janowski, B. A., Corey, D. R. RNAi factors are present and active in human cell nuclei. Cell Rep. 6 (1), 211-221 (2014).
- Zeng, Y., Cullen, B. R. RNA interference in human cells is restricted to the cytoplasm. RNA. 8 (7), 855-860 (2002).
- Yang, J., et al. CRISPR/Cas9-mediated noncoding RNA editing in human cancers. RNA Biol. 15 (1), 35-43 (2018).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355 (6320), aah7111(2017).
- Tsung, K., et al. CRISPRi screen of long non-coding RNAs identifies LINC03045 regulating glioblastoma invasion. PLoS Genet. 20 (6), e1011314(2024).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Boettcher, M., McManus, M. T. Choosing the right tool for the job: RNAi, TALEN, or CRISPR. Mol Cell. 58 (4), 575-585 (2015).
- Yeo, N. C., et al. An enhanced CRISPR repressor for targeted mammalian gene regulation. Nat Meth. 15 (8), 611-616 (2018).
- Parrish, P. C. R., et al. Discovery of synthetic lethal and tumor suppressor paralog pairs in the human genome. Cell Rep. 36 (9), 109597(2021).
- Najm, F. J., et al. Orthologous CRISPR-Cas9 enzymes for combinatorial genetic screens. Nat Biotechnol. 36 (2), 179-189 (2018).
- Petroulia, S., et al. CRISPR-inhibition screen for lncRNAs linked to melanoma growth and metastasis. bioRxiv. , (2024).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- New England Biolabs. NEBuilder HiFi DNA Assembly Reaction (E2621) v2. , (2020).
- Cortés-Ríos, J., et al. Protein quantification by bicinchoninic acid (BCA) assay follows complex kinetics and can be performed at short incubation times. Anal Biochem. 608, 113904(2020).
- Junior, N. Polyacrylamide Gel Electrophoresis (SDS-PAGE) v1. , (2019).
- Chen, X., Liu, J., Janssen, J. M., Gonçalves, M. A. F. V. The chromatin structure differentially impacts high-specificity CRISPR-Cas9 Nuclease strategies. Mol Ther Nucl Acids. 8, 558-563 (2017).
- Schep, R., et al. Impact of chromatin context on Cas9-induced DNA double-strand break repair pathway balance. Mol Cell. 81 (10), 2216-2230.e10 (2021).
- Daer, R. M., Cutts, J. P., Brafman, D. A., Haynes, K. A. The impact of chromatin dynamics on Cas9-Mediated genome editing in human cells. ACS Syn Biol. 6 (3), 428-438 (2017).
- Chavez, M., Rane, D. A., Chen, X., Qi, L. S. Stable expression of large transgenes via the knock-in of an integrase-deficient lentivirus. Nat Biomed Eng. 7 (5), 661-671 (2023).
- Ellis, J. Silencing and variegation of gammaretrovirus and lentivirus vectors. Human Gene Ther. 16 (11), 1241-1246 (2005).
- Shrestha, D., et al. Genomics and epigenetics guided identification of tissue-specific genomic safe harbors. Genome Biol. 23 (1), 199(2022).
- Chu, Z., et al. Enhanced gene transfection and induction of apoptosis in melanoma cells by branched poly(β-amino ester)s with uniformly distributed branching units. J Control Release. 367, 197-208 (2024).
- Han, S. Y., et al. Nucleofection is a highly effective gene transfer techfnique for human melanoma cell lines. Exp Dermatol. 17 (5), 405-411 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten