Method Article
完整的荧光成像定时
摘要
Streptomyces are characterized by a complex life cycle that has been experimentally challenging to study by cell biological means. Here we present a protocol to perform fluorescence time-lapse microscopy of the complete life cycle by growing Streptomyces venezuelae in a microfluidic device.
摘要
Live-cell imaging of biological processes at the single cell level has been instrumental to our current understanding of the subcellular organization of bacterial cells. However, the application of time-lapse microscopy to study the cell biological processes underpinning development in the sporulating filamentous bacteria Streptomyces has been hampered by technical difficulties.
Here we present a protocol to overcome these limitations by growing the new model species, Streptomyces venezuelae, in a commercially available microfluidic device which is connected to an inverted fluorescence widefield microscope. Unlike the classical model species, Streptomyces coelicolor, S. venezuelae sporulates in liquid, allowing the application of microfluidic growth chambers to cultivate and microscopically monitor the cellular development and differentiation of S. venezuelae over long time periods. In addition to monitoring morphological changes, the spatio-temporal distribution of fluorescently labeled target proteins can also be visualized by time-lapse microscopy. Moreover, the microfluidic platform offers the experimental flexibility to exchange the culture medium, which is used in the detailed protocol to stimulate sporulation of S. venezuelae in the microfluidic chamber. Images of the entire S. venezuelae life cycle are acquired at specific intervals and processed in the open-source software Fiji to produce movies of the recorded time-series.
引言
链霉菌是土栖细菌的特征是由多细胞,营养清除菌丝处于休眠状态,unigenomic孢子1-3涉及形态分化一种复杂的发育周期。
根据有利的生长条件,典型的链霉菌孢子开始通过挤出一个或两个芽管( 图1)发芽。这些管由尖延伸拉长,成长为被称为营养菌丝分枝菌丝网络。极地的增长和菌丝分枝由必需的蛋白质DivIVA执导。此卷曲螺旋蛋白是一个大的胞质复合物的一部分被称为polarisome,这是新的细胞包膜材料的延伸末端4-7插入至关重要。期间的营养生长,菌丝长丝成为由不频繁形成所谓的横壁8的条块。这些跨壁重新形成张塌塌米的FtsZ,微管蛋白样细胞骨架蛋白,其在大多数细菌9为细胞分裂至关重要。在链霉菌中,然而,这些营养横壁不会导致收缩和细胞-细胞分离,因此菌丝质量保持为相互连接的合胞室的一个网络。响应于营养限制和其他信号未得到很好的理解,专门气生菌丝从营养菌丝体脱离并生长到空气中3。这些结构的勃起启动发展的生殖期,在此期间,长的多的基因组的气生菌丝变得分成几十个相等大小unigenomic前孢子隔间。这种大规模的细胞分裂活动是由单一的产孢子菌丝2,10内的多个环的FtsZ的同步收缩驱动。形态分化是处于休眠状态,厚壁,色素孢子释放完毕。
T"FO:保together.within页="1">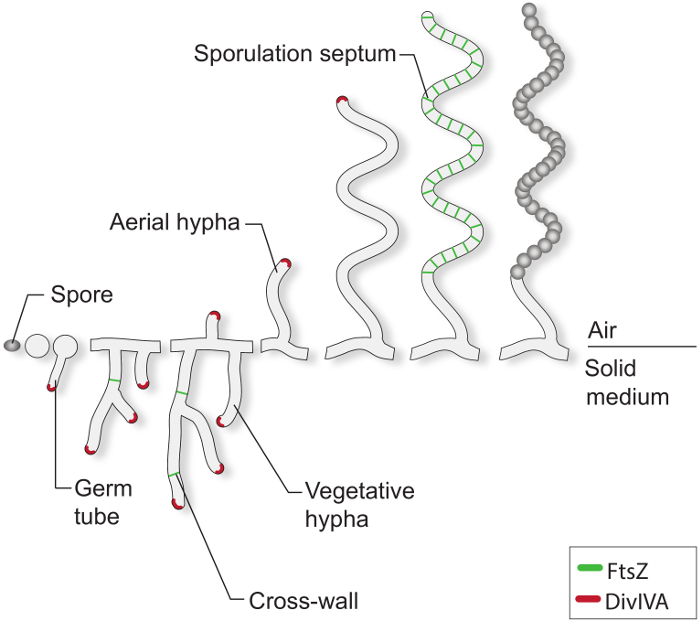
图 1: 在固体培养基上 的 链霉菌 的生命周期这是生命周期的模式的基础上S的经典研究霉菌在琼脂平板上生长。孢子的细胞发展开始与一个或两个芽管,它由前端延伸生长以形成分枝菌丝的一个网络的形成。极性生长和营养菌丝分支是由DivIVA(红色)执导。营养跨墙的形成需要的FtsZ(绿色)。响应于营养物的限制和其它信号,气生菌丝竖立。天线生长停滞是紧密结合的FtsZ环,其产生的孢子形成隔片即划分的产孢子的菌丝成盒状前孢子隔间的梯形的组件协调。这些车厢组装一厚孢壁并最终released的成熟色素孢子。
链霉菌生命周期的关键发育事件都有良好表征1,3。但是,什么是仍然很少是采用荧光延时显微镜提供洞察亚流程支撑的分化,如蛋白质本地化动态,染色体运动和发育控制细胞分裂的细胞生物学研究。 链霉菌的发展活细胞成像已经由于生命周期的复杂性和生物体的生理特征是具有挑战性的。关于营养生长以前的研究和产孢septation的初始阶段已采用氧可渗透成像腔室,或天蓝色链霉菌在显微镜阶段11-15琼脂糖支持增长。这些方法,但是,是由许多因素的限制。一些系统仅允许细胞生长的短期成像细胞前Ð荧光蛋白从供氧不足或患有生长出来焦平面由于菌丝发育的立体图案。在长期成像是可能的,培育在琼脂糖焊盘限制的实验灵活性细胞,因为细胞不能暴露于替代生长或胁迫条件,并从琼脂糖焊盘培养基中的背景荧光严重限制监视弱荧光的能力的情况下信号。
在这里,我们描述了具有优良的精度和灵敏度链完整生命周期的活细胞成像的协议。通过在连接到一个荧光广角显微镜(图2)的微流体装置成长链霉菌 ,我们现在能够监视萌发,营养生长和孢子形成septation超过高达30小时的时间段。这是通过使用新的模式生物链霉菌的极大地促进 venezuelae因为它sporulates在水下文化接近完成,从而克服了传统的模式物种S的限制天蓝色链霉菌 ,其中仅在固体培养基上16-20 sporulates。为了帮助可视化营养生长和产孢,我们共表达荧光标记的细胞极性标记DivIVA和关键细胞分裂蛋白FtsZ的版本。
我们使用的是已被成功用于分枝杆菌, 大肠杆菌 , 谷氨酸棒杆菌,枯草芽孢杆菌和酵母21-25市售微流体装置。该系统能收集在一个单一的焦平面细胞,并且允许从不同的油藏培养基的连续灌流的控制。在详细的协议,我们采取这一特点暴露S. venezuelae营养菌丝营养降档,以促进孢子。
该协议德刻划是整个链霉菌生命周期的活细胞成像,但如果具体的发育阶段是特别感兴趣的,可以选择的替代培养基条件或显微镜的设置。
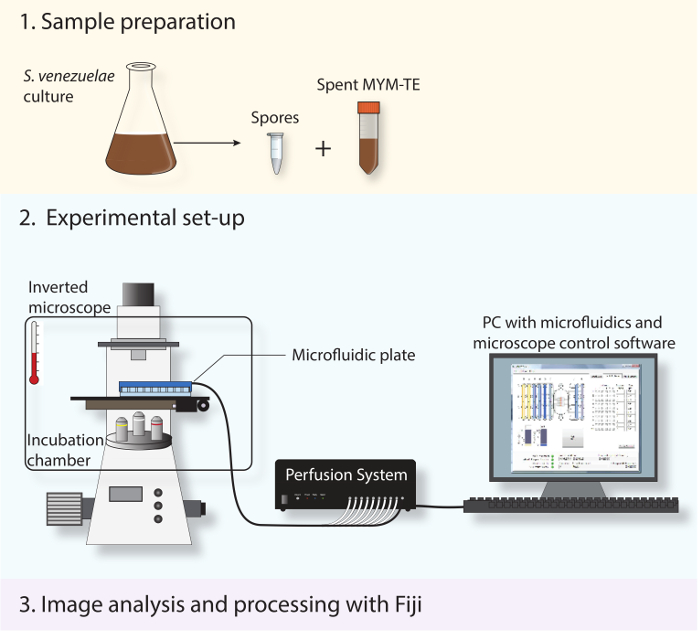
图2: 示意图描述了实验的工作流程 。在协议中所描述的三个主要步骤被示出。首先,孢子和用过的培养基是从固定相培养物制备。其次,新鲜孢子装入一个微流体系统和S. venezuelae在整个使用其发育的生命周期的全自动倒置显微镜与孵育室以保持最佳生长温度成像。三,获得的延时序列进行分析,并使用开源软件斐济处理。
研究方案
1.鲜S.隔离venezuelae孢子消耗培养基的制备
- 接种30毫升麦芽糖酵母提取物,麦芽提取物(MYM)中,辅以60微升R2微量元素溶液(TE),应变SV60 [attB位 φBT1 :: pSS209(P divIVA的10微升孢子- divIVA-mcherry P 的FtsZ -ftsZ-ypet)。对于持续增长和细胞的孢子,使用挡板烧瓶或包含一个弹簧,以便有足够的通风烧瓶中。
- 培养细胞在30℃下35-40小时和250rpm。
- 检查由液体装相差显微镜的细胞。在这一点上菌丝碎片和孢子将可见。
- 离心机1萃取池在400×g离心1分钟台式离心机以沉淀菌丝和较大细胞碎片。
- 输送约300微升上清液合作ntaining孢子悬浮液,以一个新的1.5毫升管,并保持在冰上。保留剩余的培养基中的步骤1.7。
- 稀释孢子MYM-TE的1点20分(最终浓度:0.5-5×10 7个孢子/ ml),并保持在冰上直至需要(见步骤2.3)。
注意:虽然在链霉菌孢子触发的确切情况还不是很清楚,用花了MYM-TE介质,包括从文化孢子任何未知外信号,刺激孢子。 - 过滤消毒10毫升要获得花MYM-TE剩余的培养基,是免费的孢子和菌丝体碎片组成。用无菌0.22微米的注射器过滤器和传输花费MYM-TE到无菌的15毫升管。
- 保持准备花费MYM-TE几天在4℃下,如果使用类似的生长条件附加实验都进行。
2.微流体装置的制备
注意:每个微解放军德允许多达四个独立的实验来进行。为了避免不使用的流室的污染,建立一个实验时使用的无菌溶液和工作条件。
- 除去从微流体板航运溶液,并用无菌MYM-TE冲洗孔中。
- 加入300微升MYM-TE很好1和300微升花了MYM-TE入口井2至6。
- 负载40微升来自步骤1.6在泳道的"A"的小区负载以及8稀释孢子和密封根据所述歧管到板制成品的指令。
- 启动微流体控制软件,选择合适的板型,并从入口井1设置一个流动计划,主要流道和文化室由流动介质至5日6磅每以及2分钟。
- 在微流体控制软件,创建用于实验的流协议:从入口井1中流动的MYM-TE的6小时,以允许发芽和细胞的营养生长。开关然后从井2-5花费MYM-TE中灌注在实验的剩余时间。在实验过程中保持流动压力恒定在6磅(对应于大约9微升培养基/小时)。步3.5后启动流程程序。
3.显微镜和定时协议的成立
注意:在配备有一个SCMOS照相机,金属卤化物灯,硬件自动对焦,用于96孔板的阶段保持器和一个环境室的全电动和自动化倒置广角显微镜实现此方法。
- 为了防止在开始实验后,用自动对焦问题预温环境室至30℃提前。所需的时间取决于所使用的环境的腔室,在显微镜和加热系统。开始实验前,夜间升温系统。
- 打开显微镜和显微自动化控制softwa回覆。使用高数值孔径(NA)油浸物镜为最佳的信号采集和空间分辨率,诸如100X,1.46 NA油DIC物镜。选择适当的过滤器和色镜获得黄色荧光和红色荧光蛋白融合的微分干涉对比(DIC)的图像和图像。
- 放置浸油下降到目标和浸没流体添加到成像窗口底部上的微流体板,以提高图像采集期间的细胞焦点。密封微流体装置(步骤2.5)小心安装到倒置显微镜的阶段。确保该板被牢固地放置在阶段保持器,并且不在实验的过程中移动。
- 使用嵌入式位置标记为取向带来微流体培养室的摄像窗口聚焦。聚焦在第一流动腔室的最左部分(标记为"A")与收集大小5中,对应于0.7微米陷阱高度。
- 在微流体软件,在4磅15秒称重传感器从进口以及8。通过在成像窗口移动台检查在培养室中的细胞密度。如果没有孢子被困,重复的小区装载步骤或替代地增大负载压力和/或时间,直到所需的细胞密度达到(每个成像窗1-10孢子以2048点¯x2,048像素)。避免超载文化室。
注:我们通常使用的陷阱大小5成像链 ,但尺寸4和3,请参阅供应商的手册其他建议优化小区负载过程中,我们也取得了良好的效果与陷阱。 - 在开始从步骤2.5控制软件的流程程序,并允许该微板在显微镜阶段1小时开始图像采集前中暑的平衡。
- 在显微镜控制软件,建立一个多维度的收购采取万亩在多级职位在一段时间ltiple图片:
- 对于自动保存:对图像文件自动保存指定目录。
- 用于照明设置,预先确定每个特定构建最佳照明设置。对于概述实验中,使用下面的曝光时间:DIC 150毫秒,YFP 250毫秒,RFP 100毫秒。
- 对于时间序列:成立时间序列收购序列在期望的时间点的图像。成像S的生命周期的venezuelae,选择在24小时的时间内一个40分钟的时间间隔。
- 对于第一阶段位置和自动对焦:通过移动对每个感兴趣成像位置的阶段和存储阶段的位置扫描培养室。确保单级位置位于足够远,以尽量减少漂白和phototoxicity.Typically使用多达12个位置。对于每个时间点运行自动聚焦例程以校正缓慢焦距漂移。如果在显微镜控制软件可用设置自动聚焦策略,以"通过硬件自动对焦局部表面更新"。一旦所选择的阶段的位置的Z坐标进行验证,激活硬件自动对焦。
- 开始在显微镜控制软件延时实验。
- 检查所有阶段的位置仍处于焦点在稍后的点。对于运行在几个小时的时间推移实验中,我们偶尔会使用自动对焦时,甚至观察一个阶段漂移。如果阶段位置需要重新调整,停止实验在适当的时间点,调整焦点和重新定义的成像时间间隔(步骤3.7.3)中的实验。请参阅步骤4.3如何连接的时间序列。
- 停止图像采集后24-30小时,或者当在感兴趣的区域中的菌丝已分化成孢子(参见DIC图像系列)。在软件停止流动程序和拆卸微流体装置。
- 准备使用的微流控板短期STO愤怒。去除残留MYM-TE,花了MYM-TE以及从1到6,空废井7和小区负载以及8.在无菌条件下,重新填写使用的车道"A"井和未使用的通道井("B"至在板"D"),用无菌磷酸盐缓冲盐水(PBS)中。密封该板用石蜡膜,以防止它在4℃下干燥和存储。
4.生成时间推移电影使用斐济软件
注意:我们注意到,不同的商业和自由软件包可用于处理时间推移显微镜图像,包括ZenBlue,的Metamorph,ICY,ImagePro或ImageJ的。在这里,我们专注于斐济,这是一个基于ImageJ的一个开放源码的图像处理程序,并已经提供了一些有用的预装插件。
- 从时间推移实验已经安装了斐济计算机传送图像数据。
- 启动程序,并使用与导入成像文件#34;文件>导入>生物格式"或仅仅是成像文件拖动到斐济在"生物格式导入选项",勾选框"分裂渠道"获取每个照明通道的单独图像堆栈(DIC ,RFP,YFP)。
- 可选:要为每个通道合并从在图像采集,由于重新调整(步骤3.9)突破造成独立的时间序列,打开相应的图像堆栈,并运行"图像>栈>工具>串连"(DIC,RFP,YFP )。
- 通过经由图像堆栈滚动评估时间推移数据的质量。寻找那些留在焦点随着时间的推移菌丝,显示菌丝生长,最终形成孢子(DIC堆栈)。检查DivIVA-mCherry在菌丝的提示(RFP堆栈)和FtsZ的-YPet预期的亚细胞定位形成植物人或产孢子菌丝(YFP栈)单环或多个阶梯状的结构,分别为。
- 隔离感兴趣的区域为下游加工塑料图像的NG。时间推移显微镜通常会产生可能由斐济处理放缓的大文件。因此,建议以鉴定和分离的感兴趣区域(ROI)的区域,并在图像堆栈这个较小的版本执行进一步的图像处理步骤。
- 在菜单中选择了"矩形选择"工具,并在这样的方式的显影菌丝是由整个图像序列在ROI括绘制在DIC图像堆栈一个矩形ROI。
- 重复投资回报率栈"CTRL"+"Shift"键+"D"。
- 点击YFP图像堆栈的名称栏中,选择"编辑>选项>恢复选项"菜单中的恢复从原来的DIC以前的矩形选区堆在YFP栈同样现在的位置,并用"Ctrl"键复制的投资回报率+"Shift"键+"D"。
- 重复此过程的RFP堆栈。
- 对齐图像的三个图像栈吨Ø随着时间的推移删除在XY平面舞台漂移。滚动到DIC堆栈中的最后一帧,然后从菜单中选择"插件>注册> StackReg> RigedBody"。重复此步骤的RFP和YFP图像堆栈。如果有必要,重复步骤4.5.1-4.5.3裁剪的投资回报率。
- 可选:手动调整亮度和对比度的"调整亮度和对比度工具"("Ctrl"键+"Shift"键+"C")或选择每个图像堆栈"过程>增强对比度"从菜单和应用"正常化"命令堆栈中的所有图像。应当指出的是,后者的命令将改变的像素值,因此,可能会影响到下游的图像分析。
- 可选:结合个人的堆栈(转换为RGB文件格式,见4.11)水平或垂直使用插件"图像>栈>工具>合并"。
- 添加比例尺("分析>工具>比例尺")和时间压模(#34;图像>栈>时间盖戳")。
- 保存修改后的图像栈作为".TIFF"格式的图像序列。要制作影片,保存为".AVI"。另外,利用"生物格式>生物格式导出"插件,然后选择".MOV"文件类型的QuickTime格式导出图像序列。
- 为了获得单帧图片,选择感兴趣的框架和复制的图像"CTRL"+"Shift"键+"D"。更改在"图像>类型> RGB颜色或8位"产生的图像为"RGB"或"8位"的类型和保存图像为".TIFF"。
注意:斐济提供了许多额外的功能,以进一步注释或处理时间推移系列。转到详细信息(http://fiji.sc/Fiji)斐济在线支持。
结果
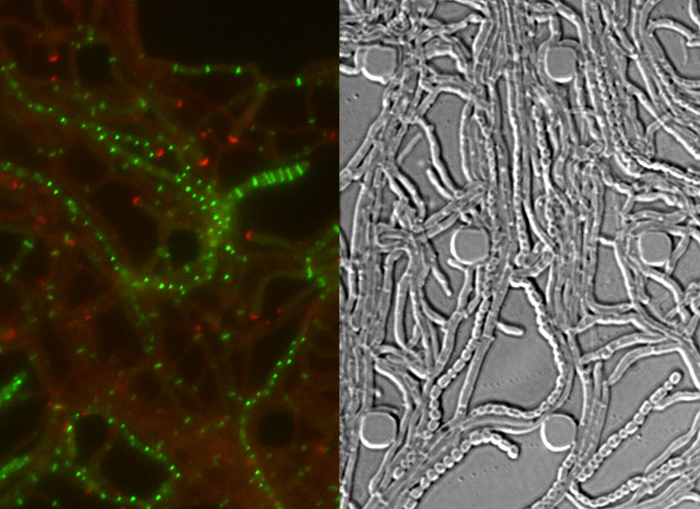
整个S的成功活细胞成像venezuelae生命周期产生一个连续的时间序列包括萌发,营养生长和孢子形成( 图3,电影1)的关键发育阶段。通过生命周期进展的可视化是通过DivIVA-mCherry和FtsZ的-YPet的不同亚细胞定位进一步增强。在发芽和营养生长,DivIVA-mCherry只积聚在生长菌丝提示或标记新形成的分支点( 图4A)。这些结果与的DivIVA 12,26的以前报道的亚细胞定位。与此相反,FtsZ的-YPet形成在生长的菌丝体( 图4B)的不规则间隔单环状结构。这些结构提供了用于非缩窄营养横壁的合成的支架,导致interconn的形成ected菌丝车厢8。菌丝成长成产孢子菌丝的细胞分化的极地DivIVA-mCherry灶消失变为可见,极增长的停滞和FtsZ的-YPet荧光随之增加( 图4C)。在孢子菌丝,FtsZ的-YPet的本土化模式发生巨大变化;第一螺旋的FtsZ-YPet长丝沿菌丝翻滚,然后,在突然的,几乎同步的事件时,螺旋结合成规则间隔的FtsZ-YPet环的一个阶梯。根据这里所述的实验条件下,这些均匀分布的FtsZ-YPet梯子持续约2小时。最后,孢子形成隔片成为微分干涉对比(DIC)图像( 图4D),并最终新孢子被释放可辨。
随后的协议描述使用斐济软件提供作为图像处理 TEP一步是如何产生的动画,从所采集的时间推移系列(电影1)出版物解释。
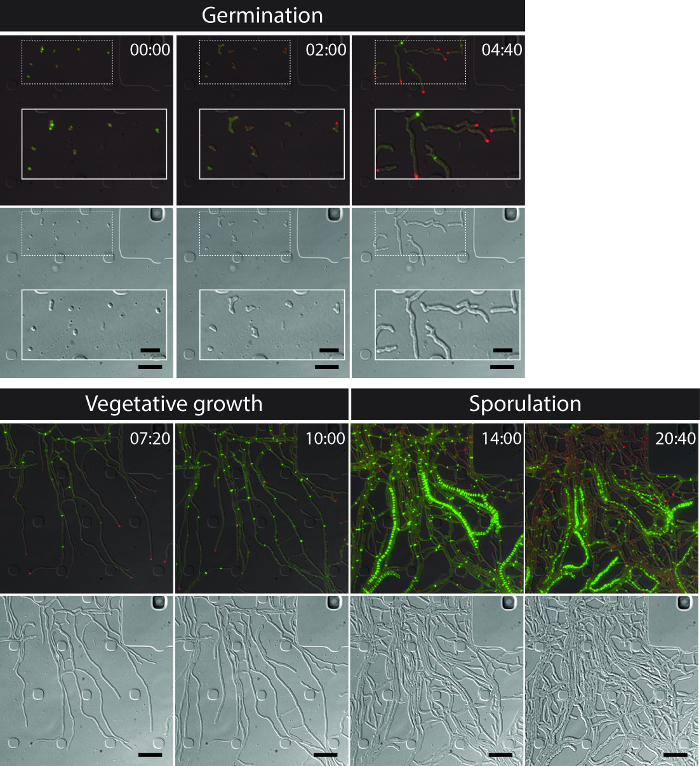
图3:从代表时间推移荧光显微镜系列 S 的快照图像 venezuelae 产生荧光标记的FtsZ(绿色)和DivIVA(红色)显示的是合并的图像中选择的帧(顶面板:RFP-YFP-DIC,底部面板:DIC)。可视化链霉菌的生命周期,包括萌发,营养生长和孢子形成。图片是从动画1.所用时间为小时:分钟。比例尺= 5微米。 请点击此处查看该图的放大版本。
D / 53863 / 53863fig4.jpg"/>
图4: 一个成功的时间推移系列 链 生命周期 的代表性结果 (A)DivIVA-mCherry的定位。显示的是从电影1与RFP和DIC渠道合并图像的两个连续时间点的快照。 DivIVA-mCherry标志着新的菌丝分支站点(实心箭头头),并在菌丝顶端直接极性生长(开放箭头)本地化。比例尺= 10微米(B)营养生长(箭头)中的FtsZ-YPet定位。的FtsZ-YPet形成单环状结构(上图)不收缩(DIC,下图)。孢子septation期间(C)的FtsZ-YPet(绿色)的本地化。 DivIVA-mCherry灶显示为红色,如下图所示经过时间(小时:分钟)。比例尺:5微米。 (D)从(C)对应的孢子菌丝DIC图像显示窗体前孢子车厢可见孢子隔膜(左图),最终成熟为孢子链(右图)的通货膨胀。时间是小时:分钟。比例尺= 5微米。 请点击此处查看该图的放大版本。
电影1:定时荧光显微镜系列 委内瑞拉链霉菌 的生命周期 的电影包括合并的RFP-YFP图像(左)和对应的微分干涉对比(DIC)图像(右)。 DivIVA-mCherry显示为红色和FtsZ的-YPet绿色。单个帧之间的时间间隔为40分钟。比例尺= 10微米。 请点击此处查看THI的电影。
讨论
链霉菌的生命周期的时间推移显微镜已经在过去的技术挑战。在这里,我们提出了一个强大的协议来执行使用荧光蛋白融合到细胞极性标记DivIVA和细胞分裂蛋白FtsZ的帮助形象化,并通过发展计划的进展跟踪( 图2)的整个生命周期中的活细胞成像。
中央该方法是S的栽培venezuelae在微流体装置,其允许与用过的培养基(花费MYM-TE)恒定介质灌注和正常生长培养基(MYM-TE)的交换。的培养条件的变化是对所描述的协议重要的,因为营养挂低档如在废培养基孔中作为任何尚未鉴定的胞外信号( 例如群体感应信号),促进孢子形成,而营养丰富的培养基的连续供给刺激植物growt(数据未显示)几乎没有任何孢子形成小时。因此,在该微流体系统中培养细胞优于琼脂糖培养细胞,因为它提供了更多的实验灵活性,并且允许响应于改变培养条件中的细菌生长的变化的长期监测。虽然微流体板被设计为一次性使用,流量,但没有接种细胞可在随后的实验中使用的信道。我们建议使用一个星期内的微流控板的所有渠道,我们经历密封歧管已被打开更长的时间片的问题。
当建立一个实验中,我们发现,新鲜制备的孢子发芽的被加载到流槽内放置2小时内,而从冷冻甘油贮存衍生孢子需要至少6小时前芽管出现(数据未显示)。这种延迟在发芽延伸实验的长度,可以与干扰设备的可用性和实验条件。同样重要的是开始与MYM-TE中灌注的实验为至少3小时,以提供足够的营养素芽管的发展和生长。灌注MYM-TE可以超出了最初的3小时,如果实验的目的是研究营养生长进行扩展。而孢子提供原料的优选的选择,短菌丝片段也可以被加载到微流控板。然而,使用剪切菌丝当加载效率显著下,经常需要几个负荷运行审查非孢子的突变体时,其可以呈现出限制。不管使用接种物的类型,不超载与孢子或菌丝片段作为本培养室将导致快速拥挤,可与媒体扩散干扰和图像分析复杂化是重要的。
规划报告蛋白的结构是很重要的carefuLLY在链霉菌中的荧光时间推移成像的使用。靶蛋白和荧光报告和N-或C-末端融合的选择之间接头长度可能是关键性的。此外,最佳实验条件,包括成像频率和曝光时间,需要预先确定每个荧光标记蛋白。S. venezuelae菌丝显示出自发荧光的绿色/黄色通道成像是在低的水平表达荧光标记的蛋白时,可能会成为问题。此外,应该指出的是,发芽和初始营养生长阶段,S期间venezuelae是短波长光( 例如 ,使用蛋白融合到太平洋时)特别敏感。
尽管在成像技术和荧光报道系统细菌的活细胞成像的连续发展,大多数的软件包( 如 MicrobeTracker,Schnitzelc厄尔,CellProfiler),用于这些种类的数据集的后续自动化处理不支持从丝状菌来源的具有多细胞生活方式27-29图像的分析。因此,有必要开发用于从链霉菌属和其他丝状菌成像数据的定量高通量分析的合适算法。
总之,这里所描述的工作表明S的巨大潜力venezuelae作为属的,因为它在液体形成孢子的能力的新的模式的发展系统。微流体培养装置简单,即使没有经验的用户使用。它提供了一个很好的平台,研究中心的链霉菌的生命周期,包括动态蛋白质定位,极化的增长,以及多菌丝形态分化成unigenomic孢子链细胞生物学过程。此外,该实验集U普还提供了一种诱人的起点,调查在开发事件需要交替培养条件,或使用荧光染料的荧光ðα-氨基酸来监测肽合成或碘化丙啶和4',6-二脒基-2-苯基这样(DAPI)以可视化的染色体组织30,31。
披露声明
The authors have nothing to disclose.
致谢
The authors thank Grant Calder for technical assistance with the microscope, Matt Bush for comments on the protocol, and the John Innes Centre for purchase of the Zeiss widefield microscope. This work was funded by BBSRC grant BB/I002197/1 (to M.J.B), by BBSRC Institute Strategic Programme Grant BB/J004561/1 to the John Innes Centre, by Swedish Research Council grant 621-2010-4463 (to K.F.), and by a Leopoldina Postdoctoral Fellowship (to S.S.).
材料
| Name | Company | Catalog Number | Comments |
| B04A CellAsic ONIX plate for bacteria cells | Merck-Millipore | B04A-03-5PK | Microfluidic culture plates |
| CellAsic ONIX Microfluidic Perfusion System and ONIX FG (version 5.0.2) | Merck-Millipore | EV-262 | The latest ONIX vesion (July 2015) and instructions on how to use the programme can be found here: http://www.merckmillipore.com |
| Axio Observer.Z1 Microscope | Zeiss | 431007-9902-000 | Fully automated and motorized inverted widefield microscope |
| Incubator XL multi S1 with Temperature Module S1 and Heating unit XL S2 | Zeiss | 411857-9061-000 | Environmental chamber surrounding the microscope |
| Plan-Apochromat 100x/1.46 Oil DIC objective | Zeiss | 420792-9800-000 | |
| Ocra FLASH 4 V2 | Hamamatsu Photonics K.K. | C11440-22CU | |
| Illuminator HXP 120V | Zeiss | 423013-9010-000 | |
| FL Filter Set 46 HE YFP shift free | Zeiss | 489046-9901-000 | Fluorescent filter set, excitation 500/25 nm, emission 535/30 nm |
| FL Filter Set 63 HE RFP shift free | Zeiss | 489063-0000-000 | Fluorescent filter set, excitation 572/25 nm, emission 629/30 nm |
| Mounting frame K-M for multiwell plates | Zeiss | 000000-1272-644 | Stage holder for microfluidic plate |
| ZEN pro 2012 | Zeiss | 410135-1002-120 | Microscope control software |
| ZEN Module Time Lapse | Zeiss | 410136-1031-110 | Software module to set up time-lapse microscopy experiments |
| ZEN Module Tiles/Positions | Zeiss | 410136-1025-110 | Software module to save specific stage positions (xzy) |
| Fiji | open-source software package | http://fiji.sc/Fiji | Generation of time-lapse movies |
| Maltose-Yeast Exctract-Malt Extract (MYM) 4 g Maltose 4 g Yeast extract 10 g Malt extract add 1 L H2O using 50 % tap water and 50 % reverse osmosis water and supplement with 200 ml of R2 trace element solution per 100 ml after autoclaving | Sporulation medium used to culture S. venezuelae SV60 | ||
| R2 Trace element solution (TE) 8 mg ZnCl2 40 mg FeCl3-6H2O 2 mg CuCl2-2H2O 2 mg MnCl2-4H2O 2 mg Na2B4O7-10H2O 2 mg (NH4)6Mo7O24-4H2O add 200 ml H2O Autoclave and store at 4 oC | Add 0.002 volumes to MYM | ||
| PBS (phosphate buffered saline) | Sigma | P4417-100TAB | Used to refill inlet wells of unused lanes in B04A plates in order to prepare plate for short-term storage. |
| 0.22 µm syringe filters | Satorius stedim | 16532-K | Preparation of spent MYM-TE |
| SV60 | John Innes Centre strain collection | S. venezuelae strain expressing divIVA-mcherry and ftsZ-ypet |
参考文献
- Bush, M. J., Tschowri, N., Schlimpert, S., Flärdh, K., Buttner, M. J. c-di-GMP signalling and the regulation of developmental transitions in streptomycetes. Nat. Rev. Microbiol. 13, 749-760 (2015).
- Jakimowicz, D., van Wezel, G. P. Cell division and DNA segregation in Streptomyces.: how to build a septum in the middle of nowhere?. Mol. Microbiol. 85, 393-404 (2012).
- McCormick, J. R., Flärdh, K. Signals and regulators that govern Streptomyces development. FEMS Microbiol. Rev. 36, 206-231 (2012).
- Flärdh, K., Richards, D. M., Hempel, A. M., Howard, M., Buttner, M. J. Regulation of apical growth and hyphal branching in Streptomyces. Curr. Opin. Microbiol. 15, 737-743 (2012).
- Hempel, A. M., et al. The Ser/Thr protein kinase AfsK regulates polar growth and hyphal branching in the filamentous bacteria Streptomyces. Proc. Natl. Acad. Sci. U S A. 109, E2371-E2379 (2012).
- Fuchino, K., et al. Dynamic gradients of an intermediate filament-like cytoskeleton are recruited by a polarity landmark during apical growth. Proc. Natl. Acad. Sci. U S A. 110, E1889-E1897 (2013).
- Holmes, N. A., et al. Coiled-coil protein Scy is a key component of a multiprotein assembly controlling polarized growth in Streptomyces. Proc. Natl. Acad. Sci. U S A. 110, E397-E406 (2013).
- McCormick, J. R., Su, E. P., Driks, A., Losick, R. Growth and viability of Streptomyces coelicolor. mutant for the cell division gene ftsZ. Mol. Microbiol. 14, 243-254 (1994).
- Margolin, W. FtsZ and the division of prokaryotic cells and organelles. Nat. Rev. Mol. Cell Biol. 6, 862-871 (2005).
- Grantcharova, N., Lustig, U., Flärdh, K. Dynamics of FtsZ assembly during sporulation in Streptomyces coelicolor A3(2). J. Bacteriol. 187, 3227-3237 (2005).
- Richards, D. M., Hempel, A. M., Flärdh, K., Buttner, M. J., Howard, M. Mechanistic basis of branch-site selection in filamentous bacteria. PLoS Comput. Biol. 8, e1002423 (2012).
- Hempel, A. M., Wang, S. B., Letek, M., Gil, J. A., Flärdh, K. Assemblies of DivIVA mark sites for hyphal branching and can establish new zones of cell wall growth in Streptomyces coelicolor. J. Bacteriol. 190, 7579-7583 (2008).
- Wolanski, M., et al. Replisome trafficking in growing vegetative hyphae of Streptomyces coelicolor A3(2). J. Bacteriol. 193, 1273-1275 (2011).
- Jyothikumar, V., Tilley, E. J., Wali, R., Herron, P. R. Time-lapse microscopy of Streptomyces coelicolor.growth and sporulation. Appl. Environ. Microbiol. 74, 6774-6781 (2008).
- Willemse, J., Borst, J. W., de Waal, E., Bisseling, T., van Wezel, G. P. Positive control of cell division: FtsZ is recruited by SsgB during sporulation of Streptomyces. Genes. Dev. 25, 89-99 (2011).
- Glazebrook, M. A., Doull, J. L., Stuttard, C., Vining, L. C. Sporulation of Streptomyces venezuelae. in submerged cultures. J. Gen. Microbiol. 136, 581-588 (1990).
- Bibb, M. J., Domonkos, A., Chandra, G., Buttner, M. J. Expression of the chaplin and rodlin hydrophobic sheath proteins in Streptomyces venezuelae.is controlled by sigma(BldN) and a cognate anti-sigma factor, RsbN. Mol. Microbiol. 84, 1033-1049 (2012).
- Al-Bassam, M. M., Bibb, M. J., Bush, M. J., Chandra, G., Buttner, M. J. Response regulator heterodimer formation controls a key stage in Streptomyces development. PLoS Genet. 10, e1004554 (2014).
- Bush, M. J., Bibb, M. J., Chandra, G., Findlay, K. C., Buttner, M. J. Genes required for aerial growth, cell division, and chromosome segregation are targets of WhiA before sporulation in Streptomyces venezuelae. MBio. 4, e00684-e00613 (2013).
- Tschowri, N., et al. Tetrameric c-di-GMP mediates effective transcription factor dimerization to control Streptomyces development. Cell. 158, 1136-1147 (2014).
- Mirouze, N., Ferret, C., Yao, Z., Chastanet, A., Carballido-Lòpez, R. MreB-Dependent Inhibition of Cell Elongation during the Escape from Competence in Bacillus subtilis. PLoS Genet. 11, e1005299 (2015).
- Zopf, C. J., Maheshri, N. Acquiring fluorescence time-lapse movies of budding yeast and analyzing single-cell dynamics using GRAFTS. J. Vis. Exp. (e50456), (2013).
- Meniche, X., et al. Subpolar addition of new cell wall is directed by DivIVA in mycobacteria. Proc. Natl. Acad. Sci. U S A. 111, E3243-E3251 (2014).
- Donovan, C., Schauss, A., Kramer, R., Bramkamp, M. Chromosome segregation impacts on cell growth and division site selection in Corynebacterium glutamicum. PLoS One. 8, e55078 (2013).
- Rojas, E., Theriot, J. A., Huang, K. C. Response of Escherichia coli. growth rate to osmotic shock. Proc. Natl. Acad. Sci. U S A. 111, 7807-7812 (2014).
- Flärdh, K. Essential role of DivIVA in polar growth and morphogenesis in Streptomyces coelicolor A3(2). Mol. Microbiol. 49, 1523-1536 (2003).
- Sliusarenko, O., Heinritz, J., Emonet, T., Jacobs-Wagner, C. High-throughput subpixel precision analysis of bacterial morphogenesis and intracellular spatio-temporal dynamics. Mol. Microbiol. 80, 612-627 (2011).
- Young, J. W., et al. Measuring single-cell gene expression dynamics in bacteria using fluorescence time-lapse microscopy. Nat. Protoc. 7, 80-88 (2012).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7, R100 (2006).
- Kuru, E., Tekkam, S., Hall, E., Brun, Y. V., Van Nieuwenhze, M. S. Synthesis of fluorescent D-amino acids and their use for probing peptidoglycan synthesis and bacterial growth in situ. Nat. Protoc. 10, 33-52 (2015).
- Szafran, M., et al. Topoisomerase I (TopA) is recruited to ParB complexes and is required for proper chromosome organization during Streptomyces coelicolor. sporulation. J. Bacteriol. 195, 4445-4455 (2013).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。