Method Article
Fluoreszenz Zeitraffer-Bildgebung des kompletten
In diesem Artikel
Zusammenfassung
Streptomyces are characterized by a complex life cycle that has been experimentally challenging to study by cell biological means. Here we present a protocol to perform fluorescence time-lapse microscopy of the complete life cycle by growing Streptomyces venezuelae in a microfluidic device.
Zusammenfassung
Live-cell imaging of biological processes at the single cell level has been instrumental to our current understanding of the subcellular organization of bacterial cells. However, the application of time-lapse microscopy to study the cell biological processes underpinning development in the sporulating filamentous bacteria Streptomyces has been hampered by technical difficulties.
Here we present a protocol to overcome these limitations by growing the new model species, Streptomyces venezuelae, in a commercially available microfluidic device which is connected to an inverted fluorescence widefield microscope. Unlike the classical model species, Streptomyces coelicolor, S. venezuelae sporulates in liquid, allowing the application of microfluidic growth chambers to cultivate and microscopically monitor the cellular development and differentiation of S. venezuelae over long time periods. In addition to monitoring morphological changes, the spatio-temporal distribution of fluorescently labeled target proteins can also be visualized by time-lapse microscopy. Moreover, the microfluidic platform offers the experimental flexibility to exchange the culture medium, which is used in the detailed protocol to stimulate sporulation of S. venezuelae in the microfluidic chamber. Images of the entire S. venezuelae life cycle are acquired at specific intervals and processed in the open-source software Fiji to produce movies of the recorded time-series.
Einleitung
Streptomyceten sind im Boden lebende Bakterien , die durch einen komplexen Entwicklungszyklus beteiligt morphologische Differenzierung von einem vielzelligen, nährstoff Spülung Myzel ruhend, unigenomic Sporen 1-3 gekennzeichnet sind.
Unter günstigen Wachstumsbedingungen, beginnt eine typische Streptomyces sporen durch Extrudieren von einem oder zwei Keimschläuchen (Abbildung 1) zu keimen. Diese Rohre durch Spitzenverlängerung verlängern und wachsen in einem verzweigten Hyphen Netzwerk wie das vegetative Myzel bekannt. Polar Wachstum und Hyphen Verzweigung wird durch die essentielles Protein DivIVA gerichtet. Diese Coiled-Coil - Protein ist Teil eines großen zytoplasmatischen Komplex die polarisome genannt, die für die Insertion von neuen Zellhüllenmaterial an der Spitze erstreckt 4-7 entscheidend ist. Während des vegetativen Wachstums werden die Hyphen Filamente compartmentalized durch die seltene Bildung von sogenannten Querwände 8. Die Bildung dieser Querwände wieder erfordert FtsZ, die Tubulin-ähnliche Zytoskelett - Protein , das für die Zellteilung in den meisten Bakterien 9 wesentlich ist. In Streptomyces jedoch diese vegetative Querwände führen nicht zu einer Einschnürung und Zell-Zell - Trennung und somit die Mycelmasse bleibt als ein Netz von miteinander verbundenen syncytial Abteilen. Als Reaktion auf einen Nährstoffmangel und andere Signale , die nicht gut verstanden sind, spezialisierte Lufthyphen brechen aus dem vegetativen Mycel entfernt und wachsen in die Luft 3. Die Errichtung dieser Strukturen initiiert die reproduktive Phase der Entwicklung, während der die lange Multi-genomischen werden Lufthyphen Dutzende von gleich großen unigenomic Präspore Fächer unterteilt. Diese massive Zellteilung Ereignis wird durch die synchrone Verengung mehrerer FtsZ Ringe innerhalb einzelner sporogenen Hyphen 2,10 angetrieben. Morphologische Differenzierung wird durch die Freisetzung von ruhend, dickwandig, pigmentierte Sporen abgeschlossen.
t "fo: keep-together.within-page =" 1 ">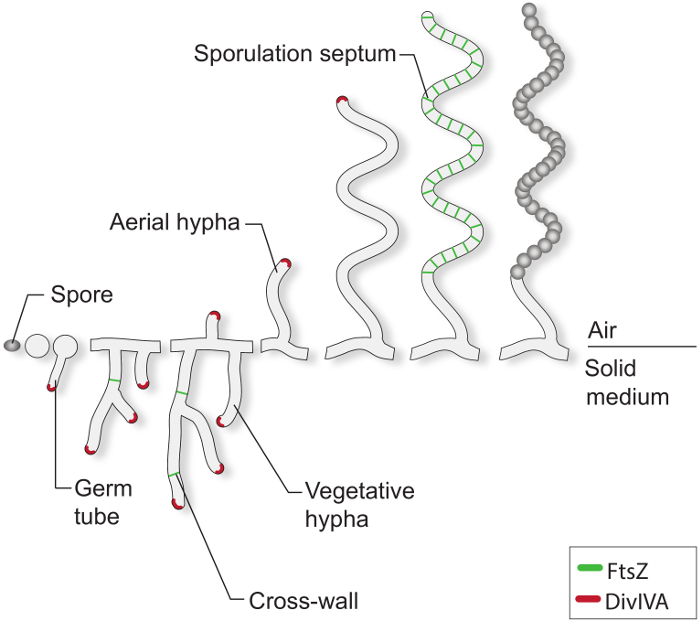
Abb . 1: Die Streptomyces - Lebenszyklus auf festen Medien Dies ist ein Modell des Lebenszyklus basiert auf der klassischen Studien von S. coelicolor auf Agarplatten wachsen. Die zelluläre Entwicklung einer Sporen beginnt mit der Bildung von einem oder zwei Keimschläuchen, die durch Spitzenverlängerung wachsen Verzweigungs Hyphen ein Netzwerk zu bilden. Polar Wachstum und Verzweigung des vegetativen Hyphen durch DivIVA (rot) gerichtet ist. Die Bildung von vegetativen Querwände erfordert FtsZ (grün). In Reaktion auf einen Nährstoffbeschränkungen und andere Signale werden Lufthyphen errichtet. Verhaftung von Luft Wachstum ist eng mit der Montage einer Leiter von FtsZ-Ringe koordiniert, die Anlass zu der Sporulation septa geben, die die sporogenen Hyphen in kastenartigen Präspore Fächer compartmentalize. Diese Abteile montieren eine dicke Spore Wand und sind schließlich Released als reife pigmentierte Sporen.
Die wichtigsten Entwicklungsereignisse des Lebenszyklus Streptomyces sind gut 1,3 charakterisiert. Was allerdings noch knapp sind zellbiologische Studien, die Fluoreszenz Zeitraffer-Mikroskopie einsetzen Einblick in die subzelluläre Prozesse zu schaffen Differenzierung zugrunde liegen, wie zum Beispiel Proteinlokalisierung Dynamik, Chromosomenbewegung und entwicklungsgesteuerte Zellteilung. Live-Cell - Imaging von Streptomyces Entwicklung ist aufgrund der Komplexität des Lebenszyklus herausfordernd und die physiologischen Eigenschaften des Organismus. Frühere Studien über das vegetative Wachstum und die Anfangsphasen der Sporulation Septierung haben sauerstoffdurchlässige Abbildungskammern oder die Agarose-gestütztes Wachstum von Streptomyces coelicolor auf einem Mikroskoptisch 11-15 eingesetzt. Diese Verfahren sind jedoch durch eine Anzahl von Faktoren begrenzt. Einige Systeme nur kurzfristige Bildgebung des Zellwachstums eine ermöglichend fluoreszierende Proteine vor Zellen leiden unter unzureichender Sauerstoffversorgung oder wachsen aus der Brennebene auf Grund der dreidimensionalen Muster von Hyphen Entwicklung. In Fällen, in denen Langzeitabbildungs möglich ist, Zellen auf Agarose pads Grenzen experimentelle Flexibilität Kultivierung, weil die Zellen nicht auf alternative Wachstums oder Stress-Bedingungen ausgesetzt werden können und die Hintergrundfluoreszenz von dem Medium in der Agarose pads schränkt die Fähigkeit schwächere Fluoreszenz zu überwachen Signale.
Hier beschreiben wir ein Protokoll für die Live-Cell - Imaging des gesamten Streptomyces Lebenszyklus mit ausgezeichneter Genauigkeit und Empfindlichkeit. Streptomyces in einer mikrofluidischen Vorrichtung verbunden mit einem Fluoreszenzmikroskop Weitfeld (Figur 2), sind wir nun in der Lage , zu überwachen Keimung vegetative Wachstum und Sporulation Septierung über einen Zeitraum von bis zu 30 h nach wächst. Dies wird stark durch die Verwendung der neuen Modellorganismus Streptomyces erleichterten venezuelae weil es sporuliert nach der Fertigstellung in Submerskultur und überwindet damit die Begrenzung des klassischen Modells Art S. coelicolor, die nur auf festen Medien 16-20 sporuliert. Um das vegetative Wachstum zu visualisieren und Sporulation wir coexprimierender FtsZ fluoreszierend markierten Versionen der Zellpolarität Marker DivIVA und der Schlüssel Zellteilungsprotein.
Wir werden mit einem handelsüblichen Mikrofluidik - Vorrichtung , die für Mykobakterien, Escherichia coli, Corynebacterium glutamicum, Bacillus subtilis und Hefe 21-25 erfolgreich eingesetzt wurde. Das System fängt Zellen in einem einzigen Brennebene und ermöglicht die Steuerung der kontinuierlichen Perfusion von Kulturmedium aus verschiedenen Reservoirs. In der detaillierten Protokoll nehmen wir die Vorteile dieser Funktion S. aussetzen venezuelae vegetative Myzel Nahrungsherunterschalten Sporulation zu fördern.
Das Protokoll deschrieben ist für Live-Cell - Imaging des gesamten Lebenszyklus Streptomyces, sondern alternative Medienbedingungen oder Mikroskopeinstellungen können , wenn bestimmte Entwicklungsstadien von besonderem Interesse sind , gewählt werden.
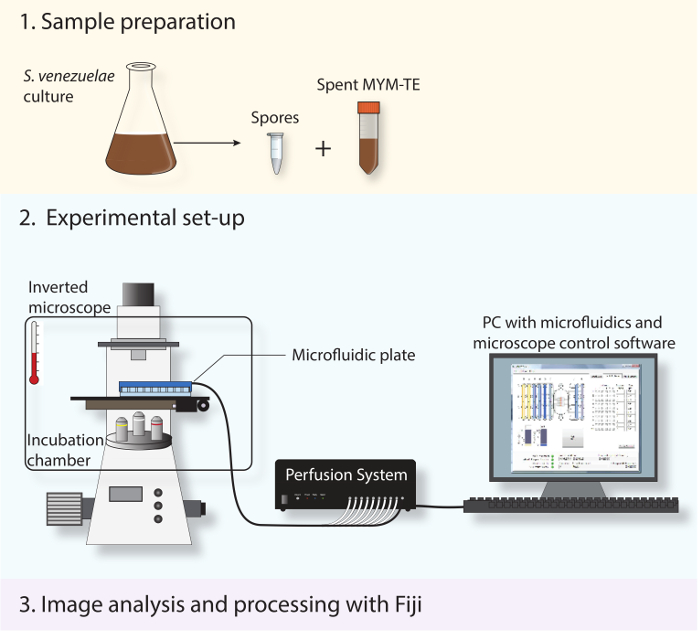
Abbildung 2: Schematische Darstellung der experimentellen Arbeitsablauf. Die drei wichtigsten Schritte in dem Protokoll beschrieben sind, gezeigt. Erstens, Sporen und verbrauchte Medium aus einer stationären Phase Kultur vorbereitet. Zweitens werden die frischen Sporen in einem mikrofluidischen System und S. geladen venezuelae ist über seine gesamte Entwicklungslebenszyklus unter Verwendung eines vollautomatischen invertiertes Mikroskop mit einer Inkubationskabine abzubildenden eine optimale Wachstumstemperatur aufrechtzuerhalten. Drittens ist die Zeitraffer-Serie erhalten analysiert und verarbeitet unter Verwendung der Open-Source-Software Fidschi.
Protokoll
1. Isolierung von Frische S. venezuelae Spores und Vorbereitung der Behandlung abgebrannter Kulturmedium
- Impfen 30 ml Maltose-Hefeextrakt-Malzextrakt (MYM) Medium, ergänzt mit 60 & mgr; l R2 Spurenelementlösung (TE), mit 10 & mgr; l Sporen des Stammes SV60 [attB φ BT1 :: pSS209 (P divIVA - divIVA-mCherry P ftsZ -ftsZ-ypet)]. Für eine konsistente Wachstum und die Sporenbildung der Zellen, mit einem Schikanekolben oder einen Kolben, der eine Feder für eine ausreichende Belüftung zu ermöglichen, enthält.
- Kulturzellen für 35-40 h bei 30 ° C und 250 Umdrehungen pro Minute.
- Untersuchen Sie die Zellen, die durch Flüssigkeit montiert Phasenkontrast-Mikroskopie. An diesem Punkt Myzelfragmenten und Sporen sichtbar.
- Zentrifuge 1 ml Zellen in einer Tischzentrifuge bei 400 · g für 1 min das Mycel und größere Zellfragmente zu pelletieren.
- Übertragen etwa 300 & mgr; l Überstand containing eine Suspension von Sporen in ein neues 1,5-ml-Tube und auf Eis halten. Halten Sie die verbleibende Kulturmedium für Schritt 1.7.
- Verdünnen Sporen 01.20 in MYM-TE (Endkonzentration: 0,5-5 x 10 7 Sporen / ml) und auf Eis halten , bis sie benötigt (siehe Schritt 2.3).
Hinweis: Obwohl die genauen Bedingungen , die Sporulation in Streptomyces auslösen nicht verstanden werden, mit verbrachte MYM-TE Medium, einschließlich aller unbekannten extrazelluläre Signale von einer sporulierenden Kultur, stimuliert Sporulation. - Filter-sterilisieren 10 ml des übrigen Kulturmedium zu MYM-TE verbrachte erhalten, die frei von Sporen und Myzelfragmenten ist. Verwenden Sie eine sterile 0,22 & mgr; m Spritzenfilter und Übertragung ausgegeben MYM-TE in ein steriles 15 ml Röhrchen.
- Halten Sie die vorbereitet verbrachte MYM-TE für ein paar Tage bei 4 ° C, wenn weitere Experimente ähnliche Wachstumsbedingungen unter Verwendung von durchgeführt werden sollen.
2. Herstellung der mikrofluidischen Vorrichtung
Hinweis: Jedes mikrofluidischen plate ermöglicht bis vier unabhängigen Experimenten bis zu werden, durchgeführt. Um eine Verunreinigung der nicht verwendeten Strömungskammern zu vermeiden, verwenden sterile Lösungen und Arbeitsbedingungen, wenn ein Experiment einrichten.
- Entfernen Versandlösung aus der mikrofluidischen Platte und spülen Sie die Vertiefungen mit sterilem MYM-TE.
- In 300 ul MYM-TE zum Einlass gut 1 und 300 & mgr; l verbrachte MYM-TE in die Vertiefungen 2 bis 6.
- Last 40 ul verdünnt Sporen aus Schritt 1.6 in Zelle Laden gut 8 der Spur "A" und die Dichtung der Verteiler an der Platte gemäß Herstelleranweisungen.
- Starten Sie die Mikrofluidik Steuerungssoftware, wählen Sie die entsprechende Plattentyp und legen ein Flussprogramm prime den Strömungskanal und Kulturkammer durch strömende Medium aus dem Einlass Brunnen 1 bis 5 bei 6 psi für 2 min pro Vertiefung.
- In der Mikrofluidik Steuersoftware ein Fluss Protokoll für das Experiment zu erstellen: MYM-TE vom Einlass und 1 für 6 h fließt für die Keimung und das vegetative Wachstum der Zellen zu ermöglichen. Schalterdann Perfusion von verbrauchtem MYM-TE von gut 2 bis 5 für die restliche Zeit des Experiments. Während des Verlaufs des Experiments halten Strömungsdruck konstant bei 6 psi (entspricht etwa 9 & mgr; l Medium / h). Starten Sie Flow-Programm nach dem Schritt 3.5.
3. Set Up des Mikroskops und der Zeitraffer-Protokoll
Hinweis: Diese Methode wurde implementiert, auf einem voll motorisierten und automatisierten inversen Weitfeld- Mikroskop ausgestattet mit einer sCMOS Kamera, einem Metall-Halogen-Lampe, einem Hardware-Autofokus, eine Bühne Halter für 96-Well-Platten und einer Klimakammer.
- Vorwärmen der Klimakammer auf 30 ° C im Voraus, um Probleme mit dem Autofokus nach dem Start des Experiments zu verhindern. Die erforderliche Zeit hängt von der Umgebungskammer verwendet wird, das Mikroskop und das Heizungssystem. Starten Sie das System in der Nacht vor dem Experiment zu erwärmen.
- Schalten Sie das Mikroskop und Mikroskopie Automatisierung und Steuerung softwaRe. Verwenden Sie eine hohe numerische Apertur (NA) -Ölimmersionsobjektiv für eine optimale Signalerfassung und räumliche Auflösung, wie ein 100X, 1,46 NA Öl DIC Ziel. Wählen Sie den entsprechenden Filter und dichromatic Spiegel Differentialinterferenzkontrast (DIC) Bilder und Bilder von gelb-fluoreszierende und rot-fluoreszierende Protein-Fusionen zu erwerben.
- Einen Tropfen Immersionsöl auf das Objektiv und fügen Immersionsflüssigkeit auf den Boden des Abbildungsfenster auf der mikrofluidischen Platte Zellfokus während der Bilderfassung zu verbessern. Halterung sorgfältig die abgedichtete Mikrofluidikvorrichtung (Schritt 2.5) auf die Phase des umgekehrten Mikroskops. Stellen Sie sicher, dass die Platte sicher in der Objektträgerhalter angeordnet ist, und nicht über den Verlauf des Experiments nicht verschiebt.
- Bringen Sie das Abbildungsfenster der mikrofluidischen Kulturkammer in den Fokus für die Orientierung der eingebetteten Position Marker. Konzentrieren sich auf die am weitesten links liegende Teil des ersten Strömungskammer (bezeichnet mit "A") mit der Falle 5 Größe, entsprechendeine Falle Höhe von 0,7 um.
- In der Mikrofluidik-Software, Wägezellen vom Einlass gut 8 bei 4 psi für 15 Sekunden. Überprüfen Sie die Zelldichte in der Kulturkammer von der Bühne über dem Abbildungsfenster bewegt. Wenn keine Sporen gefangen wurden, wiederholen Sie den Schritt Zelle Laden oder alternativ den Ladedruck zu erhöhen und / oder die Zeit, bis die gewünschte Zelldichte erreicht wird (1-10 Sporen pro Abbildungsfenster mit 2.048 x 2.048 Pixel). Vermeiden Sie die Kulturkammer zu überlasten.
Hinweis: Wir normalerweise Falle Größe 5 für die Abbildung Streptomyces verwenden, aber wir haben auch gute Ergebnisse mit Falle erhalten Größen 4 und 3. Ziehen Sie die Bedienungsanleitung des Lieferanten für weitere Vorschläge zur Optimierung des Zellenladevorgang. - Starten Sie die Strömungsprogramm in der Steuersoftware aus Schritt 2.5 und ermöglichen die mikrofluidische Platte für 1 h in dem Mikroskoptisch aufzuheizen äquilibrieren, bevor die Bildaufnahme beginnt.
- In der Mikroskopsteuerung Software, stellen Sie eine mehrdimensionale Akquisition bis mu zu nehmenltiple Bilder in mehreren Stufen Positionen im Laufe der Zeit:
- Für Automatisches Speichern: Verzeichnis für die automatische Speicherung der Bilddateien angeben.
- Für Beleuchtungseinstellungen bestimmen die optimale Beleuchtungseinstellungen für jedes spezifische Konstrukt im Voraus. Für das skizzierte Experiment, verwenden Sie die folgenden Belichtungszeiten: DIC 150 msec, YFP 250 ms, RFP 100 ms.
- For Time-Serie: eine Zeitreihe eingerichtet, um Bilder zu den gewünschten Zeitpunkten in Folge gewinnen. Für die Abbildung des Lebenszyklus von S. venezuelae, wählen Sie eine 40 min Zeitintervall über einen Zeitraum von 24 Std.
- Für Bühnenpositionen und Autofokus: Scannen der Kulturkammer von der Bühne und Speicherstufe Positionen für jede Abbildungsposition von Interesse zu bewegen. Stellen Sie sicher, dass die einzelnen Tischpositionen ausreichend voneinander entfernt sind, zu minimieren Photobleaching und phototoxicity.Typically bis 12 Positionen verbrauchen. Führen Sie Autofokus-Routine für jeden Zeitpunkt für langsame Brenn Drift korrigieren. Wenn in dem Mikroskop SteuerungssoftwareStrategie, setzen Autofokus auf "lokale Oberflächen Update von Hardware-Autofokus". Sobald die Z-Koordinaten der ausgewählten Stufe Positionen überprüft werden, aktivieren Sie die Hardware-Autofokus.
- Starten Sie den Zeitraffer-Experiment im Mikroskop Steuerungssoftware.
- Überprüfen Sie, ob alle Tischpositionen in späteren Punkte noch im Fokus sind. Für Zeitraffer-Experimente über mehrere Stunden laufen, beobachten wir gelegentlich eine Bühne Drift, auch wenn Autofokus. Wenn Stufe Positionen werden neu ausgerichtet benötigen, zu einem geeigneten Zeitpunkt den Versuch zu stoppen, um den Fokus einzustellen und das Experiment innerhalb des definierten Abbildungszeitintervall (Schritt 3.7.3) neu zu starten. Siehe Schritt 4.3, wie die Zeitreihen zu verketten.
- Stoppen Bildaufnahme nach 24-30 h oder wenn die Hyphen in der Region von Interesse in Sporen unterschieden haben (siehe DIC Bild-Serie). Stop-Fluß-Programm in der Software und zu demontieren die Mikrofluidik-Vorrichtung.
- Bereiten Sie gebrauchte mikrofluidischen Platte für kurzfristige stoRage. Entfernen restlichen MYM-TE und verbrachte MYM-TE von gut 1 bis 6, leeren Abfall gut 7 und Zellenbelastung gut 8. Unter sterilen Bedingungen wieder füllen die Vertiefungen Spur "A" verwendet und Brunnen von ungenutzten Spuren ( "B" "D") auf der Platte mit steriler Phosphat-gepufferter Saline (PBS). Verschließen Sie die Platte mit Parafilm zu verhindern, dass sie vor dem Austrocknen und lagern bei 4 ° C.
4. Erzeugung von Zeitraffer-Filmen mit Fidschi Software
Hinweis: Wir stellen fest, dass verschiedene kommerzielle und freie Softwarepakete zur Verfügung Zeitraffer-Mikroskopie-Aufnahmen einschließlich ZenBlue, Metamorph, ICY, ImagePro oder ImageJ zu verarbeiten. Hier konzentrieren wir uns auf Fidschi, die eine Open-Source-Bildbearbeitungsprogramm auf Basis von ImageJ, und die bereits bietet eine Reihe von nützlichen vorinstallierten Plugins.
- Transfer Imaging-Daten aus Ihrem Zeitraffer Experiment auf einen Computer, Fidschi installiert hat.
- Starten Sie Programm und importieren Sie die Bilddatei mit &# 34; Datei> Importieren> Bio-Formate ". Oder einfach durch die Imaging-Datei in Fidschi Ziehen im" Bio-Formate Import-Optionen ", markieren Sie das Kästchen" aufgeteilt Kanäle "ein separates Bildstapel für jeden Beleuchtungskanal zu erhalten (DIC , RFP, YFP).
- Optional: getrennte Zeitreihen zu fusionieren von einer Pause in der im Bild-Akquisition durch Refokussierung (Schritt 3.9), öffnen jeweiligen Bildstapel und führen Sie "Bild> Stacks> Tools> verketten" für jeden Kanal (DIC, RFP, YFP ).
- Bewerten Sie die Qualität der Zeitraffer-Daten, die von durch die Bildstapel blättern. Suchen Sie nach Hyphen, die im Fokus im Laufe der Zeit zeigen Myzel-Wachstum blieb und schließlich bilden Sporen (DIC-Stack). Untersuchen Sie die erwartete subzelluläre Lokalisierung für DivIVA-mCherry bei Hyphenspitzen (RFP-Stack) und FtsZ-YPet Einzelringe oder mehrere, leiterartige Strukturen in vegetative oder sporogenen Hyphen (YFP-Stack) bilden sind.
- Isolieren einer Region von Interesse für nachgeschaltete processing der Bilder. Zeitraffer-Mikroskopie erzeugt oft große Dateien, die die Verarbeitung verlangsamen kann durch Fidschi. Es ist daher eine Region of Interest (ROI) zu identifizieren und zu isolieren empfohlen und weitere Bildverarbeitungsschritte auf dieser kleinere Version des Bildstapels auszuführen.
- Wählen Sie die "Auswahlrechteck" Werkzeug im Menü und ziehen Sie einen rechteckigen ROI in dem DIC Bildstapel derart, dass die Entwicklung von Hyphen von der ROI im gesamten Bild Serie eingeschlossen sind.
- Duplizieren ROI-Stack mit "Strg" + "Shift" + "D".
- Klicken Sie auf den Namen Leiste des YFP Bildstapel und wählen Sie "Bearbeiten> Auswahl> Wiederherstellungsauswahl" im Menü mit der vorherigen rechteckige Auswahl aus dem ursprünglichen DIC wiederherzustellen Stack auf den gleichen positon in der YFP-Stack und duplizieren Sie die ROI mit "Strg" + "Shift" + "D".
- Wiederholen Sie diesen Vorgang für den RFP-Stack.
- Richten Sie die Bilder in den drei Bildstapeln to Stufe Drifts in der xy-Ebene im Laufe der Zeit zu entfernen. Blättern Sie zum letzten Bild in der DIC-Stapel und wählen Sie "Plugin> Registrierung> STACKREG> RigedBody" aus dem Menü. Wiederholen Sie diesen Schritt für die RFP und YFP Bildstapel. Bei Bedarf zuschneiden, den ROI durch 4.5.1-4.5.3 Schritt zu wiederholen.
- Optional: Stellen Sie die Helligkeit und den Kontrast manuell für jeden Bildstapel mit der "Helligkeit und Kontrast einstellen Tool" ( "Strg" + "Shift" + "C") oder wählen Sie "Prozess> Kontrast verbessern" aus dem Menü und wenden Sie die "Normalisieren" Befehl an alle Bilder in dem Stapel. Es sei darauf hingewiesen, dass dieser Befehl Pixelwerte verändern wird und somit nachgeschaltete Bildanalyse beeinträchtigen können.
- Optional: Kombinieren Sie einzelne Stapel (in den RGB-Dateiformat konvertiert, siehe 4.11) horizontal oder vertikal über das Plugin "Bild> Stacks> Tools> Kombinieren".
- Fügen Sie eine Maßstabsleiste ( "Analysieren> Tools> Scale-Bar") und eine Zeit Stempel (&# 34; Bild> Stacks> Zeit Stamper ").
- Speichern modifizierte Bildstapeln als Bildsequenz in der "Tiff" Format. Um einen Film zu produzieren, speichern als ".avi". Alternativ Export Bildserien im Quicktime-Format, die "Bio-Formate> Bio-Formate Exporter" Plugin und wählen Sie die ".mov" Dateityp verwendet wird.
- Um Bilder aus Einzelbildern zu erhalten, wählen Sie den Rahmen von Interesse und duplizieren das Bild "Strg" + "Shift" + "D". Ändern Sie den Typ des resultierenden Bildes auf "RGB" oder "8-Bit" unter "Bild> Typ> RGB-Farbe oder 8-Bit" und speichern Sie das Bild als "Tiff".
Hinweis: Fidschi bietet eine Reihe von Zusatzfunktionen weiter zu kommentieren oder Prozess Zeitraffer-Serie. Zum Fidschi Online-Support für weitere Informationen (http://fiji.sc/Fiji).
Ergebnisse
Die erfolgreiche Live-Cell - Imaging des gesamten S. venezuelae Lebenszyklus ergibt sich eine kontinuierliche Zeitreihen einschließlich der wichtigsten Entwicklungsstadien der Keimung, das vegetative Wachstum und die Sporenbildung (Abbildung 3, Film 1). Visualisierung der Progression durch den Lebenszyklus wird durch die unterschiedliche subzelluläre Lokalisation von DivIVA-mCherry und FtsZ-YPet verbessert. Während der Keimung und des vegetativen Wachstums, DivIVA-mCherry sammelt sich ausschließlich an den wachsenden Hyphenspitzen oder neu Marken bilden Verzweigungspunkte (4A). Diese Ergebnisse stehen im Einklang mit den zuvor berichteten subzellulärer Positionierung von DivIVA 12,26. Im Gegensatz dazu bildet FtsZ-YPet einzelne ringartige Strukturen in unregelmäßigen Abständen in der wachsenden Mycel (4B). Diese Strukturen bieten das Gerüst für die Synthese von nicht-constrictive vegetative Querwänden, die zur Bildung von interconnektiert Hyphen Fächer 8. Die zelluläre Differenzierung Hyphen in sporogenen Hyphen wächst durch das Verschwinden von polaren DivIVA-mCherry Brennpunkte, die Verhaftung von polaren Wachstum und die damit einhergehende Erhöhung der FtsZ-YPet Fluoreszenz (4C) sichtbar wird. In sporulierendem Hyphen, ändert sich die Lokalisierungsmuster von FtsZ-YPet dramatisch; erste Schrauben FtsZ-YPet Filamente entlang der Hyphen im Trockner und dann, in einem plötzlichen, fast synchrone Ereignis, diese Helices verbinden sich zu einer Leiter in regelmäßigen Abständen FtsZ-YPet Ringen. Unter den hier beschriebenen Versuchsbedingungen bestehen diese gleichmäßig verteilt FtsZ-YPet Leitern für ca. 2 Std. Schließlich erkennbar werden Sporulation Septen in den differentiellen Interferenzkontrast (DIC) Bilder (4D) und schließlich werden neue Sporen freigesetzt.
Das nachfolgende Protokoll beschreibt die Bildverarbeitung, die Fidschi-Software bietet als tep-für-Schritt-Erklärung, wie ein Film für die Veröffentlichung aus dem erfassten Zeitserien (Film 1) zu erzeugen.
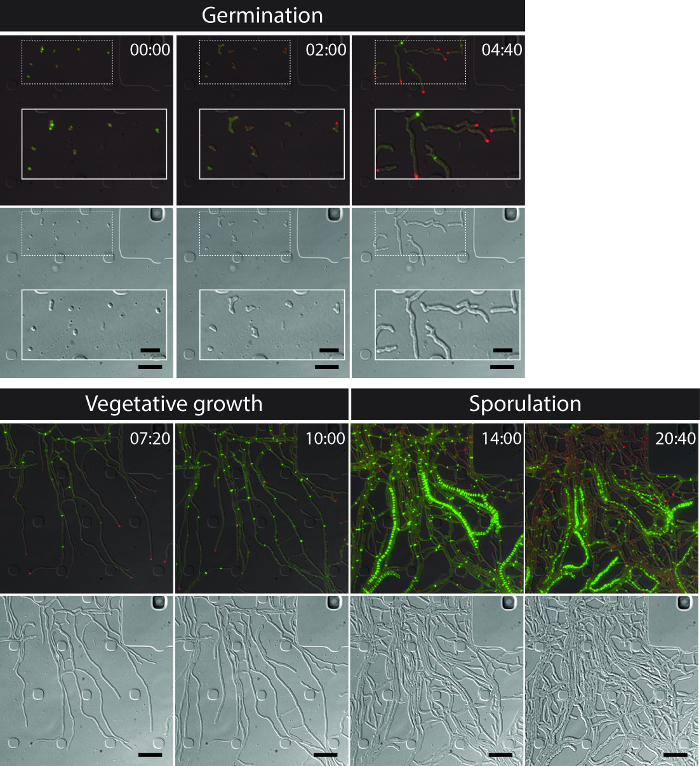
Abbildung 3: Snapshot Bilder aus einer repräsentativen Zeitraffer-Fluoreszenzmikroskopie Reihe von S. venezuelae Herstellung von fluoreszenzmarkierten FtsZ (grün) und DivIVA (rot) Gezeigt werden ausgewählte Bilder von fusionierten Bilder (oben: RFP-YFP-DIC, Bodenplatte: DIC). des Streptomyces - Lebenszyklus einschließlich der Keimung, das vegetative Wachstum und die Sporenbildung zu visualisieren. Die Bilder wurden von der Film 1. Zeit genommen ist in h: min. Maßstabsbalken = 5 & mgr; m. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
d / 53863 / 53863fig4.jpg "/>
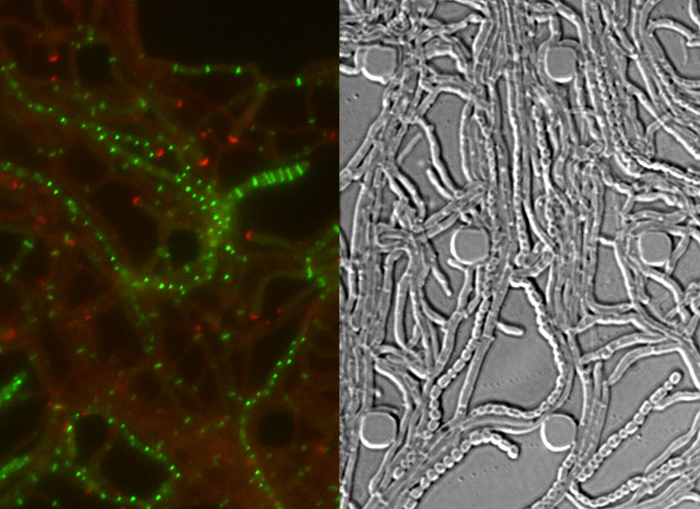
Abbildung 4: Repräsentative Ergebnisse für eine erfolgreiche Zeitraffer-Serie des Streptomyces - Lebenszyklus (A) Lokalisierung von DivIVA-mCherry.. Dargestellt sind Schnappschüsse von zwei aufeinander folgenden Zeitpunkten von der Film 1 mit fusionierten Bilder des RFP und DIC-Kanäle. DivIVA-mCherry markiert neue Hyphen Verzweigungsstellen (gefüllte Pfeilspitze) und lokalisiert an der Hyphen Spitze polaren Wachstums (offene Pfeilspitze) zu lenken. Maßstabsbalken = 10 & mgr; m (B) FtsZ-YPet Lokalisation während des vegetativen Wachstums (Pfeilspitze). FtsZ-YPet bildet einzelne ringartige Strukturen (oberes Feld), die einengen nicht (DIC, unteres Bild). (C) FtsZ-YPet (grün) Lokalisation während der Sporulation Septierung. DivIVA-mCherry Brennpunkte sind rot dargestellt, mit unten dargestellt verstrichene Zeit (hr: min). Maßstabsbalken: 5 um. (D) Entsprechende DIC Bilder des sporulierenden Hyphen von (C), die die Formation von Präspore Fächer mit sichtbaren Sporulation Septen (linkes Bild), die schließlich in eine Kette von Sporen (rechtes Bild) reifen. Die Zeit ist in h: min. Maßstabsbalken = 5 & mgr; m. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
Film 1: Zeitraffer-Fluoreszenzmikroskopie Reihe der Streptomyces venezuelae Lebenszyklus Der Film besteht aus fusionierten RFP-YFP Bilder (links) und die entsprechenden differentiellen Interferenzkontrast (DIC) Bilder (rechts).. DivIVA-mCherry ist in rot und FtsZ-YPet in grün dargestellt. Das Zeitintervall zwischen den einzelnen Frames beträgt 40 min. Maßstabsbalken = 10 & mgr; m. Bitte klicken Sie hier thi zu sehens Film.
Diskussion
Zeitraffer-Mikroskopie des Streptomyces - Lebenszyklus in der Vergangenheit technisch anspruchsvoll gewesen. Hier präsentieren wir eine robuste Protokoll Abbildung lebender Zellen des gesamten Lebenszyklus unter Verwendung von fluoreszierenden Protein - Fusionen auf die Zellpolarität Marker DivIVA und der Zellteilungsprotein FtsZ ausführen zu helfen , zu visualisieren und zu verfolgen , die Progression durch das Entwicklungsprogramm (Abbildung 2).
Im Mittelpunkt dieser Methode ist der Anbau von S. venezuelae in einer Mikrofluidik - Vorrichtung , die eine konstante Mediumperfusion und den Austausch von normalem Wachstumsmedium (MYM-TE) mit verbrauchtem Medium ermöglicht (verbrauchte MYM-TE). Die Veränderung der Kulturbedingungen ist wichtig , auf das beschriebene Protokoll , da die Ernährungsherunterschalten als Brunnen als irgendwelchen noch nicht identifizierten extrazelluläre Signale (zB Quorum Sensing - Signale) in dem Kulturmedium verbracht, Sporenbildung zu fördern, während eine kontinuierliche Versorgung mit nährstoffreichen Medium vegetative growt stimulierth mit kaum Sporenbildung (Daten nicht gezeigt). Somit ist in diesem mikrofluidischen System Kultivierung von Zellen überlegen Zellen auf Agarose-Kultivierung, weil es mehr experimentelle Flexibilität bietet und ermöglicht langfristige Überwachung von Veränderungen der Bakterienwachstum in Reaktion auf die Kulturbedingungen zu verändern. Obwohl die Mikrofluidik-Platten nur für den einmaligen Gebrauch bestimmt sind, Strömungskanäle, die mit Zellen, die nicht sind, können in den nachfolgenden Experimenten verwendet geimpft haben werden. Wir empfehlen, alle Kanäle einer mikrofluidischen Platte innerhalb einer Woche mit, wie wir Probleme erlebt haben, um den Verteiler zu Dichtungsplatten, die für längere Zeiten geöffnet wurden.
Wenn ein Experiment einrichten, fanden wir, dass frisch zubereitete innerhalb von zwei Stunden keimten die Sporen der in die Strömungskammer geladen wird, während aus einem gefrorenen Glycerolstammlösung abgeleitet Sporen mindestens 6 Stunden erforderlich, bevor Keimschläuche entstanden (Daten nicht gezeigt). Diese Verzögerung der Keimung verlängert die Länge des Experiments und können interferierenAnlagenverfügbarkeit und experimentellen Bedingungen. Es ist auch wichtig, das Experiment mit Perfusion von MYM-TE für mindestens 3 h zu starten ausreichend Nährstoffe für die Entwicklung und das Auswachsen von Keimschläuchen zu schaffen. Perfusion mit MYM-TE kann über die ursprüngliche 3 h verlängert, wenn das Experiment ausgeführt ist vegetative Wachstum zu untersuchen. Während Sporen die bevorzugte Wahl des Ausgangsmaterials zur Verfügung stellen, können kurze Hyphenfragmente auch in der mikrofluidischen Platte geladen werden. Wenn jedoch die Ladeeffizienz geschert Myzel mit Hilfe ist signifikant geringer und erfordert oft mehrere Belastungsdurchläufe, die eine Einschränkung darstellen kann, wenn nicht-sporenbildende Mutanten zu untersuchen. Unabhängig von der Art des Inokulums verwendet wird, ist es wichtig, die Kulturkammer zu überlasten mit Sporen oder hyphalen Fragmenten, da dies zu einer schnellen Überbelegung führen, die mit Medien-Diffusions stören kann und Bildanalyse erschweren.
Es ist wichtig, die Konstruktion von Reporterproteine zu planen carefully für Zeitraffer-Bildgebung Verwendung in Fluoreszenz in Streptomyces. Linkerlänge zwischen dem Zielprotein und dem Fluoreszenzreporter und die Wahl der N- oder C-terminale Fusionen können kritisch sein. Außerdem optimalen Versuchsbedingungen, einschließlich der Abbildungsfrequenz und Belichtungszeit, müssen für jedes fluoreszenzmarkierten Protein im Voraus bestimmt werden. S. venezuelae Hyphen zeigen Autofluoreszenz im grün / gelb - Kanal, der problematisch werden kann , wenn eine fluoreszenzmarkierte Protein Bildgebung , die in geringen Mengen exprimiert wird. Zusätzlich sollte es bemerkt werden , dass, während der Keimung und dem anfänglichen vegetative Wachstumsphase S. venezuelae ist besonders empfindlich für kurzwelliges Licht (zB bei der Protein - Fusionen zu CFP verwenden).
Trotz der kontinuierlichen Fortschritte in der bildgebenden Verfahren und fluoreszierenden Reportersysteme für Live-Cell - Imaging von Bakterien, die meisten Software - Pakete (zB MicrobeTracker, Schnitzelcells, CellProfiler) für die nachfolgende automatisierte Verarbeitung dieser Art von Datensätzen nicht die Analyse von 27-29 aus filamentösen Bakterien mit einem vielzelligen Lebensstil abgeleiteten Bilder unterstützen. Somit besteht ein Bedarf , einen geeigneten Algorithmus für die quantitative Analyse mit hohem Durchsatz von Bilddaten von Streptomyces und andere filamentöse Bakterien zu entwickeln.
Zusammenfassend beschrieb die Arbeit hier zeigt das immense Potenzial von S. venezuelae als neues Modell Entwicklungssystem für die Gattung, wegen seiner Fähigkeit , in Flüssigkeit zu sporulieren. Die Mikrofluidik-Kultivierungsvorrichtung ist einfach auch für ungeübte Anwender zu verwenden. Es bietet eine hervorragende Plattform , zellbiologische Prozesse im Mittelpunkt des Streptomyces - Lebenszyklus, einschließlich dynamischer Proteinlokalisierung, polarisierten Wachstum, und die morphologische Differenzierung eines vielzelligen Mycel in Ketten von unigenomic Sporen zu studieren. Darüber hinaus setzen diese experimentellen up stellt auch eine verlockende Ausgangspunkt , um Ereignisse in der Entwicklung zu untersuchen , die Kulturbedingungen erfordern abwechseln, oder die Verwendung von Fluoreszenzfarbstoffen wie fluoreszierende D-Aminosäuren Peptidoglycansynthese oder Propidiumiodid und 4 'zu überwachen, 6-diamidino-2-phenylindole (DAPI) Chromosomenorganisation 30,31 sichtbar zu machen.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
The authors thank Grant Calder for technical assistance with the microscope, Matt Bush for comments on the protocol, and the John Innes Centre for purchase of the Zeiss widefield microscope. This work was funded by BBSRC grant BB/I002197/1 (to M.J.B), by BBSRC Institute Strategic Programme Grant BB/J004561/1 to the John Innes Centre, by Swedish Research Council grant 621-2010-4463 (to K.F.), and by a Leopoldina Postdoctoral Fellowship (to S.S.).
Materialien
| Name | Company | Catalog Number | Comments |
| B04A CellAsic ONIX plate for bacteria cells | Merck-Millipore | B04A-03-5PK | Microfluidic culture plates |
| CellAsic ONIX Microfluidic Perfusion System and ONIX FG (version 5.0.2) | Merck-Millipore | EV-262 | The latest ONIX vesion (July 2015) and instructions on how to use the programme can be found here: http://www.merckmillipore.com |
| Axio Observer.Z1 Microscope | Zeiss | 431007-9902-000 | Fully automated and motorized inverted widefield microscope |
| Incubator XL multi S1 with Temperature Module S1 and Heating unit XL S2 | Zeiss | 411857-9061-000 | Environmental chamber surrounding the microscope |
| Plan-Apochromat 100x/1.46 Oil DIC objective | Zeiss | 420792-9800-000 | |
| Ocra FLASH 4 V2 | Hamamatsu Photonics K.K. | C11440-22CU | |
| Illuminator HXP 120V | Zeiss | 423013-9010-000 | |
| FL Filter Set 46 HE YFP shift free | Zeiss | 489046-9901-000 | Fluorescent filter set, excitation 500/25 nm, emission 535/30 nm |
| FL Filter Set 63 HE RFP shift free | Zeiss | 489063-0000-000 | Fluorescent filter set, excitation 572/25 nm, emission 629/30 nm |
| Mounting frame K-M for multiwell plates | Zeiss | 000000-1272-644 | Stage holder for microfluidic plate |
| ZEN pro 2012 | Zeiss | 410135-1002-120 | Microscope control software |
| ZEN Module Time Lapse | Zeiss | 410136-1031-110 | Software module to set up time-lapse microscopy experiments |
| ZEN Module Tiles/Positions | Zeiss | 410136-1025-110 | Software module to save specific stage positions (xzy) |
| Fiji | open-source software package | http://fiji.sc/Fiji | Generation of time-lapse movies |
| Maltose-Yeast Exctract-Malt Extract (MYM) 4 g Maltose 4 g Yeast extract 10 g Malt extract add 1 L H2O using 50 % tap water and 50 % reverse osmosis water and supplement with 200 ml of R2 trace element solution per 100 ml after autoclaving | Sporulation medium used to culture S. venezuelae SV60 | ||
| R2 Trace element solution (TE) 8 mg ZnCl2 40 mg FeCl3-6H2O 2 mg CuCl2-2H2O 2 mg MnCl2-4H2O 2 mg Na2B4O7-10H2O 2 mg (NH4)6Mo7O24-4H2O add 200 ml H2O Autoclave and store at 4 oC | Add 0.002 volumes to MYM | ||
| PBS (phosphate buffered saline) | Sigma | P4417-100TAB | Used to refill inlet wells of unused lanes in B04A plates in order to prepare plate for short-term storage. |
| 0.22 µm syringe filters | Satorius stedim | 16532-K | Preparation of spent MYM-TE |
| SV60 | John Innes Centre strain collection | S. venezuelae strain expressing divIVA-mcherry and ftsZ-ypet |
Referenzen
- Bush, M. J., Tschowri, N., Schlimpert, S., Flärdh, K., Buttner, M. J. c-di-GMP signalling and the regulation of developmental transitions in streptomycetes. Nat. Rev. Microbiol. 13, 749-760 (2015).
- Jakimowicz, D., van Wezel, G. P. Cell division and DNA segregation in Streptomyces.: how to build a septum in the middle of nowhere?. Mol. Microbiol. 85, 393-404 (2012).
- McCormick, J. R., Flärdh, K. Signals and regulators that govern Streptomyces development. FEMS Microbiol. Rev. 36, 206-231 (2012).
- Flärdh, K., Richards, D. M., Hempel, A. M., Howard, M., Buttner, M. J. Regulation of apical growth and hyphal branching in Streptomyces. Curr. Opin. Microbiol. 15, 737-743 (2012).
- Hempel, A. M., et al. The Ser/Thr protein kinase AfsK regulates polar growth and hyphal branching in the filamentous bacteria Streptomyces. Proc. Natl. Acad. Sci. U S A. 109, E2371-E2379 (2012).
- Fuchino, K., et al. Dynamic gradients of an intermediate filament-like cytoskeleton are recruited by a polarity landmark during apical growth. Proc. Natl. Acad. Sci. U S A. 110, E1889-E1897 (2013).
- Holmes, N. A., et al. Coiled-coil protein Scy is a key component of a multiprotein assembly controlling polarized growth in Streptomyces. Proc. Natl. Acad. Sci. U S A. 110, E397-E406 (2013).
- McCormick, J. R., Su, E. P., Driks, A., Losick, R. Growth and viability of Streptomyces coelicolor. mutant for the cell division gene ftsZ. Mol. Microbiol. 14, 243-254 (1994).
- Margolin, W. FtsZ and the division of prokaryotic cells and organelles. Nat. Rev. Mol. Cell Biol. 6, 862-871 (2005).
- Grantcharova, N., Lustig, U., Flärdh, K. Dynamics of FtsZ assembly during sporulation in Streptomyces coelicolor A3(2). J. Bacteriol. 187, 3227-3237 (2005).
- Richards, D. M., Hempel, A. M., Flärdh, K., Buttner, M. J., Howard, M. Mechanistic basis of branch-site selection in filamentous bacteria. PLoS Comput. Biol. 8, e1002423 (2012).
- Hempel, A. M., Wang, S. B., Letek, M., Gil, J. A., Flärdh, K. Assemblies of DivIVA mark sites for hyphal branching and can establish new zones of cell wall growth in Streptomyces coelicolor. J. Bacteriol. 190, 7579-7583 (2008).
- Wolanski, M., et al. Replisome trafficking in growing vegetative hyphae of Streptomyces coelicolor A3(2). J. Bacteriol. 193, 1273-1275 (2011).
- Jyothikumar, V., Tilley, E. J., Wali, R., Herron, P. R. Time-lapse microscopy of Streptomyces coelicolor.growth and sporulation. Appl. Environ. Microbiol. 74, 6774-6781 (2008).
- Willemse, J., Borst, J. W., de Waal, E., Bisseling, T., van Wezel, G. P. Positive control of cell division: FtsZ is recruited by SsgB during sporulation of Streptomyces. Genes. Dev. 25, 89-99 (2011).
- Glazebrook, M. A., Doull, J. L., Stuttard, C., Vining, L. C. Sporulation of Streptomyces venezuelae. in submerged cultures. J. Gen. Microbiol. 136, 581-588 (1990).
- Bibb, M. J., Domonkos, A., Chandra, G., Buttner, M. J. Expression of the chaplin and rodlin hydrophobic sheath proteins in Streptomyces venezuelae.is controlled by sigma(BldN) and a cognate anti-sigma factor, RsbN. Mol. Microbiol. 84, 1033-1049 (2012).
- Al-Bassam, M. M., Bibb, M. J., Bush, M. J., Chandra, G., Buttner, M. J. Response regulator heterodimer formation controls a key stage in Streptomyces development. PLoS Genet. 10, e1004554 (2014).
- Bush, M. J., Bibb, M. J., Chandra, G., Findlay, K. C., Buttner, M. J. Genes required for aerial growth, cell division, and chromosome segregation are targets of WhiA before sporulation in Streptomyces venezuelae. MBio. 4, e00684-e00613 (2013).
- Tschowri, N., et al. Tetrameric c-di-GMP mediates effective transcription factor dimerization to control Streptomyces development. Cell. 158, 1136-1147 (2014).
- Mirouze, N., Ferret, C., Yao, Z., Chastanet, A., Carballido-Lòpez, R. MreB-Dependent Inhibition of Cell Elongation during the Escape from Competence in Bacillus subtilis. PLoS Genet. 11, e1005299 (2015).
- Zopf, C. J., Maheshri, N. Acquiring fluorescence time-lapse movies of budding yeast and analyzing single-cell dynamics using GRAFTS. J. Vis. Exp. (e50456), (2013).
- Meniche, X., et al. Subpolar addition of new cell wall is directed by DivIVA in mycobacteria. Proc. Natl. Acad. Sci. U S A. 111, E3243-E3251 (2014).
- Donovan, C., Schauss, A., Kramer, R., Bramkamp, M. Chromosome segregation impacts on cell growth and division site selection in Corynebacterium glutamicum. PLoS One. 8, e55078 (2013).
- Rojas, E., Theriot, J. A., Huang, K. C. Response of Escherichia coli. growth rate to osmotic shock. Proc. Natl. Acad. Sci. U S A. 111, 7807-7812 (2014).
- Flärdh, K. Essential role of DivIVA in polar growth and morphogenesis in Streptomyces coelicolor A3(2). Mol. Microbiol. 49, 1523-1536 (2003).
- Sliusarenko, O., Heinritz, J., Emonet, T., Jacobs-Wagner, C. High-throughput subpixel precision analysis of bacterial morphogenesis and intracellular spatio-temporal dynamics. Mol. Microbiol. 80, 612-627 (2011).
- Young, J. W., et al. Measuring single-cell gene expression dynamics in bacteria using fluorescence time-lapse microscopy. Nat. Protoc. 7, 80-88 (2012).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7, R100 (2006).
- Kuru, E., Tekkam, S., Hall, E., Brun, Y. V., Van Nieuwenhze, M. S. Synthesis of fluorescent D-amino acids and their use for probing peptidoglycan synthesis and bacterial growth in situ. Nat. Protoc. 10, 33-52 (2015).
- Szafran, M., et al. Topoisomerase I (TopA) is recruited to ParB complexes and is required for proper chromosome organization during Streptomyces coelicolor. sporulation. J. Bacteriol. 195, 4445-4455 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten