需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
单细胞基因表达谱使用流式细胞仪和qPCR内部标准
摘要
We describe a method to sort single mammalian cells and to quantify the expression of up to 96 target genes of interest in each cell. This method includes the use of internal qPCR standards to enable the estimation of absolute transcript counts.
摘要
Gene expression measurements from bulk populations of cells can obscure the considerable transcriptomic variation of individual cells within those populations. Single-cell gene expression measurements can help assess the role of noise in gene expression, identify correlations in the expression of pairs of genes, and reveal subpopulations of cells that respond differently to a stimulus. Here, we describe a procedure to measure the expression of up to 96 genes in single mammalian cells isolated from a population growing in tissue culture. Cells are sorted into lysis buffer by fluorescence-activated cell sorting (FACS), and the mRNA species of interest are reverse-transcribed and amplified. Gene expression is then measured using a microfluidic real-time PCR machine, which performs up to 96 qPCR assays on up to 96 samples at a time. We also describe the generation and use of PCR amplicon standards to enable the estimation of the absolute number of each transcript. Compared with other methods of measuring gene expression in single cells, this approach allows for the quantification of more distinct transcripts than RNA FISH at a lower cost than RNA-Seq.
引言
在群体中的单个细胞可显示成均匀的生理刺激物1,2,3,4大不相同响应。细胞群体中的遗传变异是该各种反应机制之一,但也有几个非遗传因素,可以增加的响应的变异性,即使是在细胞的克隆群。例如,单个蛋白质和其他重要的信号传导分子的水平可以在小区通过小区基础而变化,从而引起变化在下游基因表达谱。此外,可发生在转录物5,6可被限制在一个相对小的数目每爆裂7,8,9的转录物的短持续时间的脉冲串的基因活化。这样随机性在基因激活可以大大促进生物反应的变异,可以在微生物10和在哺乳动物细胞中1提供选择优势,2响应于生理刺激。由于变异的遗传和非遗传来源,响应于刺激的任何给定的细胞的基因表达图谱可以由从本体响应的测量获得的平均基因表达谱大大不同。确定哪个个体细胞显示变异性响应于刺激的程度,需要对单个细胞的分离技术,用于感兴趣转录子的表达水平的测量,并将所得的表达数据的计算分析。
有用于测定基因表达的单细胞,覆盖范围广,成本几种方法,成绩单的数量探测和定量的精度。例如,单细胞RNA测序提供转录的覆盖范围和量化数千不同转录为在个体细胞中最高度表达的基因的能力的广泛的深度;然而,用这样的测序深度相关的成本可能是昂贵,虽然成本的不断降低。相反, 在原位杂交(smRNA FISH)单分子的RNA的荧光提供转录为精确定量甚至低表达以每感兴趣基因以合理的成本的基因;然而,只有靶基因的小的数目可以在通过该方法给定的细胞来测定。定量基于PCR的检测,在本协议中所述,提供这些技术之间的中间地带。这些测定使用的微流体实时PCR机在高达96的细胞的时间量化到感兴趣96转录。虽然每一个上述的方法具有所需的硬件成本,任何个别qPCR分析的成本相对低。该协议适于从一个由一个微流体实时PCR机的生产(协议ADP 41,Fluidigm公司)建议。要启用基于PCR的方法每个成绩单的绝对数量的估计,我们已经扩大了协议,使利用可在多个实验中使用准备靶基因扩增的内部控制。
作为这种技术的一个例子,通过肿瘤抑制基因p53的MCF-7人乳腺癌细胞调节的基因的表达的定量描述11。将细胞挑战与诱导DNA双链断裂的化学试剂。以前的研究已经表明,p53的应答DNA双链断裂表现出在个体细胞异质性很大,无论是在p53水平12的术语和在不同的靶基因11的激活。此外,P53调控的超过100个的表达良好表征参与许多下游途径,包括细胞周期停滞,细胞凋亡和衰老13,14靶基因。因为在每个小区中的p53介导的应答是既复杂又变量的系统的好处,从一种方法的分析中,几乎100靶基因可同时在单个细胞,如以下所述进行探测。稍作修改(如单细胞分离和裂解替代方法),该协议可以很容易地适合于研究范围广泛的哺乳动物细胞类型,转录物和细胞应答的。
通过适当的预先准备,圆细胞分类和基因表达测量的,可根据这个协议在一段三天进行。下面的时序建议:提前,选择感兴趣的成绩单,识别和验证那些transcr扩增cDNA的引物对IPTS,并准备标准,并利用这些引物引物混合物。在第1天,以下细胞治疗,收获和细胞进行排序,执行反转录和靶特异性扩增,并用外切核酸酶处理的样品以除去未掺入的引物。第2天,请对使用qPCR排序细胞质量控制。最后,在第3天,测量在使用微流体的qPCR的分选的细胞的基因表达。 图1总结了所涉及的步骤。
研究方案
1.事前准备
- 选择多达96个基因的利益,其表达的测量。
注:至少这些基因中的一个应为"持家基因",如ACTB或GAPDH,即已知在实验中所用的条件下,相对高的和恒定的水平被表达。该基因将被用来识别正排序孔中(步骤8.1)和扩增的样本(步骤10.1)。
注意:对于示例实验,良好的特点,P53的直接目标具有多种已知功能11,14和挑选了看家控制GAPDH。请参见参考文献11的靶基因,并在此研究中使用的引物序列的完整列表。 - 识别特定于感兴趣的基因的潜在的引物对( 例如,使用科学文献或引物设计工具,诸如底漆-BLAST)15。有底漆š合成并保持它们在无核酸酶水储备浓度为100μM的。
注意:这些引物应该是15-25个碱基长;产生的扩增子90-130碱基对(bp)长;跨越外显子交界处,如果存在的话,为减少扩增基因组DNA的机会;具有60±1℃的熔融温度;并具有最小的二级结构16。这些引物将被用于引发逆转录,从感兴趣的转录扩增的cDNA,并测量基因表达的DNA结合染料基的qPCR。 - 验证引物。
- 首先,从感兴趣的细胞获得的cDNA。从根据制造商的说明或标准分子生物学方法17使用RNA采收试剂盒将细胞收获的RNA。反向转录使用具有随机六聚体逆转录试剂盒的RNA成cDNA根据制造商的说明或标准方法具DS 17。
- 对于每个引物对,设置的qPCR与DNA结合染料预混,正向和反向引物,以及来自前面步骤中的cDNA,对建议的反应条件下的主混合物制造商的建议。运行使用由主混合物制造,其包括熔融曲线取得推荐一个热循环协议的实时PCR机器上这些反应。
- 在定量PCR完成后,在2%w / v琼脂糖凝胶扩增的DNA样品和可视化通过DNA嵌入染料以下的标准协议17的DNA。
- 通过构建从cDNA的连续稀释的标准曲线确定的引物对的效率根据标准协议16(在步骤1.3.1。制造)。
注:良好的引物对将产生的熔融曲线与一个单一的,定义明确的峰和单波段在预期位置的凝胶16上向上类="外部参照"> 17( 图2)。如果熔体曲线具有多个峰或一个"肩",或者如果该凝胶具有多个频带或涂抹,引物扩增的脱靶序列。重新设计已经观察到扩增的脱靶序列和重复上述验证步骤作为必要的任何引物。一个良好的引物对也将有90-110%的效率,与R 2≥0.985标准曲线16;重新设计的任何引物的量在这些范围以外的效率或R 2下降。
- 从准备纯化的DNA扩增子标准。
- 对于每一个有效的引物对:
- 使用高保真DNA聚合酶根据聚合酶制造商的引物和模板DNA的浓度,反应条件和PCR循环条件,或使用标准PCR方案建议从cDNA扩增感兴趣的区域"外部参照"> 17。
- 在2%w / v琼脂糖凝胶上扩增的cDNA和用DNA嵌入染料17可视化的频带。消费使用干净的刀片代表扩增子乐队,并放置在离心管的凝胶块。
- 净化从使用标准凝胶提取过程中,凝胶的扩增子如基于琼脂糖酶提取17或一个旋转柱凝胶提取试剂盒,按照制造商的说明。
- 使用根据制造商的说明基于荧光的双链DNA定量试剂盒测量扩增子的浓度。通过基于扩增子序列的扩增子的已知分子量除以测定的双链DNA浓度,得到每微升的扩增子分子的数目。
- 在2.0 mL的低结合离心管中,加入2微升纯化的扩增子的DNA悬浮缓冲液的体积,使得放大器的最终浓度力诚为5×10 8个分子/微升。
- 当所有扩增子都被净化,结合18微升每种纯化的扩增子在2.0 mL的低结合离心管中。添加的DNA悬浮缓冲液使1800微升的总体积;因此, 每个扩增子的浓度为5×10 6个分子/μl的。
注:这种混合物,其中包含从已知等摩尔量感兴趣的所有基因的cDNA扩增,将作为单细胞的qPCR的标准。 - 涡这种"5E6"的标准彻底,旋转10秒在2000 XG,并分为低结合PCR管20微升等分。储存在-80℃的等分试样。
- 对于每一个有效的引物对:
- 制备特定靶序列扩增(STA)引物混合物(10倍)。
- 在一个15毫升锥形管,结合25微升各100μM的股票引物(高达192)。添加的DNA悬浮缓冲液使5000微升的总体积。
注意:在该混合物中的每个引物的浓度为500纳米。分选的细胞的每个板将使用105.6微升此引物混合物向素逆转录和特定靶序列扩增。这里给出的引物混合物的体积足以让45板进行排序。最好是使引物混合物的一个足够大的体积,以便它只需要作出一次;缩放量是必要的。 - 涡旋调匀,分成110等分微升,并等分储存在-20℃。
- 在一个15毫升锥形管,结合25微升各100μM的股票引物(高达192)。添加的DNA悬浮缓冲液使5000微升的总体积。
- 制备测定混合物,每个引物对,用于在基于芯片的微流体的qPCR使用。
- 在96孔PCR板的每个孔中,结合3.0微升100μM的正向引物对于给定的基因,3.0微升相应100μM的反向引物,24.0微升的DNA悬浮缓冲液中,30.0微升2×测定装载试剂。
- 涡旋该板,旋在500 xg离心10秒,并储存在-20℃。
- 创建两个热循环程序:RTSTA(逆转录和具体的目标扩增; 表1)和EXOI(核酸外切酶I处理; 表2)。
2.治疗
- 板上的组织培养皿,使得它们将成长到所需的汇合在处理时,根据不同的实验研究条件的哺乳动物细胞。对于在本研究中提出的例子中,板4×10 5个 MCF-7的p53金星细胞每在RPMI 6cm皿18,用10%胎牛血清(FBS),100U / ml青霉素,100毫克/毫升链霉素和250毫微克/毫升两性霉素B温育直到它们达到50%汇合的细胞两天,在37℃,5%的CO 2。
- 处理细胞,以建立感兴趣根据实验研究的条件。对于本研究中提出的例子,孵化MCF-7的p53金星细胞400毫微克/毫升neocarzino他汀类药物3小时,8.5小时,14小时,或24小时。
3.裂解液制备细胞分选
注:使裂解液的单块板大约需要1小时。这是明智的制作和排序多个板块,如细胞分选可以是低效的,并产生了许多井,没有检测到的小区。
- 清洁台,移液管,管架,冷PCR板支架和手套在为PCR准备DNA洗消液。
- 通过加入股票RNA酶抑制剂对核酸 - 自由水在1.5ml管稀释RNA酶抑制剂2.64单位/微升。
注意:这允许RNA酶抑制剂添加到裂解缓冲液未经吹打子微升体积。 - 通过组合在表3中列出的组分使所述细胞裂解缓冲液。
- 转92.4微升的细胞裂解缓冲液到一个单独的管;这将是对于扩增子的标准裂解缓冲液。
- 使用LOW-结合枪头和微量离心管,准备200微升和500微升大肠杆菌 DNA稀释至3.1微克/微升和6.2微克/微升,分别用DNA悬浮缓冲液。
- 添加184.8微升3.1微克/微升大肠杆菌 DNA为细胞裂解缓冲液。
注: 大肠杆菌载体DNA用于扩大的非特异性引物结合的可能性的范围,从而减少的可能性引物二聚化将导致一个PCR产物。 - 准备标准。
- 标签低六位结合离心管为5E5,5E4,... 5e0。
- 加入90微升DNA悬浮缓冲液的"5E5"管。
- 加入90微升6.2微克/μL 大肠杆菌 DNA到每个其他五管。置于冰上所有的管子。
注:集成载体DNA导入标准使得比得上在1细胞孔的标准孔中的DNA的总质量;这减少了PRobability该标准的浓度将通过DNA受到影响结合管或吸头。 - 取从存储了"5E6"标准(各扩增子的5×10 6分子/微升,在步骤1.4中制备)的等分试样。短暂涡旋并旋转简要(5秒,在500 XG),以确保液体是在管的底部。
- 使用低结合的枪头,加入10微升5E6标准的"5E5"管。短暂涡旋,旋(5秒500 XG),并把管回到冰上。
- 从"5E5"管移取10微升进入"5E4"管。短暂涡旋,旋转(在500 xg离心5秒),并放回冰上。
- 重复这些系列稀释,每管接收来自以前管10微升,直到所有管具有标准的稀释液。
- 准备12 PCR管的一排帽。分发67微升"小区"的裂解缓冲液对每个管。盖上管后短暂离心(5秒500 XG)如果有必要。
- 使用12通道移液器从PCR管的行分发9微升"小区"裂解缓冲液进入第一7行的PCR板( 图3)的各孔中。
- 分发7微升"标准"的裂解缓冲液的板的最后一排的各孔( 图3)。
- 使用低结合枪头,根据板图( 图3)加入2微升的标准底行的每个孔中。
注:2微升5×10 0分子/μl的标准量的10分子等 - 加入2微升3.1微克/μL 大肠杆菌 DNA进10细胞和100细胞孔;加入2微升6.2微克/微升大肠杆菌 DNA入无细胞孔。
注意:每个1细胞,10-细胞和100细胞以及具有6.2皮克大肠杆菌 DNA的,近似的基因组DNA的质量在一个单一的人细胞。每个标准和无细胞以及有12.4皮克Ø˚F 大肠杆菌 DNA,大约两人类细胞的价值。 - 密封用无菌热密封,旋板为5秒,在500 xg离心,并储存在4℃直到细胞准备好排序。
4.细胞分选设置
- 编程荧光激活细胞分选仪根据板图( 图3)进行排序成96孔板;无细胞应该被分成井H1-H8或H11-H12。
- 验证细胞分选仪很好旨在进行排序成PCR板。
- 设置排序流以最小可能的角度;此设置可提高这些细胞将被分类到井的底部,而不是击中两侧的机会。
- 瞄准机器,密封一个空PCR平板,并使用测试流至PBS中"之类的"液滴到空板的密封,根据机器19的指令;液滴应该降落在日在上面的孔的中心的位置上的密封电子表面。对于最好的结果,确认整个板的长度和宽度的目的。
- 如果液滴不正确的位置降落,擦拭掉密封件的表面,根据机器制造商的说明重新校准细胞分选,并重复直到排序流中的液滴正确地沉积。
5.细胞收获和排序
- Trypsinize以适合的细胞系的细胞。例如,对于MCF-7细胞,吸从细胞培养基,用1ml 0.05%胰蛋白酶的冲洗细胞,并孵育用2ml的0.05%胰蛋白酶的5分钟将细胞在37℃和5%CO 2的。
- 8毫升细胞培养基添加到胰蛋白酶的细胞和所有10个ml的细胞悬浮液转移到15毫升管。离心机在400×g下4分钟。
- 吸出上清液并重新悬浮在PBS + 2%FBS的细胞沉淀。
注:小体积悬浮浓缩细胞,并促进细胞分选; 500微升悬浮量非常适用于在50%汇合细胞的直径6cm培养皿。 - 细胞悬液转移到一个5毫升通过40微米的过滤器流式细胞术管,移液以去除细胞团块。
- 排序单元,以确保液体是在孔的底部之前离心裂解缓冲液的板在400×g离心30秒,在4℃。
- 插入与细胞悬浮到细胞分拣机的管中。打开裂解缓冲液的板密封,并根据板图和以下的细胞分选仪制造商的说明19感兴趣的细胞仔细分类到这些板。为了确保单细胞被以最大纯度排序,运行机器中的"单一细胞"模式,并使用标准的流式细胞术了一种基于选通和侧向散射19。
注:在示例实验中,栅极基于p53基因金星荧光分离单个电池11的最亮的15%的细胞。 - 在4℃密封用1分钟一个新的,无菌的热密封和离心机在400 xg离心平板。放置板在热循环仪中,并运行RTSTA程序(参见步骤1.7)。
注:此进行反转录mRNA上从选细胞,然后放大感兴趣的成绩单的基因。
6.核酸外切酶
- 通过组合在表4中列出的组分混合稀核酸外切酶我。准备12 PCR管的一排帽。分发30.5微升稀核酸外切酶我到每个试管中。盖上试管和自旋简要地(5秒,在500 XG),以确保液体是在管的底部。
- 当RTSTA完成时,旋转样品板在500×g下5秒。使用12道移液器,分发3.6微升稀核酸外切酶我到板的各孔中。
- 密封用无菌热封和旋短暂(5秒500 XG)的板。放置板在热循环仪中,并运行EXOI程序(参见步骤1.7)。
注:此步骤后,降低特定目标扩增剩余的引物,使他们不会与将来的PCR干扰。
7.样品稀释
- 当核酸外切酶处理结束后,添加32.4微升TE缓冲液中的样品板的每个孔;这是一个可选的停止点,并且样品可以储存在-20℃下数天。
8.排序质量控制使用qPCR
注意:由于细胞分选是不完全有效,这一步是必要的,以确定该排序板的孔中实际接收的小区。然后将这些样品可用于进一步的分析。
- 测量通过qPCR每个样品中的管家基因的表达。
- 根据用于在步骤1.1中选择的看家基因中的一个聚合酶生产商的使用协议的引物制备900微升DNA结合染料的qPCR主混合物的96反应;正在使用的反应缓冲液,聚合酶,引物等 。用于该反应将取决于特定聚合酶的确切浓度。
- 分发9微升主混合物到96孔PCR板的每个孔。从样品板加入1μl样品为相应的PCR板的井。
- 运行使用由主混合物制造商建议的热循环协议或以下的标准协议20实时PCR机器上的板。
- 分析定量PCR数据20;从下面列出的排序质量控制步骤的qPCR数据的一例示于图4。
- 验证来自无细胞孔的测量值显示出低的(背景)水平基因表达或不表达的。
- 验证由1-细胞孔的测量划分明显地分为两组:具有高的基因表达,并与表达或不表达的背景水平在所有,类似于无细胞孔阴性样品阳性样品。
注:具有较高的基因表达的样品代表实际的排序细胞;那些与背景表达应被排除在进一步分析。 - 验证从10细胞和100细胞孔测量反映比正1 - 细胞孔较高的基因表达,从而为低效的排序可能导致比预期的井少生细胞的可能性。
- 验证从标准孔测量均匀分布在对数空间( 即,在(C T)的值是均匀分布)。
- 确定一切积极1-细胞样本后,做出"样品混合"板图绘制正面1策LL样品到一个新的96孔板中。包括8口井为这个板块地图上的标准。参见图5中的例子。
9.基因表达检测使用微流控的qPCR
注意:对于在本节中的每一步,吸管仅向第一站,以最小化试剂的气泡的形成。
- 打开8 PCR管的一排。如果有样本的多块板,打开8 PCR管的第二排和标签管子1-8。该行将会是汇集来自多个板的标准。
- 在1.5ml管中,与DNA的36微升结合染料样品装载试剂混合360微升的DNA结合染料的qPCR主混合物。短暂涡旋和旋转在2000×g的10秒。除以该混合物(396微升)到8的PCR管的未标记的行,将48微升在每个管中。
- 使用8道移液器,分发3.3微升主混合/上样试剂混合到每口井的一个新的96孔PCR板。标签这个板块,"样品混合。"
- 对于每个样本PCR板:
- 涡流板10秒和旋转,在500×g下10秒。取下盖子和转移2.7微升每个正单细胞样本来对样本对应得很好混合曲目板为1-细胞孔的板图上标明。
- 如果只有一个样品板,转移2.7微升每个扩增标准的其对样本对应井根据板地图混合曲目板。
- 如果有样本的多块板,转移2.7微升每个扩增标准的其对应位置的PCR管的标记的排。从样本的电流板添加标准,以任何标准已经存在。用密封膜完成后密封样品板。
- 如果有样本的多块板:
- VORTEX包含标准PCR管的排在2000×g的10秒10秒和旋转。传送从混合标准每管2.7微升入井的样本的对应根据板地图混合曲目板。
- 用的无核酸酶水2.7微升,其中板地图上显示加载NTC井。密封该样品直到需要在4℃混合曲目板和存储。
- 剥离贴纸撕下一个新的微流控芯片的qPCR的底部。仍然使用与帽控制线流体的注射器,按下每个累加器弹簧片,松开它,并用150-200微升控制线流体的注入每个累加器。
- 将在装弹机的芯片,并运行"擎天柱"的剧本。这需要约20分钟。
- 当灌注完成后,涡和自旋向下试验混合物的板和样品混合(步骤1.6准备)。
- 使用8通道移液器,根据本图中转移4.5微升每个测定混合物的进入微流体的qPCR芯片的左侧 g>的图6。吸管非常小心,以避免在孔的底部产生气泡。完成后,密封试验混合物和存储的板在-20℃以便将来使用。
- 根据图6转移4.5微升各样品混合到微流体的qPCR芯片的右侧。将芯片插入装弹机并运行"加载混合"脚本。这大约需要90分钟。
- 打开微流控实时PCR仪上,开始数据采集软件,并开启灯泡预热起来。这需要约20分钟。
- 当负载混合脚本完成,验证对面有没有线的芯片,这将表明加载问题。小心地从芯片使用透明胶带或实验室带任何灰尘颗粒。插入芯片进入微流体实时PCR机和运行数据收集脚本。使用分析软件进行检查的结果,并导出数据作进一步分析。
- 除去的量,定量PCR扩增曲线显示质量差( 即 ,对于该曲线不象标准S形扩增曲线)20的测量结果。
- 超过30质量差或零测量或其中管家基因表达(参见步骤1.1)比大多数其他样品低得多( 图7)中移除的样本。
- 对于每一个基因,估计正确的熔融峰的位置从每个样品熔融峰位置的中位数。如果测量具有位于超过1℃下从中间熔融峰,从考虑20移除测量。
- 对于每个基因,建立标准曲线来估计使用电子表格或脚本语言20每个样品的mRNA计数。
注意:在这个实验中的标准由10,100,1000,或10,000分子双链DNA的相应于每个感兴趣转录。如果逆转录是完全有效的,该标准将对应于20,200,2000,和mRNA 20000分子,因为mRNA和cDNA的是单链的。在实践中,这不是这种情况,因此,我们表达测量值作为在分子×E RT,其中E RT是逆转录的效率为单位的估计。- 每个标准孔,确定男 ,单链DNA分子的扩增前的数目(双链DNA分子的数目的两倍)( 例如,20分子,200分子等 )。
- 对于每一个标准的好,识别来自定量PCR 20(C T)值。
- 与m和(C T)的值,使用以下等式产生具有参数a和b每个基因的标准曲线进行线性回归,并找到R 2值作为拟合优度的n个指标:
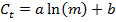
- 从标准曲线,计算每个基因的PCR效率E:
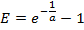
注:E的值小于1.0( 例如,E> 1.1)不是物理现实大得多;对于线性回归的R 2值远小于1.0( 例如,R 2 <0.985)的意思是标准不可靠工作。这些问题通常表明最低扩增标准低于可靠的量化的阈值,因此,最低标准应该从线性回归被排除。如果有足够的线性回归不能使用至少三个独特的标准进行,丢弃该基因的表达的测量。 - 如果该线性回归是足够的,则使用所得到的拟合参数a和b </ em>的每个基因来估计mRNA的分子数米EST(在分子×E RT为单位),用于从所测量的(C T)值每个单细胞样品中相应的基因:
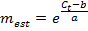
- 指定任何测量米美国东部时间 <1为m EST = 0。此外,指定任何计量与定量PCR无扩增,因此没有C 吨价为m = EST 0。
注:测量与一个m EST比最低小于或大于在回归中使用的最高标准更大的外推从标准曲线中找到。这些估计不一定是无效的,但应谨慎重视。
- 可视化的单细胞基因表达数据。
- 做一个beeswarm图(多恩,J和神圣,T.,2012年,http://www.mathworks.com/matlabcentral/filee交换网/ 37105),其中每个点代表在特定细胞的基因表达;一个例子示于图8。
- 使一个小提琴情节(多恩,J.,2012,http://www.mathworks.com/matlabcentral/fileexchange/23661),其中"小提琴"的宽度代表基因表达测量的围绕在一个特定的电平的频率分析细胞群;一个例子示于图8。
注:更复杂的分析可以探测对基因21,22之间在表达的关系,并且可以推断出基因表达的网络。
结果
该协议的一般概述示于图1,包括用于细胞治疗,单细胞通过FACS中,分离步骤产生和从单细胞裂解物,单细胞cDNA文库中的确认cDNA文库预扩增排序井,和基因表达通过qPCR测量。
在单细胞的分离和基因表达分析的准备,有必要首先确定每个感兴趣的靶基因有效的寡核苷酸引物对。 图2示出的引物的质量控...
讨论
我们已经提出,用于从培养物中生长贴壁细胞群中分离个体哺乳动物细胞和用于测定在每个小区中的大约96个基因的表达的方法。良好的事前准备是至关重要的,此方法才能正常工作。特别是,具体到感兴趣的转录(步骤1.2-1.3)设计和测试的引物对是费时,但重要的步骤,因为引物确定单细胞测量的质量。一旦可靠的引物对已经获得,它们被用来从感兴趣的转录扩增的cDNA;扩增子然后在等摩尔量?...
披露声明
The authors have nothing to disclose.
致谢
我们要感谢V.卡普尔在CCR ETIB流式细胞仪的核心,她在这个协议的开发过程中进行细胞分选援。我们也感谢M. Raffeld和CCR LP分子诊断单位和朱军和NHLBI DNA测序与基因组学核心为他们在这个协议的开发过程中进行定量PCR援助。这项研究是由美国国立卫生研究院的校内计划的支持。
材料
| Name | Company | Catalog Number | Comments |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| High Capacity cDNA Reverse Transcription Kit with RNase Inhibitor | ThermoFisher | 4374966 | |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530S | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| Quant-iT High-Sensitivity dsDNA Assay Kit | ThermoFisher | Q33120 | |
| 2.0 ml low adhesion microcentrifuge tubes | USA Scientific | 1420-2600 | |
| DNA Suspension Buffer | Teknova | T0221 | |
| Axygen 0.2 ml Maxymum Recovery Thin Wall PCR Tubes | Corning | PCR-02-L-C | |
| GE 96.96 Dynamic Array DNA Binding Dye Sample & Assay Loading Reagent Kit | Fluidigm | 100-3415 | |
| HyClone RPMI 1640 media | GE Healthcare Life Sciences | SH30027.01 | |
| Fetal Bovine Serum, Certified (US) | ThermoFisher | 16000-044 | |
| Antibiotic-Antimycotic Solution | Corning | 30-004-CI | |
| Neocarzinostatin | Sigma | N9162 | |
| ELIMINase | Decon Labs | 1101 | |
| SUPERase-In | ThermoFisher | AM2696 | |
| CellsDirect One-Step qRT-PCR Kit | ThermoFisher | 11753500 | |
| E. coli DNA | Affymetrix | 14380 10 MG | |
| ThermalSeal Sealing Film, Sterile | Excel Scientific | STR-THER-PLT | |
| BD FACSAria IIu | BD Biosciences | ||
| HyClone Trypsin 0.05% | GE Healthcare Life Sciences | SH30236.01 | |
| PBS, 1x | Corning | 21-040-CV | |
| Falcon 40 µm Cell Strainer | Corning | 352340 | |
| Exonuclease I | New England BioLabs | M0293S | |
| SsoFast EvaGreen Supermix with Low ROX | Bio-Rad | 172-5210 | |
| 96.96 Dynamic Array IFC for Gene Expression (microfluidic qPCR chip) | Fluidigm | BMK-M-96.96 | |
| IFC Controller HX (loading machine) | Fluidigm | ||
| BioMark or BioMark HD (microfluidic qPCR machine) | Fluidigm | ||
| Real-Time PCR Analysis software | Fluidigm | ||
| MATLAB software | MathWorks |
参考文献
- Feinerman, O., Veiga, J., Dorfman, J. R., Germain, R. N., Altan-Bonnet, G. Variability and Robustness in T Cell Activation from Regulated Heterogeneity in Protein Levels. Science. 321 (5892), 1081-1084 (2008).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- Geva-Zatorsky, N., Rosenfeld, N., et al. Oscillations and variability in the p53 system. Mol. Syst. Biol. 2 (1), (2006).
- Colman-Lerner, A., Gordon, A., et al. Regulated cell-to-cell variation in a cell-fate decision system. Nature. 437 (7059), 699-706 (2005).
- Chong, S., Chen, C., Ge, H., Xie, X. S. Mechanism of Transcriptional Bursting in Bacteria. Cell. 158 (2), 314-326 (2014).
- Raj, A., Peskin, C. S., Tranchina, D., Vargas, D. Y., Tyagi, S. Stochastic mRNA Synthesis in Mammalian Cells. PLoS Biol. 4 (10), e309+ (2006).
- Senecal, A., Munsky, B., et al. Transcription Factors Modulate c-Fos Transcriptional Bursts. Cell Rep. 8 (1), 75-83 (2014).
- Dey, S. S., Foley, J. E., Limsirichai, P., Schaffer, D. V., Arkin, A. P. Orthogonal control of expression mean and variance by epigenetic features at different genomic loci. Mol. Syst. Biol. 11 (5), 806+ (2015).
- Dar, R. D., Razooky, B. S., et al. Transcriptional burst frequency and burst size are equally modulated across the human genome. Proc. Natl. Acad. Sci. U.S.A. 109 (43), 17454-17459 (2012).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- Porter, J. R., Fisher, B. E., Batchelor, E. p53 Pulses Diversify Target Gene Expression Dynamics in an mRNA Half-Life-Dependent Manner and Delineate Co-regulated Target Gene Subnetworks. Cell Syst. 2 (4), 272-282 (2016).
- Lahav, G., Rosenfeld, N., et al. Dynamics of the p53-Mdm2 feedback loop in individual cells. Nat. Genet. 36 (2), 147-150 (2004).
- Levine, A. J., Oren, M. The first 30 years of p53: growing ever more complex. Nat. Rev. Cancer. 9 (10), 749-758 (2009).
- Riley, T., Sontag, E., Chen, P., Levine, A. Transcriptional control of human p53-regulated genes. Nat. Rev. Mol. Cell Biol. 9 (5), 402-412 (2008).
- Ye, J., Coulouris, G., Zaretskaya, I., Cutcutache, I., Rozen, S., Madden, T. L. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinf. 13 (1), 134 (2012).
- . . PCR Technologies: A Technical Guide. , (2014).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Batchelor, E., Mock, C. S., Bhan, I., Loewer, A., Lahav, G. Recurrent initiation: a mechanism for triggering p53 pulses in response to DNA damage. Mol. Cell. 30 (3), 277-289 (2008).
- . . Flow Cytometry: Principles and Applications. , (2007).
- . . Real-time PCR. , (2006).
- Song, L., Langfelder, P., Horvath, S. Comparison of co-expression measures: mutual information, correlation, and model based indices. BMC Bioinf. 13 (1), 328 (2012).
- Margolin, A. A., Nemenman, I., et al. ARACNE: An Algorithm for the Reconstruction of Gene Regulatory Networks in a Mammalian Cellular Context. BMC Bioinf. 7 (Suppl 1), (2006).
- Haff, L. A. Improved quantitative PCR using nested primers. PCR Methods Appl. 3 (6), 332-337 (1994).
- Hashimshony, T., Senderovich, N., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biol. 17, 77 (2016).
- Ronander, E., Bengtsson, D. C., Joergensen, L., Jensen, A. T. R., Arnot, D. E. Analysis of Single-cell Gene Transcription by RNA Fluorescent In Situ Hybridization (FISH). J. Vis. Exp. (68), e4073 (2012).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat. Methods. 5 (10), 877-879 (2008).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nat. Methods. 9 (7), 743-748 (2012).
- Battich, N., Stoeger, T., Pelkmans, L. Image-based transcriptomics in thousands of single human cells at single-molecule resolution. Nat. Methods. 10 (11), 1127-1133 (2013).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。