Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Одноклеточный экспрессии генов профилирование с помощью FACS и КПЦР с внутренними стандартами
В этой статье
Резюме
We describe a method to sort single mammalian cells and to quantify the expression of up to 96 target genes of interest in each cell. This method includes the use of internal qPCR standards to enable the estimation of absolute transcript counts.
Аннотация
Gene expression measurements from bulk populations of cells can obscure the considerable transcriptomic variation of individual cells within those populations. Single-cell gene expression measurements can help assess the role of noise in gene expression, identify correlations in the expression of pairs of genes, and reveal subpopulations of cells that respond differently to a stimulus. Here, we describe a procedure to measure the expression of up to 96 genes in single mammalian cells isolated from a population growing in tissue culture. Cells are sorted into lysis buffer by fluorescence-activated cell sorting (FACS), and the mRNA species of interest are reverse-transcribed and amplified. Gene expression is then measured using a microfluidic real-time PCR machine, which performs up to 96 qPCR assays on up to 96 samples at a time. We also describe the generation and use of PCR amplicon standards to enable the estimation of the absolute number of each transcript. Compared with other methods of measuring gene expression in single cells, this approach allows for the quantification of more distinct transcripts than RNA FISH at a lower cost than RNA-Seq.
Введение
Отдельные клетки в популяции могут показать сильно различающиеся ответы на единой физиологической стимул 1, 2, 3, 4. Генетическое изменение клеток в популяции является одним механизмом для этого разнообразия ответов, но есть также несколько не-генетические факторы, которые могут увеличить изменчивость реакций, даже в клональной популяции клеток. Например, уровни отдельных белков и других важных сигнальных молекул могут изменяться на основе ячейки за ячейкой, что приводит к изменению в последующих профилей экспрессии генов. Кроме того, активация генов может происходить при кратковременном всплесков транскриптов 5, 6 , которые могут быть ограничены относительно небольшим числом транскриптов в пакете 7, 8, 9. такиестохастич- в активации генов может в значительной степени способствовать вариабельности биологических реакций и может обеспечить селективное преимущество в микроорганизмов 10 и в клетках млекопитающих , 1, 2 в ответ на физиологический стимул. Из-за обоих генетических и негенетическими источников вариации, то профиль экспрессии генов любой данной клетки в ответ на стимул может значительно отличаться от среднего профиля экспрессии генов, полученных из измерения объемной реакции. Определение степени, в которой отдельные клетки показывают изменчивость в ответ на стимул требует методов для выделения отдельных клеток, измерение уровней экспрессии для транскриптов, представляющих интерес, и вычислительный анализ полученных данных экспрессии.
Есть несколько подходов для количественного определения экспрессии генов в единичных клетках, охватывающих широкий диапазон расходов, количество транскриптов зондировали, иТочность количественной оценки. Например, одноклеточные Секвенирование РНК предлагает большой глубины охвата транскрипта и возможность количественно оценить тысячи различных транскриптов для наиболее высоким уровнем экспрессии генов в отдельных клетках; Тем не менее, затраты, связанные с такой глубиной секвенирования может быть непомерно высокой, хотя расходы продолжают снижаться. С другой стороны , одной молекулы РНК флуоресценции в гибридизация (FISH) smRNA в предлагает точную количественную оценку транскриптов для даже с низким уровнем экспрессии генов по разумной стоимости за интерес гена; Тем не менее, лишь небольшое количество генов-мишеней, могут быть проанализированы в данной клетке с помощью этого подхода. Количественные основанные на ПЦР анализы, описанные в данном протоколе, обеспечивают золотую середину между этими методами. Эти анализы используют микрожидком ПЦР в реальном времени машину для количественного определения до 96 транскриптов, представляющих интерес в то время, в срок до 96 ячеек. В то время как каждый из вышеуказанных способов имеет необходимые затраты на оборудование, стоимость любого отдельного КПЦР анализа является сравнительнонизкий. Этот протокол адаптирован из одного предложенной производитель микрожидком ПЦР в реальном времени машины (протокол АДФ 41, Fluidigm). Чтобы включить оценку абсолютного числа каждого транскрипта в подходе ПЦР на основе, мы расширили протокол, чтобы сделать использование внутреннего контроля подготовленных ампликонов гена-мишени, которые могут быть использованы в нескольких экспериментах.
В качестве примера этой техники, количественная оценка экспрессии генов , регулируемых опухолевого супрессора р53 в MCF-7 клетках карциномы молочной железы человека описана 11. Клетки заражали химическим агентом, который индуцирует ДНК двухцепочечной разрывы. Предыдущие исследования показали , что ответ на р53 ДНК двухцепочечной разрывы проявляет большую гетерогенность в отдельных клетках, как с точки зрения уровней p53 12 и в активации различных генов - мишеней 11. Кроме того, р53 регулирует экспрессию более 100хорошо охарактеризованный целевых генов , участвующих в многочисленных последующих путей, в том числе арест клеточного цикла, апоптоз и старении 13, 14. Так как р53-опосредованный ответ в каждой клетке одновременно и сложными и изменчивыми, анализ системных преимуществ от подхода, в котором около 100 генов-мишеней могут быть проверены одновременно в отдельных клетках, таких как описанные ниже. С небольшими изменениями (например, альтернативные методы выделения одноклеточного и лизис), протокол может быть легко адаптирован для изучения широкого спектра типов клеток млекопитающих, транскриптов и клеточных реакций.
При правильной предварительной подготовки, этап сортировки клеток и измерения экспрессии генов может быть проведена в соответствии с этим протоколом в течение периода трех дней. Следующие сроки предлагается: заранее, выберите стенограммы интерес, идентифицировать и проверить пары праймеров, которые усиливают кДНК из этих TranscrДПБ, а также подготовить стандарты и грунтовки смеси с использованием этих праймеров. На 1-й день, после обработки клеток, урожай и сортировки клеток, выполняют обратную транскрипцию и конкретной целевой амплификации, и обрабатывать образцы с экзонуклеазы для удаления некорпоративные праймеров. На 2-й день, осуществлять контроль качества на отсортированных клеток с использованием КПЦР. И, наконец, на 3-й день, измерить экспрессию гена в отсортированных клеток с использованием микрожидкостных КПЦР. На рисунке 1 приведены этапы.
протокол
1. Предварительная подготовка
- Выберите до 96 генов, представляющих интерес, экспрессия которых будет измеряться.
Примечание: По крайней мере, один из этих генов должно быть "ген домашнего хозяйства", такие как ACTB или GAPDH, которые, как известно, выражается в относительно высоком и постоянном уровне в условиях, используемых в эксперименте. Этот ген будет использоваться для идентификации положительно отсортированные скважин (шаг 8.1) и усиленных образцов (шаг 10.1).
Примечание: В примере эксперимента, хорошо охарактеризован, прямой мишенью р53 с помощью множества известных функций 11, 14 и GAPDH в качестве контроля домашнего хозяйства были выбраны. Пожалуйста, см Reference 11 для полного списка генов-мишеней и последовательностей праймеров, используемых в данном исследовании. - Выявление потенциальных пар праймеров , специфичных для генов , представляющих интерес (например, с использованием научной литературы или инструмент проектирования праймера , таких как Primer-BLAST) 15. Есть грунтовкуS синтезирована и поддерживать их на складе концентрации 100 мкМ в нуклеазы без воды.
Примечание: Эти праймеры должны быть длинными 15-25 оснований; производят ампликона 90-130 пар оснований (п.о.) длинные; охватывать экзон соединение, если таковой существует, чтобы уменьшить вероятность амплификации геномной ДНК; имеют температуры плавления 60 ± 1 ° С; и имеют минимальную вторичную структуру 16. Эти праймеры будут использоваться для простого обратной транскрипции, амплификации кДНК из транскриптов, представляющих интерес, и измеряют экспрессию генов в ДНК связывания красителя на основе КПЦР. - Проверка праймеров.
- Во-первых, получение кДНК из клеток, представляющих интерес. РНК Добывается из клеток с использованием набора для уборки РНК в соответствии с инструкциями изготовителя или стандартными методами молекулярной биологии 17. Реверс-транскрибировать РНК в кДНК с использованием обратной транскрипции набора со случайными гексамерах в соответствии с инструкциями изготовителя или стандартной метоDS 17.
- Для каждой пары праймеров, установить КПЦР с ДНК-связывающий краситель основной смеси, прямого и обратного праймеров, и кДНК из предыдущей стадии, в соответствии с рекомендациями изготовителя основной смеси для предложенных условий реакции. Запуск этих реакций на ПЦР в реальном времени с использованием машины тепловой протокол езда на велосипеде, рекомендованный изготовителем смеси мастер-расплава, который включает получение кривой.
- После кПЦР, запустить усиленных образцы ДНК на 2% вес геля / об агарозном и визуализации ДНК с помощью ДНК интеркалирующего красителя следующий стандартный протокол 17.
- Определить эффективность пары праймеров путем построения стандартной кривой из серийных разведений кДНК (на шаге 1.3.1) в соответствии со стандартным протоколом 16.
Примечание: Хорошая пара праймеров даст кривую плавления с одной, хорошо выраженный пик и одной полосы в ожидаемом месте на гель 16, до класса = "" внешние ссылки> 17 (рисунок 2). Если кривая расплава имеет несколько пиков или "плечо" или, если гель имеет несколько полос или мазок, праймеры амплификации вне целевой последовательности. Редизайн любых праймеров, которые наблюдались для амплификации вне цели последовательности и повторите предыдущие шаги проверки по мере необходимости. Хорошая пара праймеров также будет иметь эффективность 90-110%, при этом R 2 ≥0.985 для стандартной кривой 16; перепроектировать любые праймеров для которых эффективность или R 2 выходят за указанные пределы.
- Подготовить стандарты из очищенного ампликонов ДНК.
- Для каждого одобренного пары праймеров:
- Amplify область интереса из кДНК с использованием ДНК-полимеразы с высокой точностью воспроизведения в соответствии с рекомендациями производителя полимеразной для концентрации праймеров и матричной ДНК, условия реакции и условия ПЦР, езда на велосипеде или с использованием стандартного протокола ПЦР"Xref"> 17.
- Запустите усиленную кДНК на 2% вес геля / об агарозном и визуализировать группу с ДНК интеркалирования краситель 17. Акцизный группу, представляющую ампликона, используя чистую лезвия бритвы и поместить гель кусок в микроцентрифужных трубки.
- Очищают ампликона из геля с использованием стандартной процедуры экстракции из геля, такого как agarase на основе экстракции 17 или набора для экстракции геля спин-колонку, в соответствии с инструкциями изготовителя.
- Измерение концентрации ампликона с использованием набора дц количественного определения флуоресценции на основе в соответствии с инструкциями изготовителя. Разделить измеренная концентрация дц по известной молекулярной массой ампликону на основе последовательности ампликона, чтобы получить число молекул ампликона на мкл.
- В 2,0 мл низкой связывающего пробирке, добавляют по 2 мкл очищенного ампликоне до объема ДНК буфера подвески таким образом, что конечная концентрация усилителяLicon составляет 5 × 10 8 молекул / мкл.
- Когда все ампликонов были очищены, объединить 18 мкл каждого очищенного ампликоне в 2,0 мл низкого связывания микроцентрифужных трубки. Добавить буфер подвески ДНК, чтобы общий объем 1800 мкл; Таким образом, концентрация каждого ампликона составляет 5 × 10 6 молекул / мкл.
Примечание: Эта смесь, которая содержит кДНК ампликонов из всех интересующих генов в известных эквимолярных количествах, будет служить в качестве стандарта в одноклеточных кПЦР. - Вихрь этот "5e6" стандарт тщательно, спина в течение 10 сек при 2000 мкг, и разделить на 20 мкл аликвоты в низких связывания трубок ПЦР. Хранить аликвот при -80 ° С.
- Для каждого одобренного пары праймеров:
- Приготовьте смесь конкретной цели амплификации (STA) праймер (10x).
- В 15 мл коническую пробирку, смешайте 25 мкл каждого 100 мкМ праймера фондовой (до 192). Добавить ДНК буфер подвески, чтобы общий объем 5000 мкл.
Обратите вниманиеконцентрация каждого праймера в этой смеси составляет 500 нМ. Каждая пластина из отсортированных клеток будет использовать 105,6 мкл этой смеси праймеров для простого обратной транскрипции и амплификации специфической целевой. Объем смеси праймеров данного здесь достаточно, чтобы сделать 45 пластин для сортировки. Желательно, чтобы сделать достаточно большой объем смеси грунтовки, так что нужно только сделать один раз; масштабировать объемы по мере необходимости. - Тщательно перемешать встряхиванием, разделить на 110 мкл аликвоты, и хранить аликвоты при -20 ° С.
- В 15 мл коническую пробирку, смешайте 25 мкл каждого 100 мкМ праймера фондовой (до 192). Добавить ДНК буфер подвески, чтобы общий объем 5000 мкл.
- Подготовить опробования смесей, по одному для каждой пары праймеров, для использования в Микрожидкостных чиповых кПЦР.
- В каждую лунку 96-луночного ПЦР планшета смешайте 3,0 мкл 100 мкМ прямого праймера для данного гена, 3,0 мкл соответствующего 100 мкМ обратного праймера, 24,0 мкл суспензионного ДНК буфера и 30,0 мкл 2x реагента для анализа загрузки ,
- Vortex эта пластинка, спина в течение 10 сек при 500 мкг, и хранить при температуре -20 ° C.
- Создание двух программ Амплифицируйте: RTSTA (обратной транскрипции и амплификации конкретной целевой, таблица 1) и EXOI (экзонуклеазная лечение I; таблица 2).
2. Лечение
- Пластина клеток млекопитающих на блюдо культуры ткани таким образом, что они будут расти до желаемой впадения во время лечения, в зависимости от условий экспериментального исследования. Для примера , приведенного в данном исследовании, плита 4 х 10 5 MCF-7 p53-Венера клеток 18 на 6 см блюдо в RPMI с 10% фетальной телячьей сыворотки (FBS), 100 ед / мл пенициллина, 100 мг / мл стрептомицина, и 250 нг / мл амфотерицина B. инкубировать клетки в течение двух дней при 37 ° с с 5% CO 2 , пока они не достигнут 50% слияния.
- Лечить клетки, чтобы установить состояние интерес, основанный на экспериментальном исследовании. Для примера, представленного в данном исследовании, инкубировать MCF-7 клеток р53-Венера с 400 нг / мл neocarzinoстатин в течение 3 ч, 8,5 ч, 14 ч, или 24 ч.
3. Буфер Подготовка Лизис для сортировки клеток
Примечание: Изготовление одной пластины буфера для лизиса занимает около 1 часа. Желательно, чтобы и сортировать кратно пластины, как сортировка клеток может быть неэффективным и дают много скважин без заметного ячейки.
- Очистите скамейки, пипеток, стойки трубы, холодные держатели ПЦР пластины и перчатки с ДНК дезактивирующим раствором при подготовке к ПЦР.
- Развести ингибитор РНКазы до 2,64 ед / мкл добавлением ингибитора РНКазы акций в нуклеазы без воды в 1,5 мл трубки.
Примечание: Это позволяет ингибитор РНКазы быть добавлен в буфер для лизиса без пипеткой суб-мкл объемы. - Сделать буфер для лизиса для ячеек путем объединения компонентов , перечисленных в таблице 3.
- Передача 92,4 мкл буфера для лизиса для клеток в отдельную пробирку; это будет лизис буфера для стандартов ампликона.
- Использование Loж-связывающим пипеток и пробирки микроцентрифужных, подготовить 200 мкл и 500 мкл кишечной палочки ДНК разбавленные до 3,1 пг / мкл и 6,2 пг / мкл, соответственно, с несущим ДНК буфера.
- Добавить 184,8 мкл 3,1 пг / мкл ДНК кишечной палочки в буфере для лизиса для клеток.
Примечание: ДНК - носитель E.coli , служит для расширения сферы охвата неспецифического связывания праймера возможностей и тем самым уменьшить вероятность того, что праймер димеризации приведет к продукту ПЦР. - Подготовить стандарты.
- Добавьте шесть низкие связывания микроцентрифужных труб как 5e5, 5e4, ... 5e0.
- Добавить 90 мкл ДНК буфера подвески к "5e5" трубки.
- Добавить 90 мкл 6,2 мкг / мкл ДНК кишечной палочки для каждого из остальных пяти трубок. Держите все пробирки на льду.
Примечание: Включение ДНК-носителя в стандарты делает суммарную массу ДНК в стандартных скважинах, сравнимых с, что в 1-клеточных скважин; это снижает прobability что концентрация стандартов будет зависеть от связывания ДНК для труб или пипеток. - Возьмем аликвоты "5e6" стандарта (5 х 10 6 молекул / мкл каждого ампликона, полученного на стадии 1.4) из хранилища. Вихревой кратко и кратко спина (5 сек при 500 х г), чтобы гарантировать, что жидкость находится в нижней части трубки.
- Использование низкого связывания кончика пипетки, добавьте 10 мкл стандарта 5e6 к "5e5" трубки. Vortex кратко, спин (5 сек при 500 мкг), и положил трубку обратно на лед.
- Внесите 10 мкл из "5e5" трубки в "5e4" трубки. Vortex кратко, спин (5 сек при 500 мкг) и положить обратно на лед.
- Повторите эти серийных разведений, с каждой трубкой, получающего по 10 мкл из предыдущей трубки, пока все трубки не имеют разбавление стандарта.
- Подготовить ряд 12 ПЦР пробирки с крышками. Распределить 67 мкл "клеток" буфера для лизиса в каждую пробирку. Трубки закрывают крышками и спином вниз кратко (5сек при 500 мкг) в случае необходимости.
- С помощью пипетки 12-канальный, распространять 9 мкл "клеток" буфера для лизиса из ряда пробирок для ПЦР в каждую лунку первых 7 рядов пластины ПЦР (рисунок 3).
- Распределить 7 мкл "стандартного" буфера для лизиса в каждую лунку последнего ряда пластины (рисунок 3).
- Используя низкие связывания наконечники пипеток, добавьте 2 мкл стандартной в каждую лунку нижнего ряда в соответствии с картой пластины (рисунок 3).
Примечание: 2 мкл 5 х 10 0 молекул / мкл стандартных составляет 10 молекул и т.д. - Добавьте 2 мкл 3,1 пг / мкл ДНК кишечной палочки в 10-клеток и 100-клеточных скважин; добавить 2 мкл 6,2 пг / мкл ДНК кишечной палочки в не-клеточных скважин.
Примечание: В каждом 1-клетка, 10-клеток, и 100-клеток также имеет 6,2 мкг E.coli ДНК, аппроксимирующих массу геномной ДНК в одной клетке человека. Каждый стандарт и не-клеток также имеет 12,4 пг ое ДНК кишечной палочки, стоит примерно в два человеческих клеток. - Уплотнение пластины, используя стерильный термическую уплотнение, спиновый в течение 5 сек при 500g, и хранят при температуре 4 ° С до тех пор, пока клетки не готовы к сортировке.
4. Настройка сортировщика Cell
- Программирование флуоресценции активированных клеток сортировщик для сортировки в 96-луночного планшета в соответствии с картой пластины (рисунок 3); клетки не должны быть отсортированы в лунки H1-H8 или Н11-Н12.
- Убедитесь в том, что ячейка сортировщика хорошо предназначена для сортировки в пластину ПЦР.
- Установите угол потока сортировки до минимально возможного; эта установка увеличивает вероятность того, что клетки будут сортироваться в днищ колодцев, а не ударил сторон.
- Направить машину, печать пустой пластины ПЦР и использовать тестовый поток на "сорта" капли PBS на печать пустую тарелку, в соответствии с инструкциями машины 19; капли должны приземлиться на гое поверхность уплотнения в положении над центром скважины. Для получения наилучших результатов, проверьте цель по всей длине и ширине пластины.
- Если капли не землю в правильном положении, вытрите их поверхность уплотнения, откалибровать сортировщик клеток в соответствии с инструкциями изготовителя станка, и повторять, пока капли в потоке сортировки не осаждаются правильно.
5. Ячейка для уборки урожая и сортировка
- Trypsinize клетки, которые необходимы для клеточной линии. Например, для клеток MCF-7, аспирацию среды из клеток, прополоскать клетки с 1 мл 0,05% трипсина и инкубировать клетки с 2 мл 0,05% трипсина в течение 5 мин при температуре 37 ° С и 5% CO 2 ,
- Добавить 8 мл клеточной культуральной среды для клеток трипсином и передать все 10 мл клеточной суспензии в 15 мл пробирку. Центрифуга в течение 4 мин при 400 х г.
- Аспирируйте супернатант и ресуспендируют осадок клеток в PBS + 2% FBS. <бр /> ПРИМЕЧАНИЕ: меньший объем ресуспендирования концентратов клеток и облегчает сортировки клеток; объем ресуспендирования 500 мкл хорошо работает на 6 см чашку клеток на 50% сплошности.
- Передачи клеточной суспензии в 5 мл проточной цитометрии трубки, пипетирования через фильтр с диаметром 40 мкм, чтобы удалить сгустки из клеток.
- Центрифуга пластины буфера для лизиса при 400 мкг в течение 30 сек при температуре 4 ° C до сортировки клеток, чтобы гарантировать, что жидкость находится на дне лунок.
- Вставьте трубку с суспензией клеток в клетки сортировщика. Открыть уплотнение на пластине буфера для лизиса и тщательно сортирует клетки , представляющие интерес в пластину в соответствии с картой пластины и следуя инструкциям производителя Клеточный сортер в 19. Для того, чтобы гарантировать , что отдельные клетки сортируются по максимальной чистоты, запустите машину в режиме "Single Cell" и использовать стандартный поток цитометрии стробирования на основе вперед и бокового рассеяния 19.
Примечание: ВПример эксперимента, ворота клетки на основе p53-Венеры флуоресценции выделить самые яркие 15% индивидуальных клеток 11. - Уплотнение плита с новым стерильным термическим уплотнением и центрифуге при 400 мкг в течение 1 мин при температуре 4 ° С. Поместите пластину в термоциклеру и запустить программу RTSTA (этап 1.7).
Примечание: Это выполняет обратную транскрипцию на мРНК из отсортированных клеток, а затем усиливает кДНК-транскриптов, представляющих интерес.
6. Лечение экзонуклеаза
- Смешайте разбавленный экзонуклеазной I путем объединения компонентов , перечисленных в таблице 4. Подготовить ряд 12 ПЦР пробирки с крышками. Распределить 30,5 мкл разбавленного экзонуклеазы I в каждую пробирку. Трубки закрывают крышками и спина на короткое время (5 сек при 500 х г), чтобы гарантировать, что жидкость находится в нижней части трубки.
- Когда RTSTA закончена, закрутить образец пластины в течение 5 сек при 500 х г. С помощью пипетки 12-канальный, распространять 3,6 мкл разбавленного экзонуклеазыЯ в каждую лунку планшета.
- Уплотнение пластины с стерильной термическим уплотнением и спина кратко (5 сек при 500 мкг). Поместите пластину в термоциклеру и запустить программу EXOI (этап 1.7).
Примечание: Этот шаг ухудшает любые праймеры, оставшиеся после определенной целевой амплификации, так что они не будут мешать будущей ПЦР.
7. Образец Разбавление
- Когда обработка экзонуклеазная закончена, добавьте 32,4 мкл ТЕ-буфера в каждую лунку для образца пластины; это дополнительная точка остановки, и образцы можно хранить при температуре -20 ° С в течение нескольких дней.
8. Сортировка Контроль качества Использование КПЦР
Примечание: Поскольку сортировка клеток не совсем эффективным, этот шаг необходим, чтобы определить, какие скважины отсортированного пластины фактически получил клетку. Эти образцы затем могут быть использованы для дальнейшего анализа.
- Измерьте экспрессию генов домашнего хозяйства в каждом образце КПЦР.
- Подготовьте 900 мкл ДНК-связывающего красителя КПЦР мастер смеси в течение 96 реакций в соответствии с протоколом с использованием праймеров производителя полимеразной для одного из генов домашнего хозяйства, выбранных на шаге 1.1; используются точные концентрации реакционного буфера, полимеразы, праймеров и т.д. для этой реакции будет зависеть от конкретной полимеразы.
- Распределить 9 мкл мастер смеси в каждую лунку 96-луночного планшета ПЦР. Добавляют 1 мкл пробы из образца пластины в соответствующую лунку пластины ПЦР.
- Запустите пластину на ПЦР в реальном времени с использованием машины тепловой протокол езда на велосипеде , предложенном производителем мастер - микс или ниже стандартного протокола 20.
- Анализ данных КПЦР 20; пример КПЦР данных из этапов контроля качества сортировки , перечисленных ниже, показан на рисунке 4.
- Убедитесь в том, что измерения от не-клеточных скважин показывают низкий (фоновый) уровеньэкспрессии генов или без выражения вообще.
- Убедитесь в том, что измерения от 1-клеточных скважин разделить четко на две группы: положительные образцы с высоким уровнем экспрессии генов и отрицательных образцов с фоновыми уровнями экспрессии или без выражения на всех, похожих на не-клеточных скважин.
Примечание: Образцы с высокой экспрессии генов представляют собой фактические отсортированные клетки; те, с фоновой экспрессии, должны быть исключены из дальнейшего анализа. - Убедитесь, что измерения от 10-клеток и 100-клеточных лунок отражают более высокую экспрессию генов, чем в положительных 1-клеточных скважин, если допустить возможность, что неэффективное сортировка может вызвать эти скважины, чтобы иметь меньше клеток, чем хотелось бы.
- Убедитесь в том, что измерения от стандартных скважин распределены равномерно в логарифмической пространстве (то есть, что значения С Т распределены равномерно).
- После определения всех положительных образцов 1-клеток, сделать "Образец" Миксы пластины карты на карту положительный результат 1-CeОбразцы Л.Л. на новый 96-луночный планшет. Включите восемь лунок для стандартов на этой пластине карте. Смотрите рисунок 5 для примера.
9. Измерение экспрессии генов Использование КПЦР микрожидком
Примечание: Для каждого шага в этом разделе пипетку только до первой остановки, чтобы свести к минимуму образование пузырьков в реагентов.
- Открыть ряд 8 пробирок для ПЦР. Если имеется несколько пластин образцов, открыть второй ряд 8 ПЦР пробирок и маркировать пробирки 1-8. Эта строка будет для объединения стандартов из нескольких пластин.
- В 1,5 мл пробирку, смешать 360 мкл ДНК-связывающего красителя КПЦР мастер смеси с 36 мкл ДНК-связывающего реагента загрузки образца красителя. Vortex кратко и спина в течение 10 сек при 2000 х г. Разделить эту смесь (396 мкл) в немаркированной ряд 8 ПЦР пробирок, помещая 48 мкл в каждую пробирку.
- С помощью пипетки 8-канальный, распространять 3,3 мкл смеси мастер-микс / загрузки образца реагента в каждую лункунового 96-луночного ПЦР планшета. Добавьте эту табличку, "Образец смеси."
- Для каждой ПЦР пластины образцов:
- Vortex планшет в течение 10 сек и спина в течение 10 сек при 500 х г. Снимите крышку и передать 2,7 мкл каждого положительного образца 1-клеток в его соответствующую лунку на образце Mixes пластины для 1-ячейки скважины, указанной на карте пластины.
- Если имеется только одна пластина из образцов, передать 2,7 мкл каждого амплифицированного стандарта на соответствующий ему хорошо на образце Mixes пластины в соответствии с картой пластины.
- Если имеется несколько пластин образцов, передавать в 2,7 мкл каждого амплифицированного стандарта в соответствующую позицию в меченой ряду пробирок для ПЦР. Добавить стандарты от текущей пластины образцов каких-то стандартов уже там. Уплотнение пластины образцов, когда делается с помощью герметизирующей пленки.
- Если имеется несколько пластин образцов:
- Вихре ряд ПЦР-пробирки, содержащие стандарты для10 сек и спина в течение 10 сек при 2000 х г. Передача 2,7 мкл из каждой пробирки смешанных стандартов в соответствующие лунки на образец Mixes пластины в соответствии с картой пластины.
- Загрузите NTC скважин с 2,7 мкл нуклеазы без воды, где указано на карте пластины. Уплотнение Образец Mixes пластины и хранят при температуре 4 ° С до тех пор, пока это необходимо.
- Пил наклейку со дна нового чипа микрожидком КПЦР. С помощью шприца из контрольной линии жидкости с крышкой еще дальше, надавите каждый аккумулятор пружину, чтобы ослабить его, и вводят каждый аккумулятор с 150-200 мкл контрольной линии жидкости.
- Вставьте чип в погрузочной машины и запустить «Prime» сценарий. Это занимает ~ 20 мин.
- Когда грунтование делается вихревые и спином вниз пластины анализа смесей (полученного на стадии 1.6) и образцов смесей.
- С помощью пипетки 8-канальный, передать 4,5 мкл каждого тест-смеси в левую сторону микрожидком чипа КПЦР в соответствии со схемой в г> Рисунок 6. Пипетка очень осторожно, чтобы избежать образования пузырьков воздуха в нижней части лунки. Когда закончите, запечатать пластину анализа смесей и хранить при температуре -20 ° C для дальнейшего использования.
- Передача 4,5 мкл каждой смеси образца с правой стороны микрожидком чипа КПЦР согласно фиг.6. Вставьте чип в погрузочной машины и запустить "Load" Mix сценарий. Это занимает ~ 90 мин.
- Включите микрожидком ПЦР в реальном времени машины, запустить программное обеспечение для сбора данных и включить лампу, чтобы согреть. Это занимает ~ 20 мин.
- Когда скрипт нагрузки Mix будет сделано, убедитесь, что нет никаких линий через чип-это может указывать на проблему загрузки. Осторожно удалите любые частицы пыли с чипом, используя скотч или ленту лаборатории. Вставьте чип в микрожидком ПЦР в реальном времени машины и запустить скрипт для сбора данных. С помощью программного обеспечения для анализа, чтобы проверить результаты и экспортировать данные для дальнейшего анализа.
- Удалите измерения , для которых кривые амплификации КПЦР показывают низкое качество (то есть, для которых кривые не похожи на стандартную сигмоидального амплификации кривую) 20.
- Удалить образцы с более чем 30 некачественное или нулевых измерений или для которых экспрессии генов домашнего хозяйства (см шаг 1.1) значительно ниже , чем в большинстве других образцов (рис 7).
- Для каждого гена, оценить правильность пика расплава местоположение в качестве медианы расплава пиковых точках из каждого образца. Если измерение имеет пик расплава , расположенный более чем на 1 ° C от медианы, удалить это измерение из рассмотрения 20.
- Для каждого гена, создать стандартную кривую для оценки мРНК подсчет в каждом образце с помощью электронных таблиц или сценариев языка 20.
Примечание: Стандарты в данном эксперименте состоят из 10, 100, 1000, или 10000 молекулиз двухцепочечной ДНК, соответствующей каждому транскрипта интерес. Если обратной транскрипции были вполне эффективными, стандарты будут соответствовать 20, 200, 2000 и 20000 молекул мРНК, так как мРНК и кДНК являются одноцепочечными. На практике это не так, поэтому мы выражаем измерения в качестве оценки в единицах молекул × E РТ, где Е RT является эффективность обратной транскрипции.- Для каждой стандартной скважины, определить м, число молекул одноцепочечной ( в два раза число молекул двухцепочечной ДНК) перед амплификации (например, 20 молекул, 200 молекул и т.д.).
- Для каждой стандартной скважины, определить C значение т из кПЦР 20.
- Со значениями т и с т, выполняют линейную регрессию , используя следующее уравнение для получения стандартной кривой с параметрами а и б для каждого гена, и найти значение R 2 в качествеп показатель СОГЛАСИИ:
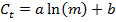
- Из стандартной кривой, вычислить эффективность ПЦР - E для каждого гена:
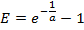
Примечание: Значение Е значительно больше , чем 1,0 (например, E> 1.1) физически не реалистично; Значение R 2 для линейной регрессии намного меньше , чем 1,0 (например, R 2 <0,985) означает , что стандарты не работают надежно. Эти проблемы, как правило, показывают, что самый низкий стандарт ампликона падает ниже порога надежной количественной оценки, и, таким образом, самый низкий стандарт, должны быть исключены из линейной регрессии. Если адекватный линейной регрессии не может быть сделано с использованием по меньшей мере, три уникальных стандартов, отбрасывать измерения экспрессии для гена. - Если линейной регрессии является адекватным, использовать полученные подходящие параметры а и б </ EM> для каждого гена , чтобы оценить количество мРНК молекул м EST (в единицах молекул × E RT) для соответствующего гена в каждом образце одноклеточного от измеренного значения C T:
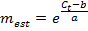
- Назначают любое измерение м EST <1 , как м EST = 0. Кроме того , назначать любое измерение, без усиления в кПЦР и , следовательно , не C значения т как м EST = 0.
Примечание: Измерения с м Эст меньше минимального или больше , чем самый высокий стандарт , используемый в регрессии найдены путем экстраполяции из стандартной кривой. Эти оценки не обязательно являются недействительными, но следует относиться с осторожностью.
- Визуализация данных экспрессии гена одноклеточных.
- Сделать beeswarm участок (Dorn, J. и свят, T., 2012, http://www.mathworks.com/matlabcentral/filee~~HEAD=dobjXchange / 37105), в котором каждая точка представляет собой экспрессию гена в определенной клетке; пример показан на рисунке 8.
- Сделайте скрипичный участок (Дорн, J., 2012, http://www.mathworks.com/matlabcentral/fileexchange/23661~~HEAD=dobj), в котором ширина "скрипка" представляет собой частоту измерений экспрессии генов вокруг определенного уровня в популяция клеток анализируемых; пример показан на рисунке 8.
Примечание: Более сложные анализы могут исследовать отношения в экспрессии между парами генов 21, 22 и можно сделать вывод экспрессии генов сетей.
Результаты
Общий обзор протокола показан на рисунке 1, в том числе шагов для лечения клеток, выделения отдельных клеток с помощью FACS, генерация и предварительной амплификации кДНК библиотек из одноклеточных лизатов, подтверждение библиотек кДНК одноклеточных в отсорти...
Обсуждение
Мы представили способ выделения отдельных клеток млекопитающих из популяции адгезивных клеток, выращенных в культуре, и будут анализироваться экспрессию приблизительно 96 генов в каждой клетке. Хорошая подготовка заранее имеет решающее значение для этого метода хорошо работать. В ча?...
Раскрытие информации
The authors have nothing to disclose.
Благодарности
Мы хотели бы поблагодарить В. Капур в CCR ETIB проточной цитометрии Ядро для ее помощи в выполнении сортировки клеток во время разработки этого протокола. Мы также благодарим М. Raffeld и блок CCR LP молекулярной диагностики и J. Zhu и НИСЛК секвенирование ДНК и геномики Сердечник за помощь в выполнении КПЦР в процессе разработки этого протокола. Это исследование было поддержано очный программы НИЗ.
Материалы
| Name | Company | Catalog Number | Comments |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| High Capacity cDNA Reverse Transcription Kit with RNase Inhibitor | ThermoFisher | 4374966 | |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530S | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| Quant-iT High-Sensitivity dsDNA Assay Kit | ThermoFisher | Q33120 | |
| 2.0 ml low adhesion microcentrifuge tubes | USA Scientific | 1420-2600 | |
| DNA Suspension Buffer | Teknova | T0221 | |
| Axygen 0.2 ml Maxymum Recovery Thin Wall PCR Tubes | Corning | PCR-02-L-C | |
| GE 96.96 Dynamic Array DNA Binding Dye Sample & Assay Loading Reagent Kit | Fluidigm | 100-3415 | |
| HyClone RPMI 1640 media | GE Healthcare Life Sciences | SH30027.01 | |
| Fetal Bovine Serum, Certified (US) | ThermoFisher | 16000-044 | |
| Antibiotic-Antimycotic Solution | Corning | 30-004-CI | |
| Neocarzinostatin | Sigma | N9162 | |
| ELIMINase | Decon Labs | 1101 | |
| SUPERase-In | ThermoFisher | AM2696 | |
| CellsDirect One-Step qRT-PCR Kit | ThermoFisher | 11753500 | |
| E. coli DNA | Affymetrix | 14380 10 MG | |
| ThermalSeal Sealing Film, Sterile | Excel Scientific | STR-THER-PLT | |
| BD FACSAria IIu | BD Biosciences | ||
| HyClone Trypsin 0.05% | GE Healthcare Life Sciences | SH30236.01 | |
| PBS, 1x | Corning | 21-040-CV | |
| Falcon 40 µm Cell Strainer | Corning | 352340 | |
| Exonuclease I | New England BioLabs | M0293S | |
| SsoFast EvaGreen Supermix with Low ROX | Bio-Rad | 172-5210 | |
| 96.96 Dynamic Array IFC for Gene Expression (microfluidic qPCR chip) | Fluidigm | BMK-M-96.96 | |
| IFC Controller HX (loading machine) | Fluidigm | ||
| BioMark or BioMark HD (microfluidic qPCR machine) | Fluidigm | ||
| Real-Time PCR Analysis software | Fluidigm | ||
| MATLAB software | MathWorks |
Ссылки
- Feinerman, O., Veiga, J., Dorfman, J. R., Germain, R. N., Altan-Bonnet, G. Variability and Robustness in T Cell Activation from Regulated Heterogeneity in Protein Levels. Science. 321 (5892), 1081-1084 (2008).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- Geva-Zatorsky, N., Rosenfeld, N., et al. Oscillations and variability in the p53 system. Mol. Syst. Biol. 2 (1), (2006).
- Colman-Lerner, A., Gordon, A., et al. Regulated cell-to-cell variation in a cell-fate decision system. Nature. 437 (7059), 699-706 (2005).
- Chong, S., Chen, C., Ge, H., Xie, X. S. Mechanism of Transcriptional Bursting in Bacteria. Cell. 158 (2), 314-326 (2014).
- Raj, A., Peskin, C. S., Tranchina, D., Vargas, D. Y., Tyagi, S. Stochastic mRNA Synthesis in Mammalian Cells. PLoS Biol. 4 (10), e309+ (2006).
- Senecal, A., Munsky, B., et al. Transcription Factors Modulate c-Fos Transcriptional Bursts. Cell Rep. 8 (1), 75-83 (2014).
- Dey, S. S., Foley, J. E., Limsirichai, P., Schaffer, D. V., Arkin, A. P. Orthogonal control of expression mean and variance by epigenetic features at different genomic loci. Mol. Syst. Biol. 11 (5), 806+ (2015).
- Dar, R. D., Razooky, B. S., et al. Transcriptional burst frequency and burst size are equally modulated across the human genome. Proc. Natl. Acad. Sci. U.S.A. 109 (43), 17454-17459 (2012).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- Porter, J. R., Fisher, B. E., Batchelor, E. p53 Pulses Diversify Target Gene Expression Dynamics in an mRNA Half-Life-Dependent Manner and Delineate Co-regulated Target Gene Subnetworks. Cell Syst. 2 (4), 272-282 (2016).
- Lahav, G., Rosenfeld, N., et al. Dynamics of the p53-Mdm2 feedback loop in individual cells. Nat. Genet. 36 (2), 147-150 (2004).
- Levine, A. J., Oren, M. The first 30 years of p53: growing ever more complex. Nat. Rev. Cancer. 9 (10), 749-758 (2009).
- Riley, T., Sontag, E., Chen, P., Levine, A. Transcriptional control of human p53-regulated genes. Nat. Rev. Mol. Cell Biol. 9 (5), 402-412 (2008).
- Ye, J., Coulouris, G., Zaretskaya, I., Cutcutache, I., Rozen, S., Madden, T. L. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinf. 13 (1), 134 (2012).
- . . PCR Technologies: A Technical Guide. , (2014).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Batchelor, E., Mock, C. S., Bhan, I., Loewer, A., Lahav, G. Recurrent initiation: a mechanism for triggering p53 pulses in response to DNA damage. Mol. Cell. 30 (3), 277-289 (2008).
- . . Flow Cytometry: Principles and Applications. , (2007).
- . . Real-time PCR. , (2006).
- Song, L., Langfelder, P., Horvath, S. Comparison of co-expression measures: mutual information, correlation, and model based indices. BMC Bioinf. 13 (1), 328 (2012).
- Margolin, A. A., Nemenman, I., et al. ARACNE: An Algorithm for the Reconstruction of Gene Regulatory Networks in a Mammalian Cellular Context. BMC Bioinf. 7 (Suppl 1), (2006).
- Haff, L. A. Improved quantitative PCR using nested primers. PCR Methods Appl. 3 (6), 332-337 (1994).
- Hashimshony, T., Senderovich, N., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biol. 17, 77 (2016).
- Ronander, E., Bengtsson, D. C., Joergensen, L., Jensen, A. T. R., Arnot, D. E. Analysis of Single-cell Gene Transcription by RNA Fluorescent In Situ Hybridization (FISH). J. Vis. Exp. (68), e4073 (2012).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat. Methods. 5 (10), 877-879 (2008).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nat. Methods. 9 (7), 743-748 (2012).
- Battich, N., Stoeger, T., Pelkmans, L. Image-based transcriptomics in thousands of single human cells at single-molecule resolution. Nat. Methods. 10 (11), 1127-1133 (2013).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены