A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
תאים יחידים Gene Expression Profiling שימוש FACS ו qPCR עם סטנדרטים פנימיים
In This Article
Summary
We describe a method to sort single mammalian cells and to quantify the expression of up to 96 target genes of interest in each cell. This method includes the use of internal qPCR standards to enable the estimation of absolute transcript counts.
Abstract
Gene expression measurements from bulk populations of cells can obscure the considerable transcriptomic variation of individual cells within those populations. Single-cell gene expression measurements can help assess the role of noise in gene expression, identify correlations in the expression of pairs of genes, and reveal subpopulations of cells that respond differently to a stimulus. Here, we describe a procedure to measure the expression of up to 96 genes in single mammalian cells isolated from a population growing in tissue culture. Cells are sorted into lysis buffer by fluorescence-activated cell sorting (FACS), and the mRNA species of interest are reverse-transcribed and amplified. Gene expression is then measured using a microfluidic real-time PCR machine, which performs up to 96 qPCR assays on up to 96 samples at a time. We also describe the generation and use of PCR amplicon standards to enable the estimation of the absolute number of each transcript. Compared with other methods of measuring gene expression in single cells, this approach allows for the quantification of more distinct transcripts than RNA FISH at a lower cost than RNA-Seq.
Introduction
תאים בודדים בתוך אוכלוסייה יכול להראות תגובות ומעולם שונות לגירוי 1 פיסיולוגי אחיד, 2, 3, 4. הווריאציה הגנטית של תאים באוכלוסייה היא מנגנון אחד עבור מגוון זה של תגובות, אבל יש גם כמה גורמים שאינם גנטיים שיכול להגדיל את ההשתנות של תגובות, גם באוכלוסייה משובטת של תאים. לדוגמא, הרמות של חלבונים בודדים מולקולות איתות חשובות אחרות יכולות להשתנות על בסיס תא אחר תא, והולידו וריאצית פרופילי ביטוי גנים במורד זרם. בנוסף, הפעלת גנים יכולה להתרחש התפרצויות קצר משך התמלילים 5, 6 שעשויות להיות מוגבל למספר קטן יחסית של תמלילים לכל פרץ 7, 8, 9. כגוןstochasticity ב הפעלת הגן יכול לתרום השתנות מאוד תגובות ביולוגיות והוא יכול לספק יתרון סלקטיבי מיקרואורגניזמים 10 ו בתאי יונקים 1, 2 מגיבים על גירוי פיזיולוגי. בשל שני המקורות הגנטיים שאינם גנטיים של וריאציה, פרופיל ביטוי גנים של כל תא נתון בתגובה לגירוי עשוי להיות שונה באופן משמעותי מפרופיל ביטוי גנים הממוצע המתקבל מדידת התגובה בתפזורת. קביעת המידה שבה תאים בודדים מראים השתנות בתגובה לגירוי דורשת טכניקות עבור הבידוד של תאים בודדים, מדידת רמות ביטוי תמלילי העניין, ועל הניתוח חישובית של נתוני הביטוי שהתקבלו.
ישנן מספר גישות מנסים לאמוד ביטוי גנים בתאים יחידים, מכסי מגוון רחב של עלויות, מספר תמלילי נחקר, ודיוק של כימות. לדוגמא, תא בודד RNA-seq מציע עומק רחב של כיסויי תמליל ואת היכולת לכמת אלף תמלילים ברורים עבור הגנים הביעו הגבוהה ביותר בתאים בודדים; עם זאת, העלות הכרוכה עומק רצף כזה עלולה להיות מרתיעה, אם כי עלויות ממשיכות לרדת. לעומת זאת, קרינת RNA מולקולה בודדת הכלאה באתרו (FISH smRNA) מציעה כימותים מדויקים של תמלילים אפילו נמוך לבטא גנים במחיר סביר לכל גן של עניין; עם זאת, רק מספר קטן של גני מטרה ניתן assayed בתא נתון על ידי גישה זו. מבחני PCR מבוססים כמוני, כמתואר בפרוטוקול זה, לספק את שביל זהב בין הטכניקות הללו. מבחנים אלו להעסיק המכונים PCR microfluidic בזמן אמת לכמת עד 96 תמלילי עניין בכל פעם עד 96 תאים. בעוד כל אחת מהשיטות הנ"ל יש עלויות חומרה נדרשות, את העלות של כל assay qPCR פרט היא יחסיתנָמוּך. פרוטוקול זה מותאם מאחד שהוצע על ידי היצרן של מכונת PCR microfluidic בזמן אמת (פרוטוקול ADP 41, Fluidigm). כדי להפעיל את הערכת מספרם המוחלט של כל תמליל בגישה מבוססת PCR, הרחבנו את הפרוטוקול לעשות שימוש לגבי הבקרה פנימית על amplicons גן המטרה המוכן שניתן להשתמש בה בכל ניסויים מרובים.
כדוגמה של טכניקה זו, כימות של ביטוי גנים מוסדר על ידי p53 מדכא גידולים בתאי סרטן שד אנושיים MCF-7 מתוארת 11. התאים מאותגרים עם סוכן כימי שגורם הפסקות פעמים גדיל DNA. מחקרים קודמים הראו כי התגובה p53 הפסקות DNA פעמיים גדיל מפגין מידה רבה של ההטרוגניות בתאים בודדים, הן מבחינת רמות ה- p53 12 ו בהפעלת גני מטרה מובחנת 11. יתר על כן, p53 מסדיר את הביטוי של למעלה מ -100היטב מאופיין גני מטרה המעורבים מסלולים רבים במורד הזרם, כולל עיכוב מחזור התא, אפופטוזיס, והזדקנות 13, 14. מאז התגובה בתיווך p53 בכל תא היא גם מורכבת ומשתנה, הניתוח של יתרונות המערכת מגישה שבה כמעט 100 גני המטרה ניתן נחקר בו זמנית בתאים בודדים, כמו זו מתוארת להלן. עם שינויים קלים (כגון שיטות חלופיות עבור בידוד תא בודד תמוגה), הפרוטוקול ניתן להתאים בקלות ללמוד מגוון רחב של סוגי תאים של יונקים, תעתיקים, ואת תגובות הסלולר.
עם הכנה מראש נכונה, סבב מיון תא ומדידת ביטוי גנים יכול להתנהל על פי פרוטוקול זה על פני תקופה של שלושה ימים. העיתוי הבא הוא הציע: מראש, בחר את תמלילי העניין, לזהות ולאמת את הזוגות פריימר כי להגביר את cDNA מאלה transcripts, ולהכין את הסטנדרטים ותערובות פריימר באמצעות פריימרים אלו. ביום 1, לאחר טיפול בתא, קציר ולמיין את התאים, לבצע שעתוק לאחור והגברת יעד ספציפי, ולטפל דגימות עם exonuclease להסיר פריימרים מאוגדים. ביום 2, לבצע בקרת איכות על תאים ממוינים באמצעות qPCR. לבסוף, ביום 3, למדוד את ביטוי הגנים בתאים המסודרים באמצעות microfluidic qPCR. איור 1 מסכם את השלבים הכרוכים.
Protocol
1. הכנה מראש
- בחר עד 96 גני העניין שביטויים יימדדו.
הערה: לפחות אחד הגנים האלה צריך להיות "גן משק," כגון ACTB או GAPDH, כי ידוע להתבטא ברמה גבוהה וקבועה יחסית בתנאים השתמשו בניסוי. גן זה ישמש לזהות בארות מסודרות באופן חיובי (שלב 8.1) דגימות מוגברות (שלב 10.1).
הערה: לצורך הניסוי לדוגמא, היטב מאופיין, מטרות ישירות של p53 עם מגוון רחב של פונקציות ידועות 11, 14 ו GAPDH כביקורת משק נבחרה. אנא ראה חומר עזר 11 לקבלת הרשימה המלאה של גני מטרה רצפים פריימר השתמשו במחקר זה. - לזהות זוגות פריימר פוטנציאל ספציפיים הגנים של עניין (למשל, באמצעות הספרות המדעית או כלי עיצוב פריימר כגון תפציץ פריימר) 15. יש פריימרזה מסונתז ולשמור אותם בריכוז מלאה של 100 מיקרומטר במי nuclease חינם.
הערה: אלה פריימרים צריכים להיות 15-25 בסיסים ארוכים; לייצר amplicon 90-130 זוגות בסיסים (bp) ארוך; משתרע על פני צומת אקסון, אם קיים כזו, כדי להקטין את הסיכוי של הגברת הדנ"א הגנומי; יש לטמפרטורת ההיתוך של 60 ± 1 ° C; ויש לי מבנה משני מינימלי 16. פריימרים אלו ישמשו שעתוק לאחור הממשלה, להגביר cDNA מן תמלילי עניין, ולמדוד ביטוי גנים בדנ"א מחייב qPCR מבוססי צבען. - אמת את פריימרים.
- ראשית, לקבל cDNA מן התאים של עניין. קציר RNA מן התאים באמצעות ערכת קצירת RNA לפי הוראות היצרן או שיטות הביולוגיה המולקולרית תקן 17. Reverse-לתמלל את RNA לתוך cDNA באמצעות ערכת שעתוק לאחור עם hexamers אקראי על פי הוראות היצרן או metho תקןDS 17.
- עבור כל זוג פריימר, להגדיר qPCR עם DNA מחייב שילוב הורי צבע, קדימה לאחור יחל ואת cDNA מהשלב הקודם, בעקבות המלצות מיקס מאסטר של היצרן עבור תנאי תגובה שהציעו. הפעל התגובות האלה על מחשב PCR בזמן אמת באמצעות פרוטוקול רכיבה תרמי בהתאם להמלצת יצרן תערובת ההורים הכולל רכישה עקומה להמס.
- לאחר qPCR, הפעל את דגימות דנ"א מוגבר על ג'ל 2% w / v agarose ו לדמיין את ה- DNA באמצעות צבע intercalating DNA הבאים פרוטוקול סטנדרטי 17.
- לקבוע את היעילות של זוג פריימר ידי בניית עקומת סטנדרט מ דילולים סדרתי של cDNA (עשה צעד 1.3.1) על פי פרוטוקול סטנדרטי 16.
הערה: זוג פריימר טוב יניב עקום להמס עם יחיד, מוגדר היטב שיא להקה אחת במיקום הצפוי על הג'ל 16, עד class = "Xref"> 17 (איור 2). אם עקום להמס יש פסגות מרובות או "כתף", או אם את הג'ל יש להקות מרובות או מריחה, פריימרים מגבירים רצף מחוץ יעד. לעצב מחדש כל פריימרים אשר נצפו כדי להגביר רצפים מחוץ היעד וחזור על צעדי אימות שקודמים לפי צורך. זוג פריימר טוב יהיה גם יעיל של 90-110%, עם R 2 ≥0.985 עבור עקומת סטנדרט 16; לעצב מחדש כל פריימרים עבורו נפילת יעילות או R 2 בחוץ בטווחים אלו.
- כן סטנדרטי amplicons DNA המטוהר.
- עבור כל זוג פריימר תוקף:
- להגביר את האזור של עניין מן cDNA באמצעות DNA פולימרז באיכות גבוהה על פי המלצות יצרן פולימראז לריכוזי פריימר ו- DNA התבנית, תנאי תגובה, ותנאי רכיבת PCR, או באמצעות פרוטוקול ה- PCR תקן"Xref"> 17.
- הפעל את cDNA מוגבר על ג'ל 2% w / v agarose ולדמיין את הלהקה עם DNA intercalating לצבוע 17. והבלו הלהקה המייצג את amplicon באמצעות סכין גילוח נקי ומניחים את פיסת ג'ל בתוך שפופרת microcentrifuge.
- לטהר את amplicon מן הג'ל באמצעות הליך מיצוי ג'ל סטנדרטי, כגון מיצוי agarase מבוסס 17 או ערכת חילוץ ג'ל ספין-טור, על פי הוראות היצרן.
- למדוד את הריכוז של amplicon באמצעות ערכת כימות dsDNA קרינה המבוססת פי הוראות היצרן. מחלק את ריכוז dsDNA שהיא נמדדת על ידי המשקל המולקולרי הידוע של amplicon מבוסס על רצף amplicon כדי לקבל את המספר של מולקולות amplicon לכל μl.
- בתוך צינור 2.0 מ"ל נמוך מחייב microfuge, להוסיף 2 μl של amplicon מטוהרים לנפח של חיץ ההשעיה DNA כך הריכוז הסופי של המגברlicon הוא 5 x 10 8 מולקולות / μl.
- כשכל amplicons צורף, לשלב 18 μl של כל amplicon מטוהר בתוך שפופרת 2.0 מיליליטר נמוך מחייב microcentrifuge. הוסף חוצץ ההשעיה DNA לעשות בנפח כולל של 1,800 μl; וכך, ריכוז של כל amplicon הוא 5 x 10 6 מולקולות / μl.
הערה: תערובת זו, אשר מכיל amplicons cDNA מכל הגנים של עניין ידוע כמויות equimolar, ישמש כסטנדרט ב תא בודד qPCR. - וורטקס "5e6" זה סטנדרטי ביסודיות, ספין למשך 10 שניות ב 2000 XG, ומחלקים ל -20 aliquots μl צינורות PCR נמוכים מחייב. אחסן את aliquots ב -80 מעלות צלזיוס.
- עבור כל זוג פריימר תוקף:
- הכן את הגברת יעד הספציפית (STA) תערובת פריימר (10x).
- בתוך צינור חרוטי 15 מ"ל, לשלב 25 μl של כל צבע יסוד 100 מיקרומטר המניות (עד 192). הוסף חוצץ ההשעיה DNA לעשות בנפח כולל של 5,000 μl.
הערה:ריכוז של כל צבע יסוד בתערובת זו הוא 500 ננומטר. כל צלחת של תאים ממוינים תשתמש 105.6 μl של תערובת פריימר זה כדי להתניע את שעתוק לאחור והגברת יעד ספציפי. היקף תערובת פריימר המתפרסם כאן הוא מספיק כדי להפוך 45 צלחות למיין. רצוי לעשות כמות גדולה מספיק של תערובת פריימר כך שהוא רק צריך להיעשות פעם; קנה המידה של כרכים לפי הצורך. - מערבבים היטב על ידי vortexing, מתחלקים 110 aliquots μl, ולאחסן את aliquots ב -20 מעלות צלזיוס.
- בתוך צינור חרוטי 15 מ"ל, לשלב 25 μl של כל צבע יסוד 100 מיקרומטר המניות (עד 192). הוסף חוצץ ההשעיה DNA לעשות בנפח כולל של 5,000 μl.
- הכן תערובות assay, אחד עבור כל זוג פריימר, לשימוש microfluidic שבב מבוסס qPCR.
- בכל טוב של צלחת 96-היטב PCR, לשלב 3.0 μl של פריימר 100 מיקרומטר קדימה עבור גן מסוים, 3.0 μl של פריימר 100 מיקרומטר הפוכה המקביל, 24.0 μl של חיץ ההשעיה DNA, 30.0 μl של מגיב טעינת assay 2x .
- וורטקס צלחת זו, ספין למשך 10 שניות ב 500 XG, ולאחסן ב -20 מעלות צלזיוס.
- צור שתי תוכניות רכיבה תרמית: RTSTA (שעתוק לאחור והגברה יעד ספציפי; טבלה 1) ו EXOI (טיפול שאני exonuclease; טבלה 2).
טיפול 2.
- פלייט בתאים יונק על צלחת בתרבית רקמה כך הם יגדלו המפגש הרצוי בעת הטיפול, בהתאם לתנאי של המחקר הניסיוני. לצורך הדוגמא המוצגת במחקר זה, צלחת 4 x 10 5 MCF-7 תאים p53-ונוס 18 לכל 6 צלחת ס"מ RPMI עם 10% בסרום שור העובר (FBS), 100 U / פניצילין מ"ל, 100 מ"ג / מ"ל סטרפטומיצין, ו ng 250 / מ"ל amphotericin B. דגירה התאים במשך יומיים ב 37 מעלות צלזיוס עם 5% CO 2 עד שהם מגיעים 50% confluency.
- פנקו את התאים כדי לקבוע את מצבו של ריבית המבוססת על מחקר ניסיוני. לצורך הדוגמא המוצגת במחקר זה, דגירה MCF-7 p53-ונוס תאים עם 400 ננוגרם / מ"ל neocarzinoסטטינים במשך 3 שעות, 8.5 שעות, 14 שעות, או 24 שעות.
3. הכנת הצפת תמוגה עבור מיון התא
הערה: ביצוע צלחת אחת של חיץ תמוגה לוקח בערך 1 שעה. רצוי לעשות ולמיין מרובי צלחות, כמו מיון תא יכול להיות יעיל להניב רבי בארות ללא תאים לזיהוי.
- נקו את הספסל, טפטפות, מתלה צינור, מחזיקי צלחת PCR קר, וכפפות עם פתרון decontaminating DNA כהכנה PCR.
- לדלל את מעכב RNAse כדי 2.64 U / μl ידי הוספת מעכב המניות RNAse כדי nuclease ללא מים בצינור 1.5 מ"ל.
הערה: פעולה זו מאפשרת RNAse מעכב שיתווספו למאגר תמוגה ללא pipetting כרכים תת-μl. - הפוך חיץ תמוגה עבור התאים על ידי שילוב של הרכיבים המפורטים בטבלה 3.
- העבר 92.4 μl של חיץ תמוגה עבור התאים לתוך צינור נפרד; זה יהיה חיץ תמוגה עבור בסטנדרטים amplicon.
- באמצעות low מחייב טיפים פיפטה וצינורות microcentrifuge, להכין 200 μl ו 500 μl של DNA E. coli מדולל ל 3.1 pg / μl ו -6.2 pg / μl, בהתאמה, עם חיץ ההשעיה DNA.
- הוסף 184.8 μl של 3.1 pg / μl E. coli DNA למאגר תמוגה עבור התאים.
הערה: דנ"א המוביל החיידק משמש כדי להרחיב את היקף אפשרויות מחייבות פריימר ספציפי ובכך להקטין את הסיכוי כי dimerization פריימר יוביל מוצר PCR. - הכן סטנדרטים.
- לייבל שישה צינורות microcentrifuge נמוכים מחייב כמו 5e5, 5e4, ... 5e0.
- להוסיף 90 μl של חיץ ההשעיה DNA אל הצינור "5e5".
- להוסיף 90 μl של 6.2 pg / μl E. coli DNA לכל אחד מחמשת צינורות אחרים. שמור את כל הצינורות על קרח.
הערה: דנ"א המוביל משלבת לתוך סטנדרטים הופך את המסה הכוללת של DNA בבארות ברמה דומה לזו של בארות 1 תאים; זה מפחית את יחסי הציבורobability כי ריכוז סטנדרטים יושפע DNA מחייב צינורות או טיפים פיפטה. - קח aliquot של התקן "5e6" (5 x 10 6 מולקולות / μl של כל amplicon, מוכן בשלב 1.4) מאחסון. בקצרה וורטקס ספין בקצרה (5 שניות ב 500 XG) על מנת להבטיח כי הנוזל הוא בתחתית של התחתית.
- בעזרת קצה פיפטה נמוך מחייב, להוסיף 10 μl לסטנדרטים 5e6 אל הצינור "5e5". וורטקס בקצרה, ספין (5 שניות ב XG 500), ולשים את הצינור בחזרה על הקרח.
- 10 פיפטה μl מ "5e5" הצינור לתוך הצינור "5e4". וורטקס בקצרה, ספין (5 שניות ב 500 XG), ומחזירי קרח.
- חזור על דילולים סדרו אלה, עם כל צינור קבלת 10 μl מהצינור הקודם, עד שיש כל הצינורות דילול לסטנדרטים.
- כן בשורה של 12 צינורות PCR עם כובעים. הפץ 67 μl של חיץ תמוגה "תא" על צינור אחד. כיסוי או צינורות ספין למטה בקצרה (5שניות ב XG 500) במידת הצורך.
- בעזרת פיפטה 12 ערוצים, להפיץ 9 μl של חיץ תמוגה "התא" משורת צינורות PCR לבאר כל 7 השורות הראשונות של צלחת PCR (איור 3).
- פזר 7 μl של חיץ תמוגה "סטנדרטי" היטב כל בשורה האחרונה של צלחת (איור 3).
- בעזרת טיפים פיפטה נמוכים מחייב, להוסיף 2 μl לסטנדרטים היטב כל בשורה התחתונה לפי מפת הצלחת (איור 3).
הערה: 2 μl של 5 x 10 0 מולקולות / μl כמויות רגילות 10 מולקולות, וכו ' - הוסף 2 μl של 3.1 pg / μl E. coli DNA לתוך בארות 10 תאים ו -100 תאים; להוסיף 2 μl של ה- DNA coli 6.2 pg / μl E. לתוך בארות ללא תאים.
הערה: כל 1 תאים, 10 תאים, ו -100 תאים יש גם 6.2 pg של E. coli DNA, המתקרבות המסה של הדנ"א הגנומי בתא אנושי אחת. יש גם רגיל ללא כל תא 12.4 pg of E. coli DNA, כשני בשווי אדם התאים. - חותם את הצלחת באמצעות חותם תרמית סטרילי, ספין במשך 5 שניות ב 500 XG, ולאחסן ב 4 מעלות צלזיוס עד התאים מוכנים למיון.
Cell 4. הגדרת סדרן
- לתכנת את סדרן תא מופעל הקרינה למיין לתוך צלחת 96-גם על פי מפת הצלחת (איור 3); לא התאים צריכים להיות מסודרים לתוך בארות H1-H8 או H11-H12.
- ודא כי סדרן התא ששינה למיון לתוך צלחת PCR.
- קבע את הזווית של זרם מהסוג למינימום האפשרי; הגדרה זו מעלה את הסיכוי שתאי ימוינו לתוך תחתית של בארות ולא פגע הצדדים.
- כדי לכוון את המכונה, לאטום צלחת PCR ריק ולהשתמש זרם מבחן "סוג" טיפות של PBS על החותם של הצלחת הריקה, בהתאם להוראות של המכונה 19; הטיפין צריכות לנחות על המשטח דואר החותם בעמדה מעל מרכז הבאר. לקבלת התוצאות הטובות ביותר, בדוק את המטרה על פני האורך והרוחב של הצלחת.
- אם הטיפות לא נוחתים במקום הנכון, למחוק אותם מעל פני השטח של החותם, לכייל מחדש את סדרן תא בהתאם להוראות של יצרן המכונה, וחזור עד טיפות בזרם סוג מופקדים כראוי.
Cell 5. קציר ומיון
- Trypsinize את התאים בהתאם הקו הסלולרי. לדוגמה, עבור תאים MCF-7, לשאוב את המדיום מתאי, לשטוף את התאים עם 1 מ"ל של טריפסין 0.05%, ו דגירה התאים עם 2 מ"ל של טריפסין 0.05% במשך 5 דקות ב 37 מעלות צלזיוס, 5% CO 2 .
- הוסף 8 מ"ל של מדיום תרבית תאים לתאי trypsinized ולהעביר את כל 10 מ"ל של השעיה לתא צינור 15 מ"ל. צנטריפוגה במשך 4 דקות ב g 400 x.
- לשאוב supernatant ו resuspend התא גלולה ב PBS + 2% FBS.
הערה: נפח resuspension קטן מרכז את התא ומקל מיון תא; נפח resuspension 500 μl עובד היטב עבור צלחת 6 ס"מ של התאים ב 50% confluency. - מעבירים את ההשעיה התא 5 מ"ל cytometry זרימה בצינור, pipetting דרך פילטר 40 מיקרומטר כדי להסיר גושים של תאים.
- צנטריפוגה צלחת של חיץ תמוגה ב 400 XG במשך 30 שניות ב 4 ° C לפני מיון התאים על מנת להבטיח כי הנוזל הוא בתחתית הבארות.
- הכנס את הצינור עם השעית התא לתוך סדרן התא. פתח את החותם על הצלחת של חיץ תמוגה ובזהירות למיין את התאים של עניין לתוך הצלחת פי מפת הצלחת וביצוע ההנחיות של יצרן סדרן תא 19. כדי להבטיח כי תאים בודדים מסודרים על טוהר מקסימלית, להפעיל את המכונה ב "תא בודד" מצב ולהשתמש זרימת תקן cytometry gating מבוסס על קדימה והצד פיזור 19.
הערה:ניסוי למשל, שער התאים המבוססים על קרינת p53-ונוס לבודד תאים בודדים 15% של מבריקי 11. - חותם את הצלחת עם חותם צנטריפוגות תרמית חדש, סטרילי ב 400 XG דקות 1 ב 4 ° C. מניח את צלחת Cycler התרמי להפעיל את תכנית RTSTA (ראה שלב 1.7).
הערה: זה מבצע שעתוק לאחור על mRNA מן התאים הממוינים ואז מגביר את cDNA של תמלילי העניין.
6. טיפול exonuclease
- מערבבים שאני exonuclease לדלל ידי שילוב הרכיבים המפורטים בטבלה 4. כן בשורה של 12 צינורות PCR עם כובעים. פזר 30.5 μl של exonuclease לדלל לי לתוך צינור אחד. כיסוי או צינורות ספין בקצרה (5 שניות ב 500 XG) על מנת להבטיח כי הנוזל הוא בתחתית של התחתית.
- כאשר RTSTA נגמר, לסובב את הצלחת מדגם במשך 5 שניות ב 500 גרם x. בעזרת פיפטה 12 ערוצים, להפיץ 3.6 μl של exonuclease לדללאני לבאר כל צלחת.
- חותם את הצלחת עם חותם תרמית סטרילית בקצרה ספין (5 שניות ב XG 500). מניח את צלחת Cycler התרמי להפעיל את תכנית EXOI (ראה שלב 1.7).
הערה: שלב זה מבזה כל פריימרים הנותרים לאחר הגברת היעד ספציפית, כך שהם לא יפריעו בעתיד PCR.
דילול מדגם 7.
- כאשר הטיפול exonuclease נגמר, להוסיף 32.4 μl של חיץ TE היטב בכל צלחת מדגם; זהו נקודת עצירה אופציונלית, וניתן לאחסן דגימות ב -20 מעלות צלזיוס במשך כמה ימים.
מיין 8. בקרת איכות באמצעות qPCR
הערה: מאחר מיון תא אינו יעיל בצורה מושלמת, צעד זה הוא הכרחי כדי לזהות אילו בארות הצלחת המיון למעשה קבלו תא. דגימות אלה לאחר מכן ניתן להשתמש לצורך ניתוח נוסף.
- מדוד את ביטוי גן משק בכל מדגם על ידי qPCR.
- כן 900 μl של תמהיל מאסטר לצבוע qPCR DNA מחייב עבור 96 תגובות על פי פריימרים באמצעות הפרוטוקול של היצרן פולימראז עבור אחד גני המשק שנבחרו בשלב 1.1; הריכוזים המדויקים של חיץ תגובה, פולימראז, יחל וכו לתגובה זו תהיה תלויה פולימראז הספציפית שבשימוש.
- פזר 9 μl של תמהיל מאסטר לכל גם צלחת 96-היטב PCR. הוסף 1 μl של מדגם מהצלחת מדגם מקביל גם של צלחת PCR.
- הפעל את הצלחת על מכונת ה- PCR בזמן אמת באמצעות פרוטוקול רכיבת תרמית שהוצע על ידי יצרן מיקס מאסטר או הבאה פרוטוקול סטנדרטי 20.
- לנתח את הנתונים qPCR 20; דוגמא נתוני qPCR ממדרגות בקרת איכות מהסוג המפורטים להלן מוצגת באיור 4.
- ודא כי מדידות מבארות אין תאים להראות רמה נמוכה (ברקע)של ביטוי גנים או אין ביטוי כלל.
- ודא כי מדידות מבארות 1 תאים לחלק בבירור לשתי קבוצות: דגימות חיוביות עם ביטוי גנים גבוה דגימות שליליות עם רמות רקע ביטוי או אין ביטוי כלל, בדומה הבארות ללא תאים.
הערה: הדגימות עם ביטוי גנים גבוהים מייצגות תאים ממוינים בפועל; אלה עם ביטוי רקע צריכים להיות מחוץ ניתוח נוסף. - ודא כי מדידות מבארות 10 תאים ו -100 תאים לשקף ביטוי גנים גבוה יותר מאשר בארות 1 התאים החיוביים, המאפשר את האפשרות כי מיון יעיל עלול לגרום הבארות הללו שתהיינה פחות תאים מרצויות.
- ודא כי מדידות מבארות התקן מופצות באופן שווה בחלל לוגריתמים (כלומר, כי ערכי t C מופצים באופן שווה).
- לאחר זיהוי כל דגימות 1 התאים החיוביים, לעשות מפת צלחת "תערובות מדגמות" למפות 1-ce החיוביתll דגימות לצלחת 96-באר חדשה. כלול שמונה בארות עבור התקנים על מפת צלחת זו. ראה איור 5 למשל.
9. ג'ין מדידת ביטוי שימוש Microfluidic qPCR
הערה: על כל צעד ושעל בסעיף זה, פיפטה רק התחנה הראשונה כדי למזער את היווצרות של בועות חומרים כימיים.
- פתח ברצף של 8 צינורות PCR. אם יש צלחות של דגימות מרובות, לפתוח בשורה השנייה של 8 צינורות PCR ואת התווית צינורות 1-8. שורה זו תהיה עבור איגום סטנדרטים מן הצלחות מרובות.
- בתוך צינור 1.5 מ"ל, לערבב 360 μl של תמהיל מאסטר qPCR לצבוע DNA מחייבת עם 36 μl של ה- DNA מחייב מגיב טעינת צבע מדגם. בקצרה ספין וורטקס למשך 10 שניות ב g x 2,000. מחלקים את התערובת (396 μl) לתוך השורה ללא תווית של 8 צינורות PCR, הצבת 48 μl בצינור אחד.
- בעזרת פיפטה 8 ערוצים, להפיץ 3.3 μl של התערובת מגיב טעינת תערובת / מדגם מאסטר לבאר כלצלחת PCR 96-באר חדשה. לייבל צלחת זו, "לדוגמא תערובות."
- עבור כל צלחת של דגימות ה- PCR:
- וורטקס הצליח למשך 10 שניות ספין למשך 10 שניות ב 500 גרם x. הסר את הכיסוי ולהעביר 2.7 μl של כל דגימת 1 תאים חיוביות המתאימה לו היטב על מדגם תערובות צלחת עבור בארות התאים 1 מצוינת על גבי מפת הצלחת.
- אם יש רק אחד צלחת של דגימות, להעביר 2.7 μl של כל תקן מוגבר שלה מתאים היטב על מדגם תערובות צלחת פי מפת הצלחת.
- אם יש הצלחות מרובות של דגימות, להעביר 2.7 μl של כל תקן מוגבר לעמדה המתאימה לו בשורה שכותרתו של צינורות PCR. להוסיף תקנים מהצלחת הנוכחית של דגימות לכל תקנים כבר שם. חותם את הצלחת של דגימות כאשר נעשה באמצעות סרט איטום.
- אם יש הצלחות מרובות של דגימות:
- וורטקס שורה של צינורות PCR המכיל תקנים10 שניות ספין למשך 10 שניות ב 2000 x ז. העבר 2.7 μl מצינור כל תקנים מעורבים לתוך מתאים היטב על מדגם תערובות צלחת פי מפת הצלחת.
- טען את בארות NTC עם 2.7 μl מי nuclease ללא בם מצוין על מפת הצלחת. חותם את המדגם תערובות צלחת ולאחסן ב 4 מעלות צלזיוס עד הצורך.
- לקלף את המדבקה מתחתית שבב qPCR microfluidic חדש. באמצעות מזרק של נוזל שליטה בקנה אחד עם כובע עדיין על, לדחוף למטה בכל שנה באביב מצברים כדי לשחרר אותו, ולהזריק כל מצברים עם 150-200 μl של נוזל פקד הקו.
- הכנס את השבב במכונת ההעמסה להריץ את הסקריפט "הראש". זה לוקח ~ 20 דקות.
- כאשר יחול נעשתה, מערבולת ספין למטה הצלחות של תערובות assay (מוכן בשלב 1.6) ותערובות מדגמות.
- בעזרת פיפטה 8 ערוצים, להעביר 4.5 μl של כל ערבוב של assay לתוך הצד השמאלי של שבב microfluidic qPCR בהתאם לתרשים ב g> איור 6. פיפטה בזהירות רבה, כדי למנוע יצירת בועות אוויר בתחתית הבארות. בסיום, לאטום את הצלחת של תערובות assay ולאחסן ב -20 ° C לשימוש עתידי.
- העבר 4.5 μl של כל ערבוב של מדגם לצד ימין של השבב microfluidic qPCR פי איור 6. הכנס את השבבים לתוך מכונת ההעמסה להריץ את הסקריפט "טען Mix". זה לוקח ~ 90 דקות.
- הפעילו את המכונה PCR microfluidic בזמן אמת, להפעיל את תוכנת איסוף נתונים, ולהדליק את המנורה כדי לחמם אותו. זה לוקח ~ 20 דקות.
- כאשר את התסריט Mix טען נעשה, לוודא שאין קווים ברחבי שבב זה היה להצביע על בעיה הטעינה. מוציאים בזהירות את כל חלקיקי אבק מהשבב באמצעות נייר דבק או קלטת מעבדה. הכנס את השבבים לתוך מכונת microfluidic בזמן אמת PCR ולהפעיל את התסריט איסוף הנתונים. השתמש בתוכנת הניתוח כדי לבדוק את התוצאות לייצא את הנתונים לניתוח נוסף.
- הסר את המדידות עבורו עקומות הגברת qPCR להראות באיכות ירודה (כלומר, שעבורם העקומות לא דומות עקומת הגברת sigmoidal הסטנדרטית) 20.
- הסר דגימות עם יותר מ -30 מדידות באיכות ירודה או אפס או לביטוי אשר גן משק (ראה שלב 1.1) הוא הרבה יותר נמוך מאשר רוב דוגמאות אחרות (איור 7).
- עבור כל גן, להעריך את מיקומה שיא להמס הנכון לפי חציון מקומות השיא להמס מכל מדגם. אם המדידה יש שיא להמיס ממוקם יותר מ 1 ° C מן החציון, להסיר המדידה כי מן התמורה 20.
- עבור כל גן, ליצור עקומת סטנדרט להעריך את ספירת mRNA במדגם כל משתמש בשפה גיליון אלקטרוני או scripting 20.
מולקולות הסטנדרטים בניסוי זה מורכב של 10, 100, 1,000 או 10,000: הערהשל dsDNA מתאים לכל אחד תמליל של עניין. אם שעתוק לאחור היה באופן מושלם ביותר, בסטנדרטים יתאימו 20, 200, 2,000, ו -20,000 מולקולות של mRNA, מאז mRNA ו cDNA הם חד-גדילים. בפועל, זה לא המקרה, אז אנו מביעים מדידות כאומדן ביחידות של מולקולות × E RT, כאשר E RT הוא היעילות של שעתוק לאחור.- עבור כל טוב תקן, לזהות מ ', את מספר המולקולות של ssDNA (כפול ממספרם של מולקולות של dsDNA) לפני ההגברה (למשל, 20 מולקולות, 200 מולקולות, וכו').
- עבור כל טוב תקן, לזהות את הערך t C מ qPCR 20.
- עם הערכים מ 't C, לבצע רגרסיה ליניארית באמצעות המשוואה הבאה כדי ליצור עקומה רגילה עם פרמטרי a ו- b עבור כל גן, ולמצוא את ערך R 2 בתורמחוון n של טיב ההתאמה:
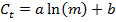
- מתוך עקומת סטנדרט, לחשב את יעילות ה- PCR E עבור כל גן:
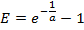
הערה: ערך של E הרבה יותר מאשר 1.0 (למשל, E> 1.1) היא לא מציאותית פיזי; ערך R 2 עבור רגרסיה ליניארית הרבה פחות מ -1.0 (למשל, R 2 <0.985) אומר כי הסטנדרטים אינם פועלים באופן מהימן. בעיות אלו מצביעות בדרך כלל כי תקן amplicon הנמוך ביותר נופל מתחת לסף של כימות אמינה, ובכך בתקן הנמוך ביותר צריך להיות שלילי מן רגרסיה ליניארית. אם רגרסיה ליניארית נאותה לא יכול להתבצע באמצעות לפחות שלושה תקנים ייחודיים, לבטל את מדידות הביטוי של הגן. - אם רגרסיה ליניארית היא נאותה, השתמש פרמטרים בכושר שהתקבל ב </ em> עבור כל גן כדי לאמוד את מספר mRNA מולקולות מ est (ביחידות של מולקולות × E RT) עבור הגן המתאים במדגם כל תא בודד מהערך t C נמדד:
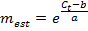
- לייעד כל est מדידה m <1 כמו מ 'est = 0. כמו כן, לייעד מדידה כלשהי ללא הגברה qPCR וכך אין ערך C t כמו מ' est = 0.
הערה: מדידות עם מ 'est פחות מ הנמוך ביותר, או יותר ברמה הגבוהה ביותר בשימוש ברגרסיה נמצאות על ידי אקסטרפולציה מעקום סטנדרטי. אומדנים אלה אינם בהכרח לא חוקיים, אבל צריך להתייחס בזהירות.
- דמיינו את נתוני התבטאות גנים תא בודד.
- הפוך מגרש beeswarm (דורן, ג'יי הקודש, ט 2012, http://www.mathworks.com/matlabcentral/fileeXchange / 37,105) שבה כל נקודה מייצגת את ביטוי הגנים בתא מסוים; דוגמה מוצגת באיור 8.
- הפוך מגרש כינור (דורן, J., 2012, http://www.mathworks.com/matlabcentral/fileexchange/23661) שבו הרוחב של "הכינור" מייצג את תדירות מדידות ביטוי גנים סביב רמה מסוימת של אוכלוסיית התאים נתחו; דוגמה מוצגת באיור 8.
הערה: ניתוחים מתוחכמים יותר יכולים לחקור את היחסים בביטוי בין הזוגות של גנים 21, 22 ו יכולים להסיק רשתות ביטוי גנים.
תוצאות
סקירה כללית של הפרוטוקול מוצגת באיור 1, כוללת צעדים לטיפול תא, הבידוד של תאים בודדים על ידי FACS, דור מראש הגברה של ספריות cDNA מ lysates תא בודד, האישור של ספריות cDNA מתא בודד ב בארות מיון, וכן מדידה של ביטוי גנים על ידי qPCR.
Discussion
הצגנו שיטה לבידוד בתאי יונקים בודדים מתוך אוכלוסייה של תאי חסיד הגדלים בתרבית ועבור מנסה לאמוד את הביטוי של כ 96 גנים בכל תא. הכנה מראש טוב הוא קריטי עבור בשיטה זו כדי לעבוד היטב. בפרט, בעיצוב פריימר בדיקות זוגות מסוימים אל תעתיקי עניין (שלבי 1.2-1.3) צורכים זמן אבל חשובי צ?...
Disclosures
The authors have nothing to disclose.
Acknowledgements
ברצוננו להודות V. קאפור ב Core cytometry זרימת CCR ETIB עבור לעזרתה בביצוע מיון התא במהלך הפיתוח של פרוטוקול זה. אנו מודים גם מ Raffeld ויחידת האבחון המולקולרי CCR LP וג 'ג' ו ואת רצף NHLBI דנ"א ג'נומיקס Core עבור לעזרתם בביצוע qPCR במהלך הפיתוח של פרוטוקול זה. מחקר זה נתמך על ידי התכנית העירונית של NIH.
Materials
| Name | Company | Catalog Number | Comments |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| High Capacity cDNA Reverse Transcription Kit with RNase Inhibitor | ThermoFisher | 4374966 | |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530S | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| Quant-iT High-Sensitivity dsDNA Assay Kit | ThermoFisher | Q33120 | |
| 2.0 ml low adhesion microcentrifuge tubes | USA Scientific | 1420-2600 | |
| DNA Suspension Buffer | Teknova | T0221 | |
| Axygen 0.2 ml Maxymum Recovery Thin Wall PCR Tubes | Corning | PCR-02-L-C | |
| GE 96.96 Dynamic Array DNA Binding Dye Sample & Assay Loading Reagent Kit | Fluidigm | 100-3415 | |
| HyClone RPMI 1640 media | GE Healthcare Life Sciences | SH30027.01 | |
| Fetal Bovine Serum, Certified (US) | ThermoFisher | 16000-044 | |
| Antibiotic-Antimycotic Solution | Corning | 30-004-CI | |
| Neocarzinostatin | Sigma | N9162 | |
| ELIMINase | Decon Labs | 1101 | |
| SUPERase-In | ThermoFisher | AM2696 | |
| CellsDirect One-Step qRT-PCR Kit | ThermoFisher | 11753500 | |
| E. coli DNA | Affymetrix | 14380 10 MG | |
| ThermalSeal Sealing Film, Sterile | Excel Scientific | STR-THER-PLT | |
| BD FACSAria IIu | BD Biosciences | ||
| HyClone Trypsin 0.05% | GE Healthcare Life Sciences | SH30236.01 | |
| PBS, 1x | Corning | 21-040-CV | |
| Falcon 40 µm Cell Strainer | Corning | 352340 | |
| Exonuclease I | New England BioLabs | M0293S | |
| SsoFast EvaGreen Supermix with Low ROX | Bio-Rad | 172-5210 | |
| 96.96 Dynamic Array IFC for Gene Expression (microfluidic qPCR chip) | Fluidigm | BMK-M-96.96 | |
| IFC Controller HX (loading machine) | Fluidigm | ||
| BioMark or BioMark HD (microfluidic qPCR machine) | Fluidigm | ||
| Real-Time PCR Analysis software | Fluidigm | ||
| MATLAB software | MathWorks |
References
- Feinerman, O., Veiga, J., Dorfman, J. R., Germain, R. N., Altan-Bonnet, G. Variability and Robustness in T Cell Activation from Regulated Heterogeneity in Protein Levels. Science. 321 (5892), 1081-1084 (2008).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- Geva-Zatorsky, N., Rosenfeld, N., et al. Oscillations and variability in the p53 system. Mol. Syst. Biol. 2 (1), (2006).
- Colman-Lerner, A., Gordon, A., et al. Regulated cell-to-cell variation in a cell-fate decision system. Nature. 437 (7059), 699-706 (2005).
- Chong, S., Chen, C., Ge, H., Xie, X. S. Mechanism of Transcriptional Bursting in Bacteria. Cell. 158 (2), 314-326 (2014).
- Raj, A., Peskin, C. S., Tranchina, D., Vargas, D. Y., Tyagi, S. Stochastic mRNA Synthesis in Mammalian Cells. PLoS Biol. 4 (10), e309+ (2006).
- Senecal, A., Munsky, B., et al. Transcription Factors Modulate c-Fos Transcriptional Bursts. Cell Rep. 8 (1), 75-83 (2014).
- Dey, S. S., Foley, J. E., Limsirichai, P., Schaffer, D. V., Arkin, A. P. Orthogonal control of expression mean and variance by epigenetic features at different genomic loci. Mol. Syst. Biol. 11 (5), 806+ (2015).
- Dar, R. D., Razooky, B. S., et al. Transcriptional burst frequency and burst size are equally modulated across the human genome. Proc. Natl. Acad. Sci. U.S.A. 109 (43), 17454-17459 (2012).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- Porter, J. R., Fisher, B. E., Batchelor, E. p53 Pulses Diversify Target Gene Expression Dynamics in an mRNA Half-Life-Dependent Manner and Delineate Co-regulated Target Gene Subnetworks. Cell Syst. 2 (4), 272-282 (2016).
- Lahav, G., Rosenfeld, N., et al. Dynamics of the p53-Mdm2 feedback loop in individual cells. Nat. Genet. 36 (2), 147-150 (2004).
- Levine, A. J., Oren, M. The first 30 years of p53: growing ever more complex. Nat. Rev. Cancer. 9 (10), 749-758 (2009).
- Riley, T., Sontag, E., Chen, P., Levine, A. Transcriptional control of human p53-regulated genes. Nat. Rev. Mol. Cell Biol. 9 (5), 402-412 (2008).
- Ye, J., Coulouris, G., Zaretskaya, I., Cutcutache, I., Rozen, S., Madden, T. L. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinf. 13 (1), 134 (2012).
- . . PCR Technologies: A Technical Guide. , (2014).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Batchelor, E., Mock, C. S., Bhan, I., Loewer, A., Lahav, G. Recurrent initiation: a mechanism for triggering p53 pulses in response to DNA damage. Mol. Cell. 30 (3), 277-289 (2008).
- . . Flow Cytometry: Principles and Applications. , (2007).
- . . Real-time PCR. , (2006).
- Song, L., Langfelder, P., Horvath, S. Comparison of co-expression measures: mutual information, correlation, and model based indices. BMC Bioinf. 13 (1), 328 (2012).
- Margolin, A. A., Nemenman, I., et al. ARACNE: An Algorithm for the Reconstruction of Gene Regulatory Networks in a Mammalian Cellular Context. BMC Bioinf. 7 (Suppl 1), (2006).
- Haff, L. A. Improved quantitative PCR using nested primers. PCR Methods Appl. 3 (6), 332-337 (1994).
- Hashimshony, T., Senderovich, N., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biol. 17, 77 (2016).
- Ronander, E., Bengtsson, D. C., Joergensen, L., Jensen, A. T. R., Arnot, D. E. Analysis of Single-cell Gene Transcription by RNA Fluorescent In Situ Hybridization (FISH). J. Vis. Exp. (68), e4073 (2012).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat. Methods. 5 (10), 877-879 (2008).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nat. Methods. 9 (7), 743-748 (2012).
- Battich, N., Stoeger, T., Pelkmans, L. Image-based transcriptomics in thousands of single human cells at single-molecule resolution. Nat. Methods. 10 (11), 1127-1133 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved