このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
内部標準でFACSおよび定量PCRを用いた単一細胞遺伝子発現プロファイリング
要約
We describe a method to sort single mammalian cells and to quantify the expression of up to 96 target genes of interest in each cell. This method includes the use of internal qPCR standards to enable the estimation of absolute transcript counts.
要約
Gene expression measurements from bulk populations of cells can obscure the considerable transcriptomic variation of individual cells within those populations. Single-cell gene expression measurements can help assess the role of noise in gene expression, identify correlations in the expression of pairs of genes, and reveal subpopulations of cells that respond differently to a stimulus. Here, we describe a procedure to measure the expression of up to 96 genes in single mammalian cells isolated from a population growing in tissue culture. Cells are sorted into lysis buffer by fluorescence-activated cell sorting (FACS), and the mRNA species of interest are reverse-transcribed and amplified. Gene expression is then measured using a microfluidic real-time PCR machine, which performs up to 96 qPCR assays on up to 96 samples at a time. We also describe the generation and use of PCR amplicon standards to enable the estimation of the absolute number of each transcript. Compared with other methods of measuring gene expression in single cells, this approach allows for the quantification of more distinct transcripts than RNA FISH at a lower cost than RNA-Seq.
概要
集団内の個々の細胞は均一な生理的刺激1、2、3、4に大幅に異なる応答を示すことができます。集団中の細胞の遺伝的変異は、応答のこの品種のための1つのメカニズムであるが、それでも細胞のクローン集団で、応答の変動性を向上させることができ、いくつかの非遺伝的要因もあります。例えば、個々のタンパク質および他の重要なシグナル伝達分子のレベルは、下流の遺伝子発現プロファイルの変化を生じる、セル単位で変化させることができます。さらに、遺伝子活性化は、バースト7,8,9あたりの転写物の比較的少数に限定することができる転写5,6の短時間のバーストで発生することができます。そのような遺伝子活性化における偶然性が大幅に生物学的応答の変動に寄与することができ、生理的刺激に応答して、微生物10におよび哺乳動物細胞1に2を選択的利点を提供することができます。変化の遺伝的および非遺伝的供給源の両方に、刺激に応答する任意の細胞の遺伝子発現プロファイルは、バルク応答の測定から得られた平均遺伝子発現プロファイルとは大きく異なっていてもよいです。個々の細胞は、刺激に応答して可変性を示す程度を決定することは、個々の細胞の単離のための技術は、目的の転写物の発現レベルを測定し、得られた発現データのコンピュータ分析を必要とします。
コストの広い範囲をカバーし、単一細胞における遺伝子発現をアッセイするためのいくつかのアプローチがあり、転写産物の数はプローブ、及び定量化の精度。例えば、単一細胞RNA-のSeqは、転写物のカバレッジと、個々の細胞の中で最も高度に発現される遺伝子のための別個の転写産物の数千を定量する能力の広い深さを提供しています。コストが減少し続けるが、しかし、このようなシーケンシングの深さに関連するコストは、法外することができます。逆に、in situハイブリダイゼーション(FISH smRNA) における単一分子RNAの蛍光は、目的の遺伝子につき合理的なコストであっても低発現遺伝子のための転写物の正確な定量を提供しています。しかしながら、標的遺伝子の少数は、このアプローチによって、所定の細胞においてアッセイすることができます。このプロトコールに記載定量的PCRベースのアッセイは、これらの技術の間の妥協点を提供します。これらのアッセイは、最大96個の細胞で一度に関心の96転写物まで定量化するために、マイクロ流体リアルタイムPCR装置を採用しています。前述の方法のそれぞれは、必要なハードウェアのコストを有しているが、任意の個々のqPCRアッセイのコストは比較的です低いです。このプロトコルは、(プロトコルADP 41、フリュー)マイクロ流体リアルタイムPCR装置の製造元が推奨する1から適応されます。 PCRベースのアプローチで各転写産物の絶対数の推定を可能にするために、我々は、複数の実験で使用できる準備標的遺伝子アンプリコンの内部統制を利用するプロトコルを拡大してきました。
この技術の一例として、MCF-7ヒト乳癌細胞において腫瘍抑制因子p53によって調節される遺伝子の発現の定量化は、11に記載されています。細胞は、DNA二本鎖切断を誘発する化学物質でチャレンジされています。以前の研究は、DNA二重鎖切断に対するp53応答は、p53レベル12の観点と異なる標的遺伝子11の活性化の両方で、個々の細胞における異質性を大いに発揮することが示されています。さらに、p53は100以上の発現を調節します細胞周期停止、アポトーシス及び老化13、14を含む多くの下流経路に関与する標的遺伝子を十分に特徴付け。各細胞におけるp53媒介性応答は、複雑かつ可変でもあるので、アプローチからシステムの利点の分析とは、約100の標的遺伝子は、以下に記載されるような個々の細胞、同時にプローブすることができます。 (例えば、単一細胞の単離および溶解のための代替方法として)わずかな変更を加えて、プロトコルは、容易に、哺乳動物の細胞型、転写、および細胞応答の広い範囲を研究するために適合させることができます。
適切な事前準備で、細胞選別および遺伝子発現測定のラウンドは、3日間にわたって、このプロトコルに従って行うことができます。 、事前に関心の転写産物を選択し、それらのtranscrからcDNAを増幅するプライマー対を識別し、検証:以下のタイミングが示唆されていますIPTSは、および標準とそれらのプライマーを用いてプライマーミックスを準備します。細胞治療の後、1日目に、収穫と逆転写および特定の標的増幅を行い、細胞を選別し、取り込まれなかったプライマーを除去するためにエキソヌクレアーゼでサンプルを扱います。 2日目、定量PCRを使用して選別した細胞上の品質管理を行います。最後に、3日目に、マイクロ流体定量PCRを使用して選別した細胞における遺伝子発現を測定します。 図1は、必要な手順をまとめたものです。
プロトコル
1.事前準備
- その発現が測定される関心の96遺伝子まで選択。
注:これらの遺伝子の少なくとも1つは、ACTBまたはGAPDHなどの「ハウスキーピング遺伝子」である必要があり、それは、実験に用いた条件下で、相対的に高い及び一定のレベルで発現することが知られています。この遺伝子は、正にソートウェル(ステップ8.1)、増幅サンプル(ステップ10.1)を識別するために使用されます。
注:例えば、実験のために、公知の機能11、14およびハウスキーピングコントロールを選択したとしてGAPDHの様々なp53の、直接の標的を十分に特徴付けられました。本研究で用いた標的遺伝子およびプライマー配列の完全なリストについては、参考文献11を参照してください。 - 15(このようなプライマー-BLASTとして科学文献またはプライマーの設計ツールを使用して、例えば )目的の遺伝子に特異的な潜在的なプライマー対を特定します。プライマーを持っています合成だとヌクレアーゼフリー水に、100μMのストック濃度でそれらを維持します。
注:これらのプライマーは15-25塩基の長さでなければなりません。アンプリコンを90〜130塩基対(bp)の長さを生成します。存在する場合、ゲノムDNAを増幅する可能性を低減するために、エクソンジャンクションにまたがります。 60±1℃の融解温度を有します。そして、最小限の二次構造16を有しています。これらのプライマーは、目的の転写物からcDNAを増幅し、染料ベースのqPCR結合DNAにおける遺伝子発現を測定し、プライム逆転写に使用されます。 - プライマーを検証します。
- まず、目的の細胞からcDNAを得ます。製造業者の取扱説明書または標準的な分子生物学的方法17に従ってRNA採取キットを用いて細胞から収穫RNA。製造元の指示または標準メトに従ってランダムヘキサマーを用いた逆転写キットを用いてRNAをcDNAに逆転写しますDS 17。
- 各プライマー対について、DNAが前方、染料のマスターミックスを結合し、逆方向プライマー、および前のステップからのcDNA、提案した反応条件のマスターミックスメーカーの推奨と次のように定量PCRを設定します。溶融曲線の取得を含んでマスターミックスのメーカーが推奨する熱サイクルプロトコルを用いたリアルタイムPCRマシン上でこれらの反応を実行します。
- 定量PCRの後、2%のw / vのアガロースゲル上で増幅されたDNAサンプルを実行し、標準的なプロトコル17以下のDNA挿入色素を介したDNAを可視化します。
- cDNAの連続希釈液から標準曲線を構築することによって、プライマー対の効率を決定する標準的なプロトコル16に従って(ステップ1.3.1で行われました)。
注:良いプライマー対は、ゲル16の期待位置に単一の、明確に定義されたピークと融解曲線と単一バンドを生じるであろう、 クラスアップ= "外部参照"> 17( 図2)。融解曲線」は、肩」は、複数のピークまたは有するか、またはゲルが複数のバンドまたは汚れがある場合、プライマーは、オフターゲット配列を増幅している場合。オフターゲット配列を増幅し、必要に応じて前の検証手順を繰り返し観察されている任意のプライマーを再設計します。良好なプライマー対は、標準曲線16のためのR 2≥0.985で、90から110まで%の効率を持つことになります。これらの範囲外の効率またはR 2の秋のための任意のプライマーを再設計。
- 精製されたDNAアンプリコンから基準を準備します。
- 各検証プライマー対の場合:
- プライマーと鋳型DNA濃度、反応条件、及びPCRサイクル条件、または標準的なPCRプロトコールを使用するためのポリメラーゼを製造業者の推奨に従って、高忠実度DNAポリメラーゼを用いてcDNAから関心領域を増幅"外部参照"> 17。
- 2%のw / vのアガロースゲル上で増幅されたcDNAを実行し、DNA挿入色素17でバンドを可視化します。きれいなカミソリの刃を使用してアンプリコンを表すバンドを切除し、マイクロチューブにゲル片を配置します。
- 製造者の指示に従って、例えば、アガラーゼベース抽出17スピンカラムゲル抽出キットなどの標準的なゲル抽出法を用いてゲルからアンプリコンを精製します。
- 製造業者の指示に従って蛍光ベースのdsDNA定量キットを使用してアンプリコンの濃度を測定します。 1μl当たり単位複製配列分子の数を得るために、アンプリコンの配列に基づいて、単位複製配列の既知の分子量測定のdsDNAの濃度を分割します。
- 2.0ミリリットルの低結合マイクロチューブでは、DNAの懸濁緩衝液の体積に精製されたアンプリコンの2μlを添加するようにアンプの最終濃度liconは5×10 8分子/μlです。
- すべてのアンプリコンは精製されている場合には、2.0ミリリットルの低結合マイクロチューブに各精製されたアンプリコンの18μLを兼ね備えています。 1,800μLの総容量を作るためにDNAを懸濁緩衝液を加えます。このように、 各アンプリコンの濃度が5×10 6分子/μlです。
注:既知の等モル量で関心のあるすべての遺伝子からのcDNAアンプリコンが含まれている。この混合物は、単一セルのqPCRにおける標準となります。 - 渦この「5E6」標準徹底的に、2000×gで10秒間スピン、低結合PCRチューブ中の20μlのアリコートに分割します。 -80℃でアリコートを保管してください。
- 各検証プライマー対の場合:
- 特定の標的増幅(STA)プライマーミックス(10倍)を準備します。
- 15ミリリットルコニカルチューブでは、(192まで)、各100μMのストックプライマー25μlのを兼ね備えています。 5,000μLの総容量を作るためにDNAを懸濁緩衝液を加えます。
注記:この混合物中の各プライマーの濃度は500 nmです。選別された細胞の各プレートは、プライム逆転写および特定の標的増幅にこのプライマーミックスの105.6μLを使用します。ここで与えられたプライマーミックスのボリュームは、ソートに45版を作るのに十分です。一度だけ行われる必要があるようにプライマー混合物の十分に大きな容積を作成することが望ましいです。必要に応じてボリュームを拡張できます。 - 110μlのアリコートに分割し、ボルテックスで混合し、-20℃でアリコートを保存します。
- 15ミリリットルコニカルチューブでは、(192まで)、各100μMのストックプライマー25μlのを兼ね備えています。 5,000μLの総容量を作るためにDNAを懸濁緩衝液を加えます。
- マイクロ流体チップベースのqPCRでの使用のために、アッセイミックス、各プライマー対のための1を準備します。
- 96ウェルPCRプレートの各ウェルには、所定の遺伝子のために対応する100μMリバースプライマーの3.0μlを、DNAの懸濁緩衝液の24.0μlを、そして2×アッセイローディング試薬の30.0μLを100μMのフォワードプライマーの3.0μLを組み合わせます。
- 渦このプレート、500×gで10秒間スピンし、-20℃で保存。
- ;およびEXOI(エキソヌクレアーゼI処理; 表2)RTSTA( 表1転写および特定の標的増幅を逆):2熱サイクルプログラムを作成します。
2.治療
- それらは実験的研究の条件に応じて、処理の際に所望のコンフルエンスまで成長するように、組織培養皿上で哺乳動物細胞をプレート。この研究で提示され、例えば、10%ウシ胎児血清(FBS)を含むRPMI中で、プレート4×10 5個のMCF-7のp53ビーナス細胞18 6当たり10cmディッシュ、100 U / mlペニシリン、100ミリグラム/ mlのストレプトマイシン、および250 ngの/ 5%CO 2で37℃で2日間mlのアムホテリシンBをインキュベート細胞彼らは50%の密集度に達するまで。
- 実験的研究に基づいて、関心の条件を確立するために、細胞を処理します。本研究で提示例えば、400 ngの/ mlのneocarzinoでMCF-7のp53-金星細胞をインキュベート3時間、8.5時間、14時間、または24時間スタチン。
細胞選別のための3の溶解緩衝液の準備
注:溶解緩衝液の単板を作るには、約1時間かかります。細胞選別は非効率的であるとは検出されなかった細胞で多くの井戸を得ることができるように、複数のプレートを作成し、ソートすることをお勧めします。
- PCRの準備のためにDNAの除染液とベンチ、ピペット、チューブラック、コールドPCRプレートホルダー、手袋を清掃してください。
- ヌクレアーゼフリー水を1.5mlチューブに株式RNase阻害剤を添加することにより、2.64 U /μlのRNアーゼ阻害剤を希釈します。
注:これは、RNアーゼ阻害剤は、サブマイクロリットル容積をピペッティングすることなく、溶解緩衝液に添加されることを可能にします。 - 表3に列挙した成分を組み合わせることによって、細胞のための溶解緩衝液を作ります。
- 別のチューブへの細胞のための溶解バッファーの92.4μLを移します。これは、アンプリコン標準の溶解バッファーになります。
- LOを使用しましたワット結合ピペットチップやマイクロ遠心チューブ、DNA懸濁液緩衝液で、それぞれ、200μlの3.1 PG /μlの6.2 PG /μlに希釈した大腸菌 DNAの500μlのを準備します。
- 細胞のための溶解緩衝液に3.1 PG /μlの大腸菌 DNAの184.8μLを加えます。
注: 大腸菌キャリアDNAは、非特異的プライマー結合の可能性の範囲を広げることにより、プライマー二量体は、PCR産物につながる可能性を低減するのに役立ちます。 - 基準を準備します。
- 5E5、5E4、... 5e0として6低結合マイクロ遠心チューブにラベルを付けます。
- 「5E5」のチューブにDNAを懸濁緩衝液の90μLを加えます。
- 他の5つの管のそれぞれに6.2 PG /μlの大腸菌 DNAの90μLを加えます。氷の上ですべてのチューブを保管してください。
注:規格にキャリアDNAを組み込むには、1セルのウェル中に匹敵する標準ウェルにおけるDNAの総質量になります。これは、PRを低減します基準濃度はチューブ又はピペットチップへのDNA結合によって影響されることobability。 - ストレージから(ステップ1.4で調製した各アンプリコンの5×10 6分子/μL、)「5E6」標準のアリコートを取ります。簡単にボルテックスし、液体がチューブの底にあることを保証するために、簡単に(500×gで5秒)スピン。
- 低結合ピペットチップを使用して、「5E5」チューブに5E6標準の10μlを添加します。簡単にボルテックス、スピン(500×gで5秒)、そして氷上に戻ってチューブを置きます。
- 「5E4」チューブに「5E5」管からのピペットを10μl。簡単にボルテックス、スピン(500×gで5秒)、そして氷上に戻しました。
- すべてのチューブは、標準の希釈になるまで、各チューブが前のチューブから10μlのを受けて、これらの連続希釈液を繰り返します。
- キャップで12 PCRチューブの行を準備します。各チューブに、「細胞」の溶解緩衝液67μLを配布します。チューブに蓋をし、簡単にスピンダウン(5500×gで秒)必要であれば。
- 12チャンネルピペットを使用して、PCRプレートの最初の7列の各ウェルにPCRチューブの行から「細胞」の溶解緩衝液( 図3)の9μLを分配します。
- プレートの最後の行( 図3)の各ウェルに、「標準」の溶解緩衝液7μLを配布します。
- 低結合ピペットチップを使用して、プレートマップ( 図3)に記載の最下行の各ウェルに標準の2μlを添加します。
注: など 10分子、5×10 0個/μlの標準量の2μlの - 10細胞および100細胞ウェルに3.1 PG /μlの大腸菌 DNAの2μLを加えます。無細胞ウェルに6.2 PG /μlの大腸菌 DNAの2μlを添加します。
注:各1セル、10セル、および100細胞はよく単一のヒト細胞では、ゲノムDNAの質量を近似、 大腸菌 DNAの6.2 pgのを持っています。各標準および無細胞ウェルが12.4 pgのO持ちますF 大腸菌 DNA、およそ2つのヒト細胞の価値があります。 - 細胞はソーティングのための準備が整うまで4℃で滅菌した熱シール、500×gで5秒間スピンし、ストアを使用してプレートをシール。
4.セルソーターのセットアップ
- プログラムプレートマップ( 図3)によれば、96ウェルプレートに選別し、蛍光活性化セルソーター。何の細胞をウェルH1-H8またはH11-H12に分類されるべきではありません。
- セルソーターは、ウェルPCRプレートに並べ替えを目的としていることを確認します。
- 可能な限り最小にソートストリームの角度を設定します。この設定は、細胞が側面を打つのではなく、井戸の底に分類される可能性を増大させます。
- マシンを目指すためには、機械19の指示に従って、空のPCRプレートを密封し、空のプレートのシールにPBSの「並べ替え」の液滴にテストストリームを使用します。液滴が目に着陸する必要がありますウェルの中央より上の位置でのシールの電子表面。最良の結果を得るために、プレートの長さと幅を横切って目的を確認します。
- 液滴が正しい位置に上陸しない場合は、シールの表面を、それらを拭き取り、機械メーカーの指示に従って、セルソーターを再キャリブレーション、およびソート・ストリーム内の液滴を正確に堆積されるまで繰り返します。
5.細胞収穫と選別
- 細胞株のような適切な細胞をトリプシン処理します。例えば、MCF-7細胞について、0.05%トリプシン1mlで細胞をすすぎ、細胞から培地を吸引し、37℃、5%CO 2で5分間0.05%トリプシン2mLで細胞をインキュベート。
- トリプシン処理した細胞に細胞培養培地を8mlを加え、15mlチューブに細胞懸濁液の全10 mlで移します。 400×gで4分間遠心分離します。
- 上清を吸引除去し、PBS + 2%FBS中で細胞ペレットを再懸濁します。
注:小さい再懸濁体積は細胞を集中し、細胞選別を容易にします。 500μlの再懸濁量は50%の集密度で細胞を6 10cmディッシュに適しています。 - 細胞の塊を除去するために、40ミクロンのフィルターを通して、チューブサイトメトリーピペッティングを流すmlの5に細胞懸濁液を転送します。
- 液体はウェルの底にあることを保証するために、細胞を選別する前に、4℃で30秒間、400×gでの溶解緩衝液プレートを遠心。
- セルソーターに細胞懸濁液をチューブを挿入します。溶解バッファーの板にシールを開き、慎重にプレートマップに従って、セルソーターの製造元の指示19を次のプレートに関心の細胞を選別。単一細胞が最大純度でソートされていることを確認するには、「単一セル」モードでマシンを実行し、前方および側方散乱光19に基づいて、標準的なフローサイトメトリーゲーティングを使用します。
注:中実験例、個々のセル11の最も明るい15%を単離するために、p53を金星の蛍光に基づいて、ゲートの細胞。 - 4℃で1分間、400×gで新しい、滅菌熱シールや遠心分離機でプレートをシール。サーマルサイクラーに板を置き、RTSTAプログラム(ステップ1.7を参照)を実行します。
注:これは分類された細胞からのmRNAの逆転写を行い、次いで、目的の転写産物のcDNAを増幅します。
6.エキソヌクレアーゼ処理
- 表4に列挙した成分を組み合わせることによって希釈エキソヌクレアーゼIを混ぜます。キャップで12 PCRチューブの行を準備します。各チューブに、希釈エキソヌクレアーゼIの30.5μLを配布します。チューブに蓋をし、液体がチューブの底にあることを保証するために、簡単に(500×gで5秒)スピン。
- RTSTAが終了すると、500×gで5秒間のサンプルプレートをスピン。 12チャンネルピペットを用い、希釈エキソヌクレアーゼの3.6μLを分配しますIプレートの各ウェルに。
- 滅菌熱シールでプレートをシールし、(500×gで5秒)を簡単にスピン。サーマルサイクラーに板を置き、EXOIプログラム(ステップ1.7を参照)を実行します。
注:彼らは将来のPCRに干渉しないように、このステップは、特定の標的増幅後に残っている任意のプライマーを劣化させます。
7.サンプル希釈
- エキソヌクレアーゼ処理が終了すると、サンプルプレートの各ウエルにTE緩衝液32.4μlを添加します。これは、任意の停止点であり、サンプルは数日間-20℃で保存することができます。
定量PCRを使用した8ソート品質管理
注:細胞選別が完全に効率的ではないので、このステップは、ソートプレートのウェルは、実際にセルを受信し識別することが必要です。これらのサンプルは、さらなる分析のために使用することができます。
- qPCRにより、各試料中のハウスキーピング遺伝子の発現を測定します。
- ステップ1.1で選択したハウスキーピング遺伝子のいずれかのプライマーを用いてポリメラーゼの製造元のプロトコルに従って96の反応のための染料のqPCRマスターミックスをDNA結合900μlのを準備します。この反応のため等の反応緩衝液、ポリメラーゼ、プライマーの正確な濃度は、使用される特定のポリメラーゼに依存するであろう。
- 96ウェルPCRプレートの各ウェルにマスターミックスの9μLを配布します。 PCRプレートの対応するウェルにサンプルプレートからサンプルを1μlを追加します。
- マスターミックス製造元が推奨する熱サイクルプロトコルを使用するか、または標準プロトコル20次のリアルタイムPCR装置にプレートを実行します。
- qPCRデータ20を分析します 。下記のソート品質制御ステップからのqPCRデータの例は図4に示されています。
- 無細胞ウェルからの測定値が低い(バックグラウンド)レベルを示していることを確認します遺伝子発現または全く発現。
- 無細胞ウェルに似た高い遺伝子発現および発現または全く発現のバックグラウンドレベルで陰性サンプルで陽性サンプル、:1 - 細胞ウェルからの測定値は、二つのグループに明確に分けることを確認します。
注:高い遺伝子発現を有するサンプルは、実際の選別された細胞を表します。バックグラウンド発現を有するものが、さらなる分析から除外すべきです。 - 10細胞および100-細胞ウェルからの測定値は、非効率的なソートがそれらのウェルが望まれるより少ない細胞を持たせることができる可能性を考慮して、正の1セルウェルよりも高い遺伝子発現を反映していることを確認します。
- 標準ウェルから測定値が均等に(C t値が均等に分布していること、すなわち )対数空間に分布していることを確認します。
- すべての正の1セルのサンプルを特定した後、正1-CEをマップするために、「サンプルミックス」のプレートマップを作ります新しい96ウェルプレート上にLLサンプル。このプレートマップ上の標準の8ウェルが含まれます。例えば、図5を参照してください。
マイクロ流体定量PCRを使用した9遺伝子発現測定
注:このセクションのすべてのステップのために、試薬中の気泡の形成を最小限にするための唯一の最初のピットストップにピペット。
- 8 PCRチューブの行を開きます。サンプルの複数のプレートがある場合、8 PCRチューブの2行目を開き、チューブ1-8を標識します。この行には、複数のプレートから基準をプールするためになります。
- 1.5mlチューブにおいて、色素サンプルローディング試薬を結合するDNAの36μlのDNA結合色素定量PCRマスターミックスの360μLを混ぜます。渦簡単にと2000×gで10秒間スピン。各チューブに48μlずつ配置し、8 PCRチューブの標識されていない行にこの混合物(396μl)を分割します。
- 8チャンネルピペットを用いて、各ウェルにマスターミックス/サンプルローディング試薬混合物の3.3μLを配布新しい96ウェルPCRプレート。このプレート、ラベル「サンプルミックスを。」
- サンプルの各PCRプレート用:
- 渦500×gで10秒間10秒とスピンのためのプレート。カバーを取り外し、そのは、サンプルに対応するウェルにそれぞれ正1 - 細胞サンプルの2.7μLを移す1-細胞ウェルのためのプレートをミックスプレートマップに示されています。
- サンプルの一つだけのプレートがある場合は、そのは、サンプルに対応するウェルに移し、各増幅された標準の2.7μlをプレートマップに従ってプレートをミックスします。
- サンプルの複数のプレートがある場合、PCRチューブの標識された列内の対応する位置にそれぞれ増幅標準の2.7μLを移します。すでにそこに任意の規格にサンプルの整流板から基準を追加します。封止フィルムを用いて行ったときのサンプルプレートをシール。
- サンプルの複数のプレートがある場合:
- 基準を含むPCRチューブの列を渦2,000×gで10秒間10秒とスピン。よくサンプルに対応するに混合標準の各チューブから2.7μLを移し、プレートマップに従ってプレートをミックスします。
- プレートマップ上に表示ヌクレアーゼフリー水2.7μlのNTC井戸をロードします。必要になるまで、サンプルを4℃でプレートや店舗をミックスシール。
- ピールステッカー新しいマイクロ流体定量PCRチップの底オフ。まだ上にキャップと制御線流体の注射器を使用して、それを緩めるために、各アキュムレータスプリングを押し下げ、制御線液の150〜200μlの各アキュムレータを注入します。
- 搭載機にチップを挿入し、「プライム」スクリプトを実行します。これは〜20分かかります。
- プライミングは、渦を行って、アッセイ混合物のプレートをスピンダウンした場合、サンプルミックス(ステップ1.6で調製しました)。
- 8チャンネルピペットを用いて、図中によるマイクロ流体定量PCRチップの左側に各アッセイミックスの4.5μLを移しますグラム>図6。ウェルの底に気泡の発生を防ぐために非常に慎重にピペット。終了したら、将来の使用のために-20℃でアッセイミックスや店舗のプレートをシール。
- 図6によるマイクロ流体定量PCRチップの右側に、各サンプルミックスの4.5μLを移します。搭載機にチップを挿入し、「ロード・ミックス "スクリプトを実行します。これは〜90分かかります。
- 、マイクロ流体リアルタイムPCR装置の電源をオンにし、データ収集ソフトウェアを起動し、それをウォームアップするためにランプを点灯。これは〜20分かかります。
- ロードミックススクリプトが実行されると、チップ-この全体の何行がないことを確認すると、ロードの問題となります。慎重にスコッチテープやラボのテープを使用して、チップからほこり粒子を除去。マイクロ流体リアルタイムPCR機にチップを挿入し、データ収集スクリプトを実行します。結果を検査し、さらなる分析のためのデータをエクスポートする解析ソフトウェアを使用してください。
- 定量PCR増幅曲線は20(曲線は、標準的なS字型増幅曲線に似ていない、すなわち 、)低品質を示している測定値を削除します。
- 30以上の低品質またはゼロ測定またはのためのハウスキーピング遺伝子の発現を有するサンプルを削除する(ステップ1.1参照)他のサンプル( 図7)の大部分よりもはるかに低いです。
- 各遺伝子について、各試料からの溶融ピーク位置の中央値として正確な融解ピーク位置を推定します。測定値が中央値から以上1°Cに位置する溶融ピークを持っている場合、考慮20からその測定値を削除します。
- 各遺伝子について、スプレッドシートやスクリプト言語20を使用して、各サンプル中のmRNAの数を推定するための標準曲線を作成します。
注:この実験での標準は10、100、千から成り、または10,000の分子dsDNAの関心の各転写物に対応します。逆転写は完全に効率的であった場合には、mRNAおよびcDNAは一本鎖であるため、基準は、20、200、2,000、およびmRNA 20,000の分子に対応します。実際には、これはそうではありませんので、我々は、E RTは逆転写の効率であるE RT、×分子単位の推定値として測定値を表現します。- 各標準ウェルについて、 メートル 、増幅前のssDNAの分子の数(dsDNAの分子の数の2倍)( 例えば、20分子、200分子など )を識別します。
- 各標準ウェルについて、定量PCR 20からのC t値を識別します。
- mおよびC tの値と、各遺伝子のパラメータaおよびbを有する標準曲線を生成するために、以下の式を用いて線形回帰を実行し、のようなR 2の値を見つけます適合度のn個の指標:
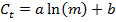
- 標準曲線から、各遺伝子のPCR効率Eを計算します :
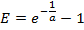
注:Eの値1.0よりもはるかに大きい( 例えば、E> 1.1)は、物理的に現実的ではありません。 1.0よりもはるかに少ない線形回帰( 例えば、R 2 <0.985)のためのR 2値は、基準が確実に動作していないことを意味します。これらの問題は、一般的に最も低いアンプリコンの標準は、信頼性の高い定量化の閾値を下回った、従って最低の標準は、線形回帰から除外されるべきであることを示しています。適切な線形回帰は、少なくとも3つのユニークな基準を使用して作製することができない場合には、遺伝子の発現の測定を捨てます。 - 線形回帰が適切である場合、得られた適合パラメータaおよびbを使用して<mRNAの数を推定するために、各遺伝子について/ em>の測定されたC tの値から、各単一細胞サンプル中の対応する遺伝子のために(E RT×分子の単位で) メートルの東側を分子:
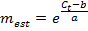
- また、m個のEST = 0として任意の測定メートルの東側 <1を指定し、= 0メートルの東側として定量PCRで増幅し、したがってなしのC t値なしで任意の測定値を指定します。
注:estの最低未満または回帰で使用される最も高い標準よりも大きいmの測定は、標準曲線から外挿によって発見されました。これらの推定値は必ずしも無効ではありませんが、慎重にみなされるべきです。
- 単一細胞の遺伝子発現データを視覚化します。
- beeswarmプロット(ドーン、J.聖、T.、2012、http://www.mathworks.com/matlabcentral/fileeを作ります各点は、特定の細胞における遺伝子発現を表す、XCHANGE / 37105);例えば、 図8に示されています。
- 「バイオリン」の幅がで特定のレベルのまわりの遺伝子発現測定の周波数を表しているバイオリンプロット(ドーン、J.、2012、http://www.mathworks.com/matlabcentral/fileexchange/23661)を作ります分析した細胞の集団。例えば、 図8に示されています。
注:より洗練された分析は、遺伝子21のペア間の発現の関係をプローブ22および遺伝子発現ネットワークを推測することができます。
結果
プロトコルの一般的な概要は、細胞治療、FACSによる単一細胞の単離、単一細胞溶解物からのcDNAライブラリーの生成および前増幅、単一細胞cDNAライブラリーを確認するステップを含む、 図1に示されていますソートされたウェル、および定量PCRにより遺伝子発現を測定します。
単一細胞の単離および遺伝子発?...
ディスカッション
我々は、培養で増殖させた接着性細胞の集団から個々の哺乳動物細胞を単離するため、各セル内の約96の遺伝子の発現をアッセイするための方法を提示しています。この方法がうまく機能するために良好な事前準備が重要です。プライマーは、単セル測定の品質を決定するように、特に興味のある転写物(1.2〜1.3ステップ)に特異的な設計およびテストプライマーペアは、時間がかかるが、?...
開示事項
The authors have nothing to disclose.
謝辞
私たちは、このプロトコルの開発中に細胞選別を行うの彼女の援助のためにCCR ETIBフローサイトメトリーコアでV.カプールに感謝したいと思います。また、このプロトコルの開発中に定量PCRを行う際の援助のためにM. RaffeldとCCR LP分子診断ユニットとJ.朱とNHLBI DNA配列決定およびゲノミクスコアに感謝します。この研究は、NIHの学内プログラムによってサポートされていました。
資料
| Name | Company | Catalog Number | Comments |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| High Capacity cDNA Reverse Transcription Kit with RNase Inhibitor | ThermoFisher | 4374966 | |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530S | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| Quant-iT High-Sensitivity dsDNA Assay Kit | ThermoFisher | Q33120 | |
| 2.0 ml low adhesion microcentrifuge tubes | USA Scientific | 1420-2600 | |
| DNA Suspension Buffer | Teknova | T0221 | |
| Axygen 0.2 ml Maxymum Recovery Thin Wall PCR Tubes | Corning | PCR-02-L-C | |
| GE 96.96 Dynamic Array DNA Binding Dye Sample & Assay Loading Reagent Kit | Fluidigm | 100-3415 | |
| HyClone RPMI 1640 media | GE Healthcare Life Sciences | SH30027.01 | |
| Fetal Bovine Serum, Certified (US) | ThermoFisher | 16000-044 | |
| Antibiotic-Antimycotic Solution | Corning | 30-004-CI | |
| Neocarzinostatin | Sigma | N9162 | |
| ELIMINase | Decon Labs | 1101 | |
| SUPERase-In | ThermoFisher | AM2696 | |
| CellsDirect One-Step qRT-PCR Kit | ThermoFisher | 11753500 | |
| E. coli DNA | Affymetrix | 14380 10 MG | |
| ThermalSeal Sealing Film, Sterile | Excel Scientific | STR-THER-PLT | |
| BD FACSAria IIu | BD Biosciences | ||
| HyClone Trypsin 0.05% | GE Healthcare Life Sciences | SH30236.01 | |
| PBS, 1x | Corning | 21-040-CV | |
| Falcon 40 µm Cell Strainer | Corning | 352340 | |
| Exonuclease I | New England BioLabs | M0293S | |
| SsoFast EvaGreen Supermix with Low ROX | Bio-Rad | 172-5210 | |
| 96.96 Dynamic Array IFC for Gene Expression (microfluidic qPCR chip) | Fluidigm | BMK-M-96.96 | |
| IFC Controller HX (loading machine) | Fluidigm | ||
| BioMark or BioMark HD (microfluidic qPCR machine) | Fluidigm | ||
| Real-Time PCR Analysis software | Fluidigm | ||
| MATLAB software | MathWorks |
参考文献
- Feinerman, O., Veiga, J., Dorfman, J. R., Germain, R. N., Altan-Bonnet, G. Variability and Robustness in T Cell Activation from Regulated Heterogeneity in Protein Levels. Science. 321 (5892), 1081-1084 (2008).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- Geva-Zatorsky, N., Rosenfeld, N., et al. Oscillations and variability in the p53 system. Mol. Syst. Biol. 2 (1), (2006).
- Colman-Lerner, A., Gordon, A., et al. Regulated cell-to-cell variation in a cell-fate decision system. Nature. 437 (7059), 699-706 (2005).
- Chong, S., Chen, C., Ge, H., Xie, X. S. Mechanism of Transcriptional Bursting in Bacteria. Cell. 158 (2), 314-326 (2014).
- Raj, A., Peskin, C. S., Tranchina, D., Vargas, D. Y., Tyagi, S. Stochastic mRNA Synthesis in Mammalian Cells. PLoS Biol. 4 (10), e309+ (2006).
- Senecal, A., Munsky, B., et al. Transcription Factors Modulate c-Fos Transcriptional Bursts. Cell Rep. 8 (1), 75-83 (2014).
- Dey, S. S., Foley, J. E., Limsirichai, P., Schaffer, D. V., Arkin, A. P. Orthogonal control of expression mean and variance by epigenetic features at different genomic loci. Mol. Syst. Biol. 11 (5), 806+ (2015).
- Dar, R. D., Razooky, B. S., et al. Transcriptional burst frequency and burst size are equally modulated across the human genome. Proc. Natl. Acad. Sci. U.S.A. 109 (43), 17454-17459 (2012).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- Porter, J. R., Fisher, B. E., Batchelor, E. p53 Pulses Diversify Target Gene Expression Dynamics in an mRNA Half-Life-Dependent Manner and Delineate Co-regulated Target Gene Subnetworks. Cell Syst. 2 (4), 272-282 (2016).
- Lahav, G., Rosenfeld, N., et al. Dynamics of the p53-Mdm2 feedback loop in individual cells. Nat. Genet. 36 (2), 147-150 (2004).
- Levine, A. J., Oren, M. The first 30 years of p53: growing ever more complex. Nat. Rev. Cancer. 9 (10), 749-758 (2009).
- Riley, T., Sontag, E., Chen, P., Levine, A. Transcriptional control of human p53-regulated genes. Nat. Rev. Mol. Cell Biol. 9 (5), 402-412 (2008).
- Ye, J., Coulouris, G., Zaretskaya, I., Cutcutache, I., Rozen, S., Madden, T. L. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinf. 13 (1), 134 (2012).
- . . PCR Technologies: A Technical Guide. , (2014).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Batchelor, E., Mock, C. S., Bhan, I., Loewer, A., Lahav, G. Recurrent initiation: a mechanism for triggering p53 pulses in response to DNA damage. Mol. Cell. 30 (3), 277-289 (2008).
- . . Flow Cytometry: Principles and Applications. , (2007).
- . . Real-time PCR. , (2006).
- Song, L., Langfelder, P., Horvath, S. Comparison of co-expression measures: mutual information, correlation, and model based indices. BMC Bioinf. 13 (1), 328 (2012).
- Margolin, A. A., Nemenman, I., et al. ARACNE: An Algorithm for the Reconstruction of Gene Regulatory Networks in a Mammalian Cellular Context. BMC Bioinf. 7 (Suppl 1), (2006).
- Haff, L. A. Improved quantitative PCR using nested primers. PCR Methods Appl. 3 (6), 332-337 (1994).
- Hashimshony, T., Senderovich, N., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biol. 17, 77 (2016).
- Ronander, E., Bengtsson, D. C., Joergensen, L., Jensen, A. T. R., Arnot, D. E. Analysis of Single-cell Gene Transcription by RNA Fluorescent In Situ Hybridization (FISH). J. Vis. Exp. (68), e4073 (2012).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat. Methods. 5 (10), 877-879 (2008).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nat. Methods. 9 (7), 743-748 (2012).
- Battich, N., Stoeger, T., Pelkmans, L. Image-based transcriptomics in thousands of single human cells at single-molecule resolution. Nat. Methods. 10 (11), 1127-1133 (2013).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved