Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Unicellulaire profil d'expression génique utilisant FACS et qPCR avec les normes internes
Dans cet article
Résumé
We describe a method to sort single mammalian cells and to quantify the expression of up to 96 target genes of interest in each cell. This method includes the use of internal qPCR standards to enable the estimation of absolute transcript counts.
Résumé
Gene expression measurements from bulk populations of cells can obscure the considerable transcriptomic variation of individual cells within those populations. Single-cell gene expression measurements can help assess the role of noise in gene expression, identify correlations in the expression of pairs of genes, and reveal subpopulations of cells that respond differently to a stimulus. Here, we describe a procedure to measure the expression of up to 96 genes in single mammalian cells isolated from a population growing in tissue culture. Cells are sorted into lysis buffer by fluorescence-activated cell sorting (FACS), and the mRNA species of interest are reverse-transcribed and amplified. Gene expression is then measured using a microfluidic real-time PCR machine, which performs up to 96 qPCR assays on up to 96 samples at a time. We also describe the generation and use of PCR amplicon standards to enable the estimation of the absolute number of each transcript. Compared with other methods of measuring gene expression in single cells, this approach allows for the quantification of more distinct transcripts than RNA FISH at a lower cost than RNA-Seq.
Introduction
Les cellules individuelles dans une population peuvent montrer des réponses très différentes à un stimulus uniforme physiologique 1, 2, 3, 4. La variation génétique des cellules dans une population est un mécanisme pour cette série de réactions, mais il existe également plusieurs facteurs non génétiques qui peuvent augmenter la variabilité des réponses, même dans une population clonale de cellules. Par exemple, les niveaux de différentes protéines et d'autres molécules de signalisation importantes peuvent varier sur une base cellule par cellule, donnant lieu à des variations dans les profils d'expression des gènes en aval. En outre, l' activation du gène peut se produire dans des éclats de courte durée des relevés de notes 5, 6 qui peuvent être limités à un nombre relativement faible de transcrits par rafale 7, 8, 9. Telstochasticité dans l' activation de gène peut grandement contribuer à la variabilité des réponses biologiques et peut fournir un avantage sélectif sur les micro - organismes 10 dans des cellules mammifères et 1, 2 répondant à un stimulus physiologique. En raison de ces deux sources génétiques et non génétiques de la variation, le profil d'expression génique d'une cellule donnée en réponse à un stimulus peut être très différente du profil d'expression génique moyenne obtenue à partir de la mesure de la réponse en vrac. Déterminer la mesure dans laquelle les cellules individuelles présentent une grande variabilité dans la réponse à un stimulus nécessite des techniques pour l'isolement de cellules individuelles, la mesure des niveaux d'expression des transcrits d'intérêt, et l'analyse informatique des données d'expression en résultant.
Il existe plusieurs approches pour le dosage de l'expression des gènes dans des cellules individuelles, couvrant un large éventail de coûts, nombre de transcrits sondé, etprécision de quantification. Par exemple, une seule cellule ARN-Seq offre une grande profondeur de la couverture de la transcription et de la capacité à quantifier des milliers de transcriptions distinctes pour les gènes les plus fortement exprimé dans des cellules individuelles; Cependant, le coût associé à une telle profondeur de séquençage peut être prohibitif, même si les coûts continuent de diminuer. A l' inverse, une seule molécule d' ARN hybridation fluorescente in situ (smRNA FISH) propose une quantification précise des transcriptions pour encore faible expression de gènes à un coût raisonnable par gène d'intérêt; Cependant, seul un petit nombre de gènes cibles peut être testée dans une cellule donnée par cette approche. tests basés sur la PCR quantitative, décrits dans le présent protocole, fournissent un juste milieu entre ces techniques. Ces tests utilisent une machine microfluidique PCR en temps réel pour quantifier jusqu'à 96 transcriptions d'intérêt à un moment dans un maximum de 96 cellules. Bien que chacune des méthodes mentionnées ci-dessus a des coûts matériels nécessaires, le coût de tout essai qPCR individuelle est relativementfaible. Ce protocole est adapté de celui suggéré par le fabricant d'une machine microfluidique PCR en temps réel (Protocole ADP 41, Fluidigm). Pour permettre l'estimation du nombre absolu de chaque transcription dans une approche basée sur la PCR, nous avons élargi le protocole à utiliser des contrôles internes de préparées amplicons de gènes cibles qui peuvent être utilisés dans de multiples expériences.
A titre d'exemple de cette technique, la quantification de l'expression des gènes régulés par le suppresseur de tumeur p53 dans les cellules MCF-7 de carcinome du sein humain 11 est décrit. Les cellules sont contestées avec un agent chimique qui induit ADN cassures double brin. Des études antérieures ont montré que la réponse de p53 à l' ADN cassures double brin présente une grande hétérogénéité dans des cellules individuelles, tant en termes de niveaux de p53 12 et dans l'activation de gènes cibles distinctes 11. En outre, p53 régule l'expression de plus de 100bien caractérisé des gènes cibles impliqués dans de nombreuses voies en aval, y compris l' arrêt du cycle cellulaire, l' apoptose et la sénescence 13, 14. Puisque la réponse médiée par p53 dans chaque cellule est à la fois complexe et variable, l'analyse des avantages du système à partir d'une approche dans laquelle près de 100 gènes cibles peut être sondé simultanément dans des cellules individuelles, telles que celles décrites ci-dessous. Avec de légères modifications (telles que les méthodes alternatives pour l'isolement de cellules isolées et une lyse), le protocole peut être facilement adapté pour étudier un large éventail de types de cellules de mammifère, des transcriptions et des réponses cellulaires.
Avec une préparation préalable appropriée, une ronde de tri cellulaire et la mesure de l'expression du gène peut être réalisée selon ce protocole sur une période de trois jours. Le calendrier suivant est proposé: à l'avance, sélectionner les transcriptions d'intérêt, d'identifier et de valider les paires d'amorces qui amplifient l'ADNc de ceux transcrIPTS, et de préparer les normes et mélanges d'amorces en utilisant ces amorces. Le jour 1, après un traitement cellulaire, la récolte et de trier les cellules, effectuer la transcription inverse et l'amplification de cible spécifique, et de traiter les échantillons avec une exonucléase pour éliminer les amorces non constituées en société. Le jour 2, effectuer un contrôle de qualité sur les cellules triées en utilisant qPCR. Enfin, le jour 3, mesurer l'expression des gènes dans les cellules triées en utilisant microfluidique qPCR. La figure 1 résume les différentes étapes.
Protocole
1. Préparation à l'avance
- Sélectionnez jusqu'à 96 gènes d'intérêt dont l'expression sera mesurée.
REMARQUE: Au moins un de ces gènes devrait être un «gène de ménage», comme ACTB ou GAPDH, qui est connu pour être exprimé à un niveau relativement élevé et constant dans les conditions utilisées dans l'expérience. Ce gène sera utilisé pour identifier positivement les puits triés (étape 8.1) et des échantillons amplifiés (étape 10.1).
NOTE: Pour l'exemple, bien caractérisé, les cibles directes de p53 avec une variété de fonctions connues 11, 14 et GAPDH comme un contrôle de ménage ont été sélectionnés. S'il vous plaît voir la référence 11 pour la liste complète des gènes cibles et des séquences d'amorces utilisées dans cette étude. - Identifier les paires possibles d'amorces spécifiques pour les gènes d'intérêt (par exemple, en utilisant la littérature scientifique ou un outil de conception d'amorce tels que Primer-BLAST) 15. Avoir l'amorces synthétisé et de les maintenir à une concentration stock de 100 pM dans de l'eau sans nucléase.
NOTE: Ces amorces doivent être 15-25 bases de long; produire un amplicon 90-130 paires de bases (pb) de long; couvrir une jonction exon, le cas échéant, pour réduire le risque d'amplifier l'ADN génomique; ont des températures de fusion de 60 ± 1 ° C; et ont une structure secondaire minimale 16. Ces amorces seront utilisées pour amorcer la transcription inverse, amplifier l'ADNc des transcrits d'intérêt, et de mesurer l'expression génique dans l'ADN de liaison qPCR à base de colorant. - Valider les amorces.
- Tout d'abord, obtenir un ADNc à partir de cellules d'intérêt. L' ARN récolté à partir des cellules en utilisant un kit d' ARN de récolte selon les instructions du fabricant ou les procédés standards de biologie moléculaire 17. Reverse-transcrire l'ARN en ADNc en utilisant un kit de transcription inverse avec des hexamères aléatoires selon les instructions du fabricant ou métho normeds 17.
- Pour chaque paire d'amorces, mis en place qPCR avec liaison à l'ADN colorant mélange maître, amorces directe et inverse, et l'ADNc de l'étape précédente, en suivant les recommandations du fabricant de mélange maître pour les conditions de réaction proposées. Exécuter ces réactions sur une machine de PCR en temps réel en utilisant un protocole de cyclage thermique recommandé par le fabricant de mélange maître qui comprend l'acquisition de la courbe à l'état fondu.
- Après la qPCR, exécutez les échantillons d' ADN amplifiés sur un gel d'agarose à 2% p / v et de visualiser l'ADN via un colorant intercalant de l' ADN suivant un protocole standard 17.
- Déterminer l'efficacité de la paire d' amorces en construisant une courbe d' étalonnage à partir de dilutions en série d'ADNc (effectuée à l' étape 1.3.1) , selon un protocole standard 16.
NOTE: Une bonne paire d'amorces donnera une courbe de fusion avec un seul pic bien défini et une seule bande à l'emplacement prévu sur le gel 16, up class = "xref"> 17 (Figure 2). Si la courbe de fusion a plusieurs pics ou une «épaule», ou si le gel a de multiples bandes ou un frottis, les amorces amplifient une séquence hors cible. Refonte des amorces qui ont été observées pour amplifier des séquences hors-cible et répétez les étapes de validation précédentes si nécessaire. Une bonne paire d'amorces aura également un rendement de 90-110%, avec R 2 ≥0.985 pour la courbe standard 16; refonte des amorces pour lesquels l'efficacité ou R 2 sont en dehors de ces plages.
- Préparer des normes de amplicons d'ADN purifiés.
- Pour chaque paire d'amorces validées:
- Amplifier la région d'intérêt à partir d'ADNc en utilisant un ADN polymérase haute fidélité selon les recommandations du fabricant de la polymérase pour amorces et l'ADN matrice concentrations, les conditions de réaction et les conditions de cycle de PCR, ou en utilisant un protocole de PCR standard"xref"> 17.
- Exécuter l'ADNc amplifié sur un gel d'agarose à 2% p / v et de visualiser la bande avec un colorant intercalant de l' ADN 17. Exciser la bande représentant l'amplicon en utilisant une lame de rasoir propre et placez le morceau de gel dans un tube à centrifuger.
- Purifier l'amplicon à partir du gel en utilisant un procédé standard d'extraction de gel, par exemple une extraction à base d' agarase-17 ou un kit d'extraction de gel de spin colonne selon les instructions du fabricant.
- Mesurer la concentration de l'amplicon en utilisant un kit de quantification d'ADNdb basé sur la fluorescence selon les instructions du fabricant. Diviser la concentration en ADNdb mesurée par le poids moléculaire connu de l'amplicon basé sur la séquence de l'amplicon pour obtenir le nombre de molécules d'amplicon par ml.
- Dans une faible liaison à microtube de 2,0 ml, ajouter 2 ul de l'amplicon purifié jusqu'à un volume de tampon de suspension d'ADN de telle sorte que la concentration finale en ampèreslicon est de 5 x 10 8 molécules / ul.
- Lorsque tous les amplicons ont été purifiés, mélanger 18 pi de chaque amplicon purifié dans une faible liaison microtube 2,0 ml. Ajouter un tampon ADN de suspension pour obtenir un volume total de 1 800 pi; Ainsi, la concentration de chaque amplicon est de 5 x 10 6 molécules / pl.
NOTE: Ce mélange, qui contient amplicons d'ADNc de tous les gènes d'intérêt en quantités équimolaires connues, servira de standard dans une seule cellule qPCR. - Vortex cette norme «5E6» à fond, rotation pendant 10 secondes à 2000 xg, et se divisent en 20 aliquotes dans des tubes à faible liaison PCR. Conserver les aliquotes à -80 ° C.
- Pour chaque paire d'amorces validées:
- Préparer l'amplification cible spécifique (STA) mélange d'amorces (10x).
- Dans un tube de 15 ml conique, mélanger 25 pi de chaque 100 uM Stock primaire (jusqu'à 192). Ajouter un tampon de suspension d'ADN pour obtenir un volume total de 5000 pi.
Noter lala concentration de chaque amorce dans ce mélange est de 500 nm. Chaque plaque de cellules triées utilisera 105,6 pi de cette amorce mélange pour amorcer la transcription inverse et l'amplification de cible spécifique. Le volume de mélange d'amorces donné ici est assez pour faire 45 plaques pour trier. Il est conseillé de faire un volume suffisant de mélange d'amorces de sorte qu'il ne doit être fait une fois; échelle les volumes nécessaires. - Mélanger soigneusement par tourbillonnement, diviser en 110 aliquotes et stocker les aliquots à -20 ° C.
- Dans un tube de 15 ml conique, mélanger 25 pi de chaque 100 uM Stock primaire (jusqu'à 192). Ajouter un tampon de suspension d'ADN pour obtenir un volume total de 5000 pi.
- Préparer des mélanges d'essai, un pour chaque paire d' amorces, pour une utilisation dans qPCR à base de puce microfluidique.
- Dans chaque puits d'une 96 puits plaque PCR, mélanger 3,0 pi de 100 pM amorce directe pour un gène donné, 3,0 pi de 100 pM amorce inverse correspondante, 24,0 pi de tampon d'ADN de suspension, et 30,0 ul de 2x réactif test de chargement .
- Vortex cette plaque, rotation pendant 10 s à 500 g, et conserver à -20 ° C.
- Créer deux programmes de cycles thermiques: RTSTA (transcription inverse et l' amplification de cible spécifique; tableau 1) et Exoi (traitement exonucléase I; tableau 2).
2. Traitement
- Plaquer les cellules de mammifère sur une boîte de culture de tissu de telle sorte qu'elles vont croître jusqu'à la confluence souhaitée au moment du traitement, en fonction des conditions de l'étude expérimentale. Pour l'exemple présenté dans cette étude, la plaque 4 x 10 5 cellules MCF-7 p53-Vénus 18 par boîte de 6 cm dans du RPMI avec 10% de sérum de veau fœtal (FBS), 100 U / ml de pénicilline, 100 mg / ml de streptomycine, et 250 ng / ml d' amphotéricine B. on incube les cellules pendant deux jours à 37 ° C avec 5% de CO2 jusqu'à ce qu'elles atteignent 50% de confluence.
- Traiter les cellules pour établir l'état de l'intérêt sur la base de l'étude expérimentale. Pour l'exemple présenté dans cette étude, incuber les cellules p53-Vénus MCF-7 avec 400 ng / ml neocarzinostatine pendant 3 h, 8,5 h, 14 h ou 24 h.
3. Lysis Buffer Préparation cellulaire tri
NOTE: Faire une seule plaque de tampon de lyse dure environ 1 heure. Il est conseillé de faire et de plaques de tri multiple, comme le tri cellulaire peut être inefficace et donner de nombreux puits sans cellule détectable.
- Nettoyez le banc, pipettes, support de tubes, supports de plaques PCR froid, et des gants avec une solution d'ADN décontaminant en préparation de la PCR.
- Diluer l'inhibiteur de RNAse à 2,64 U / ul en ajoutant un inhibiteur de stock RNAse à nucléase sans eau dans un tube de 1,5 ml.
REMARQUE: Ceci permet un inhibiteur de RNAse à être ajouté au tampon de lyse sans pipetage de volumes de sous-yl. - Faire un tampon de lyse pour les cellules en combinant les composants énumérés dans le tableau 3.
- Transférer 92,4 ul de tampon de lyse pour les cellules dans un tube séparé; ce sera le tampon de lyse pour les standards d'amplicon.
- Utilisation de low contraignant embouts de pipette et microtubes, préparer 200 pi et 500 pi d'ADN de E. coli dilué à 3,1 pg / pl et 6,2 pg / pl, respectivement, avec un tampon d' ADN de suspension.
- Ajouter 184,8 ul de / ul d' ADN de E. coli 3,1 pg du tampon de lyse pour les cellules.
NOTE: L'ADN porteur de E. coli sert à élargir le champ des possibilités de liaison d'amorces spécifiques et réduire ainsi la probabilité que la dimérisation primaire conduira à un produit de PCR. - Préparer des normes.
- Etiqueter six tubes de faible liaison centrifuger comme 5E5, 5E4, ... 5E0.
- Ajouter 90 ul de tampon de suspension de l'ADN dans le tube de "5E5".
- Ajouter 90 ul de / ul d' ADN de E. coli 6,2 pg de chacun des cinq autres tubes. Conservez tous les tubes sur la glace.
REMARQUE: L'intégration de l'ADN vecteur dans les normes rend la masse totale de l'ADN dans les puits de niveau comparable à celui dans les puits 1-cellule; ce qui réduit la probability que la concentration des normes sera affectée par liaison à l'ADN à des tubes ou des embouts de pipette. - Prélever une partie aliquote de la norme "de 5E6" (5 x 10 6 molécules / ul de chaque amplicon, préparé à l' étape 1.4) de stockage. brièvement Vortex et centrifuger brièvement (5 sec à 500 g) pour veiller à ce que le liquide est au fond du tube.
- En utilisant une pointe à faible liaison pipette, ajouter 10 pi de standard 5E6 au tube "de 5E5". Vortex brièvement, rotation (5 sec à 500 g), et de mettre le tube de retour sur la glace.
- Pipette 10 ul du "5E5" tube dans le "5E4" tube. Vortex brièvement, rotation (5 sec à 500 g), et remis sur la glace.
- Répétez ces dilutions en série, avec chaque tube de réception 10 ul du tube précédent, jusqu'à ce que tous les tubes ont une dilution de la norme.
- Préparer une rangée de 12 tubes PCR avec bouchons. Distribuer 67 pi de tampon de lyse "cellule" dans chaque tube. Boucher les tubes et centrifuger brièvement (5sec à 500 g) si nécessaire.
- En utilisant une pipette à 12 canaux, distribuer 9 pi de tampon de lyse "cellule" de la rangée de tubes PCR dans chaque puits des 7 premières lignes d'une plaque PCR (figure 3).
- Distribuer 7 pi de tampon de lyse "standard" à chaque puits de la dernière rangée de la plaque (Figure 3).
- En utilisant des pointes à faible liaison pipette, ajouter 2 pi de standard pour chaque puits de la rangée du bas selon la carte de la plaque (figure 3).
NOTE: 2 pi de 5 x 10 0 molécules / montants ul standard à 10 molécules, etc. - Ajouter 2 pl de / ul d' ADN de E. coli 3,1 pg dans les puits de 10 cellules et 100 cellules; ajouter 2 pi de / ul d' ADN de E. coli 6,2 pg dans les puits sans cellules.
Remarque: chaque cellule 1, la cellule 10 et 100 cellules a ainsi 6,2 pg d'ADN de E. coli, une approximation de la masse d'ADN génomique dans une cellule humaine. Chaque puits standard et sans cellule a 12,4 pg of ADN de E. coli, la valeur d'environ deux cellules humaines. - Sceller la plaque à l'aide d'un joint thermique stérile, rotation pendant 5 s à 500 g, et conserver à 4 ° C jusqu'à ce que les cellules sont prêtes pour le tri.
4. trieur de cellules Setup
- Programmer le trieur de cellules activées par fluorescence pour trier dans une plaque à 96 puits en fonction de la carte de la plaque (figure 3); pas de cellules doivent être triés dans les puits H1-H8 ou H11-H12.
- Vérifiez que le trieur de cellules est bien destiné à trier dans une plaque PCR.
- Réglez l'angle du flux de tri au minimum possible; ce paramètre augmente les chances que les cellules seront triées dans le fond des puits plutôt que de frapper les côtés.
- Pour viser la machine, sceller une plaque PCR vide et utiliser le courant d'essai à des gouttelettes "tri" de PBS sur le joint de la plaque vide, selon les instructions de la machine 19; les gouttelettes devraient atterrir sur the surface du joint d'étanchéité à une position au-dessus du centre du puits. Pour les meilleurs résultats, vérifiez l'objectif à travers la longueur et la largeur de la plaque.
- Si les gouttelettes ne débarquent pas dans la bonne position, les essuyer la surface du joint, recalibrer le trieur de cellules selon les instructions du fabricant de la machine, et répéter jusqu'à ce que les gouttelettes dans le courant de tri sont déposés correctement.
5. Cellule Récolte et tri
- Trypsiniser les cellules en fonction de la lignée cellulaire. Par exemple, pour les cellules MCF-7, Aspirer le milieu des cellules, laver les cellules avec 1 ml de trypsine à 0,05%, et on incube les cellules avec 2 ml de trypsine à 0,05% pendant 5 min à 37 ° C et 5% de CO 2 .
- Ajouter 8 ml de milieu de culture cellulaire pour les cellules trypsinisées et transférer tous les 10 ml de suspension cellulaire dans un tube de 15 ml. Centrifugeuse pendant 4 min à 400 x g.
- Aspirer le surnageant et remettre en suspension le culot cellulaire dans du PBS + 2% de FBS.
NOTE: Un volume de resuspension plus petit concentre les cellules et facilite le tri cellulaire; un volume de remise en suspension 500 ul fonctionne bien pour une boîte de 6 cm de cellules à 50% de confluence. - Transférer la suspension cellulaire à 5 ml d'une cytométrie de flux tube de pipetage à travers un filtre de 40 um pour éliminer les amas de cellules.
- Centrifuger la plaque de tampon de lyse à 400 xg pendant 30 secondes à 4 ° C avant le tri des cellules pour faire en sorte que le liquide se trouve au fond des puits.
- Insérer le tube avec la suspension cellulaire dans le trieur de cellules. Ouvrez le joint d' étanchéité sur la plaque de tampon de lyse et de trier soigneusement les cellules d'intérêt dans la plaque selon la carte de la plaque et en suivant les instructions du fabricant de trieur de cellules 19. Pour veiller à ce que des cellules individuelles sont triées à la pureté maximale, faire fonctionner la machine en mode "cellule unique" et utilisez débit standard cytométrie gating basé sur l' avant et le côté scatter 19.
NOTE: Dans leexemple expérience, porte les cellules sur la base p53-Vénus fluorescence pour isoler les plus brillants de 15% des cellules individuelles 11. - Sceller la plaque avec un nouveau joint d'étanchéité thermique stérile et centrifuger à 400 g pendant 1 min à 4 ° C. Placer la plaque dans le cycleur thermique et exécuter le programme de RTSTA (voir étape 1.7).
NOTE: Ceci effectue une transcription inverse sur l'ARNm des cellules triées, puis l'amplifie l'ADNc des transcrits d'intérêt.
6. Traitement exonucléase
- Mélanger dilué exonucléase I en combinant les composants énumérés dans le tableau 4. Préparer une rangée de 12 tubes PCR avec bouchons. Distribuer 30,5 pi de exonucléase I dilué dans chaque tube. Boucher les tubes et centrifuger brièvement (5 sec à 500 g) pour veiller à ce que le liquide est au fond du tube.
- Lorsque le RTSTA est terminé, tourner la plaque d'échantillon pendant 5 s à 500 x g. En utilisant une pipette à 12 canaux, distribuer 3,6 pi d'exonucléase diluéI dans chaque puits de la plaque.
- Sceller la plaque avec un joint thermique stérile et brièvement de spin (5 sec à 500 g). Placer la plaque dans le cycleur thermique et exécuter le programme de Exoi (voir étape 1.7).
REMARQUE: Cette étape se dégrade tous les amorces restantes après amplification cible spécifique afin qu'ils ne seront pas interférer avec la PCR future.
Dilution 7. Sample
- Lorsque le traitement par une exonucléase est terminée, ajouter 32,4 pi de tampon TE à chaque puits de la plaque d'échantillon; ceci est un point d'arrêt en option, et les échantillons peuvent être conservés à -20 ° C pendant plusieurs jours.
8. Trier Contrôle de la qualité utilisant qPCR
NOTE: Parce que le tri cellulaire est pas parfaitement efficace, cette étape est nécessaire pour identifier les puits de la plaque triée ont effectivement reçu une cellule. Ces échantillons peuvent ensuite être utilisés pour une analyse ultérieure.
- Mesurer l'expression du gène de ménage dans chaque échantillon par qPCR.
- Préparer 900 pi de liaison colorant qPCR mélange maître ADN pour 96 réactions selon le protocole en utilisant les amorces du fabricant de la polymérase pour l'un des gènes de ménage sélectionnés à l'étape 1.1; la concentration exacte de tampon de réaction, la polymérase, des amorces, etc. , pour cette réaction dépend de la polymérase spécifique utilisé.
- Distribuer 9 pi de mélange maître à chaque puits d'une plaque à 96 puits PCR. Ajouter 1 pi d'échantillon de la plaque échantillon au puits correspondant de la plaque de PCR.
- Exécutez la plaque sur une machine PCR en temps réel en utilisant un protocole de cyclage thermique suggéré par le fabricant principal de mixage , ou à la suite d' un protocole standard 20.
- Analyser les données qPCR 20; un exemple de données de tri qPCR des étapes de contrôle de qualité énumérés ci - dessous est représentée sur la figure 4.
- Vérifiez que les mesures à partir des puits sans cellules montrent un (arrière-plan) faible niveaude l'expression génique ou pas d'expression du tout.
- Vérifiez que les mesures de puits 1-cellules se divisent clairement en deux groupes: les échantillons positifs avec l'expression génique élevé et des échantillons négatifs avec des niveaux de fond d'expression ou pas d'expression à tous, semblables aux puits sans cellules.
NOTE: Les échantillons présentant une expression élevée du gène représentent des cellules triées réelles; ceux avec l'expression de fond doivent être exclus de l'analyse. - Vérifier que les mesures à partir des puits de cellules 10 et 100 de cellules reflètent l'expression génique plus élevée que dans les puits 1 de cellules positives, ce qui permet à la possibilité que le tri inefficace pourrait entraîner ces puits aient moins de cellules que souhaité.
- Vérifiez que les mesures des puits standards sont répartis uniformément dans l' espace logarithmique ( à savoir que les valeurs t C sont répartis uniformément).
- Après avoir identifié tous les échantillons 1-cellules positives, faire un "échantillon Mixes" carte de la plaque à la carte positifs 1-ceéchantillons ll sur une nouvelle plaque de 96 puits. Inclure huit puits pour les normes sur cette carte de la plaque. Voir la figure 5 pour un exemple.
9. Gene Expression mesure Utilisation microfluidique qPCR
NOTE: Pour chaque étape de cette section, pipette seulement au premier arrêt pour minimiser la formation de bulles dans les réactifs.
- Ouvrez une rangée de 8 tubes PCR. Si plusieurs plaques d'échantillons, ouvrir une deuxième rangée de 8 tubes PCR et étiqueter les tubes 1-8. Cette ligne sera de mise en commun des normes à partir de plusieurs plaques.
- Dans un tube de 1,5 ml, mélanger 360 pi de liaison colorant qPCR mélange maître ADN avec 36 pi de l'ADN échantillon de colorant réactif de chargement de liaison. brièvement Vortex et de spin pendant 10 secondes à 2000 x g. Divisez ce mélange (396 pi) dans la ligne de non marqué 8 tubes PCR, placer 48 ul dans chaque tube.
- En utilisant une pipette à 8 canaux, distribuer 3,3 pi du mélange de réactif de chargement mélange maître / échantillon dans chaque puitsd'une nouvelle plaque PCR 96 puits. Etiqueter cette plaque, "Sample Mixes."
- Pour chaque plaque PCR d'échantillons:
- Vortex la plaque pendant 10 secondes et de spin pendant 10 s à 500 x g. Retirer le couvercle et transférer 2,7 pi de chaque échantillon de 1 cellule positive à son puits correspondant sur l'échantillon Mixes plaque pour la cellule 1 puits indiqués sur la carte de la plaque.
- S'il n'y a qu'une plaque d'échantillons, transférer 2,7 pi de chaque norme amplifié à son puits correspondant sur l'échantillon Combine plaque selon la carte de la plaque.
- Si plusieurs plaques d'échantillons, transférer 2,7 pi de chaque norme amplifié à sa position correspondante dans la ligne marquée des tubes PCR. Ajouter les normes de la plaque actuelle des échantillons à des normes déjà là-bas. Sceller la plaque d'échantillons lorsqu'elle est effectuée en utilisant un film d'étanchéité.
- S'il y a plusieurs plaques d'échantillons:
- Vortex la rangée de tubes PCR contenant des normes pour10 sec et essorage pendant 10 secondes à 2000 x g. Transférer 2,7 pi de chaque tube de normes mixtes dans le puits correspondant sur l'échantillon Combine plaque selon la carte de la plaque.
- Charger les puits NTC avec 2,7 pi d'eau sans nucléase l'endroit indiqué sur la carte de la plaque. Sceller l'échantillon Mixes plaque et conserver à 4 ° C jusqu'à ce que nécessaire.
- Pelez l'autocollant au fond d'une nouvelle puce microfluidique qPCR. En utilisant une seringue de fluide de la ligne de commande avec le bouchon encore, pousser vers le bas de chaque ressort de l'accumulateur pour le desserrer, et injecter chaque accumulateur avec 150-200 pi de fluide de la ligne de commande.
- Insérez la puce dans la machine de chargement et exécuter le script "Prime". Cela prend ~ 20 min.
- Lorsque l'amorçage est fait, vortex et centrifuger les plaques de mélanges d'essai (préparé à l'étape 1.6) et des mélanges d'échantillons.
- À l'aide d'une pipette à 8 canaux, transférer 4,5 ul de chaque mélange d'essai dans la partie gauche de la puce microfluidique qPCR selon le schéma g> Figure 6. Pipette très soigneusement pour éviter de créer des bulles d'air au fond des puits. Une fois terminé, sceller la plaque de mélanges d'essai et conserver à -20 ° C pour une utilisation future.
- Transférer 4,5 pi de chaque échantillon mélange sur le côté droit de la puce microfluidique qPCR selon la figure 6. Insérez la puce dans la machine de chargement et exécutez le script "Charger Mix". Cela prend ~ 90 min.
- Mettez la machine microfluidique PCR en temps réel, démarrez le logiciel de collecte de données, et allumer la lampe pour la réchauffer. Cela prend ~ 20 min.
- Lorsque le script Load Mix est fait, vérifier qu'il n'y a pas de lignes à travers la puce-cela indiquerait un problème de chargement. Retirez soigneusement les particules de poussière de la puce en utilisant du ruban Scotch ou d'une bande de laboratoire. Insérez la puce dans la machine microfluidique PCR en temps réel et exécutez le script de collecte des données. Utilisez le logiciel d'analyse pour inspecter les résultats et exporter les données pour une analyse ultérieure.
- Retirez les mesures pour lesquelles les courbes d'amplification qPCR montrent une mauvaise qualité ( à savoir, pour lesquels les courbes ne ressemblent pas à la courbe d'amplification sigmoïde standard) 20.
- Retirer les échantillons avec plus de 30 mauvaise qualité ou zéro mesures ou pour lesquelles l' expression du gène de ménage (voir l' étape 1.1) est beaucoup plus faible que la majorité des autres échantillons (figure 7).
- Pour chaque gène, une estimation de l'emplacement du pic à l'état fondu correctes, la médiane des pics des emplacements à l'état fondu à partir de chaque échantillon. Si une mesure a un pic de fusion située à plus de 1 ° C de la médiane, retirer cette mesure de l' examen 20.
- Pour chaque gène, de créer une courbe d' étalonnage pour estimer le nombre d'ARNm dans chaque échantillon à l' aide d' un tableur ou un langage de script 20.
NOTE: Les normes de cette expérience sont constitués de 10, 100, 1000 ou 10.000 moléculesde dsDNA correspondant à chaque transcrit d'intérêt. Si la transcription inverse sont parfaitement efficaces, les normes correspondraient à 20, 200, 2000, et 20.000 molécules d'ARNm, étant donné que l'ARNm et l'ADNc sont simple brin. Dans la pratique, ce n'est pas le cas, alors nous exprimons des mesures comme une estimation en unités de molécules × E RT, où E RT est l'efficacité de la transcription inverse.- Pour chaque puits standard, identifier m, le nombre de molécules d'ADNss (deux fois le nombre de molécules d'ADNdb) avant l' amplification (par exemple, 20 molécules, 200 molécules, etc.).
- Pour chaque puits standard, identifier la valeur t de C de qPCR 20.
- Les valeurs pour m et C t, effectuer une régression linéaire en utilisant l'équation suivante pour générer une courbe standard avec des paramètres a et b pour chaque gène, et trouver la valeur de R 2 en tant quen indicateur de la qualité de l'ajustement:
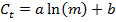
- A partir de la courbe étalon, calculer l'efficacité de la PCR pour chaque gène E:
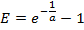
NOTE: Une valeur de E bien supérieure à 1,0 (par exemple, E> 1.1) ne sont pas physiquement réalistes; une valeur R 2 de la régression linéaire très inférieure à 1,0 (par exemple, R 2 <0,985) signifie que les normes ne fonctionnent pas de façon fiable. Ces problèmes indiquent généralement que la norme amplicon plus bas tombe en dessous d'un seuil de quantification fiable, et donc le plus bas niveau devrait être exclue de la régression linéaire. Si une régression linéaire adéquate ne peut être faite en utilisant au moins trois normes spécifiques, éliminer les mesures d'expression du gène. - Si la régression linéaire est suffisante, utilisez le paramètres d' ajustement résultant a et b </ em> pour chaque gène d'estimer le nombre d'ARNm molécules m est (en unités de molécules × E RT) pour le gène correspondant dans chaque échantillon de cellules isolées à partir de la valeur mesurée t C:
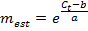
- Désigner toute mesure m is <1 m is = 0. En outre, désigner toute mesure sans amplification dans qPCR et donc pas de t valeur de C comme m is = 0.
NOTE: Les mesures avec un m is inférieur au plus bas ou supérieur à la norme la plus élevée utilisée dans la régression se trouvent par extrapolation à partir de la courbe standard. Ces estimations ne sont pas nécessairement valides, mais doivent être considérées avec prudence.
- Visualisez les données d'expression génique unicellulaires.
- Faire une parcelle de beeswarm (Dorn, J. et Saint, T., 2012, http://www.mathworks.com/matlabcentral/fileexchange / 37105) dans laquelle chaque point représente l'expression du gène dans une cellule particulière; un exemple est représenté sur la figure 8.
- Faire un terrain de violon (Dorn, J., 2012, http://www.mathworks.com/matlabcentral/fileexchange/23661) dans laquelle la largeur de la "violon" représente la fréquence des mesures d'expression génique autour d'un niveau particulier dans le population de cellules analysées; un exemple est représenté sur la figure 8.
NOTE: Des analyses plus sophistiquées peuvent sonder les relations dans l' expression entre des paires de gènes 21, 22 et peuvent déduire les réseaux d'expression des gènes.
Résultats
Un aperçu général du protocole est représentée sur la figure 1, comprenant des étapes pour le traitement des cellules, l'isolement des cellules isolées par FACS, la génération et la pré-amplification des banques d'ADNc à partir de lysats de cellule unique, la confirmation des banques d'ADNc monocellulaires dans les puits triées et la mesure de l'expression des gènes par PCR quantitative.
Discussion
Nous avons présenté un procédé pour isoler des cellules de mammifères individuels à partir d'une population de cellules adhérentes cultivées en culture et pour le dosage de l'expression d'environ 96 gènes dans chaque cellule. Une bonne préparation préalable est essentielle pour que cette méthode fonctionne bien. En particulier, la conception et les tests paires d'amorces spécifiques pour les transcriptions d'intérêt (étapes 1,2-1,3) prennent du temps, mais d'importantes étapes, c...
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
Nous tenons à remercier V. Kapoor dans le CCR ETIB cytométrie de flux de base pour son aide dans l'exécution du tri cellulaire au cours du développement de ce protocole. Nous remercions également M. l'Unité CCR LP Molecular Diagnostics Raffeld et et J. Zhu et le NHLBI séquençage de l'ADN et la génomique de base pour leur aide dans l'exécution de la qPCR lors de l'élaboration de ce protocole. Cette recherche a été soutenue par le programme intra-muros des NIH.
matériels
| Name | Company | Catalog Number | Comments |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| High Capacity cDNA Reverse Transcription Kit with RNase Inhibitor | ThermoFisher | 4374966 | |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530S | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| Quant-iT High-Sensitivity dsDNA Assay Kit | ThermoFisher | Q33120 | |
| 2.0 ml low adhesion microcentrifuge tubes | USA Scientific | 1420-2600 | |
| DNA Suspension Buffer | Teknova | T0221 | |
| Axygen 0.2 ml Maxymum Recovery Thin Wall PCR Tubes | Corning | PCR-02-L-C | |
| GE 96.96 Dynamic Array DNA Binding Dye Sample & Assay Loading Reagent Kit | Fluidigm | 100-3415 | |
| HyClone RPMI 1640 media | GE Healthcare Life Sciences | SH30027.01 | |
| Fetal Bovine Serum, Certified (US) | ThermoFisher | 16000-044 | |
| Antibiotic-Antimycotic Solution | Corning | 30-004-CI | |
| Neocarzinostatin | Sigma | N9162 | |
| ELIMINase | Decon Labs | 1101 | |
| SUPERase-In | ThermoFisher | AM2696 | |
| CellsDirect One-Step qRT-PCR Kit | ThermoFisher | 11753500 | |
| E. coli DNA | Affymetrix | 14380 10 MG | |
| ThermalSeal Sealing Film, Sterile | Excel Scientific | STR-THER-PLT | |
| BD FACSAria IIu | BD Biosciences | ||
| HyClone Trypsin 0.05% | GE Healthcare Life Sciences | SH30236.01 | |
| PBS, 1x | Corning | 21-040-CV | |
| Falcon 40 µm Cell Strainer | Corning | 352340 | |
| Exonuclease I | New England BioLabs | M0293S | |
| SsoFast EvaGreen Supermix with Low ROX | Bio-Rad | 172-5210 | |
| 96.96 Dynamic Array IFC for Gene Expression (microfluidic qPCR chip) | Fluidigm | BMK-M-96.96 | |
| IFC Controller HX (loading machine) | Fluidigm | ||
| BioMark or BioMark HD (microfluidic qPCR machine) | Fluidigm | ||
| Real-Time PCR Analysis software | Fluidigm | ||
| MATLAB software | MathWorks |
Références
- Feinerman, O., Veiga, J., Dorfman, J. R., Germain, R. N., Altan-Bonnet, G. Variability and Robustness in T Cell Activation from Regulated Heterogeneity in Protein Levels. Science. 321 (5892), 1081-1084 (2008).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- Geva-Zatorsky, N., Rosenfeld, N., et al. Oscillations and variability in the p53 system. Mol. Syst. Biol. 2 (1), (2006).
- Colman-Lerner, A., Gordon, A., et al. Regulated cell-to-cell variation in a cell-fate decision system. Nature. 437 (7059), 699-706 (2005).
- Chong, S., Chen, C., Ge, H., Xie, X. S. Mechanism of Transcriptional Bursting in Bacteria. Cell. 158 (2), 314-326 (2014).
- Raj, A., Peskin, C. S., Tranchina, D., Vargas, D. Y., Tyagi, S. Stochastic mRNA Synthesis in Mammalian Cells. PLoS Biol. 4 (10), e309+ (2006).
- Senecal, A., Munsky, B., et al. Transcription Factors Modulate c-Fos Transcriptional Bursts. Cell Rep. 8 (1), 75-83 (2014).
- Dey, S. S., Foley, J. E., Limsirichai, P., Schaffer, D. V., Arkin, A. P. Orthogonal control of expression mean and variance by epigenetic features at different genomic loci. Mol. Syst. Biol. 11 (5), 806+ (2015).
- Dar, R. D., Razooky, B. S., et al. Transcriptional burst frequency and burst size are equally modulated across the human genome. Proc. Natl. Acad. Sci. U.S.A. 109 (43), 17454-17459 (2012).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- Porter, J. R., Fisher, B. E., Batchelor, E. p53 Pulses Diversify Target Gene Expression Dynamics in an mRNA Half-Life-Dependent Manner and Delineate Co-regulated Target Gene Subnetworks. Cell Syst. 2 (4), 272-282 (2016).
- Lahav, G., Rosenfeld, N., et al. Dynamics of the p53-Mdm2 feedback loop in individual cells. Nat. Genet. 36 (2), 147-150 (2004).
- Levine, A. J., Oren, M. The first 30 years of p53: growing ever more complex. Nat. Rev. Cancer. 9 (10), 749-758 (2009).
- Riley, T., Sontag, E., Chen, P., Levine, A. Transcriptional control of human p53-regulated genes. Nat. Rev. Mol. Cell Biol. 9 (5), 402-412 (2008).
- Ye, J., Coulouris, G., Zaretskaya, I., Cutcutache, I., Rozen, S., Madden, T. L. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinf. 13 (1), 134 (2012).
- . . PCR Technologies: A Technical Guide. , (2014).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Batchelor, E., Mock, C. S., Bhan, I., Loewer, A., Lahav, G. Recurrent initiation: a mechanism for triggering p53 pulses in response to DNA damage. Mol. Cell. 30 (3), 277-289 (2008).
- . . Flow Cytometry: Principles and Applications. , (2007).
- . . Real-time PCR. , (2006).
- Song, L., Langfelder, P., Horvath, S. Comparison of co-expression measures: mutual information, correlation, and model based indices. BMC Bioinf. 13 (1), 328 (2012).
- Margolin, A. A., Nemenman, I., et al. ARACNE: An Algorithm for the Reconstruction of Gene Regulatory Networks in a Mammalian Cellular Context. BMC Bioinf. 7 (Suppl 1), (2006).
- Haff, L. A. Improved quantitative PCR using nested primers. PCR Methods Appl. 3 (6), 332-337 (1994).
- Hashimshony, T., Senderovich, N., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biol. 17, 77 (2016).
- Ronander, E., Bengtsson, D. C., Joergensen, L., Jensen, A. T. R., Arnot, D. E. Analysis of Single-cell Gene Transcription by RNA Fluorescent In Situ Hybridization (FISH). J. Vis. Exp. (68), e4073 (2012).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat. Methods. 5 (10), 877-879 (2008).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nat. Methods. 9 (7), 743-748 (2012).
- Battich, N., Stoeger, T., Pelkmans, L. Image-based transcriptomics in thousands of single human cells at single-molecule resolution. Nat. Methods. 10 (11), 1127-1133 (2013).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon