Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Einzellige Genexpressionsprofile mittels FACS und qPCR mit internen Standards
In diesem Artikel
Zusammenfassung
We describe a method to sort single mammalian cells and to quantify the expression of up to 96 target genes of interest in each cell. This method includes the use of internal qPCR standards to enable the estimation of absolute transcript counts.
Zusammenfassung
Gene expression measurements from bulk populations of cells can obscure the considerable transcriptomic variation of individual cells within those populations. Single-cell gene expression measurements can help assess the role of noise in gene expression, identify correlations in the expression of pairs of genes, and reveal subpopulations of cells that respond differently to a stimulus. Here, we describe a procedure to measure the expression of up to 96 genes in single mammalian cells isolated from a population growing in tissue culture. Cells are sorted into lysis buffer by fluorescence-activated cell sorting (FACS), and the mRNA species of interest are reverse-transcribed and amplified. Gene expression is then measured using a microfluidic real-time PCR machine, which performs up to 96 qPCR assays on up to 96 samples at a time. We also describe the generation and use of PCR amplicon standards to enable the estimation of the absolute number of each transcript. Compared with other methods of measuring gene expression in single cells, this approach allows for the quantification of more distinct transcripts than RNA FISH at a lower cost than RNA-Seq.
Einleitung
Einzelne Zellen in einer Population kann zu einem einheitlichen physiologischen Reiz 1, 2, 3, 4 sehr unterschiedliche Reaktionen zeigen. Die genetische Veränderung der Zellen in einer Population eines Mechanismus für diese Vielzahl von Antworten, aber es gibt auch einige nicht-genetischen Faktoren, die die Variabilität von Antworten erhöhen können, selbst in einer klonalen Population von Zellen. Zum Beispiel können die Ebenen der einzelnen Proteine und andere wichtige Signalmoleküle variieren auf einer Zelle-für-Zelle-Basis, was zu geben Variation in nachgeschalteten Genexpressionsprofilen. Zusätzlich können kurzzeitige Bursts von Transkripten 5 treten Genaktivierung in 6, der 7 auf eine relativ kleine Anzahl von Transkripten pro Burst begrenzt sein kann, 8, 9. Eine solchestochasticity in Genaktivierung kann stark von Schwankungen der biologischen Reaktionen beitragen und einen selektiven Vorteil in Mikroorganismen 10 und in Säugetierzellen 1, 2 reagiert auf einen physiologischen Reiz bieten kann. Aufgrund beiden genetischen und nicht-genetischen Variationsquellen kann das Genexpressionsprofil eines beliebigen gegebenen Zelle in Reaktion auf einen Reiz deutlich vom Durchschnitt Genexpressionsprofil unterscheiden sich von der Messung der Massenantwort erhalten. Das Ausmaß, in dem einzelne Zellen Variabilität in Reaktion auf einen Reiz zeigen, erfordert Techniken zur Isolierung von einzelnen Zellen, die Messung der Expressionsspiegel für Transkripte von Interesse, und die Computeranalyse der resultierenden Expressionsdaten.
Es gibt mehrere Ansätze zur Untersuchung der Genexpression in einzelnen Zellen, eine breite Palette von Kosten abdeckt, die Anzahl der Transkripte sondiert, undGenauigkeit der Quantifizierung. So bietet zum Beispiel Single-Cell-RNA-Seq eine große Tiefe transcript Abdeckung und der Möglichkeit, Tausende von verschiedenen Transkripte für den am höchsten exprimierten Gene in einzelnen Zellen zu quantifizieren; jedoch verbunden sind die Kosten, die mit solchen Sequenzierungstiefe kann unerschwinglich sein, auch wenn die Kosten weiter sinken. Im Gegensatz dazu bietet Einzelmolekül RNA Fluoreszenz in situ Hybridisierung (FISH smRNA) genaue Quantifizierung von Transkripten für sogar Low-Expression von Genen zu vernünftigen Kosten pro Gen von Interesse; jedoch nur eine geringe Anzahl von Zielgenen können durch diese Vorgehensweise in einer gegebenen Zelle untersucht werden. Quantitative PCR-basierten Assays, in diesem Protokoll beschrieben, einen Mittelweg zwischen diesen Techniken bereitzustellen. Diese Assays verwenden eine mikrofluidische Echtzeit-PCR-Maschine zu einem Zeitpunkt, zu 96 Transkripte von Interesse zu quantifizieren bis in bis zu 96 Zellen. Während jedes der vorgenannten Verfahren erforderliche Hardwarekosten hat, sind die Kosten eines einzelnen qPCR Assay relativniedrig. Dieses Protokoll wird von einem durch den Hersteller eines mikrofluidischen Echtzeit-PCR-Maschine (Protokoll ADP 41, Fluidigm) vorgeschlagen angepasst. Um die Abschätzung der absoluten Nummer jedes Transkript in einem PCR-basierten Ansatz zu ermöglichen, haben wir das Protokoll erweitert, um die Verwendung von internen Kontrollen vorbereiteter Zielgen Amplikons machen, die über mehrere Experimente verwendet werden kann.
Als ein Beispiel für diese Technik ist 11 beschrieben die Quantifizierung der Expression von Genen durch den Tumorsuppressor p53 in menschlichen MCF-7 - Brustkarzinomzellen reguliert. Die Zellen werden mit einem chemischen Mittel in Frage gestellt, die DNA-Doppelstrangbrüche induziert. Frühere Studien haben gezeigt , dass die p53 - Antwort auf DNA - Doppelstrangbrüchen eine große Heterogenität in einzelnen Zellen aufweist, sowohl in Bezug auf p53 Ebenen 12 und bei der Aktivierung verschiedener Zielgene 11. Darüber hinaus reguliert p53 die Expression von mehr als 100gut charakterisierten Zielgene in zahlreichen Downstream Wegen beteiligt, einschließlich Zellzyklusarrest, Apoptose und Seneszenz 13, 14. Da die p53-vermittelte Reaktion in jeder Zelle sowohl komplex und variabel ist, kann die Analyse des Systems profitiert von einem Ansatz, bei dem nahezu 100 Zielgene gleichzeitig in einzelnen Zellen werden untersucht, wie das unten beschrieben wird. Mit leichten Modifikationen (wie alternative Verfahren zur Einzelzellisolierung und Lyse), kann das Protokoll leicht ein breites Spektrum von Säugerzelltypen, Protokolle und zelluläre Antworten zu untersuchen angepasst werden.
Mit der richtigen rechtzeitige Vorbereitung kann eine Runde der Zellsortierung und Genexpressionsmessung durchgeführt werden, gemäß diesem Protokoll über einen Zeitraum von drei Tagen. Das folgende Zeit wird vorgeschlagen: im Voraus, die Transkripte von Interesse auswählen, zu identifizieren und die Primerpaare zu validieren, die die cDNA von jenen transcr verstärkenIPTS und bereite die Standards und Zündmittelmischungen diese Primer verwendet. Am Tag 1 nach Zellbehandlung, Ernte und sortieren Sie die Zellen, führen die reverse Transkription und spezifische Ziel-Amplifikation und Behandlung der Proben mit einer Exonuklease unincorporated Primer zu entfernen. Am Tag 2, führen an der sortierten Zellen Qualitätskontrolle mit qPCR. Schließlich, am Tag 3, messen die Genexpression in den sortierten Zellen mikrofluidischen qPCR verwendet wird. In Abbildung 1 sind die Schritte beteiligt.
Protokoll
1. Eine frühzeitige Vorbereitung
- Wählen Sie bis zu 96 Gene von Interesse, deren Expression gemessen wird.
HINWEIS: Mindestens eines dieser Gene sollte ein "housekeeping Gens" sein, wie zum Beispiel ACTB oder GAPDH, dass bekannt ist, bei einer relativ hohen und konstanten Niveau unter den Bedingungen in dem Experiment verwendet exprimiert wird. Dieses Gen wird verwendet, um positiv sortiert Vertiefungen (Schritt 8.1) und die amplifizierten Proben (Schritt 10.1) zu identifizieren.
HINWEIS: Für das Beispiel Experiment gut charakterisierte, direkte Ziele von p53 mit einer Vielzahl von bekannten Funktionen 11, 14 und GAPDH als Housekeeping Kontrolle ausgewählt wurden. Bitte siehe Referenz 11 für die komplette Liste von Zielgenen und Primersequenzen in dieser Studie verwendet. - Identifizieren Sie potenzielle Primerpaare spezifisch für die Gene von Interesse (zB mit Hilfe der wissenschaftlichen Literatur oder ein Primer - Design - Tool wie Primer-BLAST) 15. Lassen Sie die Grundierungs synthetisiert und sie bei einer Stoffkonzentration von 100 & mgr; M in nukleasefreiem Wasser halten.
HINWEIS: Diese Primer sollten 15-25 Basen lang sein; ein Amplikon 90-130 Basenpaare (bp) lang produzieren; umspannen ein Exon-Übergang, falls vorhanden, die Chance zur Verstärkung der genomischen DNA zu verringern; Schmelztemperaturen von 60 ± 1 ° C haben; und haben minimale Sekundärstruktur 16. Diese Primer werden prime reverse Transkription verwendet werden, verstärken cDNA aus den Transkripten von Interesse, und messen die Genexpression in der DNA-bindenden Farbstoff-basierte qPCR. - Überprüfen Sie die Primer.
- Zunächst erhalten cDNA aus den Zellen von Interesse. Ernte RNA aus den Zellen eine RNA Ernte Kit nach den Anweisungen des Herstellers oder molekularbiologischen Standardverfahren 17. Revers transkribiert die RNA in cDNA eine reverse Transkription-Kit mit zufälligen Hexameren unter Verwendung entsprechend den Anweisungen des Herstellers oder Standard-methods 17.
- Für jedes Primerpaar eingesetzten qPCR mit Farbstoff Master-Mix DNA-Bindung, Vorwärts- und Reverse-Primer, und die cDNA aus dem vorherigen Schritt, im Anschluss an die Master-Mix Herstellerempfehlungen vorgeschlagen Reaktionsbedingungen. Führen Sie diese Reaktionen auf einer Echtzeit-PCR-Maschine mit einem thermischen Zyklus Protokoll, das vom Master-Mix Hersteller empfohlenen verwenden, die Schmelzkurve Akquisition umfasst.
- Die amplifizierten DNA - Proben auf einem 2% w / v Agarosegel nach der qPCR, ausführen und die DNA mittels einer DNA - interkalierenden Farbstoffes visualisieren nach einem Standardprotokoll 17.
- Bestimmen die Effizienz des Primerpaars durch eine Standardkurve von seriellen Verdünnungen von cDNA Konstruktion (hergestellt in Schritt 1.3.1) entsprechend einem Standardprotokoll 16.
HINWEIS: Ein guter Primerpaar wird mit einem einzigen, gut definierte Spitze und eine einzelne Bande in der erwarteten Position auf dem Gel 16 eine Schmelzkurve ergeben, up class = "xref"> 17 (Abbildung 2). Wenn die Schmelzkurve hat mehrere Spitzen oder eine "Schulter" oder, wenn das Gel mehrere Bänder oder einen Abstrich hat, sind zu verstärken die Primer eine Off-Target-Sequenz. Redesign alle Primer, die beobachtet wurden, Off-Target-Sequenzen und wiederholen Sie die vorhergehenden Validierungsschritte wie nötig zu verstärken. Ein gutes Primerpaar wird auch eine Effizienz von 90-110% haben, wobei R 2 ≥0.985 für die Standardkurve 16; Redesign alle Primer für die der Wirkungsgrad oder R 2 fallen außerhalb dieser Bereiche.
- Bereiten Standards aus gereinigter DNA - Amplikons.
- Für jeden validierten Primerpaar:
- Erhöhen der Region von Interesse von cDNA, die eine High-Fidelity DNA-Polymerase gemäß den Empfehlungen des Polymerase-Hersteller für Primer und Templat-DNA-Konzentrationen, die Reaktionsbedingungen und PCR-Zyklusbedingungen, oder ein Standard-PCR-Protokoll"xref"> 17.
- Führen der amplifizierten cDNA auf einem 2% w / v Agarosegel und zu visualisieren , die Band mit einem DNA - interkalierenden Farbstoffes 17. Excise die Band, die die Amplikon einen sauberen Rasierklinge und legen Sie das Gelstück in einem Reaktionsgefäß.
- Man reinige das Amplikon aus dem Gel ein Standard - Gel - Extraktionsverfahren, wie beispielsweise ein Agarase-basierte Extraktion 17 oder Spin-Säule Gel Extraction Kit nach den Anweisungen des Herstellers.
- Messen der Konzentration von Amplikon eine fluoreszenzbasierte dsDNA Quantifizierungs Kit nach den Anweisungen des Herstellers. Unterteilen den gemessenen dsDNA-Konzentration durch das bekannte Molekulargewicht des Amplikons auf der Amplicon-Sequenz basiert, die Anzahl der Amplicon-Molekülen pro & mgr; l zu erhalten.
- In einem 2,0 ml schwach bindende Mikrozentrifugenröhrchen, fügen 2 ul gereinigtem Amplikon auf ein Volumen von DNA Suspensionspuffer, so daß die Endkonzentration an amplicon ist 5 x 10 8 Molekülen / ul.
- Wenn alle Amplifikate gereinigt wurden, kombinieren 18 ul jedes gereinigten Amplikon in einer 2,0-ml schwach bindende Reaktionsgefäß. In DNA Suspensionspuffer auf ein Gesamtvolumen von 1.800 & mgr; l zu machen; Somit ist die Konzentration jedes Amplikon 5 x 10 & sup6 ; Moleküle / & mgr; l.
HINWEIS: Diese Mischung, die cDNA-Amplikons aus allen Genen von Interesse in bekannten äquimolaren Mengen enthält, werden in Einzelzellen-qPCR als Standard dienen. - Vortex dieses "5E6" Standard gründlich, Spin für 10 Sekunden bei 2000 · g, und teilen sich in 20 ul Aliquots in Nieder Bindung PCR-Röhrchen. Speichern Sie die Aliquots bei -80 ° C.
- Für jeden validierten Primerpaar:
- Bereiten Sie die spezifischen Ziel - Amplifikation (STA) Primer - Mix (10x).
- In einem 15 ml konischen Röhrchen, kombinieren 25 ul jeder 100 uM Lager Primer (bis zu 192). In DNA Suspensionspuffer auf ein Gesamtvolumen von 5.000 & mgr; l zu machen.
Beachten Sie dasKonzentration jedes Primers in dieser Mischung beträgt 500 nM. Jede Platte der sortierten Zellen werden 105,6 ul dieser Primer-Mix prime die reverse Transkription und spezifische Ziel-Amplifikation verwendet werden. Das Volumen der Primermix hier gegeben ist genug 45 Platten zu machen, zu sortieren. Es ist ratsam, eine ausreichend große Menge Primermischung zu machen, so dass es nur einmal gemacht werden; skaliert die Volumina wie nötig. - Mischen Sie gründlich durch Vortexen, teilen sich in 110 ul Aliquots, und speichern Sie die Aliquots bei -20 ° C.
- In einem 15 ml konischen Röhrchen, kombinieren 25 ul jeder 100 uM Lager Primer (bis zu 192). In DNA Suspensionspuffer auf ein Gesamtvolumen von 5.000 & mgr; l zu machen.
- Bereiten Assay - Mischungen, eines für jedes Primerpaar für den Einsatz in mikrofluidischen Chip-basierte qPCR.
- In jede Vertiefung einer Platte mit 96 Vertiefungen PCR kombinieren 3,0 ul der 100 & mgr; M Vorwärtsprimer für ein gegebenes Gen, 3,0 & mgr; l des entsprechenden 100 uM reverse-Primer, 24,0 & mgr; l DNA Suspensionspuffer, und 30,0 & mgr; l 2x Assay Lade Reagenzes .
- Vortex diese Platte, Spin für 10 Sekunden bei 500 · g, und bei -20 ° C.
- Erstellen Sie zwei thermischen Zyklen Programme: RTSTA (Reverse Transkription und spezifische Ziel - Amplifikation; Tabelle 1) und ExoI (Exonuklease I - Behandlung; Tabelle 2).
2. Behandlung
- Platte die Säugerzellen auf einer Gewebekulturschale, so dass sie auf die gewünschte Konfluenz zum Zeitpunkt der Behandlung wachsen wird, abhängig von den Bedingungen der experimentellen Studie. Für das Beispiel in dieser Studie präsentierten, Platte 4 x 10 5 MCF-7 - p53-Venus Zellen 18 pro 6 cm Schale in RPMI mit 10% fötalem Rinderserum (FBS), 100 U / ml Penicillin, 100 mg / ml Streptomycin und 250 ng / ml Amphotericin B. Inkubiere die Zellen für zwei Tage bei 37 ° C mit 5% CO 2 , bis sie 50% Konfluenz erreichen.
- Behandeln Sie die Zellen, die den Zustand von Interesse auf der experimentellen Studie basiert zu etablieren. Für das Beispiel in dieser Studie vorgestellt, brüten MCF-7-p53-Venus-Zellen mit 400 ng / ml neocarzinoStatin für 3 h, 8,5 h, 14 h oder 24 h.
3. Lysispuffer Vorbereitung für Zellsortierung
HINWEIS: eine einzelne Platte Lysepuffer Herstellung dauert ca. 1 Stunde. Es ist ratsam, zu machen und zu sortieren mehrere Platten, wie Zellsortierung ineffizient sein kann und viele Brunnen ohne nachweisbare Zelle ergeben.
- Reinigen Sie die Bank, Pipetten, Röhrchen-Rack, kalt PCR Plattenhalter und Handschuhe mit DNA-Dekontaminationslösung in Vorbereitung für die PCR.
- Verdünnen Sie die RNAse-Inhibitor auf 2,64 U / ul durch Lager RNAse Inhibitor Zugabe zu nukleasefreiem Wasser in einem 1,5-ml-Röhrchen.
ANMERKUNG: Dies ermöglicht RNAse Inhibitor ohne Pipettieren Unter ul Volumina Lysepuffer hinzugefügt werden. - Machen Lysepuffer für die Zellen , die durch Vereinigung der Komponenten in Tabelle 3 aufgeführt.
- Transfer 92,4 & mgr; l des Lysepuffers für die Zellen in ein separates Rohr; Dies wird der Lysepuffer für die Amplicon-Standards sein.
- Mit low-bindenden Pipettenspitzen und Reaktionsgefäße, bereiten 200 & mgr; l und 500 & mgr; l E. coli - DNA verdünnt auf 3,1 pg / ul und 6,2 pg / ul jeweils mit Puffer DNA - Suspension.
- Hinzufügen , 184,8 & mgr; l von 3,1 pg / ul E. coli DNA zu dem Lysepuffer für die Zellen.
HINWEIS: Der E. coli - Träger - DNA dient dazu , den Umfang der nicht - spezifische Primer - Bindungsmöglichkeiten zu erweitern und dadurch die Wahrscheinlichkeit zu verringern , dass Primer Dimerisierung zu einem PCR - Produkt führen wird. - Bereiten Standards.
- Beschriften sechs schwach bindende Mikrozentrifugenröhrchen als 5e5, 5e4, 5e0 ....
- In 90 ul DNA Suspensionspuffer auf die "5e5" Rohr.
- Werden 90 & mgr; l von 6,2 pg / ul E. coli DNA an jede der anderen fünf Rohre. Halten Sie alle Röhrchen auf Eis.
HINWEIS: Die Einbeziehung Träger-DNA in die Standards macht die Gesamtmasse der DNA in den Standardvertiefungen vergleichbar mit der in der 1-Zelle Vertiefungen; Dadurch verringert sich die probability dass die Konzentration der Standards werden durch DNA-Bindung an Rohren oder Pipettenspitzen berührt. - Nehmen Sie einen aliquoten Teil der "5E6" Standard (5 x 10 6 Moleküle / ul jedes Amplikon, hergestellt in Schritt 1.4) aus dem Speicher. Vortex kurz und Spin kurz (5 sec bei 500 xg), um sicherzustellen, daß die Flüssigkeit am Boden des Röhrchens ist.
- Mit einem Low-Bindung Pipettenspitze, mit 10 & mgr; l von 5E6-Standard zum "5e5" Rohr. Vortex kurz, Spin (5 sec bei 500 · g), und setzen Sie den Schlauch wieder auf dem Eis.
- Je 10 & mgr; l aus dem "5e5" Rohr in das "5E4" Rohr. Vortex kurz, Spin (5 sec bei 500 · g) und auf Eis gelegt zurück.
- Wiederholen Sie diese Verdünnungsreihen, wobei jede Rohraufnahme 10 & mgr; l aus dem vorherigen Rohr, bis alle Rohre eine Verdünnung von Standard haben.
- Bereiten Sie eine Reihe von 12-PCR-Röhrchen mit Kappen. zu jedem Röhrchen 67 ul "Zelle" Lysepuffer verteilen. Verschließen Sie die Rohre und kurz abzentrifugieren (5sec bei 500 xg), falls erforderlich.
- Mit einem 12-Kanal - Pipette, verteilen 9 ul "Zelle" Lysepuffer aus der Reihe von PCR - Röhrchen in jede Vertiefung der ersten 7 Zeilen einer PCR - Platte (Abbildung 3).
- Verteilen 7 ul "standard" Lysepuffer zu jeder Vertiefung der letzten Reihe der Platte (3).
- Mit Low-Bindung Pipettenspitzen, fügen Sie 2 ul Standard zu jeder Vertiefung der unteren Reihe entsprechend der Plattenbelegung (Abbildung 3).
HINWEIS: 2 ul 5 x 10 0 Moleküle / ul Standard beträgt 10 Moleküle usw. - Hinzufügen 2 & mgr; l von 3,1 pg / ul E. coli DNA in die 10-Zellen - und 100-Zellen - Vertiefungen; Add 2 ul von 6,2 pg / ul E. coli - DNA in die No-Zelle Brunnen.
HINWEIS: Jedes 1-Zellen, 10-Zellen und 100-Zellen und hat 6,2 pg E. coli DNA, Annähern der Masse der genomischen DNA in einer menschlichen Zelle. Jeder Standard-und No-Zelle gut hat 12,4 pg of E. coli - DNA, etwa zwei menschlichen Wert der Zellen. - Verschließen Sie die Platte eine sterile thermische Dichtung verwendet, Spin für 5 Sekunden bei 500 × g und bei 4 ° C, bis die Zellen sind bereit für die Sortierung.
4. Cell Sorter-Setup
- Programmieren Sie die Fluoreszenz-aktivierten Zellsortierer in einer 96-Well - Platte zu sortieren nach der Platte Karte (Abbildung 3); keine Zellen sollten in die Vertiefungen H1-H8 oder H11-H12 sortiert werden.
- Stellen Sie sicher , dass die Zellsortierer auch für die Sortierung in einer PCR - Platte ausgerichtet ist.
- Den Winkel des Sortierstrom so gering wie möglich; Diese Einstellung erhöht die Chance, dass die Zellen in den Böden der Vertiefungen sortiert werden, anstatt die Seiten getroffen.
- Um die Maschine zu zielen, Siegel eine leere PCR - Platte und verwenden Sie den Teststrom zu "sortieren" Tropfen PBS auf die Dichtung der leeren Platte, entsprechend den Anweisungen der Maschine 19; die Tröpfchen auf th landen solltee Fläche der Dichtung an einer Stelle oberhalb der Mitte des Schachtes. Für die besten Ergebnisse, stellen Sie sicher das Ziel, über die Länge und Breite der Platte.
- Wenn die Tröpfchen in der richtigen Position nicht landen, wischen sie die Oberfläche der Dichtung ab, neu kalibrieren die Zellsortierer nach den Anweisungen des Maschinenherstellers, und wiederholen, bis die Tröpfchen in der Sortierstrom korrekt abgelegt werden.
5. Zellernte und Sortieren
- Trypsinize die Zellen als geeignet für die Zelllinie. Beispielsweise für MCF-7 - Zellen, anzusaugen , das Medium von den Zellen, spülen Sie die Zellen mit 1 ml von 0,05% Trypsin und Inkubation der Zellen mit 2 ml 0,05% Trypsin für 5 min bei 37 ° C und 5% CO 2 .
- 8 ml von Zellkulturmedium zu den Zellen trypsinisiert und übertragen alle 10 ml der Zellsuspension in ein 15 ml Röhrchen. Zentrifuge für 4 min bei 400 x g.
- Den Überstand aspirieren und Zellpellet in PBS + 2% FBS.
Hinweis: Ein kleiner Aufwirbelung Volumen der Zellen konzentriert und erleichtert die Zellsortierung; Ein 500 & mgr; l der Resuspension Volumen funktioniert gut für eine 6 cm-Schale von Zellen bei 50% Konfluenz. - Übertragen die Zellsuspension auf eine 5 ml-Zytometrie Rohr fließen, Pipettieren durch ein 40 um-Filter Zellklumpen zu entfernen.
- Zentrifugieren der Platte Lysepuffer bei 400 xg für 30 sec bei 4 ° C, bevor die Zellen Sortier um sicherzustellen, daß die Flüssigkeit am Boden der Vertiefungen ist.
- Legen Sie die Röhre mit der Zellsuspension in die Zellsortierer. Öffnen Sie die Dichtung auf der Platte Lysepuffer und sortieren Sie sorgfältig die Zellen von Interesse in die Platte nach der Platte Karte und im Anschluss an die Zellsortierer Anweisungen des Herstellers 19. Um sicherzustellen , dass einzelne Zellen bei der maximalen Reinheit sortiert sind, führen Sie die Maschine in "Einzelzelle" Modus und verwenden Standard - Durchflusszytometrie - Gating basierend auf Vorwärts- und Seitenstreuung 19.
HINWEIS: In derBeispiel Experiment Gate die basierend Zellen auf p53-Venus Fluoreszenz 11 die hellsten 15% der einzelnen Zellen zu isolieren. - Verschließen Sie die Platte mit einer neuen, sterilen thermische Dichtung und Zentrifuge bei 400 × g für 1 min bei 4 ° C. Die Platte wird in den Thermocycler und starten Sie das RTSTA Programm (siehe Schritt 1.7).
HINWEIS: Diese führt die Transkription von den sortierten Zellen auf der mRNA umgekehrt und verstärkt dann die cDNA der Transkripte von Interesse.
6. Exonucleasebehandlung
- Mischen verdünnter Exonuklease I , indem die Komponenten in der Tabelle 4 aufgeführt kombiniert. Bereiten Sie eine Reihe von 12-PCR-Röhrchen mit Kappen. 30,5 ul verdünnter Exonuklease verteile ich in jedes Röhrchen. Verschließe die Röhrchen und Spin kurz (5 sec bei 500 xg), um sicherzustellen, daß die Flüssigkeit am Boden des Röhrchens ist.
- Wenn die RTSTA beendet ist, drehen Sie das Probenplatte ca. 5 s bei 500 x g. Mit einem 12-Kanal-Pipette, verteilen 3,6 ul verdünnter ExonukleaseIch in jede Vertiefung der Platte.
- Verschließen Sie die Platte mit einer sterilen thermische Dichtung und Spin kurz (ca. 5 s bei 500 · g). Die Platte wird in den Thermocycler und starten Sie das ExoI Programm (siehe Schritt 1.7).
Hinweis: Dieser Schritt verschlechtert alle Primer nach spezifischen Ziel-Amplifikation verbleibenden, so dass sie nicht mit zukünftigen PCR stören.
7. Probenverdünnung
- Wenn die Exonuclease-Behandlung abgeschlossen ist, fügen 32,4 & mgr; l TE-Puffer zu jedem Well der Probenplatte; Dies ist eine optionale Haltepunkt, und die Proben können bei -20 ° C für mehrere Tage gelagert werden.
8. Sortieren Qualitätskontrolle qPCR Verwendung
Hinweis: Da Zellsortierung nicht vollkommen effizient ist, ist dieser Schritt notwendig, um festzustellen, welche Vertiefungen der Platte sortierten erhalten tatsächlich eine Zelle. Diese Proben können dann für die weitere Analyse verwendet werden.
- Messen Sie die Housekeeping - Gen - Expression in jeder Probe durch qPCR.
- Bereiten Sie 900 ul der DNA-bindenden Farbstoff qPCR-Mastermix für 96 Reaktionen nach dem Protokoll des Polymerase-Hersteller unter Verwendung von Primern für eine der Housekeeping-Gene in Schritt ausgewählt 1.1; die genauen Konzentrationen der Reaktionspuffer, Polymerase, Primer etc. für diese Reaktion hängt von der spezifischen Polymerase abhängen verwendet werden.
- Verteilen 9 ul Master-Mix zu jeder Vertiefung einer 96-Well-PCR-Platte. 1 ul Probe aus der Probenplatte auf die Vertiefung der PCR-Platte entspricht.
- Führen Sie die Platte auf einer Echtzeit - PCR - Maschine mit einem thermischen Zyklus - Protokoll durch den Master - Mix Hersteller oder nach einem Standardprotokoll 20 vorgeschlagen werden.
- Analysieren Sie den qPCR - Daten 20; ein Beispiel für qPCR Daten von den Sortierqualitätskontrollschritte unten aufgeführt ist in Abbildung 4 dargestellt.
- Stellen Sie sicher, dass die Messungen von den nicht-Zell-Brunnen zeigen einen niedrigen (Hintergrund) Ebeneder Genexpression oder gar keiner Ausdruck.
- Stellen Sie sicher, dass die Messungen von 1-Zelle Brunnen klar in zwei Gruppen unterteilen: positive Proben mit hoher Genexpression und negativen Proben mit Hintergrundwerte des Ausdrucks oder ohne Ausdruck überhaupt, ähnlich wie bei den No-Zelle Brunnen.
HINWEIS: Die Proben mit hoher Genexpression darstellen tatsächlichen sortierten Zellen; Menschen mit Hintergrund Ausdruck sollte von der weiteren Analyse ausgeschlossen werden. - Stellen Sie sicher, dass die Messungen aus den 10-Zellen und 100-Zellen-Brunnen spiegeln höhere Genexpression als in den positiven 1-Zelle Brunnen, so dass für die Möglichkeit, dass ineffiziente Sortier diese Vertiefungen dazu führen könnten, weniger Zellen zu haben als gewünscht.
- Stellen Sie sicher , dass die Messungen von den Standard - Brunnen gleichmäßig im logarithmischen Raum verteilt sind (dh , dass die C t - Werte gleichmäßig verteilt sind).
- Nachdem alle positiven 1-Zellproben zu identifizieren, ein "Sample Mixes" Platte Karte machen positive 1-ce zur Kartell Proben auf eine neue 96-Well-Platte. Fügen Sie acht Vertiefungen für Standards auf dieser Platte Karte. Siehe Abbildung 5 zeigt ein Beispiel.
9. Genexpression Messung mit Mikrofluidik-qPCR
HINWEIS: Für jeden Schritt in diesem Abschnitt Pipette nur bis zum ersten Anschlag die Bildung von Blasen in den Reagenzien zu minimieren.
- Öffnen Sie eine Reihe von 8 PCR-Röhrchen. Wenn es mehrere Platten von Proben sind, öffnen Sie eine zweite Reihe von 8 PCR-Röhrchen und beschriften Sie die Rohre 1-8. Diese Zeile wird für die Bündelung Standards aus mehreren Platten sein.
- In einem 1,5-ml-Röhrchen, mischen 360 & mgr; l DNA-bindenden Farbstoff qPCR-Mastermix mit 36 & mgr; l DNA-bindenden Farbstoff Probenbeladung Reagenz. Vortex kurz und Spin für 10 Sekunden bei 2.000 x g. Teilen Sie diese Mischung (396 ul) in die nicht-markierten Reihe von 8 PCR-Röhrchen, Platzierung 48 & mgr; l in jedes Röhrchen.
- Mit einem 8-Kanal-Pipette, verteilen 3,3 ul des Master-Mix / Probenbeladung Reagenzmischung in jede Vertiefungeines neuen 96-Well-PCR-Platte. Beschriften Sie diese Platte, "Sample Mixes."
- Für jede PCR - Platte der Proben:
- Vortex die Platte für 10 Sekunden und Spin für 10 Sekunden bei 500 x g. Entfernen Sie die Abdeckung und übertragen 2,7 ul jeder positiven 1-Zellprobe auf seine entsprechende Vertiefung auf der Probe Mischt Platte für die 1-Zelle Vertiefungen auf der Platte Karte angezeigt.
- Wenn es nur eine Platte von Proben ist, übertragen 2,7 ul jeder verstärkten Standard auf seine entsprechende Vertiefung auf der Probe Mischt Platte gemäß der Platte Karte.
- Wenn es mehrere Platten von Proben sind, übertragen 2,7 ul jeder verstärkten Standard zu seiner entsprechenden Position in der markierten Reihe von PCR-Röhrchen. In Normen von der aktuellen Platte der Proben auf alle dort bereits Standards. Verschließen Sie die Platte der Proben bei Verwendung eines Dichtungsfolie erfolgen.
- Wenn mehrere Platten der Proben:
- Vortex die Reihe von PCR-Röhrchen, die Standards für die10 sec und Spin für 10 Sekunden bei 2.000 x g. Übertragen 2,7 ul aus jedem Röhrchen von gemischten Standards in das Bohrloch entsprechend auf der Probe Mischt Platte gemäß der Platte Karte.
- Legen Sie die NTC-Brunnen mit 2,7 ul Nuklease-freies Wasser, wo auf der Platte Karte angezeigt. Siegel der Probe Mischt Platte und lagern bei 4 ° C bis benötigt.
- Ziehen Sie die Aufkleber von der Unterseite eines neuen mikrofluidischen qPCR-Chip. Unter Verwendung einer Spritze der Steuerleitung Flüssigkeit mit der Kappe noch auf, drücken Sie jeden Speicherfeder nach unten um sie zu lösen, und spritzen jeden Akkumulator mit 150-200 ul Steuerleitung Flüssigkeit.
- Setzen Sie den Chip in der Lademaschine und starten Sie das "Prime" Skript. Dies dauert ~ 20 min.
- Wenn Grundierung durchgeführt wird, Wirbel und drehen Sie die Platten von Testmischungen nach unten und Probenmischungen (in Schritt 1.6) hergestellt.
- Verwendung eines 8-Kanal-Pipette 4,5 ul jeder Testmischung in die linke Seite des mikrofluidischen qPCR-Chip gemäß der Darstellung in g> 6. Pipette sehr vorsichtig Luftblasen an der Unterseite der Vertiefungen zu schaffen zu vermeiden. Wenn Sie fertig sind, versiegeln Sie die Platte von Assay-Mischungen und bei -20 ° C für die zukünftige Verwendung.
- Übertragen 4,5 ul jeder Probenmischung auf die rechte Seite des mikrofluidischen Chips qPCR gemäß 6. Setzen Sie den Chip in die Lademaschine und führen Sie den "Load Mix" Skript. Dieser Vorgang dauert ca. 90 min.
- Schalten Sie den mikrofluidischen real-time PCR-Maschine, die Software zur Datenerfassung zu starten, und schalten Sie die Lampe um es aufzuwärmen. Dies dauert ~ 20 min.
- Wenn die Last Mix Skript ausgeführt wird, stellen Sie sicher, dass es keine Linien über den sind Chip würde dies eine Ladeproblem hinweisen. Entfernen Sie vorsichtig alle Staubpartikel aus der Verwendung von Klebeband oder Laborband-Chip. Setzen Sie den Chip in den mikrofluidischen real-time PCR-Maschine und die Datenerfassung Skript ausführen. Verwenden die Analysesoftware, um die Ergebnisse zu untersuchen und die Daten für die weitere Analyse exportiert.
- Entfernen Sie die Messungen , für die die qPCR Amplifikationskurven schlechter Qualität zeigen (dh, für die sich die Kurven nicht die Standard - S - förmigen Verstärkungskurve ähnlich) 20.
- Entfernen Sie Proben mit mehr als 30 schlechte Qualität oder Null - Messungen oder für die Housekeeping - Gen - Expression (siehe Schritt 1.1) wesentlich geringer ist als die meisten anderen Proben (Abbildung 7).
- Für jedes Gen, schätzen die richtige Schmelzpeakposition als Median der Schmelzpeak Stellen von jeder Probe. Wenn eine Messung mit einer Schmelz Peak aus dem Median liegt mehr als 1 ° C, zu entfernen , dass die Messung aus der Betrachtung 20.
- Für jedes Gen, erstellen Sie eine Standardkurve der mRNA Zahl in jeder Probe zu schätzen , eine Tabelle oder eine Skriptsprache , 20 verwendet wird .
HINWEIS: Die Standards in diesem Experiment von 10 bestehen, 100, 1.000 oder 10.000 Molekülenvon dsDNA zu jedem Transkript von Interesse entspricht. Wenn die reverse Transkription vollkommen effizient wäre, würde entsprechen die Standards bis 20, 200, 2.000 und 20.000 Molekülen von mRNA, da mRNA und cDNA sind einsträngige. In der Praxis ist dies nicht der Fall, so drücken wir Messungen als eine Schätzung in Einheiten von Molekülen × E RT, wobei E RT die Effizienz der reversen Transkription ist.- Für jeden Standard gut identifizieren m, die Anzahl der Moleküle von ssDNA (zweimal die Anzahl der Moleküle von dsDNA) vor der Verstärkung (beispielsweise 20 - Moleküle, Moleküle 200, etc.).
- Für jeden Standard gut, identifizieren die C t - Wert von qPCR 20.
- Mit den Werten für m und C t, führen Sie eine lineare Regression unter Verwendung der folgenden Gleichung eine Standardkurve mit den Parametern a und b für jedes Gen zu erzeugen, und die R 2 -Wert als ein findenn Indikator für die Güte der Anpassung:
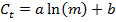
- Von der Standardkurve berechnen Sie die PCR - Effizienz E für jedes Gen:
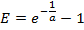
HINWEIS: Ein Wert von E viel größer als 1,0 (zB E> 1.1) ist physikalisch nicht realistisch; ein R 2 -Wert für die lineare Regressions viel weniger als 1,0 (beispielsweise R 2 <0,985) bedeutet , dass die Standards nicht zuverlässig arbeiten. Diese Probleme zeigen allgemein, daß die niedrigste Amplikon Standard unter einen Schwellenwert von zuverlässige Quantifizierung und somit die niedrigste Standard sollte aus der linearen Regression ausgeschlossen. Wird eine angemessene lineare Regression kann nicht mit mindestens drei einzigartigen Standards vorgenommen werden, um die Expressionsmessungen für das Gen zu verwerfen. - Wenn die lineare Regression ausreichend ist, verwenden Sie die Passform Parameter a und b </ em> für jedes Gen die Anzahl von mRNA - Molekülen m est (in Einheiten von Molekülen × E RT) für das entsprechende Gen in jedem Einzelzellprobe aus dem gemessenen Wert C t zu schätzen:
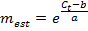
- Bezeichnen Sie jede Messung m est <1 als m est = 0. Auch bezeichnen jede Messung ohne Verstärkung in qPCR und somit Wert keinem C t als m est = 0.
HINWEIS: Die Messungen mit einem m est kleiner als die niedrigste oder größer als der höchste Standard in der Regression werden durch Extrapolation aus der Standardkurve gefunden. Diese Schätzungen sind nicht unbedingt ungültig sollte aber mit Vorsicht betrachtet werden.
- Visualisieren Sie die Einzelzell - Genexpressionsdaten.
- Machen Sie eine beeswarm Grundstück (Dorn, J. und Holy, T., 2012, http://www.mathworks.com/matlabcentral/fileexchange / 37105), bei dem jeder Punkt die Genexpression in einer bestimmten Zelle darstellt; ein Beispiel ist in 8 gezeigt.
- Machen Sie eine Violine Grundstück (Dorn, J., 2012, http://www.mathworks.com/matlabcentral/fileexchange/23661), in dem die Breite der "Violine" stellt die Frequenz der Genexpression Messungen um ein bestimmtes Niveau in der Population von Zellen analysiert; ein Beispiel ist in 8 gezeigt.
HINWEIS: Komplexere Analysen 21 die Beziehungen in der Expression zwischen Paaren von Genen untersuchen können, 22 und kann die Genexpression Netzwerke schließen.
Ergebnisse
Eine allgemeine Übersicht über das Protokoll ist in Abbildung 1, mit Schritten zur Zellbehandlung, die Isolierung von einzelnen Zellen durch FACS, die Erzeugung gezeigt und Vorverstärkung von cDNA - Bibliotheken aus single-Zelllysaten, die Bestätigung der Einzelzell - cDNA - Bibliotheken in sortierter Wells, und die Messung der Genexpression durch qPCR.
In Vorbereitung auf die Analyse Einzelzellisolierung ...
Diskussion
Wir haben zur Isolierung von einzelnen Säugerzellen aus einer Population von anhaftenden Zellen in der Kultur und zur Untersuchung der Expression von etwa 96 Gene in jeder Zelle gezüchtet ein Verfahren dargestellt. Gute rechtzeitige Vorbereitung ist entscheidend für diese Methode gut zu funktionieren. Insbesondere Entwerfen und Testen Primerpaare spezifisch für die Transkripte von Interesse (Schritte 1,2-1,3) sind zeitaufwendig, aber wichtige Schritte, wie die Primer, die Qualität der Einzelzellmessungen bestimmen....
Offenlegungen
The authors have nothing to disclose.
Danksagungen
Wir möchten, dass bei der Durchführung der Zellsortierung bei der Entwicklung dieses Protokolls V. Kapoor in der CCR ETIB Flow Cytometry Kern für ihre Hilfe zu danken. Wir danken auch M. Raffeld und die CCR LP Molecular Diagnostics Unit und J. Zhu und die NHLBI DNA-Sequenzierung und Genomics Core-für ihre Hilfe in der qPCR bei der Entwicklung dieses Protokolls durchgeführt wird. Diese Forschung wurde von der Intramural Programm des NIH unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| High Capacity cDNA Reverse Transcription Kit with RNase Inhibitor | ThermoFisher | 4374966 | |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530S | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| Quant-iT High-Sensitivity dsDNA Assay Kit | ThermoFisher | Q33120 | |
| 2.0 ml low adhesion microcentrifuge tubes | USA Scientific | 1420-2600 | |
| DNA Suspension Buffer | Teknova | T0221 | |
| Axygen 0.2 ml Maxymum Recovery Thin Wall PCR Tubes | Corning | PCR-02-L-C | |
| GE 96.96 Dynamic Array DNA Binding Dye Sample & Assay Loading Reagent Kit | Fluidigm | 100-3415 | |
| HyClone RPMI 1640 media | GE Healthcare Life Sciences | SH30027.01 | |
| Fetal Bovine Serum, Certified (US) | ThermoFisher | 16000-044 | |
| Antibiotic-Antimycotic Solution | Corning | 30-004-CI | |
| Neocarzinostatin | Sigma | N9162 | |
| ELIMINase | Decon Labs | 1101 | |
| SUPERase-In | ThermoFisher | AM2696 | |
| CellsDirect One-Step qRT-PCR Kit | ThermoFisher | 11753500 | |
| E. coli DNA | Affymetrix | 14380 10 MG | |
| ThermalSeal Sealing Film, Sterile | Excel Scientific | STR-THER-PLT | |
| BD FACSAria IIu | BD Biosciences | ||
| HyClone Trypsin 0.05% | GE Healthcare Life Sciences | SH30236.01 | |
| PBS, 1x | Corning | 21-040-CV | |
| Falcon 40 µm Cell Strainer | Corning | 352340 | |
| Exonuclease I | New England BioLabs | M0293S | |
| SsoFast EvaGreen Supermix with Low ROX | Bio-Rad | 172-5210 | |
| 96.96 Dynamic Array IFC for Gene Expression (microfluidic qPCR chip) | Fluidigm | BMK-M-96.96 | |
| IFC Controller HX (loading machine) | Fluidigm | ||
| BioMark or BioMark HD (microfluidic qPCR machine) | Fluidigm | ||
| Real-Time PCR Analysis software | Fluidigm | ||
| MATLAB software | MathWorks |
Referenzen
- Feinerman, O., Veiga, J., Dorfman, J. R., Germain, R. N., Altan-Bonnet, G. Variability and Robustness in T Cell Activation from Regulated Heterogeneity in Protein Levels. Science. 321 (5892), 1081-1084 (2008).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- Geva-Zatorsky, N., Rosenfeld, N., et al. Oscillations and variability in the p53 system. Mol. Syst. Biol. 2 (1), (2006).
- Colman-Lerner, A., Gordon, A., et al. Regulated cell-to-cell variation in a cell-fate decision system. Nature. 437 (7059), 699-706 (2005).
- Chong, S., Chen, C., Ge, H., Xie, X. S. Mechanism of Transcriptional Bursting in Bacteria. Cell. 158 (2), 314-326 (2014).
- Raj, A., Peskin, C. S., Tranchina, D., Vargas, D. Y., Tyagi, S. Stochastic mRNA Synthesis in Mammalian Cells. PLoS Biol. 4 (10), e309+ (2006).
- Senecal, A., Munsky, B., et al. Transcription Factors Modulate c-Fos Transcriptional Bursts. Cell Rep. 8 (1), 75-83 (2014).
- Dey, S. S., Foley, J. E., Limsirichai, P., Schaffer, D. V., Arkin, A. P. Orthogonal control of expression mean and variance by epigenetic features at different genomic loci. Mol. Syst. Biol. 11 (5), 806+ (2015).
- Dar, R. D., Razooky, B. S., et al. Transcriptional burst frequency and burst size are equally modulated across the human genome. Proc. Natl. Acad. Sci. U.S.A. 109 (43), 17454-17459 (2012).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- Porter, J. R., Fisher, B. E., Batchelor, E. p53 Pulses Diversify Target Gene Expression Dynamics in an mRNA Half-Life-Dependent Manner and Delineate Co-regulated Target Gene Subnetworks. Cell Syst. 2 (4), 272-282 (2016).
- Lahav, G., Rosenfeld, N., et al. Dynamics of the p53-Mdm2 feedback loop in individual cells. Nat. Genet. 36 (2), 147-150 (2004).
- Levine, A. J., Oren, M. The first 30 years of p53: growing ever more complex. Nat. Rev. Cancer. 9 (10), 749-758 (2009).
- Riley, T., Sontag, E., Chen, P., Levine, A. Transcriptional control of human p53-regulated genes. Nat. Rev. Mol. Cell Biol. 9 (5), 402-412 (2008).
- Ye, J., Coulouris, G., Zaretskaya, I., Cutcutache, I., Rozen, S., Madden, T. L. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinf. 13 (1), 134 (2012).
- . . PCR Technologies: A Technical Guide. , (2014).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Batchelor, E., Mock, C. S., Bhan, I., Loewer, A., Lahav, G. Recurrent initiation: a mechanism for triggering p53 pulses in response to DNA damage. Mol. Cell. 30 (3), 277-289 (2008).
- . . Flow Cytometry: Principles and Applications. , (2007).
- . . Real-time PCR. , (2006).
- Song, L., Langfelder, P., Horvath, S. Comparison of co-expression measures: mutual information, correlation, and model based indices. BMC Bioinf. 13 (1), 328 (2012).
- Margolin, A. A., Nemenman, I., et al. ARACNE: An Algorithm for the Reconstruction of Gene Regulatory Networks in a Mammalian Cellular Context. BMC Bioinf. 7 (Suppl 1), (2006).
- Haff, L. A. Improved quantitative PCR using nested primers. PCR Methods Appl. 3 (6), 332-337 (1994).
- Hashimshony, T., Senderovich, N., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biol. 17, 77 (2016).
- Ronander, E., Bengtsson, D. C., Joergensen, L., Jensen, A. T. R., Arnot, D. E. Analysis of Single-cell Gene Transcription by RNA Fluorescent In Situ Hybridization (FISH). J. Vis. Exp. (68), e4073 (2012).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat. Methods. 5 (10), 877-879 (2008).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nat. Methods. 9 (7), 743-748 (2012).
- Battich, N., Stoeger, T., Pelkmans, L. Image-based transcriptomics in thousands of single human cells at single-molecule resolution. Nat. Methods. 10 (11), 1127-1133 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten