È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Unicellulare profili di espressione genica Utilizzando FACS e qPCR con standard interni
In questo articolo
Riepilogo
We describe a method to sort single mammalian cells and to quantify the expression of up to 96 target genes of interest in each cell. This method includes the use of internal qPCR standards to enable the estimation of absolute transcript counts.
Abstract
Gene expression measurements from bulk populations of cells can obscure the considerable transcriptomic variation of individual cells within those populations. Single-cell gene expression measurements can help assess the role of noise in gene expression, identify correlations in the expression of pairs of genes, and reveal subpopulations of cells that respond differently to a stimulus. Here, we describe a procedure to measure the expression of up to 96 genes in single mammalian cells isolated from a population growing in tissue culture. Cells are sorted into lysis buffer by fluorescence-activated cell sorting (FACS), and the mRNA species of interest are reverse-transcribed and amplified. Gene expression is then measured using a microfluidic real-time PCR machine, which performs up to 96 qPCR assays on up to 96 samples at a time. We also describe the generation and use of PCR amplicon standards to enable the estimation of the absolute number of each transcript. Compared with other methods of measuring gene expression in single cells, this approach allows for the quantification of more distinct transcripts than RNA FISH at a lower cost than RNA-Seq.
Introduzione
Cellule individuali in una popolazione può mostrare risposte molto diverse ad uno stimolo uniforme fisiologica 1, 2, 3, 4. La variazione genetica di cellule in una popolazione è un meccanismo per questa varietà di risposte, ma ci sono anche diversi fattori non genetici che possono aumentare la variabilità delle risposte, anche in una popolazione clonale delle cellule. Ad esempio, i livelli di singole proteine e di altre importanti molecole di segnalazione possono variare su base cella per cella, dando luogo a variazioni nei profili di espressione genica a valle. Inoltre, l'attivazione del gene può avvenire in esplosioni di breve durata di trascrizioni 5, 6, che può essere limitato ad un numero relativamente piccolo di trascrizioni per raffica 7, 8, 9. Comestocasticità in attivazione del gene può contribuire notevolmente alla variabilità nelle risposte biologiche e in grado di fornire un vantaggio selettivo nei microrganismi 10 e in cellule di mammifero 1, 2 risponde a uno stimolo fisiologico. Grazie alle due fonti genetiche e non genetiche di variazione, il profilo di espressione genica di una data cella in risposta ad uno stimolo può differire notevolmente dal profilo medio espressione genica ottenuti dalla misurazione della risposta bulk. Determinare la misura in cui le singole celle mostrano variabilità in risposta ad uno stimolo richiede tecniche per l'isolamento di singole cellule, la misurazione dei livelli di espressione di trascritti di interesse, e l'analisi computazionale dei dati di espressione risultanti.
Ci sono diversi approcci per saggiare l'espressione genica in cellule singole, che coprono una vasta gamma di costi, il numero di trascrizioni sondato, ela precisione di quantificazione. Ad esempio, monocellulari RNA-Seq offre un'ampia profondità di copertura trascrizione e la capacità di quantificare migliaia di trascritti distinti per i geni più altamente espresso in singole cellule; Tuttavia, il costo associato con tale profondità sequenziamento può essere proibitivo, anche se i costi continuano a diminuire. Al contrario, una sola molecola di RNA ibridazione in situ fluorescente (FISH smRNA) offre una precisa quantificazione di trascritti per anche a bassa espressione dei geni ad un costo ragionevole per ogni gene di interesse; Tuttavia, solo un piccolo numero di geni bersaglio può essere analizzato in una data cella da questo approccio. test basati sulla PCR quantitativa, descritti in questo protocollo, forniscono una via di mezzo tra queste tecniche. Questi test impiegano un real-time PCR macchina microfluidica di quantificare fino a 96 trascrizioni di interesse in un momento in un massimo di 96 celle. Mentre ciascuno dei metodi di cui sopra ha costi hardware necessari, il costo di qualsiasi saggio individuale qPCR è relativamenteBasso. Questo protocollo è adattato da quello suggerito da un produttore di un real-time PCR macchina microfluidica (Protocollo di ADP 41, Fluidigm). Per abilitare la stima del numero assoluto di ogni trascritto in un approccio basato sulla PCR, abbiamo ampliato il protocollo di avvalersi di controlli interni di preparati ampliconi gene bersaglio che possono essere utilizzati in più esperimenti.
Come esempio di questa tecnica, la quantificazione dell'espressione dei geni regolati dal soppressore del tumore p53 in cellule MCF-7 di carcinoma mammario umano è descritto 11. Le cellule sono sfidati con un agente chimico che induce DNA rotture del doppio filamento. Studi precedenti hanno dimostrato che la risposta p53 al DNA rotture del doppio filamento presenta una grande eterogeneità in singole cellule, sia in termini di livelli di p53 12 e nell'attivazione di distinti geni bersaglio 11. Inoltre, p53 regola l'espressione di oltre 100ben caratterizzato geni bersaglio coinvolti in numerose vie a valle, tra cui l'arresto del ciclo cellulare, apoptosi, e senescenza 13, 14. Poiché la risposta p53-mediata in ogni cella è sia complessa e variabile, l'analisi delle prestazioni del sistema da un approccio in cui quasi 100 geni bersaglio può essere sondata simultaneamente in singole cellule, come quello descritto di seguito. Con lievi modifiche (quali metodi alternativi per isolamento cella singola e lisi), il protocollo può essere facilmente adattato per studiare una vasta gamma di tipi di mammiferi, cellule trascrizioni e risposte cellulari.
Con una corretta preparazione anticipata, un giro di smistamento delle cellule e misura l'espressione genica può essere condotta in base a questo protocollo per un periodo di tre giorni. Il seguente tempistica è suggerito: in anticipo, selezionare le trascrizioni di interesse, identificare e convalidare le coppie di primer che amplificano il cDNA da quelli trascrIPTS, e preparare gli standard e le miscele di primer che utilizzano tali primer. Il giorno 1, in seguito al trattamento delle cellule, raccolta e ordinare le celle, eseguire la trascrizione inversa e l'amplificazione target specifico, e trattare i campioni con una esonucleasi per rimuovere primer non incorporati. Il giorno 2, eseguire il controllo di qualità sulle cellule ordinati utilizzando qPCR. Infine, il giorno 3, misurare l'espressione genica nelle cellule ordinati usando microfluidica qPCR. La Figura 1 riassume i passi necessari.
Protocollo
1. Preparazione Advance
- Selezionare fino a 96 geni di interesse la cui espressione sarà misurato.
NOTA: Almeno uno di questi geni dovrebbe essere un "gene housekeeping", come ACTB o GAPDH, che è noto per essere espressa ad un livello relativamente elevato e costante in condizioni utilizzate nell'esperimento. Questo gene verrà utilizzato per identificare pozzetti filtrate positivamente (passo 8.1) e di campioni amplificati (passo 10.1).
NOTA: Per l'esempio esperimento, ben caratterizzato, bersagli diretti di p53 con una varietà di funzioni note 11, 14 e GAPDH come controllo di pulizia sono stati selezionati. Si prega di consultare di riferimento 11 per l'elenco completo dei geni bersaglio e sequenze di primer utilizzati in questo studio. - Identificare i potenziali coppie di primer specifici per i geni di interesse (ad esempio, utilizzando la letteratura scientifica o uno strumento di progettazione fondo come Primer-BLAST) 15. Avere il primers sintetizzato e mantenerli in un magazzino concentrazione di 100 micron di acqua priva di nucleasi.
NOTA: Questi primer dovrebbe essere lunga 15-25 basi; produrre un amplicone 90-130 coppie di basi (bp) di lunghezza; estendersi una giunzione esone, se ne esiste uno, per ridurre la possibilità di amplificare DNA genomico; hanno temperature di fusione di 60 ± 1 ° C; e hanno il minimo struttura secondaria 16. Questi primer saranno utilizzati per trascrizione inversa primo, amplificare il cDNA dalle trascrizioni di interesse, e misurare l'espressione genica nel legame al DNA qPCR dye-based. - Convalida i primer.
- Innanzitutto, ottenere cDNA dalle cellule di interesse. Harvest RNA dalle cellule utilizzando un kit di RNA di raccolta in base alle istruzioni del produttore o dei metodi di biologia molecolare standard di 17. Reverse-trascrivere l'RNA in cDNA utilizzando un kit di trascrizione inversa con esameri casuali in base alle istruzioni del produttore o metho di serieds 17.
- Per ogni coppia di primer, istituito qPCR con legame al DNA colorante master mix, in avanti e retromarcia primer, e il cDNA dal passaggio precedente, seguendo le raccomandazioni del costruttore master mix di condizioni di reazione suggerite. Eseguire queste reazioni su una macchina di PCR in tempo reale utilizzando un protocollo ciclo termico consigliato dal produttore master mix che comprende l'acquisizione della curva di fusione.
- Dopo la qPCR, eseguire i campioni di DNA amplificati su un 2% w / v gel e visualizzare il DNA tramite un colorante intercalante del DNA a seguito di un protocollo standard 17.
- Determinare l'efficienza della coppia di primer costruendo una curva standard da diluizioni seriali di cDNA (fatta allo step 1.3.1) secondo un protocollo standard 16.
NOTA: Un buon paio di primer produrrà una curva di fusione con un picco singolo ben definito e una singola banda nella posizione prevista sul gel 16, fino class = "xref"> 17 (Figura 2). Se la curva di fusione ha più picchi o una "spalla", o se il gel ha più bande o uno striscio, i primer sono amplificando una sequenza esce a lato. Riprogettare eventuali primer che sono stati osservati per amplificare le sequenze off-target e ripetere la procedura di validazione precedenti, se necessario. Un buon paio di primer avrà anche un'efficienza del 90-110%, con R 2 ≥0.985 per la curva standard 16; ridisegnare eventuali primer per cui l'efficienza o R 2 cadono fuori di questi intervalli.
- Preparare gli standard di ampliconi DNA purificato.
- Per ogni coppia di primer convalidato:
- Amplificare la regione di interesse da cDNA utilizzando una ad alta fedeltà DNA polimerasi in base alle raccomandazioni del produttore della polimerasi per le concentrazioni di primer e DNA stampo, condizioni di reazione, e le condizioni di ciclismo PCR, o utilizzando un protocollo standard di PCR"xref"> 17.
- Eseguire il cDNA amplificato su un 2% w / v gel e visualizzare la band con un DNA intercalando tintura 17. Asportare la band che rappresenta il amplicone con una lama di rasoio pulito e posizionare il pezzo di gel in una provetta.
- Purificare il amplicon dal gel utilizzando una procedura di estrazione gel standard, come estrazione agarase a base 17 o un kit di estrazione di gel spin-colonna, secondo le istruzioni del produttore.
- Misurare la concentrazione di amplicone utilizzando un kit quantificazione dsDNA basato sulla fluorescenza secondo le istruzioni del produttore. Dividere la concentrazione di dsDNA misurata dal peso molecolare noto dell'amplicone basata sulla sequenza amplicone per ottenere il numero di molecole ampliconi per microlitri.
- In 2,0 ml tubo low-binding microcentrifuga, aggiungere 2 ml di amplicone purificato ad un volume di tampone sospensione DNA tale che la concentrazione finale di amplificatorelicon è di 5 x 10 8 molecole / ml.
- Quando tutti ampliconi sono stati purificati, unire 18 ml di ogni amplicone purificati in 2,0 ml di tubo a basso legame microcentrifuga. Aggiungere tampone sospensione DNA per fare un volume totale di 1800 ml; pertanto, la concentrazione di ogni amplicone è di 5 x 10 6 molecole / ml.
NOTA: Questa miscela, che contiene ampliconi cDNA da tutti i geni di interesse in quantità equimolari noti, servirà come standard nella cella singola qPCR. - Vortex questo "5e6" standard a fondo, rotazione per 10 sec a 2.000 xg, e dividere in 20 aliquote microlitri in tubi a bassa vincolante PCR. Conservare le aliquote a -80 ° C.
- Per ogni coppia di primer convalidato:
- Preparare la Primer Mix (10x) specifica amplificazione del target (STA).
- In un tubo da 15 ml, unire 25 ml di ogni 100 micron magazzino fondo (fino a 192). Aggiungere tampone sospensione DNA per fare un volume totale di 5000 ml.
Notare laconcentrazione di ciascun primer in questa miscela è 500 nM. Ogni piatto di cellule ordinati utilizzerà 105,6 ml di questa miscela di primer per innescare la trascrizione inversa e l'amplificazione target specifico. Il volume di primer mix data qui è abbastanza per fare 45 piatti da ordinare. Si consiglia di fare una grande abbastanza volume di miscela di primer in modo che solo bisogno di essere fatta una sola volta; scalare i volumi, se necessario. - Mescolare accuratamente con il vortex, dividere in 110 microlitri aliquote, e conservare le aliquote a -20 ° C.
- In un tubo da 15 ml, unire 25 ml di ogni 100 micron magazzino fondo (fino a 192). Aggiungere tampone sospensione DNA per fare un volume totale di 5000 ml.
- Preparare le miscele test, uno per ogni coppia di primer, per l'uso in base chip microfluidico qPCR.
- In ciascun pozzetto di un pozzo 96 piastra PCR, combinare 3,0 ml di primer forward 100 pM per un dato gene, 3,0 ml di corrispondente 100 pM primer reverse, 24.0 ml di tampone sospensione DNA, e 30,0 ml di 2x dosaggio carico reattivo .
- Vortex questo piatto, rotazione per 10 sec a 500 xg, e conservare a -20 ° C.
- Creare due programmi di cicli termici: RTSTA (trascrizione inversa e l'amplificazione target specifico; Tabella 1) e EXOI (esonucleasi trattamento che ho; tabella 2).
2. Trattamento
- Piastra le cellule di mammifero su un piatto di coltura tissutale, che cresceranno alla confluenza desiderata al momento del trattamento, a seconda delle condizioni dello studio sperimentale. Per l'esempio presentato in questo studio, piastra 4 x 10 5 cellule MCF-7 p53-Venus 18 per sei centimetri piatto in RPMI con siero 10% bovino fetale (FBS), 100 U / ml di penicillina, 100 mg / ml di streptomicina, e 250 ng / ml amfotericina B. Incubare le cellule per due giorni a 37 ° C con 5% di CO 2 fino a raggiungere il 50% di confluenza.
- Trattare le cellule per stabilire la condizione di interesse basato sullo studio sperimentale. Per l'esempio presentato in questo studio, incubare cellule p53-Venus MCF-7 con 400 ng / ml neocarzinostatina per 3 ore, 8,5 ore, 14 ore o 24 ore.
3. Lysis Buffer Preparazione per Cell Sorting
NOTA: Fare un singolo piatto di tampone di lisi dura circa 1 ora. Si consiglia di effettuare e piastre sorta multipla, come l'ordinamento delle cellule può essere inefficiente e produrre molti pozzi senza cellule rilevabile.
- Pulire la panchina, pipette, cremagliera del tubo, freddi portatarga PCR, e guanti con la soluzione decontaminante DNA in preparazione per la PCR.
- Diluire l'inibitore RNAsi al 2,64 U / ml con l'aggiunta di inibitore della RNAsi per priva di nucleasi acqua in una provetta da 1,5 ml.
NOTA: Questo permette inibitore RNAsi da aggiungere al tampone di lisi senza pipettando volumi sub-microlitri. - Rendere tampone di lisi delle cellule combinando i componenti elencati nella Tabella 3.
- Trasferire 92,4 ml di tampone di lisi delle cellule in un tubo separato; questo sarà il tampone di lisi per gli standard ampliconi.
- utilizzando low-binding puntali e provette da microcentrifuga, preparare 200 ml e 500 ml di E. coli DNA diluito a 3,1 pg / ml e 6,2 pg / ml, rispettivamente, con buffer di sospensione del DNA.
- Aggiungere 184,8 ml di 3,1 pg / ml di E. coli DNA al tampone di lisi delle cellule.
NOTA: Il DNA di E. coli vettore serve ad ampliare il campo di applicazione di primer non specifico possibilità vincolanti e quindi ridurre la probabilità che dimerizzazione fondo porterà ad un prodotto di PCR. - Preparare gli standard.
- Etichettare sei tubi a bassa vincolante microcentrifuga come 5e5, 5E4, ... 5e0.
- Aggiungere 90 ml di tampone di sospensione DNA alla provetta "5e5".
- Aggiungere 90 ml di 6,2 pg / ml di E. coli DNA a ciascuno degli altri cinque tubi. Mantenere tutte le provette su ghiaccio.
NOTA: Incorporando DNA vettore nelle norme rende la massa totale di DNA nei pozzetti standard, paragonabile a quella nei pozzetti 1-cellulari; ciò riduce la probability che la concentrazione degli standard sarà influenzato da legame al DNA di tubi o punte per pipette. - Prelevare un 'aliquota dello standard "5e6" (5 x 10 6 molecole / ml di ogni amplicone, preparata nella fase 1.4) dal deposito. Vortex brevemente e ruotare brevemente (5 sec a 500 xg) per garantire che il liquido si trovi sul fondo della provetta.
- Utilizzando una punta bassa vincolante pipetta, aggiungere 10 ml di standard di 5e6 per il tubo "5e5". Vortex brevemente, rotazione (5 sec a 500 xg), e mettere il tubo di nuovo sul ghiaccio.
- Pipettare 10 microlitri dal tubo "5e5" nel tubo "5E4". Vortex brevemente, rotazione (5 sec a 500 xg), e mettere di nuovo sul ghiaccio.
- Ripetere queste diluizioni seriali, con ogni tubo ricevente 10 microlitri dal tubo precedente, fino a quando tutti i tubi hanno una diluizione di standard.
- Preparare una fila di 12 provette PCR con tappi. Distribuire 67 ml di tampone di lisi "cella" ad ogni provetta. Tappare le provette e centrifugare brevemente (5sec a 500 xg) se necessario.
- Usando una pipetta a 12 canali, distribuire 9 ml di tampone di lisi "cella" dalla fila di provette per PCR in ciascun pozzetto delle prime 7 righe di una piastra PCR (Figura 3).
- Distribuire 7 ml di tampone di lisi "standard" in ciascun pozzetto dell'ultima riga della piastra (Figura 3).
- Usando le punte basse vincolante pipetta, aggiungere 2 ml di standard per ciascun pozzetto della fila inferiore secondo la mappa piastra (Figura 3).
NOTA: 2 pl di 5 x 10 0 molecole / ml importi standard a 10 molecole, ecc - Aggiungere 2 ml di 3,1 pg / ml di E. coli DNA nei pozzetti 10-cellula e cellula-100; aggiungere 2 ml di 6,2 pg / ml di E. coli DNA nei pozzetti non-cellulari.
NOTA: Ogni 1-celle, 10 celle, e 100 cellule ben ha 6.2 pg di E. coli DNA, approssimare la massa di DNA genomico in una singola cellula umana. Ogni pozzetto di serie e non-cella ha 12.4 pg of E. coli DNA, del valore di circa due cellule umane. - Sigillare la piastra con un sigillo termico sterili, rotazione per 5 secondi a 500 xg, e conservare a 4 ° C fino a quando le cellule sono pronte per l'ordinamento.
4. Classificatore celle Setup
- Programmare il cell sorter fluorescenza attivato per ordinare in una piastra a 96 pozzetti in base alla mappa piastra (figura 3); non le cellule devono essere ordinati in pozzi H1-H11 H8 o-H12.
- Verificare che il cell sorter è ben mirato per l'ordinamento in un piatto PCR.
- Impostare l'angolo della corrente sort al minimo possibile; questa impostazione aumenta la possibilità che le cellule saranno ordinati in fondo dei pozzi, piuttosto che ha colpito i lati.
- Per dirigere la macchina, sigillare un piatto vuoto PCR e usare il flusso di test per "sort" goccioline di PBS sulla guarnizione del piatto vuoto, secondo le istruzioni della macchina 19; le goccioline dovrebbero atterrare su °e la superficie della guarnizione in una posizione sopra il centro del pozzo. Per i migliori risultati, verificare l'obiettivo tutta la lunghezza e la larghezza della piastra.
- Se le goccioline non atterrano nella posizione corretta, li pulire la superficie del sigillo, ricalibrare il cell sorter secondo le istruzioni del costruttore della macchina, e ripetere fino a quando le goccioline nel flusso tipo sono depositati in modo corretto.
5. cellulare Harvest e ordinamento
- Trypsinize le celle come appropriati per la linea cellulare. Ad esempio, per cellule MCF-7, aspirare il terreno dalle cellule, lavare le cellule con 1 ml di 0,05% tripsina, e incubare le cellule con 2 ml di 0,05% tripsina per 5 min a 37 ° C e 5% CO 2 .
- Aggiungere 8 ml di terreno di coltura cellulare alle cellule trypsinized e trasferire tutti i 10 ml di sospensione cellulare in una provetta da 15 ml. Centrifugare per 4 min a 400 x g.
- Aspirare il surnatante e risospendere il pellet cellulare in PBS + 2% FBS.
Nota: Un volume risospensione più piccolo concentra le cellule e facilita cellule di smistamento; un volume risospensione 500 microlitri funziona bene per un sei centimetri piatto di cellule al 50% di confluenza. - Trasferire la sospensione cellulare ad un 5 ml citometria a flusso tubo, di dispensazione attraverso un filtro 40 micron per rimuovere grumi di cellule.
- Centrifugare la piastra di tampone di lisi a 400 xg per 30 sec a 4 ° C prima di classificare le cellule per garantire che il liquido è al fondo dei pozzetti.
- Inserire il tubo con la sospensione cellulare in cell sorter. Aprire il sigillo sulla piastra di tampone di lisi e ordinare con attenzione le cellule di interesse nella piastra secondo la mappa piastra e seguendo le istruzioni 19 del produttore cell sorter. Per garantire che le singole cellule sono ordinati alla massima purezza, far funzionare la macchina in modalità "singola cellula" e utilizzare citometria di flusso standard gating sulla base avanti e scatter laterale 19.
NOTA: Nellaesperimento di esempio, porta le cellule in base p53-Venus fluorescenza per isolare il più luminoso 15% delle singole celle 11. - Sigillare la piastra con una nuova, sterile tenuta termica e centrifugare a 400 xg per 1 min a 4 ° C. Porre la piastra in termociclatore ed eseguire il programma RTSTA (vedi punto 1.7).
NOTA: Esegue trascrizione inversa sul mRNA dalle cellule ordinati e quindi amplifica il cDNA delle trascrizioni di interesse.
6. Il trattamento esonucleasi
- Mescola diluita esonucleasi I combinando i componenti elencati nella Tabella 4. Preparare una fila di 12 provette PCR con tappi. Distribuire 30,5 ml di diluito esonucleasi I in ogni provetta. Tappare le provette e ruotare brevemente (5 sec a 500 xg) per assicurare che il liquido si trovi sul fondo della provetta.
- Quando il RTSTA è finito, girare la piastra di campione per 5 secondi a 500 x g. Usando una pipetta a 12 canali, distribuire 3,6 microlitri di esonucleasi diluitaI in ciascun pozzetto della piastra.
- Sigillare la piastra con un sigillo termico sterile e di spin brevemente (5 sec a 500 xg). Porre la piastra in termociclatore ed eseguire il programma EXOI (vedi punto 1.7).
NOTA: Questo passaggio degrada alcun primer specifico rimanenti dopo l'amplificazione di destinazione in modo che non interferiscano con il futuro PCR.
Diluizione 7. Campione
- Quando il trattamento è terminato esonucleasi, aggiungere 32,4 ml di tampone TE a ciascun pozzetto della piastra campione; questo è un punto di arresto opzionale, ed i campioni possono essere conservati a -20 ° C per diversi giorni.
8. Sort controllo della qualità utilizzando qPCR
NOTA: Poiché selezione cellulare non è perfettamente efficiente, questo passaggio è necessario individuare quali i pozzetti della piastra ordinata effettivamente ricevuto una cella. Questi campioni possono poi essere utilizzati per ulteriori analisi.
- Misurare l'espressione genica di pulizia in ogni campione da qPCR.
- Preparare 900 ml di legame al DNA colorante qPCR master mix per 96 reazioni secondo il protocollo utilizzando primer del produttore della polimerasi per uno dei geni housekeeping selezionati al punto 1.1; le concentrazioni esatte di tampone di reazione, polimerasi, primer, ecc per questa reazione dipenderà dalla polimerasi specifica in uso.
- Distribuire 9 ml di master mix per ogni pozzetto di una piastra a 96 pozzetti PCR. Aggiungere 1 ml di campione dalla piastra del campione al corrispondente pozzetto della piastra PCR.
- Eseguire la piastra su una macchina di PCR in tempo reale utilizzando un protocollo cicli termici suggerito dal produttore Master Mix o in seguito ad un protocollo standard 20.
- Analizzare i dati qPCR 20; un esempio di qPCR dati dai ordinamento procedura di controllo della qualità elencate di seguito è mostrata in figura 4.
- Verificare che le misurazioni da pozzi non-cellule mostrano un livello (di fondo) a bassadell'espressione genica o nessuna espressione.
- Verificare che le misurazioni da pozzi 1-cellule si dividono nettamente in due gruppi: i campioni positivi con elevata espressione genica e di campioni negativi con i livelli di fondo di espressione o nessuna espressione a tutti, in modo simile ai pozzetti non-cellulari.
NOTA: I campioni con elevata espressione genica rappresentano cellule effettive ordinati; quelli con l'espressione di fondo dovrebbero essere esclusi da ulteriori analisi. - Verificare che le misurazioni dai pozzetti 10-cellula e cellula-100 riflettono genica superiore nei pozzetti 1-cellule positive, consentendo la possibilità che ordinamento inefficiente potrebbe causare quei pozzetti di avere meno cellule di quanto desiderato.
- Verificare che le misurazioni dai pozzetti standard sono distribuiti uniformemente nello spazio logaritmica (vale a dire, che i valori t C sono distribuiti in modo uniforme).
- Dopo aver identificato tutti i campioni 1-cellule positive, fare una mappa piatto "Sample Mixes" per mappare positivo 1-cell campioni su una nuova piastra a 96 pozzetti. Include otto pozzi per gli standard di questo piatto mappa. Vedere Figura 5 per un esempio.
Misurazione Espressione genica 9. Usando Microfluidic qPCR
NOTA: Per ogni passo in questa sezione, pipetta solo al primo stop per minimizzare la formazione di bolle nei reagenti.
- Aprire una fila di 8 provette PCR. Se ci sono più piatti di campioni, aprire una seconda fila di 8 tubi per PCR ed etichettare i tubi 1-8. Questa riga sarà per mettere in comune gli standard da più piatti.
- In una provetta da 1,5 ml, mescolare 360 ml di legame al DNA colorante qPCR Master Mix con 36 ml di legame al DNA del campione colorante carico reattivo. Vortex brevemente e girare per 10 secondi a 2.000 x g. Dividere questa miscela (396 ml) nella fila senza etichetta di 8 provette PCR, ponendo 48 ml in ciascun tubo.
- L'utilizzo di un pipetta a 8 canali, distribuire 3,3 microlitri della miscela dei reagenti carico Master Mix / campione in ciascun pozzettodi un nuovo 96 pozzetti PCR. Etichettare questo piatto, "Sample Mixes".
- Per ogni piastra PCR di campioni:
- Vortex la piastra per 10 secondi e girare per 10 secondi a 500 x g. Rimuovere il coperchio e trasferire 2,7 ml di ogni campione 1-cellule positive al corrispondente bene sul campione Miscele piastra per la 1-cellule pozzi indicate sulla mappa piatto.
- Se c'è solo un piatto di campioni, trasferire 2,7 ml di ciascuno standard amplificato al corrispondente bene sul campione miscele piastra secondo la mappa piatto.
- Se ci sono più piatti di campioni, trasferire 2,7 ml di ogni standard amplificato nella sua posizione corrispondente nella riga etichettata di provette PCR. Aggiungere standard dalla piastra corrente di campioni da qualsiasi standard già presenti. Sigillare la piastra di campioni quando fatto utilizzando una pellicola sigillante.
- Se ci sono più piatti di campioni:
- Agitare la fila di tubi PCR contenenti norme per10 sec e girare per 10 secondi a 2.000 x g. Trasferire 2,7 ml di ciascun tubo di standard misti nella corrispondente bene sul campione mescola piatto secondo la mappa piatto.
- Caricare i pozzi NTC con 2,7 ml di acqua priva di nucleasi dove indicato sulla mappa piatto. Sigillare il campione Mixes piatto e conservare a 4 ° C fino al momento dell'uso.
- Sbucciate l'adesivo dal fondo di un nuovo chip microfluidica qPCR. Utilizzando una siringa di liquido linea di controllo con il tappo ancora, spingere verso il basso ogni primavera accumulatore per allentarla, e iniettare ogni accumulatore con 150-200 ml di liquido linea di controllo.
- Inserire il chip nella macchina di carico ed eseguire lo script "Prime". Questo richiede ~ 20 min.
- Quando adescamento è fatto, vortice e spin giù per le tavole di miscele test (preparato nel passo 1.6) e miscele di esempio.
- Usando una pipetta a 8 canali, trasferire 4,5 ml di ciascuna miscela test nel lato sinistro del chip microfluidico qPCR secondo lo schema in g> Figura 6. Pipetta molta attenzione per evitare la creazione di bolle d'aria nella parte inferiore dei pozzetti. Al termine, sigillare la piastra di miscele di analisi e conservare a -20 ° C per un uso futuro.
- Trasferire 4,5 ml di ciascuna miscela di esempio sul lato destro del chip microfluidico qPCR di figura 6. Inserire il chip nella macchina di carico ed eseguire lo script "Load Mix". Questo richiede ~ 90 min.
- Accendere il real-time PCR macchina microfluidica, avviare il software di raccolta dati, e accendere la lampada per riscaldarlo. Questo richiede ~ 20 min.
- Quando lo script Load Mix è fatto, verificare che non vi siano linee attraverso il chip-questo indicherebbe un problema di caricamento. Rimuovere con cautela eventuali particelle di polvere dal chip utilizzando scotch o nastro di laboratorio. Inserire il chip in real-time PCR macchina microfluidica ed eseguire lo script di raccolta dei dati. Utilizzare il software di analisi per controllare i risultati ed esportare i dati per ulteriori analisi.
- Rimuovere le misurazioni per cui le curve di amplificazione qPCR mostrano scarsa qualità (cioè, per cui le curve non assomigliano alla curva standard di amplificazione sigmoidale) 20.
- Rimuovere campioni con più di 30 scarsa qualità o nulli misurazioni o per il quale l'espressione gene housekeeping (vedi passo 1.1) è molto inferiore rispetto alla maggior parte degli altri campioni (Figura 7).
- Per ogni gene, stimare la posizione melt picco preciso la mediana dei luoghi di picco melt da ciascun campione. Se una misura ha un picco di fusione si trova più di 1 ° C dalla mediana, rimuovere tale misura dalla considerazione 20.
- Per ogni gene, creare una curva standard per stimare il numero di mRNA in ciascun campione utilizzando un linguaggio di scripting foglio di calcolo o 20.
NOTA: Le norme in questo esperimento sono costituiti da 10, 100, 1.000, 10.000 o molecoledi dsDNA corrispondente ad ogni trascrizione di interesse. Se trascrizione inversa fosse perfettamente efficiente, le norme si corrispondono a 20, 200, 2000, e 20.000 molecole di mRNA, poiché mRNA e cDNA sono a singolo filamento. In pratica, questo non è il caso, in modo da esprimere misurazioni quale stima in unità di molecole × RT E, dove E RT è il rendimento di trascrizione inversa.- Per ciascun pozzetto standard identificare m, il numero di molecole di ssDNA (due volte il numero di molecole di dsDNA) prima dell'amplificazione (ad esempio, 20 molecole, 200 molecole, ecc).
- Per ogni ben di serie, identificare il valore t C da qPCR 20.
- Con i valori di m e C t, eseguire una regressione lineare utilizzando la seguente equazione per generare una curva standard con parametri b per ogni gene, e trovare il valore R 2 comeIndicatore n della bontà di adattamento:
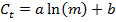
- Dalla curva standard, calcolare l'efficienza della PCR E per ogni gene:
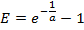
NOTA: Un valore di E molto maggiore di 1,0 (per esempio, E> 1.1) non è fisicamente realistica; un valore R 2 per la regressione lineare molto meno di 1,0 (per esempio, R 2 <0,985) significa che gli standard non funzionano in modo affidabile. Questi problemi generalmente indicano che il livello più basso amplicone scende sotto una soglia di quantificazione affidabile, e quindi il livello più basso deve essere escluso dalla regressione lineare. Se una regressione lineare adeguata non può essere effettuata utilizzando almeno tre standard unici, scartare le misurazioni di espressione per il gene. - Se la regressione lineare è adeguata, usare la risultante parametri atti a e b </ em> per ogni gene per stimare il numero di mRNA molecole m est (in unità di molecole × E RT) per il gene corrispondente a ciascun campione singola cella dal valore t C misurato:
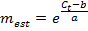
- Designare qualsiasi misura m est <1 come m est = 0. Inoltre, designare qualsiasi misura con nessuna amplificazione in qPCR e quindi nessun valore t C come m est = 0.
NOTA: Le misurazioni con un m est minore del più basso o superiore al più alto standard utilizzato nella regressione sono trovati per estrapolazione dalla curva standard. Queste stime non sono necessariamente valida, ma devono essere considerati con cautela.
- Visualizzare i dati di espressione genica unicellulari.
- Fare una trama beeswarm (Dorn, J. e S., T., 2012, http://www.mathworks.com/matlabcentral/fileexchange / 37105), in cui ciascun punto rappresenta l'espressione genica in una determinata cella; un esempio è mostrato in figura 8.
- Effettuare una trama violino (Dorn, J., 2012, http://www.mathworks.com/matlabcentral/fileexchange/23661) in cui la larghezza della "violino" rappresenta la frequenza delle misurazioni di espressione genica attorno ad un particolare livello nel popolazione di cellule analizzate; un esempio è mostrato in figura 8.
NOTA: le analisi più sofisticate possono sondare i rapporti di espressione tra coppie di geni 21, 22 e possono dedurre le reti di espressione genica.
Risultati
Una panoramica generale del protocollo è mostrato in Figura 1, compresi i passi per il trattamento della cellula, l'isolamento di singole cellule mediante FACS, la generazione e pre-amplificazione di librerie di cDNA da lisati monocellulari la conferma di librerie di cDNA monocellulari in pozzi ordinati, e la misura dell'espressione genica da qPCR.
In preparazione per l'isolamento cella singola e ...
Discussione
Abbiamo presentato un metodo per isolare le singole cellule di mammifero da una popolazione di cellule aderenti in coltura e per saggiare l'espressione di circa 96 geni in ogni cellula. Una buona preparazione anticipo è fondamentale per questo metodo di lavorare bene. In particolare, progettazione e collaudo di primer coppie specifiche per le trascrizioni di interesse (passi 1.2-1.3) sono in termini di tempo, ma importanti passi, come primer determinano la qualità delle misurazioni unicellulari. Una volta coppie d...
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
Vorremmo ringraziare V. Kapoor nel RCC ETIB Citometria a flusso core per il suo aiuto nello svolgere l'ordinamento delle cellule durante lo sviluppo di questo protocollo. Ringraziamo anche M. Raffeld e l'Unità CCR LP diagnostica molecolare e J. Zhu e l'NHLBI sequenziamento del DNA e Genomica core per il loro aiuto nello svolgimento della qPCR durante lo sviluppo di questo protocollo. Questa ricerca è stata sostenuta dal Programma intramurale del NIH.
Materiali
| Name | Company | Catalog Number | Comments |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| High Capacity cDNA Reverse Transcription Kit with RNase Inhibitor | ThermoFisher | 4374966 | |
| Phusion High-Fidelity DNA Polymerase | New England BioLabs | M0530S | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| Quant-iT High-Sensitivity dsDNA Assay Kit | ThermoFisher | Q33120 | |
| 2.0 ml low adhesion microcentrifuge tubes | USA Scientific | 1420-2600 | |
| DNA Suspension Buffer | Teknova | T0221 | |
| Axygen 0.2 ml Maxymum Recovery Thin Wall PCR Tubes | Corning | PCR-02-L-C | |
| GE 96.96 Dynamic Array DNA Binding Dye Sample & Assay Loading Reagent Kit | Fluidigm | 100-3415 | |
| HyClone RPMI 1640 media | GE Healthcare Life Sciences | SH30027.01 | |
| Fetal Bovine Serum, Certified (US) | ThermoFisher | 16000-044 | |
| Antibiotic-Antimycotic Solution | Corning | 30-004-CI | |
| Neocarzinostatin | Sigma | N9162 | |
| ELIMINase | Decon Labs | 1101 | |
| SUPERase-In | ThermoFisher | AM2696 | |
| CellsDirect One-Step qRT-PCR Kit | ThermoFisher | 11753500 | |
| E. coli DNA | Affymetrix | 14380 10 MG | |
| ThermalSeal Sealing Film, Sterile | Excel Scientific | STR-THER-PLT | |
| BD FACSAria IIu | BD Biosciences | ||
| HyClone Trypsin 0.05% | GE Healthcare Life Sciences | SH30236.01 | |
| PBS, 1x | Corning | 21-040-CV | |
| Falcon 40 µm Cell Strainer | Corning | 352340 | |
| Exonuclease I | New England BioLabs | M0293S | |
| SsoFast EvaGreen Supermix with Low ROX | Bio-Rad | 172-5210 | |
| 96.96 Dynamic Array IFC for Gene Expression (microfluidic qPCR chip) | Fluidigm | BMK-M-96.96 | |
| IFC Controller HX (loading machine) | Fluidigm | ||
| BioMark or BioMark HD (microfluidic qPCR machine) | Fluidigm | ||
| Real-Time PCR Analysis software | Fluidigm | ||
| MATLAB software | MathWorks |
Riferimenti
- Feinerman, O., Veiga, J., Dorfman, J. R., Germain, R. N., Altan-Bonnet, G. Variability and Robustness in T Cell Activation from Regulated Heterogeneity in Protein Levels. Science. 321 (5892), 1081-1084 (2008).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- Geva-Zatorsky, N., Rosenfeld, N., et al. Oscillations and variability in the p53 system. Mol. Syst. Biol. 2 (1), (2006).
- Colman-Lerner, A., Gordon, A., et al. Regulated cell-to-cell variation in a cell-fate decision system. Nature. 437 (7059), 699-706 (2005).
- Chong, S., Chen, C., Ge, H., Xie, X. S. Mechanism of Transcriptional Bursting in Bacteria. Cell. 158 (2), 314-326 (2014).
- Raj, A., Peskin, C. S., Tranchina, D., Vargas, D. Y., Tyagi, S. Stochastic mRNA Synthesis in Mammalian Cells. PLoS Biol. 4 (10), e309+ (2006).
- Senecal, A., Munsky, B., et al. Transcription Factors Modulate c-Fos Transcriptional Bursts. Cell Rep. 8 (1), 75-83 (2014).
- Dey, S. S., Foley, J. E., Limsirichai, P., Schaffer, D. V., Arkin, A. P. Orthogonal control of expression mean and variance by epigenetic features at different genomic loci. Mol. Syst. Biol. 11 (5), 806+ (2015).
- Dar, R. D., Razooky, B. S., et al. Transcriptional burst frequency and burst size are equally modulated across the human genome. Proc. Natl. Acad. Sci. U.S.A. 109 (43), 17454-17459 (2012).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- Porter, J. R., Fisher, B. E., Batchelor, E. p53 Pulses Diversify Target Gene Expression Dynamics in an mRNA Half-Life-Dependent Manner and Delineate Co-regulated Target Gene Subnetworks. Cell Syst. 2 (4), 272-282 (2016).
- Lahav, G., Rosenfeld, N., et al. Dynamics of the p53-Mdm2 feedback loop in individual cells. Nat. Genet. 36 (2), 147-150 (2004).
- Levine, A. J., Oren, M. The first 30 years of p53: growing ever more complex. Nat. Rev. Cancer. 9 (10), 749-758 (2009).
- Riley, T., Sontag, E., Chen, P., Levine, A. Transcriptional control of human p53-regulated genes. Nat. Rev. Mol. Cell Biol. 9 (5), 402-412 (2008).
- Ye, J., Coulouris, G., Zaretskaya, I., Cutcutache, I., Rozen, S., Madden, T. L. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinf. 13 (1), 134 (2012).
- . . PCR Technologies: A Technical Guide. , (2014).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Batchelor, E., Mock, C. S., Bhan, I., Loewer, A., Lahav, G. Recurrent initiation: a mechanism for triggering p53 pulses in response to DNA damage. Mol. Cell. 30 (3), 277-289 (2008).
- . . Flow Cytometry: Principles and Applications. , (2007).
- . . Real-time PCR. , (2006).
- Song, L., Langfelder, P., Horvath, S. Comparison of co-expression measures: mutual information, correlation, and model based indices. BMC Bioinf. 13 (1), 328 (2012).
- Margolin, A. A., Nemenman, I., et al. ARACNE: An Algorithm for the Reconstruction of Gene Regulatory Networks in a Mammalian Cellular Context. BMC Bioinf. 7 (Suppl 1), (2006).
- Haff, L. A. Improved quantitative PCR using nested primers. PCR Methods Appl. 3 (6), 332-337 (1994).
- Hashimshony, T., Senderovich, N., et al. CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome Biol. 17, 77 (2016).
- Ronander, E., Bengtsson, D. C., Joergensen, L., Jensen, A. T. R., Arnot, D. E. Analysis of Single-cell Gene Transcription by RNA Fluorescent In Situ Hybridization (FISH). J. Vis. Exp. (68), e4073 (2012).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat. Methods. 5 (10), 877-879 (2008).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nat. Methods. 9 (7), 743-748 (2012).
- Battich, N., Stoeger, T., Pelkmans, L. Image-based transcriptomics in thousands of single human cells at single-molecule resolution. Nat. Methods. 10 (11), 1127-1133 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon