需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
用数字全息显微镜 (DHM) 定量化低浓度微生物
摘要
数字全息显微镜 (DHM) 是一种体积技术, 允许成像样品50-100X 厚比明显微镜在可比的分辨率, 与重点进行后处理。DHM 用于识别、计数和跟踪非常低密度的微生物, 并与光学密度测量、平板计数和直接计数相比较。
摘要
准确地检测和计数稀疏细菌样品在食品、饮料和制药加工业、医疗诊断和机器人任务到太阳系其他行星和卫星的生命检测中有许多应用。目前, 稀疏细菌样品通过培养电镀或荧光显微镜进行计数。培养皿需要很长的孵育时间 (天到周), 荧光显微镜需要广泛的染色和样品的浓度。在这里, 我们演示如何使用离轴数字全息显微镜 (DHM) 在非常稀的文化中列举细菌 (100-104细胞/毫升)。首先, 讨论了定制 DHM 的构建, 并详细说明了如何构建低成本的仪器。讨论了全息术的原理, 根据仪器的光学性能特点和细菌溶液浓度 (表 2), 利用统计模型估计视频检测细胞的时间长度。.视频检测的细胞在 105, 104, 103, 和100细胞/毫升是实时演示使用 un-reconstructed 全息图。利用开源软件包对振幅和相位图像进行重建。
引言
在非常稀的样品中准确的细菌计数的测定在许多应用中是至关重要的: 几个例子是水和食品质量分析1,2,3;检测血液、脑脊液或痰液中的病原体4,5;生产医药产品, 包括无菌水6;和环境群落分析在营养环境中如开阔的海洋和沉积物7,8,9。在木星和土星的冰冷卫星上, 特别是木卫二10、11和土卫二12、13、 14, 已知有地下液态海洋。由于维京人在1978年试图在另一个星球上寻找现存的生命, 因此没有任何任务, 因此在空间飞行任务的过程中, 细菌识别和计数的技术和仪器的开发是有限的15。
传统的平板计数方法只发现培养细胞, 它能在环境菌株中代表少数物种, 有时和 #60; 1%16。盘子需要几天或几周的孵化来获得最大的成功, 这取决于应变。荧光显微镜在很大程度上取代了板数作为快速准确的微生物计数的金标准。核酸标记荧光染料, 如 4, 6-diamidino-2-吲盐酸盐 (DAPI), SYBR 绿色, 或吖啶橙绑定到核酸是典型的染料使用17,18,19, 虽然许多研究使用了克标志的荧光指标20,21,22,23,24。使用这些没有预步骤的方法, 每个 mL 会导致检测 (检测) 105单元格的限制。改进的 LoD 是可能的使用过滤。液体样品是真空过滤到膜上, 通常是聚碳酸酯和理想的黑色, 以减少背景。低背景染料, 如上面提到的 DNA 污渍, 可以直接应用于过滤器25。为了准确地按眼睛计数, 每个过滤器需要 105单元格, 这意味着对于每个 mL 的样本比 105单元更稀, 必须收集和筛选大量的样本卷。为了系统地探索过滤器的所有区域, 并因此减少了计数所需的单元数, 将检测范围推到每 mL26102单元格, 从而开发了激光扫描设备。然而, 这些在大多数实验室是不可用的, 并且需要先进的硬件和软件, 允许专家确认观察到的粒子是细菌而不是碎片。
作为参考, 成人脓毒症通常开始显示的症状和 #60; 100 细胞/毫升的血液, 和婴儿在和 #60; 10 细胞/毫升。从一个成年人的血液提取需要10毫升, 从一个婴儿, 1 毫升。pcr 方法被存在人和致病植物群脱氧核糖核酸和由 PCR 抑制的组分抑制在血液27,28。尽管有各种新兴技术, 文化仍然是诊断血流感染的金标准, 特别是在更多的农村地区或发展中国家。为了探测其他行星上的生命, 热力学计算可以估计生命的能量预算, 从而预测可能的生物量。1-100 细胞/毫升预计将热力合理的欧罗巴29。从这些数字可以很容易地看出, 大量的水溶液中的少量细胞的检测是一个重要的未决问题。
在本文中, 我们演示了检测沙雷菌和希瓦 oneidensis (野生类型和 non-motile 变种) 的浓度为 105, 104, 103, 和100细胞/毫升使用离轴数字全息显微镜 (DHM)。DHM 优于传统光学显微镜的关键优势是在高-的同时成像厚样本体积, 该实现, 样品室是0.8 毫米厚。这些样品室由烷 (soft-lithography) 由精密机械加工的铝模制成, 公差为±50µm。这代表大约100倍的改善在领域的深度在大功率光学显微镜。DHM 还提供了定量的相位信息, 允许测量光路长度 (折射率和厚度的乘积)。DHM 和类似的技术已用于监测细菌和酵母细胞周期和细菌干质量的计算30,31,32;散射差异甚至可以用来区分细菌菌株33。
我们使用的仪器是 custom-built 专门用于微生物, 如以前发布的34,35, 它的设计和构造是演示和讨论的。通过注射器泵连续向0.25 µL 容积样品室提供水溶液;流率是由相机帧率确定的, 以确保整个样本量的成像。统计计算预测的样本量必须被成像, 以便检测到一个相当数量的细胞在一定浓度。
对于细胞检测应用, 不需要将全息图重建为振幅和相位图像;对原始全息图进行了分析。这节省了大量的计算资源和磁盘空间: 重建时, 500 Mb 的全息图视频将是 1-2 Tb。然而, 我们确实讨论了通过样品的深度重建, 以确认全息图代表了所需的物种。DHM 的一个重要特点是它能够监测图像的强度和相位。在强度上几乎是透明的生物体 (如大多数生物细胞) 在相时清晰地显现出来。由于它是一种无标签的技术, 没有使用染料。这是一个dvantage 为可能的太空飞行应用, 因为染料可能不生存使命的条件, 并且-更重要地-不能假设与外星有机体一起使用, 可能不用脱氧核糖核酸或 RNA 为编码。这也是在诸如北极和南极等极端环境中工作的一个优势, 在那里, 染料可能难以带到偏远的地方, 并可能在储存时降解。图像的相位和振幅重建是使用一个开源软件包, 我们已经提供了 GitHub (洗发水) 或使用 ImageJ。
研究方案
1. 细菌的生长和计数
注意: 这适用于在适当培养基中生长的几乎任何细菌菌株 36 。在我们的例子中, 我们使用三菌株: 沙雷菌 作为一个常见的、容易识别的实验室菌株;和一个较小的, 高度能动的环境应变, 希瓦 oneidensis MR-1。为了比较检测的运动与 non-motile 细胞, non-motile 希瓦 突变体, 和 #916; FlgM, 也用于比较 37 。所有菌株都生长在性肉汤 (LB).
- 准备无菌 LB 培养基 (每公升蒸馏水:10 克 bacto 胰, 5 克 bacto 酵母, 10 克氯化钠; 过滤器或高压釜)。准备标准的100毫米直径 LB 琼脂板 (相同的配方, 中等加 15 g 琼脂每升; 蒸压釜 (121, #176; C, 20 分钟), 以杀菌和倒入时, 冷却足够处理)。在冰箱中存放介质和盘子.
- 试验前的前一天 5-6 毫升的 #34; 硕士和 #34; 培养基从新鲜细菌菌落, 使用正确的无菌和生物安全技术。在30和 #176 的振动器上孵育 (120 rpm); C 为 ~ 12 h. 潜伏期将取决于应变和生长速率。在我们的实验中, 希瓦 菌株孵育12小时;但是, 沙雷 在8小时后收获, 以确保文化将显示 mid-logarithmic 的增长.
- 实验的当天, 采取分光光度读数 (OD 600 ) 的细菌的主文化, 这是预计的范围0.6 至0.7。它应该在对数增长阶段 (如先前所确定)。如果不是, 亚文化100和 #181; L 到5毫升培养基, 并允许细胞生长到 mid-log 阶段.
- 采用主区域性样本, 并使用 Petroff-豪塞尔计数室直接计数单元格。这将枚举活的和死的细胞.
- 提取10和 #181; 未稀释的文化的样品与微和转移入房间.
- 图像下的 high-dry 物镜显微镜 (40X 和 #160; 或 63X, NA 0.7-0.8) 使用相衬.
- 统计至少20平方和平均值的细菌数.
注: 只计算那些完全在一个正方形内的细菌, 只有那些越过顶部和左边的边界 (如果你愿意的话)。如果正方形由几行分隔, 请选择一个作为边界. - 计算浓度为20平方 x 稀释因子的平均值。注意, 直接计数对浓度和 #60 不起作用; 10 7 /mL.
- 用于对菌落形成单元 (CFU) 计数的区域性进行串行稀释。这将仅枚举活动单元格.
- 将每个选定的细菌样本与无菌0.9% 盐水溶液进行连续稀释。转移20和 #181; l 的细菌溶液和稀释它与180和 #181; l 盐水。重复直到最低浓度为 ~ 10 3 单元格/mL.
- 采取100和 #181; L 从至少两个稀释建议是 10 3 和 10 4 /mL 和 #160; #8212; 在适当的固体介质板上板。与无菌摊铺机。执行每次稀释的至少3复制.
- 在适当的温度下孵育细菌过夜或直到菌落生长.
- 计数殖民地和计算殖民地形成单位 (CFU) 根据 (# 的殖民地 x 稀释因子)/体积镀 = CFU/毫升。对复制的平均 CFU.
2。DHM 的高稀释样品的制备
- 在 LB 介质板上为 DHM 和后 DHM 计数的菌落形成单元稀释的主区域性的序列 (见 1.4.1-1.4.4)。执行这双盲, 使录音的人不知道每个测量样本的浓度.
- 将细菌稀释到 10-15 毫升的最小培养基中, 这将鼓励运动 (视情况而定), 但抑制细胞分裂, 以便在实验过程中细胞的浓度不会有明显变化。这可能是0.9% 盐水或更具体的运动介质。例如, 如果使用 大肠杆菌 , 则运动介质必须包含 EDTA 38 。对于这些例子, 我们使用0.9% 盐水.
3。录制 DHM 视频
- 使用无菌注射器, 抽取约10毫升的稀释利息.
- 使用无菌管件和油管将注射器连接到 DHM 样品室.
注: custom-made 微流控室是为了使样品的一致流动通过仪器的光学通路而建造的。有关详细信息, 请参见 结果 部分。在实验之前, 样品室的所有组分应该由灭菌消毒. - 从注射器通过样品室源源不断地使用注射器泵.
注: 适当的流速取决于射流通道的尺寸、所需的吞吐量以及数据获取的限制. - 当样品流经样品室时, 以适当的帧速率连续获取全息图。将创建一个时间文件, 记录每个图像捕获的时间, 以便以后在数据分析期间使用 (协议4节)。获取所有全息图在环境温度 (23 和 #176; C)。通过执行由照相机制造商提供的预先 c++ 可执行文件来记录全息图.
注: 适当的帧速率根据选择的流速而变化。对于这个实验, 建议使用一个帧速率, 这样细菌就会被成像和 #62; 2 倍, 因为它横跨仪器和 #39; s 的视野. - 允许足够的时间让整个10毫升的样品流经 DHM.
- 为了确保在实验过程中细菌不会生长或死亡, 用100和 #181 对培养基中的富集进行接种; DHM 图像捕获。用无菌的吊具铺上, 并在适当温度下孵育24小时;计数殖民地存在.
4。计算细胞密度和检测限值
- 已获得的全息图视频, 分析数据中是否存在细菌.
- 计算全息图的时间序列的中值像素值, 然后从每个各自的像素中减去此中间数值, 以消除全息图像中的固定工件。这可以通过执行以下步骤在 ImageJ 中完成:
- 转换为32位 (Image-Type-32 位)
- 计算中值 (图像堆栈-Zproject-中值)
- 从堆栈的其余部分减去中间值 (进程-ImageCalculator 减去)
- 计算可见的通风环和焦点单元格的数目手动.在 ImageJ 中, 这是通过用点工具指向对象来完成的.
- 每系列全息图将附有一个时间文件 (在全息图像采集时记录)。使用时间文件计算在捕获图像时抽取的样本总量.
- 计算全息图的时间序列的中值像素值, 然后从每个各自的像素中减去此中间数值, 以消除全息图像中的固定工件。这可以通过执行以下步骤在 ImageJ 中完成:
- 通过将检测到的总体积的单元格总数除以图像的总数来计算单元格的密度。平均 5-10 帧用于精确统计.
5。图像重构到振幅和相位
- 选择每帧 10-200 单元格的代表性视频。装载原始 (不是中位减去) 全息图入重建软件 (例子: ImageJ 数值传播 39 ;洗发水 [GitHub]).
- 使用 ImageJ, 转到 OD-数值传播-数值衍射.
- 在提示时, 绘制一个傅里叶掩码以选择真实或虚拟图像, 并消除任何正弦噪声特征.
- 通过进入重建距离, 在适当的 z 步重建振幅和相位.
- 中值减去振幅数据以减少噪音, 如4.1.1 中所示.
- 将数据绘制为3D 多维数据集或投影。转到插件和 #8212; 卷查看器.
结果
结果应表明, DHM 能在极低的水平上检测活细菌和死菌。计数的细菌数量应与使用 Petroff-豪瑟计数室和钢板计数所得的结果一致。标准统计方法提供了各种细菌浓度不同检测方法的准确性信息。
图 1显示了 Petroff-豪塞尔计数室, 以及用于直接枚举单元格的计数室的显微结构。图 2A显示?...
讨论
全息图的数值重建: 用于全息图的数值重建, 采用了角谱法 (ASM)。这涉及到全息图的卷积与格林函数的 DHM。利用傅里叶卷积定理可以计算出在特定焦平面上的复杂波前, 如下所示:
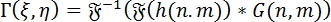 (1)
(1)
其中, 是傅里叶变换运算符, 是全息图矩阵, 是 DHM 的绿色函数, 定义为:
披露声明
作者没有什么可透露的。
致谢
作者承认戈登和贝蒂摩尔基金会授予加州理工学院4037和4038资助这项工作。
材料
| Name | Company | Catalog Number | Comments |
| Bacto Yeast | BD Biosciences | 212750 | |
| Bacto Tryptone | BD Biosciences | 211705 | |
| Sodium chloride | Sigma-Aldrich | 7710 | Many options for purchase |
| Bacto Agar | BD Biosciences | 214010 | |
| 10 cm Petri dishes | VWR | 10053-704 | |
| 15 mL culture tubes | Falcon (Corning Life Sciences) | 352002 | Loose-capped |
| Petroff-Hauser chamber | Electron Microscopy Sciences | 3920 | |
| 10 mL syringes | BD Biosciences | 309604 | Luer-Lok tip not necessary |
| Male Luer to 1/16” barbed fitting | McMaster-Carr | 51525K291 | |
| Autoclavable 1/16” ID PVC tubing for flow | Nalgene | 8000-0004 | Sold by length, purchase accordingly |
| Syringe pump | Harvard Apparatus | PHD 2000 | |
| Sample Chamber | Custom | n/a | See Materials Section |
| Holographic Microscope | Custom | n/a | See Wallace et al. |
| Open-source software | Custom | https://github.com/bmorris3/shampoo |
参考文献
- Akineden, O., Weirich, S., Abdulmawjood, A., Failing, K., Buelte, M. Application of a Fluorescence Microscopy Technique for Detecting Viable Mycobacterium avium ssp. paratuberculosis Cells in Milk. Food Anal. Methods. 8, 499-506 (2015).
- Deibl, J., Paulsen, P., Bauer, F. Rapid enumeration of total aerobic microbial counts on meat and in meat products by means of the direct epifluorescence filter technique. Wien Tierarztl Monat. 85, 327-333 (1998).
- Huang, J., Li, Y., Slavik, M. F., Tao, Y., Huff, G. R. Identification and enumeration of Salmonella on sample slides of poultry carcass wash water using image analysis with fluorescent microscopy. Transactions of the Asae. 42, 267-273 (1999).
- Durtschi, J. D., et al. Increased sensitivity of bacterial detection in cerebrospinal fluid by fluorescent staining on low-fluorescence membrane filters. J Med Microbiol. 54, 843-850 (2005).
- Panicker, R. O., Soman, B., Saini, G., Rajan, J. A Review of Automatic Methods Based on Image Processing Techniques for Tuberculosis Detection from Microscopic Sputum Smear Images. J Med Syst. 40, (2016).
- Riepl, M., et al. Applicability of solid-phase cytometry and epifluorescence microscopy for rapid assessment of the microbiological quality of dialysis water. Nephrol Dial Transplant. 26, 3640-3645 (2011).
- Hwang, C. Y., Cho, B. C. Virus-infected bacteria in oligotrophic open waters of the East Sea, Korea. Aquat Microb Ecol. 30, 1-9 (2002).
- Kepner, R. L., Pratt, J. R. Use of Fluorochromes for Direct Enumeration of Total Bacteria in Environmental-Samples - Past and Present. Microbiol Rev. 58, 603-615 (1994).
- Queric, N. V., Soltwedel, T., Arntz, W. E. Application of a rapid direct viable count method to deep-sea sediment bacteria. J Microbiol Meth. 57, 351-367 (2004).
- Prieto-Ballesteros, O., Vorobyova, E., Parro, V., Rodriguez Manfredi, J. A., Gomez, F. Strategies for detection of putative life on Europa. Adv Space Res. 48, 678-688 (2011).
- Gleeson, D. F., et al. Biosignature Detection at an Arctic Analog to Europa. Astrobiology. 12, 135-150 (2012).
- Carr, C. E., Zuber, M. T., Ruvkun, G., Ieee, . 2013 Ieee Aerospace Conference IEEE Aerospace Conference Proceedings. , (2013).
- Konstantinidis, K., et al. A lander mission to probe subglacial water on Saturn's moon Enceladus for life. Acta Astronautica. 106, 63-89 (2015).
- McKay, C. P., Anbar, A. D., Porco, C., Tsou, P. Follow the Plume: The Habitability of Enceladus. Astrobiology. 14, 352-355 (2014).
- Hoover, R. B., Hoover, R. B., Levin, G. V., Rozanov, A. Y., Wickramasinghe, N. C. . Instruments, Methods, and Missions for Astrobiology Xvii. , (2015).
- Handelsman, J. Metagenomics: application of genomics to uncultured microorganisms. Microbiol Mol Biol Rev: MMBR. 68, 669-685 (2004).
- Lebaron, P., Parthuisot, N., Catala, P. Comparison of blue nucleic acid dyes for flow cytometric enumeration of bacteria in aquatic systems. Appl Environ Microbiol. 64, 1725-1730 (1998).
- Marie, D., Partensky, F., Jacquet, S., Vaulot, D. Enumeration and cell cycle analysis of natural populations of marine picoplankton by flow cytometry using the nucleic acid stain SYBR Green I. Appl Environ Microbiol. 63, 186-193 (1997).
- Noble, R. T., Fuhrman, J. A. Use of SYBR Green I for rapid epifluorescence counts of marine viruses and bacteria. Aquat Microb Ecol. 14, 113-118 (1998).
- Forster, S., Snape, J. R., Lappin-Scott, H. M., Porter, J. Simultaneous fluorescent gram staining and activity assessment of activated sludge bacteria. Appl Environ Microbiol. 68, 4772-4779 (2002).
- Lauer, B. A., Reller, L. B., Mirrett, S. Comparison Of Acridine-Orange And Gram Stains For Detection Of Microorganisms In Cerebrospinal-Fluid And Other Clinical Specimens. J Clin Microbiol. 14, 201-205 (1981).
- Mason, D. J., Shanmuganathan, S., Mortimer, F. C., Gant, V. A. A fluorescent gram stain for flow cytometry and epifluorescence microscopy. Appl Environ Microbiol. 64, 2681-2685 (1998).
- Saida, H., Ytow, N., Seki, H. Photometric application of the Gram stain method to characterize natural bacterial populations in aquatic environments. Appl Environ Microbiol. 64, 742-747 (1998).
- Sizemore, R. K., Caldwell, J. J., Kendrick, A. S. Alternate Gram Staining Technique Using A Fluorescent Lectin. Appl Environ Microbiol. 56, 2245-2247 (1990).
- Bitton, G., Dutton, R. J., Foran, J. A. New Rapid Technique For Counting Microorganisms Directly On Membrane Filters. Stain Technology. 58, 343-346 (1983).
- Broadaway, S. C., Barton, S. A., Pyle, B. H. Rapid staining and enumeration of small numbers of total bacteria in water by solid-phase laser cytometry. Appl Environ Microbiol. 69, 4272-4273 (2003).
- Yagupsky, P., Nolte, F. S. Quantitative aspects of septicemia. Clin Microbiol Rev. 3, 269-279 (1990).
- Kang, D. K., et al. Rapid detection of single bacteria in unprocessed blood using Integrated Comprehensive Droplet Digital Detection. Nat Commun. 5, 5427 (2014).
- Hand, K. P. . Report of the Europa Lander Science Definition Team. , (2017).
- Popescu, G., et al. Optical imaging of cell mass and growth dynamics. Am J Physiol-Cell Physiol. 295, C538-C544 (2008).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci U S A. 108, 13124-13129 (2011).
- Rappaz, B., et al. Noninvasive characterization of the fission yeast cell cycle by monitoring dry mass with digital holographic microscopy. J.Biomed Opt. 14, 034049 (2009).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Opt. Express. 23, 15792-15805 (2015).
- Wallace, J. K., et al. Robust, compact implementation of an off-axis digital holographic microscope. Opt. Express. 23, 17367-17378 (2015).
- Lindensmith, C. A., et al. A Submersible, Off-Axis Holographic Microscope for Detection of Microbial Motility and Morphology in Aqueous and Icy Environments. Plos One. 11, e0147700 (2016).
- Dumas, E. M., Ozenne, V., Mielke, R. E., Nadeau, J. L. Toxicity of CdTe Quantum Dots in Bacterial Strains. IEEE Trans. NanoBiosci. 8, 58-64 (2009).
- Frisk, A., Jyot, J., Arora, S. K., Ramphal, R. Identification and functional characterization of flgM, a gene encoding the anti-sigma 28 factor in Pseudomonas aeruginosa. J Bacteriol. 184, 1514-1521 (2002).
- Adler, J., Templeton, B. The effect of environmental conditions on the motility of Escherichia coli. Journal Gen Microbiol. 46, 175-184 (1967).
- Piedrahita-Quintero, P., Castaneda, R., Garcia-Sucerquia, J. Numerical wave propagation in Image J. Appl Opt. 54, 6410-6415 (2015).
- Schnars, U., Jüptner, W. P. Digital recording and numerical reconstruction of holograms. Meas. Sci. Technol. 13, R85 (2002).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。