Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Quantifizierung von Mikroorganismen in geringen Konzentrationen mit Hilfe digitalen holographischen Mikroskopie (DHM)
In diesem Artikel
Zusammenfassung
Digitaler holographischer Mikroskopie (DHM) ist eine volumetrische Technik, die können bildgebende Proben, die 50-100 X dicker als Hellfeld Mikroskopieren mit vergleichbarer Auflösung, mit der Fokussierung durchgeführt Nachbearbeitung. Hier DHM dient zur Identifizierung, zählen, und verfolgen Mikroorganismen bei sehr niedrigen dichten und verglichen mit optischen Dichtemessungen, Keimzahl und direkte Zählung.
Zusammenfassung
Genau erkennen und zählen spärliche bakterielle Proben hat viele Anwendungen in der Lebensmittel-, Getränke- und pharmazeutischen Industrie, in der medizinischen Diagnostik und für die Lebens-Erkennung von unbemannten Missionen zu anderen Planeten und Monde des Sonnensystems. Derzeit sind spärliche bakterielle Proben von Kultur-Beschichtung oder Epifluoreszenz Mikroskopie gezählt. Kultur-Platten erfordern lange Inkubationszeit Zeit (Tage bis Wochen) und Epifluoreszenz Mikroskopie erfordert umfangreiche Färbung und Konzentration der Probe. Hier zeigen wir wie einfallenden digitalen holographischen Mikroskopie (DHM) zum Auflisten von Bakterien in sehr verdünnter Kulturen (100-104 Zellen/mL). Erstens ist der Bau von benutzerdefinierten DHM zusammen mit detaillierten Anweisungen auf den Aufbau einer kostengünstigen Instrument diskutiert. Die Grundsätze der Holographie werden besprochen, und ein statistisches Modell wird verwendet, um abzuschätzen, wie lange Videos sollte sein, Zellen, basierend auf die optische Leistungsmerkmale des Instruments und die Konzentration der bakterielle Lösung (Tabelle 2) zu erkennen . Videoerkennung von Zellen bei 105, 104, 103und 100 Zellen/mL zeigt sich in Echtzeit mit UN-rekonstruierten Hologrammen. Rekonstruktion von Amplitude und Phase Bildern zeigt sich mit einer Open-Source-Software-Paket.
Einleitung
Bestimmung der genauen bakteriellen zählt in sehr verdünnten Proben ist in vielen Anwendungen von entscheidender Bedeutung: einige Beispiele sind Wasser und Nahrung Qualität Analyse1,2,3; Nachweis von Krankheitserregern in Blut, Liquor cerebrospinalis oder Sputum4,5; Herstellung von pharmazeutischen Produkten, einschließlich steriles Wasser6; und Umwelt-Community-Analyse in oligotrophen Umgebungen wie das offene Meer und Sedimente7,8,9. Es gibt auch immer mehr Interesse an der Erkennung von möglichen erhaltene mikrobielles Leben auf den eisigen Monden von Jupiter und Saturn, besonders Europa10,11 und Enceladus12,13, 14, die bekanntermaßen Untergrund flüssigen Ozeane haben. Weil keine Mission seit Viking 1978 versucht hat, erhaltene Leben auf einem anderen Planeten zu finden, gab es begrenzte Entwicklung der Technologien und Instrumente für bakterielle Identifikation und im Weltraum-Missionen15zählen.
Traditionelle Methoden der Keimzahl finden nur kultivierbare Zellen, die eine Minderheit von Tierarten in ökologischer Belastungen manchmal darstellen können < 1 %16. Platten erfordern Tage oder Wochen Inkubation für maximalen Erfolg, abhängig von der Belastung. Epifluoreszenz Mikroskopie hat weitgehend Platte zählt als Goldstandard für die schnelle und genaue mikrobielle Aufzählung ersetzt. Nukleinsäure-Säure-labeling fluoreszierende Farbstoffe z. B. 4 ', 6-Diamidino-2-Phenylindole Dihydrochloride (DAPI), SYBR Green oder Acridin Orange, die an Nukleinsäuren binden sind typische verwendeten Farbstoffe17,18,19 , obwohl viele Studien verwenden unterzeichnen fluoreszierende Indikatoren Gramm20,21,22,23,24. Mit diesen Methoden ohne Vorkonzentrierung Stufen führt zu Nachweisgrenzen (DGS) ~ 105 Zellen pro mL. Verbesserungen in der DG sind mit Filterung möglich. Eine flüssige Probe wird Vakuum gefiltert auf eine Membran, in der Regel aus Polycarbonat und idealerweise schwarz, Hintergrund zu reduzieren. Low-Hintergrund Farbstoffe wie die oben genannten DNA-Flecken direkt auf den Filter25angewendet werden können. Für die genaue Zählung vom Auge, ~ 105 Zellen pro Filter, was, dass für mehr als 10 ~5 Zellen pro mL verdünnten Proben bedeutet erforderlich sind, erhebliche Probenvolumina gesammelt und gefiltert werden müssen. Laserscanning-Geräte wurden entwickelt, um systematisch erkunden alle Regionen des Filters und reduzieren damit die Anzahl der Zellen erforderlich für die Zählung, Nachweisgrenzen bis zu ~ 102 Zellen pro mL26schieben. Aber diese sind nicht verfügbar in den meisten Labors und benötigen anspruchsvolle Hardware als auch Software, die Experten Bestätigung zu erlauben, die Partikel beobachtet Bakterien und keine Ablagerungen sind.
Als Referenz, Erwachsene mit Sepsis zeigen Symptome bei in der Regel beginnen < 100 Zellen/mL Blut und Säuglinge bei < 10 Zellen/mL. Eine Blutprobe von einem Erwachsenen dauert 10 mL, und von einem Kleinkind, 1 mL. PCR-basierte Methoden sind durch die Anwesenheit von menschlichen und nicht-pathogenen Flora DNA und durch Hemmung der PCR-Komponenten in der Blut-27,-28gehemmt. Trotz einer Vielzahl von Zukunftstechniken bleiben Kulturen der Goldstandard für die Diagnose der Blutbahn Infektionen, vor allem in ländlichen Gebieten oder sich entwickelnden Nationen. Für die Erkennung von Leben auf anderen Planeten können Thermodynamische Berechnungen den Energiehaushalt und damit die erwarteten möglich Biomasse abschätzen. 1 - 100 Zellen/mL sollen thermodynamisch günstigen auf Europa29sein. Es ist leicht aus diesen Zahlen ersichtlich, Erkennung von sehr kleinen Zahl von Zellen in großen Mengen wässrigen Lösung eine wichtige ungelöste Problem ist.
In diesem Beitrag zeigen wir Erkennung von Serratia Marcescens und Shewanella Oneidensis (Wildtyp und nicht bewegliche Mutant) bei Konzentrationen von 105, 104, 103und 100 Zellen/mL über eine off-Axis holographische Digitalmikroskop (DHM). Der entscheidende Vorteil des DHM über traditionelle Lichtmikroskopie ist die gleichzeitige Darstellung der dicken Probenvolumen mit hoher Auflösung – in dieser Implementierung wurde die Probenkammer 0,8 mm dick. Diese Probenkammern entstanden durch die Soft-Lithographie von Polydimethylsiloxan (PDMS) aus einer Präzision bearbeitete Aluminium-Form mit einer Toleranz von ± 50 µm. Dies entspricht einer ca. 100-fold Verbesserung in der Tiefe des Feldes über Hochleistungs-Lichtmikroskopie. DHM bietet auch quantitative Phaseninformationen ermöglicht Messungen der optischen Weglänge (Produkt aus Brechungsindex und Dicke). DHM und ähnliche Techniken sind verwendet worden, für die Überwachung der Bakterien und Hefe Zellzyklus und Berechnung der bakteriellen trockene Masse30,31,32; Streuung Unterschiede können auch verwendet werden, um Bakterienstämme33zu unterscheiden.
Das Instrument, das wir verwenden ist speziell für den Einsatz mit Mikroorganismen, wie zuvor veröffentlichten34,35, nach Mass und seine Architektur und Konstruktion werden gezeigt und diskutiert. Wässrige Lösungen sind eine 0,25 µL Volumen Probenraum über Spritzenpumpe kontinuierlich zugeführt; die Durchflussrate richtet sich nach Kamera-Frame-Rate um Bildgebung des gesamten Probenvolumen zu gewährleisten. Eine statistische Berechnung prognostiziert die Anzahl der Probenvolumina, die abgebildet werden müssen, um eine beträchtliche Anzahl von Zellen in einer bestimmten Konzentration zu erkennen.
Für Handy-Anwendungen war die Rekonstruktion der Hologramme in Amplitude und Phase Bilder nicht erforderlich; Analyse wurde auf das rohe Hologramm durchgeführt. Das spart erhebliche Rechenressourcen und Speicherplatz: 500 Mb Hologramm video 1-2 Tb bei rekonstruiert werden. Jedoch diskutieren wir Rekonstruktion durch Tiefe der Probe zu bestätigen, dass die Hologramme die gewünschten Arten vertreten. Ein wichtiges Merkmal des DHM ist seine Fähigkeit, Intensität und Phase der Bilder zu überwachen. Organismen, die nahezu transparent in ihrer Intensität (wie die meisten biologischen Zellen) sind eindeutig in Phase angezeigt. Da es eine markierungsfreie Technik ist, werden keine Farbstoffe eingesetzt. Dies ist ein einDvantage für möglich Raumfahrt-Anwendungen, da Farbstoffe nicht die Voraussetzungen einer Mission überleben können und – noch wichtiger ist – kann nicht davon ausgegangen werden, mit Außerirdischen Organismen arbeiten, die nicht für die Codierung von DNA oder RNA nutzen dürfen. Es ist auch ein Vorteil für die Arbeit in extremen Umgebungen wie der Arktis und Antarktis, wo Farbstoffe können schwierig sein, zu der abgelegenen Lage bringen und bei der Lagerung beeinträchtigen können. Rekonstruktion von Bildern in Phase und Amplitude erfolgt mithilfe einer Open-Source-Software-Pakets, die wir auf GitHub (SHAMPOO) zur Verfügung gestellt haben oder ImageJ.
Protokoll
1. Wachstum und Aufzählung der Bakterien
Hinweis: Dies gilt für fast jeder Bakterienstamm in die entsprechende mittlere 36 angebaut. In unserem Beispiel verwenden wir drei Sorten: Serratia Marcescens als eine gemeinsame, leicht identifizierbare Lab Belastung; und ein kleiner, sehr bewegliche Umweltbelastung, Shewanella Oneidensis MR-1. Um die Erkennung von vergleichen bewegliche vs. nicht-bewegliche Zellen, nicht bewegliche Shewanella Mutant, Δ FlgM, dient auch zum Vergleich 37. Alle Sorten werden angebaut, in Lysogeny Brühe (LB).
- Prepare sterile LB-Medium (pro Liter destilliertem Wasser: 10 g Bacto Tryptone, 5 g Bacto Hefe, 10 g NaCl; Filter oder Autoklaven). Bereiten Sie standard 100 mm Durchmesser LB-Agar-Platten (gleiche Rezept als Medium plus 15 g Agar / l; Autoklaven (121 ° C, 20 min) zu sterilisieren und gießen Wenn kühl genug, um zu behandeln). Medium und Teller im Kühlschrank lagern.
- Am Tag vor dem Experiment Samen 5-6 mL des " Master " Kulturmedium aus einer frischen bakterielle Kolonie, mit richtig steril und biologische Verfahren. Inkubieren Sie auf einen Shaker (120 u/min) bei 30 ° C für ~ 12 h Inkubationszeit abhängig von der Belastung und des Wachstums Rate werden. In unseren Experimenten werden die Stämme Shewanella für 12 Stunden inkubiert; jedoch ist die Serratia nach 8 Stunden geerntet, um die Kultur präsentiert Mitte logarithmische Wachstum zu gewährleisten.
- Am Tag des Experiments, nehmen Sie eine photometrische Lesung (OD 600) von den bakteriellen Kultur, die voraussichtlich im Bereich von 0,6 bis 0,7 zu meistern. Es sollte in der logarithmischen Wachstumsphase (wie zuvor festgelegt). Wenn nicht, Subkultur 100 µL in 5 mL Medium und Zellwachstum in der Mitte des Log-Phase lassen.
- Nehmen Sie eine Probe des Meisters Kultur und zählen Sie die Zellen, die direkt mit einem Petroff-Hausser zählen Kammer. Das lebende und tote Zellen auflisten wird.
- Eine 10 µL Probe der unverdünnte Kultur mit einer Mikropipette und Transfer in die Kammer zu extrahieren.
- Bild unter einem hohen trocknende Objektive Mikroskop (40 X oder 63 X, NA 0,7 - 0,8) mit Phasenkontrast.
- Zählen die Bakterien auf mindestens 20 Plätzen und Durchschnitt.
Hinweis: Zählen Sie nur jene Bakterien, die vollständig in ein Quadrat und nur diese überqueren oben und links Grenzen (oder unten und rechts, wenn Sie es vorziehen). Wenn Plätze von mehreren Linien voneinander getrennt sind, wählen Sie eine als Umgrenzung. - Calculate Konzentration als der Durchschnitt von 20 Plätzen x Verdünnungsfaktor. Hinweis die direkte zählt funktionieren nicht gut für Konzentrationen < 10 7 / mL.
- Machen Verdünnungsreihen der Kultur für die Zählung der Koloniebildenden Einheiten (KBE). Dies wird lebenden Zellen nur aufzählen.
- Stellen eine serielle Verdünnung von jeder der ausgewählten bakteriellen Proben mit steriler Kochsalzlösung 0,9 % Lösung. 20 µL der bakterielle Lösung zu übertragen und mit 180 µL Kochsalzlösung verdünnen. Wiederholen Sie, bis die niedrigste Konzentration ~ 10 3 Zellen/mL.
- Nehmen 100 µL aus mindestens zwei Verdünnungen empfohlen sind 10 3 und 10 4 /mL — und Platte auf einen geeigneten Nährboden-Platte. Mit einem sterilen Spreader verteilt. Führen Sie mindestens 3 Wiederholungen jeder Verdünnung.
- Brüten auf eine entsprechende Temperatur für Ihre Bakterien über Nacht oder bis Kolonien wachsen.
- Kolonien zählen und rechnen Koloniebildenden Einheiten (KBE) nach (Anzahl der Kolonien x Verdünnungsfaktor) / Volumen plattiert = KBE/mL. Durchschnittliche KBE in den Wiederholungen.
2. Vorbereitung der hoch verdünnt Proben für DHM
- machen Verdünnungsreihen des Meisters Kultur für DHM und Post-DHM zählen der Koloniebildenden Einheiten auf LB Medien Platten (siehe 1.4.1 - 1.4.4). Diese Doppelblind-führen, so dass die Person, die die Aufnahmen nichts von der Konzentration in jeder gemessene Probe ist.
- Verdünnen die Bakterien in 10-15 mL ein minimaler Medium, die Motilität (bei Bedarf) zu fördern, sondern hemmen die Zellteilung, so dass die Konzentration der Zellen während des Experiments nicht merklich ändert. 0,9 % Kochsalzlösung oder eine spezifischere Motilität Medium möglicherweise. Beispielsweise muss bei Verwendung von Escherichia coli Motilität Medium EDTA 38 enthalten. Für diese Beispiele verwenden wir 0,9 % Kochsalzlösung.
3. Aufnahme von DHM Videos
- mit einer sterilen Spritze, in ca. 10 mL der Verdünnung von Interesse ziehen.
- Die Spritze an der DHM Probenkammer mit sterilen Armaturen und Schläuche anschließen.
Hinweis: Eine maßgeschneiderte mikrofluidischen Kammer wurde konstruiert, um die konstanten Durchfluss der Probe durch den Strahlengang des Instruments zu ermöglichen. Siehe Abschnitt "Ergebnisse" für weitere Details. Vor dem Experiment, sollten alle Komponenten der Probenraum durch Autoklavieren sterilisiert werden. - Fließen die Probe aus der Spritze durch die Probenkammer kontinuierlich über eine Spritzenpumpe.
Hinweis: Geeignete Flussraten variieren je nach fluidische Kanal Abmessungen sowie gewünschten Durchsatz und Erwerb Dateneinschränkungen. - Wie im Beispiel die Probenkammer durchströmt wird erwerben Hologramme nacheinander mit einem entsprechenden Frame-Rate. Eine Zeitstempel Datei wird erstellt werden, welche Protokolle erfassen die Zeit eines jeden Bildes später während der Datenanalyse (Abschnitt 4-Protokoll) verwendet werden. Erhalten Sie alle Hologramme bei Raumtemperatur (23° C). Hologramme durch Ausführen einer vorformulierte C++ ausführbare Datei zur Verfügung gestellt vom Kamerahersteller aufnehmen.
Hinweis: Angemessener Preise variieren je nach der Volumenstrom gewählt. Für dieses Experiment, es empfiehlt sich, eine Frame-Rate zu verwenden, so dass ein Bakterium abgebildet werden würde > 2 Mal, wie es über das Instrument gereist ' Sichtfeld s. - Lassen Sie genügend Zeit für die gesamte 10 mL der Probe durch das DHM übergeben werden.
- , Damit Bakterien nicht wachsen oder sterben während der Experimente impfen bereichert Medien Platten mit 100 µL der abgebrannten Medien Post-DHM-Bilderfassung. Mit einem sterilen Spreader verteilt und rund um die Uhr bei entsprechenden Temp inkubieren; zählen der Kolonien vorhanden.
4. Berechnung der Zelldichte und Grenzen der Erkennung
- mit den erworbenen Hologramm-Videos analysieren Sie die Daten für die Anwesenheit der Bakterien.
- Berechnen Sie den Median Pixelwert für eine Zeitreihe von Hologrammen und ziehen diese Medianwert von jedem jeweiligen Pixel, stationäre Artefakte im Hologramm zu beseitigen. Dies kann in ImageJ erfolgen, indem die folgenden Schritte ausführen:
- Konvertieren in 32-Bit (Image-Typ-32 Bit)
- berechnen der Median (Bild-Stack-Zproject-Median)
- subtrahieren den Median aus dem Rest der Stack ( Process-ImageCalculator-Subtract)
- die Anzahl der sichtbaren hellen Ringe und Zellen im Fokus manuell. In ImageJ, dies geschieht, indem Sie auf die Objekte mit dem Werkzeug Punkt.
- Jede Serie von Hologrammen von einem Zeitstempel Datei (Aufnahme mit Hologramm Erwerbszeitpunkt) begleitet werden. Verwendung die Zeitstempel Datei zur Berechnung der Gesamtbetrag der Probe gepumpt zur Zeit das Bild erfasst wurde.
- Berechnen Sie den Median Pixelwert für eine Zeitreihe von Hologrammen und ziehen diese Medianwert von jedem jeweiligen Pixel, stationäre Artefakte im Hologramm zu beseitigen. Dies kann in ImageJ erfolgen, indem die folgenden Schritte ausführen:
- Die Zelldichte zu berechnen, indem man die Gesamtzahl der Zellen erkannt durch das Gesamtvolumen der Stichprobe abgebildet. Durchschnittlich ca. 5-10 Frames für genaue Statistiken.
5. Bild-Rekonstruktion, Amplitude und Phase
- Wählen Sie repräsentative Videos mit 10-200 Zellen pro Frame. Roh (nicht Median-subtrahiert) Hologramme in der Rekonstruktion Software laden (Beispiele: numerische Vermehrung ImageJ 39; SHAMPOO [GitHub]).
- Verwendung von ImageJ, gehen Sie zum OD-numerische Propagation-numerische Beugung.
- An der Eingabeaufforderung, zeichnen Sie eine Fourier-Maske zu wählen, die realen oder virtuellen Abbild und beseitigen jede sinusförmigen Störungselemente.
- Amplitude und Phase bei entsprechenden Z-Schritte durch Eingabe der Wiederaufbau Entfernung zu rekonstruieren.
- Median subtrahieren die Amplitude Daten zur Lärmminderung in 4.1.1.
- Plot-Daten als ein 3D Cube oder Projektion. Gehen Sie auf Plugins — Volume Viewer.
Ergebnisse
Die Ergebnisse sollten die Möglichkeit, lebende und tote Bakterien auf einem sehr niedrigen Niveau von DHM zu erkennen geben. Die Anzahl der Bakterien gezählt sollte mit der erzielten Ergebnisse mit Petroff-Hauser zählen Industrie- und Platte Grafen übereinstimmen. Statistische Standardmethoden bieten Informationen über die Genauigkeit der verschiedenen Nachweismethoden bei verschiedenen bakteriellen Konzentrationen.
Diskussion
Numerische Rekonstruktion von Hologrammen: für die numerische Rekonstruktion von Hologrammen, die eckige Spektrum-Methode (ASM) verwendet. Dies beinhaltet die Faltung des Hologramms mit dem Grün-Funktion für die DHM. Die komplexe Wellenfront des Bildes an eine bestimmte Brennebene kann durch den Einsatz der Fourier-Convolution-Theorem wie folgt berechnet werden:
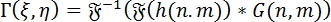 (1)
(1)
Wo
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Die Autoren erkennen die Gordon und Betty Moore Foundation Stipendien 4037 und 4038, California Institute of Technology für die Finanzierung dieser Arbeit.
Materialien
| Name | Company | Catalog Number | Comments |
| Bacto Yeast | BD Biosciences | 212750 | |
| Bacto Tryptone | BD Biosciences | 211705 | |
| Sodium chloride | Sigma-Aldrich | 7710 | Many options for purchase |
| Bacto Agar | BD Biosciences | 214010 | |
| 10 cm Petri dishes | VWR | 10053-704 | |
| 15 mL culture tubes | Falcon (Corning Life Sciences) | 352002 | Loose-capped |
| Petroff-Hauser chamber | Electron Microscopy Sciences | 3920 | |
| 10 mL syringes | BD Biosciences | 309604 | Luer-Lok tip not necessary |
| Male Luer to 1/16” barbed fitting | McMaster-Carr | 51525K291 | |
| Autoclavable 1/16” ID PVC tubing for flow | Nalgene | 8000-0004 | Sold by length, purchase accordingly |
| Syringe pump | Harvard Apparatus | PHD 2000 | |
| Sample Chamber | Custom | n/a | See Materials Section |
| Holographic Microscope | Custom | n/a | See Wallace et al. |
| Open-source software | Custom | https://github.com/bmorris3/shampoo |
Referenzen
- Akineden, O., Weirich, S., Abdulmawjood, A., Failing, K., Buelte, M. Application of a Fluorescence Microscopy Technique for Detecting Viable Mycobacterium avium ssp. paratuberculosis Cells in Milk. Food Anal. Methods. 8, 499-506 (2015).
- Deibl, J., Paulsen, P., Bauer, F. Rapid enumeration of total aerobic microbial counts on meat and in meat products by means of the direct epifluorescence filter technique. Wien Tierarztl Monat. 85, 327-333 (1998).
- Huang, J., Li, Y., Slavik, M. F., Tao, Y., Huff, G. R. Identification and enumeration of Salmonella on sample slides of poultry carcass wash water using image analysis with fluorescent microscopy. Transactions of the Asae. 42, 267-273 (1999).
- Durtschi, J. D., et al. Increased sensitivity of bacterial detection in cerebrospinal fluid by fluorescent staining on low-fluorescence membrane filters. J Med Microbiol. 54, 843-850 (2005).
- Panicker, R. O., Soman, B., Saini, G., Rajan, J. A Review of Automatic Methods Based on Image Processing Techniques for Tuberculosis Detection from Microscopic Sputum Smear Images. J Med Syst. 40, (2016).
- Riepl, M., et al. Applicability of solid-phase cytometry and epifluorescence microscopy for rapid assessment of the microbiological quality of dialysis water. Nephrol Dial Transplant. 26, 3640-3645 (2011).
- Hwang, C. Y., Cho, B. C. Virus-infected bacteria in oligotrophic open waters of the East Sea, Korea. Aquat Microb Ecol. 30, 1-9 (2002).
- Kepner, R. L., Pratt, J. R. Use of Fluorochromes for Direct Enumeration of Total Bacteria in Environmental-Samples - Past and Present. Microbiol Rev. 58, 603-615 (1994).
- Queric, N. V., Soltwedel, T., Arntz, W. E. Application of a rapid direct viable count method to deep-sea sediment bacteria. J Microbiol Meth. 57, 351-367 (2004).
- Prieto-Ballesteros, O., Vorobyova, E., Parro, V., Rodriguez Manfredi, J. A., Gomez, F. Strategies for detection of putative life on Europa. Adv Space Res. 48, 678-688 (2011).
- Gleeson, D. F., et al. Biosignature Detection at an Arctic Analog to Europa. Astrobiology. 12, 135-150 (2012).
- Carr, C. E., Zuber, M. T., Ruvkun, G., Ieee, . 2013 Ieee Aerospace Conference IEEE Aerospace Conference Proceedings. , (2013).
- Konstantinidis, K., et al. A lander mission to probe subglacial water on Saturn's moon Enceladus for life. Acta Astronautica. 106, 63-89 (2015).
- McKay, C. P., Anbar, A. D., Porco, C., Tsou, P. Follow the Plume: The Habitability of Enceladus. Astrobiology. 14, 352-355 (2014).
- Hoover, R. B., Hoover, R. B., Levin, G. V., Rozanov, A. Y., Wickramasinghe, N. C. . Instruments, Methods, and Missions for Astrobiology Xvii. , (2015).
- Handelsman, J. Metagenomics: application of genomics to uncultured microorganisms. Microbiol Mol Biol Rev: MMBR. 68, 669-685 (2004).
- Lebaron, P., Parthuisot, N., Catala, P. Comparison of blue nucleic acid dyes for flow cytometric enumeration of bacteria in aquatic systems. Appl Environ Microbiol. 64, 1725-1730 (1998).
- Marie, D., Partensky, F., Jacquet, S., Vaulot, D. Enumeration and cell cycle analysis of natural populations of marine picoplankton by flow cytometry using the nucleic acid stain SYBR Green I. Appl Environ Microbiol. 63, 186-193 (1997).
- Noble, R. T., Fuhrman, J. A. Use of SYBR Green I for rapid epifluorescence counts of marine viruses and bacteria. Aquat Microb Ecol. 14, 113-118 (1998).
- Forster, S., Snape, J. R., Lappin-Scott, H. M., Porter, J. Simultaneous fluorescent gram staining and activity assessment of activated sludge bacteria. Appl Environ Microbiol. 68, 4772-4779 (2002).
- Lauer, B. A., Reller, L. B., Mirrett, S. Comparison Of Acridine-Orange And Gram Stains For Detection Of Microorganisms In Cerebrospinal-Fluid And Other Clinical Specimens. J Clin Microbiol. 14, 201-205 (1981).
- Mason, D. J., Shanmuganathan, S., Mortimer, F. C., Gant, V. A. A fluorescent gram stain for flow cytometry and epifluorescence microscopy. Appl Environ Microbiol. 64, 2681-2685 (1998).
- Saida, H., Ytow, N., Seki, H. Photometric application of the Gram stain method to characterize natural bacterial populations in aquatic environments. Appl Environ Microbiol. 64, 742-747 (1998).
- Sizemore, R. K., Caldwell, J. J., Kendrick, A. S. Alternate Gram Staining Technique Using A Fluorescent Lectin. Appl Environ Microbiol. 56, 2245-2247 (1990).
- Bitton, G., Dutton, R. J., Foran, J. A. New Rapid Technique For Counting Microorganisms Directly On Membrane Filters. Stain Technology. 58, 343-346 (1983).
- Broadaway, S. C., Barton, S. A., Pyle, B. H. Rapid staining and enumeration of small numbers of total bacteria in water by solid-phase laser cytometry. Appl Environ Microbiol. 69, 4272-4273 (2003).
- Yagupsky, P., Nolte, F. S. Quantitative aspects of septicemia. Clin Microbiol Rev. 3, 269-279 (1990).
- Kang, D. K., et al. Rapid detection of single bacteria in unprocessed blood using Integrated Comprehensive Droplet Digital Detection. Nat Commun. 5, 5427 (2014).
- Hand, K. P. . Report of the Europa Lander Science Definition Team. , (2017).
- Popescu, G., et al. Optical imaging of cell mass and growth dynamics. Am J Physiol-Cell Physiol. 295, C538-C544 (2008).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci U S A. 108, 13124-13129 (2011).
- Rappaz, B., et al. Noninvasive characterization of the fission yeast cell cycle by monitoring dry mass with digital holographic microscopy. J.Biomed Opt. 14, 034049 (2009).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Opt. Express. 23, 15792-15805 (2015).
- Wallace, J. K., et al. Robust, compact implementation of an off-axis digital holographic microscope. Opt. Express. 23, 17367-17378 (2015).
- Lindensmith, C. A., et al. A Submersible, Off-Axis Holographic Microscope for Detection of Microbial Motility and Morphology in Aqueous and Icy Environments. Plos One. 11, e0147700 (2016).
- Dumas, E. M., Ozenne, V., Mielke, R. E., Nadeau, J. L. Toxicity of CdTe Quantum Dots in Bacterial Strains. IEEE Trans. NanoBiosci. 8, 58-64 (2009).
- Frisk, A., Jyot, J., Arora, S. K., Ramphal, R. Identification and functional characterization of flgM, a gene encoding the anti-sigma 28 factor in Pseudomonas aeruginosa. J Bacteriol. 184, 1514-1521 (2002).
- Adler, J., Templeton, B. The effect of environmental conditions on the motility of Escherichia coli. Journal Gen Microbiol. 46, 175-184 (1967).
- Piedrahita-Quintero, P., Castaneda, R., Garcia-Sucerquia, J. Numerical wave propagation in Image J. Appl Opt. 54, 6410-6415 (2015).
- Schnars, U., Jüptner, W. P. Digital recording and numerical reconstruction of holograms. Meas. Sci. Technol. 13, R85 (2002).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten