É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Quantificação de microrganismos em baixas concentrações, utilizando microscopia holográfica Digital (DHM)
Neste Artigo
Resumo
Microscopia holográfica digital (DHM) é uma técnica volumétrica que permite amostras de imagens realizadas 50-100x mais espesso que brightfield microscopia em resolução comparável, com o foco de pós-processamento. Aqui DHM é usado para identificar, contando e acompanhamento de microorganismos em muito baixas densidades e em comparação com medições de densidade óptica, contagem de placa e contagem direta.
Resumo
Com a detecção e contagem de amostras bacterianas esparsas tem muitas aplicações na comida, bebidas e indústrias de processamento farmacêutico, em diagnósticos médicos e para a detecção de vida por missões robóticas para outros planetas e luas do sistema solar. Atualmente, esparsas amostras bacterianas são contadas por microscopia de chapeamento ou epifluorescência de cultura. Placas de cultura exigem longo tempo de incubação (dias ou semanas) e microscopia de epifluorescência requer mancha extensa e concentração da amostra. Aqui, demonstramos como usar fora do eixo microscopia holográfica digital (DHM) para enumerar as bactérias em culturas muito diluídas (100-104 células/mL). Em primeiro lugar, a construção da DHM personalizado é discutida, juntamente com instruções detalhadas sobre a criação de um instrumento de baixo custo. São discutidos os princípios da holografia, e um modelo estatístico é usado para estimar quanto tempo vídeos devem ser para detectar células, com base nas características desempenho óptico do instrumento e a concentração da solução bacteriana (tabela 2) . A detecção de células em 105, 104, 103e 100 células/mL é demonstrada em tempo real usando hologramas un-reconstruídos. Reconstrução de imagens de amplitude e fase é demonstrada usando um pacote de software open-source.
Introdução
Determinação da precisa contagens bacterianas em amostras muito diluídas é crucial em muitas aplicações: alguns exemplos são a água e a comida qualidade análise1,2,3; deteção de patógenos no sangue, líquido cefalorraquidiano ou escarro4,5; produção de produtos farmacêuticos, incluindo água estéril6; e análise ambiental da Comunidade em ambientes oligotróficos como o aberto oceano e sedimentos7,8,9. Existe também uma crescente interesse para detecção de possível vida microbiana existente nas luas geladas de Júpiter e Saturno, particularmente Europa10,11 e Enceladus12,13, 14, que são conhecidos por terem subsuperficiais oceanos líquidos. Porque nenhuma missão desde Viking em 1978 tem tentado encontrar existe vida em outro planeta, tem havido desenvolvimento limitado de tecnologias e instrumentos de identificação bacteriana e contando durante espaço missões15.
Os métodos tradicionais de contagem de placa encontrar apenas células viáveis, que podem representar uma minoria das espécies em cepas ambientais, às vezes < 1%16. Placas requerem dias ou semanas de incubação para o máximo de sucesso, dependendo da estirpe. Microscopia de epifluorescência substituiu, em larga medida, contagens das placas como o padrão ouro para enumeração microbiana rápida e exata. Corantes de fluorescentes nucleic-ácido-rotulagem como laranja de acridina, dicloridrato 6-diamidino-2-phenylindole (DAPI), SYBR Green ou 4 ' que se ligam aos ácidos nucleicos são os corantes típicos usados17,18,19 , apesar de muitos estudos utilizam indicadores fluorescentes de grama assinar20,21,22,23,24. Usando esses métodos sem etapas de pré-concentração leva à limites de detecção (LoDs) de ~ 105 células por mL. Melhorias no LoD são possíveis usando filtragem. Uma amostra de líquido é filtrada vácuo em uma membrana, geralmente em policarbonato e idealmente preto para reduzir a fundo. Baixo-fundo corantes tais como as manchas de DNA mencionadas acima podem ser aplicadas directamente para o filtro de25. Para contagem exata pelo olho, as células de5 ~ 10 são necessárias por filtro, o que significa que, para amostras mais diluídas do que 10 ~5 células por mL, volumes significativos de amostra devem ser recolhidas e filtradas. Dispositivos de varredura a laser foram desenvolvidos a fim de explorar sistematicamente todas as regiões do filtro e, assim, reduzir o número de células necessárias para a contagem, empurrando os limites de detecção até ~ 102 células por mL26. No entanto, estes não estão disponíveis na maioria dos laboratórios e exigem hardware sofisticado, assim como software que permitem a confirmação de perita que observou partículas é as bactérias e não os restos.
Para referência, adultos com sepse geralmente começam a mostrar sintomas de < 100 células/mL de sangue e bebês em < 10 células/mL. Uma recolha de sangue de um adulto leva 10 mL e de uma criança, 1ml. Métodos baseados em PCR são inibidos pela presença de humano e não-patogênicas flora DNA e pelos componentes do PCR-inibindo o sangue27,28. Apesar de uma variedade de técnicas emergentes, culturas permanecem o padrão ouro para o diagnóstico de infecções da corrente sanguínea, especialmente em zonas mais rurais ou nações em desenvolvimento. Detecção de vida em outros planetas, cálculos termodinâmicos podem estimar o orçamento de energia para a vida e, portanto, a biomassa possível esperada. 1 - 100 células/mL são esperados ser termodinamicamente razoável na Europa29. Pode ser facilmente visto destes números que a detecção de um pequeno número de células em grandes quantidades de solução aquosa é um importante problema sem solução.
Neste artigo, demonstramos a detecção de Serratia marcescens e Shewanella oneidensis (tipo selvagem e non-motile mutante) em concentrações de 105, 104, 10 e 100 células/mL usando um fora do eixo3 microscópio digital holográfico (DHM). A principal vantagem da DHM por microscopia de luz tradicional é a imagem simultânea de um volume de amostra espesso em alta resolução — nessa implementação, a câmara de amostra foi de 0,8 mm de espessura. Estas câmaras de amostra foram construídas pelo macio-litografia de polidimetilsiloxano (PDMS) de um molde de alumínio de precisão usinadas com uma tolerância de ± 50 µm. Isto representa uma melhoria de aproximadamente 100 vezes em profundidade de campo através de microscopia de luz de alta potência. DHM também fornece informações de fase quantitativa, permitindo a medição do comprimento do percurso óptico (produto do índice de refração e espessura). DHM e técnicas semelhantes têm sido utilizadas para monitoramento de bacteriano e ciclo celular de levedura e cálculo de massa seca bacteriana30,31,32; diferenças de dispersão mesmo podem ser usadas para diferenciar estirpes bacterianas33.
O instrumento que usamos é costume-construído especificamente para uso com microorganismos, como publicado anteriormente34,,35, e seu design e construção são demonstrados e discutidos. Soluções aquosas são continuamente fornecidas a uma câmara de amostra de volume µ l 0,25 através de bomba de seringa; a taxa de fluxo é determinada pela taxa de quadros da câmera para assegurar imagens volume da amostra inteira. Um cálculo estatístico prevê que o número de volumes de amostra que deve ser fotografada a fim de detectar um número significativo de células em uma dada concentração.
Para aplicações de célula-deteção, reconstrução de hologramas em amplitude e fase de imagens não era exigida; análise foi realizada no holograma cru. Isso economiza espaço em disco e recursos computacionais significativos: um holograma de 500 Mb vídeo será 1 a 2 Tb quando reconstruído. No entanto, podemos discutir reconstrução através da profundidade da amostra para confirmar que os hologramas representam a espécie desejada. Uma característica importante da DHM é sua capacidade de monitorar tanto a intensidade e a fase das imagens. Organismos que são quase transparentes em intensidade (como a maioria das células biológicas) aparecem claramente em fase. Como é uma técnica livre de rótulo, não são utilizados corantes. Este é um umdvantage para aplicações de voo de espaço possível, desde que corantes podem não sobreviver as condições de uma missão e — mais importante — não pode ser presumida a trabalhar com organismos extraterrestres, que não podem usar o DNA ou RNA para codificação. Também é uma vantagem para o trabalho em ambientes extremos, tais como o Ártico e Antártico, onde corantes podem ser difíceis de trazer para o local remoto e podem degradar após armazenamento. Reconstrução de imagens em fase e amplitude é executada usando um pacote de software open-source que disponibilizamos no GitHub (SHAMPOO) ou usando o ImageJ.
Protocolo
1. crescimento e enumeração de bactérias
Nota: isso se aplica a quase qualquer estirpe bacteriana crescida no médio adequado 36. No nosso exemplo, usamos três estirpes: Serratia marcescens como uma cepa comum, fácil laboratório identificáveis; e uma menor, altamente motile tensão ambiental, Shewanella oneidensis MR-1. Para comparar a deteção de motile vs pilhas non-motile, um non-motile Shewanella mutante, Δ FlgM, também é usado para comparação 37. Todas as cepas são cultivadas em caldo lisogenia (LB).
- Preparar estéril LB médio (por litro de água destilada: 10g bacto triptona, levedura da Difco 5 g, 10 g NaCl; filtro ou autoclave). Prepare padrão 100 mm placas diâmetro LB-ágar (mesma receita como médio mais ágar 15 g por litro; autoclave (121 ° C, 20 min) para esterilizar e despeje quando fresco o suficiente para lidar com). Loja meio e placas na geladeira.
- No dia anterior o experimento semente 5-6 mL de " mestre " meio de cultura de uma colônia bacteriana fresco, usando a técnica correta de estéril e biossegurança. Incube um agitador (120 rpm), a 30 ° C para o tempo de incubação ~ 12 h. será dependente da taxa de deformação e crescimento. Em nossos experimentos, as cepas de Shewanella são incubadas durante 12 horas; no entanto, Serratia é colhida após 8 horas, para garantir a cultura estará expondo crescimento meados-logarítmica.
- No dia do experimento, fazer uma leitura espectrofotométrica (OD 600) da bacteriano dominar a cultura, que deverá estar na faixa de 0.6 a 0.7. Deve ser na fase logarítmica de crescimento (conforme determinado anteriormente). Se não, subcultura 100 µ l em 5 mL de meio e permitir o crescimento de células para a fase de log de mid.
- Tirar uma amostra do mestre cultura e contar as células diretamente usando uma contagem de Petroff-Hausser de câmara. Isto irá enumerar células vivas e mortas.
- Extrair uma amostra de 10 µ l da cultura não diluída com uma micropipeta e transferência para a câmara.
- Imagem sob um microscópio de objetiva de alta-seco (40 X ou 63 X, at 0,7 - 0,8) usando o contraste de fase.
- Contar as bactérias pelo menos 20 quadrados e média.
Nota: Conte somente as bactérias que são inteiramente dentro de um quadrado e somente aqueles atravessando o topo e limites esquerdos (ou inferior e direito, se você preferir). Se quadrados são separados por várias linhas, escolher um como uma fronteira. - Concentração de calcular a média de 20 quadrados x fator de diluição. Nota que direcionam a contagens não funcionam bem para as concentrações < 10 7 / mL.
- Fazer diluições em série da cultura para a contagem de formadoras de Colônia (UFC) de unidades. Isto irá enumerar células vivas só.
Solução de
- fazer uma diluição serial de cada uma das amostras bacterianas selecionadas com solução salina 0,9%. Transferência de 20 µ l da solução bacteriana e diluí-la com soro fisiológico 180 µ l. Repita até que a concentração mais baixa é de 10 ~ 3 células/mL.
- Levar 100 µ l de pelo menos duas diluições-sugeriu é 10 3 e 10 4 /mL — e placa sobre uma placa de cultura em meio sólido apropriado. Espalhar com um espalhador estéril. Realizar pelo menos 3 repetições de cada diluição.
- Incubar a uma temperatura adequada para seus bactérias durante a noite ou até colônias cresçam.
- a contagem das colónias e calcular unidades (CFU), de acordo com (n º de colônias x fator de diluição) formadoras / volume chapeado = CFU/mL. Média UFC sobre as repetições.
2. Preparação de amostras altamente diluído por DHM
- fazer diluições em série do mestre cultura para DHM e post-DHM contagem de unidades formadoras de colônia em placas de mídia LB (ver 1.4.1 - 1.4.4). Executar este duplo-cego, para que a pessoa que faz as gravações desconhece a concentração em cada amostra medida.
- Diluir as bactérias em 10-15 mL de um meio mínimo que irá incentivar a motilidade (conforme apropriado), mas inibir a divisão celular, para que a concentração de células não altera sensivelmente durante o experimento. Este pode ser o soro fisiológico a 0,9% ou um meio mais específico de motilidade. Por exemplo, se utilizar a Escherichia coli, médio da motilidade deve conter EDTA 38. Para estes exemplos, nós usamos o soro fisiológico a 0,9%.
3. Gravar vídeos DHM
- usando uma seringa estéril, puxe em cerca de 10 mL da diluição de interesse.
- Conectar a seringa à câmara de amostra DHM usando encaixes estéril e tubos.
Nota: Uma câmara microfluidic sob medida foi construída para permitir o fluxo consistente de amostra através do caminho óptico do instrumento. Consulte a seção de resultados para obter mais detalhes. Antes do experimento, todos os componentes da câmara de amostra devem ser esterilizados em autoclave.
A amostra da seringa através da câmara de amostra continuamente usando uma bomba de seringa de fluxo de - .
Nota: Taxas de fluxo apropriado variam dependendo das dimensões do canal fluídico, taxa de transferência desejada, bem como limitações de aquisição de dados. - Como a amostra é fluiu através da câmara de amostra, adquirir hologramas consecutivamente a uma taxa de quadro apropriado. Será criado um arquivo de carimbo de tempo quais registros o tempo de cada imagem capture para ser usado mais tarde durante a análise de dados (protocolo seção 4). Obter todos os hologramas à temperatura ambiente (23° C). Gravar hologramas executando um arquivo executável de pré-escrita C++ fornecido pelo fabricante da câmera.
Nota: Taxas de quadros adequadas variam dependendo da taxa de fluxo escolhida. Para este experimento, recomenda-se usar uma taxa de quadros, tal que uma bactéria iria ser fotografada > 2 vezes mais que viajou através do instrumento ' campo de visão de s. - Permitir tempo suficiente para o inteiro 10 mL da amostra a ser vertido a DHM.
- Para garantir que as bactérias não crescem ou morrendo durante os experimentos, inocular enriquecido placas de mídia com 100 µ l da captura de imagens de post-DHM mídia irradiado. Espalhar com um espalhador estéril e incubar a temperatura apropriada para 24 horas; contagem das colónias presentes.
4. Cálculo da densidade celular e limites de detecção
- com os vídeos adquiridos holograma, analisar os dados para a presença de bactérias.
- Calcular o valor mediano de pixel para uma série de tempo de hologramas, em seguida, subtrair deste valor mediano de cada pixel respectivo para eliminar artefatos estacionários no holograma. Isso pode ser feito no ImageJ executando as seguintes etapas:
- converter para 32-bit (bit de imagem-tipo-32) ,
- calcular a mediana (imagem-pilha-Zproject-mediana)
- subtrair a mediana do resto do (pilha Process-ImageCalculator-Subtract)
- contar o número de anéis arejados visíveis e células em foco manualmente. No ImageJ, isso é feito por apontar para os objetos com a ferramenta ponto.
- Cada série de hologramas será acompanhado por um arquivo de carimbo de tempo (gravado no momento da aquisição do holograma). Usar o arquivo de carimbo de tempo para calcular a quantidade total de amostra bombeado ao tempo a imagem foi capturado.
- Calcular o valor mediano de pixel para uma série de tempo de hologramas, em seguida, subtrair deste valor mediano de cada pixel respectivo para eliminar artefatos estacionários no holograma. Isso pode ser feito no ImageJ executando as seguintes etapas:
- Calcular a densidade de células, dividindo o número total de células detectadas pelo volume total da amostra fotografada. Média de 5-10 frames para estatísticas precisas.
5. Reconstrução de Amplitude e fase de imagem
- escolher vídeos representativos com 10-200 células por quadro. Carregar hologramas crus (não mediana-subtraído) para o software de reconstrução (exemplos: propagação numérica ImageJ 39; CHAMPÔ [GitHub]).
- Usando o ImageJ, vá para OD-numérica difração propagação numérica.
- No prompt, desenhar uma máscara de Fourier para escolher o real ou imagem virtual e eliminar quaisquer características de ruído sinusoidal.
- Reconstruir a amplitude e fase na z-medidas adequadas, inserindo a distância de reconstrução.
- Mediana subtrair os dados de amplitude para reduzir o ruído como em 4.1.1.
- Dados do enredo como um 3D cubo ou projeção. Vá para Plugins — Volume Viewer.
Resultados
Os resultados devem indicar a capacidade de detectar bactérias vivas e mortas em níveis muito baixos por DHM. O número de bactérias contadas deve coadunar-se com os resultados obtidos usando as contagem de Petroff-Hauser câmara e placa contagens. Métodos estatísticos padrão fornecem informações sobre a precisão dos métodos de deteção diferentes concentrações bacterianas.
A Figura 1 m...
Discussão
Reconstrução numérica de hologramas: para a reconstrução numérica de hologramas, é usado o método do espectro angular (ASM). Isso envolve a convolução do holograma com a função Green para a DHM. O wavefront complexo da imagem em um determinado plano focal pode ser calculada utilizando o teorema da convolução Fourier como segue:
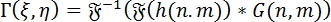 (1)
(1)
Onde
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Os autores reconhecem o Gordon e Betty Moore Foundation Grants 4037 e 4038 ao Instituto de tecnologia da Califórnia, para o financiamento deste trabalho.
Materiais
| Name | Company | Catalog Number | Comments |
| Bacto Yeast | BD Biosciences | 212750 | |
| Bacto Tryptone | BD Biosciences | 211705 | |
| Sodium chloride | Sigma-Aldrich | 7710 | Many options for purchase |
| Bacto Agar | BD Biosciences | 214010 | |
| 10 cm Petri dishes | VWR | 10053-704 | |
| 15 mL culture tubes | Falcon (Corning Life Sciences) | 352002 | Loose-capped |
| Petroff-Hauser chamber | Electron Microscopy Sciences | 3920 | |
| 10 mL syringes | BD Biosciences | 309604 | Luer-Lok tip not necessary |
| Male Luer to 1/16” barbed fitting | McMaster-Carr | 51525K291 | |
| Autoclavable 1/16” ID PVC tubing for flow | Nalgene | 8000-0004 | Sold by length, purchase accordingly |
| Syringe pump | Harvard Apparatus | PHD 2000 | |
| Sample Chamber | Custom | n/a | See Materials Section |
| Holographic Microscope | Custom | n/a | See Wallace et al. |
| Open-source software | Custom | https://github.com/bmorris3/shampoo |
Referências
- Akineden, O., Weirich, S., Abdulmawjood, A., Failing, K., Buelte, M. Application of a Fluorescence Microscopy Technique for Detecting Viable Mycobacterium avium ssp. paratuberculosis Cells in Milk. Food Anal. Methods. 8, 499-506 (2015).
- Deibl, J., Paulsen, P., Bauer, F. Rapid enumeration of total aerobic microbial counts on meat and in meat products by means of the direct epifluorescence filter technique. Wien Tierarztl Monat. 85, 327-333 (1998).
- Huang, J., Li, Y., Slavik, M. F., Tao, Y., Huff, G. R. Identification and enumeration of Salmonella on sample slides of poultry carcass wash water using image analysis with fluorescent microscopy. Transactions of the Asae. 42, 267-273 (1999).
- Durtschi, J. D., et al. Increased sensitivity of bacterial detection in cerebrospinal fluid by fluorescent staining on low-fluorescence membrane filters. J Med Microbiol. 54, 843-850 (2005).
- Panicker, R. O., Soman, B., Saini, G., Rajan, J. A Review of Automatic Methods Based on Image Processing Techniques for Tuberculosis Detection from Microscopic Sputum Smear Images. J Med Syst. 40, (2016).
- Riepl, M., et al. Applicability of solid-phase cytometry and epifluorescence microscopy for rapid assessment of the microbiological quality of dialysis water. Nephrol Dial Transplant. 26, 3640-3645 (2011).
- Hwang, C. Y., Cho, B. C. Virus-infected bacteria in oligotrophic open waters of the East Sea, Korea. Aquat Microb Ecol. 30, 1-9 (2002).
- Kepner, R. L., Pratt, J. R. Use of Fluorochromes for Direct Enumeration of Total Bacteria in Environmental-Samples - Past and Present. Microbiol Rev. 58, 603-615 (1994).
- Queric, N. V., Soltwedel, T., Arntz, W. E. Application of a rapid direct viable count method to deep-sea sediment bacteria. J Microbiol Meth. 57, 351-367 (2004).
- Prieto-Ballesteros, O., Vorobyova, E., Parro, V., Rodriguez Manfredi, J. A., Gomez, F. Strategies for detection of putative life on Europa. Adv Space Res. 48, 678-688 (2011).
- Gleeson, D. F., et al. Biosignature Detection at an Arctic Analog to Europa. Astrobiology. 12, 135-150 (2012).
- Carr, C. E., Zuber, M. T., Ruvkun, G., Ieee, . 2013 Ieee Aerospace Conference IEEE Aerospace Conference Proceedings. , (2013).
- Konstantinidis, K., et al. A lander mission to probe subglacial water on Saturn's moon Enceladus for life. Acta Astronautica. 106, 63-89 (2015).
- McKay, C. P., Anbar, A. D., Porco, C., Tsou, P. Follow the Plume: The Habitability of Enceladus. Astrobiology. 14, 352-355 (2014).
- Hoover, R. B., Hoover, R. B., Levin, G. V., Rozanov, A. Y., Wickramasinghe, N. C. . Instruments, Methods, and Missions for Astrobiology Xvii. , (2015).
- Handelsman, J. Metagenomics: application of genomics to uncultured microorganisms. Microbiol Mol Biol Rev: MMBR. 68, 669-685 (2004).
- Lebaron, P., Parthuisot, N., Catala, P. Comparison of blue nucleic acid dyes for flow cytometric enumeration of bacteria in aquatic systems. Appl Environ Microbiol. 64, 1725-1730 (1998).
- Marie, D., Partensky, F., Jacquet, S., Vaulot, D. Enumeration and cell cycle analysis of natural populations of marine picoplankton by flow cytometry using the nucleic acid stain SYBR Green I. Appl Environ Microbiol. 63, 186-193 (1997).
- Noble, R. T., Fuhrman, J. A. Use of SYBR Green I for rapid epifluorescence counts of marine viruses and bacteria. Aquat Microb Ecol. 14, 113-118 (1998).
- Forster, S., Snape, J. R., Lappin-Scott, H. M., Porter, J. Simultaneous fluorescent gram staining and activity assessment of activated sludge bacteria. Appl Environ Microbiol. 68, 4772-4779 (2002).
- Lauer, B. A., Reller, L. B., Mirrett, S. Comparison Of Acridine-Orange And Gram Stains For Detection Of Microorganisms In Cerebrospinal-Fluid And Other Clinical Specimens. J Clin Microbiol. 14, 201-205 (1981).
- Mason, D. J., Shanmuganathan, S., Mortimer, F. C., Gant, V. A. A fluorescent gram stain for flow cytometry and epifluorescence microscopy. Appl Environ Microbiol. 64, 2681-2685 (1998).
- Saida, H., Ytow, N., Seki, H. Photometric application of the Gram stain method to characterize natural bacterial populations in aquatic environments. Appl Environ Microbiol. 64, 742-747 (1998).
- Sizemore, R. K., Caldwell, J. J., Kendrick, A. S. Alternate Gram Staining Technique Using A Fluorescent Lectin. Appl Environ Microbiol. 56, 2245-2247 (1990).
- Bitton, G., Dutton, R. J., Foran, J. A. New Rapid Technique For Counting Microorganisms Directly On Membrane Filters. Stain Technology. 58, 343-346 (1983).
- Broadaway, S. C., Barton, S. A., Pyle, B. H. Rapid staining and enumeration of small numbers of total bacteria in water by solid-phase laser cytometry. Appl Environ Microbiol. 69, 4272-4273 (2003).
- Yagupsky, P., Nolte, F. S. Quantitative aspects of septicemia. Clin Microbiol Rev. 3, 269-279 (1990).
- Kang, D. K., et al. Rapid detection of single bacteria in unprocessed blood using Integrated Comprehensive Droplet Digital Detection. Nat Commun. 5, 5427 (2014).
- Hand, K. P. . Report of the Europa Lander Science Definition Team. , (2017).
- Popescu, G., et al. Optical imaging of cell mass and growth dynamics. Am J Physiol-Cell Physiol. 295, C538-C544 (2008).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci U S A. 108, 13124-13129 (2011).
- Rappaz, B., et al. Noninvasive characterization of the fission yeast cell cycle by monitoring dry mass with digital holographic microscopy. J.Biomed Opt. 14, 034049 (2009).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Opt. Express. 23, 15792-15805 (2015).
- Wallace, J. K., et al. Robust, compact implementation of an off-axis digital holographic microscope. Opt. Express. 23, 17367-17378 (2015).
- Lindensmith, C. A., et al. A Submersible, Off-Axis Holographic Microscope for Detection of Microbial Motility and Morphology in Aqueous and Icy Environments. Plos One. 11, e0147700 (2016).
- Dumas, E. M., Ozenne, V., Mielke, R. E., Nadeau, J. L. Toxicity of CdTe Quantum Dots in Bacterial Strains. IEEE Trans. NanoBiosci. 8, 58-64 (2009).
- Frisk, A., Jyot, J., Arora, S. K., Ramphal, R. Identification and functional characterization of flgM, a gene encoding the anti-sigma 28 factor in Pseudomonas aeruginosa. J Bacteriol. 184, 1514-1521 (2002).
- Adler, J., Templeton, B. The effect of environmental conditions on the motility of Escherichia coli. Journal Gen Microbiol. 46, 175-184 (1967).
- Piedrahita-Quintero, P., Castaneda, R., Garcia-Sucerquia, J. Numerical wave propagation in Image J. Appl Opt. 54, 6410-6415 (2015).
- Schnars, U., Jüptner, W. P. Digital recording and numerical reconstruction of holograms. Meas. Sci. Technol. 13, R85 (2002).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados