Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Cuantificación de microorganismos a bajas concentraciones usando microscopia holográfica Digital (DHM)
En este artículo
Resumen
La microscopia holográfica digital (DHM) es una técnica volumétrica que permite muestras de 50-100 X más gruesa que la microscopía brightfield en resolución comparable, con enfoque realizado post-processing. Aquí DHM se utiliza para identificar, contar y microorganismos en muy bajas densidades y en comparación con las mediciones de densidad óptica, gérmenes y conteo directo.
Resumen
Detecta y cuenta escasas muestras bacterianas tiene muchas aplicaciones en los alimentos, bebidas y la industria farmacéutica, diagnóstico médico y para la detección de vida por las misiones robóticas a otros planetas y lunas del sistema solar. En la actualidad, se cuentan escasas muestras bacterianas por microscopia de la galjanoplastia o epifluorescencia de cultura. Placas requieren largos tiempos de incubación (días a semanas) y microscopía de epifluorescencia requiere extensa de tinción y concentración de la muestra. Aquí, demostramos cómo se utiliza la microscopia holográfica digital fuera del eje (DHM) para enumerar bacterias en cultivos muy diluidas (100-104 células/mL). En primer lugar, se discute la construcción de la DHM personalizado, junto con instrucciones detalladas sobre la construcción de un instrumento de bajo costo. Se discuten los principios de la holografía, y se utiliza un modelo estadístico para estimar cuánto deberían videos para detectar células, basadas en las características de rendimiento óptico del instrumento y la concentración de la solución bacteriana (tabla 2) . Video detección de células a 105, 104103y 100 células/mL se demuestra en tiempo real utilizando hologramas no reconstruidos. Reconstrucción de imágenes de amplitud y fase se demuestra utilizando un paquete de software de código abierto.
Introducción
Determinación de la exacta conteos bacterianos en muestras muy diluidas es crucial en muchas aplicaciones: algunos ejemplos son alimentos y agua calidad análisis1,2,3; detección de patógenos en sangre, líquido cefalorraquídeo o esputo4,5; producción de productos farmacéuticos, incluyendo agua estéril6; y análisis de la comunidad ambiental en ambientes oligotróficos como en el abierto océano y los sedimentos7,8,9. Existe también un creciente interés en la detección de posible vida microbiana existente en las lunas heladas de Júpiter y Saturno, particularmente Europa10,11 y Encélado12,13, 14, que son conocidos por tener océanos líquidos subsuperficiales. Porque ninguna misión desde Viking en 1978 ha intentado encontrar existe vida en otro planeta, se ha limitado el desarrollo de tecnologías e instrumentos para la identificación bacteriana y conteo durante misiones de espacio15.
Los métodos tradicionales de gérmenes encuentran solamente las células cultivables, que pueden representar una minoría de la especie en las cepas ambientales, a veces < 1%16. Las placas requieren días o semanas de incubación para el máximo éxito, dependiendo de la cepa. Microscopía de epifluorescencia ha substituido en gran parte placa cuenta como estándar de oro para el recuento microbiano rápido y preciso. Etiquetado de ácido nucleico tintes fluorescentes como 4′, diclorhidrato 6-diamidino-2-phenylindole (DAPI), SYBR Green o naranja de acridina que se unen a los ácidos nucleicos son los típicos colorantes usados17,18,19 , aunque muchos estudios utilizan indicadores fluorescentes de Gram muestra20,21,22,23,24. Utilizando estos métodos sin previa concentración conduce a límites de detección (LoDs) de ~ 105 células por mL. Mejoras en LoD son posibles mediante filtración. Una muestra de líquido es filtrado a vacío sobre una membrana, generalmente policarbonato e idealmente negro para reducir el fondo. Bajo fondo colorantes tales como las manchas de ADN mencionadas pueden aplicarse directamente al filtro25. Para cuenta exacta por el ojo, ~ 105 células requiere por filtro, lo que significa que para las muestras más diluidas que ~ 105 células por mL, volúmenes de muestra significativa deben ser recogidos y filtrados. Dispositivos de escaneo láser se han desarrollado con el fin de explorar sistemáticamente todas las regiones del filtro y así reducir el número de células necesario para contar, empujando los límites de detección hasta 10 ~2 células por mL26. Sin embargo, estos no están disponibles en la mayoría de los laboratorios y requieren sofisticado hardware así como software que permitan la confirmación de expertos que observó las partículas es bacterias y no desechos.
Para referencia, adultos con sepsis generalmente comienzan con síntomas en < 100 células/mL de sangre y los bebés en < 10 células/mL. 10 mL, lleva un empate de sangre de un adulto y un niño, 1 mL. Métodos basados en PCR son inhibidos por la presencia de flora no patógena humana ADN y componentes inhibidores de PCR en la sangre de27,28. A pesar de una variedad de técnicas emergentes, las culturas siguen siendo el gold standard para el diagnóstico de infecciones del torrente sanguíneo, especialmente en las zonas más rurales o países en desarrollo. Para la detección de vida en otros planetas, cálculos termodinámicos pueden estimar el presupuesto de energía para la vida y por lo tanto la biomasa esperada posible. 1 - 100 células/mL se espera que sean termodinámicamente razonable en Europa29. Se puede fácilmente ver desde estos números que la detección de muy pequeñas cantidades de células en grandes cantidades de solución acuosa es un importante problema sin resolver.
En este trabajo demostramos la detección de marcescens del Serratia y Shewanella oneidensis (mutante de tipo salvaje y no-motile) en concentraciones de 105104103y 100 células/mL mediante un eje digital microscopio holográfico (DHM). La ventaja clave del DHM por microscopía óptica tradicional es la proyección de imagen simultánea de un volumen grueso de la muestra a alta resolución: en esta implementación, la cámara de la muestra fue de 0,8 mm de espesor. Estas cámaras de muestra fueron construidas por la litografía suave de polidimetilsiloxano (PDMS) de un molde de aluminio de precisión con una tolerancia de ± 50 μm. Esto representa una mejora de aproximadamente 100-fold en profundidad de campo sobre microscopía de luz de alta potencia. DHM también proporciona información de la fase cuantitativa, que permite las mediciones de la longitud del camino óptico (producto del índice de refracción y el espesor). DHM y técnicas similares han sido utilizadas para el control bacteriano y ciclo celular de la levadura y cálculo de masa seca bacteriana30,31,32; las diferencias de dispersión pueden utilizarse incluso para diferenciar cepas bacterianas33.
El instrumento que utilizamos es a la medida específicamente para uso con microorganismos, previamente publicados34,35, y su diseño y construcción se demostró y se discuten. Soluciones acuosas son suministradas continuamente a una cámara de muestra de volumen 0.25 μl a través de la bomba de jeringa; el caudal está determinado por la velocidad de fotogramas de la cámara para asegurar la proyección de imagen del volumen muestra todo. Un cálculo estadístico predice el número de volúmenes de muestra que debe ser reflejada con el fin de detectar un número significativo de células a una concentración dada.
Para aplicaciones de detección celular, reconstrucción de los hologramas en amplitud y fase de imágenes no fue requerida; Análisis fue realizado en el holograma crudo. Esto ahorra importantes recursos computacionales y espacio en disco: un holograma de 500 Mb video será 1 a 2 Tb cuando reconstruido. Sin embargo, discutir la reconstrucción a través de la profundidad de la muestra para confirmar que los hologramas representan la especie deseada. Una característica importante de DHM es su capacidad para monitorear la intensidad y fase de las imágenes. Organismos que son casi transparentes en intensidad (como la mayoría de las células biológica) aparecen claramente en fase. Como es una técnica libre de etiqueta, no tintes se utilizan. Se trata de un undvantage para aplicaciones de vuelo espacial posible, ya que los tintes no pueden sobrevivir las condiciones de una misión y — más importante — no puede ser asumido para trabajar con organismos extraterrestres, que no pueden usar ADN o ARN para codificar. También es una ventaja para el trabajo en ambientes extremos como el Ártico y el Antártico, donde los tintes pueden ser difícil de llevar a la ubicación remota y pueden degradar al almacenamiento. Reconstrucción de imágenes en fase y amplitud se realiza utilizando un paquete de software de código abierto que hemos hecho disponibles en GitHub (champú) o ImageJ.
Protocolo
1. crecimiento y enumeración de bacterias
Nota: esto es aplicable a casi cualquier cepa bacteriana en el medio adecuado 36. En nuestro ejemplo, utilizamos tres cepas: Serratia marcescens como una cepa de laboratorio común, fácil identificación; y una cepa ambiental más pequeña, altamente móvil, Shewanella oneidensis MR-1. Para comparar la detección de móviles frente a las células no-motile, un no-motile Shewanella mutante, Δ FlgM, también se utiliza para comparación 37. Todas las cepas se cultivan en caldo de lisogenia (LB).
- Prepare medio estéril de LB (por litro de agua destilada: 10 g bacto triptona, 5 g de bacto levadura, 10 g de NaCl; filtro o autoclave). Preparación estándar de 100 mm diámetro LB-agar placas (misma receta como medio más agar 15 g por litro, autoclave (121 ° C, 20 min) para esterilizar y servir cuando esté fría para manejar). Almacenar medio y placas en el refrigerador.
- El día antes del experimento de la semilla 5-6 mL de " amo " medio de cultivo de una colonia bacteriana fresca, utilizando técnica estéril y bioseguridad correcta. Incubar en un agitador (120 rpm) a 30 ° C tiempo de incubación de ~ 12 h. será dependiente de la cepa y tasa de crecimiento. En nuestros experimentos, las cepas de Shewanella se incuban durante 12 horas; sin embargo, Serratia se cosecha después de 8 horas, para asegurar la cultura expondrá crecimiento medio logarítmicos.
- El día del experimento, tomar una lectura espectrofotométrica (OD 600) de las bacterias dominar la cultura, que se espera que en el rango de 0.6 a 0.7. Debe estar en la fase de crecimiento logarítmico (según lo determinado previamente). Si no, el subcultivo 100 μl a 5 mL de medio y crecimiento de la célula a la fase de registro medio.
- Toma una muestra del maestro de la cultura y contar las células de la cámara directamente a través de un recuento de Petroff-Hausser. Esta enumerar las células vivas y muertas.
- Extraer una muestra de 10 μl de la cultura sin diluir con una micropipeta y transferencia en la cámara de.
- Imagen bajo un microscopio objetivo alto seco (40 X ó 63 X, NA 0.7 - 0.8) con contraste de fase.
- Contar las bacterias en menos de 20 plazas y media.
Nota: Contar sólo las bacterias que están enteramente dentro de una plaza y sólo ésos cruzando la parte superior y límites izquierdos (o parte inferior y derecha, si lo prefiere). Si las plazas están separadas por varias líneas, elegir uno como un límite de. - Concentración de calcular como el promedio de 20 plazas x factor de dilución. Nota que cuenta no funcionan bien para las concentraciones de < 10 7 / mL.
- Hacer diluciones seriadas de la cultura para el recuento de Colonia formando unidades (UFC). Esto enumerar células vivas solo.
Solución
- hacer una dilución seriada de cada una de las muestras bacterianas seleccionadas con solución salina 0.9% estéril. Transferir 20 μl de la solución bacteriana y diluir con solución salina μl 180. Repita hasta que la concentración más baja es ~ 10 3 células/mL.
- Tomar 100 μl de por lo menos dos diluciones sugeridas es 10 3 y 10/ml 4 — y la placa en una placa de medio sólido adecuado. Extienda con un esparcidor estéril. Realizar al menos 3 réplicas de cada dilución.
- Incubar a una temperatura adecuada para sus bacterias durante la noche o hasta que las colonias crecen.
- Contar las colonias y calcular colonias formando unidades (UFC) según (Nº de colonias x factor de dilución) / volumen plateado = UFC/mL. Promedio de UFC en las repeticiones.
2. Preparación de muestras altamente diluido para DHM
- hacer diluciones seriadas del maestro de la cultura para DHM y post-DHM recuento de unidades formadoras de colonias en placas de medio LB (ver 1.4.1 - 1.4.4). Realizar esta doble ciego para que la persona que realiza las grabaciones no es consciente de la concentración en cada muestra medido.
- Diluir las bacterias en 10-15 mL de un medio mínimo que estimular la motilidad (según corresponda) sino que inhiben la división celular, por lo que la concentración de células no cambia apreciablemente durante el experimento. Esto puede ser solución salina al 0.9% o un medio de movilidad más específico. Por ejemplo, si utiliza Escherichia coli, medio para motilidad debe contener EDTA 38. Para estos ejemplos, se utiliza solución salina al 0.9%.
3. Grabar Videos de DHM
- utilizando una jeringa estéril, tire unos 10 ml de la dilución de interés.
- Conectar la jeringa a la cámara de la muestra de la DHM usando los accesorios estériles y tubos.
Nota: Una cámara de microfluidos por encargo fue construida para permitir el flujo constante de muestra a través de la trayectoria óptica del instrumento. Consulte la sección resultados para más detalles. Antes del experimento, todos los componentes de la cámara de muestras deben esterilizarse en autoclave. - Flujo de la muestra de la jeringa a través de la cámara de muestra continuamente utilizando una bomba de jeringa.
Nota: Las tasas de flujo apropiado varían dependiendo dimensiones canales fluídicos, rendimiento deseado, así como limitaciones de adquisición de datos. - Como la muestra se atraviesa la cámara de muestras, adquisición de hologramas consecutivamente a una velocidad de fotogramas adecuada. Se creará un archivo de sello de tiempo que registros el tiempo de cada imagen de captura para ser utilizado más adelante durante el análisis de datos (Protocolo de la sección 4). Obtener los hologramas a temperatura ambiente (23° C). Grabar hologramas ejecutando un archivo ejecutable del C++ escrito previamente proporcionado por el fabricante de la cámara.
Nota: Marco apropiado varía según el caudal elegido. Para este experimento, se recomienda utilizar una velocidad de fotogramas que una bacteria sería reflejada > 2 veces como viajó a través del instrumento ' s campo de vista. - Permitir tiempo suficiente para la entera 10 mL de muestra a ser atravesado el DHM.
- Para garantizar que las bacterias no están creciendo o muriendo durante los experimentos, inoculan enriquecido las placas de los medios de comunicación con 100 μl de la captura de imagen gastado media post-DHM. Con un esparcidor estéril e incubar a temperatura adecuada por 24 horas; contar las colonias presentes.
4. Cálculo de la densidad celular y límites de detección
- con los videos de holograma adquirido, analizar los datos de presencia de bacterias.
- Calcular el valor del píxel mediana de una serie de hologramas de tiempo luego reste este valor medio de cada píxel correspondiente para eliminar artefactos fijos en el holograma. Esto se puede hacer en ImageJ realizando los siguientes pasos:
- convertir a 32-bit (bit de imagen-tipo-32)
- calcula la mediana (imagen-pila-Zproject-mediana)
- reste el promedio del resto de la pila ( Process-ImageCalculator-Subtract)
- cuenta el número de Anillos luminosos visibles y las células en el foco manualmente. En ImageJ, esto se hace apuntando a los objetos con la herramienta punto.
- Cada serie de hologramas irá acompañado de un archivo de sello de tiempo (registrado en el momento de la adquisición del holograma). Uso el archivo de sello de tiempo para calcular la cantidad total de muestra bombeado al tiempo la imagen fue capturado.
- Calcular el valor del píxel mediana de una serie de hologramas de tiempo luego reste este valor medio de cada píxel correspondiente para eliminar artefactos fijos en el holograma. Esto se puede hacer en ImageJ realizando los siguientes pasos:
- Calcular la densidad de la célula dividiendo el número total de células detectadas por el volumen total de la muestra fotografiada. Promedio de 5-10 cuadros de estadísticas precisas.
5. Amplitud y fase de la reconstrucción de la imagen
- elegir representante videos de 10-200 células por cuadro. Crudo (no restar mediana) hologramas de la carga en el software de reconstrucción (ejemplos: propagación numérica ImageJ 39; CHAMPÚ [GitHub]).
- Utilizar ImageJ, ir a difracción de propagación numérica numérica OD.
- En el símbolo del sistema, dibujar una máscara de Fourier para elegir la real o la imagen virtual y eliminar cualquier características de ruido sinusoidal.
- Reconstrucción de amplitud y fase en z-medidas adecuadas introduciendo la distancia reconstrucción.
- Mediana restar los datos de amplitud para reducir el ruido en 4.1.1.
- Datos 3D cubo o proyección. Ir a Plugins — volumen espectador.
Resultados
Los resultados deben indicar la capacidad para detectar las bacterias vivas y muertas en niveles muy bajos de DHM. El número de bacterias contadas debe ser coherente con los resultados obtenidos utilizando las cuentas Petroff-Hauser cuentas cámara y placa. Estándar métodos estadísticos proporcionan información sobre la exactitud de los métodos de detección distintos a diferentes concentraciones bacterianas.
Discusión
Reconstrucción numérica de hologramas: la reconstrucción numérica de hologramas, se utiliza el método de espectro angular (ASM). Se trata de la circunvolución del holograma con la función de Green para el DHM. El frente de onda compleja de la imagen en un determinado plano focal se puede calcular empleando el teorema de la circunvolución de Fourier como sigue:
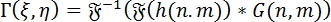 (1)
(1)
Donde
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores reconocen el Gordon y Betty Moore Foundation Becas 4037 y 4038 a la California Institute of Technology para la financiación de este trabajo.
Materiales
| Name | Company | Catalog Number | Comments |
| Bacto Yeast | BD Biosciences | 212750 | |
| Bacto Tryptone | BD Biosciences | 211705 | |
| Sodium chloride | Sigma-Aldrich | 7710 | Many options for purchase |
| Bacto Agar | BD Biosciences | 214010 | |
| 10 cm Petri dishes | VWR | 10053-704 | |
| 15 mL culture tubes | Falcon (Corning Life Sciences) | 352002 | Loose-capped |
| Petroff-Hauser chamber | Electron Microscopy Sciences | 3920 | |
| 10 mL syringes | BD Biosciences | 309604 | Luer-Lok tip not necessary |
| Male Luer to 1/16” barbed fitting | McMaster-Carr | 51525K291 | |
| Autoclavable 1/16” ID PVC tubing for flow | Nalgene | 8000-0004 | Sold by length, purchase accordingly |
| Syringe pump | Harvard Apparatus | PHD 2000 | |
| Sample Chamber | Custom | n/a | See Materials Section |
| Holographic Microscope | Custom | n/a | See Wallace et al. |
| Open-source software | Custom | https://github.com/bmorris3/shampoo |
Referencias
- Akineden, O., Weirich, S., Abdulmawjood, A., Failing, K., Buelte, M. Application of a Fluorescence Microscopy Technique for Detecting Viable Mycobacterium avium ssp. paratuberculosis Cells in Milk. Food Anal. Methods. 8, 499-506 (2015).
- Deibl, J., Paulsen, P., Bauer, F. Rapid enumeration of total aerobic microbial counts on meat and in meat products by means of the direct epifluorescence filter technique. Wien Tierarztl Monat. 85, 327-333 (1998).
- Huang, J., Li, Y., Slavik, M. F., Tao, Y., Huff, G. R. Identification and enumeration of Salmonella on sample slides of poultry carcass wash water using image analysis with fluorescent microscopy. Transactions of the Asae. 42, 267-273 (1999).
- Durtschi, J. D., et al. Increased sensitivity of bacterial detection in cerebrospinal fluid by fluorescent staining on low-fluorescence membrane filters. J Med Microbiol. 54, 843-850 (2005).
- Panicker, R. O., Soman, B., Saini, G., Rajan, J. A Review of Automatic Methods Based on Image Processing Techniques for Tuberculosis Detection from Microscopic Sputum Smear Images. J Med Syst. 40, (2016).
- Riepl, M., et al. Applicability of solid-phase cytometry and epifluorescence microscopy for rapid assessment of the microbiological quality of dialysis water. Nephrol Dial Transplant. 26, 3640-3645 (2011).
- Hwang, C. Y., Cho, B. C. Virus-infected bacteria in oligotrophic open waters of the East Sea, Korea. Aquat Microb Ecol. 30, 1-9 (2002).
- Kepner, R. L., Pratt, J. R. Use of Fluorochromes for Direct Enumeration of Total Bacteria in Environmental-Samples - Past and Present. Microbiol Rev. 58, 603-615 (1994).
- Queric, N. V., Soltwedel, T., Arntz, W. E. Application of a rapid direct viable count method to deep-sea sediment bacteria. J Microbiol Meth. 57, 351-367 (2004).
- Prieto-Ballesteros, O., Vorobyova, E., Parro, V., Rodriguez Manfredi, J. A., Gomez, F. Strategies for detection of putative life on Europa. Adv Space Res. 48, 678-688 (2011).
- Gleeson, D. F., et al. Biosignature Detection at an Arctic Analog to Europa. Astrobiology. 12, 135-150 (2012).
- Carr, C. E., Zuber, M. T., Ruvkun, G., Ieee, . 2013 Ieee Aerospace Conference IEEE Aerospace Conference Proceedings. , (2013).
- Konstantinidis, K., et al. A lander mission to probe subglacial water on Saturn's moon Enceladus for life. Acta Astronautica. 106, 63-89 (2015).
- McKay, C. P., Anbar, A. D., Porco, C., Tsou, P. Follow the Plume: The Habitability of Enceladus. Astrobiology. 14, 352-355 (2014).
- Hoover, R. B., Hoover, R. B., Levin, G. V., Rozanov, A. Y., Wickramasinghe, N. C. . Instruments, Methods, and Missions for Astrobiology Xvii. , (2015).
- Handelsman, J. Metagenomics: application of genomics to uncultured microorganisms. Microbiol Mol Biol Rev: MMBR. 68, 669-685 (2004).
- Lebaron, P., Parthuisot, N., Catala, P. Comparison of blue nucleic acid dyes for flow cytometric enumeration of bacteria in aquatic systems. Appl Environ Microbiol. 64, 1725-1730 (1998).
- Marie, D., Partensky, F., Jacquet, S., Vaulot, D. Enumeration and cell cycle analysis of natural populations of marine picoplankton by flow cytometry using the nucleic acid stain SYBR Green I. Appl Environ Microbiol. 63, 186-193 (1997).
- Noble, R. T., Fuhrman, J. A. Use of SYBR Green I for rapid epifluorescence counts of marine viruses and bacteria. Aquat Microb Ecol. 14, 113-118 (1998).
- Forster, S., Snape, J. R., Lappin-Scott, H. M., Porter, J. Simultaneous fluorescent gram staining and activity assessment of activated sludge bacteria. Appl Environ Microbiol. 68, 4772-4779 (2002).
- Lauer, B. A., Reller, L. B., Mirrett, S. Comparison Of Acridine-Orange And Gram Stains For Detection Of Microorganisms In Cerebrospinal-Fluid And Other Clinical Specimens. J Clin Microbiol. 14, 201-205 (1981).
- Mason, D. J., Shanmuganathan, S., Mortimer, F. C., Gant, V. A. A fluorescent gram stain for flow cytometry and epifluorescence microscopy. Appl Environ Microbiol. 64, 2681-2685 (1998).
- Saida, H., Ytow, N., Seki, H. Photometric application of the Gram stain method to characterize natural bacterial populations in aquatic environments. Appl Environ Microbiol. 64, 742-747 (1998).
- Sizemore, R. K., Caldwell, J. J., Kendrick, A. S. Alternate Gram Staining Technique Using A Fluorescent Lectin. Appl Environ Microbiol. 56, 2245-2247 (1990).
- Bitton, G., Dutton, R. J., Foran, J. A. New Rapid Technique For Counting Microorganisms Directly On Membrane Filters. Stain Technology. 58, 343-346 (1983).
- Broadaway, S. C., Barton, S. A., Pyle, B. H. Rapid staining and enumeration of small numbers of total bacteria in water by solid-phase laser cytometry. Appl Environ Microbiol. 69, 4272-4273 (2003).
- Yagupsky, P., Nolte, F. S. Quantitative aspects of septicemia. Clin Microbiol Rev. 3, 269-279 (1990).
- Kang, D. K., et al. Rapid detection of single bacteria in unprocessed blood using Integrated Comprehensive Droplet Digital Detection. Nat Commun. 5, 5427 (2014).
- Hand, K. P. . Report of the Europa Lander Science Definition Team. , (2017).
- Popescu, G., et al. Optical imaging of cell mass and growth dynamics. Am J Physiol-Cell Physiol. 295, C538-C544 (2008).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci U S A. 108, 13124-13129 (2011).
- Rappaz, B., et al. Noninvasive characterization of the fission yeast cell cycle by monitoring dry mass with digital holographic microscopy. J.Biomed Opt. 14, 034049 (2009).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Opt. Express. 23, 15792-15805 (2015).
- Wallace, J. K., et al. Robust, compact implementation of an off-axis digital holographic microscope. Opt. Express. 23, 17367-17378 (2015).
- Lindensmith, C. A., et al. A Submersible, Off-Axis Holographic Microscope for Detection of Microbial Motility and Morphology in Aqueous and Icy Environments. Plos One. 11, e0147700 (2016).
- Dumas, E. M., Ozenne, V., Mielke, R. E., Nadeau, J. L. Toxicity of CdTe Quantum Dots in Bacterial Strains. IEEE Trans. NanoBiosci. 8, 58-64 (2009).
- Frisk, A., Jyot, J., Arora, S. K., Ramphal, R. Identification and functional characterization of flgM, a gene encoding the anti-sigma 28 factor in Pseudomonas aeruginosa. J Bacteriol. 184, 1514-1521 (2002).
- Adler, J., Templeton, B. The effect of environmental conditions on the motility of Escherichia coli. Journal Gen Microbiol. 46, 175-184 (1967).
- Piedrahita-Quintero, P., Castaneda, R., Garcia-Sucerquia, J. Numerical wave propagation in Image J. Appl Opt. 54, 6410-6415 (2015).
- Schnars, U., Jüptner, W. P. Digital recording and numerical reconstruction of holograms. Meas. Sci. Technol. 13, R85 (2002).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados