È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Quantificare i microrganismi alle concentrazioni basse usando microscopia digitale oleografica (DHM)
In questo articolo
Riepilogo
Microscopia digitale oleografica (DHM) è una tecnica volumetrica che permette immagini campioni che 50-100 X più spessa di microscopia in campo chiaro a risoluzione paragonabile, con messa a fuoco eseguita post-elaborazione. Qui DHM viene utilizzato per l'identificazione, conteggio e microrganismi di rilevamento molto bassa densità e confrontato con misurazioni di densità ottica, germi e diretto.
Abstract
Con precisione rilevando e contando sparsi campioni batterici ha molte applicazioni nel cibo, bevanda e le industrie di trasformazione farmaceutica, nella diagnostica medica e per il rilevamento di vita tramite missioni robotiche di altri pianeti e lune del sistema solare. Attualmente, radi campioni batterici sono contati da microscopia di placcatura o epifluorescenza di cultura. Piastre di coltura richiedono lunghi tempi di incubazione (giorni o settimane), ed epifluorescenza richiede vasta macchiatura e concentrazione del campione. Qui, noi dimostrare come utilizzare fuori asse microscopia digitale oleografica (DHM) per enumerare i batteri nelle colture molto diluite (100-104 cellule/mL). In primo luogo, la costruzione del DHM personalizzato è discussa, insieme a istruzioni dettagliate sulla creazione di uno strumento di basso costo. I principi dell'olografia sono discussi, e viene utilizzato un modello statistico per stimare quanto tempo video dovrebbe essere quello di individuare le cellule, basate sulle caratteristiche prestazioni ottiche dello strumento e la concentrazione della soluzione batterica (tabella 2) . Video di rilevamento delle cellule alle 105, 104, 103e 100 cellule/mL è dimostrato in tempo reale utilizzando gli ologrammi non ricostruiti. Ricostruzione di immagini di ampiezza e fase è dimostrata utilizzando un pacchetto di software open-source.
Introduzione
Determinazione delle conte batteriche accurate in campioni molto diluite è cruciale in molte applicazioni: alcuni esempi sono acqua e cibo qualità analisi1,2,3; rilevamento di agenti patogeni nel sangue, liquido cerebrospinale o espettorato4,5; produzione di prodotti farmaceutici, tra cui acqua sterile6; e l'analisi di comunità ambientale in ambienti oligotrofici come le aperto oceano e sedimenti7,8,9. C'è anche un crescente interesse nell'individuazione di possibile esistente vita microbica sulle lune ghiacciate di Giove e Saturno, particolarmente Europa10,11 ed Encelado12,13, 14, che sono noti per avere oceani liquidi sotto la superficie. Perché nessuna missione dal Viking nel 1978 ha tentato di trovare la vita ancora esistente su un altro pianeta, c'è stato limitato sviluppo di tecnologie e strumenti per identificazione batterica ed il conteggio durante spazio missioni15.
I metodi tradizionali di germi trovano solo le celle coltivabili, che possono rappresentare una minoranza di specie in ceppi ambientali, a volte < 1%16. Piatti richiedono giorni o settimane di incubazione per il massimo successo, dipendendo il ceppo. Epifluorescenza in gran parte ha sostituito conta su piastra come il gold standard per l'enumerazione microbica rapida e accurata. Contrassegno dell'acido nucleico--coloranti fluorescenti quali 4 ′, 6-diamidino-2-phenylindole dicloridrato (DAPI), SYBR Green o arancio di acridina che si legano agli acidi nucleici sono il tipico coloranti utilizzati17,18,19 , anche se molti studi utilizzano indicatori fluorescenti di grammo Iscriviti20,21,22,23,24. Utilizzando questi metodi senza passaggi di pre-concentrazione conduce ai limiti di rilevabilità (LOD) di ~ 105 cellule per mL. Miglioramenti in LoD sono possibili mediante filtrazione. Un campione liquido è vuoto-filtrata su una membrana, solitamente in policarbonato e idealmente nero per ridurre di fondo. Basso-sfondo coloranti come le macchie di DNA cui sopra possono essere applicate direttamente al filtro25. Per conteggio preciso dall'occhio, cellule5 ~ 10 sono necessari per ciascun filtro, che significa che per i campioni più diluiti rispetto alle cellule di5 ~ 10 millilitri, volumi di campione significativo devono essere raccolto e filtrati. Dispositivi di scansione laser sono stati sviluppati al fine di esplorare sistematicamente tutte le regioni del filtro e quindi ridurre il numero di cellule necessarie per il conteggio, spingendo i limiti di rilevazione fino a ~ 102 cellule per mL26. Tuttavia, questi non sono disponibili nella maggior parte dei laboratori e richiedono hardware sofisticato così come software che consentono esperta conferma che osservate particelle è batteri e non residui.
Per riferimento, adulti con sepsi solitamente cominciano a mostrare sintomi alle < 100 cellule/mL di sangue e neonati a < 10 cellule/mL. Un prelievo di sangue da un adulto prende 10 mL e da un infante, 1 mL. Metodi basati sulla PCR sono inibiti dalla presenza della flora umana e non patogeni del DNA e di componenti d'inibizione PCR nel sangue27,28. Nonostante una varietà di tecniche emergenti, culture rimangano il gold standard per la diagnosi di infezioni del torrente sanguigno, soprattutto nelle zone più rurali o nazioni in via di sviluppo. Per la rilevazione della vita su altri pianeti, calcoli termodinamici possono stimare il bilancio di energia per la vita e, quindi, la biomassa possibile prevista. 1 - 100 cellule/mL devono essere termodinamicamente ragionevole su Europa29. Può essere facilmente visto da questi numeri che la rilevazione di molto piccolo numero di cellule in grandi quantità di soluzione acquosa è un importante problema irrisolto.
In questa carta, dimostriamo rilevamento dei marcescens del Serratia e Shewanella oneidensis (wild-type e non motili mutante) alle concentrazioni di 105, 104, 103e 100 cellule/mL utilizzando un fuori-asse microscopio digitale olografica (DHM). Il vantaggio chiave di DHM su microscopia tradizionale è la formazione immagine simultanea di un volume di campione di spessore ad alta risoluzione — in questa implementazione, la camera del campione era 0,8 mm di spessore. Queste camere di campioni sono state costruite dal soft-Litografia di polidimetilsilossano (PDMS) da uno stampo di alluminio lavorati di precisione con una tolleranza di ± 50 µm. Questo rappresenta un miglioramento di circa 100 volte in profondità di campo nel corso di microscopia ad alta potenza. DHM fornisce anche informazioni di fase quantitativa, consentendo per le misure di lunghezza del cammino ottico (prodotto di indice di rifrazione e spessore). DHM e tecniche simili sono stati utilizzati per il monitoraggio batterico e ciclo delle cellule di lievito e calcolo di batterica in massa secca30,31,32; differenze di dispersione possono anche essere usate per differenziare ceppi batterici33.
Lo strumento che usiamo è costruito su misura specificamente per l'utilizzo con microrganismi, come precedentemente pubblicati34,35, e la costruzione e sono dimostrati e discussi. Soluzioni acquose sono continuamente fornite a un 0,25 µ l volume pozzetto di misurazione tramite pompa a siringa; la portata è determinata dalla frequenza di fotogrammi fotocamera al fine di garantire la formazione immagine del volume intero campione. Un calcolo statistico predice il numero di volumi di campione che deve essere imaged al fine di rilevare un numero significativo di cellule ad una determinata concentrazione.
Per applicazioni di rilevamento delle cellule, ricostruzione di ologrammi in ampiezza e fase di immagini non è stato richiesto; analisi è stata eseguita sull'ologramma crudo. Ciò consente di risparmiare spazio su disco e risorse computazionali significative: un ologramma di 500 Mb video sarà 1-2 Tb quando ricostruito. Tuttavia, discutiamo la ricostruzione attraverso la profondità del campione per confermare che gli ologrammi rappresentano la specie desiderata. Una caratteristica importante del DHM è la sua capacità di monitorare l'intensità e la fase delle immagini. Organismi che sono quasi trasparenti in intensità (come la maggior parte delle cellule biologica) appaiono chiaramente in fase. Come è una tecnica privo di etichetta, coloranti non vengono utilizzati. Si tratta di un undvantage per applicazioni di volo spaziale possibile, dato che coloranti potrebbero non sopravvivere le condizioni di una missione e — soprattutto — non può essere assunto a lavorare con organismi extraterrestri, che non possono utilizzare DNA o RNA per la codifica. È anche un vantaggio per il lavoro in ambienti estremi come l'Artico e Antartico, dove coloranti possono essere difficili da portare nella posizione remota e possono peggiorare durante la conservazione. Ricostruzione di immagini in fase e in ampiezza viene eseguita utilizzando un pacchetto di software open-source che abbiamo reso disponibile su GitHub (SHAMPOO) o ImageJ.
Protocollo
1. crescita e l'enumerazione dei batteri
Nota: questo è applicabile a quasi qualsiasi ceppo batterico cresciuto nel medio appropriato 36. Nel nostro esempio, usiamo tre ceppi: i marcescens del Serratia come un ceppo comune, facile laboratorio identificabili; e un più piccolo ceppo ambientale altamente motile, Shewanella oneidensis MR-1. Per confrontare il rilevamento di motili vs cellule non motili, non motili Shewanella mutante, Δ FlgM, è anche utilizzato per confronto 37. Tutti i ceppi sono coltivati in brodo (LB) di Lisogenesi.
- Preparare mezzo LB sterile (per litro di acqua distillata: 10 g bacto triptone, 5 g di lievito bacto, 10 g NaCl; filtro o autoclave). Preparare standard 100 mm piastre diametro LB-agar (stessa ricetta come media più agar 15 g per litro; autoclave (121 ° C, 20 min) per sterilizzare e versare quando abbastanza fresco da gestire). Conservare medium e piastre in frigorifero.
- Il giorno prima dell'esperimento semi 5-6 mL di " Master " terreno di coltura da una colonia batterica fresca, utilizzando la tecnica sterile e biosicurezza corretta. Incubare su un agitatore (120 giri/min) a 30 ° C per il tempo di incubazione ~ 12 h. sarà dipendono il tasso di deformazione e crescita. Nei nostri esperimenti, i ceppi di Shewanella vengono incubati per 12 ore; Tuttavia, Serratia è raccolto dopo 8 ore, per garantire la cultura sarà presente crescita metà-logaritmica.
- Il giorno dell'esperimento, prendere una lettura spettrofotometrica (OD 600) del batterico Master cultura, che dovrebbe essere nel range di 0,6 a 0,7. Dovrebbe essere in fase di crescita logaritmica (come determinato in precedenza). Se non, sottocultura 100 µ l in 5 mL di terreno e consentire la crescita delle cellule alla fase di Mid-log.
- Prendere un campione del maestro di cultura e contare le celle direttamente utilizzando un conteggio di Petroff-Hausser dell'alloggiamento. Questo verrà enumerare le cellule sia dal vivere che morte.
- Estrarre un campione di 10 µ l della coltura non diluito con una micropipetta e trasferimento nella camera.
- Immagine al microscopio obiettivo alta-a secco (40 X o 63 X, NA 0,7 - 0,8) con contrasto di fase.
- Contare i batteri in almeno 20 piazze e media.
Nota: Il conteggio solo quei batteri che sono interamente all'interno di un quadrato solo quelli incrocio sopra la parte superiore e i limiti sinistro (o fondo e diritto, se si preferisce). Se piazze sono separati da linee diverse, scegliere uno come un contorno. - Concentrazione calcola la media di 20 piazze x fattore di diluizione. Si noti che diretto conteggi non funzionano bene per le concentrazioni < 10 7 / mL.
- Effettuare diluizioni seriali della cultura per il conteggio di formazione di colonie (CFU) unità. Questo enumererà cellule vive solo.
Soluzione
- fare una diluizione seriale di ciascuno dei campioni batterici selezionati con soluzione fisiologica 0,9% sterile. Trasferire 20 µ l della soluzione batterica e diluirla con soluzione fisiologica 180 µ l. Ripetere fino a quando la concentrazione minima è 3 ~ 10 cellule/mL.
- Prendere 100 µ l di almeno due diluizioni-suggerito sono 10 3 e 10 4/ml — e la piastra su una piastra di supporto solido appropriato. Stendere con una spatola sterile. Eseguire almeno 3 repliche di ogni diluizione.
- Incubare a una temperatura adeguata per il tuo batteri durante la notte o fino a quando le colonie si sviluppano.
- Colonie di contare e calcolare unità formanti colonia (UFC) secondo (# delle colonie x fattore di diluizione) / volume placcato = CFU/mL. Medio di CFU in replicati.
2. Preparazione di campioni altamente diluire per DHM
- fare diluizioni seriali del Maestro della coltura per DHM e post-DHM conteggio delle unità formanti colonie su piastre di LB media (Vedi 1.4.1 - 1.4.4). Eseguire questa prova alla cieca, in modo che la persona che fa le registrazioni non è a conoscenza della concentrazione in ogni campione misurato.
- Diluire i batteri in 10-15 mL di un medium minimo che incoraggiare la motilità (a seconda dei casi), ma inibiscono la divisione cellulare, in modo che la concentrazione di cellule non cambia sensibilmente durante l'esperimento. Questo può essere un supporto più specifico di motilità o soluzione fisiologica 0,9%. Ad esempio, se utilizzando Escherichia coli, medie di motilità devono contenere EDTA 38. Per questi esempi, utilizziamo la soluzione fisiologica allo 0,9%.
3. La registrazione di video DHM
- utilizzando una siringa sterile, tirare in circa 10 mL della diluizione di interesse.
- Collegare la siringa al pozzetto di misurazione DHM utilizzando sterile raccordi e tubi.
Nota: Una camera microfluidica su misura è stata costruita al fine di consentire il flusso costante del campione attraverso il percorso ottico dello strumento. Vedere la sezione risultati per maggiori dettagli. Prima dell'esperimento, tutti i componenti della camera del campione devono essere sterilizzati in autoclave. - Flow il campione dalla siringa attraverso la camera di campionamento continuamente utilizzando una pompa a siringa.
Nota: Portate appropriato variano a seconda di dimensioni canale fluidico, velocità effettiva desiderata, nonché limitazioni di acquisizione dati. - Come il campione viene propagato attraverso la camera del campione, acquisire ologrammi consecutivamente ad un tasso di frame appropriato. Verrà creato un file di timestamp che registri il tempo di ogni immagine acquisire per essere usato più tardi durante l'analisi dei dati (protocollo sezione 4). Ottenere tutti gli ologrammi a temperatura ambiente (23° C). L'esecuzione di un pre-scritto file eseguibile C++ fornito dal produttore della fotocamera per registrare gli ologrammi.
Nota: Frame appropriato variano a seconda del tasso di flusso scelto. Per questo esperimento, si raccomanda di utilizzare un frame rate in modo tale che un batterio sarebbe essere imaged > 2 volte come ha viaggiato attraverso lo strumento ' campo visivo s. - Consentire tempo sufficiente per l'intero 10 mL di campione per essere propagata attraverso il DHM.
- Per garantire che i batteri non crescono o morire durante gli esperimenti, inoculare arricchita piastre media con 100 µ l della cattura dell'immagine post-DHM speso media. Stendere con una spatola sterile e incubare a temperatura appropriata per 24 ore; contare le colonie presenti.
4. Calcolo della densità delle cellule e limiti di rilevabilità
- con i video acquisiti ologramma, analizzare i dati per la presenza di batteri.
- Calcolare il valore mediano pixel per una serie temporale di ologrammi quindi sottrarre questo valore mediano da ogni pixel rispettivi per eliminare gli artefatti stazionari nell'ologramma. Questo può essere fatto in ImageJ attenendosi alla procedura seguente:
- convertire a 32-bit (immagine-tipo - 32bit)
- Calcola la mediana (immagine-Stack-Zproject-mediana)
- sottrarre la mediana dal resto della pila ( Process-ImageCalculator-Subtract)
- contare il numero di anelli ariosi visibili e cellule di messa a fuoco manualmente. In ImageJ, questo è fatto puntando verso gli oggetti con lo strumento punto.
- Ogni serie di ologrammi si affiancherà un file timestamp (registrato al momento dell'acquisizione di ologramma). Uso il file timestamp per calcolare l'importo totale del campione pompato al momento l'immagine è stato catturato.
- Calcolare il valore mediano pixel per una serie temporale di ologrammi quindi sottrarre questo valore mediano da ogni pixel rispettivi per eliminare gli artefatti stazionari nell'ologramma. Questo può essere fatto in ImageJ attenendosi alla procedura seguente:
- Calcolare la densità delle cellule dividendo il numero totale di celle rilevate dal volume totale del campione imaged. Media 5-10 fotogrammi per statistiche accurate.
5. Ricostruzione di ampiezza e fase di immagine
- Scegli video rappresentativi con le cellule di 10-200 per ogni frame. Caricare il software di ricostruzione crudi (non mediana-sottratto) ologrammi (esempi: propagazione numerica ImageJ 39; SHAMPOO [GitHub]).
- Usando ImageJ, vai alla diffrazione di propagazione-numerica numerica OD.
- Al prompt, disegnare una maschera di Fourier per scegliere i reali o immagine virtuale ed eliminare tutte le funzioni sinusoidali rumore.
- Ricostruire ampiezza e fase z-procedura appropriata inserendo la distanza ricostruzione.
- Mediana sottrarre i dati di ampiezza per ridurre il rumore come in 4.1.1.
- Dati di trama come un 3D cubo o proiezione. Vai al plugin — Volume Viewer.
Risultati
I risultati dovrebbero indicare la capacità di rilevare batteri vivi e morti a livelli molto bassi di DHM. Il numero di batteri contati deve essere coerenza con i risultati ottenuti con i conteggi di camera e piastra conteggio Petroff-Hauser. Metodi statistici standard forniscono informazioni circa la precisione dei metodi di rilevazione diverse a varie concentrazioni batteriche.
La figura 1 Mostra...
Discussione
Ricostruzione digitale di ologrammi: per la ricostruzione numerica degli ologrammi, viene utilizzato il metodo di spettro angolare (ASM). Ciò comporta la convoluzione dell'ologramma con funzione di Green per DHM. Il fronte d'onda complesso dell'immagine a un particolare piano focale può essere calcolato utilizzando il teorema di convoluzione di Fourier come segue:
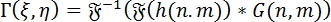 (1)
(1)
Dove
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Gli autori riconoscono la Gordon e Betty Moore Foundation sovvenzioni 4037 e 4038 al California Institute of Technology per il finanziamento di questo lavoro.
Materiali
| Name | Company | Catalog Number | Comments |
| Bacto Yeast | BD Biosciences | 212750 | |
| Bacto Tryptone | BD Biosciences | 211705 | |
| Sodium chloride | Sigma-Aldrich | 7710 | Many options for purchase |
| Bacto Agar | BD Biosciences | 214010 | |
| 10 cm Petri dishes | VWR | 10053-704 | |
| 15 mL culture tubes | Falcon (Corning Life Sciences) | 352002 | Loose-capped |
| Petroff-Hauser chamber | Electron Microscopy Sciences | 3920 | |
| 10 mL syringes | BD Biosciences | 309604 | Luer-Lok tip not necessary |
| Male Luer to 1/16” barbed fitting | McMaster-Carr | 51525K291 | |
| Autoclavable 1/16” ID PVC tubing for flow | Nalgene | 8000-0004 | Sold by length, purchase accordingly |
| Syringe pump | Harvard Apparatus | PHD 2000 | |
| Sample Chamber | Custom | n/a | See Materials Section |
| Holographic Microscope | Custom | n/a | See Wallace et al. |
| Open-source software | Custom | https://github.com/bmorris3/shampoo |
Riferimenti
- Akineden, O., Weirich, S., Abdulmawjood, A., Failing, K., Buelte, M. Application of a Fluorescence Microscopy Technique for Detecting Viable Mycobacterium avium ssp. paratuberculosis Cells in Milk. Food Anal. Methods. 8, 499-506 (2015).
- Deibl, J., Paulsen, P., Bauer, F. Rapid enumeration of total aerobic microbial counts on meat and in meat products by means of the direct epifluorescence filter technique. Wien Tierarztl Monat. 85, 327-333 (1998).
- Huang, J., Li, Y., Slavik, M. F., Tao, Y., Huff, G. R. Identification and enumeration of Salmonella on sample slides of poultry carcass wash water using image analysis with fluorescent microscopy. Transactions of the Asae. 42, 267-273 (1999).
- Durtschi, J. D., et al. Increased sensitivity of bacterial detection in cerebrospinal fluid by fluorescent staining on low-fluorescence membrane filters. J Med Microbiol. 54, 843-850 (2005).
- Panicker, R. O., Soman, B., Saini, G., Rajan, J. A Review of Automatic Methods Based on Image Processing Techniques for Tuberculosis Detection from Microscopic Sputum Smear Images. J Med Syst. 40, (2016).
- Riepl, M., et al. Applicability of solid-phase cytometry and epifluorescence microscopy for rapid assessment of the microbiological quality of dialysis water. Nephrol Dial Transplant. 26, 3640-3645 (2011).
- Hwang, C. Y., Cho, B. C. Virus-infected bacteria in oligotrophic open waters of the East Sea, Korea. Aquat Microb Ecol. 30, 1-9 (2002).
- Kepner, R. L., Pratt, J. R. Use of Fluorochromes for Direct Enumeration of Total Bacteria in Environmental-Samples - Past and Present. Microbiol Rev. 58, 603-615 (1994).
- Queric, N. V., Soltwedel, T., Arntz, W. E. Application of a rapid direct viable count method to deep-sea sediment bacteria. J Microbiol Meth. 57, 351-367 (2004).
- Prieto-Ballesteros, O., Vorobyova, E., Parro, V., Rodriguez Manfredi, J. A., Gomez, F. Strategies for detection of putative life on Europa. Adv Space Res. 48, 678-688 (2011).
- Gleeson, D. F., et al. Biosignature Detection at an Arctic Analog to Europa. Astrobiology. 12, 135-150 (2012).
- Carr, C. E., Zuber, M. T., Ruvkun, G., Ieee, . 2013 Ieee Aerospace Conference IEEE Aerospace Conference Proceedings. , (2013).
- Konstantinidis, K., et al. A lander mission to probe subglacial water on Saturn's moon Enceladus for life. Acta Astronautica. 106, 63-89 (2015).
- McKay, C. P., Anbar, A. D., Porco, C., Tsou, P. Follow the Plume: The Habitability of Enceladus. Astrobiology. 14, 352-355 (2014).
- Hoover, R. B., Hoover, R. B., Levin, G. V., Rozanov, A. Y., Wickramasinghe, N. C. . Instruments, Methods, and Missions for Astrobiology Xvii. , (2015).
- Handelsman, J. Metagenomics: application of genomics to uncultured microorganisms. Microbiol Mol Biol Rev: MMBR. 68, 669-685 (2004).
- Lebaron, P., Parthuisot, N., Catala, P. Comparison of blue nucleic acid dyes for flow cytometric enumeration of bacteria in aquatic systems. Appl Environ Microbiol. 64, 1725-1730 (1998).
- Marie, D., Partensky, F., Jacquet, S., Vaulot, D. Enumeration and cell cycle analysis of natural populations of marine picoplankton by flow cytometry using the nucleic acid stain SYBR Green I. Appl Environ Microbiol. 63, 186-193 (1997).
- Noble, R. T., Fuhrman, J. A. Use of SYBR Green I for rapid epifluorescence counts of marine viruses and bacteria. Aquat Microb Ecol. 14, 113-118 (1998).
- Forster, S., Snape, J. R., Lappin-Scott, H. M., Porter, J. Simultaneous fluorescent gram staining and activity assessment of activated sludge bacteria. Appl Environ Microbiol. 68, 4772-4779 (2002).
- Lauer, B. A., Reller, L. B., Mirrett, S. Comparison Of Acridine-Orange And Gram Stains For Detection Of Microorganisms In Cerebrospinal-Fluid And Other Clinical Specimens. J Clin Microbiol. 14, 201-205 (1981).
- Mason, D. J., Shanmuganathan, S., Mortimer, F. C., Gant, V. A. A fluorescent gram stain for flow cytometry and epifluorescence microscopy. Appl Environ Microbiol. 64, 2681-2685 (1998).
- Saida, H., Ytow, N., Seki, H. Photometric application of the Gram stain method to characterize natural bacterial populations in aquatic environments. Appl Environ Microbiol. 64, 742-747 (1998).
- Sizemore, R. K., Caldwell, J. J., Kendrick, A. S. Alternate Gram Staining Technique Using A Fluorescent Lectin. Appl Environ Microbiol. 56, 2245-2247 (1990).
- Bitton, G., Dutton, R. J., Foran, J. A. New Rapid Technique For Counting Microorganisms Directly On Membrane Filters. Stain Technology. 58, 343-346 (1983).
- Broadaway, S. C., Barton, S. A., Pyle, B. H. Rapid staining and enumeration of small numbers of total bacteria in water by solid-phase laser cytometry. Appl Environ Microbiol. 69, 4272-4273 (2003).
- Yagupsky, P., Nolte, F. S. Quantitative aspects of septicemia. Clin Microbiol Rev. 3, 269-279 (1990).
- Kang, D. K., et al. Rapid detection of single bacteria in unprocessed blood using Integrated Comprehensive Droplet Digital Detection. Nat Commun. 5, 5427 (2014).
- Hand, K. P. . Report of the Europa Lander Science Definition Team. , (2017).
- Popescu, G., et al. Optical imaging of cell mass and growth dynamics. Am J Physiol-Cell Physiol. 295, C538-C544 (2008).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci U S A. 108, 13124-13129 (2011).
- Rappaz, B., et al. Noninvasive characterization of the fission yeast cell cycle by monitoring dry mass with digital holographic microscopy. J.Biomed Opt. 14, 034049 (2009).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Opt. Express. 23, 15792-15805 (2015).
- Wallace, J. K., et al. Robust, compact implementation of an off-axis digital holographic microscope. Opt. Express. 23, 17367-17378 (2015).
- Lindensmith, C. A., et al. A Submersible, Off-Axis Holographic Microscope for Detection of Microbial Motility and Morphology in Aqueous and Icy Environments. Plos One. 11, e0147700 (2016).
- Dumas, E. M., Ozenne, V., Mielke, R. E., Nadeau, J. L. Toxicity of CdTe Quantum Dots in Bacterial Strains. IEEE Trans. NanoBiosci. 8, 58-64 (2009).
- Frisk, A., Jyot, J., Arora, S. K., Ramphal, R. Identification and functional characterization of flgM, a gene encoding the anti-sigma 28 factor in Pseudomonas aeruginosa. J Bacteriol. 184, 1514-1521 (2002).
- Adler, J., Templeton, B. The effect of environmental conditions on the motility of Escherichia coli. Journal Gen Microbiol. 46, 175-184 (1967).
- Piedrahita-Quintero, P., Castaneda, R., Garcia-Sucerquia, J. Numerical wave propagation in Image J. Appl Opt. 54, 6410-6415 (2015).
- Schnars, U., Jüptner, W. P. Digital recording and numerical reconstruction of holograms. Meas. Sci. Technol. 13, R85 (2002).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon