Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Quantification des microorganismes à des Concentrations faibles en utilisant la microscopie holographique numérique (DHM)
Dans cet article
Résumé
La microscopie holographique numérique (DHM) est une technique volumétrique qui permet aux échantillons d’imagerie 50-100 X plus épais que la microscopie fond clair à une résolution comparable, avec mise au point effectuée post-traitement. Ici DHM est utilisé pour l’identification, comptage et suivi des microorganismes à très faible densité et par rapport aux mesures de la densité optique, teneur en germes et dénombrement direct.
Résumé
Avec précision, détecter et compter les rares échantillons bactériens a de nombreuses applications dans les aliments, des boissons et des industries de transformation pharmaceutique, dans le domaine du diagnostic médical et pour la détection de la vie par les missions robotiques vers d’autres planètes et lunes du système solaire. Actuellement, les rares échantillons bactériens sont comptés par microscopie à épifluorescence ou de placage culture. Plaques de culture ont besoin de temps d’incubation fois (quelques jours ou semaines) et microscopie à épifluorescence requiert coloration étendue et la concentration de l’échantillon. Ici, nous montrent comment utiliser la microscopie holographique numérique hors-axe (DHM) pour énumérer les bactéries dans des cultures très diluées (100-104 cellules/mL). Tout d’abord, la construction de la DHM personnalisé est discutée, ainsi que des instructions détaillées sur la construction d’un instrument peu coûteux. Les principes de l’holographie sont discutés, et un modèle statistique est utilisé pour estimer combien de temps les vidéos devraient être de détecter des cellules, selon les caractéristiques de performances optiques de l’instrument et la concentration de la solution bactérienne (tableau 2) . Détection vidéo des cellules à 105, 104, 103et 100 cellules/mL est démontrée en temps réel à l’aide d’hologrammes non reconstituées. Reconstruction d’images d’amplitude et de phase est démontrée à l’aide d’un package de logiciels libres.
Introduction
Détermination de précise nombre de bactéries dans des échantillons très diluées est cruciale pour de nombreuses applications : quelques exemples sont l’eau et la nourriture qualité analyse1,2,3; détection de pathogènes dans le sang, liquide céphalo-rachidien ou expectorations4,5; production de produits pharmaceutiques, y compris l’eau stérile,6; et analyse de la communauté environnementale dans des milieux oligotrophes tels que l’ouverture océanique et les sédiments7,8,9. Il y aussi un intérêt croissant dans la détection de possibles formes de vie microbiennes existantes sur les lunes glacées de Jupiter et de Saturne, notamment Europa10,11 et Encelade12,13, 14, qui sont connus pour avoir des Océans liquides sous la surface. Parce qu’aucune mission depuis Viking en 1978 n’a tenté de trouver une vie existante sur une autre planète, il y a eu un développement limité des technologies et des instruments pour l’identification bactérienne et compter au cours de missions spatiales15.
Les méthodes traditionnelles de la teneur en germes trouver uniquement les cellules cultivables, qui peuvent représenter une minorité d’espèces dans les souches environnementales, parfois < 1 %16. Plaques nécessitent quelques jours ou semaines d’incubation pour une réussite maximale, selon la souche. Microscopie à épifluorescence a largement remplacé les dénombrements comme l’étalon-or pour dénombrement microbien rapide et précise. Acide nucléique-marquage fluorescents colorants tels que 4′, dichlorhydrate de 6-diamidino-2-phénylindole (DAPI), SYBR Green ou l’acridine orange qui se lient aux acides nucléiques sont les colorants typique utilisés17,18,19 , bien que de nombreuses études utilisent des indicateurs fluorescents de gramme signent20,21,22,23,24. Grâce à ces méthodes sans étapes de préconcentration mène aux limites de détection (LOD) ~ 105 cellules / mL. Améliorations de LoD sont possibles à l’aide de filtration. Un échantillon de liquide est filtré sous vide sur une membrane, généralement en polycarbonate et idéalement noir pour réduire le fond. Bas-fond colorants tels que les taches d’ADN mentionnés ci-dessus peuvent être appliquées directement sur le filtre25. Pour le comptage précis par oeil, 10 ~5 cellules sont tenus par le filtre, ce qui signifie que pour les échantillons plus diluées que ~ 105 cellules / mL, volumes d’échantillon significatif doivent être recueillies et filtrées. Laser à balayage ont été développées afin d’explorer systématiquement toutes les régions du filtre et ainsi réduire le nombre de cellules nécessaires pour le comptage, repousser les limites de détection vers le bas pour ~ 102 cellules par mL,26. Toutefois, ceux-ci ne sont pas disponibles dans la plupart des laboratoires et nécessitent un matériel sophistiqué ainsi que les logiciels permettant la confirmation expertise celui observé des particules sont bactéries et pas de débris.
Pour référence, adultes atteints de septicémie habituellement commencent à montrer des symptômes à < 100 cellules/mL de sang et les nourrissons à < 10 cellules/mL. Un tirage de sang d’un adulte a 10 mL et d’un nourrisson, 1 mL. Méthodes basées sur la PCR sont inhibées par la présence de la flore humaine et non pathogènes ADN et composants inhibant la PCR dans le sang27,28. Malgré la variété des techniques émergentes, les cultures restent l’étalon-or pour le diagnostic des septicémies, en particulier dans les zones plus rurales ou en voie de développement. Pour la détection de vie sur d’autres planètes, calculs thermodynamiques peuvent estimer le budget énergie pour la vie et donc de la biomasse possible attendue. 1 - 100 cellules/mL sont censés être thermodynamiquement raisonnable sur Europa29. Il peut être facilement vu de ces chiffres que la détection d’un très petit nombre de cellules de grandes quantités de solution aqueuse est un important problème non résolu.
Dans cet article, nous démontrons la détection de Serratia marcescens et Shewanella oneidensis (mutant sauvage et non mobile) à des concentrations de 105, 104, 103et 100 cellules/mL à l’aide d’un hors-axe microscope holographique numérique (DHM). Le principal avantage du DHM sur la microscopie traditionnelle est l’imagerie simultanée d’un volume d’échantillon épais à haute résolution — dans cette implémentation, la chambre de l’échantillon était de 0,8 mm d’épaisseur. Ces chambres de l’échantillon ont été construits par la douce-lithographie de polydiméthylsiloxane (PDMS) à partir d’un moule de précision en aluminium avec une tolérance de ± 50 µm. Cela représente une amélioration d’environ 100 fois en profondeur de champ sur la microscopie photonique haute puissance. DHM fournit également des informations de la phase quantitative, permettant des mesures de la longueur du trajet optique (produit de l’indice de réfraction et de l’épaisseur). DHM et des techniques similaires ont été utilisées pour la surveillance des bactéries et cycle de cellules de levure et calcul de la masse bactérienne sèche30,31,32; différences de diffusion peuvent même être utilisés pour différencier les souches bactériennes33.
L’instrument que nous utilisons est sur mesure spécifiquement pour une utilisation avec des microorganismes, comme précédemment publié34,35, et sa conception et sa construction sont démontrés et discutées. Solutions aqueuses sont fournies en permanence à une chambre de mesure de volume µL 0,25 par pousse-seringue ; le débit est déterminé par la fréquence d’images de caméra afin d’assurer l’imagerie volume de l’échantillon entier. Un calcul statistique permet de prédire le nombre de volumes d’échantillon qui doit être copié afin de détecter un nombre significatif de cellules à une concentration donnée.
Pour des applications de détection des cellules, la reconstruction des hologrammes dans l’amplitude et la phase des images n’était pas nécessaire ; analyse a été réalisée sur l’hologramme brute. Cela permet d’économiser des ressources informatiques importants et l’espace disque : un hologramme de 500 Mo vidéo sera de 1 à 2 to quand reconstruits. Toutefois, nous discutons reconstruction par profondeur de l’échantillon pour confirmer que les hologrammes représentent l’espèce désirée. Une caractéristique importante du DHM est sa capacité à contrôler l’intensité et la phase des images. Les organismes qui sont presque transparentes en intensité (comme la plupart des cellules biologiques) apparaissent clairement en phase. Comme il s’agit d’une technique exempte d’étiquette, aucun colorants ne sont utilisés. Il s’agit d’un unVantage pour des applications possibles de vol spatial, puisque les colorants ne peuvent pas survivre aux conditions d’une mission et — surtout — ne peut présumer de travailler avec des organismes extraterrestres, qui ne pourraient pas utiliser l’ADN ou ARN pour l’encodage. C’est aussi un avantage pour le travail dans des conditions extrêmes telles que l’Arctique et l’Antarctique, où les colorants peuvent être difficiles à apporter à l’emplacement distant et peuvent se dégrader après stockage. Reconstruction d’images en phase et en amplitude est effectuée à l’aide d’un package de logiciels open-source que nous proposons sur GitHub (shampooing) ou ImageJ.
Protocole
1. croissance et énumération des bactéries
Remarque : Ceci s’applique à presque n’importe quelle souche bactérienne cultivé dans le moyen approprié 36. Dans notre exemple, nous utilisons trois souches : Serratia marcescens comme étant une souche commune, easy lab identifiable ; et une souche environnementale plus petite et fortement motile, Shewanella oneidensis MR-1. Pour comparer la détection de motile vs cellules non mobile, non motiles Shewanella mutant, Δ FlgM, est également utilisé pour comparaison 37. Toutes les souches sont cultivées dans le bouillon (LB) de la lysogénie.
- Prepare stérile milieu LB (par litre d’eau distillée : 10 g bacto tryptone, 5 g de bacto levure, 10 g NaCl ; filtre ou autoclave). Préparer standard 100 mm plaques diamètre LB-agar (même recette que la moyenne plus agar 15 g par litre ; autoclave (121 ° C, 20 min) pour stériliser et verser lorsqu’elles sont suffisamment refroidies à gérer). Mettre les moyen et assiettes au réfrigérateur.
- La veille de l’expérience des graines 5 à 6 mL de " Master " milieu de culture provenant d’une colonie bactérienne fraîche, à l’aide de la technique correcte de stériles et de prévention des risques biotechnologiques. Incuber sur un agitateur (120 tr/min) à 30 ° C pour la période d’Incubation ~ 12 h. sera dépendante sur le taux de croissance et de la souche. Dans nos expériences, les souches de Shewanella sont incubés pendant 12 heures ; Cependant, Serratia est récoltée après 8 heures, pour s’assurer que la culture y exposera une croissance moyenne logarithmique.
- Le jour de l’expérience, prenez une lecture spectrophotométrique (OD 600) de la bactérie maîtriser la culture, qui devrait être de l’ordre de 0,6 à 0,7. Il devrait être dans la phase de croissance logarithmique (comme déterminé précédemment). Si non, sous-culture 100 µL dans 5 mL de milieu et permettre la croissance des cellules à la phase logarithmique.
- Prélever un échantillon du Master culture et compter les cellules directement à l’aide de Petroff-Hausser le compteur de chambre. Ceci énumère les cellules vivantes et mortes.
- Prélever un échantillon de 10 µL de la culture non dilué avec une micropipette et transfert dans la chambre de.
- Image au microscope objectif haute-sèche (40 X ou 63 X, NA 0,7 - 0,8) à l’aide de contraste de phase.
- Compter les bactéries pendant au moins 20 places et moyenne.
Remarque : Compter uniquement les bactéries qui sont entièrement dans un carré et seulement ces passage sur la partie supérieure et gauche limites (ou bas et droite, si vous préférez). Si les places sont séparées par plusieurs lignes, choisissez-le comme limite. - Concentration de calculer la moyenne de 20 places x facteur de dilution. Notez que direct des chefs d’accusation ne fonctionnent pas bien pour les concentrations < 10 7 / mL.
- Faire des dilutions successives de la culture pour le comptage des colonies (UFC) d’unités. Il énumère des cellules vivantes.
Solution
- faire une dilution en série de chaque échantillon bactériennes sélectionnées avec la solution saline 0,9 % stérile. Transférer 20 µL de la solution bactérienne et le diluer avec une solution saline de 180 µL. Répétez jusqu'à ce que la concentration minimale est 10 ~ 3 cellules/mL.
- Prendre 100 µL d’au moins deux dilutions proposées est 10 3 et 10 4/ml — et la plaque sur une plaque de support solide approprié. Étaler avec un épandeur stérile. Effectuer au moins 3 répétitions de chaque dilution.
- Incuber à une température appropriée pour vos bactéries pendant la nuit ou jusqu'à ce que les colonies grandir.
- Compter les colonies et calculer les unités (CFU) selon (nombre de colonies x facteur de dilution) formant des colonies / volume plaquée = CFU/mL. UFC en moyenne toute les réplicats.
2. Préparation des échantillons pour diluer fortement pour DHM
- faire des dilutions du Master culture pour DHM et post-DHM comptage d’unités formant des colonies sur des plaques de médias LB (voir 1.4.1 - 1.4.4). Effectuer cette double-aveugle, afin que la personne qui effectue les enregistrements n’est pas conscient de la concentration de chaque échantillon mesurée.
- Diluer les bactéries en 10 à 15 mL d’un milieu minimal qui inhibent la division cellulaire, mais encourager la motilité (le cas échéant) afin que la concentration de cellules ne change pas sensiblement pendant l’expérience. Cela peut être la solution saline à 0,9 % ou un milieu plus spécifique de la motilité. Par exemple, si vous utilisez Escherichia coli, le milieu motilité doit contenir EDTA 38. Pour ces exemples, nous utilisons la solution saline à 0,9 %.
3. Enregistrez des vidéos DHM
- à l’aide d’une seringue stérile, tirez dans environ 10 mL de la dilution de l’intérêt.
- Connecter la seringue à la chambre de mesure DHM pour tubes et raccords stériles.
Remarque : Une chambre sur mesure microfluidique a été construite afin de permettre l’écoulement uniforme d’échantillon dans le trajet optique de l’instrument. Reportez-vous à la section résultats pour plus de détails. Avant l’expérience, tous les composants de la chambre de mesure doivent être stérilisés par autoclave. - Débit de l’échantillon de la seringue à travers la chambre de mesure en continu à l’aide d’un pousse-seringue.
NOTE : Débits appropriés varient selon les dimensions du canal fluidique, débit souhaité ainsi que données acquisition limites. - Que l’échantillon est transmise par l’intermédiaire de la chambre de mesure, acquérir hologrammes consécutivement à une cadence appropriée. Un horodatage de fichier sera créé quels journaux l’heure de chaque image capture pour être utilisé plus tard au cours de l’analyse des données (protocole section 4). Obtenir tous les hologrammes à température ambiante (23° C). Enregistrer les hologrammes en exécutant un fichier exécutable C++ pré-écrits, fourni par le fabricant de la caméra.
NOTE : Taux de trame approprié varient selon le débit choisi. Pour cette expérience, il est recommandé d’utiliser une cadence telle qu’une bactérie pourrait être photographiée > 2 fois car il a voyagé à travers l’instrument ' de champ de s. - Prévoir un délai suffisant pour l’ensemble 10 mL d’échantillon de transmettre le DHM.
- Pour que les bactéries ne grandissent pas ou meurent au cours des expériences, inoculer enrichi plaques de médias avec 100µl de la capture d’image post-DHM médias usé. Étalez avec un épandeur stérile et incuber à une température appropriée pendant 24 heures ; dénombrer les colonies présentes.
4. Calcul de la densité cellulaire et les limites de détection
- avec les vidéos acquises hologramme, analyser les données pour la présence de bactéries.
- Calculer la valeur de pixel médiane d’une série chronologique d’hologrammes puis soustraire cette valeur médiane de chaque pixel respectif afin d’éliminer les artefacts stationnaires dans l’hologramme. Cela peut se faire en ImageJ en effectuant les étapes suivantes :
- convertir en 32 bits (Image-Type-32 bit)
- calculer la médiane (Image-pile-Zproject-médiane)
- soustraire la valeur médiane du reste de la pile ( Process-ImageCalculator-Subtract)
- compter le nombre de bagues lumineuses visibles et les cellules de mise au point manuellement. Dans ImageJ, cela se fait en pointant les objets avec l’outil Point.
- Chaque série d’hologrammes s’accompagnera d’un horodatage de fichier (enregistré au moment de l’acquisition de l’hologramme). Utiliser le fichier d’horodatage pour calculer le montant total de l’échantillon pompée au moment où l’image a été capturé.
- Calculer la valeur de pixel médiane d’une série chronologique d’hologrammes puis soustraire cette valeur médiane de chaque pixel respectif afin d’éliminer les artefacts stationnaires dans l’hologramme. Cela peut se faire en ImageJ en effectuant les étapes suivantes :
- Calcul de la densité cellulaire en divisant le nombre total de cellules détectées par le volume total des exemples imagés. En moyenne 5-10 cadres pour des statistiques précises.
5. Reconstruction d’Amplitude et de Phase d’images
- choisir vidéos représentatives avec 10-200 cellules par image. Charge brutes (pas médiane-soustrait) hologrammes dans le logiciel de reconstruction (exemples : ImageJ Propagation numérique 39 ; Shampooing [GitHub]).
- à l’aide de ImageJ, passer de Diffraction Propagation numérique numérique OD.
- à l’invite, dessiner un masque de Fourier pour choisir le real ou l’image virtuelle et éliminer toute caractéristique bruit sinusoïdal.
- Reconstruire l’amplitude et la phase à z-les mesures appropriées en entrant la distance de reconstruction.
- Médiane soustraire les données d’amplitude pour réduire le bruit comme dans 4.1.1.
- Tracer les données comme un 3D cube ou projection. Allez dans Plugins — Volume Viewer.
Résultats
Devrait permettre d’établir la capacité de détecter les bactéries mortes et vivantes à des niveaux très bas par DHM. Le nombre de bactéries compté doit être cohérent avec les résultats obtenus avec les comtes chambre et plaque de comptage de Petroff-Hauser. Méthodes statistiques standards fournissent des informations quant à l’exactitude des méthodes de détection différentes à différentes concentrations bactériennes.
Discussion
Reconstruction numérique d’hologrammes: pour la reconstruction numérique d’hologrammes, la méthode de spectre angulaire (ASM) est utilisée. Il s’agit de la convolution de l’hologramme avec la fonction de Green pour le DHM. Le front d’onde complexe de l’image dans un plan focal particulière peut être calculé en utilisant le théorème de Fourier Convolution comme suit :
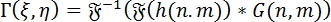 (1)
(1)
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs reconnaissent le Gordon et Betty Moore Foundation subventions 4037 et 4038 à la California Institute of Technology pour le financement de ces travaux.
matériels
| Name | Company | Catalog Number | Comments |
| Bacto Yeast | BD Biosciences | 212750 | |
| Bacto Tryptone | BD Biosciences | 211705 | |
| Sodium chloride | Sigma-Aldrich | 7710 | Many options for purchase |
| Bacto Agar | BD Biosciences | 214010 | |
| 10 cm Petri dishes | VWR | 10053-704 | |
| 15 mL culture tubes | Falcon (Corning Life Sciences) | 352002 | Loose-capped |
| Petroff-Hauser chamber | Electron Microscopy Sciences | 3920 | |
| 10 mL syringes | BD Biosciences | 309604 | Luer-Lok tip not necessary |
| Male Luer to 1/16” barbed fitting | McMaster-Carr | 51525K291 | |
| Autoclavable 1/16” ID PVC tubing for flow | Nalgene | 8000-0004 | Sold by length, purchase accordingly |
| Syringe pump | Harvard Apparatus | PHD 2000 | |
| Sample Chamber | Custom | n/a | See Materials Section |
| Holographic Microscope | Custom | n/a | See Wallace et al. |
| Open-source software | Custom | https://github.com/bmorris3/shampoo |
Références
- Akineden, O., Weirich, S., Abdulmawjood, A., Failing, K., Buelte, M. Application of a Fluorescence Microscopy Technique for Detecting Viable Mycobacterium avium ssp. paratuberculosis Cells in Milk. Food Anal. Methods. 8, 499-506 (2015).
- Deibl, J., Paulsen, P., Bauer, F. Rapid enumeration of total aerobic microbial counts on meat and in meat products by means of the direct epifluorescence filter technique. Wien Tierarztl Monat. 85, 327-333 (1998).
- Huang, J., Li, Y., Slavik, M. F., Tao, Y., Huff, G. R. Identification and enumeration of Salmonella on sample slides of poultry carcass wash water using image analysis with fluorescent microscopy. Transactions of the Asae. 42, 267-273 (1999).
- Durtschi, J. D., et al. Increased sensitivity of bacterial detection in cerebrospinal fluid by fluorescent staining on low-fluorescence membrane filters. J Med Microbiol. 54, 843-850 (2005).
- Panicker, R. O., Soman, B., Saini, G., Rajan, J. A Review of Automatic Methods Based on Image Processing Techniques for Tuberculosis Detection from Microscopic Sputum Smear Images. J Med Syst. 40, (2016).
- Riepl, M., et al. Applicability of solid-phase cytometry and epifluorescence microscopy for rapid assessment of the microbiological quality of dialysis water. Nephrol Dial Transplant. 26, 3640-3645 (2011).
- Hwang, C. Y., Cho, B. C. Virus-infected bacteria in oligotrophic open waters of the East Sea, Korea. Aquat Microb Ecol. 30, 1-9 (2002).
- Kepner, R. L., Pratt, J. R. Use of Fluorochromes for Direct Enumeration of Total Bacteria in Environmental-Samples - Past and Present. Microbiol Rev. 58, 603-615 (1994).
- Queric, N. V., Soltwedel, T., Arntz, W. E. Application of a rapid direct viable count method to deep-sea sediment bacteria. J Microbiol Meth. 57, 351-367 (2004).
- Prieto-Ballesteros, O., Vorobyova, E., Parro, V., Rodriguez Manfredi, J. A., Gomez, F. Strategies for detection of putative life on Europa. Adv Space Res. 48, 678-688 (2011).
- Gleeson, D. F., et al. Biosignature Detection at an Arctic Analog to Europa. Astrobiology. 12, 135-150 (2012).
- Carr, C. E., Zuber, M. T., Ruvkun, G., Ieee, . 2013 Ieee Aerospace Conference IEEE Aerospace Conference Proceedings. , (2013).
- Konstantinidis, K., et al. A lander mission to probe subglacial water on Saturn's moon Enceladus for life. Acta Astronautica. 106, 63-89 (2015).
- McKay, C. P., Anbar, A. D., Porco, C., Tsou, P. Follow the Plume: The Habitability of Enceladus. Astrobiology. 14, 352-355 (2014).
- Hoover, R. B., Hoover, R. B., Levin, G. V., Rozanov, A. Y., Wickramasinghe, N. C. . Instruments, Methods, and Missions for Astrobiology Xvii. , (2015).
- Handelsman, J. Metagenomics: application of genomics to uncultured microorganisms. Microbiol Mol Biol Rev: MMBR. 68, 669-685 (2004).
- Lebaron, P., Parthuisot, N., Catala, P. Comparison of blue nucleic acid dyes for flow cytometric enumeration of bacteria in aquatic systems. Appl Environ Microbiol. 64, 1725-1730 (1998).
- Marie, D., Partensky, F., Jacquet, S., Vaulot, D. Enumeration and cell cycle analysis of natural populations of marine picoplankton by flow cytometry using the nucleic acid stain SYBR Green I. Appl Environ Microbiol. 63, 186-193 (1997).
- Noble, R. T., Fuhrman, J. A. Use of SYBR Green I for rapid epifluorescence counts of marine viruses and bacteria. Aquat Microb Ecol. 14, 113-118 (1998).
- Forster, S., Snape, J. R., Lappin-Scott, H. M., Porter, J. Simultaneous fluorescent gram staining and activity assessment of activated sludge bacteria. Appl Environ Microbiol. 68, 4772-4779 (2002).
- Lauer, B. A., Reller, L. B., Mirrett, S. Comparison Of Acridine-Orange And Gram Stains For Detection Of Microorganisms In Cerebrospinal-Fluid And Other Clinical Specimens. J Clin Microbiol. 14, 201-205 (1981).
- Mason, D. J., Shanmuganathan, S., Mortimer, F. C., Gant, V. A. A fluorescent gram stain for flow cytometry and epifluorescence microscopy. Appl Environ Microbiol. 64, 2681-2685 (1998).
- Saida, H., Ytow, N., Seki, H. Photometric application of the Gram stain method to characterize natural bacterial populations in aquatic environments. Appl Environ Microbiol. 64, 742-747 (1998).
- Sizemore, R. K., Caldwell, J. J., Kendrick, A. S. Alternate Gram Staining Technique Using A Fluorescent Lectin. Appl Environ Microbiol. 56, 2245-2247 (1990).
- Bitton, G., Dutton, R. J., Foran, J. A. New Rapid Technique For Counting Microorganisms Directly On Membrane Filters. Stain Technology. 58, 343-346 (1983).
- Broadaway, S. C., Barton, S. A., Pyle, B. H. Rapid staining and enumeration of small numbers of total bacteria in water by solid-phase laser cytometry. Appl Environ Microbiol. 69, 4272-4273 (2003).
- Yagupsky, P., Nolte, F. S. Quantitative aspects of septicemia. Clin Microbiol Rev. 3, 269-279 (1990).
- Kang, D. K., et al. Rapid detection of single bacteria in unprocessed blood using Integrated Comprehensive Droplet Digital Detection. Nat Commun. 5, 5427 (2014).
- Hand, K. P. . Report of the Europa Lander Science Definition Team. , (2017).
- Popescu, G., et al. Optical imaging of cell mass and growth dynamics. Am J Physiol-Cell Physiol. 295, C538-C544 (2008).
- Mir, M., et al. Optical measurement of cycle-dependent cell growth. Proc Natl Acad Sci U S A. 108, 13124-13129 (2011).
- Rappaz, B., et al. Noninvasive characterization of the fission yeast cell cycle by monitoring dry mass with digital holographic microscopy. J.Biomed Opt. 14, 034049 (2009).
- Jo, Y., et al. Label-free identification of individual bacteria using Fourier transform light scattering. Opt. Express. 23, 15792-15805 (2015).
- Wallace, J. K., et al. Robust, compact implementation of an off-axis digital holographic microscope. Opt. Express. 23, 17367-17378 (2015).
- Lindensmith, C. A., et al. A Submersible, Off-Axis Holographic Microscope for Detection of Microbial Motility and Morphology in Aqueous and Icy Environments. Plos One. 11, e0147700 (2016).
- Dumas, E. M., Ozenne, V., Mielke, R. E., Nadeau, J. L. Toxicity of CdTe Quantum Dots in Bacterial Strains. IEEE Trans. NanoBiosci. 8, 58-64 (2009).
- Frisk, A., Jyot, J., Arora, S. K., Ramphal, R. Identification and functional characterization of flgM, a gene encoding the anti-sigma 28 factor in Pseudomonas aeruginosa. J Bacteriol. 184, 1514-1521 (2002).
- Adler, J., Templeton, B. The effect of environmental conditions on the motility of Escherichia coli. Journal Gen Microbiol. 46, 175-184 (1967).
- Piedrahita-Quintero, P., Castaneda, R., Garcia-Sucerquia, J. Numerical wave propagation in Image J. Appl Opt. 54, 6410-6415 (2015).
- Schnars, U., Jüptner, W. P. Digital recording and numerical reconstruction of holograms. Meas. Sci. Technol. 13, R85 (2002).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon