需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
分子互补亲和纯化 (BiCAP) 对多蛋白信号络合物的解剖
摘要
本手稿描述了分子互补亲和纯化 (BiCAP) 的协议。这种新方法促进了任何两种相互作用的蛋白质的特定隔离和下游蛋白质组特征, 同时排除了不复杂的个体蛋白质以及与竞争的结合伙伴形成的复合体。
摘要
蛋白质复合体的组装是许多细胞信号通路调控的中心机制。生物医学研究的一个主要重点是破译这些动态蛋白复合物如何将信号从多个来源整合起来, 以指导特定的生物反应, 以及如何在许多疾病的环境中解除管制。尽管这一关键的生化机制的重要性, 缺乏的实验技术, 可以促进具体和敏感的反褶积这些多分子信号复合物。
在这里, 这一缺陷的解决方法是通过蛋白质互补试验与构象特异 nanobody 的结合, 我们称之为分子互补亲和纯化 (BiCAP)。这种新的技术促进了特定的隔离和下游蛋白质组特征的任何一对相互作用的蛋白质, 排除不复杂的个体蛋白和复合物形成与竞争的捆绑合作伙伴。
BiCAP 技术适用于广泛的下游实验测试, 这种技术提供的高度特异性使得对蛋白质复杂装配的力学的更细致的研究比目前可能使用标准亲和纯化技术。
引言
蛋白质复合组装是维持许多信号通路1、2的时空特异性的关键过程。虽然这一管制作用的关键性质得到广泛承认, 但缺乏用于审查这些复合体的实验技术。大多数 interactomics 研究的重点是与个体蛋白质的相互作用, 或单个复杂成分的顺序浓缩。在这里, 我们提出了一种分离特定蛋白质二聚体的技术, 而不包括单个基团的成分蛋白, 以及形成了与竞争的捆绑合作伙伴3。我们称之为这种技术分子互补亲和纯化 (BiCAP), 因为它是一个以前现有的蛋白质片段互补检测, 分子荧光互补 (BiFC) 的组合, 与新的使用构象特异重组 nanobody 对 GFP 及其衍生物 (见材料表).
一个典型的蛋白质片段互补试验依赖于 "诱饵" 和 "猎物" 蛋白的表达, 融合成分裂的记者, 如荧光素酶4, β-乳糖酶5, 或绿色荧光蛋白 (GFP)6 (图 1A)。通过诱饵和猎物蛋白的相互作用, 鼓励分裂的记者领域再折叠餐巾成一个功能结构, 允许诱饵和猎物蛋白的相互作用被可视化或量化。BiCAP 是根据这种技术的版本改编的, 利用了 GFP 变种金星的片段。荧光蛋白互补检测是一种常见的方法, 以可视化的蛋白质-蛋白质相互作用的活细胞, 但直到现在已被限制在这一功能7。BiCAP 代表了这方面的一个重大进展, 因为这项技术不仅允许可视化, 而且还对由此产生的蛋白质-蛋白质相互作用的隔离和审问。
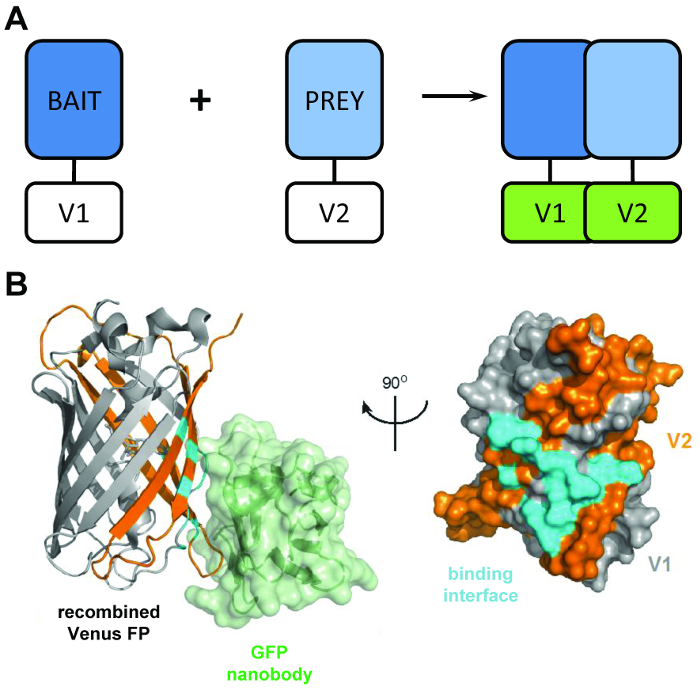
图 1: BiCAP 技术背后的结构主体.(a) 概述分子荧光互补原理的示意图, 显示 "诱饵" 和 "猎物" 蛋白, 标签为 n-端 V1 或 V2 的全长金星蛋白片段。(B) 对 GFP nanobody (绿色) 与重组金星之间的交互界面 (青色) 进行结构分析, 显示 V1 (灰色) 和 V2 (橙色) 片段 (PDB 加入 3OGO) 的位置。这一数字被重新发布 fromCroucher 等3转载的许可, 由科学促进会。请单击此处查看此图的较大版本.
BiCAP 技术利用金星的两个非荧光片段 (命名为 V1 和 V2), 它与低亲和力关联, 除非它们的融合伙伴之间发生交互作用。在此实例中, 两个拆分域再折叠餐巾到荧光的功能β桶结构 (图 1B)6。BiCAP 的关键创新来自于对重组 gfp nanobody 的介绍, 它能识别出 gfp (和金星等变种) 的β桶上的三维表位, 这只存在于正确重组和折叠的荧光 (图 1B)8。关键的是, GFP nanobody 不绑定到单独的金星碎片之一。这促进了蛋白质脂肪酸的分离, 只有在这两种蛋白质形成了自己的意志复杂, 导致更具代表性的结果比从方法, 利用化学诱导, 强迫互动9。
BiCAP 是一种功能强大的技术, 专门专注于多蛋白复合物, 这可能与一些下游应用结合, 以提高我们对这些复合体在信号传导中所起的作用的理解的粒度。.它还包括允许在原位进行蛋白质相互作用可视化的重要特征。迄今为止, BiCAP 已被证明是一种有效的方法来分析受体酪氨酸激酶 (RTK) 脂肪酸3的 interactome, 但这种方法的适应性意味着它可以在几乎任何蛋白质相互作用的情况下采取。
研究方案
1. 质粒克隆
注意: 要生成质粒载体, 将 V1 或 V2 标记融合到感兴趣基因的 N 终点或 C 终点, BiFC 的目标向量已存入 Addgene [N 末端标记: pDEST-V1-ORF (#73635), pDEST-V2-ORF (#73636)。C 终端标签: pDEST-ORF-V1 (#73637), pDEST-ORF-V2 (#73638)]。该基因/s 的兴趣将需要在特定的重组克隆兼容进入媒介 (即pDONR223 或 pENTR221), 没有停止密码子, 以进行克隆。许多相容的克隆已经在各种质粒集合中获得, 包括哺乳动物基因组收集 (https://genecollections.nci.nih.gov/MGC/)。
- 执行重组反应, 在适当的 BiFC 目标向量的 attR 站点之间插入感兴趣的基因:
- 在0.2 毫升管中, 添加 150 ng 进入向量, 150 ng BiFC 目标向量, 8 µL TE 缓冲器 (pH 8.0)。
- 添加2µL recombinase 酶组合 (见材料表)。搅拌好, 离心机简单。
- 在室温或16摄氏度的一夜内孵化1小时的反应。
- 通过添加1µL 蛋白酶 K 溶液和孵化37°c 10 分钟来停止反应。
- 用 LR 反应产物转化热休克主管细胞 (见材料表):
注: 任何基本菌株都可以在这一点上使用, 只要它不含有 Ccdb 抗毒素, 这将防止特定的选择的质粒含有插入。- 解冻有能力的细胞在冰和转移50µL 细胞成14毫升圆底聚丙烯管。
- 在细胞中加入1µL 的反应产物, 并轻轻地混合。在冰上孵化20分钟。
- 热休克在42°c 水浴四十五年代. 立即返回冰2分钟。
- 添加1毫升的 LB 培养基和孵育37°c 1 小时。
- 将其转化为含有氨苄西林 (100 µg/毫升) 的10厘米琼脂板, 并在37摄氏度一夜孵育。
- BiFC 质粒的纯化。
- 要从单个菌落中纯化质粒, 将100毫升的 LB 介质添加到大锥形瓶中, 加入氨苄西林 (100 µg/毫升)。要筛选多个菌落, 请在14毫升圆底聚丙烯管中添加5毫升的 LB 介质, 并加入氨苄西林 (100 µg/毫升)。
- 使用无菌接种循环, 选择一个单一的殖民地, 从琼脂板块。将接种循环放入 LB 介质中, 并简单混合。
- 用铝箔盖住锥形烧瓶的顶部, 在37摄氏度的时候用颤抖的一夜孵化。
- 用标准 maxiprep 或 midiprep 质粒 dna 纯化试剂盒 (见材料表) 生产转染级质粒 dna10。通过吸光度谱评价 DNA 的质量。
注: DNA 应具有 A260/A280 比 > 1.8 和 A260/A230 比 > 2.0。此时, 建议筛选多个单独的菌落, 以检查插入和标记的有效表达式。
2. 细胞培养和转染
注: 对于 BiFC 载体的转染, 重要的是要实现高效, 相对均匀的转染。该载体可能与任何标准转染试剂兼容, 并应相应地优化转染条件。为了进行质谱分析, 我们通常在10厘米的菜肴中培养细胞, 虽然这也可以按比例缩小到较小的盘子或盘子中, 而实验则需要较少的材料。
- 种子 1.0 x 106 HEK293T 细胞在 10 cm 盘与10毫升 DMEM 媒介, 补充与10% 血清和青霉素或链霉素 (1:100)。
- 将每个 BiFC 的2.5 µg 稀释成500µL 的转染缓冲器 (见材料表), 1.5 毫升离心管。
- 添加10µL 转染试剂 (见材料表)。
- 漩涡混合物为十年代, 然后简要地离心机。室温下孵育10分钟。
- 将 DNA 转染的混合物添加到盘子中, 滴状。孵化细胞足够长的时间为 BiFC 融合蛋白相互作用和为金星折叠和荧光成熟, 一般 8-24 h。
注: 金星的峰值激发是 515 nm, 其峰值发射是 528 nm, 虽然这是很容易看到使用标准的荧光显微镜设置, 以可视化 GFP 荧光。
3. 样品准备
-
收割裂解物
- 在裂解之前收集, 准备细胞裂解缓冲 [50 毫米三盐酸 (pH 7.4), 150 毫米氯化钠, 1 毫米 EDTA, 1% (v/v) 非离子洗涤剂 (见材料表)]。存储在4摄氏度。
- 在收割之前, 用蛋白酶抑制剂和磷酸酶抑制剂补充10毫升细胞裂解缓冲液 (见材料表)。在冰上保持细胞溶解缓冲。
- 在冰冷的 PBS 中洗两次细胞。吸入 PBS 并加入1毫升的冰寒补充细胞裂解缓冲液。将盘子放在冰上, 确保缓冲区在整个表面积上扩散。
- 在冰上孵育大约5分钟, 然后使用一个细胞刮板 (见材料表) 刮的细胞和转移到预冷的1.5 毫升离心管。
- 去除细胞碎片通过离心收集的裂解物在 1.8万 x g 5 分钟4摄氏度, 并转移清除上清到新鲜的离心管。
注: 在这一点上, 裂解液可以立即使用, 或储存在-80 摄氏度。此外, 还建议保持整除的粗裂解液, 使转染和亲和纯化的效率得到验证。
-
亲和纯化
注意: BiCAP 隔离步骤是使用 GFP nanobody 共轭琼脂糖珠 (见材料表) 执行。- 通过洗涤适当的体积 (20 µL 每样品 + 10 µL 过量) 在1毫升 PBS 准备琼脂糖珠。离心的珠子在 300 x g 和删除上清。
- 在每个裂解样品中加入20µL。
- 以端到端旋转的方法在4摄氏度孵化样品2小时。
注: 在这一点上, 可以为 SDS 页和西方印迹, 或者通过质谱分析来制备样品。
-
BiCAP 淋洗剂组成的制备。
- 离心 300 x g的珠子, 在细胞裂解缓冲液中洗涤三次。
- 并用重悬在50µL 适当稀释的样品缓冲器 (见材料表), 并加热样品在95°c 2-3 分钟的洗涤珠。
注: 以这种方式制备的样品可在-20 摄氏度内储存几个月。 - 对 V1 标签和 V2 标签 (见材料表), 以及任何其他感兴趣的蛋白质, 执行 SDS 页和西部印迹11 。
-
质谱 BiCAP 淋洗剂组成的制备。
注: 为进行无标签定量 (LFQ) 质谱分析, 建议在至少四份中准备样品, 以确保可靠的统计能力。- 离心的珠子在 300 x g 和洗六次细胞裂解缓冲没有洗涤剂。这是必要的, 以消除多余的蛋白质和洗涤剂, 将干扰质谱分析。立即进行下一步, 或存储在-80 °c 的珠子.
- 胰蛋白酶处理珠在60µL 缓冲 1 [2 M 尿素, 50 毫米三盐酸 (pH 7.5), 5 µg/毫升胰蛋白酶]。允许珠子消化30分钟, 在27°c 在一个 thermomixer 摇晃 800 RPM。
- 简要的离心珠, 然后收集上清和转移到离心管 (见材料表)。
- 洗涤珠在25µL 缓冲 2 [2 M 尿素, 50 毫米三 HCl (pH 7.5), 1 毫米 dithiothreitol], 为了减少束缚的蛋白质。
- 将珠子和上清液池放入一个离心管中。允许消化在室温下过夜。
- 烷基化在样品中加入20µL iodoacetamide (5 毫克/毫升的超纯水), 并在黑暗中孵化30分钟。
- 用1µL 三氟乙酸酸 (TFA) 处理每个样品, 抑制消化。这一步骤也将酸化的样品准备阶段的小费。
- 通过堆叠6层1毫米固相萃取 C18 (C18) 膜盘 (见材料表), 构造出一个200µL 微尖。为每个示例准备一个提示。
- 用甲醇湿润舞台提示, 平衡50µL 0.1% (v/v) 三氟乙酸酸 (TFA), 80% (v/v) 乙腈。
- 用50µL 0.1% (v/v) TFA 清洗提示。
- 将酸化肽加载到舞台尖端, 然后洗脱使用 0.1% (v/v) TFA, 80% (v/v) 乙腈两个步骤。
- 使用真空浓缩器蒸发样品。
- 如果需要, 将干肽贮存在-80 摄氏度。
4. 质谱法。
- 并用重悬样品在15µL 5% 甲酸, 2% 乙腈 (在超纯水)。
- 小心地将6µL 到 LC 板上, 放入 nanoLC 高效液相色谱系统 (见材料表)。
- 包装20厘米, 75 µM 内径柱与1.9 µM C18 固定相粒子 (见材料表)。将5µL 肽载入每一列。
- 洗脱肽使用乙腈的线性梯度在 250 nL/分钟140分钟以上, 并通过 nanoelectrospray 引入线性陷阱 Quadropole (LTQ) 混合质谱仪与 nanoLC HPLC 系统。
- 在140分钟的时间梯度上, 为每个扫描收集10个最丰富的离子的串联 MS 数据。随机的数据收集和交换与 BSA 运行在每个样本之间的顺序, 以尽量减少时间偏差。
5. 分析
- 使用 MaxQuant 软件版本1.2.7.4 中的默认设置处理原始 MS 数据, 并在 R 软件环境3中使用修订版本的珀尔修斯统计分析工作流分析 MaxQuant 输出。
注意: 从这一点的统计分析工作流概述在图 2中。简要地, 用 MaxQuant 识别的蛋白质的 LFQ 强度进行了变换和过滤, 然后进行规范化和归一化。二聚体对相互作用的蛋白质通过与金星控制的比较确定, 排除非特异性的背景粘合剂, 与学生的 t 测试和 Benjamini 霍赫贝格校正为多次比较。通过对单个直方图、多重回归和层次聚类的比较, 确定了数据质量。

图 2: 统计分析工作流的概要.统计分析管道流程图, 用于分析使用 MaxQuant 处理的原始质谱数据所识别的蛋白质的 LFQ 强度。绿色框: 过滤, 蓝色盒: 变换/正常化/缩放, 棕盒: 数据质量控制, 黄色框: 半定量排除/比较分析, 灰色盒: 统计分析。这一数字被重新发布 fromCroucher 等3转载的许可, 由科学促进会。请单击此处查看此图的较大版本.
结果
在使用重组克隆产生的 V1 和 V2 标记基因感兴趣的 BiFC pDEST 质粒, 共转染两个质粒含有相互作用对蛋白质将导致产生金星荧光信号后,大约8-24 小时。在没有阳性信号的情况下, 由于细胞系的选择、转染效率低或 BiFC 标记的方向不理想, 蛋白质相互作用可能不会发生。下面将讨论每个方案的疑难解答。
我们以前观察到, 在 HEK-293T 细...
讨论
BiCAP 是一种有效的分离特定蛋白脂肪酸的方法, 而不包括单个成分及其相互竞争的结合伙伴3。BiCAP 是基于一种称为 BiFC6的荧光蛋白互补试验的适应性。现有的方法, 包括 BiFC 和近距离结扎法, 已广泛用于可视化和量化活细胞中的蛋白质相互作用7, 但没有提供一种有效的方法来分离和表征脂肪酸形成的这些相互作用。
基于蛋?...
披露声明
作者没有什么可透露的
致谢
d.r. C 是一个癌症研究所新南威尔士的研究员和 d.n. S 以前是一个癌症研究所新南威尔士州研究员。本手稿中的研究结果由新南威尔士癌症研究所 (13/FRL/1-02 和 09/民防部队/2-39)、澳洲 (项目赠款 GNT1052963)、爱尔兰科学基金会 (11/SIRG/B2157)、新南威尔士州科学和医学研究办公室、来宾家庭资助。奖学金和 Mostyn 家庭基金会。J.F.H. 和 R.S. 是澳大利亚研究生奖获得者。
材料
| Name | Company | Catalog Number | Comments |
| LR Clonase II Plus enzyme | Thermo Fisher Scientific | 12538120 | Recombinase enzyme required for Gateway cloning (Step 1) into pDEST BiFC destination vectors |
| Proteinase K, recombinant, PCR grade | Thermo Fisher Scientific | EO0491 | |
| 14 mL round-bottomed polypropylene tube | Corning | 352059 | |
| Ampicillin | Roche Diagnostics Australia | 10835242001 | Stock solution prepared at 100 μg/mL in distilled water. |
| Miniprep kit | Promega Corporation | A1330 | |
| Maxiprep kit | Life Technologies Australia | K2100-07 | |
| DMEM | Gibco | 11995-073 | |
| FBS | Life Technologies Australia | 10099-141 | |
| Penicillin/Streptomycin | Life Technologies Australia | 15070-063 | |
| jetPRIME transfection buffer | Polyplus | 114-15 | |
| jetPRIME transfection reagent | Polyplus | 114-15 | |
| PhosSTOP (Phosphatase inhibitor) | Sigma-Aldrich | 4906837001 | |
| cOmplete, Mini, EDTA-free Protease inhibitor cocktail | Roche | 11873580001 | |
| Cell Scraper | Sarstedt | 83.1832 | |
| GFP-Trap_A | Chromotek Gmbh | gta-100 | GFP nanobody coupled to agarose beads |
| N-terminal GFP monoclonal antibody | Covance | MMS-118P | Will detect the V1 tag within the BiFC vectors |
| C-terminal GFP monoclonal antibody | Roche | 11814460001 | Will detect the V2 tag within the BiFC vectors |
| Sample buffer | Invitrogen | NP0008 | Supplemented with 1 mL β-mercaptoethanol. |
| Sequencing grade modified trypsin | Promega Corporation | V5117h | |
| LoBind microcentrifuge tubes | Point of Care Diagnostics | 0030 108 116 | |
| Iodoacetamide | Sigma-Aldrich | I1149-5G | Prepared at 5 mg/mL in ultrapure water |
| Trifluoroacetic Acid - Sequanal Grade | Thermo Fisher | 10628494 | |
| 3M Empore solid phase extraction C18 disks (octadecyl) - 4.7 cm | Thermo Fisher | 14-386-2 | To prepare stage tips, cut 1 mm disks using an appropriately sized hole punch. Alternatively, pre-prepared stage tips can also be purchased, see below. |
| C18 Stage Tips, 10 µL bed | Thermo Fisher | 87782 | |
| Formic acid OPTIMA for LC/MS grade 50mL | Thermo Fisher | FSBA117-50 | |
| 1.9 μm C18 ReproSil particles | Dr. Maisch GmbH | r119.aq. | Stationary phase particles |
| Acetonitrile OPTIMA LC/MS grade | Thermo Fisher | FSBA955-4 | |
| Easy-nLC HPLC | Thermo Fisher | ||
| LTQ Orbitrap Velos Pro | Thermo Fisher | ||
| Triton X-100 | Sigma-Aldrich | T8787 | Non-ionic detergent (100%) |
| DH5α cells | Thermo Fisher | Heat-shock-competent cells |
参考文献
- Kolch, W., Pitt, A. Functional proteomics to dissect tyrosine kinase signalling pathways in cancer. Nat Rev Cancer. 10 (9), 618-629 (2010).
- Pawson, T., Kofler, M. Kinome signaling through regulated protein-protein interactions in normal and cancer cells. Curr Opin Cell Biol. 21 (2), 147-153 (2009).
- Croucher, D. R., et al. Bimolecular complementation affinity purification (BiCAP) reveals dimer-specific protein interactions for ERBB2 dimers. Sci Signal. 9 (436), ra69 (2016).
- Cassonnet, P., et al. Benchmarking a luciferase complementation assay for detecting protein complexes. Nat Methods. 8 (12), 990-992 (2011).
- Rossi, F., Charlton, C. A., Blau, H. M. Monitoring protein-protein interactions in intact eukaryotic cells by beta-galactosidase complementation. Proc Natl Acad Sci U S A. 94 (16), 8405-8410 (1997).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism. J Am Chem Soc. 127 (1), 146-157 (2005).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21 (5), 539-545 (2003).
- Kubala, M. H., Kovtun, O., Alexandrov, K., Collins, B. M. Structural and thermodynamic analysis of the GFP:GFP-nanobody complex. Protein Sci. 19 (12), 2389-2401 (2010).
- Fegan, A., White, B., Carlson, J. C., Wagner, C. R. Chemically controlled protein assembly: techniques and applications. Chem Rev. 110 (6), 3315-3336 (2010).
- JoVE Science Education Database. Basic methods in cellular and molecular biology. plasmid purification. J Vis Exp. , (2017).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Shearer, R. F., et al. The E3 ubiquitin ligase UBR5 regulates centriolar satellite stability and primary cilia formation via ubiquitylation of CSPP-L. Mol Biol Cell. , (2018).
- Shannon, P., et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks. Genome Res. 13 (11), 2498-2504 (2003).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, 15690 (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。