È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Dissezione multiproteici segnalazione di purificazione di affinità di complementazione bimolecolare (BiCAP)
In questo articolo
Riepilogo
Questo manoscritto descrive il protocollo per purificazione di affinità complementazione bimolecolare (BiCAP). Questo metodo novello facilita l'isolamento specifico e caratterizzazione proteomica a valle di qualsiasi due proteine interagenti, mentre escluse ONU-complessato singole proteine, nonché complessi formati con concorrenti partner di legame.
Abstract
L'assemblaggio dei complessi della proteina è un meccanismo centrale sottostante la regolazione di molte vie di segnalazione delle cellule. Degli obiettivi principali della ricerca biomedica è decifrare come questi complessi proteici dinamica agiscono per integrare i segnali provenienti da più origini per dirigere una specifica risposta biologica, e come questo diventa deregolamentato in molte impostazioni di malattia. Nonostante l'importanza di questo meccanismo biochimico chiave, c'è una mancanza di tecniche sperimentali che possono facilitare la deconvoluzione specifico e sensibile di questi complessi di segnalazione multi-molecolari.
Qui questa lacuna viene risolto mediante la combinazione di un'analisi di complementazione della proteina con un nanobody conformazione-specifici, che abbiamo chiamato purificazione di affinità complementazione bimolecolare (BiCAP). Questa tecnica novella facilita l'isolamento specifico e caratterizzazione proteomica a valle di qualsiasi coppia di proteine interagenti, ad esclusione di ONU-complessato singole proteine e complessi formati con concorrenti partner di legame.
La tecnica di BiCAP è adattabile a una vasta gamma di analisi sperimentale a valle, e l'alto grado di specificità offerto da questa tecnica permette più sfumate indagini nelle meccaniche di assiemi complessi della proteina che è attualmente possibile utilizzando tecniche di purificazione di affinità standard.
Introduzione
Assiemi complessi della proteina sono un processo chiave nel mantenimento della specificità spatiotemporal di molti segnalazione vie1,2. Mentre la natura critica di questo ruolo normativo è ampiamente riconosciuta, c'è una mancanza di tecniche sperimentali disponibili a scrutare questi complessi. Maggior parte degli studi interattomica concentrarsi sulle interazioni con singole proteine, o l'arricchimento sequenza dei singoli componenti complessi. Qui presentiamo una tecnica per l'isolamento di un dimero di proteina specifica mentre escluse le moiety individuali di proteine componente, nonché complessi formati con associazione partner3in competizione. Abbiamo chiamato questa tecnica di purificazione di affinità complementazione bimolecolare (BiCAP), come esso è una combinazione di una precedentemente esistenti analisi di complementazione frammento della proteina, bimolecolare fluorescenza complementazione (BiFC), con l'uso novello di un nanobody ricombinante conformazione-specifici verso GFP e suoi derivati (vedere tabella materiali).
Un'analisi di complementazione del frammento proteico tipico si basa sull'espressione di proteine "esca" e "preda" fusa per dividere frammenti di giornalisti come luciferase4, β-galattosidasi5o la proteina fluorescente verde (GFP)6 ( Figura 1A). Attraverso l'interazione delle proteine esca e preda, i domini di reporter di Spalato sono incoraggiati a ripiegare in una struttura funzionale, consentendo l'interazione dell'esca e preda di proteine per essere visualizzati o quantificato. BiCAP è stato adattato da una versione di questa tecnica che ha fatto uso di frammenti della variante GFP Venus. Analisi di complementazione di proteina fluorescente sono un metodo popolare per la visualizzazione di interazioni proteina-proteina in una cellula viva, ma fino ad ora sono state limitate a questo una funzione7. BiCAP rappresenta un progresso significativo a questo proposito, come questa tecnica consente non solo per la visualizzazione, ma anche l'isolamento e interrogatori dell'interazione proteina-proteina risultante.
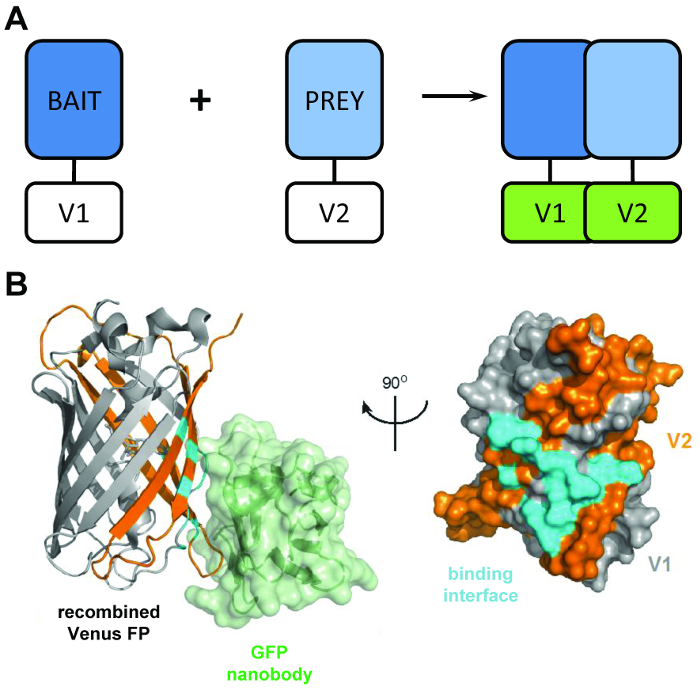
Figura 1: lo strutturale principale dietro la tecnica BiCAP. (A) uno schema che illustra il principale dietro mostrando complementazione bimolecolare fluorescenza le proteine 'esca' e 'preda' contrassegnati con il N-terminale V1 o V2 del C-terminale frammenti della proteina Venus full-length. (B) analisi strutturale di (ciano) l'interfaccia di interazione tra la GFP nanobody (verde) e Venere ricombinata, indicante la posizione delle V1 (grigio) e V2 (arancione) frammenti (PDB adesione 3OGO). Questa figura è ripubblicato fromCroucher et al.3 ristampato con il permesso di AAAS. Clicca qui per visualizzare una versione più grande di questa figura.
BiCAP tecnica fa uso di due frammenti non fluorescente di Venere (denominato V1 e V2) che associano un basso grado di affinità, a meno che non si verifica un'interazione tra i loro soci di fusione. In questo caso, i domini di due split ripiegare nella struttura funzionale del β-barilotto del fluoroforo (Figura 1B)6. L'innovazione chiave di BiCAP deriva dall'introduzione del ricombinante nanobody GFP, che riconosce un epitopo tridimensionale sul β-barilotto di GFP (e varianti come Venus) che è presente il fluoroforo correttamente ricombinato e piegato ( solo Figura 1B)8. Fondamentalmente, il nanobody GFP non viene associato a uno dei singoli frammenti di Venus. Questo facilita l'isolamento dei dimeri della proteina solo dopo che le due proteine hanno formato un complesso di loro spontanea volontà, portando a risultati più rappresentativi che quelle acquisite da metodi che fanno uso di interazioni chimicamente indotto, forzato9.
BiCAP è una tecnica potente che si concentra in particolare sui complessi della multi-proteina, che potenzialmente possono essere combinati con un numero di applicazioni a valle per migliorare il livello di dettaglio della nostra comprensione del ruolo che questi complessi svolgere nella trasduzione del segnale . Esso comprende anche la caratteristica importante di permettendo visualizzazione di proteine interazioni in situ. Ad oggi, BiCAP è stata dimostrata come un efficace metodo di analizzare Interactoma dei recettori tirosina chinasi (RTK) dimeri3, ma l'adattabilità di questo metodo significa che possa essere adottata in qualsiasi contesto di interazione della proteina.
Protocollo
1. plasmide clonazione
Nota: Per generare vettori plasmidici con i tag V1 o V2 fusi al C-terminale del gene di interesse o del N-terminale, BiFC vettori di destinazione sono stati depositati con Addgene [tag N-terminale: del pDEST-V1-ORF (#73635), del pDEST-V2-ORF (#73636). Prodotto tag: C-terminale: del pDEST-ORF-V1 (#73637), del pDEST-ORF-V2 (#73638)]. Il gene/s di interesse dovrà essere nella ricombinazione specifici vettori entrata compatibile (cioè, pDONR223 o pENTR221), la clonazione senza codoni di stop, per procedere con la clonazione. Molti cloni compatibili sono già disponibili all'interno di varie collezioni di plasmide, tra cui la collezione del genoma dei mammiferi (https://genecollections.nci.nih.gov/MGC/).
- Eseguire una reazione di ricombinazione per inserire il gene di interesse tra i siti di attR del vettore di destinazione BiFC appropriato:
- In un tubo di 0,2 mL, aggiungere 150 vettoriale di voce ng, 150 ng BiFC destinazione vettore e 8 µ l di tampone di TE (pH 8.0).
- Aggiungere 2 µ l ricombinasi enzima mix (Vedi Tabella materiali). Mescolare bene e centrifugare.
- Incubare la reazione per 1 h a temperatura ambiente o a 16 ° C durante la notte.
- Fermare la reazione aggiungendo 1 µ l di soluzione di proteinasi K e incubare a 37 ° C per 10 min.
- Trasformare cellule calore-shock-competenti (Vedi Tabella materiali) con il prodotto di reazione di LR:
Nota: Qualsiasi ceppo base sarà utilizzato presumibilmente a questo punto, purché non contenga l'antitossina Ccdb, che impedirebbe la selezione specifica di plasmidi contenenti l'inserto.- Scongelare le cellule competenti sul ghiaccio e trasferire 50 µ l di cellule in una provetta di polipropilene 14 mL a fondo tondo.
- Aggiungere 1 µ l di prodotto di reazione alle celle e mescolare delicatamente. Incubare per 20 minuti sul ghiaccio.
- Colpo in bagnomaria a 42 ° C per 45 di calore s. immediatamente restituito al ghiaccio per 2 min.
- Aggiungere 1 mL di LB media e incubare con agitazione a 37 ° C per 1 h.
- Piastra la trasformazione su una piastra di agar di 10cm contenente ampicillina (100 µ g/mL) e incubare a 37 ° C durante la notte.
- Purificazione del plasmide BiFC.
- Per purificare il plasmide da una colonia di individuo, aggiungere 100 mL di LB media in una beuta da grande e aggiungere ampicillina (100 µ g/mL). Per selezionare più colonie, aggiungere 5 mL di LB media provette in polipropilene di 14 mL a fondo tondo e aggiungere ampicillina (100 µ g/mL).
- Con un'ansa sterile inoculazione, scegliere una singola Colonia dalla piastra di agar. Mettere l'ansa per inoculazione in media LB e mescolare brevemente.
- Coprire la parte superiore del matraccio conico con carta stagnola e incubare a 37 ° C in agitazione per una notte.
- Produrre la transfezione grado plasmide DNA usando un standard maxiprep o midiprep DNA plasmidico kit di purificazione (Vedi Tabella materiali)10. Valutare la qualità del DNA di spettri di assorbanza.
Nota: DNA deve avere un rapporto A260/A280 > 1.8 e un rapporto A260/A230 > 2.0. A questo punto, si consiglia di schermo più diverse colonie verificare efficiente espressione dell'inserto e tag.
2. cultura e transfezione delle cellule
Nota: Per la transfezione dei vettori BiFC è importante ottenere un'alta efficienza e transfezione relativamente omogenea. I vettori sarà probabilmente compatibili con qualsiasi reagente di transfezione standard, e le condizioni di transfezione dovrebbero essere ottimizzate di conseguenza. Per eseguire la spettrometria di massa, abbiamo solitamente coltura cellule all'interno di piatti 10 cm, anche se questo può anche essere proporzionalmente ridotta a piatti più piccoli o piastre per esperimenti che richiedono meno materiale.
- Seme 1.0 × 106 HEK293T cellule in un 10 cm piatto con 10 mL di terreno DMEM, completati con 10% FBS e penicillina/streptomicina (1: 100).
- Diluire 2,5 µ g di ogni vettore di BiFC in 500 µ l di tampone di transfezione (Vedi Tabella materiali) in una provetta da microcentrifuga da 1,5 mL.
- Aggiungere 10 µ l di reagente di transfezione (Vedi Tabella materiali).
- Miscela di vortice per 10 s, quindi brevemente centrifuga. Incubare per 10 minuti a temperatura ambiente.
- Aggiungere la miscela di transfezione del DNA alla piastra, goccia a goccia. Incubare le cellule per una lunghezza sufficiente di tempo per le proteine di fusione BiFC interagire e per Venus pieghevole e maturazione fluoroforo, generalmente ~ 8-24 h.
Nota: L'eccitazione di picco per Venus è 515 nm e il suo picco di emissione è 528 nm, anche se questo è prontamente visto utilizzando un microscopio a fluorescenza standard impostato per visualizzare la fluorescenza di GFP.
3. preparazione del campione
-
Lisati di raccolta
- Prima della raccolta lisato, preparare il tampone di lisi delle cellule [50 mM Tris-HCl (pH 7.4), 150 mM NaCl, 1 mM EDTA, 1% (v/v) detergente non ionico (Vedi Tabella materiali)]. Conservare a 4 ° C.
- Immediatamente prima della raccolta, supplemento 10 mL di tampone di lisi delle cellule con l'inibitore della proteasi e fosfatasi inibitore (Vedi Tabella materiali). Tenere il tampone di lisi delle cellule completati sul ghiaccio.
- Lavare le cellule due volte in PBS ghiacciata. Aspirare il PBS e aggiungere 1 mL di gelida completati Buffer di lisi delle cellule. Mettere la teglia sul ghiaccio, garantendo che il buffer si sviluppa su tutta la superficie.
- Incubare in ghiaccio per circa 5 minuti, quindi utilizzare un raschietto di cella (vedere Tabella materiali) per raschiare le cellule e trasferimento in una microcentrifuga da 1,5 mL pre-refrigerati.
- Rimuovere i detriti cellulari mediante centrifugazione i raccolti lisato a 18.000 × g per 5 min a 4 ° C e trasferimento deselezionata surnatante in fresco per microcentrifuga.
Nota: A questo punto il lisato può essere utilizzato immediatamente, oppure conservato a-80 ° C. Si consiglia inoltre di mantenere un'aliquota del greggio lisato in modo che l'efficienza di trasfezione e la purificazione di affinità può essere convalidato.
-
Purificazione di affinità
Nota: Il passaggio di isolamento BiCAP viene eseguita utilizzando la GFP nanobody coniugato ai branelli di agarosio (Vedi Tabella materiali).- Preparare le perle dell'agarosi lavando un volume adeguato (20 µ l per campione) + 10 µ l in eccesso in 1 mL di PBS. Centrifugare le perline a 300 x g e rimuovere il surnatante.
- Aggiungere 20 µ l di ciascun campione lisato.
- Incubare i campioni per 2 h a 4 ° C con rotazione to-end.
Nota: A questo punto i campioni possono essere preparati per SDS-PAGE e western blotting o analisi di spettrometria di massa.
-
Preparazione di BiCAP eluente per macchiarsi occidentale.
- Centrifugare le perline a 300 x g e lavare tre volte in tampone di lisi delle cellule.
- Risospendere le perline lavate in 50 µ l di tampone di campione opportunamente diluito (Vedi Tabella materiali) e riscaldare i campioni a 95 ° C per 2-3 min.
Nota: I campioni preparati in questo modo possono essere conservati a-20 ° C per diversi mesi. - Eseguire SDS-PAGE e western blotting11 per il tag V1 e V2 Tag (Vedi tabella dei materiali), come così come qualsiasi altre proteine di interesse.
-
Preparazione di BiCAP eluente per spettrometria di massa.
Nota: Per l'analisi mediante spettrometria di massa (IFQ) privo di etichetta quantitativa si raccomanda di preparare i campioni in almeno quadruplicare per garantire potenza statistica robusta.- Centrifugare le perline a 300 x g e lavare sei volte in tampone di lisi delle cellule senza detersivo. Ciò è necessario per rimuovere l'eccesso di proteine e anche il detersivo che interferisca con l'analisi di spettrometria di massa. Procedere al passaggio successivo immediatamente, o memorizzare le perline a-80 ° C.
- Tripsinizzano perline in 60 µ l tampone 1 [2 M urea, 50 mM Tris-HCl (pH 7.5), tripsina di 5 µ g/mL]. Consentire le perline per digerire a 27 ° C in un thermomixer agitazione a 800 RPM per 30 minuti.
- Brevemente centrifugare perline, poi raccogliere il surnatante e trasferimento per microcentrifuga (Vedi Tabella materiali).
- Lavare le perle in 25 µ l tampone 2 [urea M 2, 50 mM Tris-HCl (pH 7.5), 1 mM dithiothreitol], al fine di ridurre le proteine associate.
- Piscina le perle e il surnatante in una microcentrifuga. Consentire la digestione si verifica durante la notte a temperatura ambiente.
- Alchilare i campioni con l'aggiunta di 20 µ l di iodoacetamide (5 mg/mL in acqua ultrapura) per ogni campione e incubare al buio per 30 min.
- Inibire la digestione trattando ogni campione con acido di 1 µ l trifluoroacetico (TFA). Questo passaggio sarà anche acidificare i campioni in preparazione per la fase di ribaltamento.
- Costrutto C18 fase punte di impilamento 6 strati di estrazione in fase solida 1mm C18 (ottadecil) dischi di membrana (Vedi Tabella materiali) in un suggerimento di una micropipetta 200 µ l. Preparare una punta singola per ogni campione.
- Bagnare le punte di fase con metanolo ed equilibrare con 50 µ l 0.1% (v/v) l'acido trifluoroacetico (TFA), 80% (v/v) acetonitrile.
- Lavare le punte con 50 µ l 0.1% (v/v) TFA.
- I peptidi acidificati sulle punte di fase di carico e quindi eluire utilizzando 0.1% (v/v) TFA, acetonitrile 80% (v/v) in due passaggi.
- Evaporare i campioni utilizzando un concentratore a vuoto.
- Se necessario, archiviare secchi peptidi a-80 ° C.
4. spettrometria totale.
- Risospendere i campioni in 15 µ l di acido formico al 5%, 2% acetonitrile (in acqua ultrapura).
- Accuratamente caricare 6 µ l in un piatto di LC e posto in un sistema HPLC di nanoLC (Vedi Tabella materiali).
- Confezione un 20 cm, la colonna diametro interno 75 µM con 1,9 particelle di fase stazionaria C18 µm (Vedi Tabella materiali). Caricamento del peptide su ogni colonna 5-µ l.
- Eluire peptidi utilizzando un gradiente lineare di acetonitrile 250 nL/min oltre 140 min e introdurre di nanoelectrospray in uno spettrometro di massa ibrido Quadropole trappola lineare (LTQ) accoppiato al sistema HPLC nanoLC.
- Raccogliere i dati di MS tandem per gli ioni più abbondanti di top 10 per scansione sopra una pendenza 140 minuti di tempo. Randomizzare l'ordine di raccolta di dati e di interscambio con BSA eseguita tra ogni campione per minimizzare la distorsione temporale.
5. analisi
- Elaborare i dati raw MS utilizzando impostazione predefinita all'interno di MaxQuant versione software 1.2.7.4 e analizzare MaxQuant output utilizzando una versione modificata del flusso di lavoro analisi statistica Perseo in ambiente software R3.
Nota: Il flusso di lavoro di analisi statistica da questo punto è riassunto nella Figura 2. Brevemente, intensità di IFQ delle proteine identificate utilizzando MaxQuant sono trasformati e filtrata seguita dalla normalizzazione e imputazione. Proteine d'interazione di coppie di dimero sono identificati dal confronto con un controllo di Venus, per escludere i raccoglitori di sfondo non specifici, con test t Benjamini-Hochberg correzione per i confronti multipli e dello studente. Qualità dei dati è confermata dal confronto di singoli istogrammi, regressione multipla e clustering gerarchico.

Figura 2: struttura del flusso di lavoro di analisi statistica. Diagramma di flusso della pipeline di analisi statistica utilizzata per analizzare le intensità di IFQ di proteine identificate dai dati di spettrometria di massa grezza elaborati utilizzando MaxQuant. Verde caselle: filtraggio, scatole blu: trasformazione/normalizzare/scalatura, marrone scatole: caselle di controllo di qualità, giallo di dati: finestre di analisi, grigio semi-quantitativa di esclusione/comparativa: analisi statistica. Questa figura è ripubblicato fromCroucher et al.3 ristampato con il permesso di AAAS. Clicca qui per visualizzare una versione più grande di questa figura.
Risultati
Segue l'uso della clonazione di ricombinazione per generare delle V1 e V2 tagged geni di interesse con i plasmidi del pDEST BiFC, co-transfezione di due plasmidi contenenti una coppia di proteine interagenti comporterà la generazione di un segnale fluorescente Venus dopo circa 8-24 ore. In assenza di un segnale positivo, che è possibile che l'interazione di proteine non può che si verificano a causa della scelta della linea cellulare, un'efficienza di trasfezione bassa o che l'orientam...
Discussione
BiCAP è un potente metodo per isolare i dimeri di proteina specifica escludendo i singoli componenti e loro concorrenti associazione partner3. BiCAP si basa sull'adattamento di un'analisi di complementazione di fluorescenza della proteina chiamata BiFC6. I metodi esistenti, tra cui saggi di legatura BiFC e vicinanza, sono stati ampiamente utilizzati per visualizzare e quantificare le interazioni della proteina in cellule vive7, ma non hanno fornito ...
Divulgazioni
Gli autori non hanno nulla a rivelare
Riconoscimenti
RDC è un Cancer Institute NSW Fellow e D.N.S era precedentemente un Cancer Institute NSW Fellow. I risultati di ricerca presentati in questo manoscritto sono stati finanziati da Cancer Institute NSW (13/FRL/1-02 e 09/CDF/2-39), NHMRC (progetto Grant GNT1052963), Science Foundation Ireland (11/SIRG/B2157), NSW Office of Science e ricerca medica, famiglia ospite Fellowship e Mostyn Family Foundation. J.f.h. e R.S. sono stati destinatari di un australiano premio post-laurea.
Materiali
| Name | Company | Catalog Number | Comments |
| LR Clonase II Plus enzyme | Thermo Fisher Scientific | 12538120 | Recombinase enzyme required for Gateway cloning (Step 1) into pDEST BiFC destination vectors |
| Proteinase K, recombinant, PCR grade | Thermo Fisher Scientific | EO0491 | |
| 14 mL round-bottomed polypropylene tube | Corning | 352059 | |
| Ampicillin | Roche Diagnostics Australia | 10835242001 | Stock solution prepared at 100 μg/mL in distilled water. |
| Miniprep kit | Promega Corporation | A1330 | |
| Maxiprep kit | Life Technologies Australia | K2100-07 | |
| DMEM | Gibco | 11995-073 | |
| FBS | Life Technologies Australia | 10099-141 | |
| Penicillin/Streptomycin | Life Technologies Australia | 15070-063 | |
| jetPRIME transfection buffer | Polyplus | 114-15 | |
| jetPRIME transfection reagent | Polyplus | 114-15 | |
| PhosSTOP (Phosphatase inhibitor) | Sigma-Aldrich | 4906837001 | |
| cOmplete, Mini, EDTA-free Protease inhibitor cocktail | Roche | 11873580001 | |
| Cell Scraper | Sarstedt | 83.1832 | |
| GFP-Trap_A | Chromotek Gmbh | gta-100 | GFP nanobody coupled to agarose beads |
| N-terminal GFP monoclonal antibody | Covance | MMS-118P | Will detect the V1 tag within the BiFC vectors |
| C-terminal GFP monoclonal antibody | Roche | 11814460001 | Will detect the V2 tag within the BiFC vectors |
| Sample buffer | Invitrogen | NP0008 | Supplemented with 1 mL β-mercaptoethanol. |
| Sequencing grade modified trypsin | Promega Corporation | V5117h | |
| LoBind microcentrifuge tubes | Point of Care Diagnostics | 0030 108 116 | |
| Iodoacetamide | Sigma-Aldrich | I1149-5G | Prepared at 5 mg/mL in ultrapure water |
| Trifluoroacetic Acid - Sequanal Grade | Thermo Fisher | 10628494 | |
| 3M Empore solid phase extraction C18 disks (octadecyl) - 4.7 cm | Thermo Fisher | 14-386-2 | To prepare stage tips, cut 1 mm disks using an appropriately sized hole punch. Alternatively, pre-prepared stage tips can also be purchased, see below. |
| C18 Stage Tips, 10 µL bed | Thermo Fisher | 87782 | |
| Formic acid OPTIMA for LC/MS grade 50mL | Thermo Fisher | FSBA117-50 | |
| 1.9 μm C18 ReproSil particles | Dr. Maisch GmbH | r119.aq. | Stationary phase particles |
| Acetonitrile OPTIMA LC/MS grade | Thermo Fisher | FSBA955-4 | |
| Easy-nLC HPLC | Thermo Fisher | ||
| LTQ Orbitrap Velos Pro | Thermo Fisher | ||
| Triton X-100 | Sigma-Aldrich | T8787 | Non-ionic detergent (100%) |
| DH5α cells | Thermo Fisher | Heat-shock-competent cells |
Riferimenti
- Kolch, W., Pitt, A. Functional proteomics to dissect tyrosine kinase signalling pathways in cancer. Nat Rev Cancer. 10 (9), 618-629 (2010).
- Pawson, T., Kofler, M. Kinome signaling through regulated protein-protein interactions in normal and cancer cells. Curr Opin Cell Biol. 21 (2), 147-153 (2009).
- Croucher, D. R., et al. Bimolecular complementation affinity purification (BiCAP) reveals dimer-specific protein interactions for ERBB2 dimers. Sci Signal. 9 (436), ra69 (2016).
- Cassonnet, P., et al. Benchmarking a luciferase complementation assay for detecting protein complexes. Nat Methods. 8 (12), 990-992 (2011).
- Rossi, F., Charlton, C. A., Blau, H. M. Monitoring protein-protein interactions in intact eukaryotic cells by beta-galactosidase complementation. Proc Natl Acad Sci U S A. 94 (16), 8405-8410 (1997).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism. J Am Chem Soc. 127 (1), 146-157 (2005).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21 (5), 539-545 (2003).
- Kubala, M. H., Kovtun, O., Alexandrov, K., Collins, B. M. Structural and thermodynamic analysis of the GFP:GFP-nanobody complex. Protein Sci. 19 (12), 2389-2401 (2010).
- Fegan, A., White, B., Carlson, J. C., Wagner, C. R. Chemically controlled protein assembly: techniques and applications. Chem Rev. 110 (6), 3315-3336 (2010).
- JoVE Science Education Database. Basic methods in cellular and molecular biology. plasmid purification. J Vis Exp. , (2017).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Shearer, R. F., et al. The E3 ubiquitin ligase UBR5 regulates centriolar satellite stability and primary cilia formation via ubiquitylation of CSPP-L. Mol Biol Cell. , (2018).
- Shannon, P., et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks. Genome Res. 13 (11), 2498-2504 (2003).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, 15690 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon