このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
解離性個体の親和性の浄化 (BiCAP) によって多蛋白質シグナリング複合体
要約
本稿では、二分子の相補性の親和性の浄化 (BiCAP) のプロトコルについて説明します。この手法は競合結合パートナーで形成された複合体と同様、国連複合体の個々 のタンパク質を除く、特定の分離と任意の 2 つの相互作用の蛋白質の下流のプロテオーム解析を促進します。
要約
蛋白質の複合体のアセンブリは多くの細胞シグナル伝達経路の調節中枢機構です。生物医学研究の主要な焦点は、どのようにこれらの動的蛋白質の複合体は特定の生物学的応答を送信するために複数のソースからの信号を統合する行動し、どのように多くの病気設定この自由化になる解読です。この重要な生化学的メカニズムの重要性にもかかわらずこれらの多分子シグナリング複合体の特定および敏感なデコンボリューションを容易にすることができます実験技術の欠如があります。
ここでこの欠点については、特定の構造生物学: ナノボディで、我々 が分子の相補性の親和性の浄化 (BiCAP) と呼ばれる、タンパク質の否定回路試金の組み合わせにより対処します。この手法は、特定の分離と国連複合体の個々 の蛋白質と競合結合パートナーで形成された複合体の排除の相互作用の蛋白質のペアの下流のプロテオーム解析を促進します。
BiCAP 手法は下流の実験的アッセイの広い配列に適応でき、この手法によって与えられる特異性の高いより微妙な捜査蛋白質複雑なアセンブリの力学、現在よりもを使用して標準的な親和性の浄化技術。
概要
蛋白質複雑なアセンブリは多くのシグナル伝達経路の1,2の時空間特異性を維持するために重要なプロセスです。この規制の役割の重要性が広く認識され、これらの複合体を精査する使用できる実験手法の欠如があります。Interactomics 研究のほとんどは個々 のタンパク質や各複雑なコンポーネントの連続濃縮との相互作用に焦点を当てます。ここで競争パートナー3を連結して形成される複合体と同様、タンパク質の個々 の部分を除く、特定の蛋白質の二量体の分離手法を提案します。我々 はこの手法を求めている分子の相補性の親和性の浄化 (BiCAP) それとしての小説を使って既存タンパク質フラグメント相補性アッセイは二分子の蛍光補完 (BiFC) の組み合わせである、GFP とその誘導体に向かって生物学: ナノボディでコンホメーション特異の組換え (を参照してください材料テーブル).
典型的なタンパク質フラグメント否定回路試金はルシフェラーゼ4β-ガラクトシダーゼ5、緑色蛍光タンパク質 (GFP)6 (などの記者のフラグメントに分割する溶ける「餌」と「獲物」蛋白質の表現に依存しています。図 1 a)。餌や餌の蛋白質の相互作用によって分割記者ドメインお勧め餌の対話ができるよう、機能的な構造にフォールディングを可視化したり定量化タンパク質を捕食します。BiCAP 作られたこの技術のバージョンから適応された GFP バリアント金星のフラグメントの使用します。蛍光蛋白質否定回路試金は生細胞でのタンパク質間相互作用を可視化するため人気のある方法ですが、今までこの関数を 1 つの7に限られています。BiCAP としてこの手法だけでなく、可視化が、また分離と尋問結果の蛋白質蛋白質の相互作用の点で重要な進歩を表します。
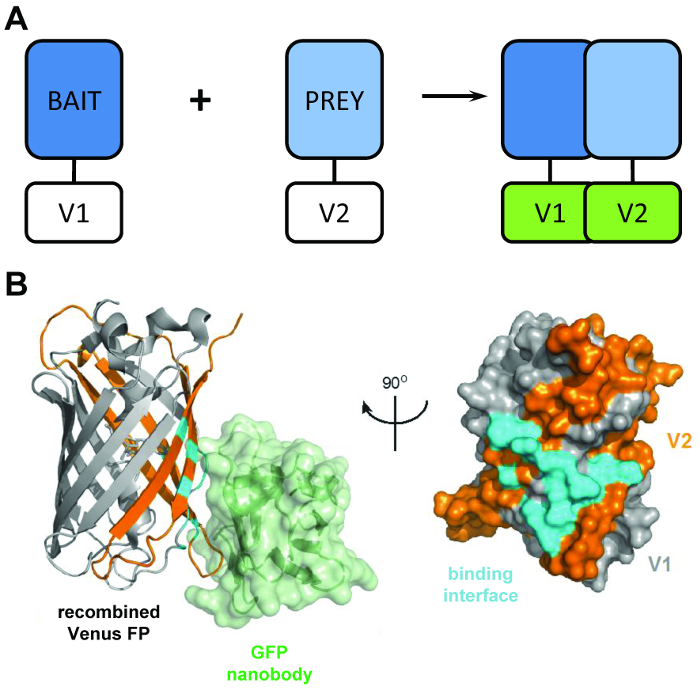
図 1: BiCAP 手法の背後にある構造のプリンシパル。(A) 分子蛍光補完表示の背後にあるプリンシパルをアウトライン図 N ターミナル V1 または V2 の C 末端タグ付き '餌' と '捕食' 蛋白質フルレングスの金星タンパク質の断片。(B) GFP 生物学: ナノボディで (緑) と (グレー) V1 と V2 (オレンジ) フラグメント (PDB 加盟 3OGO) の位置を示すられた金星の間 (シアン) 相互作用界面の構造解析。この図は再発行 fromCroucher ら3転載 AAAS から権限を持つです。この図の拡大版を表示するのにはここをクリックしてください。
フュージョン パートナー間の相互作用で発生しない限り親和性の低さに関連付ける (V1 と V2 の名前) 金星の 2 つの非蛍光性フラグメントの手法により BiCAP を使用します。このインスタンスで、2 つの分割ドメインは fluorophore (図 1 b)6の機能 β バレル構造にフォールディングします。BiCAP の重要な技術革新は、組換え GFP 生物学: ナノボディで、正しくられたと折り畳まれた蛍光体 (にある β バレル GFP (と金星などの亜種) の三次元のエピトープを認識するの導入から来る図 1 b)8。重大に、GFP の生物学: ナノボディでは個々 の金星フラグメントのいずれかにバインドされません。これは蛋白質二量体の分離を促進する後にのみ 2 つのタンパク質は、化学物質による、強制相互作用9を使用するメソッドから取得したものより代表的な結果につながる、彼ら自身の意志の複合体を形成しています。
BiCAP は特にこれらの複合体のシグナル伝達における役割の理解の粒度を改善するために下流のアプリケーション数併用できる可能性のある複数のタンパク質を中心に強力な手法.また、蛋白質の相互作用の場の可視化を許可する重要な機能が含まれます。まで BiCAP の受容体チロシン キナーゼ (RTK) ダイマー3のインタラクトームを解析の効果的な方法として実証されていますが、この方法の適応性を意味するそれはほぼすべてのタンパク質相互作用のコンテキストで採用されることができます。
プロトコル
1. プラスミッドのクローニング
注: プラスミッドのベクトルを N 末端または C 末端の興味の遺伝子の融合した V1 または V2 タグを生成するため BiFC デスティネーション ベクトルを Addgene に堆積した [N 末端タグ: pDEST-V1-ORF (#73635)、pDEST-V2-ORF (#73636)。C 末端タグ: pDEST-ORF-V1 (#73637)、pDEST-ORF-V2 (#73638)]。興味の遺伝子/s は、コドン、クローン作成を続行することがなく特定の組み変えの互換性エントリ ベクトル (すなわち、 pDONR223 または pENTR221) をクローニングにする必要があります。互換性の多くのクローンは、哺乳類のゲノムのコレクション (https://genecollections.nci.nih.gov/MGC/) を含む、さまざまなプラスミド コレクション内で使用できるいます。
- 適切な BiFC キャスト先のベクトルの attR サイト間興味の遺伝子を挿入する再結合反応を実行します。
- 0.2 mL チューブ 150 ng エントリ ベクトル、150 ng BiFC キャスト先のベクトル、および 8 μ L TE バッファー (pH 8.0) を追加します。
- 2 μ L シュプリンガー酵素ミックスを追加 (材料の表を参照してください)。よく混ぜ、遠心分離機の簡潔に。
- 一晩室温でまたは 16 ° C で 1 時間反応を孵化させなさい。
- 1 μ L プロテイナーゼ K 溶液を加え、10 分の 37 ° C で培養して反応を停止します。
- LR 反応生成物と熱衝撃有能なセル (材料の表を参照) を変換するには。
注: 任意の基本的なひずみ多分使用できますこの時点で挿入を含んでいるプラスミッドの特定の選択を妨げる Ccdb 抗毒素が含まれていない限り。- 氷の上の有能なセルを解凍し、14 mL 丸底ポリプロピレン チューブに細胞の 50 μ L を転送します。
- セルに 1 μ L の反応生成物を加え、軽く混ぜます。氷の上に 20 分間インキュベートします。
- 45 42 ° C の水浴の衝撃を熱 s. を 2 分間氷にすぐに戻る。
- ML の LB 培地 1 を追加し、37 ° c 1 時間振とうしながらインキュベートします。
- プレートのアンピシリンを含む 10 cm 寒天プレート上に変換 (100 μ g/mL) し、37 ° C で一晩インキュベートします。
- BiFC プラスミッドの浄化。
- 個々 のコロニーからのプラスミッドを浄化する大型三角フラスコに mL の LB 培地 100 を追加し、アンピシリンを追加 (100 μ g/mL)。複数のコロニーを画面に 14 mL 丸底のポリプロピレン管に 5 mL の LB 培地を追加し、アンピシリンを追加 (100 μ g/mL)。
- 滅菌接種ループを使用すると、寒天培地プレートから単一コロニーを拾います。LB メディアに接種ループを配置し、一時的に混ぜます。
- アルミ箔の三角フラスコの上部を覆うし、揺れで 37 ° C で一晩インキュベートします。
- 標準的な maxiprep または midiprep プラスミド DNA を使用してトランスフェクション グレード プラスミド DNA を作り出す浄化キット (材料の表を参照してください)10。吸光度スペクトルによる DNA の品質を評価します。
メモ: DNA が必要 A260/A280 比 > 1.8 と A260/A230 比 > 2.0。この時点で、挿入とタグの効率的な表現をチェックするため複数の個々 のコロニーを選別することをお勧めします。
2. 細胞培養とトランスフェクション
注: BiFC ベクトルの transfection のため重要です高効率、比較的均質なトランスフェクションを達成するために。ベクトルは、任意の標準的なトランスフェクション試薬と互換性がある可能性が高いとトランスフェクション条件をそれに応じて最適化する必要があります。質量分析を実行するには、私たち通常細胞の培養 10 cm 皿内でこのことができますも比例してにスケール ダウン小さな皿またはより少ない材料を必要とする実験のためのプレートが。
- 種子 1.0 × 106 HEK293T 細胞 10 cm 皿 mL の 10% を添加した DMEM 培地 10 FBS とペニシリン/ストレプトマイシン (1: 100)。
- 遺伝子導入バッファーの 500 μ L に各 BiFC ベクトルの 2.5 μ g を希釈 (材料表参照) 1.5 mL 遠心チューブに。
- トランスフェクション試薬を 10 μ l 添加 (材料の表を参照してください)。
- 10 の s、そして簡潔に遠心分離渦混合物。室温で 10 分間インキュベートします。
- プレートに滴下 DNA トランスフェクション混合物を追加します。対話する BiFC 融合蛋白質折りたたみ金星、fluorophore 成熟、一般的に 8 〜 24 時間の時間の十分な長さのための細胞を孵化させなさい。
注: 金星の最大励起、515 nm とそのピーク発光は 528 nm の, これは容易に標準的な蛍光顕微鏡 GFP 蛍光を視覚化するように設定を使用して表示されますが。
3. サンプル準備
-
Lysates を収穫
- ライセートのコレクション前にセル換散バッファーの準備 [50 mM トリス-HCl (pH 7.4) 150 mM NaCl、1 mM EDTA, 1% (v/v) 非イオン性洗剤 (材料の表を参照してください)]。4 ° C でのストア
- 収穫の直前にプロテアーゼ阻害剤・ フォスファターゼ阻害剤 (材料の表を参照してください) とセル換散バッファーの 10 mL を補足します。氷の上の補われた細胞換散バッファーを維持します。
- 氷冷 PBS で 2 回細胞を洗浄します。PBS を吸引し、冷たい補われた細胞換散バッファーの 1 つの mL を追加します。氷の上の皿を配置、表面全体の面積に広がっているバッファーを確保します。
- 細胞スクレーパーを使用して、約 5 分間氷の上を孵化させなさい (材料表を参照)、細胞をこすり中古冷蔵 1.5 mL 遠心チューブに転送します。
- 収集した遠心分離によって細胞の残骸を削除 18,000 にて 4 ° C と転送で 5 分間 × g 新鮮な遠心チューブに上清をクリアします。
注: この時点でライセート、即座に使用したり-80 ° C で保存また、トランスフェクションの親和性の浄化効率を検証可能なので原油の因数のライセートを維持することをお勧めします。
-
親和性の浄化
メモ: BiCAP 分離ステップはアガロース ビーズ (材料の表を参照してください) に共役生物学: ナノボディで GFP を使用して実行されます。- アガロース ビーズを準備するには、1 ml の PBS (サンプルあたり 20 μ L) + 10 μ L 余分な適切なボリュームを洗浄します。300 x g でビーズを遠心し、上清を除去します。
- 20 μ L を各ライセート サンプルに追加します。
- エンド ツー エンドの回転と 4 ° C で 2 時間サンプルをインキュベートします。
注: この時点で、サンプル準備できます、SDS ページおよび西部のしみが付くこと、または解析質量分析法による。
-
西部にしみが付くことのため BiCAP 溶離液の準備。
- 300 x gとセル換散バッファーで 3 回洗浄でビーズを遠心します。
- 適切に希釈サンプル バッファーの 50 μ L で洗浄のビーズを再懸濁します (材料の表を参照してください) と 2-3 分 95 ° C でサンプルの熱します。
注: この方法で作製したサンプルは-20 ° C で数ヶ月保存できます。 - SDS のページおよび興味の他の同様の蛋白質としてウエスタンブロット11 V1 タグと V2 タグ (材料の表参照) の両方を実行します。
-
BiCAP 溶離液の質量分析のための準備。
注: 少なくとものサンプルを準備すること勧め定量的ラベル無料 (LFQ) 質量分析法による分析のため, 堅牢な統計的検出力を確保するため。- 300 x g とセル換散バッファー洗剤なしで 6 回洗浄でビーズを遠心します。これは過剰なタンパク質と質量分析を妨げる洗剤両方を削除する必要は。すぐに、次のステップに進むまたは-80 ° c.ビーズを格納
- Trypsinize ビーズ 60 μ L のバッファー 1 [2 M 尿素、50 mM トリス-HCl (pH 7.5)、5 μ g/mL のトリプシン]。800 RPM で揺れ thermomixer 27 ° C で 30 分間のダイジェストにビーズを許可します。
- 簡単にビーズを遠心し、(材料表参照) 遠心チューブに上清および転送を収集します。
- 25 μ L でビーズを洗浄バッファー 2 [50 mM トリス-HCl (pH 7.5) 1 mM ジチオトレイトール 2 M 尿素] バインドされたタンパク質を減らすために。
- 1 つ遠心チューブにビーズと上澄みをプールします。室温で一晩で発生する消化力を許可します。
- ヨードアセトアミド (5 mg/mL の純水) の 20 μ L を各サンプルに追加することで、サンプルをアルキレートし、暗闇のなかで 30 分間インキュベートします。
- 1 μ L トリフルオロ酢酸 (TFA) で各サンプルを処理することによって消化を阻害します。この手順がまた舞台転換のための準備のサンプルを酸性化します。
- コンストラクト C18 ステージによる重なり 6 層 1 mm 固相抽出 C18 (オクタデシル) 200 μ L ピペット チップに膜ディスク (材料の表参照) のヒントします。各サンプルの単一の先端を準備します。
- メタノール、ステージのヒントをウェットし、50 μ L 0.1% (v/v) トリフルオロ酢酸 (TFA) 80% (v/v) アセトニ トリルと平衡します。
- 50 μ L 0.1% (v/v) TFA のヒントを洗浄します。
- ステージのヒントに酸性ペプチドを読み込み、0.1% (v/v) TFA、アセトニ トリル 80% (v/v) 2 つの手順を使用して、溶出します。
- 真空濃縮を使用してサンプルを蒸発させます。
- 必要な場合は-80 ° C で乾燥したペプチドを格納します。
4. 質量。
- (超純水) で 2% アセトニ トリル 5% ギ酸の 15 μ L のサンプルを再懸濁します。
- 慎重に LC プレートと場所に 6 μ L を安価の HPLC システムに読み込む (材料の表を参照してください)。
- パック 20 cm、75 μ M 内径列 1.9 μ m C18 固定相の粒子 (材料の表を参照してください)。各列に 5 μ L ペプチドをロードします。
- 250 nL/min 以上 140 分でアセトニ トリルの線形グラデーションを用いるペプチドの溶出し、安価の HPLC システムに結合された線形トラップ四重極 (焼入れ) ハイブリッド質量 nanoelectrospray によって紹介します。
- 時間 140 分勾配以上スキャンごとのトップ 10 最も豊富なイオンのタンデム MS データを収集します。BSA のバイアスを最小限に抑えるために各サンプルの実行とデータの収集とインターチェンジの順序をランダムします。
5. 分析
- MaxQuant ソフトウェア バージョン 1.2.7.4 内で既定の設定を使用して生の MS データを処理し、MaxQuant の R ソフトウェア環境3ペルセウス統計解析ワークフローの修正バージョンを使用して出力を分析します。
注: このポイントから統計解析のワークフローは、図 2のとおりです。簡単に、MaxQuant を使用して識別される蛋白質の LFQ 強度変換、正規化と帰属続いてフィルターします。ダイマーのペアの相互作用の蛋白質は金星制御、非特異的なバック グラウンド バインダー、スチューデント t 検定と多重比較の Benjamini ホフベルク補正を除外すると識別されます。データ品質は、個々 のヒストグラム、回帰、および階層的クラスタ リングの比較によって確認されます。

図 2: 統計解析ワークフローのアウトライン。MaxQuant を使用して処理される生の質量分析データからタンパク質の LFQ 強度を分析するための統計的解析パイプラインのフロー図。グリーン ボックス: フィルター、青色のボックス: 変換/を規準に合わせる/スケーリング、茶色のボックス: データ品質管理、黄色のボックス: 半定量的な除外・比較分析、グレー ボックス: 統計解析。この図は再発行 fromCroucher ら3転載 AAAS から権限を持つです。この図の拡大版を表示するのにはここをクリックしてください。
結果
タグ付き BiFC pDEST プラスミドを興味のある遺伝子の組換えの V1 と V2 の生成するクローニングの使用に続く、蛋白質の相互作用のペアを含む 2 つのプラスミドとの共トランスフェクション後の金星の蛍光信号の生成になります約 8 〜 24 時間。それはタンパク質間相互作用が細胞の選択により発生する可能性がない可能な肯定的な信号がない場合は、低トランスフェク?...
ディスカッション
BiCAP は、個別のコンポーネントおよび彼らの競合結合パートナー3を排除しながら特定の蛋白質の二量体を隔離するための強力な方法です。BiCAP は BiFC6と呼ばれる蛍光蛋白質否定回路試金の適応に基づいています。BiFC と近接の結紮アッセイを含む既存の方法7生きているセルの蛋白質の相互作用を定量化を視覚化して広く使用されているが...
開示事項
著者がある何も開示するには
謝辞
D.R.C がん研究所のニューサウス ウェールズ州の研究員であり D.N.S 癌研究所のニューサウス ウェールズ州の研究員であった。がん研究所 NSW (13/FRL/1-02 ・ 09/CDF/2-39)、NHMRC (プロジェクト助成金 GNT1052963)、アイルランド科学財団 (11/SIRG/B2157)、ニューサウス ウェールズ州のオフィスの科学と医学研究、客家によって資金が供給された研究成果を本稿では発表親睦とモスティン家族財団。J.F.H. と r. s. オーストラリアの大学院賞受賞者であった。
資料
| Name | Company | Catalog Number | Comments |
| LR Clonase II Plus enzyme | Thermo Fisher Scientific | 12538120 | Recombinase enzyme required for Gateway cloning (Step 1) into pDEST BiFC destination vectors |
| Proteinase K, recombinant, PCR grade | Thermo Fisher Scientific | EO0491 | |
| 14 mL round-bottomed polypropylene tube | Corning | 352059 | |
| Ampicillin | Roche Diagnostics Australia | 10835242001 | Stock solution prepared at 100 μg/mL in distilled water. |
| Miniprep kit | Promega Corporation | A1330 | |
| Maxiprep kit | Life Technologies Australia | K2100-07 | |
| DMEM | Gibco | 11995-073 | |
| FBS | Life Technologies Australia | 10099-141 | |
| Penicillin/Streptomycin | Life Technologies Australia | 15070-063 | |
| jetPRIME transfection buffer | Polyplus | 114-15 | |
| jetPRIME transfection reagent | Polyplus | 114-15 | |
| PhosSTOP (Phosphatase inhibitor) | Sigma-Aldrich | 4906837001 | |
| cOmplete, Mini, EDTA-free Protease inhibitor cocktail | Roche | 11873580001 | |
| Cell Scraper | Sarstedt | 83.1832 | |
| GFP-Trap_A | Chromotek Gmbh | gta-100 | GFP nanobody coupled to agarose beads |
| N-terminal GFP monoclonal antibody | Covance | MMS-118P | Will detect the V1 tag within the BiFC vectors |
| C-terminal GFP monoclonal antibody | Roche | 11814460001 | Will detect the V2 tag within the BiFC vectors |
| Sample buffer | Invitrogen | NP0008 | Supplemented with 1 mL β-mercaptoethanol. |
| Sequencing grade modified trypsin | Promega Corporation | V5117h | |
| LoBind microcentrifuge tubes | Point of Care Diagnostics | 0030 108 116 | |
| Iodoacetamide | Sigma-Aldrich | I1149-5G | Prepared at 5 mg/mL in ultrapure water |
| Trifluoroacetic Acid - Sequanal Grade | Thermo Fisher | 10628494 | |
| 3M Empore solid phase extraction C18 disks (octadecyl) - 4.7 cm | Thermo Fisher | 14-386-2 | To prepare stage tips, cut 1 mm disks using an appropriately sized hole punch. Alternatively, pre-prepared stage tips can also be purchased, see below. |
| C18 Stage Tips, 10 µL bed | Thermo Fisher | 87782 | |
| Formic acid OPTIMA for LC/MS grade 50mL | Thermo Fisher | FSBA117-50 | |
| 1.9 μm C18 ReproSil particles | Dr. Maisch GmbH | r119.aq. | Stationary phase particles |
| Acetonitrile OPTIMA LC/MS grade | Thermo Fisher | FSBA955-4 | |
| Easy-nLC HPLC | Thermo Fisher | ||
| LTQ Orbitrap Velos Pro | Thermo Fisher | ||
| Triton X-100 | Sigma-Aldrich | T8787 | Non-ionic detergent (100%) |
| DH5α cells | Thermo Fisher | Heat-shock-competent cells |
参考文献
- Kolch, W., Pitt, A. Functional proteomics to dissect tyrosine kinase signalling pathways in cancer. Nat Rev Cancer. 10 (9), 618-629 (2010).
- Pawson, T., Kofler, M. Kinome signaling through regulated protein-protein interactions in normal and cancer cells. Curr Opin Cell Biol. 21 (2), 147-153 (2009).
- Croucher, D. R., et al. Bimolecular complementation affinity purification (BiCAP) reveals dimer-specific protein interactions for ERBB2 dimers. Sci Signal. 9 (436), ra69 (2016).
- Cassonnet, P., et al. Benchmarking a luciferase complementation assay for detecting protein complexes. Nat Methods. 8 (12), 990-992 (2011).
- Rossi, F., Charlton, C. A., Blau, H. M. Monitoring protein-protein interactions in intact eukaryotic cells by beta-galactosidase complementation. Proc Natl Acad Sci U S A. 94 (16), 8405-8410 (1997).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism. J Am Chem Soc. 127 (1), 146-157 (2005).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21 (5), 539-545 (2003).
- Kubala, M. H., Kovtun, O., Alexandrov, K., Collins, B. M. Structural and thermodynamic analysis of the GFP:GFP-nanobody complex. Protein Sci. 19 (12), 2389-2401 (2010).
- Fegan, A., White, B., Carlson, J. C., Wagner, C. R. Chemically controlled protein assembly: techniques and applications. Chem Rev. 110 (6), 3315-3336 (2010).
- JoVE Science Education Database. Basic methods in cellular and molecular biology. plasmid purification. J Vis Exp. , (2017).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Shearer, R. F., et al. The E3 ubiquitin ligase UBR5 regulates centriolar satellite stability and primary cilia formation via ubiquitylation of CSPP-L. Mol Biol Cell. , (2018).
- Shannon, P., et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks. Genome Res. 13 (11), 2498-2504 (2003).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, 15690 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved