Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Disección señalización complejos por purificación de la afinidad de complementación Bimolecular (BiCAP)
En este artículo
Resumen
Este manuscrito describe el protocolo para la purificación de afinidad de complementación Bimolecular (BiCAP). Este novedoso método facilita el aislamiento específico y la caracterización proteómica aguas abajo de cualquier dos proteínas obran recíprocamente, mientras que excluyendo un complejado proteínas individuales como complejos formados con competidores socios de enlace.
Resumen
El ensamblaje de complejos de proteínas es un mecanismo central subyacente a la regulación de muchas vías de señalización de la célula. Un enfoque principal de la investigación biomédica es descifrar cómo estos complejos proteína dinámica actúan para integrar señales de varias fuentes para dirigir una respuesta biológica específica, y cómo esto se convierte en liberalizado en muchos contextos de la enfermedad. A pesar de la importancia de este mecanismo bioquímico clave, hay una falta de técnicas experimentales que pueden facilitar la deconvolución específico y sensible de estos complejos multi moleculares de señalización.
Esta carencia se dirige aquí a través de la combinación de un ensayo de complementación de proteínas con un nanobody específicos de conformación, que hemos llamado purificación de afinidad de complementación Bimolecular (BiCAP). Esta nueva técnica facilita el aislamiento específico y la caracterización proteómica aguas abajo de cualquier par de interacción de proteínas, a la exclusión de un complejo proteínas individuales y complejos formados con competidores socios de enlace.
La técnica del BiCAP es adaptable a una amplia gama de ensayos experimentales aguas abajo, y el alto grado de especificidad de esta técnica permite más matizada las investigaciones sobre la mecánica del conjunto complejo de proteínas que es actualmente posible utilizar técnicas de purificación de afinidad estándar.
Introducción
Conjunto complejo de proteínas es un proceso clave en el mantenimiento de la especificidad espaciotemporal de muchos señalización vías1,2. Mientras que la naturaleza crítica de esta función reguladora es ampliamente reconocida, hay una falta de técnicas experimentales para estudiar estos complejos. La mayoría interactómica estudios se centran en las interacciones con las proteínas individuales, o el enriquecimiento secuencial de componentes complejos. Presentamos aquí una técnica para el aislamiento de un dímero de la proteína específica mientras que excluyendo las moléculas individuales de la componente de proteínas así como complejos formados con competencia vinculante socios3. Hemos llamado a esta técnica de purificación de afinidad de complementación Bimolecular (BiCAP), ya que es una combinación de un ensayo de complementación de fragmento de previamente existente proteína, complementación Bimolecular de la fluorescencia (centro), con el uso nuevo de un conformación específica nanobody recombinante GFP y sus derivados (ver tabla de materiales).
Un ensayo de complementación de fragmento de proteína típica se basa en la expresión de proteínas "presa" y el "cebo" fundido para dividir fragmentos de reporteros como luciferase4, β-galactosidasa5o proteína fluorescente verde (GFP)6 ( Figura 1A). A través de la interacción de las proteínas cebo y presa, los dominios de reportero de split se anima a doblar en una estructura funcional, que permiten la interacción del cebo y presa de proteínas para ser visualizado o cuantificado. BiCAP fue adaptado de una versión de esta técnica que hace uso de fragmentos de la variante de la GFP Venus. Ensayos de complementación de proteínas fluorescentes son un método popular para la visualización de las interacciones proteína-proteína en una célula viva, pero hasta ahora se han limitado a esta un función7. BiCAP representa un avance significativo en este sentido, esta técnica permite no sólo para visualización, pero también el aislamiento y el interrogatorio de la interacción proteína-proteína resultante.
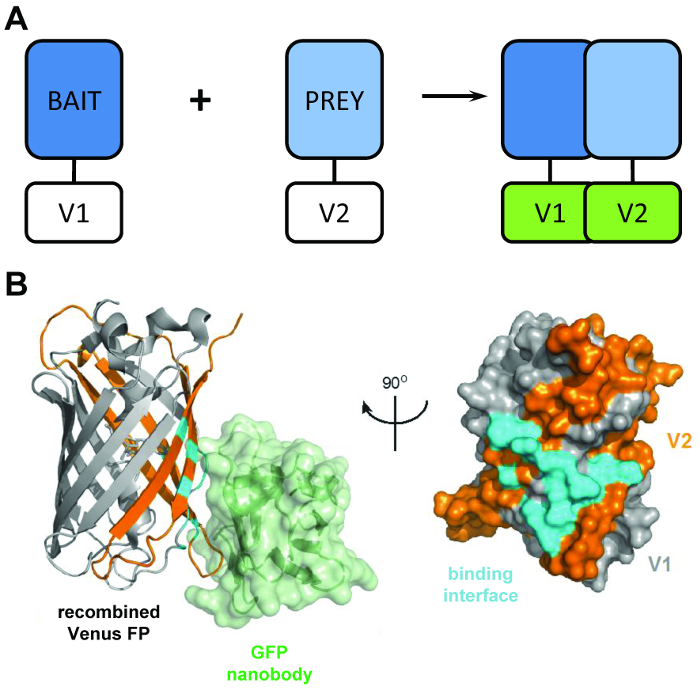
Figura 1: la estructural principal detrás de la técnica del BiCAP. (A) un esquema que al Director detrás de muestra de complementación bimolecular de la fluorescencia las proteínas 'bait' y 'presa' con la etiqueta el N-terminal V1 o V2 C-terminal fragmentos de la proteína integral de Venus. (B) análisis estructural de la interfaz de interacción (cian) entre la GFP nanobody (verde) y Venus recombinada, mostrando la posición de la V1 (gris) y fragmentos de V2 (naranja) (PDB 3OGO de adhesión). Esta cifra es republicado fromCroucher et al.3 reimpreso con permiso de la AAAS. Haga clic aquí para ver una versión más grande de esta figura.
El BiCAP técnica hace uso de dos fragmentos no fluorescente de Venus (llamado V1 y V2) que asocian con un bajo grado de afinidad, a menos que una interacción se produce entre sus socios de fusión. En este caso, los dominios de dos split doblarle en la estructura funcional del β-barril del fluoróforo (figura 1B)6. La innovación clave del BiCAP proviene de la introducción de la recombinante GFP nanobody, que reconoce un epitopo tridimensional en el β-barril de GFP (y variantes como Venus) que sólo está presente en el fluoróforo correctamente recombinado y doblado ( Figura 1B)8. Crucial, la GFP nanobody no enlazar a cualquiera de los fragmentos individuales de Venus. Esto facilita el aislamiento de dímeros de proteínas sólo después de las dos proteínas forman un complejo de su propia voluntad, llevando a resultados más representativos que los adquirió de los métodos que hacen uso de interacciones químicamente inducida, forzada9.
BiCAP es una técnica poderosa que se centra específicamente en los complejos, que potencialmente pueden combinarse con un número de aplicaciones posteriores para mejorar el nivel de detalle de nuestra comprensión de estos complejos de papel en la transducción de señal . También incluye la característica importante de permitir la visualización de las interacciones de proteínas in situ. Hasta la fecha, BiCAP se ha demostrado como un método eficaz de analizar el interactoma de receptores tirosina quinasa (RTK) dímeros3, pero la capacidad de adaptación de este método significa que pueden adoptarse en casi cualquier contexto de interacción de proteínas.
Protocolo
1. plásmido clonación
Nota: Para generar vectores plásmidos con los tags V1 o V2 fusionados a la N-terminal o c-terminal del gen de interés, centro vectores de destino han sido depositadas con Addgene [etiqueta del N-terminal: pDEST-V1-ORF (#73635), pDEST-V2-ORF (#73636). Etiqueta C-terminal: pDEST-ORF-V1 (#73637), pDEST-ORF-V2 (#73638)]. Los gene/s de interés tendrá que ser en recombinación específica vectores de entrada compatibles (es decir, pDONR223 o pENTR221), la clonación sin codones de parada, para proceder con la clonación. Muchos clones compatibles están disponibles en varias colecciones de plásmido, incluyendo la colección de genoma de mamíferos (https://genecollections.nci.nih.gov/MGC/).
- Llevar a cabo una reacción de recombinación para insertar el gen de interés entre los sitios de atributos del vector de destino centro apropiado:
- En un tubo de 0.2 mL, añadir 150 ng entrada vector, vector de destino de centro de ng 150 y 8 μl de tampón TE (pH 8.0).
- Añadir 2 μl recombinase enzima mezclar (véase Tabla de materiales). Mezclar bien y centrifugar brevemente.
- Incubar la reacción durante 1 hora a temperatura ambiente o a 16 ° C durante la noche.
- Detener la reacción agregando solución de proteinasa-K de 1 μl y incubando a 37 ° C durante 10 minutos.
- Transformar células calor-choque-competentes (véase Tabla de materiales) con el producto de reacción de LR:
Nota: Cualquier tensión básica puede concebible utilizar en este punto, siempre y cuando no contenga la antitoxina Ccdb, que impediría la selección específica de plásmidos que contienen el inserto.- Descongelar las células competentes en el hielo y transferir 50 μl de las células en un tubo de polipropileno fondo redondo de 14 mL.
- Añadir 1 μl del producto de la reacción a las células y mezclar suavemente. Incubar por 20 min en hielo.
- Choque en baño de agua de 42 ° C por 45 de calor s. inmediatamente vuelta al hielo durante 2 minutos.
- Añadir 1 mL de medio LB e incubar con agitación a 37 ° C durante 1 h.
- Placa de la transformación en una placa de agar de 10 cm conteniendo ampicilina (100 μg/mL) e incubar a 37 ° C durante la noche.
- Purificación del plásmido de centro.
- Para purificar el plásmido de una colonia individual, añadir 100 mL de medio LB a un matraz cónico grande y añadir ampicilina (100 μg/mL). Para colonias múltiples de la pantalla, añadir 5 mL de medio LB a tubos de polipropileno fondo redondo 14 mL y añadir ampicilina (100 μg/mL).
- Utilizando un asa de inoculación estéril, escoge una sola Colonia de la placa de agar. Coloque el asa de inoculación en los medios de comunicación LB y mezclar brevemente.
- Cubrir la parte superior del matraz cónico con papel de aluminio e incubar durante una noche a 37 ° C con agitación.
- Producir la transfección grado plásmido ADN utilizando un estándar maxiprep o midiprep ADN plásmido purificación kit (véase Tabla de materiales)10. Evaluar la calidad del ADN por espectros de absorbancia.
Nota: El ADN debe tener una relación A260/A280 > 1.8 y una relación A260/A230 > 2.0. En este punto, se recomienda para múltiples colonias individuales para verificar la eficiente expresión de inserción y etiqueta.
2. cultura y transfección de la célula
Nota: Para la transfección de los vectores de centro es importante lograr una alta eficiencia y transfección relativamente homogénea. Los vectores será compatibles con cualquier reactivo de transfección estándar, y las condiciones de transfección deben optimizarse en consecuencia. Para realizar la espectrometría de masas, generalmente cultura células dentro de platos de 10 cm, aunque esto puede también ser proporcionalmente reducido a pequeños platos o placas para experimentos que requieren menos material.
- Semillas 1.0 × 106 HEK293T células en 10 cm para platos con 10 mL de medio DMEM, suplementado con 10% FBS penicilina/estreptomicina (1: 100).
- Diluir 2,5 μg de cada vector de centro en 500 μl de tampón de transfección (véase Tabla de materiales) en un tubo de microcentrífuga de 1,5 mL.
- Añadir 10 μl de reactivo de transfección (véase Tabla de materiales).
- Mezcla de Vortex durante 10 s, y después brevemente centrífuga. Incubar 10 min a temperatura ambiente.
- Añadir la mezcla de la transfección de ADN a la placa, gota a gota. Incube las células durante un período suficiente de tiempo para las proteínas de fusión de centro interactuar y Venus plegamiento y maduración de fluoróforo, generalmente de 8-24 h.
Nota: La excitación máxima para Venus es 515 nm y la emisión máxima es de 528 nm, aunque esto es fácilmente visto con un microscopio fluorescente estándar para visualizar la fluorescencia de GFP.
3. preparación de la muestra
-
Cosecha de lisados
- Antes de la recogida de lisado, preparar Buffer de lisis de la célula [50 mM Tris-HCl (pH 7.4), 150 mM NaCl, 1 mM EDTA, 1% (v/v) de detergente no iónico (véase Tabla de materiales)]. Almacenar a 4 ° C.
- Inmediatamente antes de la cosecha, suplemento 10 mL de tampón de lisis celular con inhibidor de la proteasa y fosfatasa inhibidor (véase Tabla de materiales). Mantener el tampón de lisis celular suplementado en el hielo.
- Lavar las células dos veces en PBS helado. Aspire el PBS y añadir 1 mL de tampón de lisis de células complementado helada. Coloque el plato en hielo, asegurando que el búfer se extiende sobre la superficie.
- Incubar en hielo por aproximadamente 5 minutos, luego use un raspador celular (véase Tabla de materiales) para raspar las células y transferir a un tubo de microcentrífuga de 1,5 mL previamente enfriada.
- Eliminar desechos celulares por centrifugación la recogida lisado en 18.000 × g por 5 min a 4 ° C y transferencia despejó el sobrenadante en tubos de microcentrífuga fresco.
Nota: en este punto el lisado puede ser utilizado inmediatamente, o almacenado a-80 ° C. Se recomienda también mantener una alícuota de crudo lisado para que puede validarse la eficacia de transfección y la purificación de la afinidad.
-
Purificación de la afinidad
Nota: El paso de aislamiento BiCAP se realiza utilizando la GFP nanobody conjugado con perlas de agarosa (véase Tabla de materiales).- Preparar las perlas de agarosa lavando un volumen adecuado (20 μl por cada muestra) + exceso 10 μL en 1 mL de PBS. Los granos a 300 x g de centrífuga y eliminar el sobrenadante.
- Añadir 20 μl de cada muestra lisado.
- Incubar las muestras durante 2 h a 4 ° C con rotación de end-to-end.
Nota: en este punto las muestras pueden prepararse para SDS-PAGE y western blot o análisis por espectrometría de masas.
-
Preparación del BiCAP eluant para western blot.
- Centrifugue los granos a 300 x g y lavar tres veces en tampón de lisis celular.
- Resuspender los granos lavados en 50 μl de tampón de muestra adecuadamente diluido (véase Tabla de materiales) y calentar las muestras a 95 ° C durante 2-3 minutos.
Nota: Muestras preparadas de esta manera pueden conservarse a-20 ° C durante varios meses. - Realizar SDS-PAGE y western blot11 para la etiqueta de V1 y V2 etiquetas (véase tabla de materiales), como bien como cualquier otras proteínas de interés.
-
Preparación del BiCAP eluant para espectrometría de masas.
Nota: Para el análisis por espectrometría de masas (LFQ) libre de etiqueta cuantitativa se recomienda preparar las muestras en por lo menos cuadruplicar para asegurarse de poder estadístico robusto.- Centrifugue los granos a 300 x g y lavado seis veces en tampón de lisis celular sin detergente. Esto es necesario para eliminar el exceso proteína y el detergente que interferirá con el análisis de espectrometría de masas. Proceder al paso siguiente inmediatamente o almacenar los granos a-80 ° C.
- Trypsinize granos en 60 μL tampón 1 [urea de 2 M, 50 mM Tris-HCl (pH 7.5), 5 tripsina μg/mL]. Permitir que los granos digerir por 30 min a 27 ° C en un Termomezcladores sacudiendo a 800 RPM.
- Brevemente granos de centrífuga, luego recoger el sobrenadante y transferir a tubos de microcentrífuga (véase Tabla de materiales).
- Lavar los granos en 25 μl tampón 2 [urea de 2 M, 50 mM Tris-HCl (pH 7.5), 1 mM Ditiotreitol], con el fin de reducir las proteínas encuadernadas.
- Piscina los granos y el sobrenadante en tubo de uno microcentrífuga. Permita que la digestión ocurra durante la noche a temperatura ambiente.
- Alkylate las muestras añadiendo 20 μl de Yodoacetamida (5 mg/mL en agua ultrapura) a cada muestra e incubar en la oscuridad durante 30 minutos.
- Inhibir la digestión al tratar cada muestra con 1 ácido trifluoroacético μl (TFA). Este paso también será acidificar las muestras en preparación para la etapa de inflexión.
- Etapa de construcción C18 consejos por apilamientos 6 capas de extracción en fase sólida 1 mm C18 (octadecil) discos de membrana (véase Tabla de materiales) en una punta de micropipeta de 200 μl. Preparar una sola punta para cada muestra.
- Húmedas las puntas de la etapa con metanol y equilibrar con 50 μl 0,1% (v/v) de ácido trifluoroacético (TFA), 80% (v/v) de acetonitrilo.
- Lave las puntas con 50 μl 0.1% TFA (v/v).
- Los péptidos acidificados sobre las puntas de la etapa de carga y luego eluir utilizando 0,1% (v/v) TFA, 80% (v/v) de acetonitrilo en dos pasos.
- Evaporar las muestras utilizando un concentrador de vacío.
- Si es necesario, almacenar péptidos secadas a-80 ° C.
4. espectrometría de masas.
- Resuspender las muestras en 15 μl de ácido fórmico al 5%, 2% acetonitrilo (en agua ultrapura).
- Cuidadosamente la carga 6 μl en una placa de la LC y el lugar en un sistema HPLC nanoLC (véase Tabla de materiales).
- Paquete de una 20 cm, columna de diámetro interno de 75 μm con 1,9 μm C18 partículas de fase estacionaria (véase Tabla de materiales). Carga 5 péptido μL en cada columna.
- Eluir péptidos utilizando un gradiente lineal de acetonitrilo en nL/min 250 sobre 140 minutos e introducir por nanoelectrospray en un espectrómetro de masas cuadripolar de trampa lineal (LTQ) híbrido acoplado al sistema de HPLC nanoLC.
- Recoge tandem MS datos para los iones más abundantes del top 10 por escaneo de una pendiente tiempo de 140 minutos. Aleatorizar el orden de recolección de datos y el intercambio con la BSA entre cada muestra para minimizar el sesgo temporal.
5. Análisis
- Procesar datos MS utilizando por defecto en MaxQuant versión del software 1.2.7.4 y analizar MaxQuant de salida utiliza una versión modificada del flujo de trabajo de análisis estadístico de Perseo en el entorno de software de R3.
Nota: El flujo de trabajo de análisis estadístico de este punto se resume en la figura 2. Brevemente, intensidades LFQ de proteínas identificadas con MaxQuant son transformados y filtrados seguidos de la normalización y la imputación. Proteínas interacción de pares de dímero se identifican por comparación con un control de Venus, para excluir carpetas de fondo no específica, con prueba t de estudiante y Benjamini-Hochberg corrección para comparaciones múltiples. Calidad de los datos es confirmado por las comparaciones de los histogramas, regresión múltiple y clustering jerárquico.

Figura 2: esquema del flujo de trabajo de análisis estadístico. Diagrama de flujo de la tubería de análisis estadístico utilizado para analizar las intensidades LFQ de proteínas identificadas de datos de espectrometría de masas crudas procesados utilizando MaxQuant. Verde cajas: filtrado, cuadros azules: transformación/normalizar/escala, marrón cajas: cajas de datos control de calidad, amarillo: cajas de exclusión/comparativa semi-cuantitativo análisis, gris: análisis estadístico. Esta cifra es republicado fromCroucher et al.3 reimpreso con permiso de la AAAS. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
Tras el uso de la clonación de recombinación para generar de V1 y V2 con la etiqueta de genes de interés con el centro pDEST plásmidos, la transfección de dos plásmidos que contienen un par de interacción de proteínas resultará en la generación de una señal fluorescente de Venus después de aproximadamente de 8 a 24 horas. En la ausencia de una señal positiva es posible que la interacción de la proteína no puede ocurrir debido a la opción de línea celular, una eficacia de ...
Discusión
BiCAP es un método eficaz para aislar dímeros de la proteína específica mientras que excluyendo los componentes individuales y su competencia vinculante socios3. BiCAP se basa en la adaptación de un ensayo de complementación de proteínas de fluorescencia llamado Centro6. Los métodos existentes, incluyendo análisis de ligadura centro y proximidad, se han utilizado extensivamente para visualizar y cuantificar las interacciones de proteínas en células vivas
Divulgaciones
Los autores no tienen nada que revelar
Agradecimientos
D.R.C es un cáncer Instituto de NSW y D.N.S era previamente un cáncer Instituto de Nueva Gales del sur. Los resultados de la investigación presentados en este manuscrito fueron financiados por el cáncer Instituto de Nueva Gales del Sur (13/FRL/1-02 y 09/FCD/2-39), NHMRC (proyecto Grant GNT1052963), Fundación de Ciencias de España (11/GRIC/B2157), oficina de ciencia de Nueva Gales del sur e investigación médica de la familia de los Beca y Fundación de la familia Mostyn. J.F.H. y R.S. recibieron una concesión australiana del postgrado.
Materiales
| Name | Company | Catalog Number | Comments |
| LR Clonase II Plus enzyme | Thermo Fisher Scientific | 12538120 | Recombinase enzyme required for Gateway cloning (Step 1) into pDEST BiFC destination vectors |
| Proteinase K, recombinant, PCR grade | Thermo Fisher Scientific | EO0491 | |
| 14 mL round-bottomed polypropylene tube | Corning | 352059 | |
| Ampicillin | Roche Diagnostics Australia | 10835242001 | Stock solution prepared at 100 μg/mL in distilled water. |
| Miniprep kit | Promega Corporation | A1330 | |
| Maxiprep kit | Life Technologies Australia | K2100-07 | |
| DMEM | Gibco | 11995-073 | |
| FBS | Life Technologies Australia | 10099-141 | |
| Penicillin/Streptomycin | Life Technologies Australia | 15070-063 | |
| jetPRIME transfection buffer | Polyplus | 114-15 | |
| jetPRIME transfection reagent | Polyplus | 114-15 | |
| PhosSTOP (Phosphatase inhibitor) | Sigma-Aldrich | 4906837001 | |
| cOmplete, Mini, EDTA-free Protease inhibitor cocktail | Roche | 11873580001 | |
| Cell Scraper | Sarstedt | 83.1832 | |
| GFP-Trap_A | Chromotek Gmbh | gta-100 | GFP nanobody coupled to agarose beads |
| N-terminal GFP monoclonal antibody | Covance | MMS-118P | Will detect the V1 tag within the BiFC vectors |
| C-terminal GFP monoclonal antibody | Roche | 11814460001 | Will detect the V2 tag within the BiFC vectors |
| Sample buffer | Invitrogen | NP0008 | Supplemented with 1 mL β-mercaptoethanol. |
| Sequencing grade modified trypsin | Promega Corporation | V5117h | |
| LoBind microcentrifuge tubes | Point of Care Diagnostics | 0030 108 116 | |
| Iodoacetamide | Sigma-Aldrich | I1149-5G | Prepared at 5 mg/mL in ultrapure water |
| Trifluoroacetic Acid - Sequanal Grade | Thermo Fisher | 10628494 | |
| 3M Empore solid phase extraction C18 disks (octadecyl) - 4.7 cm | Thermo Fisher | 14-386-2 | To prepare stage tips, cut 1 mm disks using an appropriately sized hole punch. Alternatively, pre-prepared stage tips can also be purchased, see below. |
| C18 Stage Tips, 10 µL bed | Thermo Fisher | 87782 | |
| Formic acid OPTIMA for LC/MS grade 50mL | Thermo Fisher | FSBA117-50 | |
| 1.9 μm C18 ReproSil particles | Dr. Maisch GmbH | r119.aq. | Stationary phase particles |
| Acetonitrile OPTIMA LC/MS grade | Thermo Fisher | FSBA955-4 | |
| Easy-nLC HPLC | Thermo Fisher | ||
| LTQ Orbitrap Velos Pro | Thermo Fisher | ||
| Triton X-100 | Sigma-Aldrich | T8787 | Non-ionic detergent (100%) |
| DH5α cells | Thermo Fisher | Heat-shock-competent cells |
Referencias
- Kolch, W., Pitt, A. Functional proteomics to dissect tyrosine kinase signalling pathways in cancer. Nat Rev Cancer. 10 (9), 618-629 (2010).
- Pawson, T., Kofler, M. Kinome signaling through regulated protein-protein interactions in normal and cancer cells. Curr Opin Cell Biol. 21 (2), 147-153 (2009).
- Croucher, D. R., et al. Bimolecular complementation affinity purification (BiCAP) reveals dimer-specific protein interactions for ERBB2 dimers. Sci Signal. 9 (436), ra69 (2016).
- Cassonnet, P., et al. Benchmarking a luciferase complementation assay for detecting protein complexes. Nat Methods. 8 (12), 990-992 (2011).
- Rossi, F., Charlton, C. A., Blau, H. M. Monitoring protein-protein interactions in intact eukaryotic cells by beta-galactosidase complementation. Proc Natl Acad Sci U S A. 94 (16), 8405-8410 (1997).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism. J Am Chem Soc. 127 (1), 146-157 (2005).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21 (5), 539-545 (2003).
- Kubala, M. H., Kovtun, O., Alexandrov, K., Collins, B. M. Structural and thermodynamic analysis of the GFP:GFP-nanobody complex. Protein Sci. 19 (12), 2389-2401 (2010).
- Fegan, A., White, B., Carlson, J. C., Wagner, C. R. Chemically controlled protein assembly: techniques and applications. Chem Rev. 110 (6), 3315-3336 (2010).
- JoVE Science Education Database. Basic methods in cellular and molecular biology. plasmid purification. J Vis Exp. , (2017).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Shearer, R. F., et al. The E3 ubiquitin ligase UBR5 regulates centriolar satellite stability and primary cilia formation via ubiquitylation of CSPP-L. Mol Biol Cell. , (2018).
- Shannon, P., et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks. Genome Res. 13 (11), 2498-2504 (2003).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, 15690 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados