A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ויבתר חלבון רב מתחמי איתות על ידי טיהור זיקה קומפלמנטציה ריאקציה דו-מולקולרית (BiCAP)
In This Article
Summary
כתב יד זה מתאר את הפרוטוקול לטיהור ריאקציה דו-מולקולרית קומפלמנטציה זיקה (BiCAP). שיטה חדשנית זו מקלה על ספציפיות בידוד ואפיון פרוטיאומיה מבנית במורד הזרם של כל שני חלבונים אינטראקציה, בעוד למעט האו ם-ומורכבת חלבונים בודדים, כמו גם מתחמי הקים עם שותפים איגוד מתחרות.
Abstract
ההרכבה של מתחמי חלבון הוא מנגנון מרכזי שבבסיס ברגולציה של תאים רבים מסלולי איתות. והמוקד העיקרי של המחקר הביו-רפואי הוא בפענוח כיצד אלה מתחמי חלבון דינמי לפעול כדי לשלב את אותות ממקורות מרובים כדי לכוון תגובה ביולוגית ספציפית, איך זה הופך deregulated בהגדרות למחלות רבות. למרות חשיבותו של מנגנון הביוכימי מפתח זה, יש מחסור של טכניקות ניסיוני יכול להקל deconvolution בדיקה רגישה וספציפית של אלה מתחמי איתות רב מולקולרית.
כאן זה חסרון הוא ממוען באמצעות שילוב שיטת קומפלמנטציה עם nanobody קונפורמציה ספציפיים, אשר אנו יש להגדירו קומפלמנטציה ריאקציה דו-מולקולרית זיקה טיהור (BiCAP). טכניקה חדשנית זו מקלה על ספציפיות בידוד ואפיון פרוטיאומיה מבנית במורד הזרם של כל זוג חלבונים אינטראקציה, עד כדי של האו ם-ומורכבת חלבונים בודדים, מתחמי הקים עם שותפים איגוד מתחרות.
הטכניקה BiCAP לסגלה מגוון רחב של מבחני ניסיוני במורד הזרם, רמה גבוהה של ירידה לפרטים המוענקת על ידי טכניקה זו מאפשרת יותר הניואנסים חקירות המכניקה של חלבון הרכבה מורכב ממה שאפשרי כיום באמצעות טכניקות טיהור זיקה סטנדרטי.
Introduction
חלבונים מורכבים הרכבה הוא תהליך מפתח בשמירה על יחודיות ייתכן של רבים האיתות מסלולים1,2. מהות תפקיד רגולטורי זה קריטי ידועה ומוכרת, יש חוסר לרשות לבחון אלו מתחמי ניסיוני טכניקות. רוב המחקרים interactomics מתמקדים אינטראקציות עם חלבונים בודדים, או את העשרת רציפים של רכיבים מורכבים בודדים. כאן אנו מציגים טכניקה עבור בידודו של דיימר חלבון ספציפי בזמן למעט את moieties בודדים של רכיב חלבונים, כמו גם מתחמי הקים עם מתחרים האיגוד שותפים3. . קראנו טכניקה זו קומפלמנטציה ריאקציה דו-מולקולרית זיקה טיהור (BiCAP), כפי שהוא הוא שילוב של קיימים בעבר חלבון פרגמנט קומפלמנטציה assay, קרינה פלואורסצנטית ריאקציה דו-מולקולרית קומפלמנטציה (BiFC), עם השימוש הרומן nanobody רקומביננטי קונפורמציה ספציפי כלפי GFP ונגזרותיו (ראה טבלה של חומרים).
Assay קומפלמנטציה חלבון-קטע טיפוסי מסתמך על הביטוי של חלבונים "בית" ו- "טרף" התמזגו לפצל שברי כתבים כמו לוציפראז4, β-galactosidase5או חלבון פלואורסצנטי ירוק (GFP)6 ( איור 1A). דרך האינטראקציה של החלבונים פיתיון וטרף, התחומים כתב פיצול מומלץ לקפל לתוך מבנה פונקציונאלי, המאפשר את האינטראקציה של הפיתיון, טרף חלבונים דמיינו או לכמת. BiCAP הותאם בגרסה של טכניקה זו כי עשה שימוש שברי variant GFP ונוס. חלבון פלואורסצנטי קומפלמנטציה מבחני שיטה פופולארית להמחשת אינטראקציות חלבון בתא בשידור חי, אבל עד עכשיו היה מוגבל ל זו פונקציה אחת7. BiCAP מייצג התקדמות משמעותית בהקשר זה, כמו טכניקה זו מאפשרת לא רק עבור ויזואליזציה, אבל גם הבידוד והחקירות של אינטראקציה חלבון שנוצר.
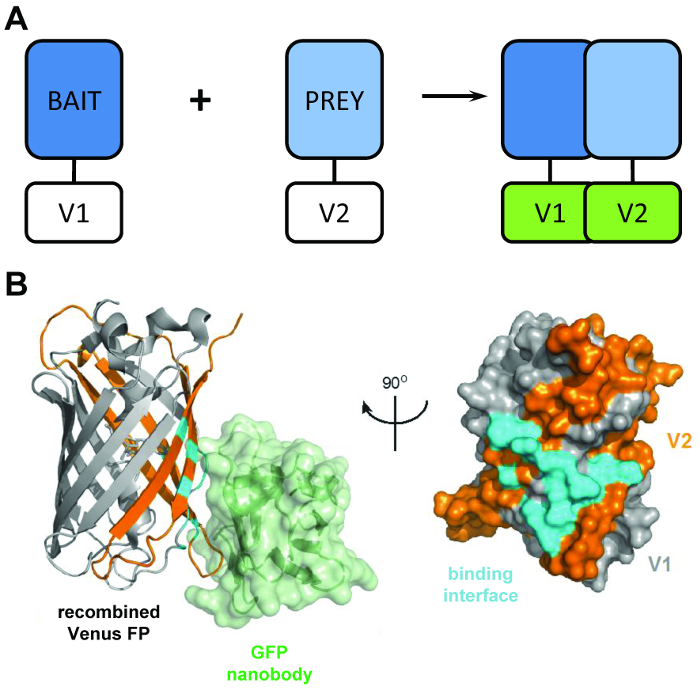
איור 1: המנהל מבניים מאחורי הטכניקה BiCAP. (א) סכימטי בהתוויית המנהלת מאחורי מראה קומפלמנטציה פלורסצנטיות ריאקציה דו-מולקולרית החלבונים "פיתיון" וגם של "טרף" מתויג עם N-מסוף V1 או C-מסוף V2 קטעים של החלבון ונוס באורך מלא. (B) ניתוח מבנה של הממשק אינטראקציה (ציאן) בין ה-GFP nanobody (ירוק) משולבות מחדש של ונוס, מציג את המיקום של V1 (אפור), שברי V2 (כתום) (PDB הצטרפותן 3OGO). איור זה הוא פרסום מחדש fromCroucher et al.3 Reprinted באישור AAAS. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
BiCAP טכניקת עושה שימוש שני קטעים שאינם-פלורסנט של ונוס (בשם V1 ו- V2) אשר מקשרים עם רמה נמוכה של זיקה אלא אם מתרחשת אינטראקציה בין בני הזוג שלהם פיוז'ן. במקרה זה, התחומים פיצול שני לקפל לתוך המבנה β-חבית התפקודי של fluorophore (איור 1B)6. החידוש מפתח של BiCAP נובע המבוא של nanobody GFP רקומביננטי, אשר מזהה epitope תלת מימדי על β-החבית של GFP (וגם משתנים כגון ונוס) אשר קיים רק fluorophore משולבות מחדש של כראוי, מקופל ( איור 1B)8. ויותר חשוב, nanobody ה-GFP אינו מאגד גם של השברים ונוס בודדים. זה מקל על בידודו של חלבון הדימרים רק לאחר שני חלבונים נוצר קומפלקס מרצונם, מובילים לתוצאות נציג יותר מאשר אלה שנרכשה מן השיטות שעושים שימוש של אינטראקציות בהשפעת כימיקלים, כפו9.
BiCAP היא טכניקה חזקה במיוחד המתמקד חלבון מרובת קומפלקסים, אשר באופן פוטנציאלי יכול להיות משולב עם מספר יישומים במורד הזרם כדי לשפר את צפיפות הרשת של הבנתנו תפקיד שאלה מתחמי לשחק אותות . זה גם כוללת את התכונה החשובה של המאפשר ויזואליזציה של אינטראקציות חלבון בתוך באתרו. עד היום הוכח BiCAP כאל שיטה יעילה של מנתח את interactome של קולטן טירוזין קינאז (RTK) הדימרים3, אבל יכולת ההתאמה של שיטה זו אומר כי זה יכול להיות מאומץ כמעט בכל הקשר אינטראקציית חלבון.
Protocol
1. פלסמיד שיבוט
הערה: כדי ליצור וקטורים פלסמיד עם התגים V1 או V2 התמזגו אמיני או קצה קרבוקסילי של הגן עניין, BiFC היעד וקטורים נצברים עם Addgene [N-מסוף תג: pDEST-V1-ORF (#73635), pDEST-V2-ORF (#73636). C-מסוף תג: pDEST-ORF-V1 (#73637), pDEST-ORF-V2 (#73638)]. הגן/s עניין תצטרך להיות ספציפי רקומבינציה שיבוט תואם ערך וקטורים (קרי, pDONR223 או pENTR221), ללא עצירה codons, להמשיך עם שיבוט. שיבוטים תואמים רבים זמינים כבר בתוך אוספים פלסמיד שונים, כולל האוסף הגנום יונקים (https://genecollections.nci.nih.gov/MGC/).
- לבצע רקומבינציה תגובה כדי להוסיף את הגן עניין בין האתרים הפו של וקטור היעד המתאים BiFC:
- בשפופרת 0.2 מ"ל, להוסיף 150 ng כניסה וקטור 150 ng BiFC היעד וקטור, µL 8 טה מאגר (pH 8.0).
- להוסיף תערובת של האנזים recombinase µL 2 (ראה טבלה של חומרים). מערבבים היטב, צנטריפוגה בקצרה.
- דגירה התגובה לשעה בטמפרטורת החדר או ב 16 מעלות צלזיוס למשך הלילה.
- לעצור את התגובה על-ידי הוספת 1 µL Proteinase-K פתרון המקננת ב 37 מעלות צלזיוס למשך 10 דקות.
- להפוך תאים חום-הלם-מוסמך (ראה טבלה של חומרים) עם המוצר התגובה LR:
הערה: כל זן בסיסי שכעיקרון ניתן להשתמש בנקודה זו, כל עוד הוא אינו מכיל טוקסין Ccdb, שימנעו את הבחירה ספציפי של פלסמידים המכיל את תותב.- תאים המוסמכת על הקרח להפשיר ולהעביר 50 µL של תאים לתוך 14 מ"ל להטלטל סיבוב פוליפרופילן צינור.
- להוסיף 1 µL של המוצר תגובת התאים ומערבבים בעדינות. תקופת דגירה של 20 דקות על קרח.
- מחממים שוק באמבטיה מים 42 מעלות צלזיוס במשך 45 ס לחזור מיד קרח למשך 2 דקות.
- להוסיף 1 מ"ל של מדיה ליברות, דגירה ברעידות-37 מעלות לשעה.
- צלחת הטרנספורמציה על צלחת אגר 10 ס מ המכיל אמפיצילין (100 µg/mL) דגירה ב 37 מעלות צלזיוס למשך הלילה.
- טיהור של פלסמיד BiFC.
- לטהר פלסמיד מהמושבה הפרט, להוסיף 100 מ של מדיה LB בקבוקון חרוט גדול ולהוסיף אמפיצילין (100 µg/mL). למסך מושבות מרובות, להוסיף 5 מ ל LB מדיה להטלטל לסיבוב צינורות פוליפרופילן 14 מ"ל ולהוסיף אמפיצילין (100 µg/mL).
- באמצעות לולאה חיסון סטרילי, לבחור מושבה בודדת מהצלחת אגר. למקם את הלולאה חיסון התקשורת LB ומערבבים בקצרה.
- מכסה העליון של הבקבוק חרוט עם רדיד אלומיניום, דגירה בין לילה ב 37 מעלות צלזיוס ברעידות.
- לייצר תרביות תאים כיתה פלסמיד הדנ א באמצעות על תקן maxiprep או midiprep פלסמיד DNA טיהור קיט (ראה טבלה של חומרים)10. להעריך את האיכות של ה-DNA על ידי ספיגת ספקטרה.
הערה: ה-DNA צריכה לקבל יחס A260/A280 > 1.8 ויחס A260/A230 > 2.0. בשלב זה, מומלץ למסך מספר מושבות בודדות כדי לבדוק ביטוי יעיל של הוספה, תג.
2. התא תרבות, תרביות תאים
הערה: על תקנים של הווקטורים BiFC חשוב להשיג יעילות גבוהה, תרביות תאים יחסית הומוגנית. הווקטורים צפויה להיות תואם עם כל ריאגנט תקנים סטנדרטיים, התנאים תרביות תאים צריך להיות מותאם בהתאם. כדי לבצע ספקטרומטר מסה, אנחנו בדרך כלל התרבות תאים בתוך מאכלים 10 ס מ, למרות שזה יכול גם באופן פרופורציונלי מדע פשוט, מנות קטנות יותר או צלחות לניסויים הדורשים פחות חומר.
- תאים6 HEK293T זרע 1.0 × 10 10 ס מ מגישים עם 10 מ"ל של מדיה DMEM, בתוספת 10% FBS ו פניצילין/סטרפטומיצין (1: 100).
- למהול 2.5 µg של כל וקטור BiFC לתוך µL 500 תקנים מאגר (ראה טבלה של חומרים) צינור microcentrifuge 1.5 mL.
- להוסיף 10 µL של תרביות תאים ריאגנט (ראה טבלה של חומרים).
- תערובת מערבולת 10 s, אז בקצרה צנטריפוגה. תקופת דגירה של 10 דקות בטמפרטורת החדר.
- להוסיף את התערובת תקנים של ה-DNA לצלחת, dropwise. דגירה את התאים עבור אורך מספיק זמן עבור החלבונים פיוז'ן BiFC אינטראקציה, ונוס קיפול והודעות fluorophore ההבשלה, בדרך כלל ~ 8-24 שעות.
הערה: עירור שיא של ונוס הוא 515 ננומטר, פליטה השיא שלה הוא 528 nm, למרות שזה נראה בקלות באמצעות מיקרוסקופ פלואורסצנטי רגיל להגדיר כדי להמחיש פלורסצנטיות GFP.
3. הכנת הדוגמא
-
Lysates קציר
- לפני lysate אוסף, להכין מאגר פירוק התא [50 מ"מ טריס-HCl (pH 7.4), 150 מ מ NaCl, 1 מ"מ EDTA, דטרגנט ללא יונית 1% (v/v) (ראה טבלה של חומרים)]. חנות ב 4 º C.
- מייד לפני הקטיף, תוספת 10 מ"ל של מאגר פירוק התא עם מעכב פרוטאז והמעכב פוספטאז (ראה טבלה של חומרים). לשמור על מאגר פירוק התא שהושלם על קרח.
- לשטוף פעמיים בתאים PBS קר כקרח. האחות של PBS ולהוסיף 1 מ"ל כקרח מאגר פירוק התא שהושלם. מניחים את המנה על קרח, המבטיח שהמאגר מורחים את פני השטח כולו.
- דגירה על קרח במשך כ 5 דקות, ואז להשתמש במגרדת תא (ראה טבלה של חומרים) לגרד את התאים ולהעביר ל צינור microcentrifuge mL 1.5 מראש צוננת.
- הסרת לכלוך התאית על ידי צריך שתוציאו את שנאספו lysate-18,000 g × עבור 5 דקות ב 4 ° C והעברת מסומנת תגובת שיקוע לתוך צינורות microcentrifuge טריים.
הערה: בשלב זה lysate שניתן להשתמש מיד או המאוחסנים ב- 80 ° c מומלץ גם כדי לשמור על aliquot של גולמי lysate כך היעילות של תרביות תאים, הטיהור זיקה הניתנים לאימות.
-
זיקה טיהור
הערה: השלב בידוד BiCAP מתבצעת באמצעות את nanobody GFP מצומדת כדי חרוזים agarose (ראה טבלה של חומרים).- הכן את החרוזים agarose על ידי נטילת אמצעי אחסון המתאים (20 µL עבור דגימה) + 10 µL עודף ב 1 מ"ל PBS. Centrifuge את החרוזים ב 300 x g ולהסיר את תגובת שיקוע.
- הוסף 20 µL כל מדגם lysate.
- דגירה בדגימות עבור 2 h ב 4 ° C עם סיבוב end-to-end.
הערה: בשלב זה הדגימות ניתן להכין עבור מרחביות-דף, סופג המערבי, או ניתוח באמצעות ספקטרומטר מסה.
-
הכנת BiCAP eluant עבור סופג המערבי.
- Centrifuge את החרוזים 300 x g ושטיפת שלוש פעמים במאגר תא פירוק.
- Resuspend את החרוזים שטף ב- 50 µL מאגר מדגם כראוי מדולל (ראה טבלה של חומרים) וחום הדגימות ב 95 מעלות צלזיוס למשך 2-3 דקות.
הערה: דגימות שהוכנו בצורה זו ניתן לאחסן ב-20 מעלות צלזיוס למשך מספר חודשים. - לבצע מרחביות-דף ומערביות סופג11 הן עבור תג V1 V2 תגיות (ראה טבלה של חומרים), כמו גם כל שאר החלבונים עניין.
-
הכנה של BiCAP eluant ספקטרומטר מסה.
הערה: לצורך ניתוח באמצעות ספקטרומטר מסה (LFQ) ללא תווית כמותיים מומלץ להכין את הדגימות לפחות quadruplicate כדי להבטיח עוצמה סטטיסטית חזקים.- Centrifuge את החרוזים 300 x g ושטיפת שש פעמים במאגר תא פירוק ללא חומרי ניקוי. הדבר נחוץ להסיר את עודף חלבון והן גם את חומרי ניקוי אשר יפריעו ספקטרומטר מסה ניתוח. המשך לשלב הבא מיד, או לאחסן את החרוזים ב-80 מעלות צלזיוס.
- Trypsinize חרוזי 60 µL 1 מאגר [2 מ' אוריאה, 50 מ מ טריס-HCl (pH 7.5), טריפסין µg/mL 5]. לאפשר את החרוזים לעכל למשך 30 דקות ב 27 ° C ב- thermomixer רועדת במהירות של 800 סל ד.
- בקצרה צנטריפוגה חרוזים, ולאחר מכן לאסוף תגובת שיקוע, העברת צינורות microcentrifuge (ראה טבלה של חומרים).
- לשטוף את החרוזים ב 25 µL מאגר 2 [2 מ' אוריאה, 50 מ מ טריס-HCl (pH 7.5), 1 מ dithiothreitol], על מנת להפחית את החלבונים מאוגד.
- בריכת חרוזים של תגובת שיקוע לתוך צינור אחד microcentrifuge. לאפשר עיכול להתרחש בין לילה בטמפרטורת החדר.
- Alkylate את הדוגמאות על-ידי הוספת 20 µL של iodoacetamide (5 מ"ג/מ"ל מים הנדסה גנטית) כל דגימה, דגירה בחושך למשך 30 דקות.
- לעכב את תהליך העיכול על ידי טיפול כל דגימה עם µL 1 trifluoroacetic חומצה (TFA). שלב זה גם acidify את הדגימות לקראת שלב המפנה.
- לבנות סי18 הבמה טיפים על ידי הערמה 6 שכבות של 1 מ מ מיצוי מעבדתי סי18 (Octadecyl) דיסקים ממברנה (ראה טבלה של חומרים) לתוך טיפ micropipette 200 µL. להכין טיפ יחיד עבור כל דגימה.
- להרטיב את הטיפים הבמה עם מתנול, equilibrate עם 50 µL 0.1% (v/v) trifluoroacetic חומצה (TFA), acetonitrile 80% (v/v).
- לשטוף את הטיפים עם 50 µL 0.1% (v/v) TFA.
- לטעון את פפטידים acidified אל העצות הבמה, ואז elute באמצעות 0.1% (v/v) TFA, acetonitrile 80% (v/v) בשני שלבים.
- להתנדף הדגימות באמצעות רכז ואקום.
- אם יש צורך, לאחסן פפטידים יבשים ב-80 מעלות צלזיוס.
4. ספקטרומטריית.
- Resuspend דוגמאות ב 15 µL 5% חומצה פורמית, 2% acetonitrile (במים הנדסה גנטית).
- בזהירות לטעון µL 6 על גבי צלחת LC ומקום לתוך מערכת HPLC nanoLC (ראה טבלה של חומרים).
- חבילת a 20 ס מ, 75 מיקרומטר עמודה הקוטר הפנימי עם 1.9 מיקרומטר סי18 שלב נייח חלקיקים (ראה טבלה של חומרים). לטעון פפטיד 5 µL על גבי כל עמודה.
- Elute פפטידים באמצעות מעבר צבע ליניארי של acetonitrile ב nL 250/דקה מעל 140 דקות, להציג על-ידי nanoelectrospray לתוך Quadropole מלכודת ליניארי (LTQ) היברידית ספקטרומטר מסה בשילוב מערכת HPLC nanoLC.
- לאסוף נתונים MS טנדם עבור היונים הנפוץ ביותר top 10 לכל סריקה מעבר הדרגתי זמן של 140 דקות. אקראי הסדר של איסוף נתונים, מחלף עם BSA בין כל דגימה כדי למזער את הטיית טמפורלית.
5. ניתוח
- לעבד נתונים MS raw באמצעות הגדרת ברירת המחדל בתוך MaxQuant גרסת תוכנה 1.2.7.4 ולנתח את הפלט באמצעות הגירסה של פרסאוס זרימת העבודה ניתוח סטטיסטי של סביבת תוכנה R3MaxQuant.
הערה: ניתוח סטטיסטי זרימת העבודה מנקודה זו מסוכם באיור2. בקצרה, LFQ עוצמות של חלבונים מזוהה באמצעות MaxQuant טרנספורמציה, מסוננים שהופעלו על-ידי נרמול מעומק. חלבונים אינטראקציה של זוגות דיימר מזוהים by comparison with פקד ונוס, כדי לא לכלול רקע ספציפי קלסרים, עם מבחן t של סטודנט, בנימיני-הוכברג תיקון להשוואות מרובות. איכות הנתונים אושר על ידי השוואות היסטוגרמות נפרדות, רגרסיה מרובים ו clustering הירארכי.

איור 2: חלוקה של זרימת העבודה ניתוח סטטיסטי. תרשים זרימה של צינור ניתוח סטטיסטי המשמש כדי לנתח את עוצמות LFQ של חלבונים מזוהה מנתונים גולמיים ספקטרומטר מסה מעובד באמצעות MaxQuant. ירוק תיבות: סינון, הקופסאות הכחולות: תיבות טרנספורמציה/נרמול/שינוי קנה מידה, חום: תיבות של בקרת איכות, צהוב נתונים: הדרה חצי כמותית/השוואתי ניתוח, אפור תיבות: ניתוח סטטיסטי. איור זה הוא פרסום מחדש fromCroucher et al.3 Reprinted באישור AAAS. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
תוצאות
בעקבות השימוש רקומבינציה שכפול ליצירת של V1 ו- V2 מתויג הגנים של עניין עם פלסמידים pDEST BiFC, תרביות תאים שותף של פלסמידים 2 המכיל זוג אינטראקציה של חלבונים יביא הדור של אות ניאון ונוס לאחר כ- 8-24 שעות. בהיעדר סימן חיובי שזה אפשרי כי לא שמתבצעים האינטראקציה חלבון בשל בחירת תאים בט...
Discussion
BiCAP היא שיטה חזקה עבור בידוד חלבון ספציפי הדימרים שיתופם של רכיבים בודדים ו שלהם שותפים מתחרות מחייב3. BiCAP מבוסס על עיבוד של קרינה פלואורסצנטית assay קומפלמנטציה חלבון הנקרא BiFC6. שיטות קיימות, כולל מבחני מצדו BiFC וקירבה, נעשה שימוש נרחב כדי להמחיש ולכמת אינטראקציות חל?...
Disclosures
המחברים אין לחשוף
Acknowledgements
D.R.C הוא בחור NSW מכון הסרטן, D.N.S היה בעבר בחור NSW מכון הסרטן. ממצאי המחקר הציג כתב יד זה במימון NSW מכון הסרטן (13/FRL/1-02 ו- 09/CDF/2-39), NHMRC (פרוייקט גרנט GNT1052963), קרן מדעי לאירלנד (11/SIRG/B2157), NSW Office של מדע ומחקר רפואי, חוות המשפחה אחווה, קרן משפחת המרגיעה. J.F.H. R.S. היו מקבלי פרס לתארים מתקדמים אוסטרלי.
Materials
| Name | Company | Catalog Number | Comments |
| LR Clonase II Plus enzyme | Thermo Fisher Scientific | 12538120 | Recombinase enzyme required for Gateway cloning (Step 1) into pDEST BiFC destination vectors |
| Proteinase K, recombinant, PCR grade | Thermo Fisher Scientific | EO0491 | |
| 14 mL round-bottomed polypropylene tube | Corning | 352059 | |
| Ampicillin | Roche Diagnostics Australia | 10835242001 | Stock solution prepared at 100 μg/mL in distilled water. |
| Miniprep kit | Promega Corporation | A1330 | |
| Maxiprep kit | Life Technologies Australia | K2100-07 | |
| DMEM | Gibco | 11995-073 | |
| FBS | Life Technologies Australia | 10099-141 | |
| Penicillin/Streptomycin | Life Technologies Australia | 15070-063 | |
| jetPRIME transfection buffer | Polyplus | 114-15 | |
| jetPRIME transfection reagent | Polyplus | 114-15 | |
| PhosSTOP (Phosphatase inhibitor) | Sigma-Aldrich | 4906837001 | |
| cOmplete, Mini, EDTA-free Protease inhibitor cocktail | Roche | 11873580001 | |
| Cell Scraper | Sarstedt | 83.1832 | |
| GFP-Trap_A | Chromotek Gmbh | gta-100 | GFP nanobody coupled to agarose beads |
| N-terminal GFP monoclonal antibody | Covance | MMS-118P | Will detect the V1 tag within the BiFC vectors |
| C-terminal GFP monoclonal antibody | Roche | 11814460001 | Will detect the V2 tag within the BiFC vectors |
| Sample buffer | Invitrogen | NP0008 | Supplemented with 1 mL β-mercaptoethanol. |
| Sequencing grade modified trypsin | Promega Corporation | V5117h | |
| LoBind microcentrifuge tubes | Point of Care Diagnostics | 0030 108 116 | |
| Iodoacetamide | Sigma-Aldrich | I1149-5G | Prepared at 5 mg/mL in ultrapure water |
| Trifluoroacetic Acid - Sequanal Grade | Thermo Fisher | 10628494 | |
| 3M Empore solid phase extraction C18 disks (octadecyl) - 4.7 cm | Thermo Fisher | 14-386-2 | To prepare stage tips, cut 1 mm disks using an appropriately sized hole punch. Alternatively, pre-prepared stage tips can also be purchased, see below. |
| C18 Stage Tips, 10 µL bed | Thermo Fisher | 87782 | |
| Formic acid OPTIMA for LC/MS grade 50mL | Thermo Fisher | FSBA117-50 | |
| 1.9 μm C18 ReproSil particles | Dr. Maisch GmbH | r119.aq. | Stationary phase particles |
| Acetonitrile OPTIMA LC/MS grade | Thermo Fisher | FSBA955-4 | |
| Easy-nLC HPLC | Thermo Fisher | ||
| LTQ Orbitrap Velos Pro | Thermo Fisher | ||
| Triton X-100 | Sigma-Aldrich | T8787 | Non-ionic detergent (100%) |
| DH5α cells | Thermo Fisher | Heat-shock-competent cells |
References
- Kolch, W., Pitt, A. Functional proteomics to dissect tyrosine kinase signalling pathways in cancer. Nat Rev Cancer. 10 (9), 618-629 (2010).
- Pawson, T., Kofler, M. Kinome signaling through regulated protein-protein interactions in normal and cancer cells. Curr Opin Cell Biol. 21 (2), 147-153 (2009).
- Croucher, D. R., et al. Bimolecular complementation affinity purification (BiCAP) reveals dimer-specific protein interactions for ERBB2 dimers. Sci Signal. 9 (436), ra69 (2016).
- Cassonnet, P., et al. Benchmarking a luciferase complementation assay for detecting protein complexes. Nat Methods. 8 (12), 990-992 (2011).
- Rossi, F., Charlton, C. A., Blau, H. M. Monitoring protein-protein interactions in intact eukaryotic cells by beta-galactosidase complementation. Proc Natl Acad Sci U S A. 94 (16), 8405-8410 (1997).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism. J Am Chem Soc. 127 (1), 146-157 (2005).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21 (5), 539-545 (2003).
- Kubala, M. H., Kovtun, O., Alexandrov, K., Collins, B. M. Structural and thermodynamic analysis of the GFP:GFP-nanobody complex. Protein Sci. 19 (12), 2389-2401 (2010).
- Fegan, A., White, B., Carlson, J. C., Wagner, C. R. Chemically controlled protein assembly: techniques and applications. Chem Rev. 110 (6), 3315-3336 (2010).
- JoVE Science Education Database. Basic methods in cellular and molecular biology. plasmid purification. J Vis Exp. , (2017).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Shearer, R. F., et al. The E3 ubiquitin ligase UBR5 regulates centriolar satellite stability and primary cilia formation via ubiquitylation of CSPP-L. Mol Biol Cell. , (2018).
- Shannon, P., et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks. Genome Res. 13 (11), 2498-2504 (2003).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, 15690 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved