需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
用于筛选小分子抑制剂的NADH耦合ATPase分析的半高通量自适应
摘要
一种烟酰胺腺苷二核苷酸(NADH)与ATPase的检测已应用于小分子肌苷抑制剂的半高通量筛选。此动力学测定以 384 孔微孔板格式运行,总反应体积仅为每孔 20 μL。该平台应适用于几乎任何ADP生产酶。
摘要
ATPase酶利用三磷酸腺苷中储存的游利用能量,催化体内各种不会自发发生的内发性生化过程。这些蛋白质对细胞生活的各个方面都至关重要,包括新陈代谢、细胞分裂、对环境变化的反应和运动。这里提出的协议描述了一种烟酰胺腺苷二核苷酸(NADH)耦合ATPase测定,该检测已适用于小分子ATPase抑制剂的半高通量筛选。该测定已应用于心脏和骨骼肌肌苷II,两个基于行为蛋白的分子运动ATPases,作为原理证明。ATP的水解通过测定中的酶反应与NADH的氧化结合。首先,由 ATPase 生成的 ADP 通过丙酮激酶 (PK) 再生为 ATP。PK催化磷酸二苯丙酸酯(PEP)与丙酮类酯的平行过渡。随后,乳酸通过乳酸脱氢酶(LDH)降低为乳酸盐,同时催化NADH的氧化。因此,ATP浓度的下降与NADH浓度的下降直接相关,随后是NADH内在荧光的变化。只要在反应系统中有PEP,ADP浓度仍然很低,避免由其自己的产品抑制ATPase酶。此外,ATP 浓度几乎保持不变,产生线性时间课程。荧光被持续监测,从而便于估计数据的质量,并有助于过滤出潜在的伪影(例如,由复合沉淀或热变化引起的)。
引言
肌苷是水解三磷酸腺苷(ATP)的机械化学能量传感器,用于在真核细胞1、2中沿活性素细胞骨架的丝状物产生定向运动。它们在结构和动力学上都适应了细胞内的各种功能,如细胞器的传输、肌肉收缩或细胞骨骼张力的产生1,2。肌苷超级家族由属于人类基因组3、4中12个不同肌苷类的+40肌苷基因表示。肌苷类的成员在一系列高度多样化的疾病中扮演着不同的角色,如几种癌症、神经系统疾病、骨骼肌病和肥大性心肌病5、6。鉴于这些分子马达的生理和病理功能很多,它们越来越被公认为各种条件的药物靶点,这并不奇怪。最近,在发现新的肌苷抑制剂8、9、10和活化剂11方面取得了重大进展,并改善了现有抑制剂12的特性。13,14,15.
烟酰胺腺苷二核苷酸(NADH)耦合ATPase测定早已用于测量各种酶的ATPase活性,如肉质性视网膜Ca2+泵ATPase 16,DNA修复ATPase Rad5417,AAA+ATPase p9718或微管马达运动体19。测定采用 ATP 再生循环。ATPase产生的二磷酸腺苷(ADP)通过丙酮酸激酶(PK)再生为ATP,将一个磷酸二醇酸(PEP)分子并联转化为丙酮酸盐。随后,通过乳酸脱氢酶(LDH)将丙酮酸酯降低为乳酸盐。这反过来又氧化了NADH的一个分子到NAD。因此,NADH浓度作为时间的函数的降低等于ATP水解率。只要 PEP 可用,ATP 再生循环就使 ATP 浓度几乎保持不变,ADP 浓度处于低水平。这导致线性时间课程,使得确定初始反应速率变得简单,并有助于避免ADP19对产品抑制。虽然NADH耦合ATPase测定已经适应了96井格式20,但高反应量(±150 μL)由于试剂需求较高,使得它不太适合快速筛选大量化合物。替代方法,如麦芽绿测定19,21,它依赖于检测由ATPase酶产生的磷酸盐,被证明更适合小型化和高通量筛选22,23,24.然而,端点测定更有可能受到几个工件的影响(下文讨论),如果没有全日制课程,这些伪像可能仍未被发现。
在这里,NADH耦合ATPase测定已针对小分子抑制剂的半高通量筛选进行了优化。骨骼和心肌肌苷II和肌苷抑制剂blebbisatin8,副阿米诺巴他丁13和准硝基布利他妥丁12用于证明测定的力量,它依赖于NADH荧光作为读出。该协议适用于筛选专注于任何ADP生产酶的项目。
研究方案
1. 制备库存溶液和试剂
- 通过在蒸馏水中溶解结晶DTT至最终浓度为1000 mM,制备二硫二硫醇(DTT)库存溶液。使用 1 M NaOH 溶液将 pHH 调整为 7.0。在-20°C下,以-20°C的分量和储存。
- 通过将蒸馏水中的结晶ATP溶解至100 mM的最终浓度,制备ATP库存溶液。使用 1 M NaOH 溶液将 pHH 调整为 7.0。在-20°C下,以-20°C的分量和储存。
- 制备含有70mM 3-(N-变形)丙烷酸(MOPS)、10mM MgCl 2、0.9mM乙二醇-乙二醇(β-氨基乙醚)-N、N、N、N-四乙酸(EGTA)和3mMNaN3的10xNADH缓冲液。使用 1 M NaOH 溶液将 pHH 调整为 7.0。储存在4°C。
- 制备含有10 mM MOPS和0.1 mM EGTA的1x肌苷缓冲液。使用 1 M NaOH 溶液将 pHH 调整为 7.0。储存在4°C。将牛血清白蛋白 (BSA) 和 DTT 添加到 0.1% 的最终浓度(w/v%)和 1 mM,分别在使用前。
- 准备含有4 mM MOPS、0.1 mM EGTA、2 mM MgCl2和3 mM NaN3的1x肌素缓冲液。使用 1 M NaOH 溶液将 pHH 调整为 7.0。储存在4°C。将 BSA 和 DTT 添加到 0.1% 的最终浓度(包括/v%)和 1 mM,分别在使用前。
- 通过在 10x NADH 缓冲液中溶解结晶 NADH 到最终浓度为 5.5 mM,制备 NADH 库存溶液。在-20°C下,以-20°C的分量和储存。
- 通过将 10x NADH 缓冲液中的结晶 PEP 溶解到 50 mM 的最终浓度,制备 PEP 库存溶液。在-20°C下,以-20°C的分量和储存。
- 通过在甘油和10x NADH缓冲液混合物中溶解冻干LDH粉末制备LDH库存溶液(50%:50%)到最终浓度为2000 U/mL。离心溶液,以去除任何未溶解的蛋白质存在(7,197 x g, 20 °C, 10 分钟)。小心地将上清液转移到干净的离心管中。在-20°C下,以-20°C的分量和储存。
- 通过在甘油和10x NADH缓冲液混合物中溶解冻干PK粉末制备PK库存溶液(50%:50%)最终浓度为10000 U/mL。离心溶液,以去除任何未溶解的蛋白质存在(7,197 x g, 20 °C, 10 分钟)。小心地将上清液转移到干净的离心管中。在-20°C下,以-20°C的分量和储存。
- 通过加入100μL蒸馏水,重新形成冻干心脏和骨骼肌肌肌酸II样品,以获得10mg/mL库存溶液,分别对应于±37.9 μM和+40.8 μM肌苷浓度(单体)。有关详细信息,请参阅制造商的说明。
- 从兔子肌肉丙酮粉制备F-actin,如帕迪和斯普迪奇25所述。
2. 测量小分子抑制剂的ATPase活性和抑制作用
- 准备复合板。
- 溶解优质二甲基硫酸盐(DMSO)中感兴趣的化合物。
- 从 DMSO 中的 10 mM 化合物浓度开始创建 15 步串行 1:2 稀释。
- 使用多通道移液器将样品转移到三联体(每块12.5 μL)的384孔聚丙烯板中。在复合板上对一个化合物(而不是三列)使用两行,以尽量减少可能受边缘效应影响的井数。将每个化合物的第二行的最后三口作为负控制(仅限 DMSO)。请勿将板上的第一行和最后一行用于复合稀释。
- 将纯 DMSO 传输到第一行的井中(保留用于 NADH 校准)。
- 使用最后一行进行正控制。
注:此处使用了DMSO中浓度为4 mM的副阿米诺比沙丁。
- 通过稀释Actin缓冲液中的Actin股票溶液,为每个测定板(384孔黑壁聚苯乙烯微板)制备4500μL的20μM稀释剂溶液。使用 5 mL 移液器上下移液 30x,彻底混合溶液,通过打破活性素细丝来降低粘度和异质性。离心溶液,以去除任何沉淀的蛋白质存在(7,197 x g,20 °C, 10 分钟)。小心地将上清液转移到干净的离心管中。
- 准备含有LDH和PK酶("酶混合")的主混合物。对于每个测定板,将171.4μL的LDH溶液、171.4μL的PK溶液和3189.3μL或3252.9 μL肌苷缓冲液分别结合在15 mL锥形离心管中,用于涉及心脏或骨骼肌肌黄蛋白II的测定。此时不要添加任何肌苷,以避免聚集和降水。
- 准备包含所有基板的主混合物("基板混合")。对于每个板,将 ATP 的 162.1 μL、162.1 μL 的 PEP 和 324.1 μL 的 NADH 溶液组合在 15 mL 锥形离心管中。此时不要添加 actin 以避免聚集和沉淀。
- 创建七步串行 1:2 稀释 NADH,以便从 250 μM 开始校准。
- 将12.3 μL的NADH库存溶液与257.7μL肌苷缓冲液混合在1.5 mL微离心管中。
- 亚细亚135 μL肌苷缓冲液放入7个1.5 mL微离心管中。
- 将135 μL溶液从第一管转移到第二管中,并通过移液混合。重复,直到达到第7管。
- 使用最后一根管作为无 NADH 控制(仅限缓冲器)。
- 使用 8 通道移液器,将 20 μL 的 NADH 校准解决方案以三联成片将 20 μL 传输到化片的第一行。
- 在酶混合物中加入68 μL的心脏或4.2 μL的骨骼肌肌肌苷II。漩涡简要。
- 除第一行外,使用自动分配器将制备的肌苷酶混合物的8.4 μL分给测定盘的每个孔。
- 使用配备 100 nL 销工具头的自动液体处理系统,将 100 nL 溶液从复合板转移到含有酶混合物的测定板。
- 使用微板摇床在室温1200rpm下摇动测定板1分钟。
- 在基材混合物中加入4,052 μL的离心反应液溶液。漩涡简要。
- 将 11.6 μL 的 actin-基质混合物放入测定板(第一行除外)的每个孔中,使用自动分配器启动酶反应。
- 使用微板摇床在室温1200rpm下摇动测定板1分钟。
- 将测定板在 101 x g下离心 30 s。
- 确保板式读卡器的内部温度稳定在 25°C。装入板并摇动 30 s。此摇动步骤是必要的,使液体表面的形状在每个孔中相似,并留出时间使板达到测量温度。
- 以 45 秒的间隔记录 NADH 荧光 30 分钟扫描板。使用 380 nm、10 nm 带宽激励滤波器和 470 nm 24 nm 带宽发射滤波器,以及 425 nm 截止双色镜。在高浓度模式下运行测量。在运行检测之前,优化闪烁次数、探测器增益、板尺寸和测量高度。
注: 最终测定条件为 300 nM 心脏/20 nM 骨骼肌肌肌苷 II,10 μM 肌素,40 U/mL LDH,200 U/mL PK,220 μM NADH,1 mM PEP,1 mM ATP 在缓冲液中包含 10 mM MOPS (pH = 7.0),2 mM MgCl2,0.15 mM EGTA,0.1 毫克/毫克 BSA,0.5% (v/v) DMSO 和 1 mM DTT。总体积为 20 μL/井。最高最终化合物浓度为50μM.20μM对αnoblebisatin,在0.5%DMSO中充当正对照,仅0.5%DMSO是负对照。 所有测量均以三次进行。
3. 分析数据
- 根据每口井的时间绘制观察到的荧光强度。
- 执行简单的线性回归,以确定每个井的荧光响应的斜率和截距。斜率与 ATP (NADH) 消耗率成正比,而截距与测量开始时的 NADH 浓度成正比(t = 0 s)。
- 通过绘制板块第一行获得的截距与 NADH 浓度的截距,为 NADH 构建校准曲线。确保截距线性取决于 NADH 浓度。
注: 截距估计 t = 0 s 处的实际荧光强度,其可信度远高于 t = 0 s 的原始荧光强度读数的平均值。 - 执行简单的线性回归以获得 NADH 校准线的斜率和截距。
注: 截距描述荧光背景信号(不存在 NADH),而斜率对应于该特定实验中 1 M NADH 溶液的外推/理论荧光强度。 - 将其余油井获得的荧光响应斜率除以 NADH 校准线的斜率,以将荧光变化转换为 ATP 消耗率。
- 根据抑制剂的浓度绘制 ATP 消耗率图。
- 要确定抑制常数,请使用适当的统计软件将剂量-响应数据拟合到以下二次方程,对应于简单的一对一结合平衡模型:
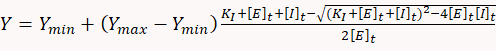
其中Y是 ATP 消费率,Y min是 ATP 消费率,没有抑制剂,Y最大值是理论 ATP 消耗率在 100% 抑制,K I是抑制常数,[E] t和[I]t分别是酶(肌苷)和抑制剂的总浓度。
结果
用于筛选实验的典型板布局图如图1所示。第一行和最后一行分别保留用于NADH校准和正控制(20μM准阿米诺比沙丁,0.5%DMSO)。其余行(B到O)用于测试化合物的抑制活性。在这里,从DMSO中的10 mM化合物浓度开始的15步序1:2稀释被制备并从复合板转移到测定板,使测定板上的最高最终化合物浓度为50μM(以0.5%DMSO为单位)。两行用于获取一种化合?...
讨论
协议中的关键步骤
通过运行多个仅带负控制(无抑制剂的 ATPase 反应)的板,优化板材布局。仔细检查结果,查看反应速率的模式。例如,这些可能由"非结合"板的亲水表面涂层的边缘效应和/或缺陷引起。如果观察到图案,请更改板类型和/或板布局,以尽量减少伪影。例如,典型的剂量反应曲线(带三重的 16 浓度,总计 48 点)可以排列在 384 孔板上的三列或两?...
披露声明
作者没有什么可透露的。
致谢
这项工作得到了国家神经疾病和中风研究所和国家药物滥用研究所NS096833(CAM)的资助。
材料
| Name | Company | Catalog Number | Comments |
| 384-well Low Flange Black Flat Bottom Polystyrene NBS Microplate | Corning | 3575 | |
| ATP (Adenosine 5′-triphosphate disodium salt hydrate) | Sigma | A7699 | |
| Aurora FRD-IB Dispenser | Aurora Discovery, Inc. | 00017425 | |
| Biomek NXP Multichannel Laboratory Automation Workstation | Beckman Coulter | A31841 | |
| Blebbistatin | AMRI | N/A | Custom synthesis |
| BSA (Bovine Serum Albumin, Protease-Free) | Akron Biotech | AK1391 | |
| Centrifuge 5430 R, refrigerated, with Rotor FA-35-6-30 | Eppendorf | 022620663 | |
| Centrifuge 5430, non-refrigerated, with Rotor A-2-MTP | Eppendorf | 022620568 | |
| DMSO (Dimethyl sulfoxide) | Sigma | D2650 | |
| DTT (DL-Dithiothreitol) | Sigma | D5545 | |
| E1 ClipTip Multichannel Pipette; 384-format; 8-channel | Thermo Scientific | 4672010 | |
| E1 ClipTip Multichannel Pipette; 96-format; 8-channel | Thermo Scientific | 4672080 | |
| EGTA (Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid) | Sigma | E3889 | |
| EnVision 2104 Multilabel Plate Reader | PerkinElmer | 2104-0010 | |
| Glycerol | Sigma | G2025 | |
| LDH (L-Lactic Dehydrogenase from rabbit muscle) | Sigma | L1254 | |
| MgCl2.6H2O (Magnesium chloride hexahydrate) | Sigma | M2670 | |
| Microplate Shaker | VWR | 12620-926 | |
| Microplate, 384 well, PP, Small Volume, Deep Well, Natural | Greiner Bio-One | 784201 | |
| MOPS (3-(N-Morpholino)propanesulfonic acid) | Sigma | M1254 | |
| Myosin Motor Protein (full length) (Bovine cardiac muscle) | Cytoskeleton | MY03 | |
| Myosin Motor Protein (full length) (Rabbit skeletal muscle) | Cytoskeleton | MY02 | |
| NADH (β-Nicotinamide adenine dinucleotide, reduced disodium salt hydrate) | Sigma | N8129 | |
| NaN3 (Sodium azide) | Sigma | 71289 | |
| NaOH (Sodium hydroxide) | Sigma | S8045 | |
| Optical Filter CFP 470/24nm (Emission) | PerkinElmer | 2100-5850 | Barcode 240 |
| Optical Filter Fura2 380/10nm (Excitation) | PerkinElmer | 2100-5390 | Barcode 112 |
| Optical Module: Beta Lactamase | PerkinElmer | 2100-4270 | Barcode 418 |
| OriginPro 2017 software | OriginLab | N/A | |
| para-Aminoblebbistatin | AMRI | N/A | Custom synthesis |
| para-Nitroblebbistatin | AMRI | N/A | Custom synthesis |
| PEP (Phospho(enol)pyruvic acid monopotassium salt) | Sigma | P7127 | |
| PK (Pyruvate Kinase from rabbit muscle) | Sigma | P9136 | |
| Rabbit Muscle Acetone Powder | Pel Freez Biologicals | 41995-2 |
参考文献
- Heissler, S. M., Sellers, J. R. Kinetic Adaptations of Myosins for Their Diverse Cellular Functions. Traffic. 17 (8), 839-859 (2016).
- Hartman, M. A., Spudich, J. A. The myosin superfamily at a glance. Journal of Cell Science. 125 (Pt 7), 1627-1632 (2012).
- Berg, J. S., Powell, B. C., Cheney, R. E. A millennial myosin census. Molecular Biology of the Cell. 12 (4), 780-794 (2001).
- Sebe-Pedros, A., Grau-Bove, X., Richards, T. A., Ruiz-Trillo, I. Evolution and classification of myosins, a paneukaryotic whole-genome approach. Genome Biology and Evolution. 6 (2), 290-305 (2014).
- Newell-Litwa, K. A., Horwitz, R., Lamers, M. L. Non-muscle myosin II in disease: mechanisms and therapeutic opportunities. Disease Models & Mechanisms. 8 (12), 1495-1515 (2015).
- He, Y. M., Gu, M. M. Research progress of myosin heavy chain genes in human genetic diseases. Yi Chuan. 39 (10), 877-887 (2017).
- Rauscher, A. A., Gyimesi, M., Kovacs, M., Malnasi-Csizmadia, A. Targeting Myosin by Blebbistatin Derivatives: Optimization and Pharmacological Potential. Trends in Biochemical Sciences. 43 (9), 700-713 (2018).
- Straight, A. F., et al. Dissecting temporal and spatial control of cytokinesis with a myosin II Inhibitor. Science. 299 (5613), 1743-1747 (2003).
- Sirigu, S., et al. Highly selective inhibition of myosin motors provides the basis of potential therapeutic application. Proceedings of the National Academy of Sciences of the United States of America. 113 (47), E7448-E7455 (2016).
- Green, E. M., et al. A small-molecule inhibitor of sarcomere contractility suppresses hypertrophic cardiomyopathy in mice. Science. 351 (6273), 617-621 (2016).
- Morgan, B. P., et al. Discovery of omecamtiv mecarbil the first, selective, small molecule activator of cardiac Myosin. ACS Medicinal Chemistry Letters. 1 (9), 472-477 (2010).
- Kepiro, M., et al. para-Nitroblebbistatin, the non-cytotoxic and photostable myosin II inhibitor. Angewandte Chemie International Edition. 53 (31), 8211-8215 (2014).
- Varkuti, B. H., et al. A highly soluble, non-phototoxic, non-fluorescent blebbistatin derivative. Scientific Reports. 6, 26141 (2016).
- Verhasselt, S., et al. Discovery of (S)-3'-hydroxyblebbistatin and (S)-3'-aminoblebbistatin: polar myosin II inhibitors with superior research tool properties. Organic and Biomolecular Chemistry. 15 (9), 2104-2118 (2017).
- Verhasselt, S., Roman, B. I., Bracke, M. E., Stevens, C. V. Improved synthesis and comparative analysis of the tool properties of new and existing D-ring modified (S)-blebbistatin analogs. European Journal of Medicinal Chemistry. 136, 85-103 (2017).
- Warren, G. B., Toon, P. A., Birdsall, N. J., Lee, A. G., Metcalfe, J. C. Reconstitution of a calcium pump using defined membrane components. Proceedings of the National Academy of Sciences of the United States of America. 71 (3), 622-626 (1974).
- Kiianitsa, K., Solinger, J. A., Heyer, W. D. Rad54 protein exerts diverse modes of ATPase activity on duplex DNA partially and fully covered with Rad51 protein. Journal of Biological Chemistry. 277 (48), 46205-46215 (2002).
- Hanzelmann, P., Schindelin, H. Structural Basis of ATP Hydrolysis and Intersubunit Signaling in the AAA+ ATPase p97. Structure. 24 (1), 127-139 (2016).
- Hackney, D. D., Jiang, W. Assays for kinesin microtubule-stimulated ATPase activity. Methods in Molecular Biology. 164, 65-71 (2001).
- Kiianitsa, K., Solinger, J. A., Heyer, W. D. NADH-coupled microplate photometric assay for kinetic studies of ATP-hydrolyzing enzymes with low and high specific activities. Analytical Biochemistry. 321 (2), 266-271 (2003).
- Carter, S. G., Karl, D. W. Inorganic phosphate assay with malachite green: an improvement and evaluation. Journal of Biochemical and Biophysical Methods. 7 (1), 7-13 (1982).
- Henkel, R. D., VandeBerg, J. L., Walsh, R. A. A microassay for ATPase. Analytical Biochemistry. 169 (2), 312-318 (1988).
- Rowlands, M. G., et al. High-throughput screening assay for inhibitors of heat-shock protein 90 ATPase activity. Analytical Biochemistry. 327 (2), 176-183 (2004).
- Rule, C. S., Patrick, M., Sandkvist, M. Measuring In Vitro ATPase Activity for Enzymatic Characterization. Journal of Visualized Experiments. (114), 54305 (2016).
- Pardee, J. D., Spudich, J. A. Purification of muscle actin. Methods in Cell Biology. 24, 271-289 (1982).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Kovacs, M., Toth, J., Hetenyi, C., Malnasi-Csizmadia, A., Sellers, J. R. Mechanism of blebbistatin inhibition of myosin II. Chem Journal of Biological Chemistry. 279 (34), 35557-35563 (2004).
- Allingham, J. S., Smith, R., Rayment, I. The structural basis of blebbistatin inhibition and specificity for myosin II. Nature Structural & Molecular Biology. 12 (4), 378-379 (2005).
- Kettlun, A. M., et al. Purification and Characterization of 2 Isoapyrases from Solanum-Tuberosum Var Ultimus. Phytochemistry. 31 (11), 3691-3696 (1992).
- Hulme, E. C., Trevethick, M. A. Ligand binding assays at equilibrium: validation and interpretation. British Journal of Pharmacology. 161 (6), 1219-1237 (2010).
- Motulsky, H. J., Neubig, R. R. Analyzing binding data. Current Protocols in Neuroscience. 52 (1), 7.5.1-7.5.65 (2010).
- Sehgal, P., Olesen, C., Moller, J. V. ATPase Activity Measurements by an Enzyme-Coupled Spectrophotometric Assay. Methods in Molecular Biology. 1377, 105-109 (2016).
- Solinger, J. A., Lutz, G., Sugiyama, T., Kowalczykowski, S. C., Heyer, W. D. Rad54 protein stimulates heteroduplex DNA formation in the synaptic phase of DNA strand exchange via specific interactions with the presynaptic Rad51 nucleoprotein filament. Journal of Molecular Biology. 307 (5), 1207-1221 (2001).
- Banik, U., Roy, S. A continuous fluorimetric assay for ATPase activity. Biochemistry Journal. 266 (2), 611-614 (1990).
- Xiao, Y. X., Yang, W. X. KIFC1: a promising chemotherapy target for cancer treatment?. Oncotarget. 7 (30), 48656-48670 (2016).
- See, S. K., et al. Cytoplasmic Dynein Antagonists with Improved Potency and Isoform Selectivity. ACS Chemical Biology. 11 (1), 53-60 (2016).
- Datta, A., Brosh, R. M. New Insights Into DNA Helicases as Druggable Targets for Cancer Therapy. Frontiers in Molecular Biosciences. 5, 59 (2018).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。