Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Adaptation semi-haute débit de l'analyse ATPase couplée au NADH pour le dépistage des petits inhibiteurs des molécules
Dans cet article
Résumé
Un test ATPase couplé à la nicotinamide adenine (NADH) a été adapté au dépistage semi-élevé de la myosine par petite molécule. Cet essai cinétique est exécuté dans un format microplate de 384 puits avec des volumes de réaction totaux de seulement 20 L par puits. La plate-forme devrait s'appliquer à pratiquement n'importe quelle enzyme productrice d'ADP.
Résumé
Les enzymes ATPase utilisent l'énergie libre stockée dans le triphosphate d'adénosine pour catalyser une grande variété de processus biochimiques endergoniques in vivo qui ne se produiraient pas spontanément. Ces protéines sont cruciales pour essentiellement tous les aspects de la vie cellulaire, y compris le métabolisme, la division cellulaire, les réponses aux changements environnementaux et le mouvement. Le protocole présenté ici décrit un nicotinamide adenine dinucleotide (NADH)-couplé ATPase-assay qui a été adapté au criblage semi-haut de débit des inhibiteurs de petite molécule d'ATPase. L'analyse a été appliquée à la myosine du muscle cardiaque et squelettique II, deux ATPases à base d'actine, comme preuve de principe. L'hydrolyse de l'ATP est couplée à l'oxydation de NADH par des réactions enzymatiques dans l'effort. Tout d'abord, l'ADP généré par l'ATPase est régénéré en ATP par pyruvate kinase (PK). PK catalyse la transition du phosphoenolpyruvate (PEP) à la pyruvate en parallèle. Par la suite, le pyruvate est réduit au lactate par la déshydrogénase de lactate (LDH), qui catalyse l'oxydation du NADH en parallèle. Ainsi, la diminution de la concentration d'ATP est directement corrélée à la diminution de la concentration de NADH, qui est suivie du changement à la fluorescence intrinsèque de NADH. Tant que la PPE est disponible dans le système de réaction, la concentration d'ADP reste très faible, évitant l'inhibition de l'enzyme ATPase par son propre produit. En outre, la concentration atp reste presque constante, donnant des cours de temps linéaires. La fluorescence est surveillée en permanence, ce qui permet d'estimer facilement la qualité des données et aide à filtrer les artefacts potentiels (p. ex., résultant de précipitations composées ou de changements thermiques).
Introduction
Les myosines sont des transducteurs d'énergie mécanochimique qui hydrolysent l'adénosine triphosphate (ATP) pour générer un mouvement directionnel le long des filaments du cytosquelette d'actine chez les eucaryotes1,2. Ils se sont adaptés structurellement et kinétiquement à leurs différentes fonctions intracellulaires, telles que le transport d'organites, la contraction musculaire ou la génération de tension cytosquelettique1,2. La superfamille de myosine est représentée par 40 gènes de myosine appartenant à 12 classes distinctes de myosine dans le génome humain3,4. Les membres des classes de myosine jouent divers rôles dans un ensemble très divers de désordres, tels que plusieurs cancers, désordres neurologiques, myopathies squelettiques, et cardiomyopathie hypertrophique5,6. Étant donné le grand nombre de fonctions physiologiques et pathologiques de ces moteurs moléculaires, il n'est pas surprenant qu'ils soient de plus en plus reconnus comme cibles médicamenteuses pour une variété de conditions7. Des progrès significatifs ont été réalisés récemment dans la découverte de nouveaux inhibiteurs de la myosine8,9,10 et activateurs11, et d'améliorer les propriétés des existants12, 13 (en) , 14 (en) , 15.
L'analyse ATPase couplée à la nicotinamide adenine (NADH) a longtemps été utilisée pour mesurer l'activité ATPase de diverses enzymes, telles que le réticulum sarcoplasmique Ca2 pompe ATPase16, la réparation de l'ADN ATPase Rad5417, l'AAA ATPase p9718 ou la kinésine moteur microtubule19. L'analyse utilise un cycle de régénération ATP. L'adénosine diphosphate (ADP) généré par l'ATPase est régénéré en ATP par pyruvate kinase (PK), qui transforme une molécule de phosphoenolpyruvate (PEP) en pyruvate en parallèle. Par la suite, le pyruvate est réduit au lactate par la déshydrogénase de lactate (LDH). Cela, à son tour, oxyde une molécule de NADH à NAD. Par conséquent, la diminution de la concentration de NADH en fonction du temps équivaut au taux d'hydrolyse atp. Le cycle de régénération de l'ATP maintient la concentration d'ATP presque constante et la concentration ADP faible tant que le PEP est disponible. Il en résulte des cours de temps linéaires, ce qui rend simple de déterminer les taux de réaction initiaux et aide à éviter l'inhibition du produit par ADP19. Bien que l'assay ATPase couplé au NADH ait déjà été adapté à un format de 96 puits20, les volumes de réaction élevés (150 l) le rendent relativement cher en raison de la forte demande de réactifs, ce qui le rend moins propice à un dépistage rapide d'un grand nombre de Composés. D'autres méthodes, telles que l'essai vert malachite19,21, qui repose sur la détection du phosphate produit par l'enzyme ATPase, se sont avérées plus appropriées pour la miniaturisation et le dépistage à haut débit22 , 23 Ans, états-unis , 24. Cependant, un point de terminaison est plus susceptible d'être affecté par plusieurs artefacts (discutés ci-dessous), qui peuvent rester inconnus en l'absence de cours à temps plein.
Ici, l'analyse ATPase couplée nADH a été optimisée pour le criblage semi-haut de débit des inhibiteurs de petite molécule. La myosine des muscles squelettiques et cardiaques II et les inhibiteurs de la myosine blebbistatin8, para-aminoblebbistatin13 et para-nitroblebbistatin12 sont utilisés pour démontrer la puissance de l'avertissement, qui repose sur NADH fluorescence comme une lecture. Ce protocole est propice aux projets de dépistage axés sur les enzymes productrices d'ADP.
Protocole
1. Préparation de solutions de stock et de réactifs
- Préparer la solution de stock de dithiothreitol (TNT) en dissolvant la TNT cristalline dans de l'eau distillée à une concentration finale de 1000 mM. Ajuster le pH à 7,0 avec une solution NaOH de 1 M. Aliquot et magasin à -20 oC.
- Préparer la solution de stock ATP en dissolvant l'ATP cristallin dans de l'eau distillée à une concentration finale de 100 mM. Ajuster le pH à 7,0 avec une solution NaOH de 1 M. Aliquot et magasin à -20 oC.
- Préparer 10x tampon NADH contenant 70 mM 3-(N-morpholino)propanesulfonic acid (MOPS), 10 mM MgCl2, 0,9 mM d'éthylène glycol-bis (ether aminoéthyle)-N,N,N,N,Nô,N'-tétraacetic acid (EGTA), et 3 mM NaN3. Ajuster le pH à 7,0 avec une solution NaOH de 1 M. Conserver à 4 oC.
- Préparer 1x tampon de myosine contenant 10 mM MOPS et 0,1 mM EGTA. Ajuster le pH à 7,0 avec une solution NaOH de 1 M. Conserver à 4 oC. Ajouter l'albumine de sérum bovin (BSA) et la TNT à une concentration finale de 0,1 % (w/v%) et 1 mm, respectivement, avant utilisation.
- Préparer 1x tampon d'actine contenant 4 mM MOPS, 0,1 mM EGTA, 2 mM MgCl2, et 3 mM NaN3. Ajuster le pH à 7,0 avec une solution NaOH de 1 M. Conserver à 4 oC. Ajouter bSA et TNT à une concentration finale de 0,1 % (w/v%) et 1 mm, respectivement, avant utilisation.
- Préparer la solution de stock NADH en dissolvant le NADH cristallin dans le tampon 10x NADH à une concentration finale de 5,5 mM. Aliquot et magasin à -20 oC.
- Préparer la solution de stock PEP en dissolvant le PEP cristallin dans un tampon NADH 10x jusqu'à une concentration finale de 50 mM. Aliquot et magasin à -20 oC.
- Préparer la solution de stock LDH en dissolvant la poudre lyophilisée LDH dans un mélange de glycérol et de tampon NADH 10x (50%:50%) à une concentration finale de 2000 U/mL. Centrifuger la solution pour éliminer toute protéine non dissoute présente (7 197 x g, 20 oC, 10 min). Transférer soigneusement le supernatant dans un tube de centrifugeuse propre. Aliquot et magasin à -20 oC.
- Préparer la solution de stock PK en dissolvant la poudre de PK lyophilisée dans un mélange de glycérol et de tampon NADH 10x (50%:50%) à une concentration finale de 10000 U/mL. Centrifuger la solution pour éliminer toute protéine non dissoute présente (7 197 x g, 20 oC, 10 min). Transférer soigneusement le supernatant dans un tube de centrifugeuse propre. Aliquot et magasin à -20 oC.
- Reconstituer les échantillons de myosine du muscle cardiaque et squelettique lyophilisé en ajoutant 100 échantillons d'eau distillée ll pour obtenir des solutions de stock de 10 mg/mL correspondant respectivement à 37,9 m et 40,8 millions de myosines (monomeric). Pour plus de détails, consultez les instructions du fabricant.
- Préparer F-actin à partir de poudre d'acétone musculaire de lapin tel que décrit par Pardee et Spudich25.
2. Mesurer les activités atpases et les effets inhibiteurs des inhibiteurs des petites molécules
- Préparer la plaque composée.
- Dissoudre les composés d'intérêt dans le diméthylsulfoxide (DMSO) de haute qualité.
- Créez des dilutions en série 1:2 en 15 étapes à partir de 10 mM de concentration composée dans DMSO.
- Transférer les échantillons dans une plaque de polypropylène de 384 puits dans des tripliciats (12,5 l chacun) à l'aide d'une pipette multicanal. Utilisez deux rangées sur la plaque composée pour un composé (au lieu de trois colonnes) pour minimiser le nombre de puits potentiellement affectés par les effets de bord. Utilisez les trois derniers puits de la deuxième rangée pour chaque composé comme contrôle négatif (DMSO seulement). N'utilisez pas la première et la dernière rangée sur la plaque pour les dilutions composées.
- Transférer d'un DMSO pur dans les puits de la première rangée (réservé à l'étalonnage NADH).
- Utilisez la dernière rangée pour un contrôle positif.
REMARQUE: Para-aminoblebbistatin à 4 mM de concentration dans DMSO a été utilisé ici.
- Préparer 4500 l de 20 m de solution d'actine diluée pour chaque plaque d'analyse (384 puits microplaque de polystyrène à parois noires) en diluant la solution de stock d'actine dans le tampon d'actine. Mélanger la solution à fond en faisant monter et descendre 30x à l'aide d'une pipette de 5 ml pour réduire la viscosité et l'hétérogénéité en cassant les filaments d'actine. Centrifuger la solution pour éliminer toute protéine précipitée présente (7 197 x g, 20 oC, 10 min). Transférer délicatement le supernatant dans un tube de centrifugeuse propre.
- Préparer le mélange maître contenant des enzymes LDH et PK (« mélange d'enzymes »). Pour chaque plaque d'essai, combinez 171,4 L de solution LDH, 171,4 L de solution PK et 3189,3 L ou 3252,9 L de tampon de myosine pour des analyses impliquant des myosines cardiaques ou squelettiques, respectivement, dans un tube de centrifugeuse conique de 15 ml. N'ajoutez pas de myosine à ce stade pour éviter l'agrégation et les précipitations.
- Préparer le mélange maître contenant tous les substrats (« mélange de substrat »). Pour chaque assiette, combiner 162,1 L d'ATP, 162,1 l de PEP et 324,1 L de solution NADH dans un tube conique de centrifugeuse de 15 ml. N'ajoutez pas d'actine à ce stade pour éviter l'agrégation et les précipitations.
- Créez des dilutions en série 1:2 en sept étapes de NADH pour l'étalonnage à partir de 250 M.
- Mélanger 12,3 L de solution de stock NADH avec 257,7 l de tampon de myosine dans un tube microcentrifuge de 1,5 mL.
- Aliquot 135 l de tampon de myosine en sept tubes microcentrifuges de 1,5 ml.
- Transférer 135 l de solution du premier tube dans le second et mélanger par pipetting. Répéter l'opération jusqu'à ce qu'elle atteigne le tube de 7 e.
- Utilisez le dernier tube comme contrôle sans NADH (tampon seulement).
- À l'aide d'une pipette à 8 canaux, transférer 20 L des solutions d'étalonnage NADH dans la première rangée de la plaque d'assay dans des tripliciats.
- Ajouter 68 l de myosine cardiaque ou 4,2 l de myosine du muscle squelettique II au mélange d'enzymes. Vortex brièvement.
- À l'exception de la première rangée, distribuez 8,4 l du mélange de myosine-enzyme préparé dans chaque puits de la plaque d'essai à l'aide d'un distributeur automatisé.
- Transférer 100 nL de solutions de la plaque composée à la plaque d'essai contenant un mélange d'enzymes à l'aide d'un système automatisé de manipulation liquide équipé d'une tête d'outil de broche de 100 nL.
- Agiter la plaque d'analyse pendant 1 min à température ambiante à 1200 tr/min à l'aide d'un shaker microplaque.
- Ajouter 4 052 L de la solution d'actine centrifugeau au mélange de substrat. Vortex brièvement.
- Distribuer 11,6 L de mélange actin-substrat dans chaque puits de la plaque d'analyse (sauf première rangée) pour démarrer la réaction enzymatique à l'aide d'un distributeur automatisé.
- Agiter la plaque d'analyse pendant 1 min à température ambiante à 1200 tr/min à l'aide d'un shaker microplaque.
- Centrifuger la plaque d'assay à 101 x g pour 30 s.
- Assurez-vous que la température intérieure du lecteur de plaque a été stabilisée à 25 oC. Chargez la plaque et secouez pendant encore 30 s. Cette étape de secousse est nécessaire pour rendre la forme de la surface liquide similaire dans chaque puits et laisse le temps à la plaque d'atteindre la température de mesure.
- Enregistrez la fluorescence NADH pendant 30 min en scannant la plaque en 45 s intervalles. Utilisez un filtre d'excitation de bande passante de 380 nm, 10 nm et un filtre d'émission de bande passante de 470 nm et 24 nm en conjonction avec un miroir dichroïque coupé de 425 nm. Exécutez la mesure en mode haute concentration. Optimisez le nombre de flashs, le gain du détecteur, les dimensions de la plaque et la hauteur de mesure avant d'exécuter les essais.
REMARQUE : Les conditions finales d'essais sont 300 nM cardiaque/20 nM myosine de muscle squelettique II, 10 'M actin, 40 U/mL LDH, 200 U/mL PK, 220 'M NADH, 1 mM PEP, 1 mM ATP dans un tampon contenant 10 mM MOPS (pH '7.0), 2 m MgCl2 , 0,15 mM EGTA, 0,1 mg/mL BSA, 0,5 % (v/v) DMSO et 1 mM DTT. Le volume total est de 20 l/puits. La concentration composée finale la plus élevée est de 50 M. 20 m para-aminoblebbistatin dans 0,5% DMSO sert de contrôle positif et 0,5% DMSO seul est le contrôle négatif. Toutes les mesures sont effectuées en tripliques.
3. Analyse des données
- Tracer l'intensité de fluorescence observée contre le temps pour chaque puits.
- Effectuer une régression linéaire simple pour déterminer la pente et intercepter les réponses de fluorescence pour chaque puits. La pente est proportionnelle au taux de consommation de l'ATP (NADH), tandis que l'interception est proportionnelle à la concentration de NADH au début de la mesure (t ' 0 s).
- Construire une courbe d'étalonnage pour NADH en traçant les interceptions obtenues pour la première rangée de la plaque contre la concentration de NADH. Assurez-vous que les interceptions dépendent linéairement de la concentration nADH.
REMARQUE : Les interceptions estiment les intensités réelles de fluorescence à t ' 0 s avec beaucoup plus de confiance que la moyenne de l'intensité de fluorescence brute se lit à t - 0 s. - Effectuer une simple régression linéaire pour obtenir la pente et intercepter la ligne d'étalonnage NADH.
REMARQUE : L'interception décrit le signal de fond de fluorescence (aucun NADH présent), alors que la pente correspond à l'intensité extrapolée/théorique de fluorescence d'une solution de 1 M NADH dans cette expérience particulière. - Divisez la pente de la réponse de fluorescence obtenue pour le reste des puits par la pente de la ligne d'étalonnage NADH pour convertir les changements de fluorescence aux taux de consommation d'ATP.
- Tracer les taux de consommation d'ATP par rapport à la concentration de l'inhibiteur.
- Pour déterminer les constantes inhibitrices, utilisez un logiciel statistique approprié pour adapter les données dose-réponse à l'équation quadratique suivante correspondant à un modèle d'équilibre contraignant simple :
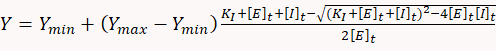
où Y est le taux de consommation ATP, Ymin est le taux de consommation ATP int l'absence d'inhibiteur, Ymax est le taux théorique de consommation ATP à 100% d'inhibition, KI est la constante inhibitrice , [E]t et [i]t sont la concentration totale de l'enzyme (myosine) et de l'inhibiteur, respectivement.
Résultats
La carte typique de mise en page des plaques utilisée pour les expériences de dépistage est illustrée dans la figure 1. Les première et dernière rangées sont réservées à l'étalonnage et au contrôle positif du NADH (20 M para-aminoblebbistatin, 0,5% de DMSO), respectivement. Les lignes restantes (B à O) sont utilisées pour tester l'activité inhibitrice des composés. Ici, les dilutions en série 1:2 en quinze étapes à partir de 10 mM...
Discussion
Étapes critiques du protocole
Optimisez la disposition des plaques en exécutant plusieurs plaques avec un contrôle négatif seulement (réaction ATPase sans inhibiteur). Inspectez soigneusement les résultats pour les tendances des taux de réaction. Par exemple, ceux-ci peuvent résulter d'effets de bord et/ou d'imperfections dans le revêtement hydrophile de surface des plaques « non contraignantes ». Si un modèle est observé, changez le type de plaque et/ou la dispos...
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Ce travail a été soutenu par une subvention du National Institute of Neurological Disorders and Stroke et du National Institute on Drug Abuse NS096833 (CAM).
matériels
| Name | Company | Catalog Number | Comments |
| 384-well Low Flange Black Flat Bottom Polystyrene NBS Microplate | Corning | 3575 | |
| ATP (Adenosine 5′-triphosphate disodium salt hydrate) | Sigma | A7699 | |
| Aurora FRD-IB Dispenser | Aurora Discovery, Inc. | 00017425 | |
| Biomek NXP Multichannel Laboratory Automation Workstation | Beckman Coulter | A31841 | |
| Blebbistatin | AMRI | N/A | Custom synthesis |
| BSA (Bovine Serum Albumin, Protease-Free) | Akron Biotech | AK1391 | |
| Centrifuge 5430 R, refrigerated, with Rotor FA-35-6-30 | Eppendorf | 022620663 | |
| Centrifuge 5430, non-refrigerated, with Rotor A-2-MTP | Eppendorf | 022620568 | |
| DMSO (Dimethyl sulfoxide) | Sigma | D2650 | |
| DTT (DL-Dithiothreitol) | Sigma | D5545 | |
| E1 ClipTip Multichannel Pipette; 384-format; 8-channel | Thermo Scientific | 4672010 | |
| E1 ClipTip Multichannel Pipette; 96-format; 8-channel | Thermo Scientific | 4672080 | |
| EGTA (Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid) | Sigma | E3889 | |
| EnVision 2104 Multilabel Plate Reader | PerkinElmer | 2104-0010 | |
| Glycerol | Sigma | G2025 | |
| LDH (L-Lactic Dehydrogenase from rabbit muscle) | Sigma | L1254 | |
| MgCl2.6H2O (Magnesium chloride hexahydrate) | Sigma | M2670 | |
| Microplate Shaker | VWR | 12620-926 | |
| Microplate, 384 well, PP, Small Volume, Deep Well, Natural | Greiner Bio-One | 784201 | |
| MOPS (3-(N-Morpholino)propanesulfonic acid) | Sigma | M1254 | |
| Myosin Motor Protein (full length) (Bovine cardiac muscle) | Cytoskeleton | MY03 | |
| Myosin Motor Protein (full length) (Rabbit skeletal muscle) | Cytoskeleton | MY02 | |
| NADH (β-Nicotinamide adenine dinucleotide, reduced disodium salt hydrate) | Sigma | N8129 | |
| NaN3 (Sodium azide) | Sigma | 71289 | |
| NaOH (Sodium hydroxide) | Sigma | S8045 | |
| Optical Filter CFP 470/24nm (Emission) | PerkinElmer | 2100-5850 | Barcode 240 |
| Optical Filter Fura2 380/10nm (Excitation) | PerkinElmer | 2100-5390 | Barcode 112 |
| Optical Module: Beta Lactamase | PerkinElmer | 2100-4270 | Barcode 418 |
| OriginPro 2017 software | OriginLab | N/A | |
| para-Aminoblebbistatin | AMRI | N/A | Custom synthesis |
| para-Nitroblebbistatin | AMRI | N/A | Custom synthesis |
| PEP (Phospho(enol)pyruvic acid monopotassium salt) | Sigma | P7127 | |
| PK (Pyruvate Kinase from rabbit muscle) | Sigma | P9136 | |
| Rabbit Muscle Acetone Powder | Pel Freez Biologicals | 41995-2 |
Références
- Heissler, S. M., Sellers, J. R. Kinetic Adaptations of Myosins for Their Diverse Cellular Functions. Traffic. 17 (8), 839-859 (2016).
- Hartman, M. A., Spudich, J. A. The myosin superfamily at a glance. Journal of Cell Science. 125 (Pt 7), 1627-1632 (2012).
- Berg, J. S., Powell, B. C., Cheney, R. E. A millennial myosin census. Molecular Biology of the Cell. 12 (4), 780-794 (2001).
- Sebe-Pedros, A., Grau-Bove, X., Richards, T. A., Ruiz-Trillo, I. Evolution and classification of myosins, a paneukaryotic whole-genome approach. Genome Biology and Evolution. 6 (2), 290-305 (2014).
- Newell-Litwa, K. A., Horwitz, R., Lamers, M. L. Non-muscle myosin II in disease: mechanisms and therapeutic opportunities. Disease Models & Mechanisms. 8 (12), 1495-1515 (2015).
- He, Y. M., Gu, M. M. Research progress of myosin heavy chain genes in human genetic diseases. Yi Chuan. 39 (10), 877-887 (2017).
- Rauscher, A. A., Gyimesi, M., Kovacs, M., Malnasi-Csizmadia, A. Targeting Myosin by Blebbistatin Derivatives: Optimization and Pharmacological Potential. Trends in Biochemical Sciences. 43 (9), 700-713 (2018).
- Straight, A. F., et al. Dissecting temporal and spatial control of cytokinesis with a myosin II Inhibitor. Science. 299 (5613), 1743-1747 (2003).
- Sirigu, S., et al. Highly selective inhibition of myosin motors provides the basis of potential therapeutic application. Proceedings of the National Academy of Sciences of the United States of America. 113 (47), E7448-E7455 (2016).
- Green, E. M., et al. A small-molecule inhibitor of sarcomere contractility suppresses hypertrophic cardiomyopathy in mice. Science. 351 (6273), 617-621 (2016).
- Morgan, B. P., et al. Discovery of omecamtiv mecarbil the first, selective, small molecule activator of cardiac Myosin. ACS Medicinal Chemistry Letters. 1 (9), 472-477 (2010).
- Kepiro, M., et al. para-Nitroblebbistatin, the non-cytotoxic and photostable myosin II inhibitor. Angewandte Chemie International Edition. 53 (31), 8211-8215 (2014).
- Varkuti, B. H., et al. A highly soluble, non-phototoxic, non-fluorescent blebbistatin derivative. Scientific Reports. 6, 26141 (2016).
- Verhasselt, S., et al. Discovery of (S)-3'-hydroxyblebbistatin and (S)-3'-aminoblebbistatin: polar myosin II inhibitors with superior research tool properties. Organic and Biomolecular Chemistry. 15 (9), 2104-2118 (2017).
- Verhasselt, S., Roman, B. I., Bracke, M. E., Stevens, C. V. Improved synthesis and comparative analysis of the tool properties of new and existing D-ring modified (S)-blebbistatin analogs. European Journal of Medicinal Chemistry. 136, 85-103 (2017).
- Warren, G. B., Toon, P. A., Birdsall, N. J., Lee, A. G., Metcalfe, J. C. Reconstitution of a calcium pump using defined membrane components. Proceedings of the National Academy of Sciences of the United States of America. 71 (3), 622-626 (1974).
- Kiianitsa, K., Solinger, J. A., Heyer, W. D. Rad54 protein exerts diverse modes of ATPase activity on duplex DNA partially and fully covered with Rad51 protein. Journal of Biological Chemistry. 277 (48), 46205-46215 (2002).
- Hanzelmann, P., Schindelin, H. Structural Basis of ATP Hydrolysis and Intersubunit Signaling in the AAA+ ATPase p97. Structure. 24 (1), 127-139 (2016).
- Hackney, D. D., Jiang, W. Assays for kinesin microtubule-stimulated ATPase activity. Methods in Molecular Biology. 164, 65-71 (2001).
- Kiianitsa, K., Solinger, J. A., Heyer, W. D. NADH-coupled microplate photometric assay for kinetic studies of ATP-hydrolyzing enzymes with low and high specific activities. Analytical Biochemistry. 321 (2), 266-271 (2003).
- Carter, S. G., Karl, D. W. Inorganic phosphate assay with malachite green: an improvement and evaluation. Journal of Biochemical and Biophysical Methods. 7 (1), 7-13 (1982).
- Henkel, R. D., VandeBerg, J. L., Walsh, R. A. A microassay for ATPase. Analytical Biochemistry. 169 (2), 312-318 (1988).
- Rowlands, M. G., et al. High-throughput screening assay for inhibitors of heat-shock protein 90 ATPase activity. Analytical Biochemistry. 327 (2), 176-183 (2004).
- Rule, C. S., Patrick, M., Sandkvist, M. Measuring In Vitro ATPase Activity for Enzymatic Characterization. Journal of Visualized Experiments. (114), 54305 (2016).
- Pardee, J. D., Spudich, J. A. Purification of muscle actin. Methods in Cell Biology. 24, 271-289 (1982).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Kovacs, M., Toth, J., Hetenyi, C., Malnasi-Csizmadia, A., Sellers, J. R. Mechanism of blebbistatin inhibition of myosin II. Chem Journal of Biological Chemistry. 279 (34), 35557-35563 (2004).
- Allingham, J. S., Smith, R., Rayment, I. The structural basis of blebbistatin inhibition and specificity for myosin II. Nature Structural & Molecular Biology. 12 (4), 378-379 (2005).
- Kettlun, A. M., et al. Purification and Characterization of 2 Isoapyrases from Solanum-Tuberosum Var Ultimus. Phytochemistry. 31 (11), 3691-3696 (1992).
- Hulme, E. C., Trevethick, M. A. Ligand binding assays at equilibrium: validation and interpretation. British Journal of Pharmacology. 161 (6), 1219-1237 (2010).
- Motulsky, H. J., Neubig, R. R. Analyzing binding data. Current Protocols in Neuroscience. 52 (1), 7.5.1-7.5.65 (2010).
- Sehgal, P., Olesen, C., Moller, J. V. ATPase Activity Measurements by an Enzyme-Coupled Spectrophotometric Assay. Methods in Molecular Biology. 1377, 105-109 (2016).
- Solinger, J. A., Lutz, G., Sugiyama, T., Kowalczykowski, S. C., Heyer, W. D. Rad54 protein stimulates heteroduplex DNA formation in the synaptic phase of DNA strand exchange via specific interactions with the presynaptic Rad51 nucleoprotein filament. Journal of Molecular Biology. 307 (5), 1207-1221 (2001).
- Banik, U., Roy, S. A continuous fluorimetric assay for ATPase activity. Biochemistry Journal. 266 (2), 611-614 (1990).
- Xiao, Y. X., Yang, W. X. KIFC1: a promising chemotherapy target for cancer treatment?. Oncotarget. 7 (30), 48656-48670 (2016).
- See, S. K., et al. Cytoplasmic Dynein Antagonists with Improved Potency and Isoform Selectivity. ACS Chemical Biology. 11 (1), 53-60 (2016).
- Datta, A., Brosh, R. M. New Insights Into DNA Helicases as Druggable Targets for Cancer Therapy. Frontiers in Molecular Biosciences. 5, 59 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon