È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Adattamento semi-alto/throughput del saggio ATPase accoppiato NADH per lo screening degli inibitori delle piccole molecole
In questo articolo
Riepilogo
Un saggio ATPase accoppiato a nicotinamide (NADH) è stato adattato allo screening semi-alto flusso di inibitori della miosina. Questo saggio cinetico viene eseguito in un formato a microplacca 384 pozzetti con volumi di reazione totali di soli 20 l per pozzo. La piattaforma dovrebbe essere applicabile praticamente a qualsiasi enzima che produce ADP.
Abstract
Gli enzimi ATPase utilizzano l'energia libera immagazzinata nel triposfato adenosina per catalizzare un'ampia varietà di processi biochimici endergonici in vivo che non si verificherebbero spontaneamente. Queste proteine sono fondamentali per essenzialmente tutti gli aspetti della vita cellulare, tra cui il metabolismo, la divisione cellulare, le risposte ai cambiamenti ambientali e il movimento. Il protocollo qui presentato descrive un saggio ATPase accoppiato a nicotinamide (NADH) che è stato adattato allo screening semi-alto della produttività degli inibitori ATPase a piccole molecole. Il saggio è stato applicato alla miosina cardiaca e scheletrica di miosina II, due ATPases motori molecolari basati su actin, come prova di principio. L'idrolisi dell'ATP è accoppiata all'ossidazione di NADH da reazioni enzimatiche nel saggio. In primo luogo, l'ADP generato dall'ATPase viene rigenerato in ATP dalla chinasi pirativae (PK). PK catalizza la transizione del fosforolpyruvate (PEP) alla pirofavia in parallelo. Successivamente, il pirate viene ridotto al lattato mediante dehydrogenase di lattato (LDH), che catalizza l'ossidazione di NADH in parallelo. Pertanto, la diminuzione della concentrazione di ATP è direttamente correlata alla diminuzione della concentrazione di NADH, seguita da un cambiamento alla fluorescenza intrinseca di NADH. Finché la PEP è disponibile nel sistema di reazione, la concentrazione di ADP rimane molto bassa, evitando l'inibizione dell'enzima ATPase da parte del proprio prodotto. Inoltre, la concentrazione ATP rimane quasi costante, producendo corsi temporali lineari. La fluorescenza viene monitorata continuamente, il che consente una facile stima della qualità dei dati e aiuta a filtrare i potenziali artefatti (ad esempio, derivanti da precipitazioni composte o cambiamenti termici).
Introduzione
I miosini sono trasduttori di energia meccanochimica che idrolizzano il tripfosfato di adenosina (ATP) per generare movimento direzionale lungo i filamenti del citoscheletro actinos in eucarioti1,2. Si sono adattati sia strutturalmente che kineticamente alle loro varie funzioni intracellulari, come il trasporto di organelli, la contrazione muscolare o la generazione di tensione citoscheletrica1,2. La superfamiglia della miosina è rappresentata da 40 geni della miosina appartenenti a classi di miosina distinte da 12 dollari nel genoma umano3,4. I membri delle classi di miosina svolgono vari ruoli in una serie molto diversificata di disturbi, come diversi tumori, disturbi neurologici, miopatie scheletriche e cardiomiopatia ipertrofica5,6. Dato il gran numero di funzioni fisiologiche e patologiche di questi motori molecolari, non è sorprendente che stiano diventando sempre più riconosciuti come bersagli farmacologici per una varietà di condizioni7. Recentemente sono stati compiuti progressi significativi nella scoperta di nuovi inibitori della miosina8,9,10 e attivatori11, e per migliorare le proprietà di quelli esistenti12, 13 del sistema , 14 Del sistema , 15.
Il saggio ATPase (NADH) accoppiato alla nicotinammide adenina (NADH) è stato a lungo utilizzato per misurare l'attività ATPase di vari enzimi, come il reticulum sarccoplasmico Ca2o pompa ATPase16, la riparazione del DNA ATPase Rad5417, l'AAA ATPase p9718 o il motore microtubulo kinesin19. Il saggio utilizza un ciclo di rigenerazione ATP. Il difosfato di adenosina (ADP) generato dall'ATPase viene rigenerato all'ATP dalla chinasi pirata (PK), che trasforma una molecola di fosforenolpyruvate (PEP) in pyruvate in parallelo. Successivamente, il pirate viene ridotto al lattato dalla dehydrogenasi lattato (LDH). Che, a sua volta, ossida una molecola di NADH a NAD. Pertanto, la diminuzione della concentrazione di NADH in funzione del tempo è uguale al tasso di idrolisi ATP. Il ciclo di rigenerazione ATP mantiene la concentrazione ATP quasi costante e la concentrazione di ADP bassa fino a quando è disponibile PEP. Questo si traduce in corsi di tempo lineari, rendendo semplice determinare i tassi di reazione iniziali e aiuta a evitare l'inibizione del prodotto da ADP19. Anche se il test ATPase accoppiato con NADH è già stato adattato ad un formatoa96 ben20, gli elevati volumi di reazione (150 dollari) lo rendono relativamente costoso a causa dell'elevata domanda di reagenti, rendendolo meno suscettibile di screening rapido di un gran numero di Composti. I metodi alternativi, come il test verde malessere19,21, che si basa sulla rilevazione del fosfato prodotto dall'enzima ATPase, si sono dimostrati più adatti per la miniaturizzazione e lo screening ad alto valore22 , 23 del 23 o , 24.Tuttavia, è più probabile che un'espressione endpoint sia influenzata da diversi artefatti (discussi di seguito), che potrebbero rimanere sconosciuti in assenza di corsi a tempo pieno.
In questo caso, il saggio ATPase accoppiato nADH è stato ottimizzato per lo screening semi-alto della produttività degli inibitori delle piccole molecole. La miosina scheletrica e cardiaca muscolare miosina II e gli inibitori della miosina blebbistatin8, para-aminoblebbistatin13 e para-nitroblebbistatin12 sono utilizzati per dimostrare la potenza del saggio, che si basa su NADH fluorescenza come una lettura. Questo protocollo è suscettibile di vagliare progetti incentrati su qualsiasi enzimi che producono ADP.
Protocollo
1. Preparazione di soluzioni e reagenti azionari
- Preparare la soluzione di stock dithiothreitol (DTT) sciogliendo il DTT cristallino in acqua distillata fino a una concentrazione finale di 1000 mM. Regolare il pH a 7.0 con una soluzione NaOH da 1 M. Aliquota e conservare a -20 gradi centigradi.
- Preparare la soluzione di riserva ATP sciogliendo l'ATP cristallino nell'acqua distillata fino a una concentrazione finale di 100 mM. Regolare il pH a 7.0 con una soluzione NaOH da 1 M. Aliquota e conservare a -20 gradi centigradi.
- Preparare il buffer 10x NADH contenente 70 mM 3-(N-morpholino)propanesulfonico acid (MOPS), 10 mM MgCl2, 0,9 m etilene glicol-bis(Etere n-aminoethyl)-N,N'n'tetraacetic acid (EGTA) e 3 mM NaN3. Regolare il pH a 7.0 con una soluzione NaOH da 1 M. Conservare a 4 gradi centigradi.
- Preparare 1x buffer di miosina contenente 10 mM MOPS e 0,1 mM EGTA. Regolare il pH a 7.0 con una soluzione NaOH da 1 M. Conservare a 4 gradi centigradi. Aggiungere l'albumina del siero bovino (BSA) e la DTT a una concentrazione finale dello 0,1% (w/v%) e 1 mM, rispettivamente, prima dell'uso.
- Preparare 1x buffer di actin contenente 4 mM MOPS, 0,1 mM EGTA, 2 mM MgCl2e 3 mM NaN3. Regolare il pH a 7.0 con una soluzione NaOH da 1 M. Conservare a 4 gradi centigradi. Aggiungere BSA e DTT a una concentrazione finale dello 0,1% (w/v%) e 1 mM, rispettivamente, prima dell'uso.
- Preparare la soluzione di stock NADH sciogliendo il nADH cristallino nel buffer 10x NADH a una concentrazione finale di 5,5 mM. Aliquota e conservare a -20 gradi centigradi.
- Preparare la soluzione stock PEP sciogliendo pep cristallino nel buffer 10x NADH ad una concentrazione finale di 50 mM. Aliquota e conservare a -20 gradi centigradi.
- Preparare la soluzione stock LDH sciogliendo la polvere LDH lofilia in una miscela di glicerolo e buffer 10x NADH (50%:50%) ad una concentrazione finale di 2000 U/mL. Centrifugare la soluzione per rimuovere qualsiasi proteina non disciolta presente (7.197 x g, 20 c, 10 min). Trasferire il supernatante in un tubo di centrifuga pulito con attenzione. Aliquota e conservare a -20 gradi centigradi.
- Preparare la soluzione PK stock sciogliendo la polvere di PK liofilizzata in una miscela di glicerolo e 10x buffer NADH (50%:50%) ad una concentrazione finale di 10000 U/mL. Centrifugare la soluzione per rimuovere qualsiasi proteina non disciolta presente (7.197 x g, 20 c, 10 min). Trasferire il supernatante in un tubo di centrifuga pulito con attenzione. Aliquota e conservare a -20 gradi centigradi.
- Ricostituire i campioni di miosina del muscolo leofilato cardiaco e scheletrico aggiungendo rispettivamente 100 acqua distillata l'acqua per ottenere 10 soluzioni di stock mg/mL corrispondenti rispettivamente a 37,9 e 40,8 M di concentrazioni di miosina (monomerico). Per ulteriori dettagli, vedere le istruzioni del produttore.
- Preparare F-actin da polvere di acetone muscolo coniglio come descritto da Pardee e Spudich25.
2. Misurazione delle attività ATPase e degli effetti inibitori degli inibitori delle piccole molecole
- Preparare la piastra composta.
- Sciogliere composti di interesse in dimethylsulfossido di alta qualità (DMSO).
- Creare diluizioni seriali 1:2 in 13 fasi a partire dalla concentrazione composta di 10 mM in DMSO.
- Trasferire i campioni su una piastra di polipropilene 384 pozzetto in triplice (12,5 l ciascuno) utilizzando una pipetta multicanale. Utilizzare due file sulla piastra composta per un composto (invece di tre colonne) per ridurre al minimo il numero di pozzi potenzialmente influenzati dagli effetti del bordo. Utilizzare gli ultimi tre pozze nella seconda riga per ogni composto come controllo negativo (solo DMSO). Non utilizzare la prima e l'ultima fila sulla piastra per le diluizioni composte.
- Trasferire DMSO puro nei pozze della prima fila (riservato per la calibrazione NADH).
- Utilizzare l'ultima riga per il controllo positivo.
NOTA: Qui è stata utilizzata lapara-aminoblebbistatin a 4 mM di concentrazione nel DMSO.
- Preparare una soluzione di actina diluita per ogni piastra di analisi (piastra di asini 384-well black-wall microptirone) diluendo la soluzione di riserva di actina nel buffer di actina. Mescolare accuratamente la soluzione pipetting up e down 30x utilizzando una pipetta da 5 mL per ridurre la viscosità e l'eterogeneità rompendo i filamenti di actina. Centrifugare la soluzione per rimuovere qualsiasi proteina precipitata presente (7.197 x g, 20 c, 10 min). Trasferire con attenzione il supernatante in un tubo di centrifuga pulito.
- Preparare il mix master contenente enzimi LDH e PK ("mix di enzimi"). Per ogni plato di analisi, unire 171,4 l di soluzione LDH, 171,4 l di soluzione PK e 3189,3 l o 3252,9 l di buffer di miosina per i saggi che coinvolgono la miosina del muscolo cardiaco o scheletrico II, rispettivamente, in un tubo centrifuga connico 15 mL. Non aggiungere alcuna miosina a questo punto per evitare l'aggregazione e le precipitazioni.
- Preparare la miscela principale contenente tutti i substrati ("mix di substrati"). Per ogni piastra, unire 162,1 gradi di ATP, 162,1 l di PEP e 324,1 L di soluzione NADH in un tubo di centrifuga conica da 15 mL. Non aggiungere actin a questo punto per evitare l'aggregazione e le precipitazioni.
- Creare diluizioni seriali 1:2 in sette passaggi di NADH per la calibrazione a partire da 250 M.
- Mescolare la soluzione di stock da 12,3 litri di NADH con 257,7 tamponanti di miosina in un tubo di microcentrifuga da 1,5 ml.
- Aliquota 135 L di buffer di miosina in sette tubi di microcentrifuga da 1,5 ml.
- Trasferire 135 l di soluzione dal primo tubo al secondo e mescolare con la pipettatura. Ripetere fino a raggiungere il tubo 7 th.
- Utilizzare l'ultimo tubo come controllo no-NADH (solo buffer).
- Utilizzando una pipetta a 8 canali, trasferire 20 gradi l delle soluzioni di calibrazione NADH nella prima fila della piastra di analisi in triplicati.
- Aggiungere al mix di enzimi 68 -L di cardiaco o 4,2 gradi di miosina muscolare scheletrica. Vortice brevemente.
- Ad eccezione della prima fila, erogare 8,4 litri della miscela miosina-enzima preparata in ogni pozzo della piastra di analisi utilizzando un dispenser automatizzato.
- Trasferire 100 nL di soluzioni dalla piastra composta alla piastra di analisi contenente mix di enzimi utilizzando un sistema di movimentazione liquida automatizzato dotato di una testa di utensile perno da 100 nL.
- Agitare la piastra di analisi per 1 min a temperatura ambiente a 1200 giri/mm utilizzando uno shaker a microplacca.
- Aggiungere alla miscela di substrato 4.052 l della soluzione di actina centrifuga. Vortice brevemente.
- Distribuisci 11,6 l di miscela actina-substrato in ogni pozzetto della piastra di analisi (tranne la prima fila) per avviare la reazione enzimatica utilizzando un dispenser automatizzato.
- Agitare la piastra di analisi per 1 min a temperatura ambiente a 1200 giri/mm utilizzando uno shaker a microplacca.
- Centrifugare la piastra di saggio a 101 x g per 30 s.
- Assicurarsi che la temperatura interna del lettore di placche sia stata stabilizzata a 25 gradi centigradi. Caricare la piastra e agitare per altri 30 s. Questa fase di agitazione è necessaria per rendere la forma della superficie liquida simile in ogni pozzo e consente alla piastra di raggiungere la temperatura di misurazione.
- Registrare la fluorescenza NADH per la scansione della piastra a 30 min a intervalli di 45 s. Utilizzare un filtro di eccitazione larghezza di banda 380 nm e un filtro di emissione larghezza di banda da 24 nm di 470 nm in combinazione con un mirror dicroico tagliato a 425 nm. Eseguire la misurazione in modalità ad alta concentrazione. Ottimizzare il numero di lampi, guadagno del rilevatore, dimensioni della piastra e altezza di misurazione prima di eseguire i saggi.
NOTA: le condizioni di analisi finale sono 300 nM cardiaco/20 nM muscolo scheletrico miosina II, 10 actina m, 40 U/mL LDH, 200 U/mL PK, 220M - NADH, 1 mM PEP, 1 mM ATP in un buffer contenente 10 mM MOPS (pH 7,0), 2 mM MGCl2 , 0,15 mM EGTA, 0,1 mg/mL BSA, 0,5% (v/v) DMSO e 1 mM DTT. Il volume totale è di 20 l/well. La più alta concentrazione finale di composti è 50 m. 20 m para-aminoblebbistatin in 0,5% DMSO serve come controllo positivo e 0,5% DMSO da solo è il controllo negativo. Tutte le misurazioni vengono effettuate in triplicati.
3. Analisi dei dati
- Tracciare l'intensità di fluorescenza osservata rispetto al tempo per ogni pozzo.
- Eseguire una regressione lineare semplice per determinare la pendenza e l'intercetta delle risposte di fluorescenza per ogni pozzo. La pendenza è proporzionale al tasso di consumo atP (NADH), mentre l'intercetta è proporzionale alla concentrazione di NADH all'inizio della misurazione (t - 0 s).
- Costruire una curva di calibrazione per NADH tracciando le intercettazioni ottenute per la prima fila della piastra contro la concentrazione di NADH. Assicurarsi che le intercettazioni dipendano linearmente dalla concentrazione di NADH.
NOTA: Le intercettazioni stimano le intensità di fluorescenza reale a t s 0 s con molta più sicurezza rispetto alla media dell'intensità di fluorescenza grezza si legge a 0 s. - Eseguire la regressione lineare semplice per ottenere la pendenza e l'intercetta della linea di calibrazione NADH.
NOTA: L'intercetta descrive il segnale di fondo a fluorescenza (nessun NADH presente), mentre la pendenza corrisponde all'intensità di fluorescenza estrapolata/teorica di una soluzione NADH da 1 M in quel particolare esperimento. - Dividere la pendenza della risposta di fluorescenza ottenuta per il resto dei pozzi per la pendenza della linea di calibrazione NADH per convertire le variazioni di fluorescenza nei tassi di consumo ATP.
- Tracciare i tassi di consumo dell'ATP rispetto alla concentrazione dell'inibitore.
- Per determinare le costanti inibitorie, utilizzare un software statistico appropriato per adattare i dati dose-risposta alla seguente equazione quadratica corrispondente a un semplice modello di equilibrio di legame uno a uno:
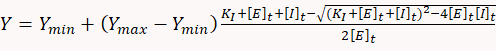
dove Y è il tasso di consumo ATP, Ymin è il tasso di consumo ATP inassenza dell'inibitore, Ymax è il tasso teorico di consumo ATP al 100% inibizione, KI è la costante inibitoria , [E]t e [I]t sono rispettivamente la concentrazione totale dell'enzima (miosina) e dell'inibitore.
Risultati
La tipica mappa di layout delle lame utilizzata per gli esperimenti di screening è illustrata nella Figura1. La prima e l'ultima riga sono riservate rispettivamente alla calibrazione NADH e al controllo positivo (20 m para-aminoblebbistatin, 0,5% DMSO). Le righe rimanenti (da B a O) vengono utilizzate per testare l'attività inibitoria dei composti. Qui, le diluizioni seriali 1:2 in quindici in fasi a partire dalla concentrazione composta di 10 mM i...
Discussione
Passaggi critici nel protocollo
Ottimizzare il layout della piastra eseguendo più piastre con controllo negativo (reazione ATPase senza inibitore). Esaminare attentamente i risultati per i modelli nei tassi di reazione. Ad esempio, questi possono derivare da effetti di bordo e/o imperfezioni nel rivestimento superficiale idrofilo di piastre "non vincolanti". Se si osserva un motivo, modificare il tipo di piastra e/o il layout della lastra per ridurre al minimo gli artefatti. ...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto da una sovvenzione dell'Istituto Nazionale di Disturbi Neurologici e Ictus e Istituto Nazionale per l'abuso di droga NS096833 (CAM).
Materiali
| Name | Company | Catalog Number | Comments |
| 384-well Low Flange Black Flat Bottom Polystyrene NBS Microplate | Corning | 3575 | |
| ATP (Adenosine 5′-triphosphate disodium salt hydrate) | Sigma | A7699 | |
| Aurora FRD-IB Dispenser | Aurora Discovery, Inc. | 00017425 | |
| Biomek NXP Multichannel Laboratory Automation Workstation | Beckman Coulter | A31841 | |
| Blebbistatin | AMRI | N/A | Custom synthesis |
| BSA (Bovine Serum Albumin, Protease-Free) | Akron Biotech | AK1391 | |
| Centrifuge 5430 R, refrigerated, with Rotor FA-35-6-30 | Eppendorf | 022620663 | |
| Centrifuge 5430, non-refrigerated, with Rotor A-2-MTP | Eppendorf | 022620568 | |
| DMSO (Dimethyl sulfoxide) | Sigma | D2650 | |
| DTT (DL-Dithiothreitol) | Sigma | D5545 | |
| E1 ClipTip Multichannel Pipette; 384-format; 8-channel | Thermo Scientific | 4672010 | |
| E1 ClipTip Multichannel Pipette; 96-format; 8-channel | Thermo Scientific | 4672080 | |
| EGTA (Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid) | Sigma | E3889 | |
| EnVision 2104 Multilabel Plate Reader | PerkinElmer | 2104-0010 | |
| Glycerol | Sigma | G2025 | |
| LDH (L-Lactic Dehydrogenase from rabbit muscle) | Sigma | L1254 | |
| MgCl2.6H2O (Magnesium chloride hexahydrate) | Sigma | M2670 | |
| Microplate Shaker | VWR | 12620-926 | |
| Microplate, 384 well, PP, Small Volume, Deep Well, Natural | Greiner Bio-One | 784201 | |
| MOPS (3-(N-Morpholino)propanesulfonic acid) | Sigma | M1254 | |
| Myosin Motor Protein (full length) (Bovine cardiac muscle) | Cytoskeleton | MY03 | |
| Myosin Motor Protein (full length) (Rabbit skeletal muscle) | Cytoskeleton | MY02 | |
| NADH (β-Nicotinamide adenine dinucleotide, reduced disodium salt hydrate) | Sigma | N8129 | |
| NaN3 (Sodium azide) | Sigma | 71289 | |
| NaOH (Sodium hydroxide) | Sigma | S8045 | |
| Optical Filter CFP 470/24nm (Emission) | PerkinElmer | 2100-5850 | Barcode 240 |
| Optical Filter Fura2 380/10nm (Excitation) | PerkinElmer | 2100-5390 | Barcode 112 |
| Optical Module: Beta Lactamase | PerkinElmer | 2100-4270 | Barcode 418 |
| OriginPro 2017 software | OriginLab | N/A | |
| para-Aminoblebbistatin | AMRI | N/A | Custom synthesis |
| para-Nitroblebbistatin | AMRI | N/A | Custom synthesis |
| PEP (Phospho(enol)pyruvic acid monopotassium salt) | Sigma | P7127 | |
| PK (Pyruvate Kinase from rabbit muscle) | Sigma | P9136 | |
| Rabbit Muscle Acetone Powder | Pel Freez Biologicals | 41995-2 |
Riferimenti
- Heissler, S. M., Sellers, J. R. Kinetic Adaptations of Myosins for Their Diverse Cellular Functions. Traffic. 17 (8), 839-859 (2016).
- Hartman, M. A., Spudich, J. A. The myosin superfamily at a glance. Journal of Cell Science. 125 (Pt 7), 1627-1632 (2012).
- Berg, J. S., Powell, B. C., Cheney, R. E. A millennial myosin census. Molecular Biology of the Cell. 12 (4), 780-794 (2001).
- Sebe-Pedros, A., Grau-Bove, X., Richards, T. A., Ruiz-Trillo, I. Evolution and classification of myosins, a paneukaryotic whole-genome approach. Genome Biology and Evolution. 6 (2), 290-305 (2014).
- Newell-Litwa, K. A., Horwitz, R., Lamers, M. L. Non-muscle myosin II in disease: mechanisms and therapeutic opportunities. Disease Models & Mechanisms. 8 (12), 1495-1515 (2015).
- He, Y. M., Gu, M. M. Research progress of myosin heavy chain genes in human genetic diseases. Yi Chuan. 39 (10), 877-887 (2017).
- Rauscher, A. A., Gyimesi, M., Kovacs, M., Malnasi-Csizmadia, A. Targeting Myosin by Blebbistatin Derivatives: Optimization and Pharmacological Potential. Trends in Biochemical Sciences. 43 (9), 700-713 (2018).
- Straight, A. F., et al. Dissecting temporal and spatial control of cytokinesis with a myosin II Inhibitor. Science. 299 (5613), 1743-1747 (2003).
- Sirigu, S., et al. Highly selective inhibition of myosin motors provides the basis of potential therapeutic application. Proceedings of the National Academy of Sciences of the United States of America. 113 (47), E7448-E7455 (2016).
- Green, E. M., et al. A small-molecule inhibitor of sarcomere contractility suppresses hypertrophic cardiomyopathy in mice. Science. 351 (6273), 617-621 (2016).
- Morgan, B. P., et al. Discovery of omecamtiv mecarbil the first, selective, small molecule activator of cardiac Myosin. ACS Medicinal Chemistry Letters. 1 (9), 472-477 (2010).
- Kepiro, M., et al. para-Nitroblebbistatin, the non-cytotoxic and photostable myosin II inhibitor. Angewandte Chemie International Edition. 53 (31), 8211-8215 (2014).
- Varkuti, B. H., et al. A highly soluble, non-phototoxic, non-fluorescent blebbistatin derivative. Scientific Reports. 6, 26141 (2016).
- Verhasselt, S., et al. Discovery of (S)-3'-hydroxyblebbistatin and (S)-3'-aminoblebbistatin: polar myosin II inhibitors with superior research tool properties. Organic and Biomolecular Chemistry. 15 (9), 2104-2118 (2017).
- Verhasselt, S., Roman, B. I., Bracke, M. E., Stevens, C. V. Improved synthesis and comparative analysis of the tool properties of new and existing D-ring modified (S)-blebbistatin analogs. European Journal of Medicinal Chemistry. 136, 85-103 (2017).
- Warren, G. B., Toon, P. A., Birdsall, N. J., Lee, A. G., Metcalfe, J. C. Reconstitution of a calcium pump using defined membrane components. Proceedings of the National Academy of Sciences of the United States of America. 71 (3), 622-626 (1974).
- Kiianitsa, K., Solinger, J. A., Heyer, W. D. Rad54 protein exerts diverse modes of ATPase activity on duplex DNA partially and fully covered with Rad51 protein. Journal of Biological Chemistry. 277 (48), 46205-46215 (2002).
- Hanzelmann, P., Schindelin, H. Structural Basis of ATP Hydrolysis and Intersubunit Signaling in the AAA+ ATPase p97. Structure. 24 (1), 127-139 (2016).
- Hackney, D. D., Jiang, W. Assays for kinesin microtubule-stimulated ATPase activity. Methods in Molecular Biology. 164, 65-71 (2001).
- Kiianitsa, K., Solinger, J. A., Heyer, W. D. NADH-coupled microplate photometric assay for kinetic studies of ATP-hydrolyzing enzymes with low and high specific activities. Analytical Biochemistry. 321 (2), 266-271 (2003).
- Carter, S. G., Karl, D. W. Inorganic phosphate assay with malachite green: an improvement and evaluation. Journal of Biochemical and Biophysical Methods. 7 (1), 7-13 (1982).
- Henkel, R. D., VandeBerg, J. L., Walsh, R. A. A microassay for ATPase. Analytical Biochemistry. 169 (2), 312-318 (1988).
- Rowlands, M. G., et al. High-throughput screening assay for inhibitors of heat-shock protein 90 ATPase activity. Analytical Biochemistry. 327 (2), 176-183 (2004).
- Rule, C. S., Patrick, M., Sandkvist, M. Measuring In Vitro ATPase Activity for Enzymatic Characterization. Journal of Visualized Experiments. (114), 54305 (2016).
- Pardee, J. D., Spudich, J. A. Purification of muscle actin. Methods in Cell Biology. 24, 271-289 (1982).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Kovacs, M., Toth, J., Hetenyi, C., Malnasi-Csizmadia, A., Sellers, J. R. Mechanism of blebbistatin inhibition of myosin II. Chem Journal of Biological Chemistry. 279 (34), 35557-35563 (2004).
- Allingham, J. S., Smith, R., Rayment, I. The structural basis of blebbistatin inhibition and specificity for myosin II. Nature Structural & Molecular Biology. 12 (4), 378-379 (2005).
- Kettlun, A. M., et al. Purification and Characterization of 2 Isoapyrases from Solanum-Tuberosum Var Ultimus. Phytochemistry. 31 (11), 3691-3696 (1992).
- Hulme, E. C., Trevethick, M. A. Ligand binding assays at equilibrium: validation and interpretation. British Journal of Pharmacology. 161 (6), 1219-1237 (2010).
- Motulsky, H. J., Neubig, R. R. Analyzing binding data. Current Protocols in Neuroscience. 52 (1), 7.5.1-7.5.65 (2010).
- Sehgal, P., Olesen, C., Moller, J. V. ATPase Activity Measurements by an Enzyme-Coupled Spectrophotometric Assay. Methods in Molecular Biology. 1377, 105-109 (2016).
- Solinger, J. A., Lutz, G., Sugiyama, T., Kowalczykowski, S. C., Heyer, W. D. Rad54 protein stimulates heteroduplex DNA formation in the synaptic phase of DNA strand exchange via specific interactions with the presynaptic Rad51 nucleoprotein filament. Journal of Molecular Biology. 307 (5), 1207-1221 (2001).
- Banik, U., Roy, S. A continuous fluorimetric assay for ATPase activity. Biochemistry Journal. 266 (2), 611-614 (1990).
- Xiao, Y. X., Yang, W. X. KIFC1: a promising chemotherapy target for cancer treatment?. Oncotarget. 7 (30), 48656-48670 (2016).
- See, S. K., et al. Cytoplasmic Dynein Antagonists with Improved Potency and Isoform Selectivity. ACS Chemical Biology. 11 (1), 53-60 (2016).
- Datta, A., Brosh, R. M. New Insights Into DNA Helicases as Druggable Targets for Cancer Therapy. Frontiers in Molecular Biosciences. 5, 59 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon