Method Article
基于生物传感器的高通量生物跨越和生物信息学分析战略,用于全球药物-蛋白质相互作用验证
摘要
这项研究旨在提出一个确定药物肽相互作用的战略。该战略涉及基于石英晶体微平衡(QCM)生物传感器的药物识别短肽的生物跨越,然后进行生物信息学分析,以定量评估为蛋白质上药物识别和药物结合位点注释而获得的信息。
摘要
受体和酶蛋白是重要的生物分子,是生物活性小分子的结合靶点。因此,快速和全球验证药物-蛋白质相互作用是非常可取的,不仅用于了解治疗疗效背后的分子机制,而且可用于评估药物特性,如吸附、分布、新陈代谢、排泄和用于临床的毒性(ADMET)。在这里,我们提出了一个基于生物传感器的高通量策略,用于T7噬菌体显示的短肽的生物跨越,可以很容易地显示在噬菌体上。随后分析含有短段的肽的氨基酸序列,作为"破碎的遗物",在受体配体接触(RELIC)套件中使用生物信息学程序的药物结合部位。本文阐述了将这种方法应用于两种临床批准的药物,一种抗肿瘤的伊里诺特坎和一种抗流感的奥塞塔米韦,从而解释了收集药物识别肽序列和突出目标蛋白的药物结合位点的详细过程。此处描述的策略可用于任何感兴趣的小分子。
引言
确定药物结合靶点对于药物的发展以及了解疾病的分子机制至关重要。特别是受体和酶蛋白是生物活性小分子最重要的分子靶点1。虽然亲和力捕获是识别药物结合蛋白2的成熟技术,但技术限制,如蛋白质的低溶解性,往往阻碍药物靶点2的验证。最重要的是,固定的小分子失去了对接所需的自由度,并且可能无法进入位于内部的结合位点对较大的目标蛋白质。此外,蛋白质错折叠、无法分析共同结晶条件以及分子大小的限制往往妨碍使用X射线晶体学、核磁共振(NMR)和其他此类实验分析来研究药物-蛋白质相互作用。
使用T7噬菌体显示生物平移是确定小分子诱饵3蛋白质结合位点的有效方法。特别是,T7噬菌体显示的随机肽库,可以通过将合成DNA插入多克隆站点来构建,是有效的。与显示蛋白质的T7噬菌体库相比,短肽可以很容易地设计成在没有物理限制的情况下显示在T7噬菌体上,它可以与固定在固体支撑2上的任何小分子药物进行绝育接触。此外,在T7噬菌体显示生物跨越平台中引入石英晶体微平衡(QCM)生物传感器,通过监测QCM频率4、5的降低,可以识别短肽与药物的这种微弱但具体的相互作用。然后,通过感染宿主大肠杆菌(BLT5615) 直接恢复绑定 T7 噬菌体,并确定编码亲和力选择肽的区域的 DNA 序列,该肽含有药物识别短段。随后对肽群体氨基酸序列的分析提供了有关药物识别的信息。在西里科对齐中,获救的氨基酸序列可用于获取有关所选蛋白质组内药物的生物靶点的信息。这种对药物具有亲和力的蛋白质片段的高通量鉴定,可以用来启发性地重建药物结合的部位,其方式类似于从陶器碎片6中重建古代文物。特别是,当传统的蛋白组学方法失败时,这种独特的方法是有用的。
在这里,我们提出了一个基于生物传感器的策略,用于T7噬菌体显示肽的生物跨越和生物信息学分析,以实现小分子的目标验证。除了对传统方法的技术限制外,该策略还能够根据相同的协议识别目标蛋白上任何感兴趣的小分子的药物结合位点。
研究方案
注:以下是使用QCM生物传感器筛查药物识别T7噬菌体并通过大肠杆菌(BLT5615)感染恢复筛查噬菌体的步骤。合成形成自组装单层(SAM)的小分子衍生物和建造T7噬菌体显示的15-mer随机肽库的协议可以在其他地方找到6,7。
1. QCM传感器芯片的准备
- 在 27-MHz QCM 仪器的振荡器上安装陶瓷传感器芯片,并在小分子固定之前记录空气相中的内在频率 (Hz)。
- 分离芯片,将一种小分子衍生物的溶液(70% 乙醇中的 1 mM)滴入 20 μL,该衍生物使用移液器将 SAM 放到传感器芯片的金电极上。
警告:金电极(Au,0.1毫米厚,2.5毫米,即4.9毫米2)所在的传感器芯片晶体非常薄,容易破裂(SiO 2,0.06毫米厚,9毫米 i.d.)。因此,移液器小心。 - 在室温下(约20°C)在培养皿中离开1小时,盘中带有湿润的组织,不受室灯的遮挡。
- 用超纯净水轻轻清洗电极表面:然后,用注射器或空气除尘器吹空气,去除水滴。
- 为 QCM 仪器设置传感器芯片,并记录空气相中频率的降低,以测量已固定的小分子的量。
注:至少,100 Hz的内在频率是成功的小分子固定(1 Hz固定30 pg)的必要条件。
2. 使用QCM生物传感器的T7噬菌体库的生物跨越(图1)
- 在 QCM 生物传感器上设置带有专用磁搅拌器的 cuvette,并将 8 mL 的反应缓冲器(10 mM Tris-HCl、200 mM NaCl、pH 7-8)倒入 cuvette 中。
- 将 QCM 传感器芯片连接到振荡器上,然后拉下振荡器的手臂,将芯片浸入以 1000 rpm 的速度搅拌的缓冲器中。
- 开始监控个人计算机 (PC) 上的 QCM 频率,并等待传感器图平衡到频率漂移的 3 Hz/min 左右。
- 将 T7 噬菌体库 (1- 2 × 1010 pfu/mL) 的 8 μL 注入 cuvette(最终浓度:1 -2 × 107 pfu/mL),并在传感器上标记注射点。
- 监测T7噬菌体与金电极表面固定的小分子的结合导致的频率降低10分钟。
- 停止 QCM 频率监视器,将传感器芯片从振荡器中移开,并通过吹空气和/或用湿巾擦去缓冲区。
- 将传感器芯片放入湿润的培养皿中,将 大肠杆菌 (BLT5615) 悬架的 20 微升(OD600 = 0.5-1.0)在 37 °C 下摇晃后,将日志相中的 IPTG 添加到 1mM)主机单元到金电极上。
- 合上盘子的盖子,用铝箔盖住,挡住光线。
- 在 96 井微板搅拌机 (1000-1500 rpm) 上以 37 °C 孵化菜 30 分钟,以增强绑定 T7 噬菌体的恢复。
- 恢复解决方案的 20 μL,并将其悬挂到 200 μL 的 LB 介质中。
注:在此步骤中获得的样本可在一周内保存在 4 °C。 - 根据制造商说明书 8、9中描述的一般程序,准备一系列噬菌体溶液的稀释系列,用于斑块分离和 DNA 测序。
- 用浸泡有1%钠硫酸钠溶液的棉签擦拭金电极表面。
- 用洗涤瓶中的超纯水清洗金表面,然后用注射器或空气除尘器吹空气以去除水滴。
- 将 5 μL 的食人鱼溶液 (Conc. H2SO4: 30% H2O2 = 3:1) 放在金色表面上, 离开 5 分钟。
- 用水再次清洗金表面,然后用湿巾吹空气和/或擦拭干燥。
- 重复步骤 2.14 和 2.15。
注意:在使用前立即准备食人鱼溶液。小心使用这种液体,因为它是一种非常强的酸。治疗时间超过5分钟会侵蚀传感器芯片。
3. 生物信息学分析使用受体利甘接触 (RELIC) 程序套件 (图 2)10,11
- 使用 MS Windows 操作系统在 PC 上解控独立 RELIC 程序。
- 对齐使用药物选择的 15 美人肽亲和力的氨基酸序列,或在每个文本格式文件(名称.txt)中从未筛选的父库中随机选择。
- 使用 FASTA 格式在每个文本文件中键入单个或多个蛋白质的氨基酸序列,或从任何蛋白质数据库(例如 UniProt (http://www.uniprot.org/) 或药物银行 (https://www.drugbank.ca/) 以 FASTA 格式下载数据库文本文件。
- 将文本文件(和用于 HETERO 对齐的 PDB 文件)放在运行每个 RELIC 程序所需的文件夹中。
- 单击独立文件夹中的 AADIV、INFO、MOTIF、匹配、异地对齐、快速分析和 FASTAskan 的可执行文件(程序.exe),打开 FTN95 的个人版本。
- 在命令消息中键入适当的文件名以及扩展(名称.txt),以执行每个程序并获取所需的文本格式文件。
- 将生成的文本文件导出到电子表格软件(例如 Excel),以生成使用 BLOSUM62 计算的信息内容图 (INFO) 或累积相似性分数(匹配、HETERO 对齐)。
注:原始RELIC服务器(http://relic.bio.anl.gov)不再可用,一些独立类型的RELIC程序,工作在PC上与Windows平台可以从相应的作者(tkksg@rs.noda.tus.ac.jp)获得。
结果
两种临床批准药物的代表性结果见图3。Irinotecan (图 3A),一种用于治疗晚期结直肠癌和非小细胞肺癌的天然胰腺素水溶性前列腺素, 在肝脏中转化为 SN-38, 抑制癌细胞12中的托莫索酶 I .此外,这种化合物直接抑制乙酰胆碱酯酶(AChE)13,14。通过该战略,QCM基于单周期生物跨度子集确定了29种多肽,这些肽将Iri固定为SAM。随后,29肽和AChE的对齐为Y121、Q225、F290、E327、H440和Y442带来了最高分,并突出了三维结构中的部分。这些氨基酸残留物与组成 ACHE 的 Iri 结合位点的残留物一致。在催化三合会(S221、E353和H467)附近的催化三合会(S221、E353和H467)中成功识别出E99、L100、L252、L305、L305、I387和V474的同一子集,表明这些氨基酸在Iri15的去灭过程中构成了伊里识别的脚手架。催化部位的这种氨基酸残留物不能直接使用传统的X射线晶体学或NMR分析来识别,因为酶反应进行得很顺利,在一般实验条件下不会稳定地形成静态复合物。因此,使用为一种药物确定的亲和力选择肽,可以组合检测多种蛋白质的药物结合位点,包括可能与特定药物代谢反应期间可能与酶形成的中间复合物中的位点。
图3B 显示了为奥塞尔塔米韦(Osel)获得的其他结果,奥塞尔是一种抗流感药物,被激活到奥塞尔塔米韦卡博基拉特,这反过来又抑制了流感病毒16的神经酰胺酶(NA)。识别覆盖QCM传感器芯片金电极表面的27种肽成功地探测到了NA16中的奥塞尔结合位点。此绑定站点由非结构化肽循环组成,在与 Osel 对接时可能会经历动态运动。T7 噬菌体上的 Osel 识别肽在与 QCM 传感器芯片的黄金电极表面固定的 Osel 结合时可能会模仿这种动态对接。在患有流感的年轻患者中,神经精神病学不良事件(NPAEs)已被识别出来,这对患者的影响可能与疾病本身有关,而不是与药物有关。研究表明,Osel通过血脑屏障(BBB)17中的多药耐药性(MDR)蛋白从啮齿动物的中枢神经系统(CNS)中积极出口。事实上,在我们的研究中,除了其他运输剂、神经递质相关酶和受体外,该类MDR的蛋白质之一也显示出高分(在药物银行1.0蛋白质数据库18中,4,396种蛋白质中占前5%)。这些蛋白质对奥塞尔不良反应外观的药理学意义正在调查中。
到目前为止,靶蛋白上的单小分子结合位点和多个小分子结合位点已经成功识别出六种小分子药物,这些药物一直在使用我们的策略(图4)。对于Brz2001和罗西霉素(RXM),使用不同的肽池、数字和氨基酸序列来识别目标蛋白上相同的药物结合位点,其数量和氨基酸序列完全不同。此外,为 Iri、RXM 和 Osel 获得的单个肽池导致识别出每种药物不同蛋白质的多个结合位点,如 Iri 的 AChE 和 CE(图 3A)20、RXM19、21的血管素和 CYP3A4,以及 Osel 的 NA 和 MDR 相关蛋白质。一个未知的分子靶点被确定为抗肿瘤化合物多克索鲁比辛(FANCF)22,和抗血管大滑剂抗生素RXM(血管素)19。
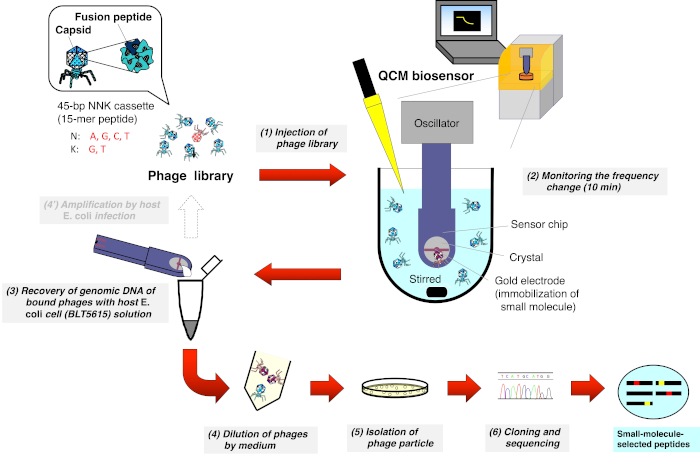
图1:T7噬菌体显示肽库QCM生物传感器生物跨越的示意图表示。显示随机肽的T7噬菌体库被注射到含有缓冲区(搅拌下)的cuvette中,其中QCM生物传感器芯片浸入其中,频率稳定。在监测由于T7噬菌体与传感器芯片的金电极表面固定的小分子结合而降低频率后,传感器芯片与振荡器分离。然后,在宿主大肠杆菌(BLT5615)感染后,从绑定T7噬菌体中直接恢复DNA。由此产生的T7噬菌体通过斑块形成进行分离,最后,T7噬菌体上显示的药物亲和力选择肽的氨基酸序列根据一般噬菌体显示方法确定。请点击这里查看此数字的较大版本。
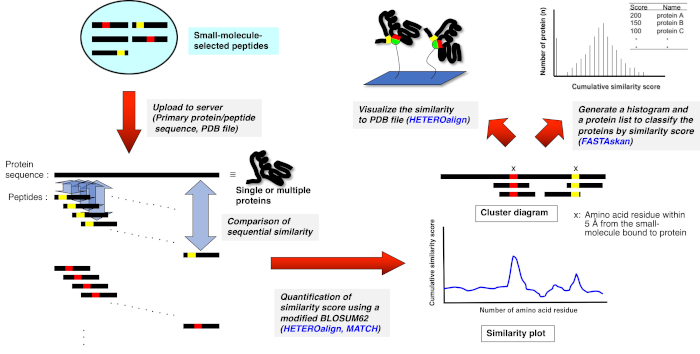
图2: 药物选择肽与单一或多重蛋白质序列比较的定量评估的示意图表示。药物选择的肽序列分别与单一和多重蛋白质的原氨基酸序列对齐,并且根据经过修改的 BLOSUM62 矩阵通过对齐累计评分每 3 -5 个氨基酸集的相似性。由此产生的图或图表示构成蛋白质上潜在药物结合部位的残留物或部分。使用适当的RELIC程序进行进一步分析,突出显示三维结构上的绑定位点(如果有 PDB 文件),或对可能是绑定目标的整个蛋白质进行排名(HETERO 对齐程序当前不可用)。 请点击这里查看此数字的较大版本。
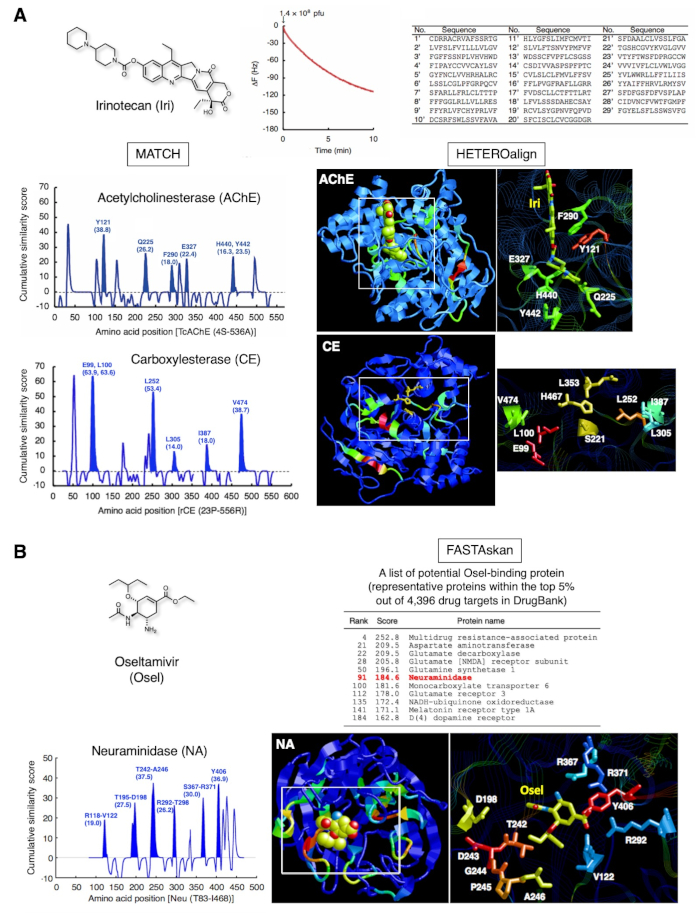
图3:肽收集及后续生物信息学分析的代表性结果。(A) 伊里诺特坎 (抗肿瘤前列腺药物, 托波索酶 I 抑制剂).T7噬菌体交互被监测10分钟,以减少QCM频率。被绑定T7噬菌体的DNA被恢复并测序,以确定相应的氨基酸序列。使用单周期生物跨度子集收集的 29 个 15 mer 肽的氨基酸序列突出了构成 ACHE [PDB ID 1U65] 的 Iri 结合位点的氨基酸。使用相同的 29 肽进行进一步评估,突出了 CE 催化三合会的邻近氨基酸残留物(去除灭菌的脚手架残留物),这是一种将 Iri 转换为 SN-38(活动形式)的肝脏酶 [PDB ID: 1K4Y]。从未筛选的父库 7 中随机选择的 103 个肽的相似性分数7,从这些分数中减去19个,以消除库偏差。这些数字转载自参考20,并经埃尔塞维尔许可。(B)奥塞塔米韦(抗流感药物)。含有奥塞尔识别氨基酸的27种肽突出了新氨基酶(NA)(病毒酶)的奥塞尔结合部位的无序部分[PDB ID:2HT7]。对药物银行1.018中27种肽和4,396种蛋白质的序列相似性的全球验证显示,NA在前5%范围内,此外还有与中枢神经系统功能相关的宿主人类蛋白质。请点击这里查看此数字的较大版本。
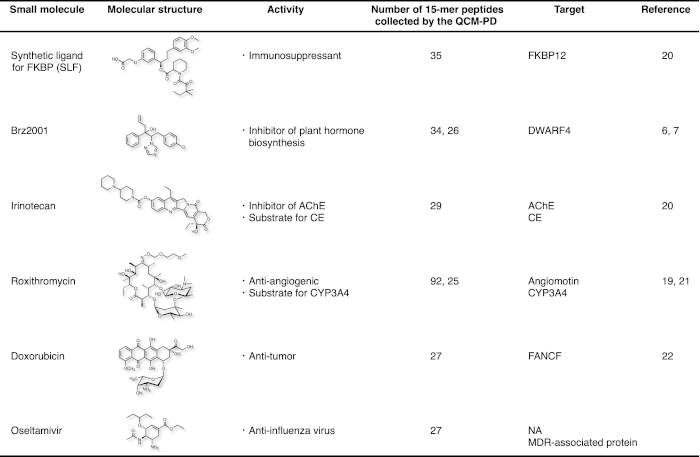
图4:小分子的总结,小分子的结合目标是利用这一策略确定的。请点击这里查看此数字的较大版本。
讨论
在这里,提出了一个基于QCM生物传感器的药物识别肽生物跨度策略,随后是利用已识别的肽验证药物-蛋白质相互作用的生物信息学分析。在生物传感器的金电极上设计用于固定的小分子衍生物是一个重要步骤,因为引入的链接器可能会阻碍识别药物的肽的结合和收集。为了避免这种情况,引入链接器不同位置的衍生工具准备了23个。或者,为了固定疏水小分子,传感器芯片浸入10厘米的培养皿中的散装水中,小分子的20 μL溶液(二甲基硫化物中的10mM溶液)被丢弃在生物传感器的金电极上,覆盖其表面,并孵育5分钟。这允许保留小分子的亚百赫兹内在频率,在生物跨越期间保持至少10分钟。事实上,使用这种固定,奥塞尔亲和力选择肽清楚地突出了在NA的奥塞尔绑定网站(图3)。
T7噬菌体用于准备肽库在这里是基因工程使用NNK15盒式,编码32个鳕鱼的所有20个标准氨基酸,并抑制出现2停止科顿(UAA,UGA),只出现UAG(图1)6,7。这对展示 15mer 全长肽和增加库的多样性非常重要。T7噬菌体显示系统的技术显示限制为107-109 T7噬菌体。然而,15mer肽库的多样性理论上是20 15(3.27×1019):因此,它不能用于完整的图书馆建设。然而,相似性搜索或挖掘保存的图案允许检测氨基酸,包括蛋白质的药物结合位点,即使这种有限的多肽多样性在图书馆。此外,3-5氨基酸延伸在库肽(外观速率在1/203和1/205之间,这可以通过T7噬菌体显示系统实现)参与小分子药物的识别:因此,不需要将肽序列与构成靶蛋白药物结合部位的15-mer氨基酸序列的100%匹配。事实上,大约30种亲和力选择的肽成功地突出了每个测试药物的目标蛋白的结合位点(图4)。因此,使用父 T7 噬菌体库(1.7 × 107 pfu/mL)的多样性可用于启发式地重建药物绑定站点。
通常,3-5份T7噬菌体显示的序列与含有药物识别氨基酸延伸的15-mer氨基酸序列的序列相同,这些序列出现在任意隔离的16块斑块内,表明根据我们的协议选择亲和力是成功的。这表明,在隔离的96个斑块(数字与微板格式相关)中收集了18-30个不同的药物识别肽序列,这些序列随后通过DNA测序和获得相应的氨基酸序列进行识别。在目前的策略中,将 8 μL 的 T7 噬菌体库注入包含 8 mL 缓冲区(库的稀释 1000 倍)的 cuvette 适合减少 T7 噬菌体的非特定绑定。为了增加亲和力选择肽的多样性,重复多次单周期选择,每次筛查使用 16 或 32 个斑块隔离,证明比一次从单个溶液中分离更有效。例如,为了有效地收集大约30种不同顺序的亲和力选择肽,进行了3-6套单周期选择,在每个实验中分离了16或32个斑块。所有 16 或 32 个 T7 噬菌体斑块中相同序列的外观表示意外检测背景或可能作为结转污染。相比之下,许多肽比15毫米长度短的T7噬菌体没有相同序列或外观的T7噬菌体,这表明T7噬菌体在人群中非特异性出现的可能性很高。由于QCM频率降低即使在此类情况下也发生到同样的程度,因此应通过对分离的T7噬菌体的DNA进行测序,然后对肽的氨基酸序列进行生物信息学分析,对选择的成功率进行全面评估。此外,与传统的T7噬菌体显示程序不同,重复轮选择效果较差,因为T7噬菌体的变化和数量很小,即使在重复放大和选择步骤23后也不会集中。
重要的是,这种方法适用于在人类蛋白质组、致病病毒甚至植物中挖掘小分子结合位点。有趣的是,T7噬菌体上可能非结构化的多肽短显示长度可以模仿蛋白质肽在与小分子对接时的分子动力学:这可以反映动态绑定24。除了传统方法的技术局限性外,这种适用于小分子相同方案的策略可能会扩大可药蛋白质组,并提供有关药物-蛋白质相互作用分析的更多粒度。
应考虑这种方法的某些技术局限性。有机合成是生物传感器芯片金电极表面小分子固定的必要条件。对于有机化学的非专家,一些固定试剂在商业上可用于通过耦合机械修复小分子。此外,肽的某些无稽之谈部分可能导致检测出部分蛋白质与药物对接无关,因为假阳性。这在经验上与富含白氨酸或血管残留物的β-表或富含白氨酸的域相对应,当T7噬菌体的副本被编码时,这些领域被编码的鳕鱼比其他标准氨基酸多。控制库肽长度可能会控制误报的发生。相比之下,可能有些情况下,未检测到用于对接的药物结合部位中的氨基酸残留物,如使用 X 射线晶体学或 NMR 分析所示。这可以通过收集更多的药物识别肽或改变金电极上小分子固定的方向来解决。
许多与药物使用的主要和次要影响有关的药物-蛋白质相互作用在蛋白质组中可能尚未确定:此外,负责药物吸收、分配、新陈代谢、排泄和毒性的酶和运输剂也可能仍然不明。蛋白质结合并不总是对药物的生物活性负责。因此,结合生物检测的其他信息,将改善对药物的主要和不良影响负责的基本药物靶点的识别。进一步适应这种简洁的技术将提高挖掘各种小分子药物的蛋白质结合位点的实用性和通量。本文介绍的方法不仅有助于开展相关领域的基础研究,而且有助于阐明药物在临床应用中疗效或其他生物效应的分子机制。
披露声明
作者没有利益冲突可以披露。
致谢
作者感谢河野洋平博士和石川海藤博士提供奥司他韦,以及李·马科夫斯基博士提供独立的RELIC项目。作者还感谢川口泰子为QCM实验提供技术援助。这项工作得到了JSPS卡肯希赠款编号17K01363(Y.T.)的部分支持。
数据可用性声明
独立RELIC程序和药物亲和力选择肽的序列数据,以及本文中使用的蛋白质组数据库中的蛋白质序列,可应作者的要求(tkksg@rs.noda.tus.ac.jp)获得。
材料
| Name | Company | Catalog Number | Comments |
| AFFINIXQN | ULVAC, Inc. (Tokyo, Japan) | QCM2008-STKIT | Contains Glass cuvette, stir magnet, operation and analysis software with a Windows PC |
| AADIV | Northeastern University (Lee Makowski) | AADIV.exe | Calculates the frequency of occurrence of each of the 20 amino acids at each recombinant insert position, as well as the overall position-independent frequency of each amino acid within that set of peptide sequences. Also roughly estimates the sequence diversity of a display library by statistical sampling method based upon sequences obtained from a limited number of randomly sampled members of the library. |
| Ceramic Sensor Chip | ULVAC, Inc. (Tokyo, Japan) | QCMST27C | 4 sensor chips/package |
| Dimethyl sulfoxide | Sigma-Aldrich (St. Louis, MO, USA) | D8418 | |
| Ethanol | Merck (Kenilworth, NJ, USA) | 09-0850 | |
| FASTAcon | Northeastern University (Lee Makowski) | FASTAcon.exe | Identifies proteins from a population with short consensus sequences. |
| FASTAskan | Northeastern University (Lee Makowski) | FASTAskan.exe | Lists proteins with high similarity to a peptide population. |
| Immiblization kit for AFFINIX | ULVAC, Inc. (Tokyo, Japan) | QCMIMKT | SAM reagent and amine coupling reagent |
| INFO | Northeastern University (Lee Makowski) | INFO.exe | Provides mathematical measure of the probability of observing a particular peptide sequence by random chance (i.e., nonspecific binding) as opposed to by selection for a specific property (affinity to small molecule). |
| Liquid LB medium | Sigma-Aldrich (St. Louis, MO, USA) | L3522 | Autoclave for 20 min |
| MATCH | Northeastern University (Lee Makowski) | MATCH.exe | Identifies any stretches of amino acid residues within a particular protein that exhibit significant similarity to a group of affinity-selected peptides. Outputs as cluster dia- gram and cumulative similarity plot calculated from a modified BLOSUM62 matrix with a short window (5–6 amino acids in length). |
| MOTIF1 | Northeastern University (Lee Makowski) | MOTIF1.exe | Searches for three continuous amino acid sequence motifs within a peptide population. |
| MOTIF2 | Northeastern University (Lee Makowski) | MOTIF2.exe | Searches for patterns of three amino acids and does not allow conservative amino acid substitutions, but does allow identical gap lengths. |
| NaCl | Merck (Kenilworth, NJ, USA) | S3014 | |
| Receptor ligand contacts (RELIC) | Argonne National Laboratory (Lemont, IL, USA) | https://www.relic.anl.gov | Currently unavailable (Stand-alone program can be used from correspondence author upon request) |
| Tris | Merck (Kenilworth, NJ, USA) | 252859 |
参考文献
- Santos, R., et al. A comprehensive map of molecular drug targets. Nature Reviews Drug Discovery. 16 (1), 19-34 (2017).
- Ziegler, S., Pries, V., Hedberg, C., Waldmann, H. Target identification for small bioactive molecules: finding the needle in the haystack. Angewandte Chemie International Edition (English). 52 (10), 2744-2792 (2013).
- Piggott, A. M., Karuso, P. Identifying the cellular targets of natural products using T7 phage display. Natural Product Reports. 33 (5), 626-636 (2016).
- Takakusagi, Y., Takakusagi, K., Sakaguchi, K., Sugawara, F. Phage display technology for target determination of small-molecule therapeutics: an update. Expert Opinion on Drug Discovery. 15 (10), 1199-1211 (2020).
- Takakusagi, Y., Takakusagi, K., Sugawara, F., Sakaguchi, K. Use of phage display technology for the determination of the targets for small-molecule therapeutics. Expert Opinion on Drug Discovery. 5 (4), 361-389 (2010).
- Takakusagi, Y., Takakusagi, K., Sugawara, F., Sakaguchi, K. Using the QCM Biosensor-Based T7 Phage Display Combined with Bioinformatics Analysis for Target Identification of Bioactive Small Molecule. Methods in Molecular Biology. 1795, 159-172 (2018).
- Takakusagi, Y., et al. Mapping a disordered portion of the Brz2001-binding site on a plant monooxygenase, DWARF4, using a quartz-crystal microbalance biosensor-based T7 phage display. ASSAY and Drug Devevelopment Technologies. 11 (3), 206-215 (2013).
- Novagen. T7 Select® System Manual. Novagen. , TB178 1009JN (2009).
- Novagen. OrientExpressTM cDNA Manual. Novagen. , TB247 1109JN (2009).
- Mandava, S., Makowski, L., Devarapalli, S., Uzubell, J., Rodi, D. J. RELIC--a bioinformatics server for combinatorial peptide analysis and identification of protein-ligand interaction sites. Proteomics. 4 (5), 1439-1460 (2004).
- Makowski, L. Phage Nanobiotechnology. Petrenko, V. A., Smith, G. P. , RSC Publishing. Ch. 3 33-54 (2011).
- Garcia-Carbonero, R., Supko, J. G. Current perspectives on the clinical experience, pharmacology, and continued development of the camptothecins. Clinical Cancer Research. 8 (3), 641-661 (2002).
- Harel, M., et al. The crystal structure of the complex of the anticancer prodrug 7-ethyl-10-[4-(1-piperidino)-1-piperidino]-carbonyloxycamptothecin (CPT-11) with Torpedo californica acetylcholinesterase provides a molecular explanation for its cholinergic action. Molecular Pharmacology. 67 (6), 1874-1881 (2005).
- Dodds, H. M., Rivory, L. P. The mechanism for the inhibition of acetylcholinesterases by irinotecan (CPT-11). Molecular Pharmacology. 56 (6), 1346-1353 (1999).
- Bencharit, S., et al. Structural insights into CPT-11 activation by mammalian carboxylesterases. Nature Structural Biology. 9 (5), 337-342 (2002).
- Kim, C. U., et al. Influenza neuraminidase inhibitors possessing a novel hydrophobic interaction in the enzyme active site: design, synthesis, and structural analysis of carbocyclic sialic acid analogues with potent anti-influenza activity. Journal of the American Chemical Society. 119 (4), 681-690 (1997).
- Hoffmann, G., et al. Nonclinical pharmacokinetics of oseltamivir and oseltamivir carboxylate in the central nervous system. Antimicrobial Agents and Chemotherapy. 53 (11), 4753-4761 (2009).
- Wishart, D. S., et al. DrugBank 5.0: a major update to the DrugBank database for 2018. Nucleic Acids Research. 46 (1), 1074-1082 (2018).
- Takakusagi, K., et al. Multimodal biopanning of T7 phage-displayed peptides reveals angiomotin as a potential receptor of the anti-angiogenic macrolide Roxithromycin. European Journal of Medicinal Chemistry. 90, 809-821 (2015).
- Takakusagi, Y., et al. Efficient one-cycle affinity selection of binding proteins or peptides specific for a small-molecule using a T7 phage display pool. Bioorganic and Medicinal Chemistry. 16 (22), 9837-9846 (2008).
- Takakusagi, Y., Suzuki, A., Sugawara, F., Kobayashi, S., Sakaguchi, K. Self-assembled monolayer (SAM) of small organic molecule for efficient random-peptide phage display selection using a cuvette type quartz-crystal micobalance (QCM) device. World Journal of Engineering. 5, 1005-1006 (2009).
- Kusayanagi, T., et al. The antitumor agent doxorubicin binds to Fanconi anemia group F protein. Bioorganic and Medicinal Chemistry. 20 (21), 6248-6255 (2012).
- Takakusagi, Y., et al. Identification of C10 biotinylated camptothecin (CPT-10-B) binding peptides using T7 phage display screen on a QCM device. Bioorganic and Medicinal Chemistry. 15 (24), 7590-7598 (2007).
- Rodi, D. J., et al. Identification of small molecule binding sites within proteins using phage display technology. Combinatorial Chemistry and High Throughput Screening. 4 (7), 553-572 (2001).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。