Method Article
Biosensor-basierte Hochdurchsatz-Biopanning- und Bioinformatik-Analysestrategie zur globalen Validierung von Wechselwirkungen zwischen Arzneimittel und Proteinen
In diesem Artikel
Zusammenfassung
Diese Studie zielte darauf ab, eine Strategie zur Identifizierung von Wechselwirkungen zwischen Medikamenten und Peptid zu präsentieren. Die Strategie umfasst die Biopanierung von medikamentenerkennenden kurzinomischen Kurzpeptiden auf der Grundlage eines Mikrobilanzsensors (QCM) in Quarzkristall, gefolgt von einer Bioinformatik-Analyse zur quantitativen Bewertung der für die Arzneimittelerkennung und Anmerkung der Arzneimittelbindungsstellen auf Proteinen erhaltenen Informationen.
Zusammenfassung
Rezeptoren und Enzymproteine sind wichtige Biomoleküle, die als Bindungsziele für bioaktive kleine Moleküle fungieren. Daher ist die schnelle und globale Validierung der Wechselwirkungen zwischen Arzneimittel und Protein enden sehr wünschenswert, um nicht nur die molekularen Mechanismen zu verstehen, die der therapeutischen Wirksamkeit zugrunde liegen, sondern auch für die Bewertung von Arzneimittelmerkmalen wie Adsorption, Verteilung, Stoffwechsel, Ausscheidung und Toxizität (ADMET) für den klinischen Einsatz. Hier stellen wir eine biosensorbasierte Hochdurchsatzstrategie für die Biopannierung von T7-Phagen-angezeigten kurzen Peptiden vor, die leicht auf dem Phagendeckel angezeigt werden können. Eine nachträgliche Analyse der Aminosäuresequenzen von Peptiden, die kurze Segmente enthalten, als "gebrochene Relikte", der arzneimittelbindenden Stellen mit Bioinformatik-Programmen in der Rezeptligaand-Kontakt-Suite (RELIC) gezeigt. Durch die Anwendung dieser Methode auf zwei klinisch zugelassene Medikamente, einen Anti-Tumor-Irinotecan und ein Anti-Grippe-Oseltamivir, wird der detaillierte Prozess zum Sammeln der arzneimittelerkennenden Peptidsequenzen und zur Hervorhebung der wirkstoffbindenden Stellen der Zielproteine in diesem Papier erläutert. Die hier beschriebene Strategie kann für alle kleinen Moleküle von Interesse angewendet werden.
Einleitung
Die Identifizierung von medikamentenbindenden Zielen ist ein wesentlicher Bestandteil für die Entwicklung von Medikamenten sowie für das Verständnis der molekularen Mechanismen von Krankheiten. Insbesondere Rezeptor- und Enzymproteine sind die wichtigsten molekularen Ziele bioaktiver kleiner Moleküle1. Obwohl Affinitätserfassung eine etablierte Technik zur Identifizierung der arzneimittelbindenden Proteine2ist, behindern technische Einschränkungen, wie z. B. die geringe Löslichkeit von Proteinen, oft die Validierung von Wirkstoffzielen2. Am wichtigsten ist, dass die immobilisierten kleinen Moleküle den für das Andocken notwendigen Freiheitsgrad verlieren und für die intern lokalisierten Bindungsstellen auf größeren Zielproteinen unzugänglich sein können. Darüber hinaus behindern Proteinfehlfaltung, Unfähigkeit, Kokristallisationsbedingungen zu analysieren, und Einschränkungen aufgrund molekularer Größe häufig die Verwendung von Röntgenkristallographie, Kernspinresonanz (NMR) und anderen solchen experimentellen Analysen zur Untersuchung von Wechselwirkungen zwischen Medikamenten und Proteinen.
Die Verwendung des T7 Phagen-Display-Biopannings ist eine effiziente Möglichkeit, die Bindungsstelle auf Proteinen für kleine Molekülköder zu bestimmen3. Insbesondere eine vom T7-Phagen angezeigte zufällige Peptidbibliothek, die durch Einsetzen synthetischer DNA in eine Multiklonierungsstelle aufgebaut werden kann, ist wirksam. Im Vergleich zur T7-Phagenbibliothek, die Proteine anzeigt, können die kurzen Peptide ohne physikalische Einschränkungen einfach auf dem T7-Phagenkapsid angezeigt werden, das sterisch mit jedem kleinen Molekül-Medikament in Berührung kommen kann, das auf einer festen Stütze fixiert ist2. Darüber hinaus ermöglicht die Einführung eines Quarz-Kristall-Mikrobilanz-Biosensors (QCM) in die Biopanning-Plattform t7-Phagendisplay die Identifizierung solcher schwachen, aber spezifischen Wechselwirkungen von kurzen Peptiden mit Medikamenten durch Überwachung der Reduzierung der QCM-Frequenz4,5. Der gebundene T7-Phagen wird dann direkt durch Infizierung des Wirts Escherichia coli (BLT5615) wiederhergestellt, und die DNA-Sequenz der Region, die das affinitätsausgewählte Peptid kodiert, das drogenerkennende Kurzsegmente beherbergt, wird bestimmt. Die nachfolgende Analyse der Aminosäuresequenz der Peptidpopulation liefert Informationen zur Arzneimittelerkennung. In silico paarweise Ausrichtung der geretteten AminosäureSequenzen können verwendet werden, um Informationen über das biologische Ziel des Medikaments innerhalb eines ausgewählten Proteomzuerhaltens zu erhalten. Diese hohe Durchsatzidentifikation von Proteinfragmenten mit Affinität zu einem Medikament kann verwendet werden, um die drogenbindende Stelle heuristisch zu rekonstruieren, ähnlich wie die Rekonstruktion eines alten Artefakts aus Keramikscherben6. Insbesondere kann dieser einzigartige Ansatz nützlich sein, wenn herkömmliche Proteomik-Ansätze fehlschlagen.
Hier stellen wir eine biosensorbasierte Strategie zur Biopannierung von T7-Phagen-Peptiden und Bioinformatik-Analysen zur Zielvalidierung der kleinen Moleküle vor. Über die technischen Beschränkungen konventioneller Methoden hinaus ermöglicht diese Strategie die Identifizierung von Wirkstoffbindungsstellen auf Zielproteinen für jedes kleine Molekül von Interesse nach dem identischen Protokoll.
Protokoll
HINWEIS: Im Folgenden sind die Schritte zum Screening von T7-Phagen mit Hilfe eines QCM-Biosensors und zur Wiederherstellung der gescreenten Phagen mittels E. coli -Infektion (BLT5615). Die Protokolle für die Synthese eines Derivats eines kleinen Moleküls, das eine selbst zusammengesetzte Monoschicht (SAM) bildet, und für den Aufbau der T7-Phagen-angezeigten 15-mer-Zufallspeptidbibliothek finden Sie an anderer Stelle6,7.
1. Vorbereitung des QCM-Sensorchips
- Befestigen Sie einen Keramik-Sensorchip am Oszillator eines 27-MHz-QCM-Geräts und zeichnen Sie die Eigenfrequenz (Hz) in der Luftphase vor der kleinen Molekülimmobilisierung auf.
- Lösen Sie den Chip und lassen Sie 20 l einer Lösung (1 mM in 70% Ethanol) eines kleinen Molekülderivats, das SAM bildet, mit einer Pipette auf die Goldelektrode des Sensorchips fallen.
VORSICHT: Der Sensorchip-Kristall, in dem sich die Goldelektrode (Au, 0,1 mm dick, 2,5 mm i.d., 4,9 mm2) befindet, ist extrem dünn und kann leicht reißen (SiO2, 0,06 mm dick, 9 mm i.d.). Daher Pipette sorgfältig. - 1 h bei Raumtemperatur (ca. 20 °C) in einer Petrischale mit befeuchteten Gewebe und vor Raumbeleuchtung abgeschirmt lassen.
- Waschen Sie die Elektrodenoberfläche sanft mit reinem Wasser; entfernen Sie dann die Wassertropfen, indem Sie Luft mit einer Spritze oder einem Luftstaubsauger blasen.
- Richten Sie den Sensorchip für das QCM-Gerät ein und zeichnen Sie die Frequenzreduzierung in der Luftphase auf, um die Menge des kleinen Moleküls zu messen, das immobilisiert wurde.
HINWEIS: Mindestens 100 Hz Eigenfrequenz ist für eine erfolgreiche kleine Molekülimmobilisierung notwendig (1 Hz immobilisiert 30 pg).
2. Biopanning der T7-Phagenbibliothek mit einem QCM-Biosensor (Abbildung 1)
- Setzen Sie eine Küvette mit einem speziellen Magnetrührer auf den QCM-Biosensor und gießen Sie 8 ml des Reaktionspuffers (10 mM Tris-HCl, 200 mM NaCl, pH 7-8) in die Küvette.
- Befestigen Sie den QCM-Sensorchip am Oszillator und ziehen Sie den Arm des Oszillators nach unten, um den Chip in den Bei1000-Rpm-Gerührer gerührten Puffer einzutauchen.
- Beginnen Sie mit der Überwachung der QCM-Frequenz auf dem PC und warten Sie, bis das Sensorgramm auf etwa 3 Hz/min der Frequenzdrift gleicht.
- In jizieren Sie 8 l einer T7-Phagenbibliothek (1—2 × 1010 pfu/ml) in die Küvette (Endkonzentration: 1-2 × 107 pfu/ml) und markieren Sie den Injektionspunkt auf dem Sensor.
- Überwachen Sie die Frequenzreduktion, die durch die Bindung von T7-Phagen an das kleine Molekül verursacht wird, das 10 min auf der Goldelektrodenoberfläche immobilisiert ist.
- Stoppen Sie den QCM-Frequenzmonitor, lösen Sie den Sensorchip vom Oszillator und entfernen Sie den Puffer, indem Sie Luft blasen und/oder mit Tüchern ableiten.
- Legen Sie den Sensorchip in eine feuchte Petrischale und legen Sie die 20 L der Suspension von E. coli (BLT5615) (OD600 = 0,5—1,0 nach Schütteln bei 37 °C durch Zugabe von IPTG zu 1 mM) Wirtszellen in der Log-Phase auf die Goldelektrode ab.
- Schließen Sie den Deckel der Schale und bedecken Sie ihn mit Aluminiumfolie, um Licht zu blockieren.
- Inkubieren Sie die Schale bei 37 °C für 30 min auf einem 96-Well-Mikroplattenmischer (1000-1500 Rpm) zur Verbesserung der Wiederherstellung der gebundenen T7 Phagen.
- Stellen Sie die 20 L der Lösung wieder her, und setzen Sie sie in 200 l LB-Medium aus.
HINWEIS: Die in diesem Schritt erhaltenen Proben können bei 4 °C um eine Woche aufbewahrt werden. - Bereiten Sie eine Verdünnungsserie der Phagenlösung für plaque isolation und DNA-Sequenzierung nach dem allgemeinen Verfahren in der Herstelleranleitungbeschrieben 8,9.
- Wischen Sie die Goldelektrodenoberfläche mit einem Wattestäbchen ab, der mit 1% Natriumdodecylsulfat-Lösung getränkt ist.
- Waschen Sie die Goldoberfläche mit reinem Wasser aus der Waschflasche und entfernen Sie dann Wassertropfen, indem Sie Luft mit einer Spritze oder einem Luftstaubsauger blasen.
- Tropfen Sie 5 l Piranha-Lösung (Conc. H2SO4: 30% H2O2 = 3:1) auf die Goldoberfläche fallen und 5 min abgeben.
- Waschen Sie die Goldoberfläche wieder mit Wasser und trocknen Sie dann, indem Sie Luft blasen und/oder mit Tüchern ableiten.
- Wiederholen Sie die Schritte 2.14 und 2.15.
VORSICHT: Bereiten Sie die Piranha-Lösung unmittelbar vor der Anwendung vor. Verwenden Sie diese Flüssigkeit sorgfältig, da es eine sehr starke Säure ist. Die Behandlung über 5 min erodiert den Sensorchip.
3. Bioinformatik-Analyse mit Rezeptorligand Contact (RELIC) Programmsuite (Abbildung 2)10,11
- Entpacken Sie das eigenständige RELIC-Programm auf einem PC mit einem MS Windows-Betriebssystem.
- Richten Sie die Aminosäuresequenzen der 15-mer-Peptide-Affinitätsaffinität aus, die mit dem Medikament ausgewählt oder zufällig aus der nicht gescreenten übergeordneten Bibliothek in jeder Textformatdatei ausgewählt wurde (Name.txt).
- Geben Sie die Aminosäuresequenz einzelner oder mehrerer Proteine in jede Textdatei im FASTA-Format ein, oder laden Sie die Datenbanktextdateien im FASTA-Format aus einer beliebigen Proteindatenbank (z. B. UniProt (http://www.uniprot.org/) oder DrugBank (https://www.drugbank.ca/) herunter.
- Platzieren Sie die Textdateien (und PDB-Dateien für HETEROalign) in dem Ordner, der zum Ausführen jedes RELIC-Programms erforderlich ist.
- Klicken Sie im unabhängigen Ordner auf die ausführbare Datei (Programm.exe) für AADIV, INFO, MOTIF, MATCH, HETEROalign, FASTAcon und FASTAskan, um die persönliche Version von FTN95 zu öffnen.
- Geben Sie den entsprechenden Dateinamen zusammen mit der Erweiterung (Name.txt) in die Befehlsmeldung ein, um jedes Programm auszuführen und die erforderliche Textformatdatei abzuerhalten.
- Exportieren Sie die resultierende Textdatei in eine Tabellenkalkulationssoftware (z. B. Excel), um ein Diagramm mit Informationsinhalten (INFO) oder kumulativen Ähnlichkeitswerten zu generieren, die mit einem BLOSUM62 (MATCH, HETEROalign) berechnet wurden.
HINWEIS: Der ursprüngliche RELIC-Server (http://relic.bio.anl.gov) ist nicht mehr verfügbar und einige eigenständige RELIC-Programme, die auf PCs mit einer Windows-Plattform funktionieren, können vom entsprechenden Autor (tkksg@rs.noda.tus.ac.jp) bezogen werden.
Ergebnisse
Die repräsentativen Ergebnisse für zwei klinisch zugelassene Medikamente sind in Abbildung 3dargestellt. Irinotecan (Abbildung 3A), ein wasserlösliches Prodrug des natürlichen Camptothecins zur Behandlung von fortgeschrittenem Dickdarmkrebs und nicht-kleinzelligem Lungenkrebs, wird in der Leber in SN-38 umgewandelt, was Topoisomerase I in Krebszellen12hemmt. Darüber hinaus hemmt diese Verbindung direkt Acetylcholinesterase (AChE)13,14. Durch die Strategie wurden 29 Peptide, die Iri als SAM immobilisiert erkannten, von QCM-Biosensor-basierten Ein-Zyklus-Biopanning-Teilmengen identifiziert. Die anschließende paarweise Ausrichtung der 29 Peptide und AChE ergab maximale Werte für Y121, Q225, F290, E327, H440 und Y442 und hob den Teil in der dreidimensionalen Struktur hervor. Diese Aminosäurerückstände stimmten mit denen überein, die die Iri-bindende Stelle von AChE ausmachten. Dieselbe Untergruppe von Peptiden identifizierte erfolgreich E99, L100, L252, L305, I387 und V474 in der Nähe der katalytischen Triade (S221, E353 und H467) in carboxylesterase (CE), was darauf hindeutet, dass diese Aminosäuren ein Gerüst zur Iri-Erkennung bei der Esterung von Iri15bilden. Solche Aminosäurerückstände an der katalytischen Stelle können nicht direkt mit konventioneller Röntgenkristallographie oder NMR-Analyse identifiziert werden, da die Enzymreaktion reibungslos verläuft und den statischen Komplex unter allgemeinen experimentellen Bedingungen nicht stabil bildet. Somit ist der kombinatorische Nachweis von medikamentenbindenden Stellen mehrerer Proteine, einschließlich der in den Zwischenkomplexen, die möglicherweise mit Enzymen während der Stoffwechselreaktionen für ein bestimmtes Medikament gebildet werden, mit den für ein Medikament bestimmten Affinitäts-ausgewählten Peptiden möglich.
Abbildung 3B zeigt die anderen Ergebnisse für Oseltamivir (Osel), ein Anti-Grippe-Medikament, das gegen Oseltamivircarboxylat aktiviert wird, das wiederum die Neuraminidase (NA) des Influenzavirus16hemmt. Die 27 Peptide, die Osel auf der QCM-Sensorchip-Goldelektrodenoberfläche erkannten, erkannten erfolgreich die Osel-Bindungsstelle in NA16. Diese Bindungsstelle besteht aus unstrukturierten Peptidschleifen, die beim Andocken mit Osel möglicherweise einer dynamischen Bewegung unterzogen werden. Die Osel-erkennenden Peptide auf dem T7-Phagen-Capsid könnten dieses dynamische Andocken imitieren, wenn sie an die Osel-Basierte an der Goldelektrodenoberfläche des QCM-Sensorchips binden. Neuropsychiatrische Nebenwirkungen (NPAEs) wurden bei jungen Patienten mit Influenza identifiziert, die möglicherweise mit der Krankheit selbst und nicht mit dem Medikament zusammenhängen. Studien haben gezeigt, dass Osel aktiv aus dem zentralnervösen System (ZNS) von Nagetieren über Multidrug Resistance (MDR) Protein in der Blut-Hirn-Schranke (BBB)17exportiert wird. Tatsächlich zeigte eines der Proteine der Klasse dieser MDR in unserer Studie einen hohen Wert (Top 5% von 4.396 in der DrugBank 1.0 Proteindatenbank18), zusätzlich zu anderen Transportern, Neurotransmitter-bezogenen Enzymen und Rezeptoren. Die pharmakologische Bedeutung dieser Proteine im Hinblick auf das Auftreten von Nebenwirkungen von Osel wird untersucht.
Bisher wurden einzelne und mehrere kleine Molekülbindungsstellen an den Zielproteinen erfolgreich für sechs kleine Molekülmedikamente identifiziert, die unsere Strategie nutzten (Abbildung 4). Für Brz2001 und Roxithromycin (RXM) wurden identische Wirkstoffbindungsstellen auf einem Zielprotein mit verschiedenen Peptidenpools identifiziert, deren Anzahl und Aminosäuresequenzen vollständig variierten7,19. Darüber hinaus führten einzelne Peptidpools für Iri, RXM und Osel zur Identifizierung mehrerer Bindungsstellen an verschiedenen Proteinen für jedes Medikament, wie AChE und CE für Iri (Abbildung 3A)20, Angiomotin und CYP3A4 für RXM19,21und NA und MDR-assoziiertes Protein für Osel. Für die Anti-Tumor-Verbindung Doxorubicin (FANCF)22und das antiangiogene Makrolid-Antibiotikum RXM (Angiomotin)19wurde ein unbekanntes molekulares Ziel identifiziert.
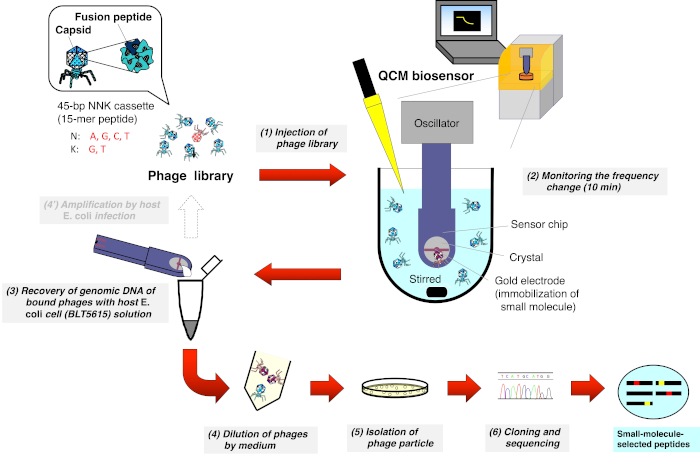
Abbildung 1: Schematische Darstellung der QCM-Biosensor-basierten Biopannierung der T7-Phagen-angezeigten Peptidbibliothek. Eine T7-Phagenbibliothek, die zufällige Peptide anzeigt, wird in die Küvette injiziert, die den Puffer (unter Rühren) enthält, in den der QCM-Biosensorchip getaucht und die Frequenz stabilisiert wird. Nach der Überwachung der Frequenzreduktion durch Bindung von T7-Phagen an kleine Moleküle, die auf der Goldelektrodenoberfläche des Sensorchips immobilisiert sind, wird der Sensorchip vom Oszillator abgetrennt. DIE DNA aus dem gebundenen T7-Phagen wird dann nach einer E. coli-Infektion (BLT5615) direkt wiederhergestellt. Die resultierenden T7-Phagen werden über Plaquebildung isoliert und schließlich wird die Aminosäuresequenz des auf dem T7-Phagenkapsid angezeigten Wirkstoffaffinitäts-ausgewählten Peptids nach der allgemeinen Phagenanzeigemethode bestimmt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
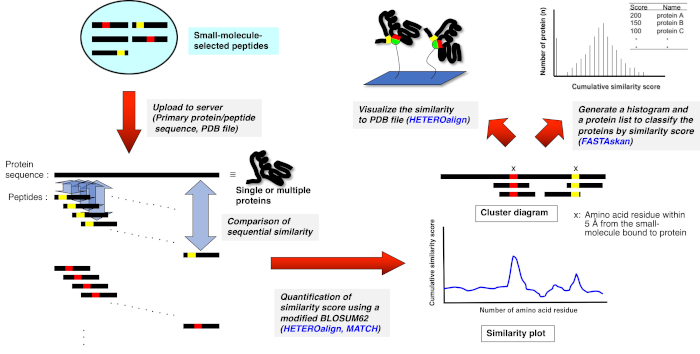
Abbildung 2: Schematische Darstellung der quantitativen Bewertung des Sequenzvergleichs zwischen medikamentös ausgewählten Peptiden und einzelnen oder mehreren Proteinen. Die medikamentös ausgewählten Peptidsequenzen werden jeweils an den primären Aminosäuresequenzen einzelner und mehrerer Proteine ausgerichtet, und die Ähnlichkeit in jedem 3-5 Aminosäure-Set wird kumulativ über eine paarweise Ausrichtung gemäß einer modifizierten BLOSUM62-Matrix bewertet. Das resultierende Diagramm zeigt die Rückstände oder Portionen an, die eine potenzielle wirkstoffbindende Stelle auf dem Protein darstellen. Eine weitere Analyse mit einem entsprechenden RELIC-Programm hebt die Bindungsseite auf der dreidimensionalen Struktur hervor (falls PDB-Datei verfügbar ist) oder ordnet ganze Proteine ein, die möglicherweise das Bindungsziel sind (HETEROalign-Programm ist derzeit nicht verfügbar). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
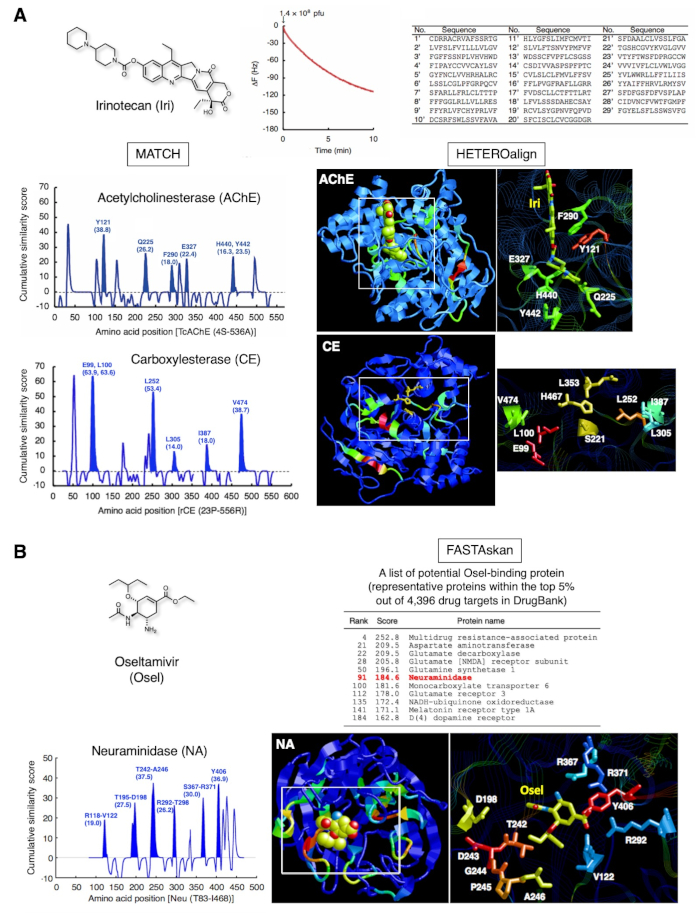
Abbildung 3: Repräsentative Ergebnisse der Peptidsammlung und der anschließenden Bioinformatikanalyse. (A) Irinotecan (Anti-Tumor-Prodrug, Topoisomerase-I-Hemmer). Die T7-Phagen-Interaktion wurde 10 min lang als Verringerung der QCM-Frequenz überwacht. Die DNA des gebundenen T7-Phagens wurde zurückgewonnen und sequenziert, um die entsprechende Aminosäuresequenz zu bestimmen. Die Aminosäuresequenzen von 29 15-mer Peptiden, die mit einer Teilmenge der Ein-Zyklus-Biopanning gesammelt wurden, hoben die Aminosäuren hervor, aus denen die Iri-bindungsstelle von AChE besteht [PDB ID 1U65]. Eine weitere Bewertung mit den gleichen 29 Peptiden zeigte die benachbarten Aminosäurerückstände (Gerüstrückstände zur Entesterung) der katalytischen Triade in CE, einem Leberenzym, das Iri in SN-38 (aktive Form) umwandelt [PDB-ID: 1K4Y]. Ähnlichkeitswerte von 103 zufällig ausgewählten Peptiden aus der nicht gescreenten übergeordneten Bibliothek7,19 wurden von diesen Partituren subtrahiert, um Bibliotheksverzerrungen zu entfernen. Diese Zahlen stammen aus Ref. 20, mit Genehmigung von Elsevier. (B) Oseltamivir (Anti-Grippe-Medikament). Die 27 Peptide, die Osel-erkennende Aminosäuren enthielten, zeigten ungeordnete Teile der Osel-Bindungsstelle für Neuraminidase (NA) (Virusenzym) [PDB ID: 2HT7]. Die globale Validierung der Sequenzähnlichkeit zwischen 27 Peptiden und 4.396 Proteinen in DrugBank 1.018 ergab, dass NA im oberen Bereich von 5% liegt, zusätzlich zu den menschlichen Wirtsproteinen, die mit den Funktionen des zentralen Nervensystems verbunden sind. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
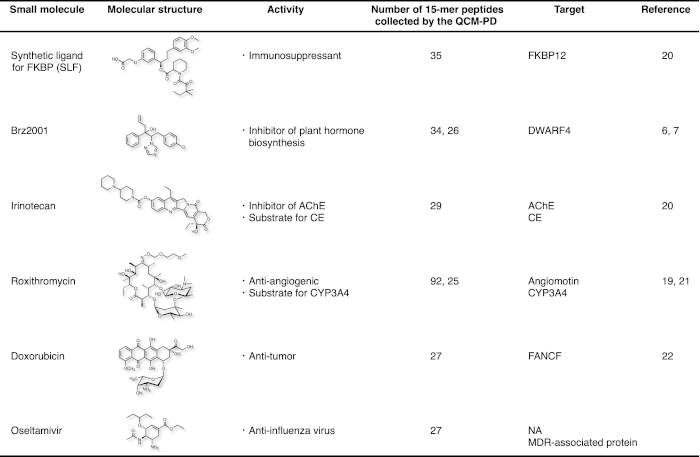
Abbildung 4:Zusammenfassung derkleinen Moleküle, deren Bindungsziele mit dieser Strategie identifiziert wurden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Hier wurde eine Strategie für die QCM-Biosensien von arzneimittelerkennenden Peptiden vorgestellt, gefolgt von einer Bioinformatik-Analyse zur Validierung von Arzneimittel-Protein-Wechselwirkungen unter Verwendung der identifizierten Peptide. Die Entwicklung der kleinen Molekülderivate zur Immobilisierung auf der Goldelektrode des Biosensors ist ein wichtiger Schritt, da der eingeführte Linker die Bindung und Sammlung des Peptids, das das Medikament erkennt, behindern kann. Um dies zu vermeiden, werden Derivate mit unterschiedlichen Positionen des eingeführtenLinkers 23vorbereitet. Alternativ wird der Sensorchip zur Immobilisierung hydrophober kleiner Moleküle in massenhaftes Wasser in eine 10 cm Petrischale getaucht und eine 20-L-Lösung des kleinen Moleküls (10 mM Lösung in Dimethylsulfoxid) auf die Goldelektrode des Biosensors fallen, um seine Oberfläche zu bedecken, und für 5 min inkubiert. Dies ermöglicht die Retention einer intrinsischen Frequenz von kleinen Molekülen mit unter hundert Hertz, die während der Biopannierung mindestens 10 min gehalten wird. Tatsächlich haben die von Osel-Affinität ausgewählten Peptide mit einer solchen Immobilisierung die Osel-Bindungsstelle in NA deutlich hervorgehoben (Abbildung 3).
Der T7-Phagen, der hier für die Vorbereitung der Peptidbibliothek verwendet wird, ist gentechnisch verändert mit der NNK15 Kassette, die 32 Codons für alle 20 Standard-Aminosäuren kodiert und die Entstehung von 2 Stop-Codons (UAA, UGA) unterdrückt und nur UAG (Abbildung 1)6,7. Dies ist wichtig für die Anzeige von 15-mer-Peptiden in voller Länge und die Erhöhung der Vielfalt der Bibliothek. Das Phagenanzeigesystem T7 hat eine technische Anzeigegrenze von 107—109 T7 Phagen. Die Vielfalt der 15-mer-Peptidbibliothek beträgt jedoch theoretisch 2015 (3,27 × 1019); daher kann es nicht für den kompletten Bibliotheksbau verwendet werden. Dennoch ermöglicht die Ähnlichkeitssuche oder der Abbau konservierter Motive den Nachweis der Aminosäuren, die die wirkstoffbindenden Stellen von Proteinen umfassen, auch bei dieser begrenzten Vielfalt an Peptiden in der Bibliothek. Darüber hinaus sind 3-5 Aminosäure-Dehnungen innerhalb des Bibliothekspeptids (die Erscheinungsrate liegt zwischen 1/203 und 1/ 205, die mit dem T7-Phagenanzeigesystem realisiert werden kann) an der Erkennung kleiner Molekülmedikamente beteiligt; Daher ist eine 100%ige Übereinstimmung der Peptidsequenzen mit 15-mer-Aminosäuresequenzen, die die Wirkstoffbindungsstelle des Zielproteins bilden, nicht erforderlich. Tatsächlich haben etwa 30 affinitätsausgewählte Peptide erfolgreich die Bindungsstelle des Zielproteins für jedes getestete Medikament hervorgehoben (Abbildung 4). So kann die Vielfalt der verwendeten Elternbibliothek T7 (1,7 × 107 pfu/ml) genutzt werden, um die drogenbindende Stelle heuristisch zu rekonstruieren.
Typischerweise entstanden innerhalb der 16 Plaques willkürlich isolierte Kopien von T7-Phagen, die die gleichen Sequenzen wie die der 15-mer-Aminosäuresequenzen zeigten, die drogenerkennende Aminosäure-Strecken beherbergen. Dies deutet darauf hin, dass 18-30 verschiedene medikamentös erkennende Peptidsequenzen innerhalb der 96 Plaques isoliert werden (die Zahl ist mit dem Mikroplattenformat verbunden), die anschließend mittels Sequenzierung der DNA und der entsprechenden Aminosäuresequenz identifiziert werden. Nach der vorliegenden Strategie eignet sich die Injektion von 8 l der T7-Phagenbibliothek in die Küvette mit 8 ml Puffer (1000-fache Verdünnung der Bibliothek) zur Reduzierung der unspezifischen Bindung von T7-Phagen. Um die Vielfalt der affinitätsausgewählten Peptide zu erhöhen, erwies sich die mehrfache Wiederholung der Ein-Zyklus-Auswahl und die Verwendung von 16 oder 32 Plaqueisolationen pro Screening als effektiver als die Isolierung von einer einzigen Lösung zu einem Zeitpunkt. Um beispielsweise etwa 30 unterschiedlich sequenzierte, affinitätsausgewählte Peptide effektiv zu sammeln, wurden 3–6 Sätze von Ein-Zyklus-Auswahl durchgeführt, 16 oder 32 Plaques wurden in jedem Experiment isoliert. Das Auftreten identischer Sequenzen in allen 16 oder 32 T7 Phagen-Plaques ist auf eine versehentliche Erkennung des Hintergrunds oder eine Kontamination als Übertragung hindeutet. Im Gegensatz dazu deutet das Fehlen von T7-Phagen mit der gleichen Reihenfolge oder Erscheinung vieler T7-Phagen mit kürzeren Peptiden als die 15-mer-Länge darauf hin, dass T7-Phagen in der Population nicht spezifisch mit hoher Wahrscheinlichkeit entstanden sind. Da die QCM-Frequenzreduktion auch in solchen Fällen im gleichen Ausmaß erfolgt, sollte der Erfolg der Selektion umfassend bewertet werden, indem die DNA des isolierten T7-Phagens sequenziert wird, gefolgt von einer bioinformatischen Analyse der Aminosäuresequenzen der Peptide. Darüber hinaus sind im Gegensatz zum herkömmlichen T7-Phagenanzeigeprotokoll wiederholte Selektionsrunden weniger effektiv, da die Variation und Anzahl der T7-Phagen klein sind und auch nach Wiederholung der Verstärkungs- und Selektionsschritte23nicht konzentriert sind.
Wichtig ist, dass diese Methode für den Abbau kleiner Molekül-bindender Stellen in den Proteomen von Menschen, pathogenen Viren und sogar Pflanzen anwendbar ist. Interessanterweise kann die möglicherweise unstrukturierte kurze Anzeigelänge von Peptiden auf dem T7-Phagenkapsid die molekulare Dynamik von Peptiden von Proteinen während des Andockens mit einem kleinen Molekül imitieren; dies kann die dynamische Bindung24widerspiegeln. Über die technischen Grenzen konventioneller Methoden hinaus kann diese Strategie, die auf identische Protokolle für kleine Moleküle anwendbar ist, das medikamentöse Proteom erweitern und eine größere Granularität in Bezug auf die Wirkstoff-Protein-Interaktionsanalyse bieten.
Bestimmte technische Einschränkungen dieses Ansatzes sollten in Betracht gezogen werden. Die organische Synthese ist für die kleine Molekülimmobilisierung auf der Goldelektrodenoberfläche des Biosensorchips notwendig. Für die Nicht-Experten in der organischen Chemie sind einige Immobilisierungsreagenzien kommerziell erhältlich, um das kleine Molekül durch Kopplung mechanisch zu fixieren. Darüber hinaus können bestimmte Unsinnsanteile der Peptide dazu führen, dass ein Teil des Proteins, der für das Drogendocken nicht relevant ist, als falsch positive Werte nachweisbar ist. Dies entspricht empirisch Beta-Blatt- oder Leucin-reichen Domänen reich an Leucin- oder Valin-Rückständen, die bei der Herstellung von Kopien des T7-Phagens durch mehr Codons als andere Standard-Aminosäuren kodiert werden. Die Steuerung der Bibliothekspeptidlänge kann das Auftreten falschpositiver Positivmeldungen steuern. Im Gegensatz dazu kann es Fälle geben, in denen Aminosäurerückstände an der Wirkstoffbindungsstelle, die am Andocken beteiligt sind, wie mit Röntgenkristallographie oder NMR-Analyse nachgewiesen, nicht nachgewiesen werden. Dies kann gelöst werden, indem eine größere Anzahl der arzneimittelerkennenden Peptide gesammelt oder die Richtung der Fixierung der kleinen Moleküle auf der Goldelektrode geändert wird.
Viele Wechselwirkungen zwischen Arzneimittel und Proteinen, die mit den Haupt- und Sekundärwirkungen des Drogenkonsums zusammenhängen, können im Proteom noch nicht identifiziert werden; Außerdem könnten Enzyme und Transporter, die für die Wirkstoffaufnahme, -verteilung, -stoffwechsel, -ausscheidung und -toxizität verantwortlich sind, ebenfalls noch nicht identifiziert werden. Proteinbindung ist nicht immer verantwortlich für die Bioaktivität eines Arzneimittels. So wird eine Kombination anderer Informationen aus biologischen Assays die Identifizierung wesentlicher Arzneimittelziele verbessern, die für die wichtigsten und nachteiligen Auswirkungen von Arzneimitteln verantwortlich sind. Weitere Anpassungen dieser prägnanten Technik werden die Praktikabilität und den Durchsatz für den Abbau der Proteinbindungsstellen einer breiten Palette kleiner Molekülmedikamente erhöhen. Die hier vorgestellte Methode wird weitgehend dazu beitragen, nicht nur Grundlagenforschungen in den verwandten Bereichen durchzuführen, sondern auch die molekularen Mechanismen zu klären, die der therapeutischen Wirksamkeit oder anderen biologischen Wirkungen von Arzneimitteln im klinischen Einsatz zugrunde liegen.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Der Autor dankt Drs. Yujiro Hayashi und Hayato Ishikawa für die Bereitstellung von Oseltamivir und Dr. Lee Makowski für die Bereitstellung der eigenständigen RELIC-Programme. Der Autor würdigt auch Tetsuya Kawagoe für die technische Unterstützung des QCM-Experiments. Diese Arbeit wurde teilweise von JSPS KAKENHI Grant Number 17K01363 (Y.T.) unterstützt.
DATENVERFÜGBARKEITS-ANWEISUNG
Die eigenständigen RELIC-Programme und die Sequenzdaten von affinitätsausgewählten Peptiden für Medikamente sowie die in dieser Arbeit verwendeten Proteinsequenzen aus der Proteom-Datenbank sind bei diesem Autor auf Anfrage erhältlich (tkksg@rs.noda.tus.ac.jp).
Materialien
| Name | Company | Catalog Number | Comments |
| AFFINIXQN | ULVAC, Inc. (Tokyo, Japan) | QCM2008-STKIT | Contains Glass cuvette, stir magnet, operation and analysis software with a Windows PC |
| AADIV | Northeastern University (Lee Makowski) | AADIV.exe | Calculates the frequency of occurrence of each of the 20 amino acids at each recombinant insert position, as well as the overall position-independent frequency of each amino acid within that set of peptide sequences. Also roughly estimates the sequence diversity of a display library by statistical sampling method based upon sequences obtained from a limited number of randomly sampled members of the library. |
| Ceramic Sensor Chip | ULVAC, Inc. (Tokyo, Japan) | QCMST27C | 4 sensor chips/package |
| Dimethyl sulfoxide | Sigma-Aldrich (St. Louis, MO, USA) | D8418 | |
| Ethanol | Merck (Kenilworth, NJ, USA) | 09-0850 | |
| FASTAcon | Northeastern University (Lee Makowski) | FASTAcon.exe | Identifies proteins from a population with short consensus sequences. |
| FASTAskan | Northeastern University (Lee Makowski) | FASTAskan.exe | Lists proteins with high similarity to a peptide population. |
| Immiblization kit for AFFINIX | ULVAC, Inc. (Tokyo, Japan) | QCMIMKT | SAM reagent and amine coupling reagent |
| INFO | Northeastern University (Lee Makowski) | INFO.exe | Provides mathematical measure of the probability of observing a particular peptide sequence by random chance (i.e., nonspecific binding) as opposed to by selection for a specific property (affinity to small molecule). |
| Liquid LB medium | Sigma-Aldrich (St. Louis, MO, USA) | L3522 | Autoclave for 20 min |
| MATCH | Northeastern University (Lee Makowski) | MATCH.exe | Identifies any stretches of amino acid residues within a particular protein that exhibit significant similarity to a group of affinity-selected peptides. Outputs as cluster dia- gram and cumulative similarity plot calculated from a modified BLOSUM62 matrix with a short window (5–6 amino acids in length). |
| MOTIF1 | Northeastern University (Lee Makowski) | MOTIF1.exe | Searches for three continuous amino acid sequence motifs within a peptide population. |
| MOTIF2 | Northeastern University (Lee Makowski) | MOTIF2.exe | Searches for patterns of three amino acids and does not allow conservative amino acid substitutions, but does allow identical gap lengths. |
| NaCl | Merck (Kenilworth, NJ, USA) | S3014 | |
| Receptor ligand contacts (RELIC) | Argonne National Laboratory (Lemont, IL, USA) | https://www.relic.anl.gov | Currently unavailable (Stand-alone program can be used from correspondence author upon request) |
| Tris | Merck (Kenilworth, NJ, USA) | 252859 |
Referenzen
- Santos, R., et al. A comprehensive map of molecular drug targets. Nature Reviews Drug Discovery. 16 (1), 19-34 (2017).
- Ziegler, S., Pries, V., Hedberg, C., Waldmann, H. Target identification for small bioactive molecules: finding the needle in the haystack. Angewandte Chemie International Edition (English). 52 (10), 2744-2792 (2013).
- Piggott, A. M., Karuso, P. Identifying the cellular targets of natural products using T7 phage display. Natural Product Reports. 33 (5), 626-636 (2016).
- Takakusagi, Y., Takakusagi, K., Sakaguchi, K., Sugawara, F. Phage display technology for target determination of small-molecule therapeutics: an update. Expert Opinion on Drug Discovery. 15 (10), 1199-1211 (2020).
- Takakusagi, Y., Takakusagi, K., Sugawara, F., Sakaguchi, K. Use of phage display technology for the determination of the targets for small-molecule therapeutics. Expert Opinion on Drug Discovery. 5 (4), 361-389 (2010).
- Takakusagi, Y., Takakusagi, K., Sugawara, F., Sakaguchi, K. Using the QCM Biosensor-Based T7 Phage Display Combined with Bioinformatics Analysis for Target Identification of Bioactive Small Molecule. Methods in Molecular Biology. 1795, 159-172 (2018).
- Takakusagi, Y., et al. Mapping a disordered portion of the Brz2001-binding site on a plant monooxygenase, DWARF4, using a quartz-crystal microbalance biosensor-based T7 phage display. ASSAY and Drug Devevelopment Technologies. 11 (3), 206-215 (2013).
- Novagen. T7 Select® System Manual. Novagen. , TB178 1009JN (2009).
- Novagen. OrientExpressTM cDNA Manual. Novagen. , TB247 1109JN (2009).
- Mandava, S., Makowski, L., Devarapalli, S., Uzubell, J., Rodi, D. J. RELIC--a bioinformatics server for combinatorial peptide analysis and identification of protein-ligand interaction sites. Proteomics. 4 (5), 1439-1460 (2004).
- Makowski, L. Phage Nanobiotechnology. Petrenko, V. A., Smith, G. P. , RSC Publishing. Ch. 3 33-54 (2011).
- Garcia-Carbonero, R., Supko, J. G. Current perspectives on the clinical experience, pharmacology, and continued development of the camptothecins. Clinical Cancer Research. 8 (3), 641-661 (2002).
- Harel, M., et al. The crystal structure of the complex of the anticancer prodrug 7-ethyl-10-[4-(1-piperidino)-1-piperidino]-carbonyloxycamptothecin (CPT-11) with Torpedo californica acetylcholinesterase provides a molecular explanation for its cholinergic action. Molecular Pharmacology. 67 (6), 1874-1881 (2005).
- Dodds, H. M., Rivory, L. P. The mechanism for the inhibition of acetylcholinesterases by irinotecan (CPT-11). Molecular Pharmacology. 56 (6), 1346-1353 (1999).
- Bencharit, S., et al. Structural insights into CPT-11 activation by mammalian carboxylesterases. Nature Structural Biology. 9 (5), 337-342 (2002).
- Kim, C. U., et al. Influenza neuraminidase inhibitors possessing a novel hydrophobic interaction in the enzyme active site: design, synthesis, and structural analysis of carbocyclic sialic acid analogues with potent anti-influenza activity. Journal of the American Chemical Society. 119 (4), 681-690 (1997).
- Hoffmann, G., et al. Nonclinical pharmacokinetics of oseltamivir and oseltamivir carboxylate in the central nervous system. Antimicrobial Agents and Chemotherapy. 53 (11), 4753-4761 (2009).
- Wishart, D. S., et al. DrugBank 5.0: a major update to the DrugBank database for 2018. Nucleic Acids Research. 46 (1), 1074-1082 (2018).
- Takakusagi, K., et al. Multimodal biopanning of T7 phage-displayed peptides reveals angiomotin as a potential receptor of the anti-angiogenic macrolide Roxithromycin. European Journal of Medicinal Chemistry. 90, 809-821 (2015).
- Takakusagi, Y., et al. Efficient one-cycle affinity selection of binding proteins or peptides specific for a small-molecule using a T7 phage display pool. Bioorganic and Medicinal Chemistry. 16 (22), 9837-9846 (2008).
- Takakusagi, Y., Suzuki, A., Sugawara, F., Kobayashi, S., Sakaguchi, K. Self-assembled monolayer (SAM) of small organic molecule for efficient random-peptide phage display selection using a cuvette type quartz-crystal micobalance (QCM) device. World Journal of Engineering. 5, 1005-1006 (2009).
- Kusayanagi, T., et al. The antitumor agent doxorubicin binds to Fanconi anemia group F protein. Bioorganic and Medicinal Chemistry. 20 (21), 6248-6255 (2012).
- Takakusagi, Y., et al. Identification of C10 biotinylated camptothecin (CPT-10-B) binding peptides using T7 phage display screen on a QCM device. Bioorganic and Medicinal Chemistry. 15 (24), 7590-7598 (2007).
- Rodi, D. J., et al. Identification of small molecule binding sites within proteins using phage display technology. Combinatorial Chemistry and High Throughput Screening. 4 (7), 553-572 (2001).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten