Method Article
用于生物膜材料异质性 3 维分析的基于 Bead 运动的计算框架
摘要
我们描述了一种分析和量化 1 μm 羧酸盐珠子通过异质细菌生物膜的运动模式的方法。运动模式的比较可用于量化生物膜材料特性的差异。
摘要
在不同细菌种类的生物膜中,在不同生长条件下的同一物种内以及用基质修饰分子处理后,已经观察到细菌生物膜材料特性的差异。为了更好地定量 3D 生物膜的材料特性,开发了一种实验和计算工作流程,并应用于检查 粪肠球菌、肠道沙门氏菌 血清型鼠伤寒和 大肠杆菌 生物膜之间的差异,以及淀粉样蛋白卷曲在确认肠杆菌科生物膜刚性中的作用。在 20 分钟内在 20 μm 3D 生物膜中跟踪生物膜中 1 μm 羧酸盐珠子的时空动力学。使用 ImageJ 中的 Mosaic 插件处理 4D 图像堆栈,以生成珠子运动的 3D 轨迹数据。该轨迹数据使用新开发的 Bead Evaluator 工具箱进行分析,其中运动数据(包括轨迹寿命、珠子速度、沿轨迹的细胞密度和边界框信息)被计算并存储在 csv 文件中。本文介绍了从实验设置和图像记录到微珠轨迹计算和分析的工作流程。与 curli 突变体和肠球菌生物膜相比,含 curli 的生物膜的结构导致更稳定的珠子相互作用和更少的珠子运动。在测量珠子速度和轨迹边界框体积时,珠子运动似乎并不强烈依赖于细胞密度,这支持了生物膜的其他材料特性控制珠子动力学的假设。该技术广泛适用于定量不同基质组成的生物膜以及基质修饰处理前后生物膜的差异。
引言
细菌生物膜作为人类微生物群的一部分无处不在,并不断与分子相互作用。这些分子的大小从 1 nm 抗生素和 1-3 mm 细菌到胃肠道中较大的纤维颗粒不等。单物种或多物种生物膜的组成会影响材料特性,从而影响颗粒通过生物膜的运动模式 1,2,3,4,5。一个例子是细菌淀粉样蛋白,它具有保守的纤维状交叉 β 片结构6。卷曲淀粉样蛋白由肠道细菌表达,例如大肠埃希菌和肠道沙门氏菌血清型 鼠伤寒,并且已在多个其他细菌门中检测到基因7。生物膜的各种材料特性都受 curli的影响 8,9。Curli 直接与基质的其他组分相互作用,例如细胞外 DNA (eDNA) 和纤维素10,11。Curli 围绕细胞并影响细胞膜刚度12 和生物膜的整体粘弹性13。Curli 通过与纤连蛋白结合来介导拉伸强度的增加,从而导致玻璃表面紧密结合的增加14。进入的噬菌体与 curli 结合并限制噬菌体侵入生物膜15。
当使用 Multitest 涂层孔玻片分析大约 20 μm 厚的粪肠球菌、大肠杆菌和 S 时。使用共聚焦显微镜的鼠伤寒生物膜,大肠杆菌、S 之间的明显差异。可以观察到鼠伤寒 10,16 和粪肠球菌生物膜(当前研究)。虽然肠杆菌科生物膜具有高水平的刚性并且细胞密度低的区域易于成像,但使用线和帧平均获得清晰的高分辨率粪肠球菌生物膜图像需要对载玻片施加压力,以诱导足够的表面张力,以便在成像过程中保持细胞稳定性。细菌淀粉样蛋白(如 curli)形成高度有序的结构,表明它们可能相对坚硬17。这激发了淀粉样蛋白卷曲可能诱导大肠杆菌和 S 僵硬的假设。鼠伤寒生物膜。没有明确的证据表明 E. faecalis 在研究的条件下表达淀粉样蛋白。蛋白质 Esp 是一种与粪肠球菌的更多致病菌株相关的 pilin 基因,最近显示可产生淀粉样蛋白结构18;然而,使用 BLASTN 和 BLASTP 搜索,在这些研究中使用的粪肠球菌共生型菌株 OG1RF 中未检测到该基因。OG1RF 产生的信息素 cOB1 可以形成淀粉样蛋白结构19。然而,在给定的生物膜生长条件和先前用于 S 的淀粉样蛋白检测方法下。 鼠伤寒淀粉样蛋白染色10 在粪肠球菌中,无法检测到 OG1RF 淀粉样蛋白(数据未显示)。开发了一种新的四维 (4D) 成像技术,用于比较粘性粪肠球菌、大肠杆菌和鼠伤寒沙门氏菌之间的整体材料特性,以及使用 S 的淀粉样蛋白突变体确定淀粉样蛋白对肠杆菌科生物膜的贡献。 鼠伤寒和大肠杆菌。
过去,荧光珠成功地用于使用微流变学 20,21,22,23,24,25 在二维 (2D) 中分析生物膜的材料特性。这可以通过研究生物膜中不同深度的 2D 光学切片26 应用于三维生物膜。目前的技术是为了随着时间的推移在 3D 中跟踪 1 μm 微尺度的微珠,以用于 4D 建模。部分基本原理是使用 4D 建模来了解质粒在胃肠道微生物群落中的移动的总体概念。使用直径为 1 μm 的带荧光电荷的羧酸盐珠子,因为它们在大小和电荷方面与粪肠球菌非常对应,粪肠球菌是选择的用于质粒移动和维持的模式生物27, 28。开发了一种 4D 测定法来量化生物膜的物理特性(图 1A)。在设计的方法中,将珠子添加到生物膜中,并在 10-20 分钟内通过 10-20 μm 厚的生物膜记录它们的时空轨迹。然后使用新开发的工具箱根据轨迹长度、珠子速度、轨迹边界框体积(包含轨迹的最小框)和边界框细胞密度来量化 3D 中的珠子轨迹。以下协议可用于生成细菌和珠子的 4D 图像数据含有生物膜,使用 ImageJ29 和插件 Mosaic 预处理数据,以及使用 Bead Evaluator 工具箱分析珠子轨迹。
该技术具有多种应用,可用于检查材料特性以及跟踪颗粒和细菌的三维运动。例如,该技术的早期版本用于表征针对 curli 的单克隆抗体对生物膜结构完整性的影响16。完整版具有多种工具,可提供对生物膜材料特性的更详细分析,并继续用于检查单克隆抗体处理对生物膜的影响。不同电荷的粒子可用于检查生物膜的材料电荷特性以及粒子在具有不同基质组成的生物膜中的运动。这可用于比较 2D 微流变学的结果,这些结果揭示了我们在未流动的生物膜中观察到的珠子移动的材料特性。该技术也可用于具有不同生物膜组成区域的混合物种生物膜。生物膜可以在微流体设备和流动池中的流动下进行实时成像,以检查静态和流动生物膜之间材料特性的变化以及流动对颗粒运动的影响。该技术还可以应用于荧光标记的细菌,以表征外源细菌通过生物膜群落的运动。使用三种颜色,荧光标记的供体细菌、荧光标记的受体细菌和荧光标记的质粒可用于跟踪质粒的运动、对接和转移。
研究方案
1. 生物膜制备
注:用于分析的生物膜可以使用任何允许在光学玻璃表面形成生物膜的方法生长。生物膜结构应充分粘附在光学表面,以便在方案的洗涤和/或安装步骤中结构不会被破坏。下面描述了 96 孔光学底板和 24 孔板中 12 mm 玻璃盖玻片的技术。其他选项包括不同尺寸的光学底板和带和不带流量的光流室。
- 生物膜设置
- 将细菌生长培养基添加到板的孔中。对于这项研究,对于 E。 粪便,向 24 孔板中加入 2 mL 的 Todd-Hewitt (TH),向视底 96 孔板中加入 0.4 mL 的 TH。对于 S。鼠伤寒、 大肠杆菌 和同基因 csgAB curli 突变体,向孔中加入 0.7 mL 无盐 Luria 肉汤 (LB)。如果使用光学底腔室,请继续执行步骤 1.2。
- 将 12 mm #1.5 玻璃光学盖玻片放入培养皿中,并用乙醇覆盖。
- 使用镊子取下盖玻片,用火焰点燃剩余的酒精。让酒精燃烧掉。只需用火焰点燃它们;不要将盖玻片放在火焰中,因为它会破裂。在将盖玻片放入孔中之前,让盖玻片冷却 10-20 秒,以防止开裂。
- 将盖玻片以一定角度放入装有培养基的孔中,以防止其躺在培养基顶部。不要将盖玻片添加到干燥的孔中,然后添加培养基,因为这会导致盖玻片粘在孔的底部。
- 使用无菌移液器吸头小心地将盖玻片推至含有培养基的孔的底部。
注意:请记住将光学玻璃盖玻片或光学底板的厚度(#1 或 #1.5)与共聚焦显微镜光学元件的厚度相匹配。
- 在适当的条件下孵育生物膜以促进生物膜生长。在这些研究中,在 37 °C 下将 粪肠杆菌 生物膜作为静态培养物生长,并在 30 °C 下有氧生长 大肠杆菌 生物膜。
- 在这些研究中,在早晨和傍晚用培养基切换,将 粪肠球菌 生物膜培养 2 天。为防止损坏或移动生物膜,请小心地倾斜板。将移液器吸头放在孔的底部边缘附近,然后慢慢吸出培养基。以相同的方式加入第一 mL 新鲜培养基。在培养基/孔界面附近缓慢加入第二个 mL。
- 在这些研究中,使用生长条件来实现最佳的卷发生产。生长 S。 鼠伤寒生物膜在 28 °C 下孵育 6-8 天,倾斜孵育,使生物膜附着在载玻片上方约 2/3 处,然后在气液界面上生长为薄膜。这是在没有更换介质的情况下完成的。为防止培养基变干,请将 24 孔板放入带水盘的腔室中。
2. 4D 成像
- 生物膜支架的准备
- 在 PBS 中以 1:50 的比例稀释 Crimson 1 μm 羧酸盐 FluoSpheres 微珠(在 1 mL PBS 中加入 2 x 107 个微珠)。如果使用光学底腔室,则将稀释度为 1 μL 的 Syto9 添加到 300 μL 微珠制备物中。
- (可选)如果生长培养基具有自发荧光,请清洗生物膜以去除生长培养基的痕迹。在这些实验中,用 1 mL PBS 洗涤两次,小心地倾斜板,将移液器吸头放在孔的底部边缘附近,然后慢慢吸出培养基。通过将尖端放在底部边缘附近加入 PBS,然后慢慢填充孔。除了 24 孔板中的盖玻片外,还可以在光学底孔上使用该技术。
- 去除培养基或 PBS。将 2.1.1 中制备的稀释 Crimson 珠子添加到生物膜中。在这些研究中,将 1 mL 珠子 (2x107) 添加到盖玻片中,将 0.2 mL 珠子 (4x106) 和 Syto9 添加到 96 孔光学底板中。
- 在室温下孵育 1 分钟以允许珠子结合。
- 去除珠子并用 PBS 轻轻洗涤生物膜一次,以去除未结合的珠子。在这些研究中,对于盖玻片,用 1 mL PBS 轻轻洗涤,用 1 mL PBS 重新填充孔,然后进行 2.1.6。对于 96 孔光学底板,用 0.2 mL PBS 轻轻洗涤生物膜,然后用 0.2 mL PBS 重新填充孔。光学底部腔室现在可以成像,因此请继续执行步骤 2.2。
- (如果使用盖玻片)将 1 μL Syto9(绿色荧光 DNA 染色剂;根据制造商的说明稀释)添加到包被的 10 孔多测试载玻片的孔中心。这些涂层盖玻片的孔深为 23-25 μm。
- (如果使用盖玻片)使用酒精火焰灭菌镊子,小心地从孔中取出盖玻片,然后倒置到含有 Syto9 滴剂的孔上。将盖玻片留在 1 mL PBS 中更容易从孔中取出,并有助于从生物膜中洗出未结合的珠子。
- (如果使用盖玻片)用指甲油小心密封盖玻片,不要滑动或按下盖玻片,这会导致表面张力,从而停止在更粘稠、更不坚硬的生物膜中移动。
- (如果使用盖玻片)让指甲油干燥。用 70% 乙醇小心擦拭盖玻片的外表面。由于上述原因,无需对盖玻片施加任何压力即可擦拭。
- 共聚焦成像
注意:在这些研究中,使用配备 TCS 共聚焦系统和 63 倍物镜的倒置光谱成像激光扫描共聚焦显微镜。该示波器将用于生成 4D 视频(图 1)。3D 生物膜将由通过 18-20 μm 厚的生物膜以 0.5 μm 步骤捕获的 Z 切片组成,产生 36-40 个 Z 切片。每个 3D 生物膜需要 50-60 秒来捕获。切片一起将构成一个框架,该框架可以可视化为 3D 生物膜。此过程将重复 20 次以生成 4D 延时视频,总跟踪时间为 18-20 分钟。- 设置范围以捕获荧光基团。在这些研究中,用 488 nm 激光激发 Syto9(细菌 DNA 染色)并测量 495 至 540 nm 的发射(Leica Sp5 是一种光谱成像显微镜)。用 633 nm 激光激发深红色(红色)微珠,并测量 650 nm 至 700 nm 的发射波长。可以调整这些荧光基团设置以捕获任何所需的荧光基团。
- 选择 xyzt 成像模式。
- 确定生物膜中具有高密度和低密度区域混合物的区域,以捕获生物膜厚和薄区域的粘弹性差异。
- 设置 18-20 μm 厚的 Z 堆栈。在盖玻片安装的生物膜中,避免生物膜的最顶部和最底部接触玻璃,以避免珠子捕获伪影(请参阅讨论)。
- 调整增益和偏移,以在生物膜最亮的点中使用强度的整个动态范围。这最大限度地减少了来自生物膜下层的信号重叠。
- 将切片厚度设置为 0.5 μm。这允许快速成像而不会丢失微珠信息。
- 将分辨率设置为 512 x 512 (0.48 μm)。这允许快速成像,但生成的图像具有足够的分辨率,可以看到生物膜结构和珠子运动细节。
- 最大限度地缩短成像时间。
- 设置为捕获 20 个堆栈。
- 另存为 .lif(或类似的共聚焦文件)。可以在 ImageJ 中生成 4D 电影。
3. 使用 ImageJ 生成 4D 生物膜视频
- 使用以下设置在 ImageJ 中打开 .lif 文件:使用 Hyperstack 查看堆栈,堆栈顺序 XYCZT,颜色模式:彩色,选中自动缩放。然后按 OK。
- 选择 Image > Color > Split channels。
- 选择 “图像>颜色>合并通道”>“创建合成”>确定。
- 选择 3D 查看器>插件 > 通道 取消选中蓝色 > 确定。
- 选中 Edit > Show content > Show bounding box 选中。
- 使用鼠标单击并按住以选择图像。然后在继续按住单击按钮的同时旋转图像。旋转图像,使生物膜的底部在底部,并且角度支持可视化珠子。然后释放图像。
- 按红色 Record 窗口底部的按钮录制视频。
- 使用 jpeg 作为压缩,另存为 .avi 文件。
4. 生成轨迹数据
- 安装开源工具 ImageJ (https://imagej.net/Fiji) 和粒子跟踪插件 Mosaic (https://imagej.net/MOSAICsuite)。
- 导入包含微珠和细菌两个通道的 .lif 文件。拆分通道并单独保存文件。
- 在 ImageJ 中,将图像体素大小(x、y 和 z 维度)和时间步长存储到文本文件中。
- 在 ImageJ 中,转到 Plugins |Mosaic 并启动 Particle Tracker 2D/3D。
- 输入以下参数:半径:3,截止:0.003,Per/Abs:0.12,链接范围:2,置换:10.00,动力学设置为 Brownian。请参阅结果以了解在这些实验中选择 Brownian 的基本原理。
- 生成轨迹并将轨迹列表导出为 .csv 文件。
5. 分析轨迹
- 安装 VRL Studio (https://vrl-studio.mihosoft.eu)
- 下载 Biofilm 项目 (https:// neurobox3d.github.io/Biofilm/) 并在 VRL Studio 中启动
- 在 ImportData 中加载轨迹文件
- 在 ProcessTrajectories 中指定 x、y 和 z 像素大小(使用 ImageJ 查找这些值)。
- 在 ComputeVelocity 中指定帧间隔(使用 ImageJ 查找此值)。
- 在 Comdensity 中加载细菌 tiff 文件(请参阅步骤 3.2)。
- 在 SaveVelocityDataToFile 中设置速度数据的输出路径,在 SaveTrajectoryDataToFile 中设置轨迹数据的输出路径。
- 调用 SaveVelocityDataToFile 和 SaveTrajectoryDataToFile。
- 将数据导入 Excel 进行分析。这些数据将包括轨迹长度、轨迹寿命、轨迹边界框尺寸和体积、平均胶条速度和方差。该分析使用 Syto9 标记细菌的通道计算加权变量,以计算局部(在给定的轨迹边界框内)细胞密度。分析结果计算加权平均速度和方差,以及加权平均和边界框方差的方差。
结果
该方法用于检验 curli 可能赋予 大肠杆菌 和 S 刚性的假设。鼠伤寒生物膜,减少共聚焦显微镜实验期间的珠子移动。当前工具箱用于比较 粪肠球菌 共生型菌株 OG1RF 与 肠道沙门氏菌 血清型鼠伤寒、 大肠杆菌 及其各自的同基因卷曲突变体的材料特性(图 1B 和 补充视频 1、补充视频 2、补充视频 3、补充视频 4、补充视频 5、补充视频 6).生物膜材料特性在刚性(例如,curli 与 eDNA 结合)或带负电荷的珠子与生物膜细胞和基质材料之间的静电和疏水相互作用以及细胞密度方面可能有所不同。
再现性
生物膜工具箱在 VRL-Studio32 中使用 Groovy30 和 Java31 进行编程,实现了模块化工作流程设计,并自动生成所有计算组件的用户界面 (UI)。这允许自动化工作流程,在分析结果时消除实验者无意中引起的偏差。
使用 MSD 确认生物膜中的运动类型
为了使用 Particle Tracker 2D/3D 分析轨迹,可以使用不同的动力学设置来分析不同的拉延筋运动类型。对于这些研究,选择了“布朗运动”(即扩散驱动运动)设置,因为粪肠球菌是一种不运动的细菌,大肠杆菌和沙门氏菌在生物膜中不表达鞭毛,并且实验在没有流动的情况下在封闭系统中进行。这个设置可以通过计算出的珠子的均方位移 (MSD) 来进一步验证。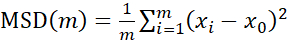 使用定义,其中 m 是轨迹段的数量,可以计算 MSD 在每个轨迹过程中的变化。线性轨迹表示扩散珠运动(图 2A)。使用二次最小二乘拟合,计算生物膜中所有珠子的平均运动模式,显示主导线性顺序并验证被动扩散作为驱动力(图 2A-2F)。
使用定义,其中 m 是轨迹段的数量,可以计算 MSD 在每个轨迹过程中的变化。线性轨迹表示扩散珠运动(图 2A)。使用二次最小二乘拟合,计算生物膜中所有珠子的平均运动模式,显示主导线性顺序并验证被动扩散作为驱动力(图 2A-2F)。
边界框分析。
该工具箱使用 ImageJ Mosaic 和 Particle Tracker 2D/3D 生成轨迹(第 4 步),然后使用自动生物膜分析管道生成有关珠子轨迹的重要数据,这些数据可用于比较生物膜材料特性。通过构建包含轨迹的最小框并测量其体积来测量以 μm3 为单位的边界框体积(图 3)。
粪肠球菌生物膜具有更多的珠子运动,边界框值为 1-6000 μm3(图 3B、3C 和 3D)。结果证实,与在光学玻璃孔底部生长并直接成像(图 3D)的生物膜相比,安装在具有约 25 μm 涂层孔的涂层载玻片上的玻璃盖玻片上看到的运动产生了相同的结果,几乎没有差异。唯一的区别是,在粪肠球菌生物膜的安装盖玻片顶部附近,寿命大于 10 分钟的稳定轨迹,但同时可以记录小的边界框,而在光学底板中,可以记录选定数量的具有更高迁移率的珠子。综上所述,这表明安装载玻片可能已经改变了系统在生物膜顶部的表面张力,而不是安装在盖玻片生物膜中的载玻片,这最终降低了一些珠子在粘度较低的生物膜区域中的流动性(图 3B、3C、3D 和 3I)。属于这一类的轨迹恰好是一个非常小的百分比,即使有如此少量的捕获珠子,安装的盖玻片上粪肠球菌的平均 MSD 也略高于从光学底板生物膜计算的 MSD(图 3)。
S. 斑疹伤寒和大肠杆菌珠轨迹具有较小的边界框体积,为 0-10 μm3(图 3A、3B、3E 和 3F),与同基因 curli 突变体相比,大肠杆菌的边界框为 1-6000 μm3,S 的边界框为 1-5000 μm3。 鼠伤寒(图 3A、3B、3F 和 3H),显示较大的珠子迁移率。这些结果表明,淀粉样蛋白的存在与生物膜的刚度增加相关,并且与视频中缺乏明显的生物膜运动一致。即使在生物膜的低密度区域,边界框体积也始终较小 (0-10 μm3)。这一观察结果与之前的观察结果一致,即 curli 可以存在于生物膜10 的低细胞密度区域。
无法比较肠杆菌科生物膜在光学底板上的行为,因为它们在气液界面上以表膜的形式生长(步骤 1.2.2)。当使用盖玻片时,薄膜在界面处附着在盖玻片上,当取下盖玻片时,薄膜位于盖玻片上,形成一个单一的图像表面。在倾斜生长的光学底板中,在液体仍在孔中的情况下进行成像。这意味着薄膜仍然漂浮在光学底部上方,并使薄膜脱离了 Leica Sp5 等倒置镜的工作深度。去除足够的介质以将生物膜带入显微镜的工作深度会导致样品在 20 分钟的成像过程中变干。
总体而言,这些图证实了补充电影中的视觉观察结果,并与观察到的 MSD 差异一致(图 3I 和 3J)。
轨迹寿命
轨迹寿命测量为记录磁珠的连续帧数(图 3)。
在更粘稠、液体状的 粪肠球 菌生物膜中,所有珠子的轨迹寿命都短于 10 分钟,而 粪肠球 菌生物膜的大多数轨迹在 2-5 分钟之间。然而,在整个成像时间窗口内,可以通过目视检查 粪肠球 菌生物膜来定位具有记录短轨迹寿命的珠子(补充视频 1 和 2)。因此,珠子有可能沿着注册的轨迹移动,间歇性地从生物膜上解离并终止轨迹,并与生物膜重新结合,此时开始新的轨迹。这最终会导致在生物膜中持续存在珠子的情况下缩短轨迹寿命。需要注意的是,使用这种技术,轨迹寿命,尤其是在粘性生物膜中,往往会低估珠子与生物膜相关的总时间。
在 S 中。 鼠伤寒生物膜具有较小的边界框体积,大多数珠子(约 80%)具有 16-20 帧的长轨迹寿命,相当于大约 15-20 分钟的实时(图 3A、3G 和 3H)。与这些相反,同基因 curli 突变生物膜携带更多的移动珠子,边界框体积范围在 1-6000 μm3 (E. coli) 和 1-5000 μm3 (S. 鼠伤寒)(图 3A、3B、3F 和 3H)。然而,与具有 >70% 轨迹的 E . faecalis 生物膜具有大于 10 μm3 的边界框体积相比,肠 杆菌科 生物膜仅记录了 30% 的珠子轨迹,边界框体积超过 10 μm3。尽管 curli 突变生物膜中的总体珠子轨迹寿命较小,但一些轨迹反映了大量的珠子运动和较长的轨迹寿命(图 3H)。这一观察结果表明,这种变化可能对应于不同的生物膜材料特性(如粘弹性)和/或颗粒表面化学变化(如电荷)。
胶条轨迹长度和速度分析
轨迹长度是微珠行进距离的量度,单位为 μm。该测量值与珠子移动速度(以 μm/s 为单位)一致。与较大的边界框体积一致, 粪肠球 菌生物膜中的珠子的轨迹长 10 倍,为 5-20 μm,而含有 curli 的生物膜中的 <4 μm。与较短的轨迹一致(图 4A)。 屎肠杆菌 珠子测得的速度高出 15 倍,大多数珠子的速度在 0.01-0.15 μm/s 之间,而速度<0.006 μm/s(图 4B)。尽管如此,与 粪肠球菌 生物膜相比,curli 突变生物膜测量的总体速度较低且轨迹较短,但与含有亲本菌株的 curli 相比,其轨迹更长,速度更高(图 4A 和 4B)。
值得注意的是,curli30 的纤维晶格状结构可能以各向异性方式影响迁移率,减少 xy 平面的运动并允许增加 z 方向的迁移率(图 2G)。在含有 curli 的生物膜中,属于大约 50 个独特珠子的大轨迹池(大约 800 个)与马赛克粒子跟踪的局限性一致,将这些快速移动的珠子中的每一个计为 x、y 和 z 上的单个珠子。需要更多的研究和软件开发来证实这一观察结果。
微珠运动对细胞密度的依赖性分析
珠子运动对细胞密度的依赖性是通过使用加权平均速度和方差,以及边界框体积的平均值/加权平均值和方差来确定的。Syto9 标记细菌的第二个成像通道用于计算加权速度中的局部细胞密度。通过在每个轨迹边缘的边界框上平均 Syto9 体素数据来计算细胞密度(图 5,右)。因此,磁珠速度可以通过边缘(局部)细胞密度进行加权。有多种类型的染色剂可用于可视化细菌,包括细胞壁、膜和 DNA 含量的染色剂。为了确定细胞密度,选择 Syto9 是因为无论可视化哪个光学 Z 切片,它都能提供最一致的信号。包膜染色剂(细胞壁和膜)将根据 Z 切片的位置给出不同的信号。如果 Z 切片包括细胞的顶部或底部,则信号将比 Z 切片穿过细胞中间时更强,因为 Z 切片仅对细胞的轮廓进行染色。
来自红色通道的珠子轨迹被跟踪 20 帧,其中单个轨迹的最小寿命为 2 帧,最大寿命为 20 帧,有 19 个轨迹段连接帧(图 5)。为了研究微珠迁移率的细胞密度依赖性,确定了每个体素的 GFP 强度(512x512 体素图像中的每个单独测量值)。珠子轨迹每个段周围的细胞密度计算为该段边界框中的局部平均密度。
对于一些生物膜,可以记录具有统计学意义的密度依赖性(图 6),最突出的是生长在玻璃盖玻片上并倒置到多孔载玻片上的粪肠球菌生物膜(图 6A)。相反,生长在 96 孔板底部的粪肠球菌生物膜(图 6B)显示没有密度依赖性。总之,这表明由于安装在多孔载玻片上,高流动性的粪肠球菌生物膜可能会被轻微压缩,这与移动速度更快的珠子数量减少以及那些被困在生物膜顶部的小边界框体积的珠子相对于载玻片一致(图 3C 与图 3D)。沙门氏菌和大肠杆菌生物膜(图 6C 和 6D)及其同基因突变体(图 6E 和 6F)均显示出轻微或无细胞密度依赖性。
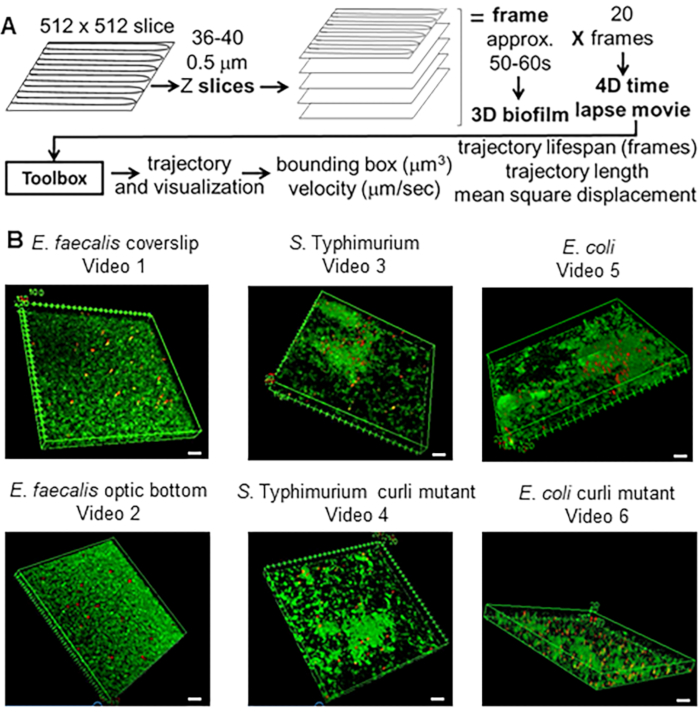
图 1.成像和分析管道(步骤 2-4) (A) 生物膜按照 2.2 中的概述进行成像。使用成像的生物膜(见 3),如 4 中所述生成珠子轨迹。使用轨迹,使用分析工具箱计算相关数据(见 5)(B) 按照步骤 1 中的描述在盖玻片上生长生物膜(粪肠球菌, 视频 1), S。 鼠伤寒(视频 3)、 大肠杆菌 (视频 5)和同基因卷曲突变体(视频 4、视频 6)或光学底部 96 孔板(粪肠杆菌 底部, 视频 2)。白色比例尺为 20 毫米。此图经 (31) 许可转载。 请单击此处查看此图的较大版本。
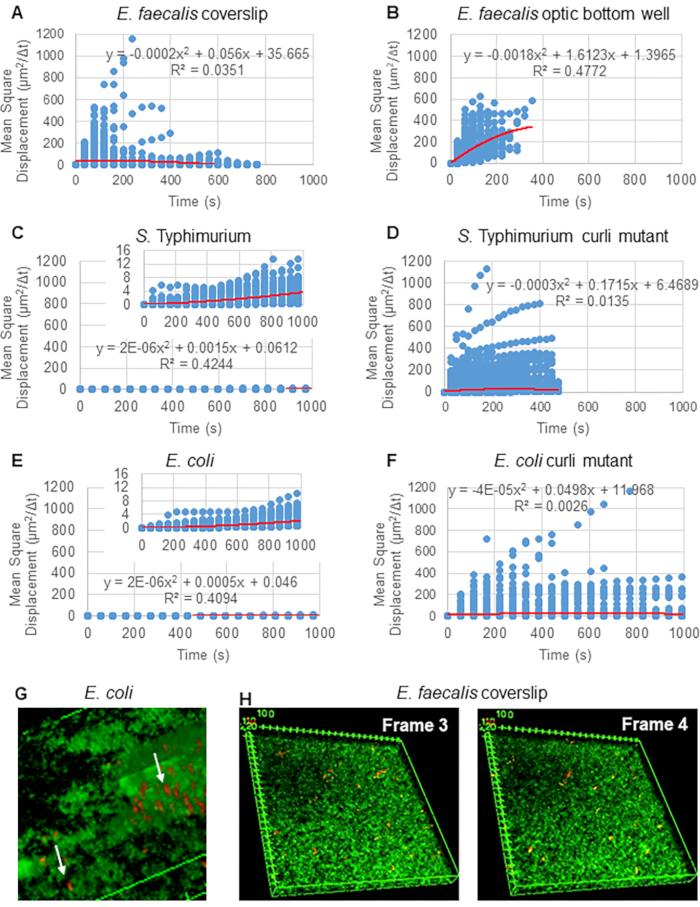
图 2.确定生物膜中的珠子运动本质上是扩散的(布朗运动,参见步骤 5)(AF)MSD 数据显示线性行为(红线)以验证布朗运动。根据步骤 1 生长生物膜 (G) 在 大肠 杆菌和鼠伤寒 沙门氏菌 生物膜中观察到的椭圆珠子运动示例 从 大肠杆菌 4D 生物膜测定的一个框架中获取 (H) 从 粪肠杆菌 光学底井获取的第 3 帧和第 4 帧之间珠子图案发生巨大变化的示例。请注意,生物膜本身会引发一些流动(视频 1 和 2),这使得第 3 帧和第 4 帧看起来方向不同。此图经 (31) 许可转载。 请单击此处查看此图的较大版本。
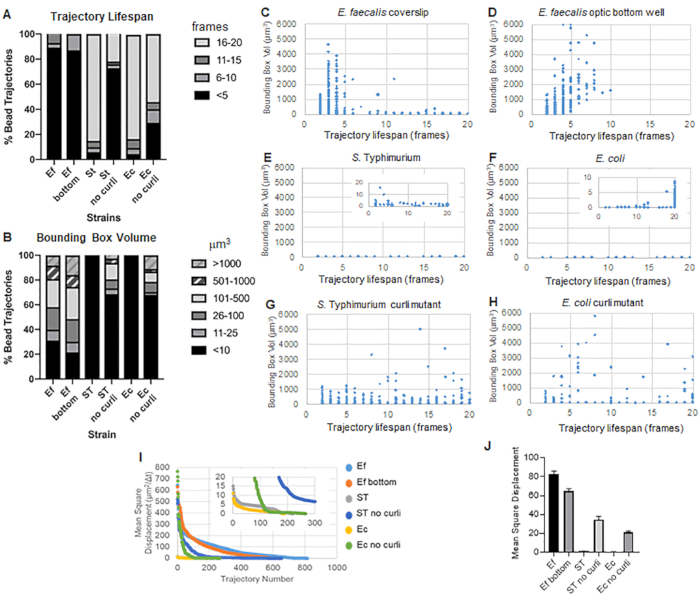
图 3.使用边界框和轨迹寿命分析刚度的差异。 按照步骤 1 中的描述在盖玻片上生长生物膜(粪肠球菌, 视频 1), S。 鼠伤寒(视频 3)、 大肠杆菌 (视频 5)和同基因卷曲突变体(视频 4、视频 6)或光学底部 96 孔板(粪肠杆菌 底部, 视频 2)。轨迹寿命以珠子总轨迹的百分比 (A) 和散点图 (CH) 以及边界框体积(在步骤 5 中计算) (I) 不同生物膜中珠子 MSD 的比较 (H) 每种生物膜的平均 MSD。条形表示数据的 95% 置信区间。此图经 (31) 许可转载。 请单击此处查看此图的较大版本。
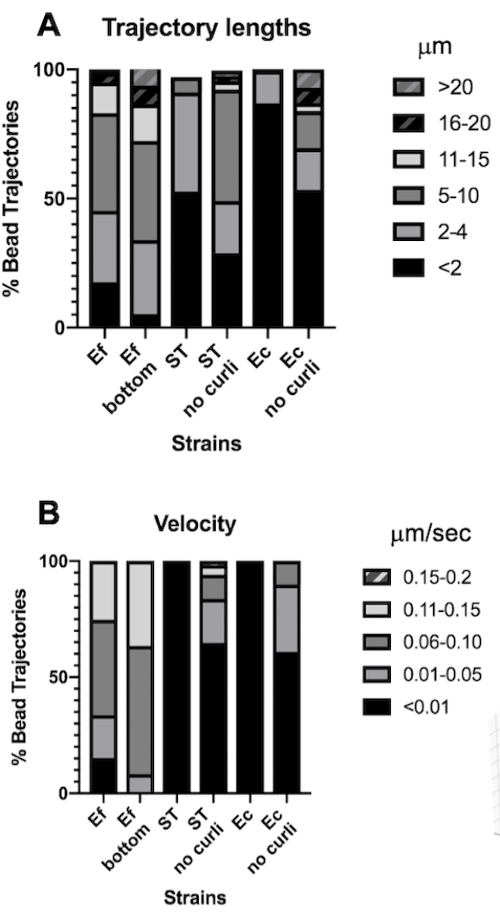
图 4.分析轨迹长度和珠子速度的差异。 按照步骤 1 中的描述生长生物膜。轨迹长度以 μm 为单位,以珠总轨迹 (A) 的百分比表示。速度以 μm/s 表示,以珠总轨迹的百分比 (B) 表示。此图经 (31) 许可改编。 请单击此处查看此图的较大版本。
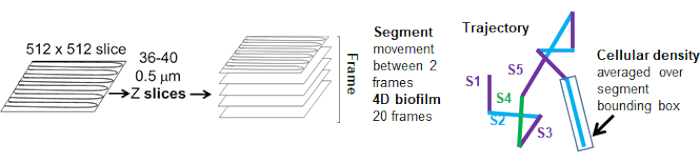
图 5.轨迹段分析概述。 此图经 (31) 许可改编。 请单击此处查看此图的较大版本。
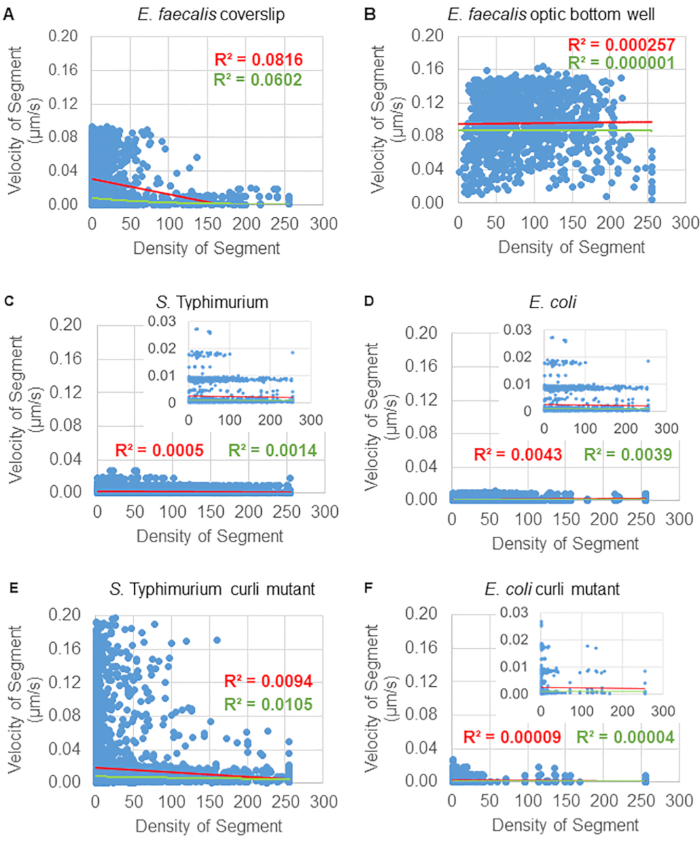
图 6.使用生物膜分析管道研究生物膜密度对珠子速度的影响(步骤 5)。 结果表明,磁珠速度并不完全依赖于细胞密度。按照步骤 1 中的描述生长生物膜。在单个轨迹段的尺度上分析轨迹(图 5)。对于每个片段,以 μm/s 为单位的珠子速度与边界框细胞密度(边界框内每个体素的平均 GFP)作图。红线表示线性回归,绿线表示指数回归轨迹。此图经 (31) 许可转载。 请单击此处查看此图的较大版本。
补充视频 1. 在 1.5 厚的光学玻璃盖玻片上生长的 24 小时粪肠球菌 OG1RF 生物膜的 4D 视频。4D 延时视频是使用显微镜以 512x512 分辨率生成的。通过以 0.5 μm 的步长对生物膜的约 20 μm 厚区域进行成像,生成 40 张图像的 Z 系列。每个 Z 系列都是一帧,需要 50-60 秒来捕捉。捕获一系列 20 个连续帧以生成 4D 视频。视频播放速度约为 120 倍。视频代表至少 6 个独立实验。请点击此处下载此视频。
补充视频 2.在 96 孔光学底板上生长的 24 小时粪肠球菌 OG1RF 生物膜的 4D 视频。4D 延时视频是使用显微镜以 512x512 分辨率生成的。通过以 0.5 μm 的步长对生物膜的约 20 μm 厚区域进行成像,生成 40 张图像的 Z 系列。每个 Z 系列都是一帧,需要 50-60 秒来捕捉。捕获一系列 20 个连续帧以生成 4D 视频。视频播放速度约为 120 倍。视频代表 3 个独立实验。请点击这里下载此内容 视频.
补充视频 3. 肠道沙门氏菌血清型 鼠伤寒生物膜 ATCC 14028 在 1.5 厚的光学玻璃盖玻片上生长 6-7 天的 4D 视频。4D 延时视频是使用显微镜以 512x512 分辨率生成的。通过以 0.5 μm 的步长对生物膜的约 20 μm 厚区域进行成像,生成 40 张图像的 Z 系列。每个 Z 系列都是一帧,需要 50-60 秒来捕捉。捕获一系列 20 个连续帧以生成 4D 视频。视频播放速度约为 120 倍。视频代表 3 个独立实验。请点击这里下载此内容 视频.
补充视频 4. 肠道沙门氏菌血清型 鼠伤寒生物膜 ATCC 14028 curli (csgBA) 突变体的 4D 视频在 1.5 厚的光学玻璃盖玻片上生长 6-7 天。4D 延时视频是使用显微镜以 512x512 分辨率生成的。通过以 0.5 μm 的步长对生物膜的约 20 μm 厚区域进行成像,生成 40 张图像的 Z 系列。每个 Z 系列都是一帧,需要 50-60 秒来捕捉。捕获一系列 20 个连续帧以生成 4D 视频。视频播放速度约为 120 倍。视频代表 3 个独立实验。请点击这里下载此内容 视频.
补充视频 5. 大肠杆菌 UTI89 在 1.5 厚的光学玻璃盖玻片上生长 6-7 天的 4D 视频。4D 延时视频是使用显微镜以 512x512 分辨率生成的。通过以 0.5 μm 的步长对生物膜的约 20 μm 厚区域进行成像,生成 40 张图像的 Z 系列。每个 Z 系列都是一帧,需要 50-60 秒来捕捉。捕获一系列 20 个连续帧以生成 4D 视频。视频播放速度约为 120 倍。视频代表 3 个独立实验。请点击这里下载此内容 视频.
补充视频 6.4D 视频 大肠杆菌 UTI89 curli (csgBA) 突变体在 1.5 厚的光学玻璃盖玻片上生长 6-7 天。 4D 延时视频是使用显微镜以 512x512 分辨率生成的。通过以 0.5 μm 的步长对生物膜的约 20 μm 厚区域进行成像,生成 40 张图像的 Z 系列。每个 Z 系列都是一帧,需要 50-60 秒来捕捉。捕获一系列 20 个连续帧以生成 4D 视频。视频播放速度约为 120 倍。视频代表 3 个独立实验。请点击这里下载此内容 视频.
讨论
关键步骤和故障排除
该技术的最大挑战是使用带有非常粘稠的生物膜(如 粪肠球菌) 的安装盖玻片。需要小心准确地将盖玻片放在多孔载玻片上,无需重新定位。在密封步骤中,需要小心防止向下推盖玻片或意外地将其推/滑过载玻片表面。任何移动或压力都可能产生表面张力并阻止粘性生物膜的运动。如果可能,通过将光学底部井上的生物膜成像与盖玻片支架来比较生物膜材料特性,这将允许技术评估。如果作正确,盖玻片安装非常类似于 粪肠球菌光学底板中的生物膜。
此外,当使用已安装的盖玻片时,应避免对生物膜的界面进行成像,盖玻片位于底部或载玻片位于最顶部。当使用倒置的内窥镜时,盖玻片位于底部,生物膜底部可能会有被困的珠子靠在盖玻片上。这些珠子穿过生物膜,即使在轻轻洗涤后也会被困在盖玻片上。它们的 x、y 和 z 坐标为 0,边界框坐标为 0。然而,对于某些应用,例如检查处理后的生物膜完整性,这些数据点可以用作工具。珠子穿过生物膜的厚区域到达盖玻片底部的能力可用于评估处理后的生物膜完整性(与 Tükel 实验室合作准备的手稿)。在生物膜的顶部,在像 E. faecalis 这样的粘性生物膜中,我们有一些证据表明盖玻片施加了压实。这限制了一些微珠在载玻片界面处的移动,并可能给微珠运动分析带来了一些密度依赖性。
洗涤步骤对于生物膜是必要的,因为生长培养基在绿色通道中具有很强的自发荧光。我们选择使用多余的磁珠,并通过洗涤去除未结合的磁珠,以最大限度地提高相关磁珠,从而获得观察区域的最准确表征。
获得所需数据集所需的微珠和洗涤次数需要根据经验确定。生物膜中存在过多的珠子会产生难以分析的不可能的大数据集。珠子太少的存在不会产生生物膜环境的全面采样。然而,控制添加的珠子数量(1 mL PBS 中2x10 7 个珠子)和使用洗涤步骤,导致与生物膜相关的珠子数量 (40-140) 相对一致,具体取决于其结构、空间排列和组成。
在研究具有混合粘性和刚性区域的生物膜时,随着时间的推移,珠子可能会被困在刚性区域。在这种情况下,需要在添加珠子后立即开始成像。这通常无法使用盖玻片完成,但需要光学底板或流通池,在添加磁珠和洗涤步骤后可以立即进行成像。
修改和未来应用
使用微流控设备。 在我们的研究中,为研究肠杆菌科生物膜建立的最佳条件要求生物膜在气液界面处生长为表膜。这限制了光学底板和微流体设备在研究中的使用。然而,当生物膜形成条件允许时,生物膜可以在微流体室或流动池中生长。然后可以清洗生物膜,并通过微流体装置引入珠子,对生物膜的破坏最小。
在生物膜生长过程中添加珠子。 我们选择将多余的珠子添加到生物膜中,然后通过温和洗涤去除未结合的珠子,以优化分析过程中存在的珠子数量。在粘性粪 肠球菌 生物膜中,珠子可能在 20 分钟的成像时间内解离并重新结合。如果在生物膜生长过程中的不同时间添加少量珠子,则有可能将珠子捕获在生物膜中,从而更准确地表征更粘稠的生物膜中的生物膜运动。
选择要映像的区域。对于材料特性的研究,最好选择生物膜的厚区域和薄区域。然而,在研究处理过的生物膜的材料特性变化时,可以对厚的汇合区域进行成像,以确定这些区域中粘弹性和珠子渗透的变化。在这种情况下,寻找穿透生物膜并最终被困在盖玻片上的珠子是生物膜破坏的有用措施。
流下成像。使用光学玻璃流通池或微流体设备,可以对流动下生物膜中珠子或细菌的运动进行成像。这可以通过不同的方式完成。可以通过将珠子注射到整个腔室中,然后进行短暂孵育以使珠子与生物膜结合来完成。可以通过洗涤去除未结合的珠子,并在有或没有流动的情况下对生物膜进行成像。相反,可以将少量珠子引入腔室的一侧,并且可以在流动下跟踪它们穿过生物膜和在生物膜中的移动。使用 flow 时,在选择 Mosaic 珠子跟踪设置(步骤 4.5)时需要谨慎。在目前的研究中,动力学设置为布朗。MSD 计算证实,运动可能是扩散性的,因此 Brownian 是合适的设置。
基质染色。在目前的研究中,用 Syto9 染色检查的是细胞密度,而不是生物膜结构密度。例如,淀粉样蛋白的存在可能会增加生物膜基质材料的密度。运动对淀粉样蛋白密度的依赖性可以通过使用荧光基质染色剂代替 Syto9 来确定。
荧光标记的细菌。 荧光标记的细菌可用于追踪外源细菌通过生物膜的运动(例如,含质粒的细菌)。荧光标记的细菌(如 肠球菌 )的挑战在于它们会形成单链、双球菌和短链,这使得准确追踪细菌的能力变得复杂。如果细菌具有单细胞形态,这个过程会更容易。
局限性
轨迹可视化和拼接的限制。
该方法的一个限制是轨迹可视化和拼接。重建和分析的轨迹由 x、y、z 点坐标组成,其中后续点定义这些点之间的线性路径。这种分段线性轨迹的可视化可以通过各种工具来实现。一种方法是将 Python 和 Jupyter 笔记本与 Python 插件 Pandas 和 Matplotlib 一起使用。虽然可以在最初发表该技术的 Journal of Bacteriology 文章中可视化单个现有轨迹34,但在未来的研究中仍然存在重大限制。
目前,重建轨迹的数量大于生物膜中的珠子数量,这意味着多个轨迹可能对应于一个珠子。这可能是由一帧中的微弱共聚焦信号引起的,其中 Mosaic 将终止轨迹并启动第二帧。这可能记录为一个珠子的多个较短轨迹,尤其是在粘度较低的生物膜中。轨迹数量多的另一个原因是缺少轨迹拼接。特别是在 粪肠球 菌光学底孔生物膜中,珠子在成像过程中在视觉上与生物膜保持关联(补充视频 2)。然而,没有超过 10 个轨迹,超过 90% 的轨迹的寿命为 5 帧或更短(图 3D)。如果该软件仅用于分析超过定义长度的轨迹(例如,在追踪能够转移质粒的细胞时),则可以自动从数据集中删除较短的轨迹。但是,还有其他目的,拼接轨迹可能非常重要。最后,由于沿 Z 方向的快速移动导致椭圆形珠子,无法将珠子的快速移动作为单个轨迹进行跟踪,可能会导致肠杆菌科生物膜中出现更多轨迹(图 2G)。快速各向异性运动扰乱缝合轨迹的可能性对于研究肠杆菌科中卷曲淀粉样蛋白基质的影响非常重要。
意义
开发了一种计算工作流程来研究微珠轨迹,以比较 3D 生物膜的材料特性。该工作流程使研究人员能够确定可用于异质生物膜中流体动力学计算建模的关键参数。借助这个开源微珠评估器,可以研究细菌淀粉样蛋白卷曲对材料性能的影响,显示卷曲导致生物膜基质刚度增加。在更一般的背景下,评估器可用于研究由生物膜处理或不同环境条件(如流动)引起的生物膜结构变化。例如,该工具正在与 Tükel 实验室(LKSOM 天普大学)合作,用于分析单克隆抗体治疗对生物膜结构破坏的影响。微珠评估器工具箱使用 VRL-Studio 以模块化方式完全适应和扩展,以进一步增强和扩展其功能。
披露声明
作者没有什么可披露的。
致谢
GQ 和 BAB 实验室的工作没有从公共、商业或非营利部门的任何资助机构获得任何特定资助。作者感谢 Isaac Klapper 博士(天普大学数学系)的有益讨论和 Çagla Tükel(天普大学微生物学和免疫学系)在包含该技术的初始出版物中提供的肠杆菌科专业知识。
材料
| Name | Company | Catalog Number | Comments |
| 96-well plates, No. 1.5 Uncoated Coverslip, 5 mm Glass Diameter | MatTek | P96G1.55F | |
| Fisherbrand Cover Glasses: Circles | Fisher Scientific | 12-293-232P | 1.5 optic glass coverslip |
| Invitrogen Syto 9 Green Fluorescent Nucleic Acid Stain | Invitrogen | S34854 | |
| Molecular Probes FluoSpheres Carboxylate-modified Microspheres, 1 um, crimson fluorescent (625/645) | Molecular Probes | F8816 |
参考文献
- Huang, R., Li, M., Gregory, R. L. Bacterial interactions in dental biofilm. Virulence. 2, 435-444 (2011).
- Nadell, C. D., Drescher, K., Wingreen, N. S., Bassler, B. L. Extracellular matrix structure governs invasion resistance in bacterial biofilms. ISME Journal. 9, 1700-1709 (2015).
- Billings, N., Birjiniuk, A., Samad, T. S., Doyle, P. S., Ribbeck, K. Material properties of biofilms - key methods for understanding permeability and mechanics. Reports on Progress in Physics. 78, 036601 (2015).
- Araújo, G. R. d. e. S., Viana, N. B., Gómez, F., Pontes, B., Frases, S. The mechanical properties of microbial surfaces and biofilms. The Cell Surface. 5, 100028 (2019).
- Tallawi, M., Opitz, M., Lieleg, O. Modulation of the mechanical properties of bacterial biofilms in response to environmental challenges. Biomaterials Science. 5, 887-900 (2017).
- Tursi, S. A., Tükel, &. #. 1. 9. 9. ;. Curli-Containing Enteric Biofilms Inside and Out, Matrix Composition, Immune Recognition, and Disease Implications. Microbiology and Molecular Biology Reviews. 82, (2018).
- Dueholm, M. S., Albertsen, M., Otzen, D., Nielsen, P. H. Curli functional amyloid systems are phylogenetically widespread and display large diversity in operon and protein structure. PLoS One. 7 (12), 51274 (2012).
- Hung, C., et al. Escherichia coli biofilms have an organized and complex extracellular matrix structure. mBio. 4, 00645 (2013).
- Kikuchi, T., Mizunoe, Y., Takade, A., Naito, S., Yoshida, S. Curli fibers are required for development of biofilm architecture in Escherichia coli K-12 and enhance bacterial adherence to human uroepithelial cells. Microbiology and Immunology. 49, 875-884 (2005).
- Gallo, P. M., et al. Amyloid-DNA Composites of Bacterial Biofilms Stimulate Autoimmunity. Immunity. 42, 1171-1184 (2015).
- Serra, D. O., Richter, A. M., Hengge, R. Cellulose as an architectural element in spatially structured Escherichia coli biofilms. Journal of Bacteriology. 195, 5540-5554 (2013).
- Oh, Y. J. Characterization of curli A production on living bacterial surfaces by scanning probe microscopy. Biophysical Journal. 103, 1666-1671 (2012).
- Lembré, P., Di Martino, P., Vendrely, C. Amyloid peptides derived from CsgA and FapC modify the viscoelastic properties of biofilm model matrices. Biofouling. 30, 415-426 (2014).
- Oh, Y. J., et al. Curli mediate bacterial adhesion to fibronectin via tensile multiple bonds. Scientific Reports. 6, 33909 (2016).
- Vidakovic, L., Singh, P. K., Hartmann, R., Nadell, C. D., Drescher, K. Dynamic biofilm architecture confers individual and collective mechanisms of viral protection. Nature Microbiology. 3, 26-31 (2018).
- Tursi, S. A., et al. Salmonella Typhimurium biofilm disruption by a human antibody that binds a pan-amyloid epitope on curli. Nature Communications. 11, 1007 (2020).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15 (8), 1007978 (2019).
- Taglialegna, A., et al. The biofilm-associated surface protein Esp of Enterococcus faecalis forms amyloid-like fibers. Npj Biofilms and Microbiomes. 6, 15 (2020).
- Gour, S., Kumar, V., Rana, M., Yadav, J. K. Pheromone peptide cOB1 from native Enterococcus faecalis forms amyloid-like structures, A new paradigm for peptide pheromones. Journal of Peptide Science. 25, 3178 (2019).
- Stoodley, P., Debeer, D., Lewandowski, Z. Liquid flow in biofilm systems. Applied and Environmental Microbiology. 60, 2711-2716 (1994).
- Birjiniuk, A., et al. Single particle tracking reveals spatial and dynamic organization of the E. coli biofilm matrix. New Journal of Physics. 16, 085014 (2014).
- Chew, S. C., et al. Dynamic remodeling of microbial biofilms by functionally distinct exopolysaccharides. mBio. 5, 01536 (2014).
- Cao, H., et al. Revealing region-specific biofilm viscoelastic properties by means of a micro-rheological approach. Npj Biofilms and Microbiomes. 2, 5 (2016).
- Galy, O., et al. Mapping of bacterial biofilm local mechanics by magnetic microparticle actuation. Biophysical Journal. 103, 1400-1408 (2012).
- Rogers, S. S., vander Walle, C., Waigh, T. A. Microrheology of bacterial biofilms in vitro, Staphylococcus aureus and Pseudomonas aeruginosa. Langmuir. 24, 13549-13555 (2008).
- Hart, J. W., Waigh, T. A., Lu, J. R., Roberts, I. S. Microrheology and Spatial Heterogeneity of Staphylococcus aureus Biofilms Modulated by Hydrodynamic Shear and Biofilm-Degrading Enzymes. Langmuir. 35 (9), 3553-3561 (2019).
- van Merode, A. E. J., van der Mei, H. C., Busscher, H. J., Krom, B. P. Influence of culture heterogeneity in cell surface charge on adhesion and biofilm formation by Enterococcus faecalis. Journal of Bacteriology. 188, 2421-2426 (2006).
- Tariq, M., Bruijs, C., Kok, J., Krom, B. P. Link between Culture Zeta Potential Homogeneity and Ebp in Enterococcus faecalis. Applied and Environmental Microbiology. 78, 2282-2288 (2012).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ, 25 years of image analysis. Nature Methods. 9, 671-675 (2012).
- . Oracle.com Available from: https://docs.oracle.com/en/java (2021)
- Hoffer, M., Poliwoda, C., Wittum, G. Visual reflection library, a framework for declarative GUI programming on the Java platform. Computing and Visualization in Science. 16, 181-192 (2013).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15, 1007978 (2019).
- Malhotra, K., et al. Development of a New Bead Movement-Based Computational Framework Shows that Bacterial Amyloid Curli Reduces Bead Mobility in Biofilms. Journal of Bacteriology. 202, 00253 (2020).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。