Method Article
생물막 재료 이질성의 3차원 분석을 위한 비드 이동 기반 계산 프레임워크
요약
이질적인 박테리아 생물막을 통해 1μm 카르복실레이트 비드의 이동 패턴을 분석하고 정량화하는 방법을 설명합니다. 이동 패턴의 비교는 생물막의 재료 특성의 차이를 정량화하는 데 사용할 수 있습니다.
초록
박테리아 생물막의 재료 특성의 차이는 서로 다른 박테리아 종의 생물막, 동일한 종 내에서 다른 성장 조건 하에서 그리고 매트릭스 변형 분자로 처리 한 후에 관찰되었습니다. 3D 생물막의 재료 특성을 더 잘 정량화하기 위해 실험 및 계산 워크플로우를 개발 및 적용하여 Enterococcus faecalis, Salmonella enterica serotype Typhimurium 및 Escherichia coli 생물막 간의 차이점과 Enterobacteriaceae 생물막에 대한 강성을 확인하는 데 있어 amyloid curli의 역할을 조사했습니다. 생물막에서 1μm 카르복실레이트 비드의 시공간 역학은 20분 동안 20μm 3D 생물막에서 추적되었습니다. 4D 이미지 스택은 비드 움직임의 3D 궤적 데이터를 생성하기 위해 ImageJ의 Mosaic 플러그인을 사용하여 처리되었습니다. 이 궤적 데이터는 새로 개발된 Bead Evaluator 도구 상자로 분석되었으며, 여기서 궤적 수명, 비드 속도, 궤적을 따른 셀 밀도 및 바운딩 박스 정보를 포함한 이동 데이터가 계산되어 csv 파일에 저장되었습니다. 이 백서에서는 실험 설정 및 이미지 기록부터 비드 궤적 계산 및 분석에 이르는 워크플로우를 제시합니다. curli를 함유한 생물막의 구조는 curli 돌연변이 및 장구균 생물막보다 더 안정적인 bead interaction과 더 적은 bead movement를 가져왔습니다. 비드 속도와 궤적 경계 상자 부피를 측정할 때 비드 이동은 세포 밀도에 크게 의존하지 않는 것으로 보였으며, 이는 생물막의 다른 재료 특성이 비드 역학을 제어한다는 가설을 뒷받침합니다. 이 기술은 매트릭스 변형 처리 전후의 바이오필름뿐만 아니라 다양한 매트릭스 조성의 바이오필름의 차이를 정량화하는 데 널리 적용할 수 있습니다.
서문
박테리아 생물막은 인간 미생물총(microbiota)의 일부로 어디에나 존재하며 분자와 지속적으로 상호 작용합니다. 이 분자는 1nm 항생제와 1-3mm 박테리아에서 위장관의 더 큰 섬유 입자에 이르기까지 크기가 다양합니다. 단일 또는 다종 생물막의 조성은 재료 특성에 영향을 미치고 따라서 생물막을 통한 입자의 이동 패턴에 영향을 미칩니다 1,2,3,4,5. 한 가지 예는 보존된 섬유소 교차 베타 시트구조 6을 가진 박테리아 아밀로이드입니다. Amyloid curli는 Escherichia coli 및 Salmonella enterica serotype Typhimurium과 같은 장 세균에 의해 발현되며 유전자는 여러 다른 박테리아 문에서 검출되었습니다7. 생물막의 다양한 재료 특성은 curli 8,9의 영향을 받습니다. Curli는 세포외 DNA(eDNA) 및 셀룰로오스10,11과 같은 기질의 다른 구성 요소와 직접 상호 작용합니다. Curli는 세포를 둘러싸고 세포막 강성(cellmembrane rigidity)12 및 생물막(biofilm) 13의 전반적인 점탄성 특성에 영향을 미칩니다. Curli는 피브로넥틴(fibronectin)에 결합하여 인장 강도를 증가시켜 강력한 유리 표면 부착을 증가시킵니다14. 유입되는 박테리오파지는 컬리(curli)에 결합하여 파지가 생물막으로 침입하는 것을 제한합니다15.
약 20μm 두께의 엔테로코커스 패칼리스(Enterococcus faecalis), 대장균(E. coli) 및 S를 분석하기 위해 다중 테스트 코팅된 웰 슬라이드를 사용하는 경우. 컨포칼 현미경을 사용한 Typhimurium 생물막, E. coli, S의 명확한 차이점. Typhimurium 10,16 및 E. faecalis 생물막(현재 연구)을 관찰할 수 있습니다. Enterobacteriaceae 종의 생물막은 강성이 높고 세포 밀도가 낮은 영역은 이미지화하기 쉬웠지만, 라인 및 프레임 평균화를 사용하여 E. faecalis 생물막의 선명한 고해상도 사진을 얻으려면 이미징 과정에서 세포 안정성을 위한 충분한 표면 장력을 유도하기 위해 슬라이드에 압력을 가해야 했습니다. 컬리(curli)와 같은 박테리아 아밀로이드는 고도로 정렬된 구조를 형성하는데, 이는 상대적으로 단단할 수 있음을 시사한다17. 이것은 아밀로이드 컬리가 대장균과 S에 경직을 유발할 수 있다는 가설에 동기를 부여했습니다. 티피무리움 생물막. E. faecalis가 연구된 조건에서 아밀로이드를 발현하고 있다는 명확한 증거는 없었습니다. E. faecalis의 더 많은 병원성 균주와 관련된 필린 유전자인 단백질 Esp는 최근 아밀로이드 구조를 생성하는 것으로 나타났습니다18; 그러나 blastn 및 blastp 검색을 사용하여 이 유전자는 이러한 연구에 사용된 E. faecalis 공생 유형 균주 OG1RF에서 검출되지 않았습니다. OG1RF에 의해 생성된 페로몬 cOB1은 아밀로이드와 유사한 구조를 형성할 수 있습니다19. 그러나, 주어진 생물막 성장 조건과 이전에 S에 사용되던 아밀로이드 검출 방법으로는. E. faecalis, OG1RF 아밀로이드에서10을 염색하는 Typhimurium amyloids는 검출할 수 없습니다(데이터는 표시되지 않음). 새로운 4차원(4D) 이미지 기법은 점성이 있는 E. faecalis, E. coli 및 S. Typhimurium 간의 전반적인 재료 특성을 비교하고 S의 아밀로이드 돌연변이를 사용하여 Enterobacteriaceae 생물막에 대한 아밀로이드의 기여도를 결정하기 위해 개발되었습니다. 티피무리움과 대장균.
과거에는 미세유 변학20,21,22,23,24,25을 사용하여 2차원(2D)으로 생물막의 재료 특성을 분석하는 데 형광 비드가 성공적으로 사용되었습니다. 이는 생물막(26)의 다양한 깊이에서 2D 광학 슬라이스를 연구함으로써 3차원 생물막에 적용할 수 있습니다. 현재 기술은 4D 모델링에 사용하기 위해 시간이 지남에 따라 3D에서 1μm 마이크로 스케일 비드를 추적하기 위해 개발되었습니다. 그 근거 중 하나는 4D 모델링을 사용하여 위장 미생물군 군집을 통한 플라스미드의 이동을 이해한다는 중요한 개념이었습니다. 1 μm 직경의 형광 하전을 띤 카르복실레이트 비드를 사용했는데, 이는 크기와 전하가 플라스미드 이동 및 유지를 위해 선택된 모델 유기체인 E. faecalis와 잘 일치하기 때문입니다27, 28. 생물막의 물리적 특성을 정량화하기 위한 4D 분석이 개발되었습니다(그림 1A). 고안된 방법론에서는 비드를 생물막에 첨가하고 10-20분 동안 10-20μm 두께의 생물막을 통해 시공간 궤적을 기록했습니다. 그런 다음 3D의 비드 궤적은 새로 개발된 도구 상자를 사용하여 궤적 길이, 비드 속도, 궤적 경계 상자 부피(궤적을 포함하는 최소 상자) 및 경계 상자 세포 밀도 측면에서 정량화되었습니다. 다음 프로토콜을 사용하여 박테리아 및 비드 함유 생물막의 4D 이미지 데이터를 생성하고, ImageJ29 및 플러그인 Mosaic으로 데이터를 전처리하고, Bead Evaluator 도구 상자로 비드 궤적을 분석할 수 있습니다.
이 기술은 재료 특성을 검사하고 입자 및 박테리아의 움직임을 3차원으로 추적하기 위한 여러 응용 분야가 있습니다. 예를 들어, 이 기술의 초기 버전은 생물막의 구조적 무결성에 대한 curli에 대한 단클론 항체의 효과를 특성화하는 데 사용되었습니다16. 정식 버전에는 생물막 재료 특성에 대한 보다 자세한 분석을 제공하는 여러 도구가 있으며 생물막에 대한 단클론 항체 처리의 효과를 조사하는 데 계속 사용되고 있습니다. 서로 다른 전하를 가진 입자를 사용하여 생물막의 물질 전하 특성과 서로 다른 매트릭스 조성을 가진 생물막을 통한 입자의 이동을 검사할 수 있습니다. 이는 유동이 없는 생물막에서 관찰된 비드의 이동을 담당하는 재료 특성을 밝히는 2D 미세유변학의 결과를 비교하는 데 사용할 수 있습니다. 이 기술은 또한 다른 생물막 조성의 영역을 가진 혼합 종 생물막에 사용될 수 있습니다. 바이오필름은 미세유체 장치 및 플로우 셀에서 플로우 하에서 라이브 이미징을 수행하여 정적 바이오필름과 유동 바이오필름 사이의 재료 특성 변화와 입자 이동에 대한 흐름의 영향을 조사할 수 있습니다. 이 기술은 또한 형광 표지된 박테리아에 적용하여 생물막 커뮤니티를 통한 외인성 박테리아의 움직임을 특성화할 수 있습니다. 형광 표지된 donor bacteria, fluorescently labeled recipient bacteria 및 fluorescently labeled plasmids의 세 가지 색상을 사용하여 plasmid의 이동, 도킹 및 이동을 추적할 수 있습니다.
프로토콜
1. 생물막 준비
참고: 분석을 위한 생물막은 광학 유리 표면에 생물막이 형성될 수 있는 모든 방법을 사용하여 성장할 수 있습니다. 생물막 구조는 프로토콜의 세척 및/또는 장착 단계 중에 구조가 중단되지 않도록 광학 표면에 충분히 부착되어야 합니다. 아래에서는 24웰 플레이트에서 96웰 광학 바닥 플레이트와 12mm 유리 커버슬립에 대한 기술을 설명합니다. 다른 옵션으로는 다양한 크기의 광학 바닥 플레이트와 흐름이 있거나 없는 광학 흐름 챔버가 있습니다.
- 생물막 설정
- 플레이트의 웰에 박테리아 성장 배지를 추가합니다. 이 연구에서는 E. 예를 들어, 24웰 플레이트에 2mL의 Todd-Hewitt(TH)를 추가하고 0.4mL의 TH를 광학 바닥 96웰 플레이트에 추가합니다. S의 경우. 티피무리움(Typhimurium), 대장균(E. coli ) 및 동인성 csgAB curli 돌연변이체는 0.7mL의 무염 루리아 육수(LB)를 웰에 첨가합니다. 광학 바닥 챔버를 사용하는 경우 1.2단계로 진행합니다.
- 페트리 접시에 12mm #1.5 유리 광학 커버슬립을 놓고 에탄올로 덮습니다.
- 집게를 사용하여 커버슬립을 제거하고 불꽃을 사용하여 남은 알코올에 불을 붙입니다. 알코올이 타도록 하십시오. 불꽃을 사용하여 불을 붙이기만 하면 됩니다. 커버슬립은 깨질 수 있으므로 화염 속에 넣지 마십시오. 균열을 방지하기 위해 덮개 슬립을 우물에 넣기 전에 10-20초 동안 식히십시오.
- 매체가 들어 있는 웰에 커버 슬립을 비스듬히 놓아 매체 위에 놓이지 않도록 합니다. 커버슬립을 마른 우물에 넣지 말고 중간을 넣으면 커버슬립이 우물 바닥에 달라붙을 수 있습니다.
- 멸균 피펫 팁을 사용하여 커버슬립을 매체가 들어 있는 웰 바닥으로 조심스럽게 밀어 넣습니다.
참고: 광학 유리 커버슬립 또는 광학 바닥판의 두께(#1 또는 #1.5)를 컨포칼 현미경 광학 장치의 두께와 일치시키는 것을 잊지 마십시오.
- 생물막 성장을 위한 적절한 조건에서 생물막을 배양합니다. 이 연구에서는 37°C에서 E. faecalis 생물막을 정적 배양으로 성장시키고 30°C에서 E. coli 생물막을 호기적으로 성장시킵니다.
- 이 연구에서는 아침과 이른 저녁에 중간 스위치를 사용하여 2일 동안 E. faecalis 생물막을 성장시킵니다. 생물막이 손상되거나 빠지는 것을 방지하려면 플레이트를 조심스럽게 기울이십시오. 피펫 팁을 웰의 아래쪽 가장자리 근처에 놓고 매체를 천천히 꺼냅니다. 같은 방식으로 새 배지의 첫 번째 mL를 추가합니다. 두 번째 mL를 배지/웰 계면 근처에 천천히 추가합니다.
- 이 연구에서는 최적의 컬리 생산을 위해 성장 조건을 사용합니다. 성장 S. 28°C에서 6-8일 동안 Typhimurium 생물막을 비스듬히 배양하여 생물막이 슬라이드의 약 2/3 지점에 부착된 다음 공기-액체 계면에서 펠리클로 자랄 수 있도록 했습니다. 이 작업은 중간 변경 없이 수행되었습니다. 매체가 마르는 것을 방지하려면 24웰 플레이트를 물 팬이 있는 챔버에 놓습니다.
2. 4D 이미징
- 생물막 마운트의 준비
- PBS에서 Crimson 1μm 카르복실레이트 FluoSpheres 비드 1:50을 희석합니다(PBS 1mL에 2 x 107 비드). 광학 하단 챔버를 사용하는 경우 1 μL - 300 μL의 비드 제제로 희석하여 Syto9을 추가합니다.
- (선택 사항) 성장 배지에 자가형광이 있는 경우 생물막을 세척하여 성장 배지의 흔적을 제거합니다. 이 실험에서는 1mL의 PBS로 두 번 세척하고 플레이트를 조심스럽게 기울이고 피펫 팁을 웰의 하단 가장자리 근처에 놓고 배지를 천천히 빼냅니다. 팁을 아래쪽 가장자리 근처에 놓고 PBS를 추가하고 우물을 천천히 채웁니다. 이 기법은 24웰 플레이트의 커버슬립 외에도 광학 바닥 웰에서 사용합니다.
- 매체 또는 PBS를 제거합니다. 2.1.1에서 준비된 희석된 크림슨 비드를 생물막에 첨가합니다. 이 연구에서는 커버슬립에 1mL의 비드(2x107)를 추가하고 0.2mL의 비드(4x106) 및 Syto9를 96웰 광학 바닥 플레이트에 추가합니다.
- 비드 결합을 허용하기 위해 실온에서 1분 동안 배양합니다.
- 비드를 제거하고 PBS로 바이오필름을 한 번 부드럽게 세척하여 연결되지 않은 비드를 제거합니다. 이 연구에서 커버슬립의 경우 1mL의 PBS로 부드럽게 세척하고 1mL PBS로 웰을 다시 채운 다음 2.1.6으로 진행합니다. 96웰 광학 바닥 플레이트의 경우 0.2mL의 PBS로 바이오필름을 부드럽게 세척하고 0.2mL의 PBS로 웰을 다시 채웁니다. 이제 광학 바닥 챔버를 이미지화할 준비가 되었으므로 2.2단계로 진행합니다.
- (커버슬립을 사용하는 경우) 1μL의 Syto9(녹색 형광 DNA 염색, 제조업체의 지침에 따라 희석됨)를 코팅된 10웰 다중 테스트 슬라이드의 웰 중앙에 추가합니다. 이 코팅된 커버슬립의 깊이는 23-25μm입니다.
- (커버슬립을 사용하는 경우) 알코올 화염 살균 겸자를 사용하여 우물에서 커버슬립을 조심스럽게 제거하고 Syto9 방울이 들어 있는 우물에 뒤집습니다. PBS 1mL에 커버슬립을 그대로 두면 웰에서 더 쉽게 제거할 수 있고 생물막에서 연관되지 않은 비드를 씻어내는 데 도움이 됩니다.
- (커버슬립을 사용하는 경우) 커버슬립을 미끄러지거나 누르지 않고 손톱 광택제로 커버슬립을 조심스럽게 밀봉하면 표면 장력이 발생하여 점성이 높고 덜 단단한 생물막의 움직임을 멈출 수 있습니다.
- (커버슬립을 사용하는 경우) 매니큐어를 말리십시오. 커버슬립의 외부 표면을 70% 에탄올로 조심스럽게 닦습니다. 위에 명시된 이유로 커버슬립에 압력을 가하지 않고 닦으십시오.
- 컨포칼 이미징
참고: 이 연구에서는 63x 대물렌즈와 함께 TCS 컨포칼 시스템이 장착된 도립 스펙트럼 이미징 레이저 스캐닝 컨포칼 현미경을 사용하십시오. 이 스코프는 4D 비디오를 생성하는 데 사용됩니다(그림 1). 3D 생물막은 18-20μm 두께의 생물막을 통해 0.5μm 단계로 캡처된 Z 슬라이스로 구성되어 36-40 Z 슬라이스를 생성합니다. 각 3D 생물막을 캡처하는 데 50-60초가 걸립니다. 조각은 함께 3D 생물막으로 시각화할 수 있는 프레임을 구성합니다. 이 과정을 20회 반복하여 총 추적 시간 18-20분 동안 4D 타임랩스 비디오를 생성합니다.- 형광단을 캡처하도록 범위를 설정합니다. 이 연구에서는 488nm 레이저로 Syto9(박테리아 DNA 염색)를 여기시키고 495-540nm의 방출을 측정합니다(Leica Sp5는 분광 이미징 현미경입니다). 633nm 레이저로 진홍색(빨간색) 비드를 여기하고 650nm에서 700nm까지의 방출을 측정합니다. 이러한 형광단 설정은 원하는 형광단을 포착하도록 조정할 수 있습니다.
- xyzt 이미징 모드를 선택합니다.
- 더 높은 밀도의 영역과 더 낮은 밀도의 영역이 혼합된 생물막의 영역을 식별하여 생물막의 두꺼운 영역과 얇은 영역에서 점탄성 특성의 차이를 포착합니다.
- 18-20μm 두께의 Z 스택을 설정합니다. 커버슬립 장착 바이오필름에서 비드 트랩 아티팩트를 방지하기 위해 바이오필름의 맨 위와 맨 아래가 유리에 닿는 것을 피하십시오(토론 참조).
- 게인과 오프셋을 조정하여 생물막의 가장 밝은 지점에서 강도의 전체 동적 범위를 사용합니다. 이것은 생물막의 낮은 층에서 신호 겹침을 최소화합니다.
- 슬라이스 두께를 0.5μm로 설정합니다. 이를 통해 비드 정보의 손실 없이 신속한 이미징이 가능합니다.
- 해상도를 512 x 512(0.48μm)로 설정합니다. 이를 통해 신속한 이미징이 가능하면서도 생물막 구조와 비드 이동 세부 사항을 볼 수 있는 충분한 해상도의 이미지를 생성할 수 있습니다.
- 이미징 시간을 최소화합니다.
- 20중첩을 캡처하도록 설정합니다.
- .lif(또는 유사한 컨포칼 파일)로 저장합니다. 4D 동영상은 ImageJ에서 생성할 수 있습니다.
3. ImageJ를 가진 4D biofilm 영상 생성하기
- ImageJ에서 다음 설정으로 .lif 파일을 엽니다: 하이퍼스택이 있는 스택 보기, 스택 순서 XYCZT, 색상 모드: 색상화, 자동 크기 조정 선택. 그런 다음 확인을 누릅니다.
- 이미지 > 색상 > 분할 채널을 선택합니다.
- 이미지 > 색상 > 채널 병합 > 복합 > 만들기를 선택합니다.
- 플러그인 > 3D 뷰어 > 채널을 선택하고 파란색 > 확인을 선택 취소합니다.
- 편집을 선택하고 > 콘텐츠 표시를 선택합니다> 테두리 상자 표시를 선택합니다.
- 마우스 길게 클릭하여 이미지를 선택합니다. 그런 다음 클릭 버튼을 계속 누른 상태에서 이미지를 회전합니다. 생물막의 바닥이 바닥에 오도록 이미지를 회전하고 각도가 비드 시각화를 지원하도록 합니다. 그런 다음 이미지를 해제합니다.
- 창 하단의 빨간색 녹화 버튼을 눌러 비디오를 녹화합니다.
- jpeg를 압축으로 사용하여 .avi 파일로 저장합니다.
4. 궤적 데이터 생성
- 오픈 소스 도구 ImageJ(https://imagej.net/Fiji) 및 입자 추적 플러그인 Mosaic(https://imagej.net/MOSAICsuite)를 설치합니다.
- 비드와 박테리아에 대한 두 채널이 포함된 .lif 파일을 가져옵니다. 채널을 분할하고 파일을 별도로 저장합니다.
- ImageJ에서 이미지 복셀 크기(x, y 및 z 치수)와 시간 단계 크기를 텍스트 파일에 저장합니다.
- ImageJ에서 플러그인 | 모자이크 를 만들고 파티클 트래커 2D/3D를 실행합니다.
- 역학을 Brownian으로 설정하고 반경: 3, 컷오프: 0.003, Per/Abs: 0.12, 링크 범위: 2, 디스플레이스: 10.00 매개변수를 입력합니다. 이 실험에서 Brownian을 선택한 이유에 대한 결과를 참조하십시오.
- 궤적을 생성하고 궤적 목록을 .csv 파일로 내보냅니다.
5. 궤적 분석
- VRL Studio 설치(https://vrl-studio.mihosoft.eu)
- Biofilm 프로젝트(https:// neurobox3d.github.io/Biofilm/)를 다운로드하고 VRL Studio에서 실행
- ImportData에서 궤적 파일을 로드합니다.
- ProcessTrajectories에서 x, y 및 z 픽셀 크기(ImageJ를 사용하여 이러한 값 찾기)를 지정합니다.
- ComputeVelocity에서 프레임 간격을 지정합니다(ImageJ를 사용하여 이 값을 찾습니다).
- Comdensity에서 박테리아 tiff 파일(3.2단계 참조)을 로드합니다.
- SaveVelocityDataToFile에서 속도 데이터의 출력 경로를 설정하고 SaveTrajectoryDataToFile에서 궤적 데이터를 설정합니다.
- SaveVelocityDataToFile 및 SaveTrajectoryDataToFile을 호출합니다.
- 분석을 위해 데이터를 Excel로 가져옵니다. 이러한 데이터에는 궤적 길이, 궤적 수명, 궤적 경계 상자 치수 및 부피, 평균 비드 속도 및 분산이 포함됩니다. 이 분석은 Syto9 표지 박테리아에 대한 채널을 사용하여 가중 변수를 계산하여 국부적(주어진 궤적 경계 상자 내) 세포 밀도를 계산합니다. 분석 결과 가중 평균, 속도 및 분산, 가중 평균 및 경계 상자 분산의 분산이 계산됩니다.
결과
이 방법은 curli가 E. coli와 S에 강성을 부여할 수 있다는 가설을 테스트하는 데 사용되었습니다. 티피무리움 생물막, 컨포칼 현미경 실험 중 비드 움직임을 감소시킵니다. 현재 도구 상자는 Enterococcus faecalis 공생형 균주 OG1RF의 재료 특성을 Salmonella enterica serotype Typhimurium, E. coli 및 각각의 동종 curli 돌연변이와 비교하는 데 사용되었습니다(그림 1B 및 보충 동영상 1, 보충 동영상 2, 보충 동영상 3, 보충 동영상 4, 보충 동영상 5, 보충 동영상 6 ). 생물막 물질 특성은 강성(예: eDNA에 결합된 curli) 또는 음전하를 띤 비드와 생물막 세포 및 매트릭스 물질 간의 정전기 및 소수성 상호 작용, 세포 밀도와 관련하여 잠재적으로 달라질 수 있습니다.
재현성
Biofilm 도구 상자는 VRL-Studio32 내의 Groovy30 및 Java31로 프로그래밍되어 모든 계산 구성 요소의 자동 UI(사용자 인터페이스) 생성을 통해 모듈식 워크플로 설계를 가능하게 합니다. 이를 통해 자동화된 워크플로우를 통해 결과를 분석할 때 의도하지 않은 실험자 유발 편향을 제거할 수 있었습니다.
MSD를 사용하여 생물막의 동작 유형을 확인합니다.
Particle Tracker 2D/3D를 사용한 궤적 분석의 경우, 다양한 비드 이동 유형 분석을 위한 다양한 역학 설정을 사용할 수 있습니다. 이러한 연구를 위해 E. faecalis는 비운동성 박테리아이고, E. coli와 Salmonella는 생물막에서 편모를 발현하지 않으며, 실험은 흐름이 없는 폐쇄된 시스템에서 수행되었기 때문에 "브라운 운동"(즉, 확산 구동 운동) 설정이 선택되었습니다. 이 설정은 비드의 계산된 평균 제곱 변위(MSD)에 의해 추가로 검증될 수 있습니다. m이 궤적 세그먼트의 수인 정의를 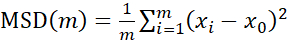 사용하여 각 궤적 과정에 대한 MSD의 변화를 계산할 수 있습니다. 선형 궤적은 확산 비드의 움직임을 나타냅니다(그림 2A). 2차 최소 제곱 피팅을 사용하여 생물막에 있는 모든 비드의 평균 이동 패턴을 계산하여 지배적인 선형 순서를 보여주고 수동 확산을 원동력으로 검증했습니다(그림 2A-2F).
사용하여 각 궤적 과정에 대한 MSD의 변화를 계산할 수 있습니다. 선형 궤적은 확산 비드의 움직임을 나타냅니다(그림 2A). 2차 최소 제곱 피팅을 사용하여 생물막에 있는 모든 비드의 평균 이동 패턴을 계산하여 지배적인 선형 순서를 보여주고 수동 확산을 원동력으로 검증했습니다(그림 2A-2F).
바운딩 박스 분석.
이 툴박스는 ImageJ Mosaic 및 Particle Tracker 2D/3D를 사용하여 궤적을 생성하고(4단계) 자동화된 바이오필름 분석 파이프라인을 사용하여 바이오필름 재료 특성을 비교하는 데 사용할 수 있는 비드 궤적에 대한 중요한 데이터를 생성합니다. μm3 단위의 bounding box volume은 궤적을 포함하는 최소 box를 구성하고 그 부피를 측정하여 측정되었습니다(그림 3).
E. faecalis 생물막은 1-6000 μm3 의 bounding box 값으로 더 많은 bead 움직임을 가지고 있습니다 (그림 3B, 3C 및 3D). 그 결과, 약 25μm의 코팅된 웰이 있는 코팅된 슬라이드에 장착된 유리 커버슬립에서 보이는 움직임(그림 3C)과 광학 유리 웰 바닥에서 성장하여 직접 이미징된 생물막(그림 3D)에서 보이는 움직임이 거의 차이가 없는 동일한 결과를 산출한다는 것을 확인했습니다. 유일한 차이점은 E. faecalis 생물막의 장착된 커버슬립 상단 부근에서는 수명이 10분 이상인 안정적인 궤적을 기록하지만 동시에 작은 바운딩 박스를 등록할 수 있는 반면, 광학 바닥 플레이트에서는 이동성이 더 높은 선별된 수의 비드를 등록할 수 있다는 것입니다. 종합하면, 이는 유리 슬라이드를 장착하면 장착된 커버슬립 생물막의 슬라이드에 대한 생물막 상단의 시스템 표면 장력이 변경되었을 수 있으며, 이는 궁극적으로 점성이 낮은 생물막 영역에서 일부 비드의 이동성을 감소시켰을 수 있음을 시사합니다(그림 3B, 3C, 3D 및 3I). 이 범주에 속하는 궤적은 매우 작은 비율이었으며, 이렇게 적은 수의 갇힌 비드에도 불구하고 장착된 커버슬립에 있는 E. faecalis 의 평균 MSD는 광학 바닥판 생물막에서 계산된 MSD보다 약간 높았습니다(그림 3).
에스. Typhimurium 및 E. coli bead 궤적은 E. coli의 경우 1-6000 μm3 및 S의 경우 1-5000 μm3 의 bounding box를 가진 isogenic curli mutant에 비해 0-10 μm3 의 더 작은 bounding box volume (그림 3A, 3B, 3E 및 3F)를 가졌습니다. 티피무리움(그림 3A, 3B, 3F 및 3H)은 더 큰 비드 이동성을 보여줍니다. 이러한 결과는 아밀로이드의 존재가 생물막의 강성 증가와 상관관계가 있으며 비디오에서 눈에 띄는 생물막 움직임이 없는 것과 일치함을 시사했습니다. 바운딩 박스 부피는 생물막의 저밀도 영역에서도 일관되게 작았습니다(0-10 μm3). 이러한 관찰은 curli가 생물막10의 낮은 세포 밀도 영역에 존재할 수 있다는 이전 관찰과 일치합니다.
장내세균(Enterobacteriaceae) 생물막은 공기-액체 계면에서 펠리클(pellicle)로 자라기 때문에 광학 바닥판에서 장내세균(Enterobacteriaceae biofilm)의 거동을 비교할 수 없었습니다(단계 1.2.2). 커버슬립을 사용할 때, 펠리클은 인터페이스에서 커버슬립에 부착되고, 커버슬립이 제거되었을 때 펠리클이 커버슬립 위에 놓여 단일 이미지 표면을 생성했습니다. 비스듬히 성장한 광학 바닥판에서 이미징은 우물 안에 여전히 액체가 있는 상태에서 수행되었습니다. 이는 펠리클이 여전히 광학 바닥 위에 떠 있고 Leica Sp5와 같은 거꾸로 된 스코프의 작업 깊이에서 펠리클을 만든다는 것을 의미합니다. 생물막을 현미경의 작업 깊이로 가져올 수 있을 만큼 충분한 매체를 제거하면 20분의 이미징 프로세스 동안 표본이 건조되었습니다.
전반적으로 플롯은 보충 동영상의 시각적 관찰을 확인하며 관찰된 MSD 차이와 일치합니다(그림 3I 및 3J).
궤적 수명
궤적 수명은 비드가 등록된 연속된 프레임의 수로 측정되었습니다(그림 3).
점성이 더 높고 유체와 같은 E. faecalis 생물막에서 모든 비드의 궤적 수명은 10분 미만이었고 대부분의 궤적은 E. faecalis 생물막에 대해 2-5분 사이였습니다. 그러나 짧은 궤적 수명이 등록된 비드는 전체 이미징 시간 창에 걸쳐 E. faecalis 생물막에서 육안 검사를 통해 찾을 수 있습니다(보충 비디오 1 및 2). 따라서, 비드가 등록된 궤적을 따라 이동하면서 간헐적으로 바이오필름에서 분리되고 궤적을 종료하며 바이오필름과 재결합하는 것이 가능하며, 이 지점에서 새로운 궤적이 시작됩니다. 이것은 궁극적으로 생물막에 있는 beads의 지속적인 존재 하에서 짧은 탄도 수명으로 이끌어 낼 것입니다. 이 기술을 사용하면 특히 점성이 있는 생물막에서 궤적 수명이 비드가 생물막과 관련된 총 시간을 과소평가하는 경향이 있다는 점에 유의하는 것이 중요합니다.
S에서. bounding box 부피가 더 작은 Typhimurium 생물막의 대다수(약 80%)는 실시간으로 약 15-20분에 해당하는 16-20프레임의 긴 궤적 수명을 가졌습니다(그림 3A, 3G 및 3H). 이와는 반대로, 동종 curli 돌연변이 생물막은 1-6000 μm3 (E. coli) 및 1-5000 μm3 (S. Typhimurium) (그림 3A, 3B, 3F 및 3H). 그러나 경계 상자 부피가 10μm3보다 큰 >70% 궤적을 가진 E. faecalis 생물막과 달리 Enterobacteriaceae 종 생물막은 경계 상자 부피가 10μm3 이상인 30%의 비드 궤적만 등록했습니다. curli 돌연변이 생물막에서 전체 비드 궤적 수명은 더 작았지만, 일부 궤적은 상당한 비드 이동과 긴 궤적 수명을 반영했습니다(그림 3H). 이러한 관찰은 이러한 변동성이 점탄성과 같은 다양한 생물막 재료 특성 및/또는 전하와 같은 입자 표면 화학적 변화에 해당할 수 있음을 나타낼 수 있습니다.
비드 궤적 길이 및 속도 분석
궤적 길이는 비드가 이동한 거리를 μm 단위로 측정한 것입니다. 이 측정은 비드 이동 속도(μm/s)와 일치합니다. 더 큰 bounding box 부피와 일치하게, E. faecalis 생물막의 비드는 curli를 포함하는 생물막의 <4μm에 비해 5-20μm로 10배 더 긴 궤적을 가졌습니다. 더 짧은 궤적과 일치합니다(그림 4A). E. faecalis 비드는 최대 15배 더 높은 속도를 측정했으며, 대부분의 비드는 <0.006μm/s의 속도에 비해 0.01-0.15μm/s 범위의 속도를 가졌습니다(그림 4B). 그럼에도 불구하고, curli 돌연변이 생물막은 E. faecalis 생물막에 비해 전반적으로 더 낮은 속도와 더 짧은 궤적을 측정했지만, 부모 균주를 포함하는 curli보다 더 긴 궤적과 더 높은 속도를 측정했습니다(그림 4A 및 4B).
주목할 만한 것은 curli30 의 섬유소 격자와 같은 구조가 비등방성 방식으로 이동성에 영향을 미칠 수 있으며, xy-평면에서의 움직임을 감소시키고 z-방향의 이동성을 증가시킬 수 있다는 사실입니다(그림 2G). curli를 함유한 생물막에서 약 50개의 고유한 구슬에 속하는 큰 궤적 풀(약 800개)은 이러한 빠르게 움직이는 구슬 각각을 x, y 및 z의 단일 구슬로 계산하는 모자이크 입자 추적의 한계와 일치할 것입니다. 이 관찰을 확인하기 위해서는 추가 연구 및 소프트웨어 개발이 필요할 것입니다.
세포 밀도에 대한 비드 이동 의존성 분석
세포 밀도에 대한 비드 이동의 의존성은 가중 평균 속도 및 분산, 평균/가중 평균 및 bounding box volume의 분산을 사용하여 결정되었습니다. Syto9 표지 박테리아에 대한 두 번째 이미징 채널은 가중 속도 계산에서 국소 세포 밀도를 계산하는 데 사용되었습니다. 세포 밀도는 각 궤적 가장자리의 경계 상자에 대한 Syto9 복셀 데이터의 평균을 계산하여 계산되었습니다(그림 5, 오른쪽). 따라서, 비드 속도는 가장자리 방향(국부적) 세포 밀도에 의해 가중치가 부여될 수 있습니다. 박테리아를 시각화하는 데 사용할 수 있는 여러 유형의 염색에는 세포벽, 막 및 DNA 함량에 대한 염색을 포함하여 사용할 수 있습니다. 세포 밀도를 결정하기 위해 Syto9을 선택한 이유는 어떤 광학 Z 슬라이스를 시각화하든 가장 일관된 신호를 제공하기 때문입니다. 봉투 염색(세포벽 및 막)은 Z 슬라이스의 위치에 따라 다른 신호를 제공합니다. Z 슬라이스가 셀의 상단 또는 하단을 포함하는 경우, Z 슬라이스가 셀의 윤곽선만 염색되는 셀의 중앙을 통과하는 경우보다 신호가 더 강합니다.
빨간색 채널의 비드 궤적은 20개 프레임에 대해 추적되었으며, 여기서 개별 궤적은 최소 2프레임의 수명을 가지며 프레임을 연결하는 19개의 궤적 세그먼트가 있는 최대 20개의 프레임을 가졌습니다(그림 5). 비드 이동도의 세포 밀도 의존성을 연구하기 위해 복셀당 GFP 강도(512x512 복셀 이미지의 각 개별 측정)를 측정했습니다. 비드 궤적의 각 세그먼트 주위의 세포 밀도는 세그먼트의 bounding box에서 국부적으로 평균 밀도로 계산되었습니다.
일부 생물막의 경우 통계적으로 유의한 밀도 의존성을 문서화할 수 있으며(그림 6), 가장 두드러진 것은 유리 커버슬립에서 성장하고 멀티웰 슬라이드로 반전된 E. faecalis 생물막입니다(그림 6A). 반대로, 96 웰 플레이트의 바닥에서 성장한 E. faecalis 생물막(그림 6B)은 밀도 의존성을 나타내지 않았습니다. 결론적으로, 이는 매우 유동적인 E. faecalis 생물막이 멀티웰 슬라이드에 장착되어 잠재적으로 약간 압축될 수 있음을 시사하며, 이는 더 빠르게 움직이는 비드의 수와 유리 슬라이드에 대한 생물막 상단에 갇힌 작은 경계 상자 부피를 가진 비드의 수가 감소하는 것과 일치합니다(그림 3C 대 그림 3D). 살모넬라균 및 대장균 생물막(그림 6, C 및 6D)과 이들의 동종 돌연변이(그림 6E 및 6F)는 모두 세포 밀도 의존성이 경미하거나 전혀 없는 것으로 나타났습니다.
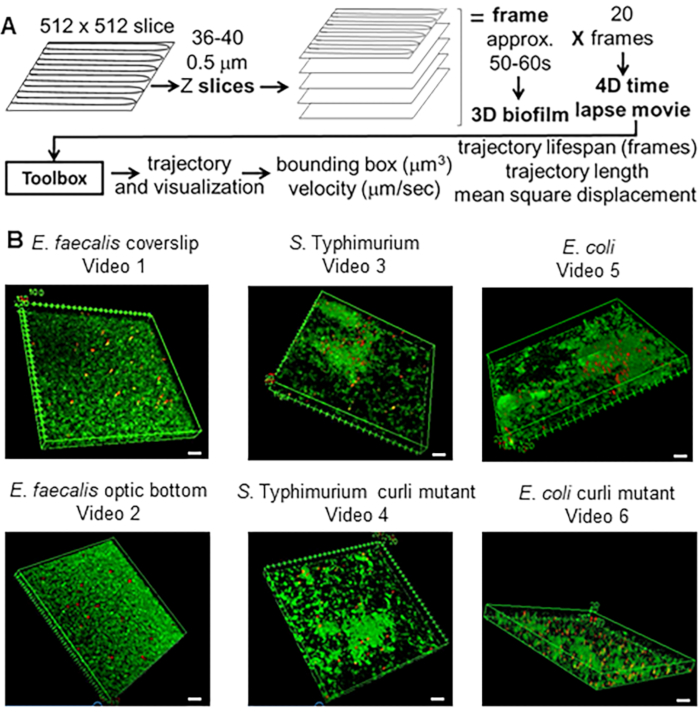
그림 1. 이미징 및 분석 파이프라인(2-4단계) (A) 생물막은 2.2에 설명된 대로 이미지화됩니다. 이미징된 생물막(3 참조)을 사용하여 4에 설명된 바와 같이 비드 궤적을 생성했습니다. 궤적을 사용하여 분석 도구 상자(5 참조)로 관련 데이터를 계산했습니다(B) 바이오필름은 커버슬립(E. faecalis, 비디오 1)에 대한 1단계에서 설명한 대로 성장했습니다 . 티피무리움(동영상 3), 대장균 (동영상 5) 및 동원성 컬리 돌연변이(동영상 4, 동영상 6) 또는 광학 바닥 96웰 플레이트(E. faecalis 하단, 동영상 2). 흰색 눈금 막대는 20mm입니다. 이 그림은 (31)의 허락을 받아 복제한 것입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
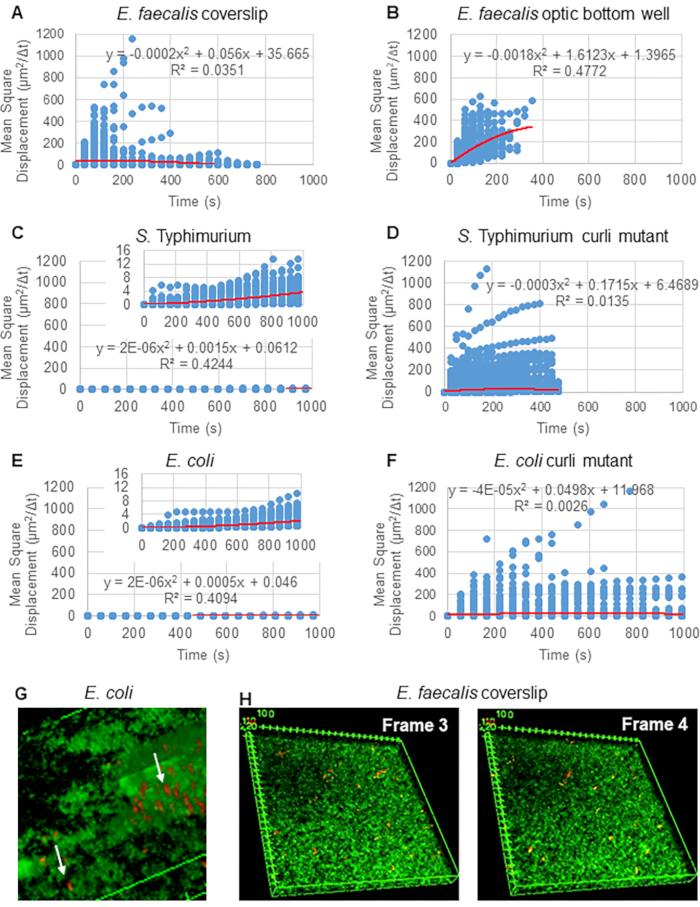
그림 2. 생물막의 비드 이동은 자연에서 확산되는 것으로 결정되었습니다(브라운 운동, 5단계 참조) (A-F) MSD 데이터는 브라운 운동을 검증하기 위한 선형 거동(빨간색 선)을 보여줍니다. 생물막은 단계 1 (G) E . coli 및 S. Typhimurium에서 관찰된 타원형 비드 이동의 예, E. coli 4D 생물막 분석의 한 프레임에서 취한 생물막(H) E. faecalis 광학 바닥 웰에서 가져온 프레임 3과 4 사이의 비드 패턴의 큰 변화의 예에 따라 성장했습니다. 생물막 자체가 약간의 흐름(비디오 1 및 2)을 유도하여 프레임 3과 4가 다른 방향인 것처럼 보입니다. 이 그림은 (31)의 허락을 받아 복제한 것입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
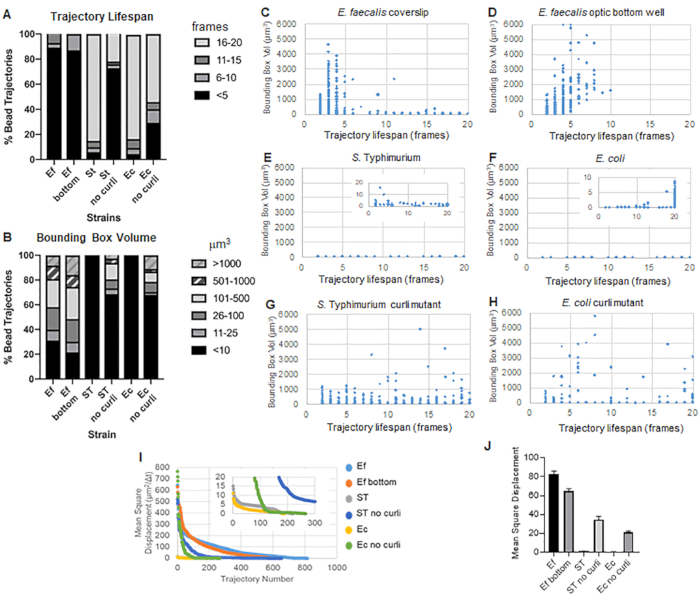
그림 3. bounding boxes 및 궤적 수명을 사용한 강성의 차이를 분석합니다. 생물막은 커버슬립(coverslips)의 1단계(E. faecalis, Video 1), S. 티피무리움(동영상 3), 대장균(동영상 5) 및 동원성 컬리 돌연변이(동영상 4, 동영상 6) 또는 광학 바닥 96웰 플레이트(E. faecalis 하단, 동영상 2). 궤적 수명은 bounding box volume(5단계에서 계산) (I) 서로 다른 생물막의 비드 MSD 비교 (H) 각 유형의 생물막의 평균 MSD와 함께 총 비드 궤적(A) 및 산란 그래프(C-H)의 %로 표시됩니다. 막대는 데이터의 95% 신뢰 구간을 나타냅니다. 이 그림은 (31)의 허락을 받아 복제한 것입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
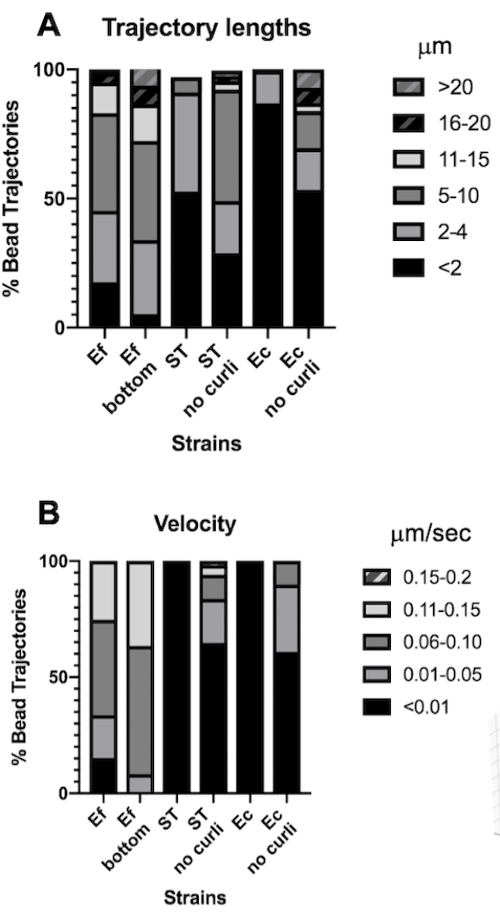
그림 4. 궤적 길이와 비드 속도의 차이 분석. 생물막은 단계 1에 기술된 바와 같이 성장시켰다. 궤적 길이는 μm로 표시되며 총 비드 궤적의 %(A)로 표시됩니다. 속도는 μm/s로 표시되며 총 비드 궤적의 %(B)로 표시됩니다. 이 그림은 (31)의 허락을 받아 수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
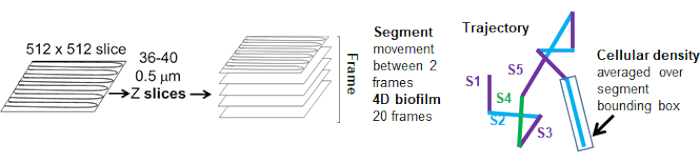
그림 5. 궤적 세그먼트 분석의 개요. 이 그림은 (31)의 허락을 받아 수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
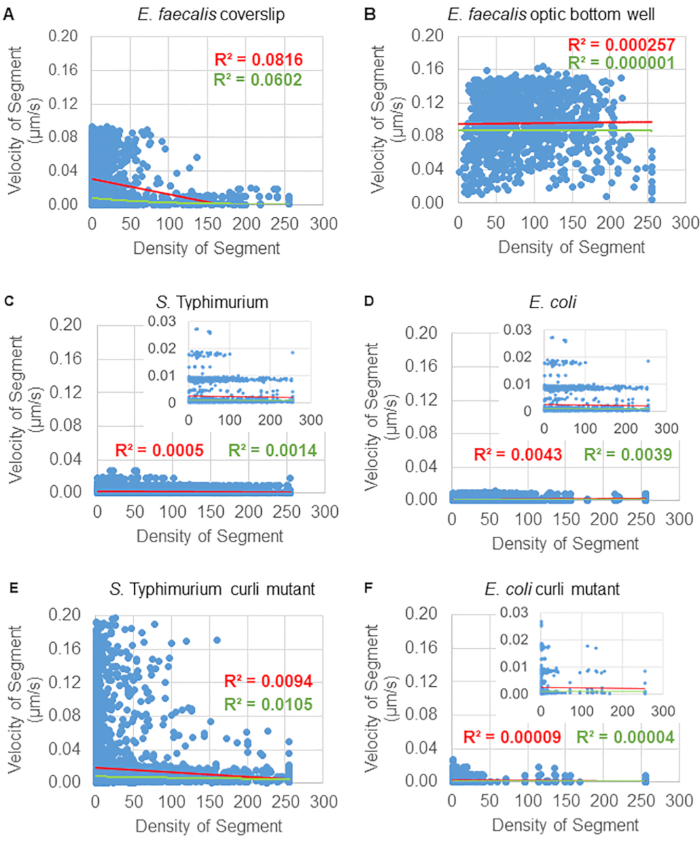
그림 6. 생물막 분석 파이프라인을 사용하여 비드 속도에 대한 생물막 밀도의 영향을 연구합니다(5단계). 결과는 비드 속도가 전적으로 세포 밀도에 의존하는 것은 아니라는 것을 보여주었습니다. 생물막은 단계 1에 기술된 바와 같이 성장시켰다. 궤적은 개별 궤적 세그먼트의 규모로 분석되었습니다(그림 5). 각 세그먼트에 대해 μm/s 단위의 비드 속도를 바운딩 박스 세포 밀도(바운딩 박스 내 복셀당 평균 GFP)에 대해 표시했습니다. 빨간색 선은 선형 회귀를 나타내고 녹색 선은 지수 회귀 추적을 나타냅니다. 이 그림은 (31)의 허락을 받아 복제한 것입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 비디오 1. 1.5 두께의 광학 유리 커버 슬립에서 성장한 24 시간 E. faecalis OG1RF 생물막의 4D 비디오.4D 타임랩스 비디오는 512x512 해상도의 현미경을 사용하여 생성되었습니다. 40개의 이미지로 구성된 Z 시리즈는 0.5μm 단계로 약 20μm 두께의 생물막 영역을 이미징하여 생성되었습니다. 각 Z 시리즈는 하나의 프레임이었고 캡처하는 데 50-60초가 필요했습니다. 4D 비디오를 제작하기 위해 일련의 20개의 연속된 프레임을 캡처했습니다. 비디오 재생은 약 120x입니다. 동영상은 최소 6개의 독립적인 실험을 대표합니다. 이 비디오를 다운로드하려면 여기를 클릭하십시오.
보충 비디오 2. 96웰 광학 바닥판에서 성장한 24시간 E. faecalis OG1RF 생물막의 4D 비디오. 4D 타임랩스 비디오는 512x512 해상도의 현미경을 사용하여 생성되었습니다. 40개의 이미지로 구성된 Z 시리즈는 0.5μm 단계로 약 20μm 두께의 생물막 영역을 이미징하여 생성되었습니다. 각 Z 시리즈는 하나의 프레임이었고 캡처하는 데 50-60초가 필요했습니다. 4D 비디오를 제작하기 위해 일련의 20개의 연속된 프레임을 캡처했습니다. 비디오 재생은 약 120x입니다. 비디오는 3개의 독립적인 실험을 대표합니다.이것을 다운로드하려면 여기를 클릭하십시오. 비디오.
보충 비디오 3. 6-7일 동안 1.5 두께의 광학 유리 커버슬립에서 자란 Salmonella enterica 혈청형 Typhimurium 생물막 ATCC 14028 의 4D 비디오.4D 타임랩스 비디오는 512x512 해상도의 현미경을 사용하여 생성되었습니다. 40개의 이미지로 구성된 Z 시리즈는 0.5μm 단계로 약 20μm 두께의 생물막 영역을 이미징하여 생성되었습니다. 각 Z 시리즈는 하나의 프레임이었고 캡처하는 데 50-60초가 필요했습니다. 4D 비디오를 제작하기 위해 일련의 20개의 연속된 프레임을 캡처했습니다. 비디오 재생은 약 120x입니다. 비디오는 3개의 독립적인 실험을 대표합니다.이것을 다운로드하려면 여기를 클릭하십시오. 비디오.
보충 비디오: 4. Salmonella enterica 혈청형 Typhimurium biofilms ATCC 14028 curli (csgBA) 돌연변이의 4D 비디오를 1.5 두께의 광학 유리 커버 슬립에서 6-7 일 동안 성장시켰다. 4D 타임랩스 비디오는 512x512 해상도의 현미경을 사용하여 생성되었습니다. 40개의 이미지로 구성된 Z 시리즈는 0.5μm 단계로 약 20μm 두께의 생물막 영역을 이미징하여 생성되었습니다. 각 Z 시리즈는 하나의 프레임이었고 캡처하는 데 50-60초가 필요했습니다. 4D 비디오를 제작하기 위해 일련의 20개의 연속된 프레임을 캡처했습니다. 비디오 재생은 약 120x입니다. 비디오는 3개의 독립적인 실험을 대표합니다. 이것을 다운로드하려면 여기를 클릭하십시오. 비디오.
보충 비디오 5. 6-7일 동안 1.5 두께의 광학 유리 커버슬립에서 자란 E. coli UTI89의 4D 비디오. 4D 타임랩스 비디오는 512x512 해상도의 현미경을 사용하여 생성되었습니다. 40개의 이미지로 구성된 Z 시리즈는 0.5μm 단계로 약 20μm 두께의 생물막 영역을 이미징하여 생성되었습니다. 각 Z 시리즈는 하나의 프레임이었고 캡처하는 데 50-60초가 필요했습니다. 4D 비디오를 제작하기 위해 일련의 20개의 연속된 프레임을 캡처했습니다. 비디오 재생은 약 120x입니다. 비디오는 3개의 독립적인 실험을 대표합니다.이것을 다운로드하려면 여기를 클릭하십시오. 비디오.
보충 비디오 6. 4D 비디오 E. coli UTI89 curli (csgBA) 돌연변이가 1.5 두께의 광학 유리 커버 슬립에서 6-7 일 동안 자랐습니다. 4D 타임랩스 비디오는 512x512 해상도의 현미경을 사용하여 생성되었습니다. 40개의 이미지로 구성된 Z 시리즈는 0.5μm 단계로 약 20μm 두께의 생물막 영역을 이미징하여 생성되었습니다. 각 Z 시리즈는 하나의 프레임이었고 캡처하는 데 50-60초가 필요했습니다. 4D 비디오를 제작하기 위해 일련의 20개의 연속된 프레임을 캡처했습니다. 비디오 재생은 약 120x입니다. 비디오는 3개의 독립적인 실험을 대표합니다.이것을 다운로드하려면 여기를 클릭하십시오. 비디오.
토론
중요한 단계 및 문제 해결
이 기술의 가장 큰 과제는 E. faecalis 와 같은 점성이 매우 높은 생물막이 있는 장착된 커버슬립을 사용하는 것입니다.커버슬립은 위치를 변경하지 않고 멀티웰 슬라이드에 신중하고 정확하게 배치해야 합니다. 밀봉 단계에서는 커버슬립을 아래로 누르거나 슬라이드 표면을 가로질러 실수로 밀거나 미끄러지지 않도록 주의해야 합니다. 모든 움직임이나 압력은 표면 장력을 생성하고 점성 생물막의 움직임을 차단할 수 있습니다. 가능한 경우, 광학 바닥 웰의 생물막을 커버슬립 마운트에 이미징하여 생물막 재료 특성을 비교하면 기술 평가가 가능합니다. 올바르게 수행되었을 때, 커버슬립 마운트는 E. faecalis의 광학 바닥 플레이트에 있는 생물막과 매우 유사했습니다.
또한, 장착된 커버슬립을 사용할 때, 하단의 커버슬립 또는 맨 위의 슬라이드와 바이오필름의 계면에 대한 이미징은 피해야 합니다. 거꾸로 된 내시경을 사용할 때 커버슬립이 바닥에 있는 경우 커버슬립에 대한 생물막 바닥에 갇힌 비드가 있을 수 있습니다. 이 구슬은 생물막을 통과하여 부드럽게 세척한 후에도 커버슬립에 갇히게 됩니다. x, y 및 z 좌표는 0이고 경계 상자 좌표는 0입니다. 그러나 처리 후 생물막 무결성 검사와 같은 특정 응용 분야의 경우 이러한 데이터 포인트를 도구로 사용할 수 있습니다. 두꺼운 생물막 영역을 통해 커버슬립 바닥까지 침투하는 비드의 능력은 처리 후 생물막 무결성을 평가하는 데 사용할 수 있습니다(Tükel 실험실과 공동으로 준비 중인 원고). 생물막의 맨 위에서, E. faecalis 와 같은 점성이 있는 생물막에서 우리는 커버슬립에 의해 부과된 압축의 증거를 가지고 있었습니다. 이로 인해 유리 슬라이드 계면에서 일부 비드의 이동이 제한되었으며 비드 이동 분석에 약간의 밀도 의존성이 발생했을 수 있습니다.
세척 단계는 성장 배지가 녹색 채널에서 강한 자가형광을 갖기 때문에 생물막에 필요했습니다. 우리는 관찰된 영역의 가장 정확한 특성화를 얻기 위해 관련 비드를 최대화하기 위해 과도한 비드를 사용하고 세척을 통해 연관되지 않은 비드를 제거하기로 선택합니다.
원하는 데이터 세트를 얻는 데 필요한 비드 및 세척 횟수는 경험적으로 결정해야 합니다. 생물막에 너무 많은 비드가 존재하면 분석하기 어려운 불가능할 정도로 큰 데이터 세트가 생성됩니다. 비드가 너무 적으면 생물막 환경의 철저한 샘플링이 생성되지 않습니다. 그러나, 첨가된 비드의 수(PBS 1mL에 2x107 비드)의 제어와 세척 단계의 사용은 구조, 공간 배열 및 구성에 따라 생물막과 관련된 비교적 일관된 수의 비드(40-140)를 생성했습니다.
점성 영역과 강성 영역이 혼합된 생물막을 연구할 때 비드는 시간이 지남에 따라 강성 영역에 갇힐 수 있습니다. 이 경우 비드를 추가한 직후 이미징을 시작해야 합니다. 이는 종종 커버슬립을 사용하여 달성할 수 없지만 광학 바닥 플레이트 또는 플로우 셀이 필요하며, 비드 추가 및 세척 단계 직후에 이미징을 수행할 수 있습니다.
수정 사항 및 향후 적용 사례
미세유체 장치의 사용. 우리의 연구에서, Enterobacteriaceae 생물막 연구를 위해 확립된 최적의 조건은 공기-액체 계면에서 펠리클로서 생물막의 성장을 필요로 했습니다. 이로 인해 연구에서 광학 바닥 플레이트와 미세유체 장치의 사용이 제한되었습니다. 그러나 생물막 형성 조건이 허락하는 경우 생물막은 미세유체 챔버 또는 플로우 셀에서 성장할 수 있습니다. 그런 다음 생물막을 세척하고 생물막의 파괴를 최소화하면서 미세유체 장치를 통해 비드를 도입할 수 있었습니다.
생물막 성장 중 비드 추가. 우리는 생물막에 과도한 비드를 추가한 다음 분석 중에 존재하는 비드의 수를 최적화하기 위해 부드러운 세척으로 연결되지 않은 비드를 제거하기로 결정했습니다. 점성이 있는 E. faecalis 생물막에서 비드가 20분의 이미징 시간 동안 분리되었다가 재결합될 수 있습니다. 생물막 성장 중 서로 다른 시간에 적은 수의 비드가 추가되는 경우, 비드를 생물막에 가두어 점성이 더 높은 생물막에서 생물막 이동을 보다 정확하게 특성화할 수 있습니다.
이미지화할 영역을 선택합니다. 재료 특성에 대한 연구를 위해서는 생물막의 두껍고 얇은 영역을 선택하는 것이 가장 좋습니다. 그러나 처리된 생물막의 재료 특성 변화를 연구할 때 두꺼운 합류 영역을 이미지화하여 해당 영역의 점탄성 특성과 비드 침투의 변화를 결정할 수 있습니다. 이 경우, 생물막을 관통하여 커버슬립에 갇힌 비드를 찾는 것이 생물막 파괴의 유용한 척도입니다.
흐름에 따른 이미징. 광학 유리 플로우 셀 또는 미세유체 장치를 사용하여 유동 중인 생물막에서 비드 또는 박테리아의 움직임을 이미지화할 수 있습니다. 이것은 다양한 방법으로 수행할 수 있습니다. 그것은 전체 챔버에 비드를 주입한 다음 비드와 생물막의 결합을 허용하기 위해 짧은 배양으로 수행할 수 있습니다. 연관되지 않은 비드는 세척을 통해 제거할 수 있으며 유동 여부에 관계없이 이미징된 생물막을 제거할 수 있습니다. 반대로, 소수의 비드가 챔버의 한쪽 면으로 유입될 수 있으며 생물막을 통한 및 생물막 내에서의 이동을 흐름 하에서 추적할 수 있습니다. 플로우를 사용할 때 모자이크 비드 추적 설정을 선택할 때 주의해야 합니다(4.5단계). 현재 연구에서 역학 설정은 브라운이었습니다. MSD 계산에 따르면 이 움직임은 확산될 가능성이 있어 Brownian이 적절한 설정입니다.
매트릭스 염색. 현재 연구에서 Syto9을 사용한 염색은 생물막 구조 밀도가 아닌 세포 밀도를 검사합니다. 예를 들어, 아밀로이드의 존재는 생물막의 매트릭스 물질의 밀도를 증가시킬 가능성이 있습니다. 아밀로이드 밀도에 대한 움직임의 의존성은 Syto9 대신 형광 매트릭스 염색을 사용하여 결정할 수 있습니다.
형광 표지 박테리아. 형광 표지된 박테리아는 생물막을 통한 외인성 박테리아(예: 플라스미드 함유 박테리아)의 이동을 추적하는 데 사용할 수 있습니다. 엔테로코씨(Enterococci )와 같은 형광 표지 박테리아의 문제는 이러한 박테리아가 단일체, 이중구균(diplococci) 및 짧은 사슬을 형성하여 박테리아를 정확하게 추적하는 능력을 복잡하게 만든다는 것입니다. 이 과정은 박테리아가 단세포 형태를 가지고 있다면 더 쉬울 것입니다.
제한
궤적 시각화 및 스티칭의 한계.
이 방법의 한 가지 한계는 궤적 시각화와 스티칭입니다. 재구성 및 분석된 궤적은 x, y, z 점 좌표로 구성되며, 여기서 후속 점은 이러한 점 사이의 선형 경로를 정의합니다. 이러한 조각별 선형 궤적의 시각화는 다양한 도구를 통해 달성할 수 있습니다. 한 가지 접근 방식은 Python 및 Jupyter 노트북을 Python 플러그인, Pandas 및 Matplotlib와 함께 사용하는 것이었습니다. 이 기술이 처음 발표된 Journal of Bacteriology 논문에서 개별 기존 궤적을 시각화하는 것이 가능했지만34 향후 연구에서 다루어질 중요한 제한 사항이 여전히 있습니다.
현재 재구성된 궤적의 수는 생물막의 비드 수보다 크며, 이는 여러 궤적이 하나의 비드에 해당할 수 있음을 의미합니다. 이는 Mosaic가 궤적을 종료하고 두 번째 궤도를 시작하는 한 프레임의 약한 공초점 신호로 인해 발생할 수 있습니다. 이것은 특히 점성이 낮은 생물막에서 하나의 비드에 대해 여러 개의 더 짧은 궤적으로 등록될 수 있습니다. 궤적 수가 많은 또 다른 원인은 궤적 스티칭이 부족하기 때문입니다. 특히 E. faecalis optic bottom well biofilms에서 beads는 이미징 중에 biofilm과 시각적으로 연결된 상태로 남아 있습니다(보충 비디오 2). 그러나 10보다 긴 궤적은 없었고 궤적의 90% 이상이 5프레임 이하의 수명을 가졌습니다(그림 3D). 소프트웨어를 사용하여 정의된 길이 이상의 궤적만 분석하는 경우(예: 플라스미드를 전달할 수 있는 셀을 추적하는 경우) 데이터 세트에서 더 짧은 궤적을 자동으로 제거할 수 있습니다. 그러나 궤적을 꿰매는 것이 매우 중요할 수 있는 다른 목적이 있습니다. 마지막으로, 단일 궤적으로 빠른 비드 이동을 추적할 수 없기 때문에 Z 방향의 빠른 움직임으로 인해 장내세균 생물막에서 더 많은 궤적이 생성되어 타원형 비드가 생성될 수 있습니다(그림 2G). 빠른 이방성 움직임에 의해 봉합 궤적이 중단될 가능성은 장내세균에서 curli 아밀로이드 기질의 효과를 연구하는 데 중요할 것입니다.
중요성
3D 생물막의 재료 특성을 비교하기 위해 비드 궤적을 연구하기 위해 컴퓨터 워크플로우가 개발되었습니다. 이 워크플로우를 통해 연구원들은 이질적인 생물막에서 유체 역학의 컴퓨터 모델링에 사용할 수 있는 중요한 매개변수를 식별할 수 있습니다. 이 오픈 소스 비드 평가자의 도움으로 박테리아 아밀로이드 curli가 재료 특성에 미치는 영향을 연구하여 curli로 인한 생물막 매트릭스 강성이 증가했음을 보여줄 수 있습니다. 보다 일반적인 맥락에서, 평가자는 생물막 처리 또는 흐름과 같은 다양한 환경 조건에 의해 유도된 생물막 구조의 변화를 연구하는 데 사용할 수 있습니다. 예를 들어, 이 도구는 Tükel 실험실(LKSOM Temple University)과 협력하여 생물막 구조의 파괴에 대한 단클론 항체 처리의 효과를 분석하는 데 사용되고 있습니다. 비드 평가기 도구 상자는 VRL-Studio를 사용하여 기능을 더욱 향상시키고 확장하기 위해 모듈식 방식으로 완전히 조정 및 확장할 수 있습니다.
공개
저자는 공개할 내용이 없습니다.
감사의 말
GQ 및 BAB 연구소에서의 연구는 공공, 상업 또는 비영리 부문의 자금 지원 기관으로부터 특정 보조금을 받지 않았습니다. 저자는 이 기술을 포함하는 초기 출판물에 대한 장내세균 전문 지식을 제공한 Isaac Klapper 박사(템플 대학교 수학과)와 Çagla Tükel(템플 대학교 미생물학 및 면역학과)에게 감사를 표합니다.
자료
| Name | Company | Catalog Number | Comments |
| 96-well plates, No. 1.5 Uncoated Coverslip, 5 mm Glass Diameter | MatTek | P96G1.55F | |
| Fisherbrand Cover Glasses: Circles | Fisher Scientific | 12-293-232P | 1.5 optic glass coverslip |
| Invitrogen Syto 9 Green Fluorescent Nucleic Acid Stain | Invitrogen | S34854 | |
| Molecular Probes FluoSpheres Carboxylate-modified Microspheres, 1 um, crimson fluorescent (625/645) | Molecular Probes | F8816 |
참고문헌
- Huang, R., Li, M., Gregory, R. L. Bacterial interactions in dental biofilm. Virulence. 2, 435-444 (2011).
- Nadell, C. D., Drescher, K., Wingreen, N. S., Bassler, B. L. Extracellular matrix structure governs invasion resistance in bacterial biofilms. ISME Journal. 9, 1700-1709 (2015).
- Billings, N., Birjiniuk, A., Samad, T. S., Doyle, P. S., Ribbeck, K. Material properties of biofilms - key methods for understanding permeability and mechanics. Reports on Progress in Physics. 78, 036601 (2015).
- Araújo, G. R. d. e. S., Viana, N. B., Gómez, F., Pontes, B., Frases, S. The mechanical properties of microbial surfaces and biofilms. The Cell Surface. 5, 100028 (2019).
- Tallawi, M., Opitz, M., Lieleg, O. Modulation of the mechanical properties of bacterial biofilms in response to environmental challenges. Biomaterials Science. 5, 887-900 (2017).
- Tursi, S. A., Tükel, &. #. 1. 9. 9. ;. Curli-Containing Enteric Biofilms Inside and Out, Matrix Composition, Immune Recognition, and Disease Implications. Microbiology and Molecular Biology Reviews. 82, (2018).
- Dueholm, M. S., Albertsen, M., Otzen, D., Nielsen, P. H. Curli functional amyloid systems are phylogenetically widespread and display large diversity in operon and protein structure. PLoS One. 7 (12), 51274 (2012).
- Hung, C., et al. Escherichia coli biofilms have an organized and complex extracellular matrix structure. mBio. 4, 00645 (2013).
- Kikuchi, T., Mizunoe, Y., Takade, A., Naito, S., Yoshida, S. Curli fibers are required for development of biofilm architecture in Escherichia coli K-12 and enhance bacterial adherence to human uroepithelial cells. Microbiology and Immunology. 49, 875-884 (2005).
- Gallo, P. M., et al. Amyloid-DNA Composites of Bacterial Biofilms Stimulate Autoimmunity. Immunity. 42, 1171-1184 (2015).
- Serra, D. O., Richter, A. M., Hengge, R. Cellulose as an architectural element in spatially structured Escherichia coli biofilms. Journal of Bacteriology. 195, 5540-5554 (2013).
- Oh, Y. J. Characterization of curli A production on living bacterial surfaces by scanning probe microscopy. Biophysical Journal. 103, 1666-1671 (2012).
- Lembré, P., Di Martino, P., Vendrely, C. Amyloid peptides derived from CsgA and FapC modify the viscoelastic properties of biofilm model matrices. Biofouling. 30, 415-426 (2014).
- Oh, Y. J., et al. Curli mediate bacterial adhesion to fibronectin via tensile multiple bonds. Scientific Reports. 6, 33909 (2016).
- Vidakovic, L., Singh, P. K., Hartmann, R., Nadell, C. D., Drescher, K. Dynamic biofilm architecture confers individual and collective mechanisms of viral protection. Nature Microbiology. 3, 26-31 (2018).
- Tursi, S. A., et al. Salmonella Typhimurium biofilm disruption by a human antibody that binds a pan-amyloid epitope on curli. Nature Communications. 11, 1007 (2020).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15 (8), 1007978 (2019).
- Taglialegna, A., et al. The biofilm-associated surface protein Esp of Enterococcus faecalis forms amyloid-like fibers. Npj Biofilms and Microbiomes. 6, 15 (2020).
- Gour, S., Kumar, V., Rana, M., Yadav, J. K. Pheromone peptide cOB1 from native Enterococcus faecalis forms amyloid-like structures, A new paradigm for peptide pheromones. Journal of Peptide Science. 25, 3178 (2019).
- Stoodley, P., Debeer, D., Lewandowski, Z. Liquid flow in biofilm systems. Applied and Environmental Microbiology. 60, 2711-2716 (1994).
- Birjiniuk, A., et al. Single particle tracking reveals spatial and dynamic organization of the E. coli biofilm matrix. New Journal of Physics. 16, 085014 (2014).
- Chew, S. C., et al. Dynamic remodeling of microbial biofilms by functionally distinct exopolysaccharides. mBio. 5, 01536 (2014).
- Cao, H., et al. Revealing region-specific biofilm viscoelastic properties by means of a micro-rheological approach. Npj Biofilms and Microbiomes. 2, 5 (2016).
- Galy, O., et al. Mapping of bacterial biofilm local mechanics by magnetic microparticle actuation. Biophysical Journal. 103, 1400-1408 (2012).
- Rogers, S. S., vander Walle, C., Waigh, T. A. Microrheology of bacterial biofilms in vitro, Staphylococcus aureus and Pseudomonas aeruginosa. Langmuir. 24, 13549-13555 (2008).
- Hart, J. W., Waigh, T. A., Lu, J. R., Roberts, I. S. Microrheology and Spatial Heterogeneity of Staphylococcus aureus Biofilms Modulated by Hydrodynamic Shear and Biofilm-Degrading Enzymes. Langmuir. 35 (9), 3553-3561 (2019).
- van Merode, A. E. J., van der Mei, H. C., Busscher, H. J., Krom, B. P. Influence of culture heterogeneity in cell surface charge on adhesion and biofilm formation by Enterococcus faecalis. Journal of Bacteriology. 188, 2421-2426 (2006).
- Tariq, M., Bruijs, C., Kok, J., Krom, B. P. Link between Culture Zeta Potential Homogeneity and Ebp in Enterococcus faecalis. Applied and Environmental Microbiology. 78, 2282-2288 (2012).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ, 25 years of image analysis. Nature Methods. 9, 671-675 (2012).
- . Oracle.com Available from: https://docs.oracle.com/en/java (2021)
- Hoffer, M., Poliwoda, C., Wittum, G. Visual reflection library, a framework for declarative GUI programming on the Java platform. Computing and Visualization in Science. 16, 181-192 (2013).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15, 1007978 (2019).
- Malhotra, K., et al. Development of a New Bead Movement-Based Computational Framework Shows that Bacterial Amyloid Curli Reduces Bead Mobility in Biofilms. Journal of Bacteriology. 202, 00253 (2020).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유