Method Article
バイオフィルム材料の不均一性の3次元解析のためのビーズ運動に基づく計算フレームワーク
要約
1 μmのカルボン酸ビーズの移動パターンを不均一な細菌バイオフィルムを通じて分析および定量する方法について説明します。移動パターンの比較は、バイオフィルムの材料特性の違いを定量化するために使用できます。
要約
細菌バイオフィルムの材料特性の違いは、異なる細菌種のバイオフィルム、同じ種内で異なる成長条件下で、およびマトリックス修飾分子で処理した後で観察されています。3Dバイオフィルムの材料特性をより定量化するために、 Enterococcus faecalis、Salmonella enterica 血清型、チフス菌、 大腸菌 のバイオフィルムの違い、および腸内細菌科バイオフィルムの剛性を確認する上でのアミロイドカーリの役割を調べるための実験的および計算ワークフローが開発および適用されました。バイオフィルム中の1μmカルボン酸ビーズの時空間ダイナミクスを、20μmの3Dバイオフィルムで20分間にわたって追跡しました。4D画像スタックは、ImageJのMosaicプラグインを使用して処理され、ビーズの動きの3D軌道データが生成されました。この軌跡データは、新たに開発されたBead Evaluatorツールボックスで解析され、軌跡の寿命、ビード速度、軌跡に沿った細胞密度、バウンディングボックス情報などの移動データが計算され、csvファイルに保存されました。この論文では、実験のセットアップと画像記録からビード軌道の計算と分析までのワークフローについて説明します。カーリ含有バイオフィルムの構造は、カーリ変異体および腸球菌バイオフィルムよりも安定したビーズ相互作用とより少ないビーズの動きをもたらしました。ビーズの速度と軌道バウンディングボックスの体積を測定すると、ビーズの動きは細胞密度に強く依存しているようには見えず、バイオフィルムの他の材料特性がビーズのダイナミクスを制御しているという仮説を支持しています。この手法は、マトリックス修飾処理の前後のバイオフィルムだけでなく、異なるマトリックス組成のバイオフィルムの違いの定量に広く適用できます。
概要
細菌のバイオフィルムは、ヒトの微生物叢の一部として遍在し、分子と絶えず相互作用します。これらの分子のサイズは、1 nmの抗生物質や1〜3 mmの細菌から、胃腸管内のより大きな繊維粒子までさまざまです。単一または複数種のバイオフィルムの組成は、材料特性に影響を与え、したがってバイオフィルム1,2,3,4,5を通る粒子の移動パターンに影響を与えます。一例は細菌性アミロイドであり、これは保存された線維性交差βシート構造6を有する。アミロイドカーリは、大腸菌やサルモネラ菌の血清型であるチフス菌などの腸内細菌によって発現され、遺伝子は他の複数の細菌門で検出されています7。バイオフィルムのさまざまな材料特性は、カーリ8,9の影響を受けます。Curliは、細胞外DNA(eDNA)やセルロース10,11など、マトリックスの他の成分と直接相互作用します。カーリーは細胞を取り囲み、細胞膜の硬直性12およびバイオフィルム13の全体的な粘弾性特性に影響を与える。カーリーは、フィブロネクチンに結合することにより引張強度の増加を媒介し、その結果、強力なガラス表面付着が増加します14。入ってくるバクテリオファージはカーリに結合し、バイオフィルムへのファージの侵入を制限します15。
マルチテストコーティングウェルスライドを使用して、厚さ約20 μmのEnterococcus faecalis、E. coli、およびSを分析する場合。共焦点顕微鏡を用いたチフス菌のバイオフィルム、大腸菌とS.の明確な違い。Typhimurium10,16およびE. faecalisのバイオフィルム(現在の研究)が観察されました。腸内細菌科のバイオフィルムは剛性が高く、細胞密度の低い領域もイメージングが容易でしたが、ラインとフレームの平均化を使用してE. faecalisのバイオフィルムの鮮明で高解像度の写真を取得するには、イメージングプロセス中に細胞の安定性に十分な表面張力を誘導するためにスライドに圧力を加える必要がありました。カーリのような細菌性アミロイドは高度に秩序化された構造を形成し、それらが比較的硬直している可能性を示唆している17。これは、アミロイドカーリが大腸菌とSの硬直を誘発している可能性があるという仮説を動機付けました。チフス菌のバイオフィルム。E. faecalisが研究された条件下でアミロイドを発現していたという明確な証拠はなかった。Espタンパク質は、E. faecalisのより病原性の高い株に関連するピリン遺伝子であり、最近、アミロイド構造を産生することが示されました18。しかし、blastnおよびblastp検索を使用して、この遺伝子は、これらの研究で使用されたE.faecalis共生型OG1RF株では検出されませんでした。OG1RFによって産生されるフェロモンcOB1は、アミロイド様構造を形成することができる19。しかし、与えられたバイオフィルムの成長条件とアミロイド検出法では、以前にS. E. faecalis, OG1RFアミロイドの10を染色したチフス菌のアミロイドは検出できなかった(データは示さず)。粘性のあるE. faecalis、E. coli、およびS. typhimuriumの全体的な材料特性を比較するとともに、Sのアミロイド変異体を使用して腸内細菌科のバイオフィルムへのアミロイドの寄与を決定するために、新しい4次元(4D)画像技術が開発されました。 チフス菌と大腸菌。
過去には、蛍光ビーズは、マイクロレオロジー20,21,22,23,24,25を使用してバイオフィルムの材料特性を2次元(2D)で分析するために成功裏に使用されていました。これは、バイオフィルム26の様々な深さにおける2D光学スライスを研究することにより、3次元バイオフィルムに適用することができる。現在の手法は、4Dモデリングで使用するために、1μmのマイクロスケールビーズを3Dで経時的に追跡するために開発されました。その根拠の一部は、4Dモデリングを使用して胃腸内細菌叢コミュニティを介したプラスミドの動きを理解するという包括的な概念でした。直径1μmの蛍光電荷カルボン酸ビーズを使用したのは、これらのビーズがサイズと電荷の点で、プラスミドの移動と維持のための選択されたモデル生物であるE.faecalisによく対応しているためである27、28。バイオフィルムの物理的特性を定量化するための4Dアッセイを開発しました(図1A)。考案された方法論では、バイオフィルムにビーズを添加し、その時空間軌跡を厚さ10〜20μmのバイオフィルムを通じて10〜20分間にわたって記録しました。次に、3Dのビーズ軌道を、新しく開発されたツールボックスを使用して、軌道の長さ、ビーズ速度、軌道バウンディングボックスの体積(軌道を含む最小ボックス)、およびバウンディングボックスのセルラー密度の観点から定量化しました。次のプロトコルを使用して、バイオフィルムを含む細菌とビーズの4D画像データを生成し、ImageJ29とプラグインMosaicでデータを前処理し、Bead Evaluatorツールボックスでビーズの軌跡を解析できます。
この手法には、材料特性の調査だけでなく、粒子や細菌の動きを3次元で追跡するための複数の用途があります。例えば、この技術の初期バージョンは、バイオフィルム16の構造的完全性に対するカーリに対するモノクローナル抗体の影響を特徴付けるために使用された。フルバージョンには、バイオフィルム材料の特性をより詳細に分析するための複数のツールがあり、バイオフィルムに対するモノクローナル抗体治療の効果を調べるために引き続き使用されています。異なる電荷の粒子を使用して、バイオフィルムの材料電荷特性と、異なるマトリックス組成のバイオフィルムを通る粒子の移動を調べることができます。これは、流れていないバイオフィルムで観察されたビーズの動きの原因となる材料特性を明らかにする2Dマイクロレオロジーの結果を比較するために使用できます。この手法は、バイオフィルム組成の異なる領域を持つ混合種のバイオフィルムにも使用できます。バイオフィルムは、マイクロ流体デバイスやフローセルで流れの下でライブイメージングでき、静的バイオフィルムとフローバイオフィルムの間の材料特性の変化や、粒子の動きに対する流れの影響を調べることができます。この技術は、蛍光標識された細菌にも適用して、バイオフィルムコミュニティを介した外因性細菌の動きを特徴付けることもできます。蛍光標識されたドナー細菌、蛍光標識されたレシピエント細菌、および蛍光標識されたプラスミドの3色を使用して、プラスミドの移動、ドッキング、および移動を追跡できます。
プロトコル
1. バイオフィルムの調製
注:分析用のバイオフィルムは、バイオフィルムを光学ガラス表面上に形成できる任意の方法を使用して成長させることができます。バイオフィルム構造は、プロトコルの洗浄および/または取り付けステップ中に構造が破壊されないように、光学面に十分に接着する必要があります。以下では、24ウェルプレートの96ウェル光学ボトムプレートと12mmガラスカバースリップの手法について説明します。その他のオプションには、さまざまなサイズの光学底板と、フロー付きまたはフローなしのオプティックフローチャンバーが含まれます。
- バイオフィルムのセットアップ
- プレートのウェルに細菌増殖培地を追加します。この研究では、 E. faecalis、24ウェルプレートに2mLのTodd-Hewitt(TH)を加え、0.4mLのTHを光学ボトム96ウェルプレートに加えます。 Sの場合。チフス菌、 大腸菌 、および同質遺伝子 のcsgAB カーリ変異体は、0.7 mLの無塩ルリアブロス(LB)をウェルに加えます。光学ボトムチャンバーを使用する場合は、ステップ1.2に進みます。
- 12 mm #1.5ガラス光学カバースリップをペトリ皿に入れ、エタノールで覆います。
- 鉗子を使用してカバーガラスをはがし、炎を使用して残りのアルコールを火に点火します。アルコールが燃え尽きるのを待ちます。炎を使って火をつけるだけです。カバースリップを炎の中に持たせないでください。ひび割れを防ぐために、カバーガラスをウェルに入れる前に、カバーガラスを10〜20秒間冷ましてください。
- カバースリップを培地が入ったウェルに斜めに置き、培地の上に置かないようにします。カバースリップを乾いたウェルに追加してからメディウムを追加すると、カバースリップがウェルの底にくっつくため、使用しないでください。
- 滅菌ピペットチップを使用して、カバースリップを培地を含むウェルの底に慎重に押し込みます。
注意: 光学ガラスカバースリップまたは光学底板の厚さ(#1または#1.5)を共焦点顕微鏡光学系の厚さに合わせることを忘れないでください。
- バイオフィルムを適切な条件下でインキュベートし、バイオフィルムを成長させます。これらの研究では、 E. faecalis バイオフィルムを 37 °C で静的培養物として成長させ、30 °C で E. coli バイオフィルムを好気的に成長させます。
- これらの研究では、 E. faecalis バイオフィルムを 2 日間、朝と夕方に媒体スイッチで成長させます。バイオフィルムの損傷や剥がれを防ぐため、プレートを慎重に傾けてください。ピペットチップをウェルの下端近くに置き、培地をゆっくりと引き出します。同じ方法で最初のmLの新鮮な培地を追加します。2 mL 目を培地/ウェル界面付近にゆっくりと加えます。
- これらの研究では、最適なカーリー生産のための成長条件を使用します。Sを育てます 。 チフス菌のバイオフィルムを28°Cで6〜8日間インキュベートし、傾斜させてバイオフィルムをスライドの約2/3に付着させ、その後、気液界面上のペリクルとして成長させました。これは、中程度の変更なしで行われました。培地が乾燥するのを防ぐために、24ウェルプレートをウォーターパン付きのチャンバーに入れます。
2. 4Dイメージング
- バイオフィルムマウントの準備
- クリムゾン1μmカルボン酸FluoSpheresビーズをPBSで1:50希釈します(PBS1 mLに2 x 107 ビーズ)。光学ボトムチャンバーを使用する場合は、1 μLから300 μLのビーズ調製物に希釈してSyto9を添加します。
- (オプション)バイオフィルムを洗浄して、増殖培地に自家蛍光がある場合は、増殖培地の痕跡を取り除きます。これらの実験では、1 mLのPBSで2回洗浄し、プレートを慎重に傾け、ピペットチップをウェルの下端近くに置き、培地をゆっくりと引き出します。先端を下端近くに配置してPBSを追加し、ゆっくりとウェルを満たします。この手法は、24ウェルプレートのカバースリップに加えて、光学ボトムウェルにも使用します。
- メディアまたはPBSを取り外します。2.1.1で調製した希釈したCrimsonビーズをバイオフィルムに加えます。これらの研究では、カバーガラスに1 mLのビーズ(2x107)を、0.2 mLのビーズ(4x106)とSyto9を96ウェル光学ボトムプレートに加えます。
- 室温で1分間インキュベートし、ビーズが結合します。
- ビーズを取り除き、バイオフィルムをPBSで一度優しく洗い、関連付けられていないビーズを取り除きます。これらの研究では、カバースリップについて、1 mLのPBSで優しく洗浄し、ウェルに1 mLのPBSを補充し、2.1.6に進みます。96ウェル光学ボトムプレートの場合、バイオフィルムを0.2 mLのPBSで優しく洗浄し、ウェルに0.2 mLのPBSを補充します。これで光学底部チャンバーをイメージングする準備が整いましたので、手順2.2に進みます。
- (カバースリップを使用する場合)コーティングされた10ウェルマルチテストスライド上のウェルの中央に、1 μLのSyto9(緑色蛍光DNA染色剤、メーカーの指示に従って希釈)を加えます。これらのコーティングされたカバーガラスは、23-25μmの井戸深さを有する。
- (カバースリップを使用する場合)アルコール炎滅菌鉗子を使用して、カバースリップをウェルから慎重に取り外し、Syto9ドロップを含むウェルに反転させます。カバーガラスを1mLのPBSに入れたままにしておくと、ウェルから取り出しやすくなり、バイオフィルムから無関係なビーズを洗い流すことができます。
- (カバースリップを使用する場合)カバーガラスをスライドさせたり押し下げたりしないように、フィンガーマニキュアでカバーガラスを慎重に密封します。これにより、表面張力が発生し、粘性が高く、剛性の低いバイオフィルムの動きが停止する可能性があります。
- (カバースリップを使用する場合)マニキュアを乾かします。カバーガラスの外面を70%エタノールで丁寧に拭きます。上記の理由により、カバーガラスに圧力をかけずに拭いてください。
- 共焦点イメージング
注:これらの研究では、63倍対物レンズを備えたTCS共焦点システムを備えた逆スペクトルイメージングレーザー走査型共焦点顕微鏡を使用します。このスコープは、4Dビデオの生成に使用されます(図1)。3D バイオフィルムは、厚さ 18 〜 20 μm のバイオフィルムを介して 0.5 μm ステップでキャプチャされた Z スライスで構成され、36 〜 40 枚の Z スライスが生成されます。各3Dバイオフィルムのキャプチャには50〜60秒かかります。スライスが合わさってフレームを構成し、3Dバイオフィルムとして視覚化できます。このプロセスを 20 回繰り返して、合計 18 分から 20 分の追跡時間で 4D タイム ラプス ビデオを生成します。- 蛍光色素を捕捉するようにスコープを設定します。これらの研究では、Syto9(細菌のDNA染色)を488 nmレーザーで励起し、495〜540 nmの発光を測定します(ライカSp5はスペクトルイメージング顕微鏡です)。633 nmレーザーで深紅色(赤)ビーズを励起し、650 nmから700 nmまでの発光を測定します。これらの蛍光色素の設定は、任意の蛍光色素を捕捉するように調整できます。
- xyzt イメージングモードを選択します。
- 高密度領域と低密度領域が混在するバイオフィルムの領域を特定し、バイオフィルムの厚い領域と薄い領域の粘弾性特性の違いを捉えます。
- Zスタックを18-20μmの厚さにセットします。カバースリップに取り付けられたバイオフィルムでは、バイオフィルムの最上部と下部がガラスに接触しないようにして、ビーズの閉じ込めアーティファクトを回避します(ディスカッションを参照してください)。
- ゲインとオフセットを調整して、バイオフィルムの最も明るいスポットの強度のダイナミックレンジ全体を使用します。これにより、バイオフィルムの下層からのシグナルの重なりが最小限に抑えられます。
- スライスの厚さを0.5μmに設定します。これにより、ビーズ情報を失うことなく迅速なイメージングが可能になります。
- 解像度を 512 x 512 (0.48 μm) に設定します。これにより、迅速なイメージングが可能になり、バイオフィルムの構造とビーズの動きの詳細を確認するのに十分な解像度の画像が生成されます。
- イメージング時間を最小限に抑えます。
- 20スタックをキャプチャするように設定します。
- .lif(または同様の共焦点ファイル)として保存します。4DムービーはImageJで生成することができます。
3. ImageJによる4Dバイオフィルムビデオの生成
- ImageJ で .lif ファイルを開き、Hyperstack でスタックを表示、スタック順序 XYCZT、カラー モード: 色付け、自動スケールをオンに設定します。次に 、[OK]を押します。
- 「イメージ」>「カラー」>「分割チャンネル」を選択します。
- [イメージ] > [カラー] > [チャンネルを結合] > [合成を作成] > [OK] を選択します。
- 「プラグイン」>「3Dビューア」>「チャンネル」を選択し、青色>「OK」のチェックを外します。
- [編集] > [コンテンツを表示] をオンにし、[境界ボックスを表示] >オンにします。
- マウスをクリックしたままにすると、画像が選択されます。次に、クリックボタンを押し続けながら画像を回転させます。バイオフィルムの底が底にくるように画像を回転させ、ビーズの視覚化をサポートする角度にします。次に、イメージを解放します。
- ウィンドウの下部にある赤い [録画 ]ボタンを押して、ビデオを録画します。
- 圧縮としてjpegを使用して.aviファイルとして保存します。
4. 軌跡データの生成
- オープンソースツールのImageJ(https://imagej.net/Fiji)とパーティクルトラッキングプラグインのMosaic(https://imagej.net/MOSAICsuite)をインストールします。
- ビーズとバクテリアの 2 つのチャンネルを含む .lif ファイルをインポートします。チャンネルを分割し、ファイルを別々に保存します。
- ImageJ で、画像のボクセル サイズ (X、Y、Z ディメンション) と時間ステップ サイズをテキスト ファイルに格納します。
- ImageJで、[ プラグイン] |Particle Tracker 2D/3Dをモザイクして起動します。
- 次のパラメーターを入力します:Radius:3、Cutoff:0.003、Per/Abs:0.12、Link Range:2、Displacement:10.00、ダイナミクスはBrownianに設定されています。これらの実験でブラウンを選択した根拠については、結果をご覧ください。
- 軌道を生成し、軌道リストを.csvファイルとしてエクスポートします。
5. 軌道の解析
- VRL Studioのインストール(https://vrl-studio.mihosoft.eu)
- バイオフィルムプロジェクト(https:// neurobox3d.github.io/Biofilm/)をダウンロードし、VRL Studioで起動
- 軌跡ファイルを ImportData に読み込みます
- ProcessTrajectoriesでx、y、zのピクセルサイズを指定します(ImageJを使用してこれらの値を見つけます)。
- フレーム間隔を ComputeVelocity で指定します (ImageJ を使用してこの値を見つけます)。
- バクテリアのtiffファイルをComdensityにロードします(手順3.2を参照)。
- 速度データの出力パスを SaveVelocityDataToFile に、軌跡データの出力パスを SaveTrajectoryDataToFile に設定します。
- SaveVelocityDataToFile と SaveTrajectoryDataToFile を呼び出します。
- 分析のためにデータをExcelにインポートします。これらのデータには、軌道の長さ、軌道の寿命、軌道のバウンディングボックスの寸法と体積、平均ビード速度と分散が含まれます。この解析では、Syto9 標識細菌のチャネルを使用して重み付け変数を計算し、局所的な (特定の軌跡境界ボックス内) 細胞密度を計算します。この解析の結果、加重平均速度と分散、およびバウンディングボックス分散の加重平均と分散が計算されます。
結果
この方法は、カーリが大腸菌とSに剛性を与える可能性があるという仮説を検証するために使用されました。チフス菌のバイオフィルムは、共焦点顕微鏡実験中のビーズの動きを減少させます。現在のツールボックスを使用して、Enterococcus faecalis commensal型OG1RF株の材料特性をサルモネラ菌血清型Typhimurium、大腸菌、およびそれぞれの同質遺伝子カーリ変異体と比較しました(図1Bおよび補足ビデオ1、補足ビデオ2、補足ビデオ3、補足ビデオ4、補足ビデオ5、補足ビデオ6 ).バイオフィルム材料の特性は、剛性(eDNAに結合したカーリーなど)や、負に帯電したビーズとバイオフィルム細胞およびマトリックス材料との間の静電的および疎水性相互作用、および細胞密度に関して異なる可能性があります。
再現
Biofilmツールボックスは、VRL-Studio32内のGroovy30およびJava31でプログラムされ、すべての計算コンポーネントのユーザーインターフェース(UI)の自動生成によるモジュール式ワークフロー設計を可能にしました。これにより、ワークフローの自動化が可能になり、結果を分析する際に実験者によって誘発される意図しないバイアスが排除されました。
MSDを用いたバイオフィルムの動きの種類確認
Particle Tracker 2D/3Dを使用した軌道の解析では、ビーズの動きの種類ごとにさまざまなダイナミクス設定が可能です。これらの研究では、E. faecalisが非運動性細菌であり、大腸菌とサルモネラ菌はバイオフィルムにべん毛を発現しないことから、「ブラウン運動」(すなわち拡散駆動運動)の設定を選択し、実験は流れのない閉鎖系で行われました。この設定は、ビーズの計算された平均二乗変位(MSD)によってさらに検証できます。定義 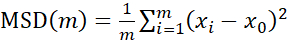 (m は軌跡セグメントの数) を使用して、各軌跡のコースにおける MSD の変化を計算できます。線形軌道は、拡散ビードの動きを示します(図2A)。2次最小二乗フィッティングを使用して、バイオフィルム内のすべてのビーズの平均移動パターンを計算し、支配的な線形秩序を示し、駆動力として受動的拡散を検証しました(図2A-2F)。
(m は軌跡セグメントの数) を使用して、各軌跡のコースにおける MSD の変化を計算できます。線形軌道は、拡散ビードの動きを示します(図2A)。2次最小二乗フィッティングを使用して、バイオフィルム内のすべてのビーズの平均移動パターンを計算し、支配的な線形秩序を示し、駆動力として受動的拡散を検証しました(図2A-2F)。
バウンディングボックス解析。
このツールボックスは、ImageJ Mosaic と Particle Tracker 2D/3D を使用して軌道を生成し (ステップ 4)、次に自動バイオフィルム解析パイプラインを使用して、バイオフィルム材料の特性を比較するために使用できるビード軌道に関する重要なデータを生成します。μm3 単位のバウンディング ボックスの体積は、軌道を含む最小ボックスを構築し、その体積を測定することによって測定されました (図 3)。
E. faecalisバイオ フィルムは、バウンディングボックスの値が1〜6000μm3 で、より多くのビーズの動きを示します(図3B、3C、および3D)。この結果から、約25 μmのコーティングウェル(図3C)でコーティングされたスライドに取り付けられたガラスカバースリップで見られる動きと、光学ガラスウェルの底部で成長して直接イメージングされたバイオフィルム(図3D)では、ほとんど違いなく同等の結果が得られることが確認されています。唯一の違いは、 E. faecalis biofilmsの取り付けカバースリップの上部近くには、寿命が10分を超える安定した軌道を記録できると同時に、小さなバウンディングボックスを登録できるのに対し、光学底板では、移動度の高いビーズを選択数登録できることでした。まとめると、これは、スライドガラスをマウントすると、取り付けられたカバースリップバイオフィルムのスライドに対するバイオフィルムの上部のシステムの表面張力が変化し、最終的に粘度の低いバイオフィルム領域の一部のビーズの移動性が低下した可能性があることを示唆しています(図3B、3C、3Dおよび3I)。このカテゴリーに分類される軌道はたまたま非常に小さな割合であり、この少数のトラップビーズでも、取り付けられたカバースリップ上の E.faecalis の平均MSDは、光学底板バイオフィルムから計算されたMSDよりもわずかに高かった(図3)。
S. チフス菌および大腸菌のビーズ軌道は、大腸菌のバウンディングボックスが1〜6000μm3、Sのバウンディングボックスが1〜5000μm3の同質遺伝子カーリ変異体と比較して、0〜10μm3(図3A、3B、3E、および3F)のバウンディングボックス容積が小さかった。チフス菌(図3A、3B、3F、3H)は、より大きなビーズの移動性を示しています。これらの結果は、アミロイドの存在がバイオフィルムの剛性の増加と相関し、ビデオで顕著なバイオフィルムの動きがないことと一致していることを示唆しました。バウンディングボックスの体積は、バイオフィルムの低密度領域でも一貫して小さかった(0〜10μm3)。この観察結果は、バイオフィルム10の低細胞密度領域にcurliが存在する可能性があるという以前の観察結果と一致しています。
腸内細菌科のバイオフィルムは、気液界面上でペリクルとして成長するため、光学底板上での挙動を比較することはできませんでした(ステップ1.2.2)。カバースリップを使用する場合、ペリクルは界面でカバースリップに取り付けられ、カバースリップを取り外すと、ペリクルがカバースリップ上に置かれ、単一の画像面が作成されます。斜めに成長させた光学底板で、井戸内に液体が残っている状態でイメージングを行いました。これは、ペリクルがまだ光学底部より上に浮いており、ライカSp5のような倒立スコープの作業深度からペリクルが外れていることを意味しています。バイオフィルムを顕微鏡の作業深度に持ち込むのに十分な媒体を取り除くと、20分間のイメージングプロセスで標本が乾燥してしまいました。
全体として、プロットは補足動画の視覚的観察を確認し、観察されたMSDの差と一致しています(図3I および3J)。
軌道の寿命
軌跡の寿命は、ビーズが登録された連続フレームの数として測定されました(図3)。
より粘性のある流体のような E.faecalis バイオフィルムでは、すべてのビーズの軌道寿命が10分未満であり、 E.faecalis バイオフィルムの軌道の大部分は2〜5分の範囲でした。しかし、軌跡の寿命が短いビーズは、全イメージング時間枠にわたって E. faecalis バイオフィルムの目視検査によって特定できる可能性があります(補足ビデオ1および2)。したがって、ビーズが登録された軌道に沿って移動し、バイオフィルムから断続的に解離して軌道を終了し、バイオフィルムと再結合し、その時点で新しい軌道が開始される可能性がある。これは最終的に、バイオフィルムにビーズが連続して存在すると、軌道寿命が短くなります。この手法を使用すると、特に粘性のあるバイオフィルムでは、軌道の寿命がビーズがバイオフィルムに関連付けられている合計時間を過小評価する傾向があることに注意することが重要です。
Sで。 バウンディングボックスの体積が小さいチフス菌のバイオフィルムは、ビーズの大部分(約80%)が16〜20フレームの長い軌道寿命を持ち、これはリアルタイムで約15〜20分に相当します(図3A、3G、3H)。これらとは対照的に、アイソジェニックカーリ変異体バイオフィルムは、バウンディングボックス容積が1〜6000μm3(大腸菌)から1〜5000μm3(S. チフス菌)(図3A、3B、3F、3H)。しかし、E. faecalis バイオフィルムが >70% の軌道を持ち、バウンディング ボックスの体積が 10 μm3 を超えるのとは対照的に、腸内細菌科の生物フィルムは、バウンディング ボックスの体積が 10 μm3 を超えるビーズの軌跡を 30% しか記録しませんでした。全体的なビーズの軌跡寿命はcurli変異体のバイオフィルムでは短かったが、一部の軌跡はかなりのビーズの動きと長い軌跡寿命を反映していた(図3H)。この観察結果は、この変動性が、粘弾性などのバイオフィルム材料特性の変化、および/または電荷などの粒子表面化学の変化に対応している可能性があることを示している可能性があります。
ビードの軌道の長さと速度の解析
軌道の長さは、ビーズが移動した距離をμm単位で測定したものです。この測定値は、μm/s単位のビーズ移動速度と一致しています。バウンディングボックスの体積が大きいことと同様に、 E. faecalis バイオフィルムのビーズは、カーリを含むバイオフィルムの<4μmに対して、5〜20μmと10倍長い軌道を示しました。短い軌道と一致します(図4A)。 E. faecalis ビーズは、最大15倍の高速速度を測定し、大部分のビーズは0.01〜0.15μm/sの範囲の速度を持ち、速度は0.006μm/s<図4B)でした。それにもかかわらず、カーリ変異体バイオフィルムは、 E. faecalis バイオフィルムと比較して全体的に低い速度と短い軌道を測定しましたが、親株を含むカーリよりも長い軌道と高い速度を測定しました(図4A および4B)。
注目すべきは、curli30 の繊維状格子状構造が異方性で移動性に影響を及ぼし、XY平面内の移動性を減少させ、z方向への移動性の増加を可能にするという事実である(図2G)。カールを含むバイオフィルムの約50個のユニークなビーズに属する大きな軌道プール(約800)は、モザイク粒子追跡の制限と一致しており、これらの高速で移動するビーズのそれぞれをx、y、zの1つのビーズとしてカウントします。この観察結果を確認するためには、さらなる研究とソフトウェア開発が必要になるでしょう。
ビーズの移動性における細胞密度依存性の解析
ビーズの動きの細胞密度への依存性は、加重平均速度と分散、およびバウンディングボックス体積の平均/加重平均と分散を使用して決定されました。Syto9標識細菌の2番目のイメージングチャネルを使用して、重み付け速度の計算における局所細胞密度を計算しました。セルラー密度は、各軌跡エッジのバウンディングボックス上のSyto9ボクセルデータを平均化することによって計算されました(図5、右)。したがって、ビーズ速度は、エッジワイズ(局所)セル密度によって重み付けできます。細菌の可視化に使用できる染色剤には、細胞壁の染色、膜、DNA含量の染色など、複数の種類があります。細胞密度を決定するためにSyto9が選択されたのは、どの光学Zスライスが視覚化されているかに関係なく、最も一貫した信号を提供するためです。エンベロープ染色(細胞壁および膜)は、Zスライスの位置によって異なるシグナルを発します。Zスライスにセルの上部または下部が含まれている場合、Zスライスがセルの中央を通り、セルの輪郭のみが染色されている場合よりも信号が強くなります。
赤チャネルからのビード軌道は 20 フレーム追跡され、個々の軌跡の最小寿命は 2 フレーム、最大寿命は 20 フレームで、フレームを接続する 19 の軌道セグメントがありました(図 5)。ビーズの移動度の細胞密度依存性を研究するために、ボクセルあたりのGFP強度(512x512ボクセル画像の個々の測定値のそれぞれ)を決定しました。ビーズ軌跡の各セグメントの周囲のセル密度は、セグメントのバウンディングボックス内の局所平均密度として計算されました。
一部のバイオフィルムでは、統計的に有意な密度依存性が文書化され(図6)、最も顕著なのは、ガラスカバーガラス上で成長させ、マルチウェルスライドに反転させたE.faecalisバイオフィルムです(図6A)。それどころか、96ウェルプレートの底部で成長したE.faecalisバイオフィルム(図6B)は、密度依存性を示さなかった。結論として、これは、流動性の高いE. faecalisバイオフィルムがマルチウェルスライドにマウントされているためにわずかに圧縮される可能性があることを示唆しており、これは、より速く移動するビーズの数の減少と、スライドガラスに対してバイオフィルムの上部に閉じ込められる小さなバウンディングボックスボリュームを持つビーズと一致しています(図3Cと図3D)。サルモネラ菌と大腸菌のバイオフィルム(図6、C、6D)、およびそれらの同質遺伝子変異体(図6E、6F)は、細胞密度依存性が軽度または全くないことを示しました。
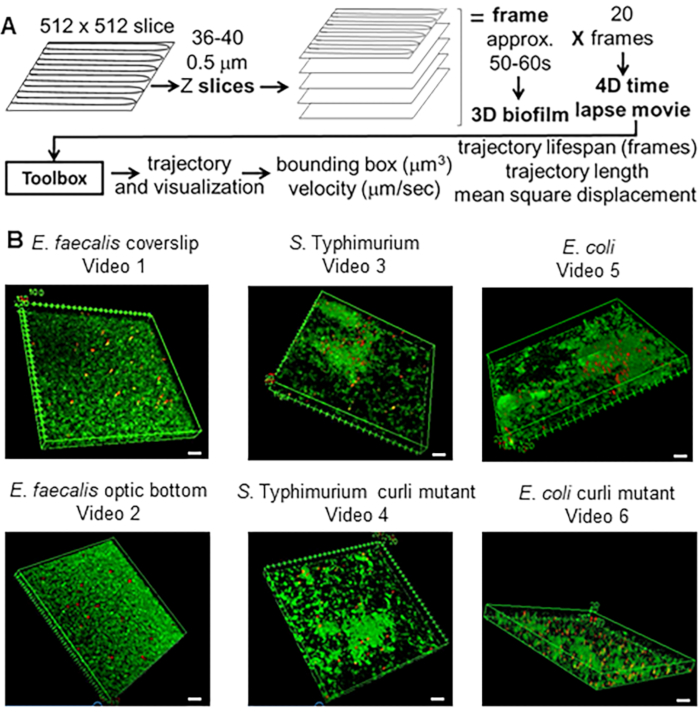
図 1.イメージングおよび解析パイプライン(ステップ2-4)(A)バイオフィルムは、2.2で概説されているようにイメージングされます。画像化されたバイオフィルム(3参照)を使用して、4で説明したようにビーズの軌跡を生成しました。軌跡を使用して、関連データを分析ツールボックス(5を参照)で計算しました(B)バイオフィルムは、カバースリップ(E.faecalis、 ビデオ1)、Sのステップ1で説明されているように成長しました 。 チフス菌(ビデオ3)、 大腸菌 (ビデオ5)、および同遺伝子のカーリ変異体(ビデオ4、ビデオ6)、または光学底部96ウェルプレート(E.faecalis ボトム、 ビデオ2)。白いスケールバーは20mmです。この図は(31)の許可を得て転載しています。 この図の拡大版を表示するには、ここをクリックしてください。
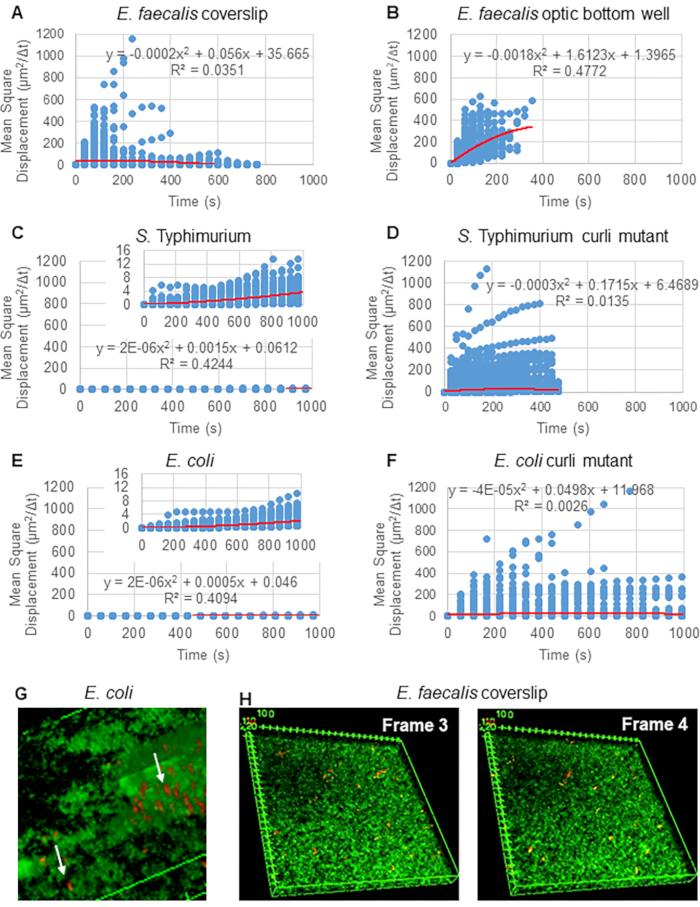
図 2.バイオフィルム内のビーズの動きは、本質的に拡散性であると判断されました(ブラウン運動、ステップ5を参照)(A-F)MSDデータは、ブラウン運動を検証するための線形挙動(赤い線)を示しています。バイオフィルムは、ステップ1(G)に従って成長させた: 大腸菌 およびチフス 菌 のバイオフィルムで観察された楕円形のビーズの動きの例、大 腸菌 4Dバイオフィルムアッセイ(H) E.faecalis 光学底井戸から採取したフレーム3とフレーム4の間のビーズパターンの大きな変化の例。バイオフィルム自体が何らかの流れを引き出し(ビデオ1と2)、フレーム3とフレーム4の向きが異なるように見えることに注意してください。この図は(31)の許可を得て転載しています。 この図の拡大版を表示するには、ここをクリックしてください。
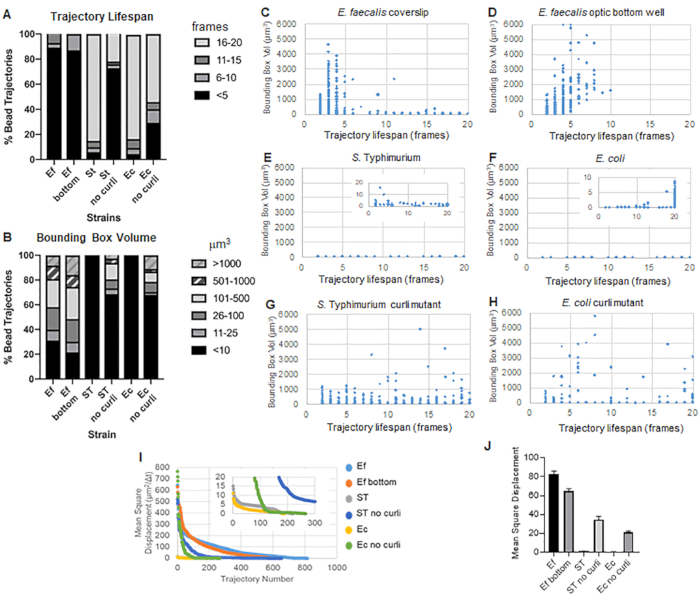
図 3.バウンディングボックスと軌道寿命を使用した剛性の違いの解析。 バイオフィルムは、ステップ1で説明したように、カバースリップ(E. faecalis, Video 1), S. チフス菌(ビデオ3)、 大腸菌 (ビデオ5)、および同遺伝子のカーリ変異体(ビデオ4、ビデオ6)、または光学底部96ウェルプレート(E.faecalis ボトム、 ビデオ2)。軌跡の寿命は、総ビーズ軌跡(A)および散布図(C-H)の%で表され、バウンディングボックスの体積(ステップ5で計算)(I)異なるバイオフィルム内のビーズMSDの比較(H)各タイプのバイオフィルムの平均MSDs。バーは、データの95%信頼区間を示します。この図は(31)の許可を得て転載しています。 この図の拡大版を表示するには、ここをクリックしてください。
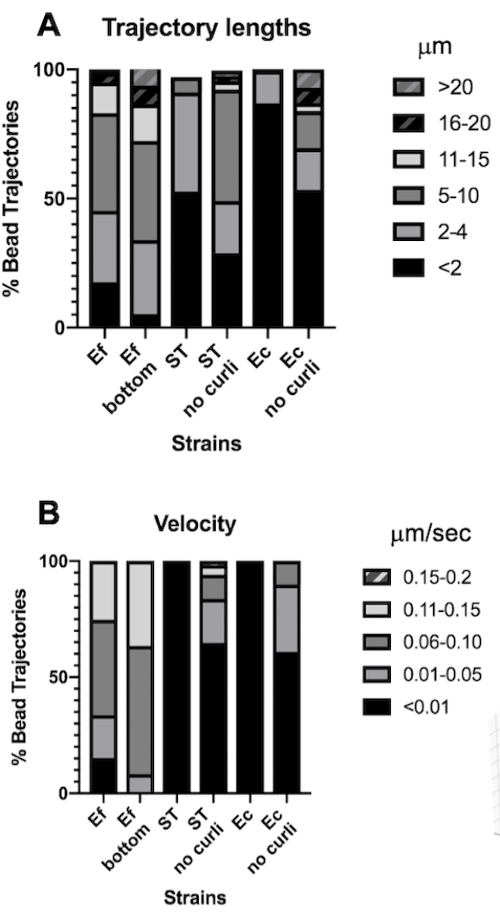
図 4.軌道の長さとビード速度の違いの解析。 バイオフィルムは、ステップ1で説明したように成長させました。軌道の長さは μm で示され、全ビード軌道の % として示されます (A)。速度はμm/sで表され、全ビード軌道の%で表されます(B)。この図は、(31)の許可を得て改作したものです。 この図の拡大版を表示するには、ここをクリックしてください。
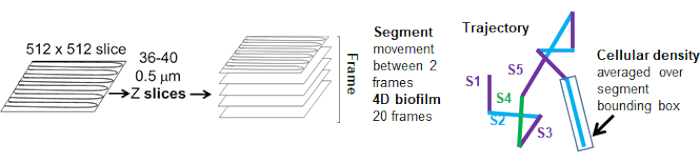
図 5.軌跡セグメント解析の概要。 この図は、(31)の許可を得て改作したものです。 この図の拡大版を表示するには、ここをクリックしてください。
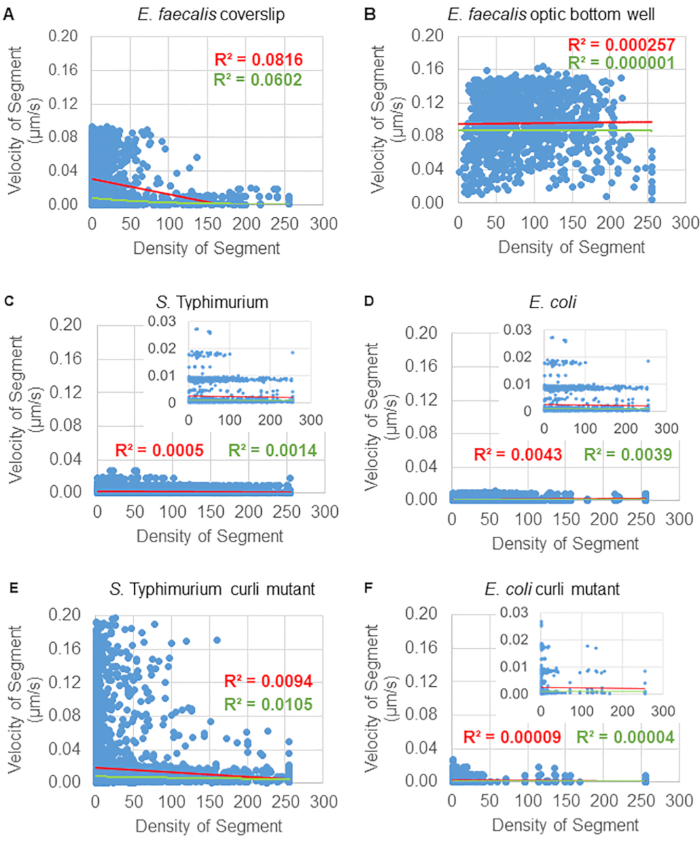
図 6.バイオフィルム分析パイプラインを使用して、バイオフィルム密度がビーズ速度に及ぼす影響を研究します(ステップ5)。 その結果、ビーズ速度は細胞密度だけに依存するわけではないことが示されました。バイオフィルムは、ステップ1で説明したように成長させました。軌跡は、個々の軌跡セグメントのスケールで解析されました(図5)。各セグメントについて、ビーズ速度(μm/s)をバウンディングボックスの細胞密度(バウンディングボックス内のボクセルあたりの平均GFP)に対してプロットしました。赤い線は線形回帰を示し、緑の線は指数回帰トレースを示します。この図は(31)の許可を得て転載しています。 この図の拡大版を表示するには、ここをクリックしてください。
補足ビデオ 1.厚さ1.5の光学ガラスカバースリップ上に成長した 24時間E.faecalis OG1RFバイオフィルムの4Dビデオ。4Dタイムラプスビデオは、顕微鏡を使用して512x512の解像度で生成されました。40枚の画像のZシリーズは、バイオフィルムの厚さ約20μmの領域を0.5μmステップでイメージングすることにより生成されました。各Zシリーズは1フレームで、キャプチャには50〜60秒が必要でした。20 個の連続したフレームをキャプチャして、4D ビデオを作成しました。ビデオの再生は約120倍速です。ビデオは、少なくとも 6 つの独立した実験の代表的なものです。このビデオをダウンロードするには、ここをクリックしてください。
補足ビデオ 2. 96ウェル光学底板上に成長した24時間E. faecalis OG1RFバイオフィルムの4Dビデオ。4Dタイムラプスビデオは、顕微鏡を使用して512x512の解像度で生成されました。40枚の画像のZシリーズは、バイオフィルムの厚さ約20μmの領域を0.5μmステップでイメージングすることにより生成されました。各Zシリーズは1フレームで、キャプチャには50〜60秒が必要でした。20 個の連続したフレームをキャプチャして、4D ビデオを作成しました。ビデオの再生は約120倍速です。ビデオは、3つの独立した実験の代表的なものです。これをダウンロードするには、ここをクリックしてください ビデオ.
補足ビデオ 3. サルモネラ菌の血清型であるチフス菌のバイオフィルムATCC 14028を1.5枚の厚い光学ガラスカバースリップで6〜7日間成長させた4Dビデオ。4Dタイムラプスビデオは、顕微鏡を使用して512x512の解像度で生成されました。40枚の画像のZシリーズは、バイオフィルムの厚さ約20μmの領域を0.5μmステップでイメージングすることにより生成されました。各Zシリーズは1フレームで、キャプチャには50〜60秒が必要でした。20 個の連続したフレームをキャプチャして、4D ビデオを作成しました。ビデオの再生は約120倍速です。ビデオは、3つの独立した実験の代表的なものです。これをダウンロードするには、ここをクリックしてください ビデオ.
補足 ビデオ 4. サルモネラ・エンテリカ 血清型 Typhimurium biofilms ATCC 14028 curli (csgBA) 変異体の4Dビデオを、1.5枚の厚い光学ガラスカバースリップで6〜7日間増殖させた。 4Dタイムラプスビデオは、顕微鏡を使用して512x512の解像度で生成されました。40枚の画像のZシリーズは、バイオフィルムの厚さ約20μmの領域を0.5μmステップでイメージングすることにより生成されました。各Zシリーズは1フレームで、キャプチャには50〜60秒が必要でした。20 個の連続したフレームをキャプチャして、4D ビデオを作成しました。ビデオの再生は約120倍速です。ビデオは、3つの独立した実験の代表的なものです。 これをダウンロードするには、ここをクリックしてください ビデオ.
補足ビデオ 5.1.5厚の光学ガラスカバースリップで6〜7日間成長した大腸菌UTI89の4Dビデオ。4Dタイムラプスビデオは、顕微鏡を使用して512x512の解像度で生成されました。40枚の画像のZシリーズは、バイオフィルムの厚さ約20μmの領域を0.5μmステップでイメージングすることにより生成されました。各Zシリーズは1フレームで、キャプチャには50〜60秒が必要でした。20 個の連続したフレームをキャプチャして、4D ビデオを作成しました。ビデオの再生は約120倍速です。ビデオは、3つの独立した実験の代表的なものです。これをダウンロードするには、ここをクリックしてください ビデオ.
補足ビデオ 6. 4Dビデオ E. coli UTI89 curli (csgBA) 変異体を1.5枚の厚い光学ガラスカバースリップで6〜7日間増殖。 4Dタイムラプスビデオは、顕微鏡を使用して512x512の解像度で生成されました。40枚の画像のZシリーズは、バイオフィルムの厚さ約20μmの領域を0.5μmステップでイメージングすることにより生成されました。各Zシリーズは1フレームで、キャプチャには50〜60秒が必要でした。20 個の連続したフレームをキャプチャして、4D ビデオを作成しました。ビデオの再生は約120倍速です。ビデオは、3つの独立した実験の代表的なものです。これをダウンロードするには、ここをクリックしてください ビデオ.
ディスカッション
重要な手順とトラブルシューティング
この技術の最大の課題は、 E. faecalis のような非常に粘性のあるバイオフィルムを貼ったカバースリップを使用することです。カバースリップは、位置を変えることなく、マルチウェルスライドに慎重かつ正確に配置する必要があります。シーリングステップでは、カバーガラスを押し下げたり、誤ってスライド面を押したり滑らせたりしないように注意する必要があります。どんな動きや圧力でも、表面張力が発生し、粘性のあるバイオフィルムの動きが妨げられる可能性があります。可能であれば、光学ボトムウェル上のバイオフィルムをカバースリップマウントにイメージングすることにより、バイオフィルムの材料特性を比較することで、技術評価が可能になります。正しく機能すると、カバースリップマウントは 、E. faecalisの光学底板のバイオフィルムに非常によく似ていました。
また、マウント型カバースリップを使用する場合は、バイオフィルムの界面を下部のカバースリップや最上部にスライドしてイメージングすることは避けてください。倒立スコープを使用すると、カバーガラスが下部にあると、バイオフィルムの基部にカバーガラスに対するビーズが閉じ込められる可能性があります。これらのビーズはバイオフィルムを通過し、優しく洗ってもカバーガラスに引っ掛かってしまいます。x、y、z 座標は 0 で、バウンディング ボックスの座標は 0 です。ただし、処理後のバイオフィルムの完全性の調査など、特定のアプリケーションでは、これらのデータポイントをツールとして使用できます。ビーズがバイオフィルムの厚い領域を通ってカバーガラスの底まで浸透する能力は、処理後のバイオフィルムの完全性を評価するために使用できます(Tükel研究室と共同で作成中の原稿)。バイオフィルムの上部には、 E. faecalis のような粘性のあるバイオフィルムで、カバースリップによって押し付けられた圧縮の証拠がいくつかありました。これにより、スライドガラス界面での一部のビーズの動きが制限され、ビーズの動きの解析に密度依存性が生じた可能性があります。
バイオフィルムには、成長培地が緑色チャネルで強い自家蛍光を示すため、洗浄ステップが必要でした。余分なビーズを使用し、関連するビーズを洗浄して関連付けられていないビーズを取り除くことを選択し、関連するビーズを最大化して、観察された領域の最も正確な特性評価を取得します。
目的のデータセットを取得するために必要なビーズと洗浄の数は、経験的に決定する必要があります。バイオフィルムにビーズが多すぎると、分析が困難な非常に大きなデータセットが生成されます。ビーズが少なすぎると、バイオフィルム環境の徹底的なサンプリングは得られません。しかし、添加するビーズの数(PBS1 mLに2x107 ビーズ)を制御し、洗浄ステップを使用すると、バイオフィルムの構造、空間配置、および組成に応じて、バイオフィルムと結合するビーズの数(40〜140)が比較的一定になりました。
粘性領域と硬質領域が混在するバイオフィルムを研究する場合、時間の経過とともにビーズが硬質領域に閉じ込められる可能性があります。この場合、ビーズを添加した直後にイメージングを開始する必要があります。これは多くの場合、カバースリップを使用して達成することはできませんが、ビーズの添加と洗浄ステップの直後にイメージングを行うことができる光学式ボトムプレートまたはフローセルが必要です。
変更点と将来のアプリケーション
マイクロ流体デバイスの使用。 私たちの研究では、腸内細菌科のバイオフィルムの研究に最適な条件を確立するには、バイオフィルムを気液界面でのペリクルとして成長させる必要がありました。これにより、研究では光学式底板とマイクロ流体デバイスの使用が制限されました。しかし、バイオフィルム形成条件が許せば、バイオフィルムはマイクロ流体チャンバーまたはフローセルで成長させることができます。その後、バイオフィルムを洗浄し、バイオフィルムの破壊を最小限に抑えながら、マイクロ流体デバイスを介してビーズを導入できます。
バイオフィルムの成長中のビーズの添加。 バイオフィルムに余分なビーズを添加し、その後、分析中に存在するビーズの数を最適化するために、穏やかな洗浄によって関連のないビーズを除去することを選択しました。粘性のある E. faecalis バイオフィルムでは、20分間のイメージング時間中にビーズが解離および再結合する可能性があります。バイオフィルムの成長中に異なる時期に少量のビーズを添加すると、バイオフィルムにビーズをトラップできる可能性があり、より粘性の高いバイオフィルムでのバイオフィルムの動きをより正確に評価できます。
イメージ化する領域の選択。材料特性の研究には、バイオフィルムの厚い領域と薄い領域を選択するのが最善です。ただし、処理されたバイオフィルムの材料特性の変化を研究する場合、厚いコンフルエント領域を画像化して、それらの領域の粘弾性特性とビーズ浸透の変化を決定する場合があります。この場合、バイオフィルムを貫通してカバーガラスに引っかかったビーズを探すことは、バイオフィルムの乱れの有用な尺度です。
フロー下でのイメージング。光学ガラスフローセルやマイクロ流体デバイスを用いて、バイオフィルム内のビーズやバクテリアの動きを流動状態で画像化することができます。これはさまざまな方法で行うことができます。これは、ビーズをチャンバー全体に注入し、その後、ビーズをバイオフィルムと関連付けるための短時間のインキュベーションによって行うことができます。関連付けられていないビーズは、洗浄によって除去でき、バイオフィルムはフローの有無にかかわらずイメージングされます。逆に、少数のビーズをチャンバーの片側に導入し、バイオフィルム内およびバイオフィルム内でのそれらの動きをフロー下で追跡することができます。フローを使用する場合は、モザイクビーズのトラッキング設定を選択する際に注意が必要です(ステップ4.5)。現在の研究では、ダイナミクスの設定はブラウンでした。MSDの計算では、この動きは拡散性である可能性が高いことが確認されており、ブラウニアンが適切な環境となった。
マトリックス染色。現在の研究では、Syto9による染色では、バイオフィルム構造の密度ではなく、細胞密度が調べられています。例えば、アミロイドの存在は、バイオフィルムのマトリックス材料の密度を増加させる可能性があります。アミロイド密度への移動依存性は、Syto9の代わりに蛍光マトリックス染色を使用することで決定できます。
蛍光標識されたバクテリア。 蛍光標識された細菌は、バイオフィルムを介した外因性細菌(プラスミド含有細菌など)の動きを追跡するために使用できます。 腸球菌 などの蛍光標識細菌の課題は、単鎖、双球菌、短鎖を形成するため、細菌を正確に追跡する能力が複雑になることです。このプロセスは、細菌が単一細胞の形態を持っている場合、より簡単になります。
制限
軌道の視覚化とスティッチングの制限。
この方法の1つの制限は、軌道の視覚化とスティッチングです。再構築および解析された軌跡は、x、y、z 点の座標で構成され、後続の点によってこれらの点間の線形パスが定義されます。このような区分線形軌跡の視覚化は、さまざまなツールで実現できます。1 つの方法は、Python と Jupyter ノートブックを Python プラグイン (Pandas、Matplotlib) と共に使用することでした。この手法が最初に発表されたJournal of Bacteriologyの記事34では、個々の既存の軌跡を視覚化することが可能でしたが、将来の研究で対処される重大な制限がまだありました。
現在、再構築された軌跡の数はバイオフィルム内のビーズの数よりも多く、1つのビーズに複数の軌跡が対応している可能性があります。これは、1つのフレームで共焦点信号が弱いため、Mosaicが軌道を終了して2番目の軌道を開始することが原因である可能性があります。これは、特に粘性の低いバイオフィルムでは、1つのビーズに対して複数の短い軌道として登録される場合があります。弾道の数が多いもう一つの原因は、弾道のステッチが不足していることです。特に E. faecalis 光学ボトムウェルバイオフィルムでは、イメージング中にビーズがバイオフィルムと視覚的に関連付けられたままになります(補足ビデオ2)。しかし、10を超える軌道はなく、軌道の90%以上が寿命が5フレーム以下でした(図3D)。ソフトウェアを使用して定義された長さを超える軌跡のみを解析する場合(たとえば、プラスミドを転送できる細胞を追跡する場合)、より短い軌跡をデータセットから自動的に削除できます。ただし、軌道のステッチが非常に重要な目的は他にもあります。最後に、ビーズの急速な動きを単一の軌道として追跡できないと、Z方向に急速に移動して楕円形のビーズが発生するため、腸内細菌科のバイオフィルムの軌道が増える可能性があります(図2G)。急速な異方性運動によって妨害された縫合軌道の可能性は、腸内細菌科のカールアミロイドマトリックスの影響を研究するために重要になります。
意味
3Dバイオフィルムの材料特性を比較するために、ビーズの軌跡を研究するための計算ワークフローが開発されました。このワークフローにより、研究者は、不均一なバイオフィルムの流体力学の計算モデリングに使用できる重要なパラメーターを特定できます。このオープンソースのビーズ評価ツールの助けを借りて、バクテリアのアミロイドカーリが材料特性に及ぼす影響を研究することができ、カーリによるバイオフィルムマトリックスの剛性の増加を示すことができました。より一般的な文脈では、評価器を使用して、バイオフィルム処理または流れなどのさまざまな環境条件によって引き起こされるバイオフィルム構造の変化を研究することができます。例えば、このツールは、テュケル研究所(LKSOMテンプル大学)と共同で、モノクローナル抗体治療がバイオフィルム構造の破壊に及ぼす影響を分析するために使用されています。ビーズエバリュエーターツールボックスは、VRL-Studioを使用してモジュール式に完全に適応および拡張でき、その機能をさらに強化および拡張できます。
開示事項
著者は何も開示していません。
謝辞
GQおよびBABラボでの研究は、公共、商業、または非営利セクターの資金提供機関から特定の助成金を受け取っていません。著者らは、この技術を含む最初の発表において、有益な議論をしてくれたIsaac Klapper博士(テンプル大学数学科)と腸内細菌科の専門知識を提供してくれたÇagla Tükel(テンプル大学微生物学および免疫学部)に感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 96-well plates, No. 1.5 Uncoated Coverslip, 5 mm Glass Diameter | MatTek | P96G1.55F | |
| Fisherbrand Cover Glasses: Circles | Fisher Scientific | 12-293-232P | 1.5 optic glass coverslip |
| Invitrogen Syto 9 Green Fluorescent Nucleic Acid Stain | Invitrogen | S34854 | |
| Molecular Probes FluoSpheres Carboxylate-modified Microspheres, 1 um, crimson fluorescent (625/645) | Molecular Probes | F8816 |
参考文献
- Huang, R., Li, M., Gregory, R. L. Bacterial interactions in dental biofilm. Virulence. 2, 435-444 (2011).
- Nadell, C. D., Drescher, K., Wingreen, N. S., Bassler, B. L. Extracellular matrix structure governs invasion resistance in bacterial biofilms. ISME Journal. 9, 1700-1709 (2015).
- Billings, N., Birjiniuk, A., Samad, T. S., Doyle, P. S., Ribbeck, K. Material properties of biofilms - key methods for understanding permeability and mechanics. Reports on Progress in Physics. 78, 036601 (2015).
- Araújo, G. R. d. e. S., Viana, N. B., Gómez, F., Pontes, B., Frases, S. The mechanical properties of microbial surfaces and biofilms. The Cell Surface. 5, 100028 (2019).
- Tallawi, M., Opitz, M., Lieleg, O. Modulation of the mechanical properties of bacterial biofilms in response to environmental challenges. Biomaterials Science. 5, 887-900 (2017).
- Tursi, S. A., Tükel, &. #. 1. 9. 9. ;. Curli-Containing Enteric Biofilms Inside and Out, Matrix Composition, Immune Recognition, and Disease Implications. Microbiology and Molecular Biology Reviews. 82, (2018).
- Dueholm, M. S., Albertsen, M., Otzen, D., Nielsen, P. H. Curli functional amyloid systems are phylogenetically widespread and display large diversity in operon and protein structure. PLoS One. 7 (12), 51274 (2012).
- Hung, C., et al. Escherichia coli biofilms have an organized and complex extracellular matrix structure. mBio. 4, 00645 (2013).
- Kikuchi, T., Mizunoe, Y., Takade, A., Naito, S., Yoshida, S. Curli fibers are required for development of biofilm architecture in Escherichia coli K-12 and enhance bacterial adherence to human uroepithelial cells. Microbiology and Immunology. 49, 875-884 (2005).
- Gallo, P. M., et al. Amyloid-DNA Composites of Bacterial Biofilms Stimulate Autoimmunity. Immunity. 42, 1171-1184 (2015).
- Serra, D. O., Richter, A. M., Hengge, R. Cellulose as an architectural element in spatially structured Escherichia coli biofilms. Journal of Bacteriology. 195, 5540-5554 (2013).
- Oh, Y. J. Characterization of curli A production on living bacterial surfaces by scanning probe microscopy. Biophysical Journal. 103, 1666-1671 (2012).
- Lembré, P., Di Martino, P., Vendrely, C. Amyloid peptides derived from CsgA and FapC modify the viscoelastic properties of biofilm model matrices. Biofouling. 30, 415-426 (2014).
- Oh, Y. J., et al. Curli mediate bacterial adhesion to fibronectin via tensile multiple bonds. Scientific Reports. 6, 33909 (2016).
- Vidakovic, L., Singh, P. K., Hartmann, R., Nadell, C. D., Drescher, K. Dynamic biofilm architecture confers individual and collective mechanisms of viral protection. Nature Microbiology. 3, 26-31 (2018).
- Tursi, S. A., et al. Salmonella Typhimurium biofilm disruption by a human antibody that binds a pan-amyloid epitope on curli. Nature Communications. 11, 1007 (2020).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15 (8), 1007978 (2019).
- Taglialegna, A., et al. The biofilm-associated surface protein Esp of Enterococcus faecalis forms amyloid-like fibers. Npj Biofilms and Microbiomes. 6, 15 (2020).
- Gour, S., Kumar, V., Rana, M., Yadav, J. K. Pheromone peptide cOB1 from native Enterococcus faecalis forms amyloid-like structures, A new paradigm for peptide pheromones. Journal of Peptide Science. 25, 3178 (2019).
- Stoodley, P., Debeer, D., Lewandowski, Z. Liquid flow in biofilm systems. Applied and Environmental Microbiology. 60, 2711-2716 (1994).
- Birjiniuk, A., et al. Single particle tracking reveals spatial and dynamic organization of the E. coli biofilm matrix. New Journal of Physics. 16, 085014 (2014).
- Chew, S. C., et al. Dynamic remodeling of microbial biofilms by functionally distinct exopolysaccharides. mBio. 5, 01536 (2014).
- Cao, H., et al. Revealing region-specific biofilm viscoelastic properties by means of a micro-rheological approach. Npj Biofilms and Microbiomes. 2, 5 (2016).
- Galy, O., et al. Mapping of bacterial biofilm local mechanics by magnetic microparticle actuation. Biophysical Journal. 103, 1400-1408 (2012).
- Rogers, S. S., vander Walle, C., Waigh, T. A. Microrheology of bacterial biofilms in vitro, Staphylococcus aureus and Pseudomonas aeruginosa. Langmuir. 24, 13549-13555 (2008).
- Hart, J. W., Waigh, T. A., Lu, J. R., Roberts, I. S. Microrheology and Spatial Heterogeneity of Staphylococcus aureus Biofilms Modulated by Hydrodynamic Shear and Biofilm-Degrading Enzymes. Langmuir. 35 (9), 3553-3561 (2019).
- van Merode, A. E. J., van der Mei, H. C., Busscher, H. J., Krom, B. P. Influence of culture heterogeneity in cell surface charge on adhesion and biofilm formation by Enterococcus faecalis. Journal of Bacteriology. 188, 2421-2426 (2006).
- Tariq, M., Bruijs, C., Kok, J., Krom, B. P. Link between Culture Zeta Potential Homogeneity and Ebp in Enterococcus faecalis. Applied and Environmental Microbiology. 78, 2282-2288 (2012).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ, 25 years of image analysis. Nature Methods. 9, 671-675 (2012).
- . Oracle.com Available from: https://docs.oracle.com/en/java (2021)
- Hoffer, M., Poliwoda, C., Wittum, G. Visual reflection library, a framework for declarative GUI programming on the Java platform. Computing and Visualization in Science. 16, 181-192 (2013).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15, 1007978 (2019).
- Malhotra, K., et al. Development of a New Bead Movement-Based Computational Framework Shows that Bacterial Amyloid Curli Reduces Bead Mobility in Biofilms. Journal of Bacteriology. 202, 00253 (2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved