Method Article
Un framework computazionale basato sul movimento delle perle per l'analisi tridimensionale dell'eterogeneità dei materiali del biofilm
In questo articolo
Riepilogo
Descriviamo un metodo per analizzare e quantificare il modello di movimento di perle di carbossilato da 1 μm attraverso biofilm batterici eterogenei. Il confronto dei modelli di movimento può essere utilizzato per quantificare le differenze nelle proprietà dei materiali dei biofilm.
Abstract
Differenze nelle proprietà dei materiali dei biofilm batterici sono state osservate in biofilm di diverse specie batteriche, all'interno della stessa specie in diverse condizioni di crescita e dopo il trattamento con molecole modificanti la matrice. Per quantificare meglio le proprietà dei materiali dei biofilm 3D, è stato sviluppato e applicato un flusso di lavoro sperimentale e computazionale per esaminare le differenze tra i biofilm di Enterococcus faecalis, Salmonella enterica sierotipo Typhimurium ed Escherichia coli , nonché il ruolo del ricciolo amiloide nel confermare la rigidità dei biofilm di Enterobacteriaceae. La dinamica spazio-temporale delle perle di carbossilato da 1 μm nei biofilm è stata monitorata in biofilm 3D da 20 μm per 20 minuti. Le pile di immagini 4D sono state elaborate utilizzando il plug-in Mosaic in ImageJ per produrre dati di traiettoria 3D del movimento del tallone. Questi dati di traiettoria sono stati analizzati con un toolbox di nuova concezione Bead Evaluator, in cui i dati di movimento, tra cui la durata della traiettoria, la velocità del tallone, la densità delle celle lungo le traiettorie e le informazioni sul riquadro di delimitazione sono stati calcolati e memorizzati in file csv. Questo articolo presenta il flusso di lavoro dalla configurazione sperimentale e dalla registrazione delle immagini al calcolo e all'analisi della traiettoria del cordone. La struttura dei biofilm contenenti curli ha portato a interazioni più stabili con le perle e a un minore movimento delle perle rispetto ai biofilm curli-mutanti ed enterococcici. Il movimento del cordone non sembra dipendere fortemente dalla densità cellulare quando si misura la velocità del cordone e il volume del riquadro di delimitazione della traiettoria, supportando l'ipotesi che altre proprietà del materiale dei biofilm controllino la dinamica del microschino. Questa tecnica è ampiamente applicabile per quantificare le differenze nei biofilm di diverse composizioni di matrici, nonché nei biofilm prima e dopo i trattamenti di modifica della matrice.
Introduzione
I biofilm batterici sono onnipresenti come parte del microbiota umano e interagiscono continuamente con le molecole. Queste molecole variano in dimensioni da antibiotici da 1 nm e batteri da 1-3 mm a particelle più grandi di fibra nel tratto gastrointestinale. La composizione di biofilm monospecie o multispecie influenza le proprietà del materiale e quindi il modello di movimento delle particelle attraverso i biofilm 1,2,3,4,5. Un esempio sono gli amiloidi batterici, che hanno una struttura conservata del foglio cross-beta fibrillare6. Il curli amiloide è espresso da batteri enterici come Escherichia coli e Salmonella enterica sierotipo Typhimurium e geni sono stati rilevati in molti altri phylum batterici7. Varie proprietà dei materiali dei biofilm sono influenzate da curli 8,9. Curli interagisce direttamente con altri componenti della matrice come il DNA extracellulare (eDNA) e la cellulosa10,11. Il curli circonda le cellule e influisce sulla rigidità della membrana cellulare12 e sulle proprietà viscoelastiche complessive del biofilm13. Curli media l'aumento della resistenza alla trazione legandosi alla fibronectina, con conseguente aumento del forte attaccamento alla superficie del vetro14. I batteriofagi in arrivo si legano ai curli e limitano l'invasione dei fagi nei biofilm15.
Quando si utilizzano vetrini rivestiti multitest per analizzare Enterococcus faecalis, E. coli e S di circa 20 μm di spessore. Biofilm di Typhimurium utilizzando la microscopia confocale, chiare differenze tra E. coli, S. Potrebbero essere osservati biofilm di Typhimurium10,16 ed E. faecalis (studio attuale). Mentre i biofilm delle specie di Enterobacteriaceae avevano un alto livello di rigidità e le aree con bassa densità cellulare erano facili da visualizzare, ottenere immagini chiare ad alta risoluzione dei biofilm di E. faecalis utilizzando la media delle linee e dei fotogrammi richiedeva l'applicazione di una pressione sul vetrino per indurre una tensione superficiale sufficiente per la stabilità cellulare durante il processo di imaging. Gli amiloidi batterici come i curli formano strutture altamente ordinate, suggerendo che potrebbero essere relativamente rigide17. Ciò ha motivato l'ipotesi che il ricciolo amiloide potesse indurre rigidità in E. coli e S. Biofilm di Typhimurium. Non c'erano prove chiare che E. faecalis esprimesse amiloidi nelle condizioni studiate. La proteina Esp, un gene della pilina associato a ceppi più patogeni di E. faecalis, ha recentemente dimostrato di produrre strutture amiloidi18; tuttavia, utilizzando ricerche blastn e blastp, questo gene non è stato rilevato nel ceppo di tipo commensale di E. faecalis OG1RF utilizzato in questi studi. Il feromone cOB1, prodotto da OG1RF, può formare strutture simili all'amiloide19. Tuttavia, con le condizioni di crescita del biofilm date e i metodi di rilevamento dell'amiloide precedentemente utilizzati per S. Colorazione dell'amiloide Typhimurium10 in E. faecalis, non è stato possibile rilevare gli amiloidi OG1RF (dati non mostrati). È stata sviluppata una nuova tecnica di immagine quadridimensionale (4D) per confrontare le proprietà complessive del materiale tra i viscosi E. faecalis, E. coli e S. Typhimurium, nonché per determinare il contributo dell'amiloide ai biofilm delle Enterobacteriaceae utilizzando mutanti amiloidi di S. Typhimurium ed E. coli.
In passato, le perle fluorescenti sono state utilizzate con successo per analizzare le proprietà dei materiali dei biofilm in due dimensioni (2D) utilizzando la microreologia 20,21,22,23,24,25. Questo può essere applicato a un biofilm tridimensionale studiando fette ottiche 2D a varie profondità nel biofilm26. L'attuale tecnica è stata sviluppata per tracciare nel tempo microsfere su microscala da 1 μm in 3D per l'uso nella modellazione 4D. Parte della logica era il concetto generale di utilizzare la modellazione 4D per comprendere il movimento dei plasmidi attraverso le comunità del microbiota gastrointestinale. Sono state utilizzate perle di carbossilato caricate fluorescentmente con un diametro di 1 μm poiché queste corrispondono bene, per dimensioni e carica, a E. faecalis, l'organismo modello scelto per il movimento e il mantenimento dei plasmidi27, 28. È stato sviluppato un saggio 4D per quantificare le proprietà fisiche dei biofilm (Figura 1A). Nella metodologia ideata, le perle sono state aggiunte ai biofilm e le loro traiettorie spazio-temporali sono state registrate attraverso biofilm spessi 10-20 μm nel corso di 10-20 minuti. Le traiettorie dei microanelli in 3D sono state quindi quantificate in termini di lunghezza della traiettoria, velocità del cordone, volume del riquadro di delimitazione della traiettoria (riquadro minimo contenente la traiettoria) e densità cellulare del riquadro di delimitazione utilizzando un toolbox di nuova concezione. Il seguente protocollo può essere utilizzato per generare dati di immagini 4D di batteri e biofilm contenenti perline, per pre-elaborare i dati con ImageJ29 e il plug-in Mosaic e per analizzare le traiettorie delle perline con un toolbox di valutazione delle perline.
Questa tecnica ha molteplici applicazioni per l'esame delle proprietà dei materiali e per il monitoraggio del movimento di particelle e batteri in tre dimensioni. Ad esempio, una prima versione di questa tecnica è stata utilizzata per caratterizzare l'effetto di anticorpi monoclonali diretti contro il curli sull'integrità strutturale dei biofilm16. La versione completa dispone di diversi strumenti per fornire un'analisi più dettagliata delle proprietà del materiale del biofilm e continua ad essere utilizzata per esaminare gli effetti del trattamento con anticorpi monoclonali sui biofilm. Particelle di diverse cariche possono essere utilizzate per esaminare le proprietà di carica del materiale dei biofilm e il movimento delle particelle attraverso biofilm con diverse composizioni di matrici. Questo potrebbe essere utilizzato per confrontare i risultati della microreologia 2D che rivelano le proprietà del materiale responsabili del movimento delle perle che abbiamo osservato nei biofilm che non erano sotto flusso. Questa tecnica potrebbe essere utilizzata anche su biofilm di specie miste con regioni di diversa composizione del biofilm. I biofilm possono essere riprodotti in tempo reale sotto flusso in dispositivi microfluidici e celle a flusso per esaminare i cambiamenti nelle proprietà del materiale tra biofilm statici e a flusso, nonché l'effetto del flusso sul movimento delle particelle. Le tecniche possono anche essere applicate a batteri marcati in fluorescenza per caratterizzare il movimento di batteri esogeni attraverso una comunità di biofilm. Utilizzando tre colori, i batteri donatori marcati in fluorescenza, i batteri riceventi marcati in fluorescenza e i plasmidi marcati in fluorescenza potrebbero essere utilizzati per tracciare il movimento, l'attracco e il trasferimento dei plasmidi.
Protocollo
1. Preparazione del biofilm
NOTA: I biofilm per l'analisi possono essere coltivati utilizzando qualsiasi metodo che consenta la formazione del biofilm su una superficie di vetro ottico. La struttura del biofilm deve aderire alla superficie ottica in modo che la struttura non venga interrotta durante le fasi di lavaggio e/o montaggio del protocollo. Di seguito viene descritta la tecnica per piastre inferiori ottiche a 96 pozzetti e vetrini coprioggetti da 12 mm in piastre a 24 pozzetti. Altre opzioni includono piastre di fondo ottiche di diverse dimensioni e camere di flusso ottico con e senza flusso.
- Impostazione del biofilm
- Aggiungere il terreno di coltura batterica ai pozzetti della piastra. Per questo studio, per E. aggiungere 2 mL di Todd-Hewitt (TH) a una piastra a 24 pozzetti e 0,4 mL di TH a piastre a 96 pozzetti a fondo ottico. Per S. Typhimurium, E. coli e i mutanti isogenici csgAB curli, aggiungono 0,7 ml di brodo Luria (LB) senza sale ai pozzetti. Se si utilizzano camere ottiche inferiori, procedere al passaggio 1.2.
- Mettere i vetrini coprioggetti ottici in vetro #1.5 da 12 mm in una capsula di Petri e coprirli con etanolo.
- Usando una pinza, rimuovere il vetrino coprioggetti e usare una fiamma per dare fuoco all'alcol rimasto. Lascia che l'alcol bruci. Basta usare la fiamma per dar loro fuoco; Non tenere il vetrino coprioggetti nella fiamma perché si spezzerà. Lasciare raffreddare il vetrino coprioggetti per 10-20 secondi prima di metterlo nel pozzetto per evitare screpolature.
- Posizionare il vetrino coprioggetti nel pozzetto contenente il substrato inclinandolo per evitare che si adagi sopra il substrato. Non aggiungere il vetrino coprioggetti in un pozzetto asciutto e poi aggiungere del terreno perché ciò farà sì che il vetrino coprioggetti si attacchi al fondo del pozzetto.
- Utilizzare una punta di pipetta sterile per spingere con cautela il vetrino coprioggetti sul fondo del pozzetto contenente il terreno.
NOTA: Ricordarsi di far corrispondere lo spessore (#1 o #1,5) del vetrino coprioggetto in vetro ottico o della piastra inferiore ottica allo spessore dell'ottica del microscopio confocale.
- Incubare il biofilm in condizioni adeguate per la crescita del biofilm. In questi studi, coltivare i biofilm di E. faecalis come colture statiche a 37 °C e far crescere i biofilm di E. coli in modo aerobico a 30 °C.
- In questi studi, coltivare biofilm di E. faecalis per 2 giorni con interruttori medi al mattino e alla sera presto. Per evitare di danneggiare o spostare il biofilm, inclinare accuratamente la piastra. Posizionare la punta della pipetta vicino al bordo inferiore del pozzetto ed estrarre lentamente il terreno. Aggiungere il primo ml di terreno fresco allo stesso modo. Aggiungere lentamente il secondo mL vicino all'interfaccia mezzo/pozzetto.
- In questi studi, utilizzare le condizioni di crescita per una produzione ottimale di ricci. Crescere S. I biofilm di Typhimurium a 28 °C per 6-8 giorni sono stati incubati su un'inclinazione che ha permesso al biofilm di attaccarsi a circa 2/3 del percorso lungo il vetrino e quindi crescere come una pellicola sull'interfaccia aria-liquido. Questo è stato fatto senza un cambiamento medio. Per evitare che il fluido si secchi, posizionare la piastra a 24 pozzetti in una camera con una vaschetta per l'acqua.
2. Imaging 4D
- Preparazione del supporto per biofilm
- Diluire le perle Crimson 1 μm carbossilato FluoSpheres 1:50 in PBS (2 x 107 perle in 1 mL di PBS). Se si utilizzano camere ottiche a fondo, aggiungere Syto9 a una diluizione di 1 μl a 300 μl di preparazione del microsfone.
- (Facoltativo) Lavare il biofilm per rimuovere le tracce di terreno di crescita se il terreno di coltura è autofluorescente. In questi esperimenti, lavare due volte con 1 mL di PBS, inclinando accuratamente la piastra, posizionando la punta della pipetta vicino al bordo inferiore del pozzetto ed estraendo lentamente il terreno. Aggiungere PBS posizionando la punta vicino al bordo inferiore e riempire lentamente il pozzetto. Utilizzare questa tecnica su pozzetti sul fondo ottico oltre ai vetrini coprioggetti in una piastra a 24 pozzetti.
- Rimuovere il mezzo o il PBS. Aggiungere al biofilm le perle Crimson diluite preparate in 2.1.1. In questi studi, aggiungere 1 mL di perle (2x107) ai vetrini coprioggetti e 0,2 mL di perline (4x106) e Syto9 a piastre di fondo ottico a 96 pozzetti.
- Incubare per 1 minuto a temperatura ambiente per consentire l'associazione delle microsfere.
- Rimuovere le perline e lavare delicatamente il biofilm una volta con PBS per rimuovere le perle non associate. In questi studi, per il vetrino coprioggetti, lavare delicatamente con 1 mL di PBS, riempire nuovamente il pozzetto con 1 mL di PBS e procedere al punto 2.1.6. Per le piastre ottiche inferiori a 96 pozzetti, lavare delicatamente il biofilm con 0,2 mL di PBS e riempire nuovamente il pozzetto con 0,2 mL di PBS. La camera inferiore ottica è ora pronta per l'acquisizione, quindi procedere al passaggio 2.2.
- (Se si utilizza un vetrino coprioggetti) Aggiungere 1 μL di Syto9 (colorazione di DNA fluorescente verde; diluito secondo le istruzioni del produttore) al centro di un pozzetto su un vetrino multitest rivestito a 10 pozzetti. Questi vetrini coprioggetti rivestiti hanno profondità di 23-25 μm.
- (Se si utilizza un vetrino coprioggetti) Utilizzando una pinza sterilizzata a fiamma ad alcool, rimuovere con cautela il vetrino coprioggetti dal pozzetto e capovolgerlo sul pozzetto contenente la goccia Syto9. Lasciare il vetrino coprioggetti in 1 mL di PBS facilita la rimozione dal pozzetto e aiuta a lavare le perle non associate dal biofilm.
- (Se si utilizza un vetrino coprioggetti) Sigillare accuratamente il vetrino coprioggetti con lo smalto per unghie senza scivolare o premere sul vetrino, che può causare una tensione superficiale che interromperà il movimento in biofilm più viscosi e meno rigidi.
- (Se si utilizza un vetrino coprioggetti) Lascia asciugare lo smalto. Pulire accuratamente la superficie esterna del vetrino coprioggetti con etanolo al 70%. Pulire senza esercitare alcuna pressione sul vetrino coprioggetti per i motivi sopra indicati.
- Imaging confocale
NOTA: In questi studi, utilizzare un microscopio confocale a scansione laser a scansione spettrale invertita dotato di un sistema confocale TCS con l'obiettivo 63x. L'oscilloscopio verrà utilizzato per generare un video 4D (Figura 1). Il biofilm 3D sarà composto da fette Z catturate in passi di 0,5 μm attraverso biofilm spessi 18-20 μm, generando fette 36-40 Z. Ogni biofilm 3D impiegherà 50-60 secondi per essere catturato. Le fette insieme comporranno un fotogramma, che può essere visualizzato come un biofilm 3D. Questo processo verrà ripetuto 20 volte per generare il video time lapse 4D per un tempo di tracciamento totale di 18-20 minuti.- Impostare il cannocchiale per catturare i fluorofori. In questi studi, eccitare Syto9 (colorazione del DNA batterico) con un laser a 488 nm e misurare l'emissione da 495 a 540 nm (il Leica Sp5 è un microscopio per immagini spettrali). Eccita le perle cremisi (rosse) con un laser a 633 nm e misura l'emissione da 650 nm a 700 nm. Queste impostazioni del fluoroforo possono essere regolate per catturare i fluorofori desiderati.
- Scegli la modalità di imaging xyzt.
- Identificare una regione del biofilm con una miscela di regioni a densità più alta e più bassa per catturare le differenze nelle proprietà viscoelastiche nelle regioni spesse e sottili del biofilm.
- Impostare una pila Z di 18-20 μm di spessore. In un biofilm montato su vetrino coprioggetti, evitare che la parte superiore e inferiore del biofilm tocchino contro il vetro per evitare artefatti da intrappolamento delle perle (vedere la discussione).
- Regola il guadagno e l'offset per utilizzare l'intera gamma dinamica dell'intensità nel punto più luminoso del biofilm. Ciò riduce al minimo la sovrapposizione del segnale dagli strati inferiori del biofilm.
- Impostare lo spessore della fetta su 0,5 μm. Ciò consente un imaging rapido senza la perdita di informazioni sul cordone.
- Impostare la risoluzione su 512 x 512 (0,48 μm). Ciò consente un imaging rapido, ma genera immagini con una risoluzione sufficiente per vedere la struttura del biofilm e i dettagli del movimento delle perline.
- Riduci al minimo il tempo di imaging.
- Impostato per catturare 20 pile.
- Salva come .lif (o file confocale simile). Il filmato 4D può essere generato in ImageJ.
3. Generazione del video del biofilm 4D con ImageJ
- Aprire il file .lif in ImageJ con le seguenti impostazioni: Visualizza stack con Hyperstack, Ordine stack XYCZT, modalità colore: colorato, scala automatica selezionata. Quindi premere OK.
- Seleziona Immagine > Colore > Dividi canali.
- Selezionate Immagine > Colore > Unisci canali > Crea > composito OK.
- Seleziona Plugin > visualizzatore 3D > Canali deseleziona blu > OK.
- Seleziona Modifica > Mostra contenuto selezionato > Mostra riquadro di delimitazione selezionato.
- Utilizzare il clic del mouse e tenere premuto per selezionare l'immagine. Quindi ruotare l'immagine continuando a tenere premuto il pulsante di clic. Ruota l'immagine in modo che la parte inferiore del biofilm sia sul fondo e l'angolo supporti la visualizzazione delle perline. Quindi rilasciare l'immagine.
- Premi il pulsante rosso Registra nella parte inferiore della finestra per registrare un video.
- Salva come .avi-file utilizzando jpeg come compressione.
4. Generazione dei dati di traiettoria
- Installa lo strumento open source ImageJ (https://imagej.net/Fiji) e il plug-in di tracciamento delle particelle Mosaic (https://imagej.net/MOSAICsuite).
- Importare il file .lif contenente i due canali per perline e batteri. Dividi i canali e salva i file separatamente.
- In ImageJ, memorizza le dimensioni del voxel dell'immagine (dimensioni x, y e z) e le dimensioni del passo temporale in un file di testo.
- In ImageJ, vai a Plugin | Mosaic e avvia il Particle Tracker 2D/3D.
- Inserire i seguenti parametri: Raggio: 3, Taglio: 0,003, Per/Abs: 0,12, Intervallo di collegamento: 2, Cilindrata: 10,00 con la dinamica impostata su Brownian. Si prega di vedere i risultati per il razionale dietro la scelta del browniano in questi esperimenti.
- Generate le traiettorie ed esportate l'elenco delle traiettorie come file .csv.
5. Analisi delle traiettorie
- Installazione di VRL Studio (https://vrl-studio.mihosoft.eu)
- Scarica il progetto Biofilm (https:// neurobox3d.github.io/Biofilm/) e lancialo in VRL Studio
- Caricare il file della traiettoria in ImportData
- Specificate la dimensione dei pixel x, y e z (utilizzate ImageJ per individuare questi valori) in ProcessTrajectories.
- Specificare l'intervallo di fotogrammi in ComputeVelocity (utilizzare ImageJ per individuare questo valore).
- Caricare il file tiff batterico (vedere il passaggio 3.2) in Comdensity.
- Impostare i percorsi di output per i dati di velocità in SaveVelocityDataToFile e i dati di traiettoria in SaveTrajectoryDataToFile.
- Richiamare SaveVelocityDataToFile e SaveTrajectoryDataToFile.
- Importa i dati in Excel per l'analisi. Questi dati includeranno le lunghezze delle traiettorie, la durata delle traiettorie, le dimensioni e i volumi del riquadro di delimitazione delle traiettorie, le velocità medie dei cordoni e le varianze. L'analisi calcola le variabili ponderate utilizzando il canale per i batteri marcati con Syto9 per calcolare le densità cellulari locali (entro determinati riquadri di delimitazione della traiettoria). L'analisi consente di calcolare le velocità e le varianze medie ponderate, nonché le medie ponderate e le varianze della varianza del riquadro di delimitazione.
Risultati
Questo metodo è stato utilizzato per testare l'ipotesi che i ricci possano conferire rigidità a E. coli e S. Biofilm di Typhimurium, che riducono il movimento delle perle durante gli esperimenti di microscopia confocale. L'attuale toolbox è stato utilizzato per confrontare le proprietà del materiale del ceppo di tipo commensale OG1RF di Enterococcus faecalis con il sierotipo di Salmonella enterica Typhimurium, E. coli e i rispettivi mutanti curli isogenici (Figura 1B e Video supplementare 1, Video supplementare 2, Video supplementare 3, Video supplementare 4, Video supplementare 5, Video supplementare 6). Le proprietà del materiale del biofilm potrebbero potenzialmente differire per quanto riguarda la rigidità (ad esempio, i riccioli legati all'eDNA) o le interazioni elettrostatiche e idrofobiche tra le perle caricate negativamente e le celle del biofilm e i materiali della matrice, nonché la densità cellulare.
Riproducibilità
Il toolbox Biofilm è stato programmato in Groovy30 e Java31 all'interno di VRL-Studio32 , consentendo una progettazione modulare del flusso di lavoro con generazione automatica dell'interfaccia utente (UI) di tutti i componenti computazionali. Ciò ha consentito un flusso di lavoro automatizzato, eliminando i pregiudizi indotti dallo sperimentatore durante l'analisi dei risultati.
Uso di MSD per confermare il tipo di movimento nei biofilm
Per l'analisi delle traiettorie utilizzando Particle Tracker 2D/3D, sono disponibili diverse impostazioni dinamiche per l'analisi di diversi tipi di movimento del cordone. Per questi studi, è stata scelta l'impostazione "Brownian motion" (cioè movimento guidato dalla diffusione) poiché E. faecalis è un batterio non mobile, E. coli e Salmonella non esprimono flagelli nei biofilm e gli esperimenti sono stati eseguiti in un sistema chiuso in assenza di flusso. Questa impostazione potrebbe essere ulteriormente convalidata dagli spostamenti quadratici medi calcolati (MSD) delle perline. Utilizzando la definizione 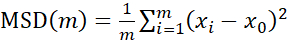 dove m è il numero di segmenti di traiettoria, è possibile calcolare la variazione della MSD nel corso di ciascuna traiettoria. Le traiettorie lineari indicano il movimento diffusivo del cordone (Figura 2A). Utilizzando l'adattamento quadratico dei minimi quadrati, è stato calcolato il modello di movimento medio di tutte le perle nel biofilm, mostrando l'ordine lineare dominante e convalidando la diffusione passiva come forza motrice (Figura 2A-2F).
dove m è il numero di segmenti di traiettoria, è possibile calcolare la variazione della MSD nel corso di ciascuna traiettoria. Le traiettorie lineari indicano il movimento diffusivo del cordone (Figura 2A). Utilizzando l'adattamento quadratico dei minimi quadrati, è stato calcolato il modello di movimento medio di tutte le perle nel biofilm, mostrando l'ordine lineare dominante e convalidando la diffusione passiva come forza motrice (Figura 2A-2F).
Analisi del riquadro di delimitazione.
Il toolbox utilizza ImageJ Mosaic e Particle Tracker 2D/3D per generare traiettorie (Passaggio 4) e quindi, utilizzando la pipeline di analisi automatizzata del biofilm, genera dati importanti sulle traiettorie delle perline che possono essere utilizzate per confrontare le proprietà del materiale del biofilm. Il volume del riquadro di delimitazione in μm3 è stato misurato costruendo il riquadro minimo che contiene una traiettoria e misurandone il volume (Figura 3).
I biofilm di E. faecalis hanno un maggiore movimento delle perle con valori di bounding box di 1-6000 μm3 (Figura 3B, 3C e 3D). I risultati confermano che il movimento osservato in un vetrino coprioggetti montato su un vetrino rivestito con un pozzetto rivestito di circa 25 μm (Figura 3C) rispetto ai biofilm cresciuti sul fondo dei pozzetti in vetro ottico e ripresi direttamente (Figura 3D) produce risultati equivalenti con poche differenze. L'unica differenza era che vicino alla parte superiore del vetrino coprioggetti montato di E. faecalis biofilms si potevano registrare traiettorie stabili con durata di vita superiore a 10 minuti, ma allo stesso tempo piccoli riquadri di delimitazione, mentre nella piastra inferiore ottica poteva essere registrato un numero selezionato di perline con maggiore mobilità. Nel complesso, ciò suggerisce che il montaggio del vetrino potrebbe aver modificato la tensione superficiale del sistema nella parte superiore del biofilm rispetto al vetrino nel biofilm del vetrino montato, che alla fine ha ridotto la mobilità di alcune perle nelle regioni del biofilm meno viscose (Figura 3B, 3C, 3D e 3I). Le traiettorie che rientrano in questa categoria erano una percentuale molto piccola e, anche con questo piccolo numero di perle intrappolate, la MSD media di E. faecalis su un vetrino coprioggetti montato era leggermente superiore alla MSD calcolata da un biofilm ottico su piastra inferiore (Figura 3).
S. Le traiettorie delle perline di Typhimurium ed E. coli avevano volumi di bounding box più piccoli di 0-10 μm3 (Figura 3A, 3B, 3E e 3F), rispetto ai mutanti curli isogenici con bounding box di 1-6000 μm3 per E. coli e 1-5000 μm3 per S. Typhimurium (Figura 3A, 3B, 3F e 3H), che dimostra una maggiore mobilità del tallone. Questi risultati hanno suggerito che la presenza dell'amiloide era correlata con una maggiore rigidità nei biofilm ed era coerente con la mancanza di un notevole movimento del biofilm nei video. I volumi del riquadro di delimitazione erano costantemente piccoli (0-10 μm3) anche nelle regioni a bassa densità del biofilm. Questa osservazione è coerente con le precedenti osservazioni secondo cui i curli possono essere presenti nelle regioni a bassa densità cellulare del biofilm10.
Non è stato possibile confrontare il comportamento dei biofilm delle Enterobacteriaceae sulle piastre di fondo ottiche perché crescono come pellicole sull'interfaccia aria-liquido (Passaggio 1.2.2). Quando si utilizza un vetrino coprioggetti, la pellicola si attacca al vetrino all'interfaccia e quando il vetrino coprioggetti viene rimosso, la pellicola viene adagiata sul vetrino coprioggetti creando un'unica superficie dell'immagine. In una piastra di fondo ottica cresciuta in inclinazione, l'imaging è stato eseguito con il liquido ancora nel pozzetto. Ciò significa che la pellicola sta ancora fluttuando sopra il fondo dell'ottica e fa uscire la pellicola dalla profondità di lavoro di un cannocchiale invertito come il Leica Sp5. La rimozione di una quantità sufficiente di terreno per portare il biofilm nella profondità di lavoro del microscopio ha causato l'essiccazione del campione nel corso del processo di imaging di 20 minuti.
Nel complesso, i grafici confermano le osservazioni visive nei filmati supplementari e sono coerenti con le differenze MSD osservate (Figura 3I e 3J).
Durata della traiettoria
La durata della traiettoria è stata misurata come il numero di fotogrammi consecutivi in cui è stato registrato un cordone (Figura 3).
Nei biofilm più viscosi, simili a fluidi, di E. faecalis , tutte le perle avevano una durata di vita della traiettoria inferiore a 10 minuti e la maggior parte delle traiettorie variava tra 2 e 5 minuti per i biofilm di E. faecalis . Tuttavia, le perle con una breve durata di vita della traiettoria registrata potrebbero essere localizzate mediante ispezione visiva nei biofilm di E. faecalis durante la finestra temporale totale dell'imaging (Video supplementari 1 e 2). Pertanto, è possibile che le perle si muovano lungo una traiettoria registrata, dissociandosi in modo intermittente dal biofilm e terminando una traiettoria, e riassociandosi con il biofilm, a quel punto viene avviata una nuova traiettoria. Ciò porterebbe in ultima analisi a una breve durata della traiettoria in presenza continua di perle nel biofilm. È importante notare che utilizzando questa tecnica, la durata della traiettoria, specialmente in un biofilm viscoso, tende a sottostimare il tempo totale in cui una perlina è associata al biofilm.
Nel S. I biofilm di Typhimurium, che avevano volumi di bounding box più piccoli, la maggior parte delle perle (circa l'80%) aveva una lunga durata della traiettoria di 16-20 fotogrammi, corrispondente a circa 15-20 minuti in tempo reale (Figura 3A, 3G e 3H). Al contrario di questi, i biofilm isogenici curli mutanti trasportano perle più mobili con volumi di bounding box compresi tra 1-6000 μm3 (E. coli) e 1-5000 μm3 (S. Typhimurium) (Figura 3A, 3B, 3F e 3H). A differenza dei biofilm di E. faecalis con traiettorie del >70% con volumi di bounding box superiori a 10 μm3, tuttavia, i biofilm delle specie di Enterobacteriaceae hanno registrato solo il 30% di traiettorie di bead con volumi di bounding box superiori a 10 μm3. Anche se la durata complessiva della traiettoria delle perle era inferiore nei biofilm curli mutanti, alcune traiettorie riflettevano un movimento sostanziale delle perle e una lunga durata della traiettoria (Figura 3H). Questa osservazione potrebbe indicare che questa variabilità può corrispondere a diverse proprietà del materiale del biofilm, come la viscoelasticità, e/o a cambiamenti chimici della superficie delle particelle come la carica.
Analisi delle lunghezze e delle velocità delle traiettorie dei cordoni
La lunghezza della traiettoria è una misura della distanza percorsa dalle perline in μm. Questa misura è coerente con la velocità di movimento del cordone in μm/s. Coerentemente con i volumi più grandi del bounding box, le perle nei biofilm di E. faecalis avevano traiettorie 10 volte più lunghe, 5-20 μm, rispetto a <4 μm nei biofilm contenenti curli. Coerente con le traiettorie più brevi (Figura 4A). Le perle di E. faecalis hanno misurato velocità fino a 15 volte superiori con una maggior parte delle perle con velocità comprese tra 0,01 e 0,15 μm/s rispetto a velocità <0,006 μm/s (Figura 4B). Ciononostante, i biofilm curli mutanti hanno misurato complessivamente velocità più basse e traiettorie più brevi rispetto ai biofilm di E. faecalis , ma traiettorie più lunghe e velocità più elevate rispetto ai ceppi parentali contenenti curli (Figura 4A e 4B).
Degno di nota è il fatto che la struttura reticolare fibrillare dei riccioli30 può influenzare la mobilità in modo anisotropo, riducendo il movimento nel piano xy e consentendo una maggiore mobilità nella direzione z (Figura 2G). L'ampio pool di traiettorie (circa 800) appartenente a circa 50 perle uniche in biofilm contenenti curli sarebbe coerente con i limiti del Mosaic Particle Tracking, contando ciascuna di queste perle in rapido movimento come una singola perlina in x, y e z. Saranno necessarie ulteriori ricerche e lo sviluppo di software per confermare questa osservazione.
Analisi della dipendenza del movimento delle perle dalla densità cellulare
La dipendenza del movimento delle perle dalla densità cellulare è stata determinata utilizzando le velocità e le varianze medie ponderate, nonché le medie/medie ponderate e le varianze dei volumi dei box di delimitazione. Il secondo canale di imaging per i batteri marcati con Syto9 è stato utilizzato per calcolare le densità cellulari locali nel calcolo delle velocità ponderate. La densità cellulare è stata calcolata calcolando la media dei dati del voxel Syto9 sul riquadro di delimitazione di ciascun bordo della traiettoria (Figura 5, a destra). Pertanto, la velocità del cordone può essere ponderata in base alle densità delle celle (locali) lungo il bordo. Esistono diversi tipi di colorazioni che potrebbero essere utilizzate per visualizzare i batteri, comprese le colorazioni per la parete cellulare, le membrane e il contenuto di DNA. Per determinare la densità cellulare, Syto9 è stato scelto perché fornisce il segnale più consistente, indipendentemente dalla sezione Z ottica visualizzata. Le colorazioni dell'involucro (parete cellulare e membrana) daranno un segnale diverso a seconda della posizione della fetta Z. Se la sezione Z include la parte superiore o inferiore della cella, il segnale sarà più forte rispetto a quando la sezione Z passa attraverso il centro della cella dove è colorato solo il contorno della cella.
Le traiettorie del cordone dal canale rosso sono state tracciate per 20 fotogrammi, dove le traiettorie individuali avevano una durata minima di 2 fotogrammi e un massimo di 20 fotogrammi con 19 segmenti di traiettoria che collegavano i telai (Figura 5). Per studiare la dipendenza dalla densità cellulare della mobilità delle perline, è stata determinata l'intensità GFP per voxel (ciascuna delle singole misurazioni nell'immagine del voxel 512x512). La densità delle celle attorno a ciascun segmento della traiettoria del cordone è stata calcolata come la densità media locale nel riquadro di delimitazione del segmento.
Per alcuni biofilm, è stato possibile documentare una dipendenza dalla densità statisticamente significativa (Figura 6), in particolare per i biofilm di E. faecalis che sono stati cresciuti su un vetrino coprioggetti e capovolti su un vetrino multipozzetto (Figura 6A). Al contrario, i biofilm di E. faecalis che sono stati cresciuti sul fondo di una piastra a 96 pozzetti (Figura 6B) non hanno mostrato alcuna dipendenza dalla densità. In conclusione, ciò suggerisce che i biofilm altamente fluidi di E. faecalis potrebbero potenzialmente essere leggermente compressi a causa del montaggio su un vetrino multipozzetto, il che è coerente con una riduzione del numero di perle che si muovono più rapidamente e di quelle con piccoli volumi di bounding box che sono intrappolati nella parte superiore del biofilm contro il vetrino (Figura 3C vs Figura 3D). Sia i biofilm di Salmonella ed E. coli (Figura 6C e 6D) che i loro mutanti isogenici (Figura 6E e 6F) hanno mostrato una dipendenza dalla densità cellulare minore o nulla.
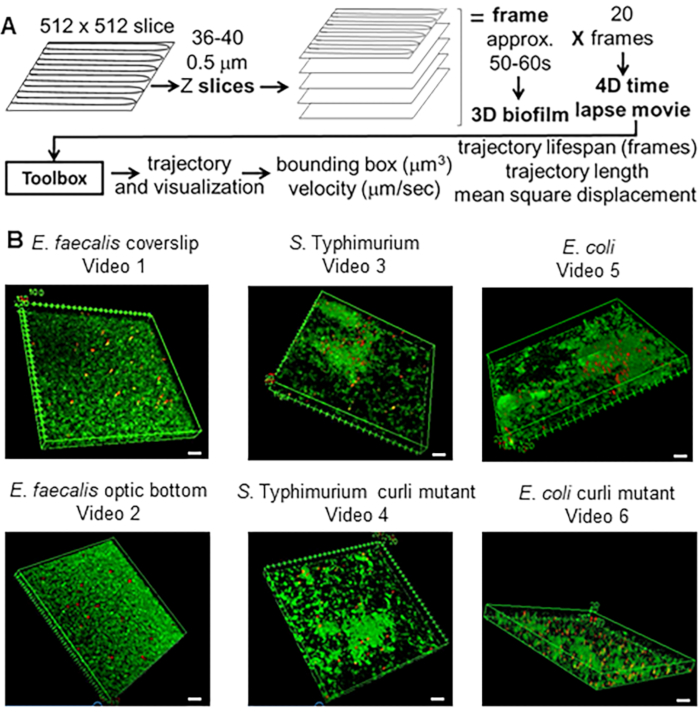
Figura 1. Pipeline di imaging e analisi (passaggi 2-4) (A) I biofilm vengono visualizzati come descritto in 2.2. Utilizzando i biofilm ripresi (vedi 3) sono state generate le traiettorie delle perle come descritto al punto 4. Utilizzando le traiettorie, i dati pertinenti sono stati calcolati con il toolbox di analisi (vedi 5) (B) I biofilm sono stati coltivati come descritto nella fase 1 sui vetrini coprioggetti (E. faecalis, Video 1), S. Typhimurium (Video 3), E. coli (Video 5) e mutanti curli isogeni (Video 4, Video 6) o in una piastra a 96 pozzetti con fondo ottico (E. faecalis in basso, Video 2). La barra della scala bianca è di 20 mm. Questa figura è riprodotta con il permesso di (31). Clicca qui per visualizzare una versione più grande di questa figura.
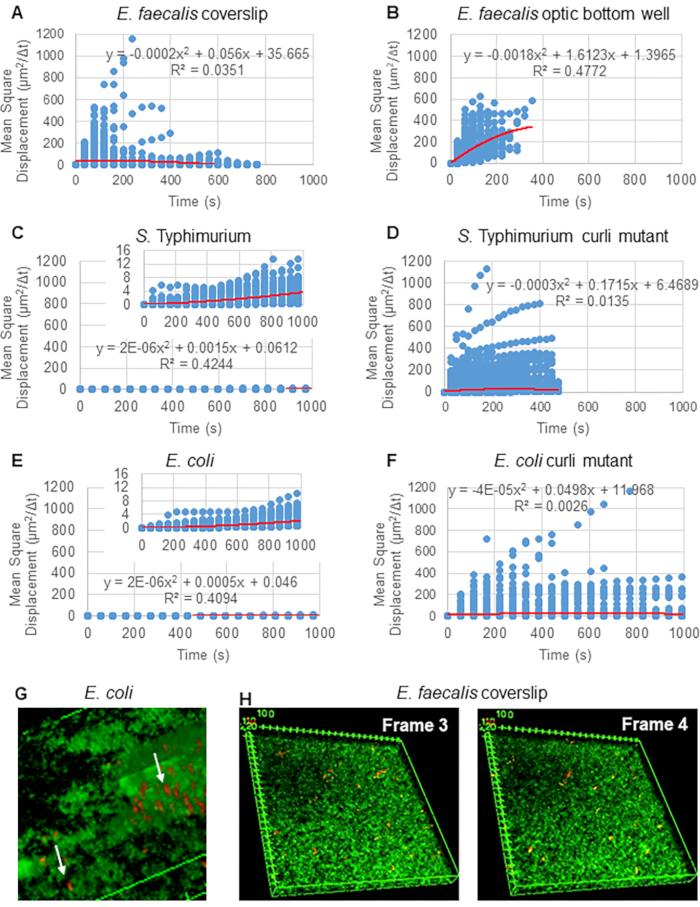
Figura 2. È stato determinato che il movimento del cordone nei biofilm è di natura diffusiva (moto browniano, vedi passaggio 5) (A-F) I dati MSD mostrano un comportamento lineare (linea rossa) per convalidare il moto browniano. I biofilm sono stati coltivati secondo la fase 1 (G) Esempio di movimento delle perle ellittiche osservato nei biofilm di E. coli e S. Typhimurium prelevati da un fotogramma del saggio del biofilm 4D di E. coli (H) Esempio di grandi cambiamenti nei modelli di perline tra il fotogramma 3 e 4 prelevati dal pozzetto di fondo ottico di E. faecalis . Si noti che il biofilm stesso suscita un certo flusso (Video 1 e 2), il che fa sembrare che i fotogrammi 3 e 4 siano orientati in modo diverso. Questa figura è riprodotta con il permesso di (31). Clicca qui per visualizzare una versione più grande di questa figura.
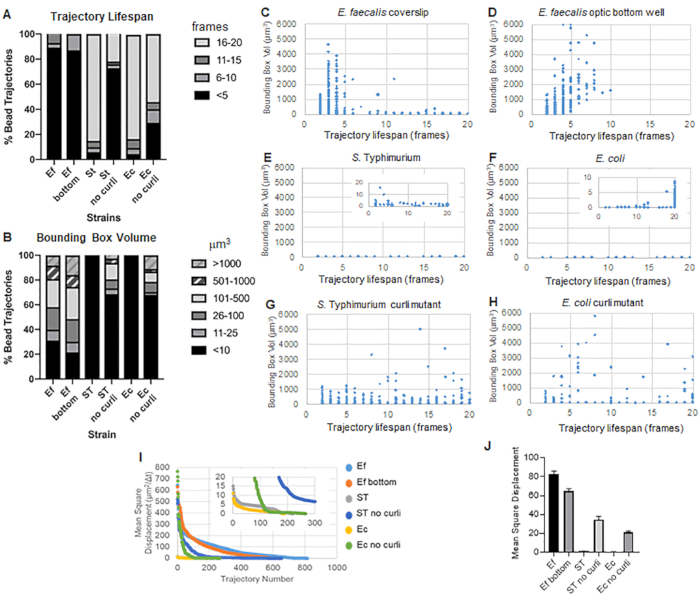
Figura 3. Analisi delle differenze di rigidezza utilizzando i riquadri di delimitazione e la durata della traiettoria. I biofilm sono stati coltivati come descritto nella fase 1 sui vetrini coprioggetti (E. faecalis, Video 1), S. Typhimurium (Video 3), E. coli (Video 5) e mutanti curli isogeni (Video 4, Video 6) o in una piastra a 96 pozzetti con fondo ottico (E. faecalis in basso, Video 2). La durata delle traiettorie è presentata in % delle traiettorie totali delle perle (A) e dei grafici a dispersione (C-H), insieme ai volumi del riquadro di delimitazione (calcolati nella fase 5) (I) Confronto dei DMS delle microsfere nei diversi biofilm (H) DMS medi di ciascun tipo di biofilm. Le barre indicano l'intervallo di confidenza del 95% dei dati. Questa figura è riprodotta con il permesso di (31). Clicca qui per visualizzare una versione più grande di questa figura.
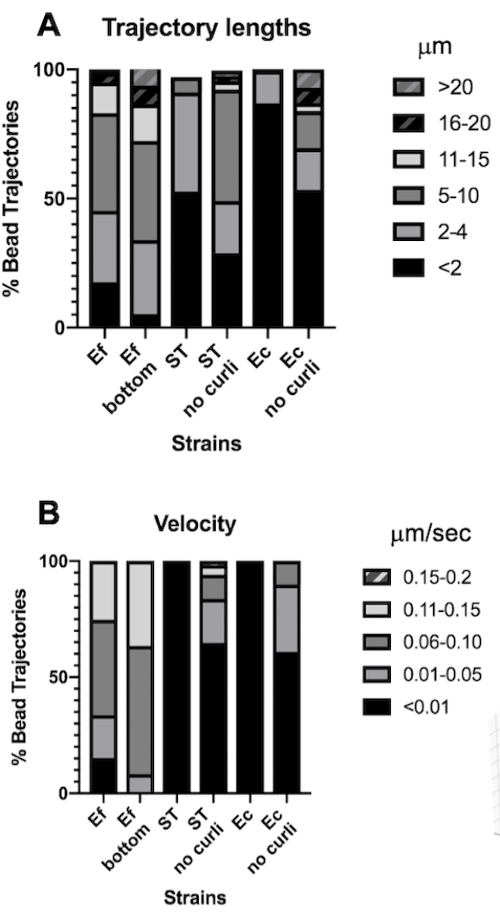
Figura 4. Analisi delle differenze nella lunghezza della traiettoria e nella velocità del cordone. I biofilm sono stati coltivati come descritto nella fase 1. Le lunghezze delle traiettorie sono indicate in μm e sono presentate come % delle traiettorie totali del cordone (A). La velocità è indicata in μm/s e presentata come % delle traiettorie totali del cordone (B). Questa figura è adattata con il permesso di (31). Clicca qui per visualizzare una versione più grande di questa figura.
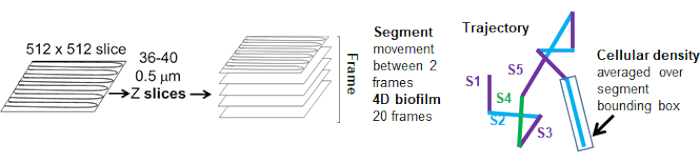
Figura 5. Cenni sull'analisi del segmento di traiettoria. Questa figura è adattata con il permesso di (31). Clicca qui per visualizzare una versione più grande di questa figura.
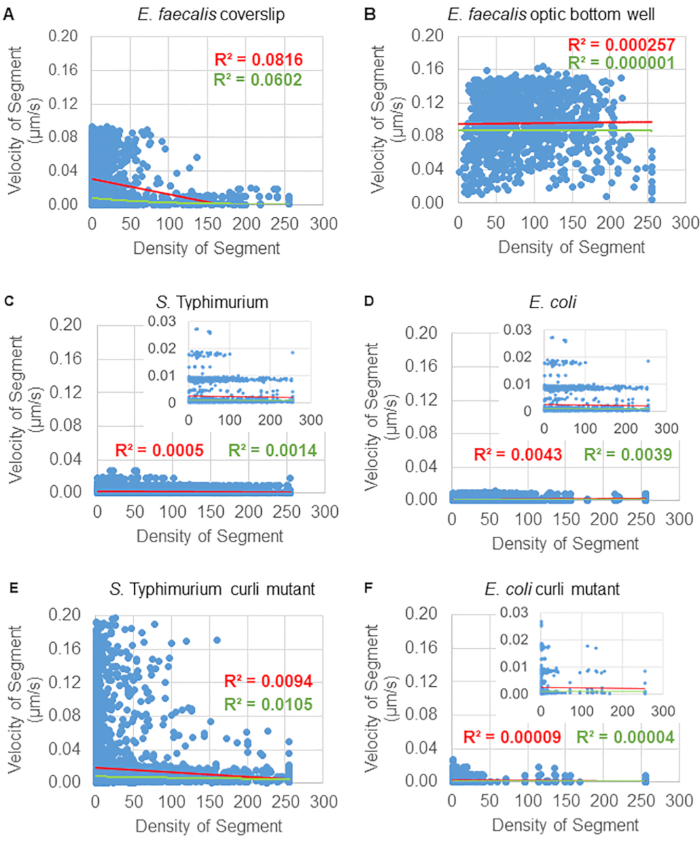
Figura 6. Studio dell'effetto della densità del biofilm sulla velocità delle microsfere utilizzando la pipeline di analisi del biofilm (fase 5). I risultati hanno indicato che la velocità del microscotto non dipende esclusivamente dalla densità cellulare. I biofilm sono stati coltivati come descritto nella fase 1. Le traiettorie sono state analizzate alla scala dei singoli segmenti di traiettoria (Figura 5). Per ogni segmento, la velocità del cordone in μm/s è stata tracciata rispetto alla densità cellulare del riquadro di delimitazione (GFP media per voxel all'interno del riquadro di delimitazione). La linea rossa mostra la regressione lineare e la linea verde la traccia della regressione esponenziale. Questa figura è riprodotta con il permesso di (31). Clicca qui per visualizzare una versione più grande di questa figura.
Video supplementare 1. Video 4D di biofilm di E. faecalis OG1RF nelle 24 ore coltivato su un vetrino coprioggetti in vetro ottico spesso 1,5. Il video time lapse 4D è stato generato utilizzando un microscopio con una risoluzione di 512x512. Una serie Z di 40 immagini è stata generata visualizzando una regione spessa circa 20 μm di un biofilm con incrementi di 0,5 μm. Ogni serie Z era di un fotogramma e richiedeva 50-60 secondi per l'acquisizione. Una serie di 20 fotogrammi contigui è stata catturata per produrre un video 4D. La riproduzione video è a circa 120x. Il video è rappresentativo di almeno 6 esperimenti indipendenti. Clicca qui per scaricare questo video.
Video supplementare 2. Video 4D di un biofilm di E. faecalis OG1RF delle 24 ore coltivato su una piastra inferiore ottica a 96 pozzetti. Il video time lapse 4D è stato generato utilizzando un microscopio con una risoluzione di 512x512. Una serie Z di 40 immagini è stata generata visualizzando una regione spessa circa 20 μm di un biofilm con incrementi di 0,5 μm. Ogni serie Z era di un fotogramma e richiedeva 50-60 secondi per l'acquisizione. Una serie di 20 fotogrammi contigui è stata catturata per produrre un video 4D. La riproduzione video è a circa 120x. Il video è rappresentativo di 3 esperimenti indipendenti.Clicca qui per scaricare questo video.
Video supplementare 3. Video 4D di un biofilm ATCC 14028 del sierotipo di Salmonella enterica Typhimurium cresciuto su vetrini coprioggetto spessi 1,5 per 6-7 giorni. Il video time lapse 4D è stato generato utilizzando un microscopio con una risoluzione di 512x512. Una serie Z di 40 immagini è stata generata visualizzando una regione spessa circa 20 μm di un biofilm con incrementi di 0,5 μm. Ogni serie Z era di un fotogramma e richiedeva 50-60 secondi per l'acquisizione. Una serie di 20 fotogrammi contigui è stata catturata per produrre un video 4D. La riproduzione video è a circa 120x. Il video è rappresentativo di 3 esperimenti indipendenti.Clicca qui per scaricare questo video.
Video supplementare 4. Video 4D del sierotipo Typhimurium biofilm ATCC 14028 curli (csgBA) mutante di Salmonella enterica sono stati coltivati su vetrini coprioggetti spessi 1,5 per 6-7 giorni. Il video time lapse 4D è stato generato utilizzando un microscopio con una risoluzione di 512x512. Una serie Z di 40 immagini è stata generata visualizzando una regione spessa circa 20 μm di un biofilm con incrementi di 0,5 μm. Ogni serie Z era di un fotogramma e richiedeva 50-60 secondi per l'acquisizione. Una serie di 20 fotogrammi contigui è stata catturata per produrre un video 4D. La riproduzione video è a circa 120x. Il video è rappresentativo di 3 esperimenti indipendenti.Clicca qui per scaricare questo video.
Video supplementare 5. Video 4D di E. coli UTI89 coltivato su vetrini coprioggetti in vetro ottico spesso 1,5 per 6-7 giorni. Il video time lapse 4D è stato generato utilizzando un microscopio con una risoluzione di 512x512. Una serie Z di 40 immagini è stata generata visualizzando una regione spessa circa 20 μm di un biofilm con incrementi di 0,5 μm. Ogni serie Z era di un fotogramma e richiedeva 50-60 secondi per l'acquisizione. Una serie di 20 fotogrammi contigui è stata catturata per produrre un video 4D. La riproduzione video è a circa 120x. Il video è rappresentativo di 3 esperimenti indipendenti. Clicca qui per scaricare questo video.
Video supplementare 6. Video 4D Il mutante di E. coli UTI89 curli (csgBA) è cresciuto su vetrini coprioggetti spessi 1,5 per 6-7 giorni. Il video time lapse 4D è stato generato utilizzando un microscopio con una risoluzione di 512x512. Una serie Z di 40 immagini è stata generata visualizzando una regione spessa circa 20 μm di un biofilm con incrementi di 0,5 μm. Ogni serie Z era di un fotogramma e richiedeva 50-60 secondi per l'acquisizione. Una serie di 20 fotogrammi contigui è stata catturata per produrre un video 4D. La riproduzione video è a circa 120x. Il video è rappresentativo di 3 esperimenti indipendenti.Clicca qui per scaricare questo video.
Discussione
Passaggi critici e risoluzione dei problemi
La sfida più grande di questa tecnica è l'utilizzo di un vetrino coprioggetti montato con un biofilm molto viscoso come E. faecalis. Il vetrino coprioggetti deve essere posizionato con cura e precisione sul vetrino multipozzetto senza riposizionarlo. Durante la fase di sigillatura, è necessario prestare attenzione per evitare di spingere verso il basso il vetrino coprioggetti o di spingerlo/farlo scivolare accidentalmente sulla superficie del vetrino. Qualsiasi movimento o pressione può creare tensione superficiale e bloccare il movimento di un biofilm viscoso. Se possibile, il confronto delle proprietà del materiale del biofilm mediante l'imaging di un biofilm su un pozzetto inferiore ottico con una montatura del vetrino coprioggetti consentirà la valutazione della tecnica. Se eseguita correttamente, una montatura per vetrino coprioggetti assomigliava molto a un biofilm in una piastra inferiore ottica per E. faecalis.
Inoltre, quando si utilizza un vetrino coprioggetti montato, è necessario evitare l'imaging delle interfacce del biofilm con il vetrino coprioggetti nella parte inferiore o il vetrino nella parte superiore. Quando si utilizza un cannocchiale invertito, con il vetrino coprioggetti nella parte inferiore, possono esserci perline intrappolate alla base del biofilm contro il vetrino coprioggetti. Queste perle passano attraverso il biofilm e rimangono intrappolate contro il vetrino coprioggetti anche dopo un lavaggio delicato. Hanno coordinate x, y e z pari a 0 e coordinate del riquadro di delimitazione pari a 0. Tuttavia, per alcune applicazioni, come l'esame dell'integrità del biofilm dopo il trattamento, questi punti dati possono essere utilizzati come strumento. La capacità delle perle di penetrare attraverso una spessa regione di biofilm fino al fondo del vetrino coprioggetti può essere utilizzata per valutare l'integrità del biofilm dopo il trattamento (manoscritto in preparazione in collaborazione con il laboratorio Tükel). Nella parte superiore del biofilm, in un biofilm viscoso come E. faecalis avevamo alcune prove di compattazione imposta dal vetrino coprioggetti. Ciò ha limitato il movimento di alcune perle all'interfaccia del vetrino e potrebbe aver introdotto una certa dipendenza dalla densità nell'analisi del movimento delle perline.
Le fasi di lavaggio sono state necessarie per i biofilm perché il terreno di coltura ha una forte autofluorescenza nel canale verde. Scegliamo di utilizzare le perle in eccesso e di rimuovere le perle non associate mediante lavaggio per massimizzare le perle associate per ottenere la caratterizzazione più accurata delle regioni osservate.
Il numero di perle e lavaggi necessari per ottenere i set di dati desiderati deve essere determinato empiricamente. La presenza di troppe perle in un biofilm genera set di dati incredibilmente grandi che sono difficili da analizzare. La presenza di un numero insufficiente di perle non genera un campionamento approfondito degli ambienti del biofilm. Tuttavia, il controllo del numero di perle aggiunte (2x107 perle in 1 mL di PBS) e l'uso delle fasi di lavaggio, hanno portato a un numero relativamente consistente di perle (40-140) che si associano al biofilm a seconda della sua struttura, disposizione spaziale e composizione.
Quando si studiano biofilm con regioni miste viscose e rigide, le perle possono rimanere intrappolate nelle regioni rigide nel tempo. In questo caso, l'imaging deve essere avviato immediatamente dopo l'aggiunta delle perline. Questo spesso non può essere ottenuto utilizzando i vetrini coprioggetti, ma richiede piastre a fondo ottico o celle a flusso in cui l'imaging può essere eseguito immediatamente dopo l'aggiunta delle perle e le fasi di lavaggio.
Modifiche e applicazioni future
Utilizzo di dispositivi microfluidici. Nei nostri studi, le condizioni ottimali stabilite per lo studio dei biofilm delle Enterobacteriaceae richiedevano la crescita del biofilm sotto forma di pellicola all'interfaccia aria-liquido. Ciò ha limitato l'uso di piastre ottiche inferiori e dispositivi microfluidici negli studi. Tuttavia, quando le condizioni di formazione del biofilm lo consentono, i biofilm possono essere coltivati in camere microfluidiche o celle a flusso. I biofilm potrebbero quindi essere lavati e le perle introdotte attraverso il dispositivo microfluidico con una minima interruzione del biofilm.
Aggiunta di perle durante la crescita del biofilm. Abbiamo scelto di aggiungere le perle in eccesso ai biofilm, per poi rimuovere le perle non associate mediante lavaggio delicato per ottimizzare il numero di perle presenti durante l'analisi. Nei biofilm viscosi di E. faecalis , è possibile che le perle si siano dissociate e riassociate durante il tempo di imaging di 20 minuti. Se un numero ridotto di perle viene aggiunto in momenti diversi durante la crescita del biofilm, potrebbe essere possibile intrappolare le perle nel biofilm, consentendo una caratterizzazione più accurata del movimento del biofilm in biofilm più viscosi.
Scelta della regione da fotografare. Per gli studi sulle proprietà dei materiali, è meglio scegliere regioni spesse e sottili del biofilm. Tuttavia, quando si studiano i cambiamenti nelle proprietà del materiale di un biofilm trattato, è possibile visualizzare regioni confluenti spesse per determinare i cambiamenti nelle proprietà viscoelastiche e nella penetrazione del cordone in tali regioni. In questo caso, la ricerca di perle che sono penetrate nel biofilm e sono finite intrappolate contro il vetrino coprioggetti è una misura utile della rottura del biofilm.
Imaging sotto flusso. Utilizzando celle a flusso in vetro ottico o dispositivi microfluidici, è possibile visualizzare il movimento di perle o batteri in un biofilm sotto flusso. Questo può essere fatto in diversi modi. Può essere fatto mediante iniezione di perle nell'intera camera seguita da una breve incubazione per consentire l'associazione delle perle con il biofilm. Le perle non associate possono essere rimosse mediante lavaggio e il biofilm può essere visualizzato con o senza flusso. Al contrario, un piccolo numero di perle può essere introdotto in un lato della camera e il loro movimento attraverso e nel biofilm può essere tracciato sotto flusso. Quando si utilizza il flusso, è necessario prestare attenzione nella scelta delle impostazioni di tracciamento delle perline Mosaic (Passaggio 4.5). Negli studi attuali l'impostazione dinamica era browniana. I calcoli MSD hanno confermato che il movimento era probabilmente diffuso, rendendo il browniano l'impostazione appropriata.
Colorazione della matrice. Negli studi attuali, la colorazione con Syto9 esamina la densità cellulare e non la densità della struttura del biofilm. Ad esempio, la presenza di amiloidi probabilmente aumenta la densità del materiale della matrice del biofilm. La dipendenza del movimento dalla densità dell'amiloide potrebbe essere determinata utilizzando coloranti fluorescenti della matrice al posto di Syto9.
Batteri marcati in fluorescenza. I batteri marcati in fluorescenza possono essere utilizzati per tracciare il movimento dei batteri esogeni attraverso i biofilm (ad esempio, batteri contenenti plasmidi). La sfida con i batteri marcati in fluorescenza, come gli enterococchi, è che formano singoli, diplococchi e catene corte, il che complica la capacità di tracciare con precisione i batteri. Questo processo sarebbe più facile se i batteri avessero una morfologia a singola cellula.
Limitazioni
Limitazioni nella visualizzazione della traiettoria e nell'unione.
Una limitazione del metodo è la visualizzazione della traiettoria e la cucitura. Le traiettorie ricostruite e analizzate sono costituite da coordinate dei punti x, y, z, dove i punti successivi definiscono il percorso lineare tra questi punti. La visualizzazione di tali traiettorie lineari a tratti può essere ottenuta con vari strumenti. Un approccio è stato quello di utilizzare i notebook Python e Jupyter insieme ai plugin Python, Pandas e Matplotlib. Sebbene sia stato possibile visualizzare le singole traiettorie esistenti nell'articolo del Journal of Bacteriology in cui questa tecnica è stata originariamente pubblicata34, c'erano ancora limitazioni significative che verranno affrontate nella ricerca futura.
Attualmente, il numero di traiettorie ricostruite è maggiore del numero di perle nel biofilm, il che significa che più traiettorie possono corrispondere a una perlina. Ciò può essere causato da un debole segnale confocale in un fotogramma in cui Mosaic terminerà una traiettoria e ne avvierà una seconda. Questo può essere registrato come più traiettorie più brevi per un perline, specialmente nei biofilm meno viscosi. Un'altra causa del gran numero di traiettorie è la mancanza di cuciture della traiettoria. Soprattutto nei biofilm di E. faecalis optic bottom well, le perle rimangono visivamente associate al biofilm durante l'imaging (Video supplementare 2). Tuttavia, non c'erano traiettorie più lunghe di 10 e oltre il 90% delle traiettorie aveva una durata di 5 fotogrammi o meno (Figura 3D). Se il software viene utilizzato per analizzare solo le traiettorie al di sopra di una lunghezza definita (ad esempio, quando si tracciano cellule in grado di trasferire plasmidi), le traiettorie più brevi possono essere automaticamente rimosse dal set di dati. Tuttavia, ci sono altri scopi per i quali la cucitura delle traiettorie può essere molto importante. Infine, l'incapacità di tracciare il movimento rapido delle perle come una singola traiettoria potrebbe comportare più traiettorie nei biofilm delle Enterobacteriaceae a causa del rapido movimento nella direzione Z che si traduce in perle di forma ellittica (Figura 2G). La possibilità di traiettorie di sutura interrotte da un rapido movimento anisotropo sarà importante per studiare l'effetto della matrice amiloide curli nelle Enterobacteriaceae.
Significato
È stato sviluppato un flusso di lavoro computazionale per studiare le traiettorie delle perline per confrontare le proprietà dei materiali dei biofilm 3D. Il flusso di lavoro consente ai ricercatori di identificare i parametri critici che possono essere utilizzati nella modellazione computazionale della fluidodinamica in biofilm eterogenei. Con l'aiuto di questo analizzatore di microsfere open-source, è stato possibile studiare l'effetto del curli amiloide batterico sulle proprietà del materiale, mostrando una maggiore rigidità della matrice del biofilm dovuta al curli. In un contesto più generale, il valutatore può essere utilizzato per studiare i cambiamenti nella struttura del biofilm indotti dal trattamento del biofilm o da diverse condizioni ambientali, come il flusso. Ad esempio, lo strumento viene utilizzato per analizzare l'effetto del trattamento con anticorpi monoclonali sulla distruzione delle strutture del biofilm in collaborazione con il laboratorio Tükel (LKSOM Temple University). La cassetta degli attrezzi per il valutatore di perline è completamente adattabile ed estendibile in modo modulare utilizzando VRL-Studio per migliorare ed estendere ulteriormente le sue funzioni.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Il lavoro nei laboratori GQ e BAB non ha ricevuto sovvenzioni specifiche da alcuna agenzia di finanziamento nei settori pubblico, commerciale o senza scopo di lucro. Gli autori ringraziano Isaac Klapper, Ph.D (Dipartimento di Matematica, Temple University) per l'utile discussione e Çagla Tükel (Dipartimento di Microbiologia e Immunologia, Temple University) per l'esperienza nelle Enterobacteriaceae nella pubblicazione iniziale di questa tecnica.
Materiali
| Name | Company | Catalog Number | Comments |
| 96-well plates, No. 1.5 Uncoated Coverslip, 5 mm Glass Diameter | MatTek | P96G1.55F | |
| Fisherbrand Cover Glasses: Circles | Fisher Scientific | 12-293-232P | 1.5 optic glass coverslip |
| Invitrogen Syto 9 Green Fluorescent Nucleic Acid Stain | Invitrogen | S34854 | |
| Molecular Probes FluoSpheres Carboxylate-modified Microspheres, 1 um, crimson fluorescent (625/645) | Molecular Probes | F8816 |
Riferimenti
- Huang, R., Li, M., Gregory, R. L. Bacterial interactions in dental biofilm. Virulence. 2, 435-444 (2011).
- Nadell, C. D., Drescher, K., Wingreen, N. S., Bassler, B. L. Extracellular matrix structure governs invasion resistance in bacterial biofilms. ISME Journal. 9, 1700-1709 (2015).
- Billings, N., Birjiniuk, A., Samad, T. S., Doyle, P. S., Ribbeck, K. Material properties of biofilms - key methods for understanding permeability and mechanics. Reports on Progress in Physics. 78, 036601 (2015).
- Araújo, G. R. d. e. S., Viana, N. B., Gómez, F., Pontes, B., Frases, S. The mechanical properties of microbial surfaces and biofilms. The Cell Surface. 5, 100028 (2019).
- Tallawi, M., Opitz, M., Lieleg, O. Modulation of the mechanical properties of bacterial biofilms in response to environmental challenges. Biomaterials Science. 5, 887-900 (2017).
- Tursi, S. A., Tükel, &. #. 1. 9. 9. ;. Curli-Containing Enteric Biofilms Inside and Out, Matrix Composition, Immune Recognition, and Disease Implications. Microbiology and Molecular Biology Reviews. 82, (2018).
- Dueholm, M. S., Albertsen, M., Otzen, D., Nielsen, P. H. Curli functional amyloid systems are phylogenetically widespread and display large diversity in operon and protein structure. PLoS One. 7 (12), 51274 (2012).
- Hung, C., et al. Escherichia coli biofilms have an organized and complex extracellular matrix structure. mBio. 4, 00645 (2013).
- Kikuchi, T., Mizunoe, Y., Takade, A., Naito, S., Yoshida, S. Curli fibers are required for development of biofilm architecture in Escherichia coli K-12 and enhance bacterial adherence to human uroepithelial cells. Microbiology and Immunology. 49, 875-884 (2005).
- Gallo, P. M., et al. Amyloid-DNA Composites of Bacterial Biofilms Stimulate Autoimmunity. Immunity. 42, 1171-1184 (2015).
- Serra, D. O., Richter, A. M., Hengge, R. Cellulose as an architectural element in spatially structured Escherichia coli biofilms. Journal of Bacteriology. 195, 5540-5554 (2013).
- Oh, Y. J. Characterization of curli A production on living bacterial surfaces by scanning probe microscopy. Biophysical Journal. 103, 1666-1671 (2012).
- Lembré, P., Di Martino, P., Vendrely, C. Amyloid peptides derived from CsgA and FapC modify the viscoelastic properties of biofilm model matrices. Biofouling. 30, 415-426 (2014).
- Oh, Y. J., et al. Curli mediate bacterial adhesion to fibronectin via tensile multiple bonds. Scientific Reports. 6, 33909 (2016).
- Vidakovic, L., Singh, P. K., Hartmann, R., Nadell, C. D., Drescher, K. Dynamic biofilm architecture confers individual and collective mechanisms of viral protection. Nature Microbiology. 3, 26-31 (2018).
- Tursi, S. A., et al. Salmonella Typhimurium biofilm disruption by a human antibody that binds a pan-amyloid epitope on curli. Nature Communications. 11, 1007 (2020).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15 (8), 1007978 (2019).
- Taglialegna, A., et al. The biofilm-associated surface protein Esp of Enterococcus faecalis forms amyloid-like fibers. Npj Biofilms and Microbiomes. 6, 15 (2020).
- Gour, S., Kumar, V., Rana, M., Yadav, J. K. Pheromone peptide cOB1 from native Enterococcus faecalis forms amyloid-like structures, A new paradigm for peptide pheromones. Journal of Peptide Science. 25, 3178 (2019).
- Stoodley, P., Debeer, D., Lewandowski, Z. Liquid flow in biofilm systems. Applied and Environmental Microbiology. 60, 2711-2716 (1994).
- Birjiniuk, A., et al. Single particle tracking reveals spatial and dynamic organization of the E. coli biofilm matrix. New Journal of Physics. 16, 085014 (2014).
- Chew, S. C., et al. Dynamic remodeling of microbial biofilms by functionally distinct exopolysaccharides. mBio. 5, 01536 (2014).
- Cao, H., et al. Revealing region-specific biofilm viscoelastic properties by means of a micro-rheological approach. Npj Biofilms and Microbiomes. 2, 5 (2016).
- Galy, O., et al. Mapping of bacterial biofilm local mechanics by magnetic microparticle actuation. Biophysical Journal. 103, 1400-1408 (2012).
- Rogers, S. S., vander Walle, C., Waigh, T. A. Microrheology of bacterial biofilms in vitro, Staphylococcus aureus and Pseudomonas aeruginosa. Langmuir. 24, 13549-13555 (2008).
- Hart, J. W., Waigh, T. A., Lu, J. R., Roberts, I. S. Microrheology and Spatial Heterogeneity of Staphylococcus aureus Biofilms Modulated by Hydrodynamic Shear and Biofilm-Degrading Enzymes. Langmuir. 35 (9), 3553-3561 (2019).
- van Merode, A. E. J., van der Mei, H. C., Busscher, H. J., Krom, B. P. Influence of culture heterogeneity in cell surface charge on adhesion and biofilm formation by Enterococcus faecalis. Journal of Bacteriology. 188, 2421-2426 (2006).
- Tariq, M., Bruijs, C., Kok, J., Krom, B. P. Link between Culture Zeta Potential Homogeneity and Ebp in Enterococcus faecalis. Applied and Environmental Microbiology. 78, 2282-2288 (2012).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ, 25 years of image analysis. Nature Methods. 9, 671-675 (2012).
- . Oracle.com Available from: https://docs.oracle.com/en/java (2021)
- Hoffer, M., Poliwoda, C., Wittum, G. Visual reflection library, a framework for declarative GUI programming on the Java platform. Computing and Visualization in Science. 16, 181-192 (2013).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15, 1007978 (2019).
- Malhotra, K., et al. Development of a New Bead Movement-Based Computational Framework Shows that Bacterial Amyloid Curli Reduces Bead Mobility in Biofilms. Journal of Bacteriology. 202, 00253 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon