Method Article
Uma estrutura computacional baseada em movimento de contas para análise tridimensional da heterogeneidade do material de biofilme
Neste Artigo
Resumo
Descrevemos um método para analisar e quantificar o padrão de movimento de grânulos de carboxilato de 1 μm através de biofilmes bacterianos heterogêneos. A comparação dos padrões de movimento pode ser usada para quantificar as diferenças nas propriedades dos materiais dos biofilmes.
Resumo
Diferenças nas propriedades materiais dos biofilmes bacterianos foram observadas em biofilmes de diferentes espécies bacterianas, dentro da mesma espécie sob diferentes condições de crescimento e após tratamento com moléculas modificadoras da matriz. Para melhor quantificar as propriedades do material dos biofilmes 3D, um fluxo de trabalho experimental e computacional foi desenvolvido e aplicado para examinar as diferenças entre os biofilmes de Enterococcus faecalis, Salmonella enterica sorotipo Typhimurium e Escherichia coli , bem como o papel do curli amilóide na confirmação da rigidez dos biofilmes de Enterobacteriaceae. A dinâmica espaço-temporal de esferas de carboxilato de 1 μm em biofilmes foi rastreada em biofilmes 3D de 20 μm durante 20 minutos. As pilhas de imagens 4D foram processadas usando o plug-in Mosaic no ImageJ para produzir dados de trajetória 3D do movimento do cordão. Esses dados de trajetória foram analisados com uma caixa de ferramentas Bead Evaluator recém-desenvolvida, onde os dados de movimento, incluindo vida útil da trajetória, velocidades do bead, densidades de células ao longo das trajetórias e informações da caixa delimitadora foram calculados e armazenados em arquivos csv. Este artigo apresenta o fluxo de trabalho desde a configuração experimental e gravação de imagens até o cálculo e análise da trajetória do cordão. A estrutura dos biofilmes contendo cachos resultou em interações mais estáveis com os grânulos e menos movimento dos grânulos do que nos biofilmes mutantes e enterocócicos. O movimento do grânulo não pareceu fortemente dependente da densidade celular ao medir a velocidade do grânulo e o volume da caixa delimitadora da trajetória, apoiando a hipótese de que outras propriedades materiais dos biofilmes controlam a dinâmica do grânulo. Esta técnica é amplamente aplicável para quantificar diferenças em biofilmes de diferentes composições de matriz, bem como biofilmes antes e depois de tratamentos modificadores de matriz.
Introdução
Os biofilmes bacterianos são onipresentes como parte da microbiota humana e interagem continuamente com as moléculas. Essas moléculas variam em tamanho, desde antibióticos de 1 nm e bactérias de 1-3 mm até partículas maiores de fibra no trato gastrointestinal. A composição de biofilmes de uma ou várias espécies afeta as propriedades do material e, portanto, o padrão de movimento das partículas através dos biofilmes 1,2,3,4,5. Um exemplo são os amilóides bacterianos, que têm uma estrutura de folha beta cruzada fibrilar conservada6. O curli amilóide é expresso por bactérias entéricas, como Escherichia coli e Salmonella enterica, sorotipo Typhimurium, e genes foram detectados em vários outros filos bacterianos7. Várias propriedades materiais dos biofilmes são afetadas pelo curli 8,9. Curli interage diretamente com outros componentes da matriz, como DNA extracelular (eDNA) e celulose10,11. O curli envolve as células e afeta a rigidez da membrana celular12 e as propriedades viscoelásticas gerais do biofilme13. Curli medeia o aumento da resistência à tração ligando-se à fibronectina, resultando em um aumento na forte fixação da superfície do vidro14. Os bacteriófagos que chegam se ligam ao curli e limitam a invasão de fagos em biofilmes15.
Ao usar lâminas de poço revestido multiteste para analisar Enterococcus faecalis, E. coli e S. com aproximadamente 20 μm de espessura. Biofilmes de Typhimurium usando microscopia confocal, diferenças claras entre E. coli, S. Foram observados biofilmesde Typhimurium 10,16 e E. faecalis (estudo atual). Embora os biofilmes das espécies de Enterobacteriaceae tivessem um alto nível de rigidez e as áreas com baixa densidade celular fossem fáceis de visualizar, a obtenção de imagens nítidas de alta resolução de biofilmes de E. faecalis usando a média de linha e quadro exigiu a aplicação de pressão na lâmina para induzir tensão superficial suficiente para a estabilidade celular durante o processo de imagem. Os amiloides bacterianos, como o curli, formam estruturas altamente ordenadas, sugerindo que podem ser relativamente rígidos17. Isso motivou a hipótese de que o curli amilóide poderia estar induzindo rigidez em E. coli e S. Biofilmes de Typhimurium. Não houve evidências claras de que E. faecalis estava expressando amilóides nas condições estudadas. A proteína Esp, um gene de pilina associado a cepas mais patogênicas de E. faecalis, demonstrou recentemente produzir estruturas amilóides18; no entanto, usando pesquisas de blastn e blastp, esse gene não foi detectado na cepa OG1RF do tipo comensal de E. faecalis usada nesses estudos. O feromônio cOB1, produzido por OG1RF, pode formar estruturas semelhantes a amiloide19. No entanto, com as condições de crescimento de biofilme fornecidas e os métodos de detecção de amilóide usados anteriormente para S. A coloração amiloide de Typhimurium10 em E. faecalis, OG1RF amilóides não pôde ser detectada (dados não mostrados). Uma nova técnica de imagem quadridimensional (4D) foi desenvolvida para comparar as propriedades gerais do material entre os viscosos E. faecalis, E. coli e S. Typhimurium, bem como para determinar a contribuição da amilóide para os biofilmes de Enterobacteriaceae usando mutantes amilóides de S. Typhimurium e E. coli.
No passado, as esferas fluorescentes eram usadas com sucesso para analisar as propriedades do material de biofilmes em duas dimensões (2D) usando microrreologia 20,21,22,23,24,25. Isso pode ser aplicado a um biofilme tridimensional estudando fatias ópticas 2D em várias profundidades no biofilme26. A técnica atual foi desenvolvida para rastrear contas em microescala de 1 μm em 3D ao longo do tempo para uso em modelagem 4D. Parte da justificativa era o conceito abrangente de usar modelagem 4D para entender o movimento de plasmídeos através das comunidades de microbiota gastrointestinal. Foram utilizados grânulos de carboxilato carregados fluorescentemente com um diâmetro de 1 μm, uma vez que correspondem bem, no que diz respeito ao tamanho e carga, a E. faecalis, o organismo modelo escolhido para movimentação e manutenção do plasmídeo27, 28. Um ensaio 4D para quantificar as propriedades físicas dos biofilmes foi desenvolvido (Figura 1A). Na metodologia elaborada, grânulos foram adicionados aos biofilmes e suas trajetórias espaço-temporais foram registradas através de biofilmes de 10-20 μm de espessura ao longo de 10-20 minutos. As trajetórias do cordão em 3D foram então quantificadas em termos de comprimento da trajetória, velocidade do cordão, volume da caixa delimitadora da trajetória (caixa mínima contendo a trajetória) e densidade celular da caixa delimitadora usando uma caixa de ferramentas recém-desenvolvida. O protocolo a seguir pode ser empregado para gerar dados de imagem 4D de bactérias e biofilmes contendo esferas, para pré-processar os dados com o ImageJ29 e o plug-in Mosaic e para analisar as trajetórias das esferas com uma caixa de ferramentas Bead Evaluator.
Essa técnica tem várias aplicações para examinar as propriedades do material, bem como rastrear o movimento de partículas e bactérias em três dimensões. Por exemplo, uma versão inicial dessa técnica foi usada para caracterizar o efeito de anticorpos monoclonais direcionados contra o curli na integridade estrutural dos biofilmes16. A versão completa possui várias ferramentas para fornecer uma análise mais detalhada das propriedades do material do biofilme e continua a ser usada para examinar os efeitos do tratamento com anticorpos monoclonais em biofilmes. Partículas de diferentes cargas podem ser usadas para examinar as propriedades de carga do material dos biofilmes e o movimento de partículas através de biofilmes com diferentes composições de matriz. Isso poderia ser usado para comparar os resultados da microrreologia 2D que revelam as propriedades do material responsável pelo movimento dos grânulos que observamos em biofilmes que não estavam sob fluxo. Esta técnica também pode ser usada em biofilmes de espécies mistas com regiões de diferentes composições de biofilme. Os biofilmes podem ser imagens ao vivo sob fluxo em dispositivos microfluídicos e células de fluxo para examinar as mudanças nas propriedades do material entre biofilmes estáticos e de fluxo, bem como o efeito do fluxo no movimento das partículas. As técnicas também podem ser aplicadas a bactérias marcadas com fluorescência para caracterizar o movimento de bactérias exógenas através de uma comunidade de biofilme. Usando três cores, bactérias doadoras marcadas com fluorescência, bactérias receptoras marcadas com fluorescência e plasmídeos marcados com fluorescência podem ser usados para rastrear movimento, acoplamento e transferência de plasmídeos.
Protocolo
1. Preparação de biofilme
NOTA: Os biofilmes para análise podem ser cultivados usando qualquer método que permita que o biofilme se forme em uma superfície de vidro óptico. A estrutura do biofilme deve aderir à superfície óptica o suficiente para que a estrutura não seja interrompida durante as etapas de lavagem e/ou montagem do protocolo. Abaixo descreve a técnica para placas de fundo óptico de 96 poços e lamínulas de vidro de 12 mm em placas de 24 poços. Outras opções incluem placas de fundo óptico de tamanhos diferentes e câmaras de fluxo óptico com e sem fluxo.
- Configuração de biofilme
- Adicione meio de crescimento bacteriano aos poços da placa. Para este estudo, para E. faecalis, adicione 2 mL de Todd-Hewitt (TH) a uma placa de 24 poços e adicione 0,4 mL de TH às placas de fundo óptico de 96 poços. Para S. Typhimurium, E. coli e os mutantes isogênicos csgAB curli, adicionam 0,7 mL de caldo Luria sem sal (LB) aos poços. Se estiver usando câmaras ópticas inferiores, prossiga para a Etapa 1.2.
- Coloque lamínulas ópticas de vidro de 12 mm # 1,5 em uma placa de Petri e cubra-as com etanol.
- Usando uma pinça, remova a lamínula e use uma chama para acender o álcool restante no fogo. Deixe o álcool queimar. Basta usar a chama para incendiá-los; Não segure a lamínula na chama, pois ela rachará. Deixe a lamínula esfriar por 10-20 s antes de colocá-la no poço para evitar rachaduras.
- Coloque a lamínula no poço que contém o meio em um ângulo para evitar que ele fique em cima do meio. Não adicione a lamínula a um poço seco e, em seguida, adicione médio, pois isso fará com que a lamínula grude no fundo do poço.
- Use uma ponta de pipeta estéril para empurrar cuidadosamente a lamínula para o fundo do poço que contém o meio.
NOTA: Lembre-se de combinar a espessura (# 1 ou # 1.5) da lamínula de vidro óptico ou placa inferior óptica com a espessura da óptica do microscópio confocal.
- Incube o biofilme em condições adequadas para o crescimento do biofilme. Nesses estudos, cultive biofilmes de E. faecalis como culturas estáticas a 37 ° C e cultive biofilmes de E. coli aeróbicos a 30 ° C.
- Nesses estudos, cultive biofilmes de E. faecalis por 2 dias com interruptores médios pela manhã e no início da noite. Para evitar danificar ou desalojar o biofilme, incline cuidadosamente a placa. Coloque a ponta da pipeta perto da borda inferior do poço e retire lentamente o meio. Adicione o primeiro mL de meio fresco da mesma maneira. Adicione o segundo mL lentamente perto da interface meio/poço.
- Nesses estudos, use condições de crescimento para uma produção ideal de caracóis. Crescer S. Biofilmes de Typhimurium a 28 ° C por 6-8 dias incubados em uma inclinação que permitiu que o biofilme se fixasse em cerca de 2/3 do caminho até a lâmina e depois crescesse como uma película na interface ar-líquido. Isso foi feito sem uma mudança de meio. Para evitar que o meio seque, coloque a placa de 24 poços em uma câmara com uma panela de água.
2. Imagem 4D
- Preparação da montagem de biofilme
- Dilua os grânulos FluoSpheres de carmesim de 1 μm de carboxilato 1:50 em PBS (2 x 107 grânulos em 1 mL de PBS). Se estiver usando câmaras ópticas de fundo, adicione Syto9 a uma diluição de 1 μL a 300 μL de preparação de esferas.
- (opcional) Lave o biofilme para remover vestígios de meio de crescimento se o meio de crescimento tiver autofluorescência. Nesses experimentos, lave duas vezes com 1 mL de PBS, inclinando cuidadosamente a placa, colocando a ponta da pipeta perto da borda inferior do poço e retirando lentamente o meio. Adicione PBS colocando a ponta perto da borda inferior e encha lentamente o poço. Use esta técnica em poços ópticos de fundo, além de lamínulas em uma placa de 24 poços.
- Remova o meio ou PBS. Adicionar ao biofilme esferas de carmesim diluídas preparadas em 2.1.1. Nesses estudos, adicione 1 mL de grânulos (2x107) às lamínulas e 0,2 mL de grânulos (4x106) e Syto9 a placas de fundo óptico de 96 poços.
- Incube por 1 min em temperatura ambiente para permitir a associação do grânulo.
- Remova as contas e lave suavemente o biofilme uma vez com PBS para remover as contas não associadas. Nestes estudos, para a lamínula, lave suavemente com 1 mL de PBS, encha novamente o poço com 1 mL de PBS e prossiga para 2.1.6. Para as placas de fundo óptico de 96 poços, lave suavemente o biofilme com 0,2 mL de PBS e reabasteça o poço com 0,2 mL de PBS. A câmara inferior óptica agora está pronta para a imagem, então prossiga para a etapa 2.2.
- (Se estiver usando uma lamínula) Adicione 1 μL de Syto9 (coloração verde de DNA fluorescente; diluído de acordo com as instruções do fabricante) no centro de um poço em uma lâmina multiteste revestida de 10 poços. Essas lamínulas revestidas têm profundidades de poço de 23-25 μm.
- (Se estiver usando uma lamínula) Usando uma pinça esterilizada por chama de álcool, remova cuidadosamente a lamínula do poço e inverta no poço que contém a gota Syto9. Deixar a lamínula em 1 mL de PBS facilita a remoção do poço e ajuda a lavar os grânulos não associados do biofilme.
- (Se estiver usando uma lamínula) Sele cuidadosamente a lamínula com esmalte sem deslizar ou pressionar a lamínula, o que pode causar tensão superficial que interromperá o movimento em biofilmes mais viscosos e menos rígidos.
- (Se estiver usando uma lamínula) Deixe o esmalte secar. Limpe cuidadosamente a superfície externa da lamínula com etanol a 70%. Limpe sem aplicar pressão na lamínula pelos motivos mencionados acima.
- Imagem confocal
NOTA: Nestes estudos, use um microscópio confocal de varredura a laser de imagem espectral invertida equipado com um sistema confocal TCS com a objetiva de 63x. O osciloscópio será usado para gerar um vídeo 4D (Figura 1). O biofilme 3D será composto por fatias de Z capturadas em etapas de 0,5 μm através de biofilmes com 18-20 μm de espessura, gerando 36-40 fatias de Z. Cada biofilme 3D levará de 50 a 60 segundos para ser capturado. As fatias juntas formarão um quadro, que pode ser visualizado como um biofilme 3D. Este processo será repetido 20 vezes para gerar o vídeo de lapso de tempo 4D para um tempo total de rastreamento de 18 a 20 minutos.- Defina o osciloscópio para capturar fluoróforos. Nesses estudos, excite Syto9 (coloração de DNA bacteriano) com um laser de 488 nm e meça a emissão de 495 a 540 nm (o Leica Sp5 é um microscópio de imagem espectral). Excite contas carmesim (vermelhas) com um laser de 633 nm e meça a emissão de 650 nm a 700 nm. Essas configurações de fluoróforo podem ser ajustadas para capturar quaisquer fluoróforos desejados.
- Escolha o modo de imagem xyzt.
- Identifique uma região do biofilme com uma mistura de regiões de maior e menor densidade para capturar diferenças nas propriedades viscoelásticas em regiões espessas e finas do biofilme.
- Defina uma pilha Z de 18-20 μm de espessura. Em um biofilme montado em lamínula, evite que a parte superior e inferior do biofilme se toquem no vidro para evitar artefatos de retenção de esferas (consulte a discussão).
- Ajuste o ganho e o deslocamento para usar toda a faixa dinâmica da intensidade no ponto mais brilhante do biofilme. Isso minimiza a sobreposição de sinal das camadas inferiores do biofilme.
- Defina a espessura da fatia para 0,5 μm. Isso permite imagens rápidas sem a perda de informações do cordão.
- Defina a resolução para 512 x 512 (0,48 μm). Isso permite imagens rápidas, mas gera imagens com resolução suficiente para ver a estrutura do biofilme e os detalhes do movimento do grânulo.
- Minimize o tempo de imagem.
- Defina para capturar 20 pilhas.
- Salve como .lif (ou arquivo confocal semelhante). O filme 4D pode ser gerado em ImageJ.
3. Gerando o vídeo de biofilme 4D com ImageJ
- Abra o arquivo .lif no ImageJ com as seguintes configurações: Exibir pilha com Hyperstack, Ordem de pilha XYCZT, modo de cor: colorido, dimensionamento automático marcado. Em seguida, pressione OK.
- Selecione Imagem > Cor > Dividir canais.
- Selecione Imagem > Cor > Mesclar canais > Criar composição > OK.
- Selecione Plugins > visualizador 3D > Canais desmarque azul > OK.
- Selecione Editar > Mostrar conteúdo marcada > Mostrar caixa delimitadora marcada.
- Use o clique do mouse e segure para selecionar a imagem. Em seguida, gire a imagem enquanto mantém pressionado o botão de clique. Gire a imagem para que a parte inferior do biofilme fique na parte inferior e o ângulo suporte a visualização das contas. Em seguida, solte a imagem.
- Pressione o botão vermelho Gravar na parte inferior da janela para gravar um vídeo.
- Salve como arquivo .avi usando jpeg como compactação.
4. Geração de dados de trajetória
- Instale a ferramenta de código aberto ImageJ (https://imagej.net/Fiji) e o plug-in de rastreamento de partículas Mosaic (https://imagej.net/MOSAICsuite).
- Importe o arquivo .lif que contém os dois canais para grânulos e bactérias. Divida os canais e salve os arquivos separadamente.
- No ImageJ, armazene o tamanho do voxel da imagem (dimensões x, y e z) e o tamanho do intervalo de tempo em um arquivo de texto.
- No ImageJ, vá para Plugins | Mosaic e inicie o Particle Tracker 2D/3D.
- Insira os seguintes parâmetros: Raio: 3, Corte: 0,003, Por/Abs: 0,12, Faixa de link: 2, Deslocamento: 10,00 com a dinâmica definida em Brownian. Por favor, veja os resultados para a lógica por trás da escolha de Brownian nesses experimentos.
- Gere trajetórias e exporte a lista de trajetórias como arquivos .csv.
5. Analisando trajetórias
- Instale o VRL Studio (https://vrl-studio.mihosoft.eu)
- Baixe o projeto Biofilm (https:// neurobox3d.github.io/Biofilm/) e lance no VRL Studio
- Carregar o arquivo de trajetória em ImportData
- Especifique o tamanho do pixel x, y e z (use ImageJ para localizar esses valores) em ProcessTrajectories.
- Especifique o intervalo de quadros em ComputeVelocity (use ImageJ para localizar esse valor).
- Carregue o arquivo tiff bacteriano (consulte a etapa 3.2) em Comdensity.
- Defina caminhos de saída para dados de velocidade em SaveVelocityDataToFile e dados de trajetória em SaveTrajectoryDataToFile.
- Invoque SaveVelocityDataToFile e SaveTrajectoryDataToFile.
- Importe dados para o Excel para análise. Esses dados incluirão comprimentos de trajetória, vida útil da trajetória, dimensões e volumes da caixa delimitadora da trajetória, velocidades médias do cordão e variações. A análise calcula variáveis ponderadas usando o canal para bactérias marcadas com Syto9 para calcular as densidades celulares locais (dentro de caixas delimitadoras de trajetória dada). A análise resulta no cálculo de velocidades e variâncias médias ponderadas, bem como médias ponderadas e variâncias da variância da caixa delimitadora.
Resultados
Este método foi usado para testar a hipótese de que o curli pode conferir rigidez a E. coli e S. Biofilmes de Typhimurium, reduzindo o movimento do grânulo durante experimentos de microscopia confocal. A caixa de ferramentas atual foi usada para comparar as propriedades do material da cepa OG1RF do tipo comensal de Enterococcus faecalis com o sorotipo Typhimurium, E. coli e seus respectivos mutantes curli isogênicos (Figura 1B e Vídeo Suplementar 1, Vídeo Suplementar 2, Vídeo Suplementar 3, Vídeo Suplementar 4, Vídeo Suplementar 5, Vídeo Suplementar 6). As propriedades do material de biofilme podem diferir em relação à rigidez (por exemplo, ondulação ligada ao eDNA) ou interações eletrostáticas e hidrofóbicas entre as esferas carregadas negativamente e as células de biofilme e materiais de matriz, bem como a densidade celular.
Reprodutibilidade
A caixa de ferramentas Biofilm foi programada em Groovy30 e Java31 dentro do VRL-Studio32 , permitindo um design de fluxo de trabalho modular com geração automática de interface de usuário (UI) de todos os componentes computacionais. Isso permitiu um fluxo de trabalho automatizado, removendo o viés não intencional induzido pelo experimentador ao analisar os resultados.
Uso de MSD para confirmar o tipo de movimento nos biofilmes
Para análise de trajetórias usando o Particle Tracker 2D/3D, diferentes configurações dinâmicas para análise de diferentes tipos de movimento do cordão estão disponíveis. Para esses estudos, a configuração "movimento browniano" (ou seja, movimento impulsionado por difusão) foi escolhida, uma vez que E. faecalis é uma bactéria imóvel, E. coli e Salmonella não expressam flagelos em biofilmes, e os experimentos foram realizados em um sistema fechado na ausência de fluxo. Essa configuração pode ser validada ainda mais pelos deslocamentos quadrados médios calculados (MSD) dos grânulos. Usando a definição 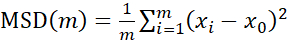 em que m é o número de segmentos de trajetória, a mudança do MSD ao longo de cada trajetória pode ser calculada. As trajetórias lineares indicam movimento difusivo do cordão (Figura 2A). Usando o ajuste quadrático de mínimos quadrados, o padrão médio de movimento de todas as contas no biofilme foi calculado, mostrando a ordem linear dominante e validando a difusão passiva como força motriz (Figura 2A-2F).
em que m é o número de segmentos de trajetória, a mudança do MSD ao longo de cada trajetória pode ser calculada. As trajetórias lineares indicam movimento difusivo do cordão (Figura 2A). Usando o ajuste quadrático de mínimos quadrados, o padrão médio de movimento de todas as contas no biofilme foi calculado, mostrando a ordem linear dominante e validando a difusão passiva como força motriz (Figura 2A-2F).
Análise de caixa delimitadora.
A caixa de ferramentas usa o ImageJ Mosaic e o Particle Tracker 2D/3D para gerar trajetórias (Etapa 4) e, em seguida, usando o pipeline automatizado de análise de biofilme, gera dados importantes sobre as trajetórias do grânulo que podem ser usados para comparar as propriedades do material de biofilme. O volume da caixa delimitadora em μm3 foi medido construindo a caixa mínima que contém uma trajetória e medindo seu volume (Figura 3).
Os biofilmes de E. faecalis têm mais movimento de esferas com valores de caixa delimitadora de 1-6000 μm3 (Figura 3B, 3C e 3D). Os resultados confirmam que o movimento visto em uma lamínula de vidro montada em uma lâmina revestida com um poço revestido de aproximadamente 25 μm (Figura 3C) versus biofilmes cultivados no fundo de poços de vidro óptico e fotografados diretamente (Figura 3D) produzem resultados equivalentes com poucas diferenças. A única diferença era que perto do topo da lamínula montada de biofilmes de E. faecalis trajetórias estáveis com vida útil superior a 10 minutos, mas ao mesmo tempo pequenas caixas delimitadoras podiam ser registradas, enquanto na placa inferior óptica um número seleto de contas com maior mobilidade podia ser registrado. Em conjunto, isso sugere que a montagem da lâmina de vidro pode ter alterado a tensão superficial do sistema na parte superior do biofilme contra a lâmina no biofilme de lamínula montado, o que acabou diminuindo a mobilidade de alguns grânulos em regiões de biofilme menos viscosas (Figura 3B, 3C, 3D e 3I). As trajetórias que se enquadram nessa categoria foram uma porcentagem muito pequena e, mesmo com esse pequeno número de contas presas, o MSD médio de E. faecalis em uma lamínula montada foi ligeiramente maior do que o MSD calculado a partir de um biofilme de placa de fundo óptico (Figura 3).
S. As trajetórias de contas de Typhimurium e E. coli tiveram volumes de caixa delimitadora menores de 0-10 μm3 (Figura 3A, 3B, 3E e 3F), em comparação com mutantes curli isogênicos com caixas delimitadoras de 1-6000 μm3 para E. coli e 1-5000 μm3 para S. Tifinoúrio (Figura 3A, 3B, 3F e 3H), demonstrando maior mobilidade do grânulo. Esses resultados sugeriram que a presença do amiloide se correlacionou com o aumento da rigidez nos biofilmes e foi consistente com a falta de movimento notável do biofilme nos vídeos. Os volumes da caixa delimitadora foram consistentemente pequenos (0-10 μm3), mesmo em regiões de baixa densidade do biofilme. Esta observação é consistente com observações anteriores de que o curli pode estar presente em regiões de baixa densidade celular do biofilme10.
Não foi possível comparar o comportamento dos biofilmes de Enterobacteriaceae em placas ópticas de fundo porque eles crescem como películas na interface ar-líquido (Etapa 1.2.2). Ao usar uma lamínula, a película é anexada à lamínula na interface e quando a lamínula foi removida, a película foi colocada na lamínula, criando uma única superfície de imagem. Em uma placa de fundo óptico cultivada em uma inclinação, a imagem foi feita com o líquido ainda no poço. Isso significa que a película ainda está flutuando acima do fundo óptico e faz com que a película saia da profundidade de trabalho de um osciloscópio invertido, como o Leica Sp5. A remoção de meio suficiente para trazer o biofilme para a profundidade de trabalho do microscópio fez com que a amostra secasse durante o processo de imagem de 20 minutos.
No geral, os gráficos confirmam as observações visuais nos filmes suplementares e são consistentes com as diferenças de MSD observadas (Figura 3I e 3J).
Vida útil da trajetória
A vida útil da trajetória foi medida como o número de quadros consecutivos em que um cordão foi registrado (Figura 3).
Nos biofilmes de E. faecalis mais viscosos e fluidos, todos os grânulos tiveram uma vida útil de trajetória inferior a 10 minutos e a maioria das trajetórias variou entre 2-5 minutos para biofilmes de E. faecalis . No entanto, grânulos com vida útil de trajetória curta registrada podem ser localizados por inspeção visual em biofilmes de E. faecalis durante a janela de tempo total da imagem (Vídeo Suplementar 1 e 2). Assim, é possível que as contas se movam ao longo de uma trajetória registrada, desassociando-se intermitentemente do biofilme e encerrando uma trajetória, e reassociando-se ao biofilme, momento em que uma nova trajetória é iniciada. Em última análise, isso levaria a uma vida útil de trajetória curta sob a presença contínua de grânulos no biofilme. É importante notar que, usando essa técnica, a vida útil da trajetória, especialmente em um biofilme viscoso, tende a subestimar o tempo total que um grânulo está associado ao biofilme.
Em S. Os biofilmes de Typhimurium, que tinham volumes de caixa delimitadores menores, a maioria dos grânulos (cerca de 80%) tinha longa vida útil de 16-20 quadros, correspondendo a cerca de 15-20 minutos em tempo real (Figura 3A, 3G e 3H). Ao contrário destes, os biofilmes mutantes de curli isogênicos carregavam mais esferas móveis com volumes de caixa delimitadora variando entre 1-6000 μm3 (E. coli) e 1-5000 μm3 (S. Timorfo) (Figura 3A, 3B, 3F e 3H). Em contraste com os biofilmes de E. faecalis com trajetórias de >70% com volumes de caixa delimitadora maiores que 10 μm3, no entanto, os biofilmes de espécies de Enterobacteriaceae registraram apenas 30% de trajetórias de grânulos com volumes de caixa delimitadora acima de 10 μm3. Embora a vida útil geral da trajetória do grânulo fosse menor em biofilmes mutantes ondulados, algumas trajetórias refletiam movimento substancial do grânulo e longa vida útil da trajetória (Figura 3H). Essa observação pode indicar que essa variabilidade pode corresponder a propriedades variáveis do material de biofilme, como viscoelasticidade, e/ou alterações químicas na superfície das partículas, como carga.
Análise dos comprimentos e velocidades da trajetória do cordão
O comprimento da trajetória é uma medida da distância percorrida pelos grânulos em μm. Esta medição é consistente com a velocidade do movimento do cordão em μm / s. Consistente com os maiores volumes de caixa delimitadora, as esferas em biofilmes de E. faecalis tiveram trajetórias 10 vezes mais longas, 5-20 μm, contra <4 μm em biofilmes contendo curli. Consistente com as trajetórias mais curtas (Figura 4A). Os grânulos de E. faecalis mediram velocidades até 15x maiores, com a maioria dos grânulos tendo velocidades na faixa de 0,01-0,15 μm / s versus velocidades <0,006 μm / s (Figura 4B). No entanto, os biofilmes mutantes curli mediram velocidades gerais mais baixas e trajetórias mais curtas em comparação com os biofilmes de E. faecalis , mas trajetórias mais longas e velocidades mais altas do que as cepas parentais contendo curli ( Figura 4A e 4B ).
Destaca-se o fato de que a estrutura fibrilar em forma de rede do curli30 pode afetar a mobilidade de forma anisotrópica, reduzindo o movimento no plano xy e permitindo maior mobilidade na direção z (Figura 2G). O grande pool de trajetória (aproximadamente 800) pertencente a cerca de 50 contas únicas em biofilmes contendo ondulações seria consistente com as limitações do Mosaic Particle Tracking, contando cada uma dessas contas em movimento rápido como uma única conta em x, y e z. Pesquisas adicionais e desenvolvimento de software serão necessários para confirmar essa observação.
Análise da dependência do movimento do grânulo na densidade celular
A dependência do movimento do cordão na densidade celular foi determinada usando velocidades e variâncias médias ponderadas, bem como médias/médias ponderadas e variâncias dos volumes das caixas delimitadoras. O segundo canal de imagem para bactérias marcadas com Syto9 foi usado para calcular as densidades celulares locais no cálculo das velocidades ponderadas. A densidade celular foi calculada calculando a média dos dados de voxel Syto9 sobre a caixa delimitadora de cada borda da trajetória (Figura 5, à direita). Assim, a velocidade do cordão pode ser ponderada pelas densidades celulares (locais) de borda. Existem vários tipos de manchas que podem ser usadas para visualizar bactérias, incluindo manchas na parede celular, membranas e conteúdo de DNA. Para determinar a densidade celular, o Syto9 foi escolhido porque fornece o sinal mais consistente, independentemente de qual fatia Z óptica está sendo visualizada. As colorações de envelope (parede celular e membrana) darão um sinal diferente dependendo da posição da fatia Z. Se a fatia Z incluir a parte superior ou inferior da célula, o sinal será mais forte do que se a fatia Z estiver no meio da célula, onde apenas o contorno da célula está manchado.
As trajetórias do cordão do canal vermelho foram rastreadas por 20 quadros, onde as trajetórias individuais tiveram uma vida útil mínima de 2 quadros e um máximo de 20 quadros com 19 segmentos de trajetória conectando os quadros (Figura 5). Para estudar a dependência da densidade celular da mobilidade do grânulo, a intensidade da GFP por voxel (cada uma das medições individuais na imagem do voxel 512x512) foi determinada. A densidade celular em torno de cada segmento da trajetória do grânulo foi calculada como a densidade média local na caixa delimitadora do segmento.
Para alguns biofilmes, a dependência de densidade estatisticamente significativa pode ser documentada (Figura 6), mais proeminentemente para biofilmes de E. faecalis que foram cultivados em uma lamínula de vidro e invertidos em uma lâmina de vários poços (Figura 6A). Pelo contrário, os biofilmes de E. faecalis que foram cultivados no fundo de uma placa de 96 poços (Figura 6B) não mostraram dependência de densidade. Em conclusão, isso sugere que os biofilmes de E. faecalis altamente fluidos podem ser ligeiramente comprimidos devido à montagem em uma lâmina de vários poços, o que é consistente com uma redução no número de grânulos que se movem mais rapidamente e aqueles com pequenos volumes de caixa delimitadora que ficam presos no topo do biofilme contra a lâmina de vidro (Figura 3C vs Figura 3D). Tanto os biofilmes de Salmonella e E. coli (Figura 6C e 6D) quanto seus mutantes isogênicos (Figura 6E e 6F) mostraram menor ou nenhuma dependência de densidade celular.
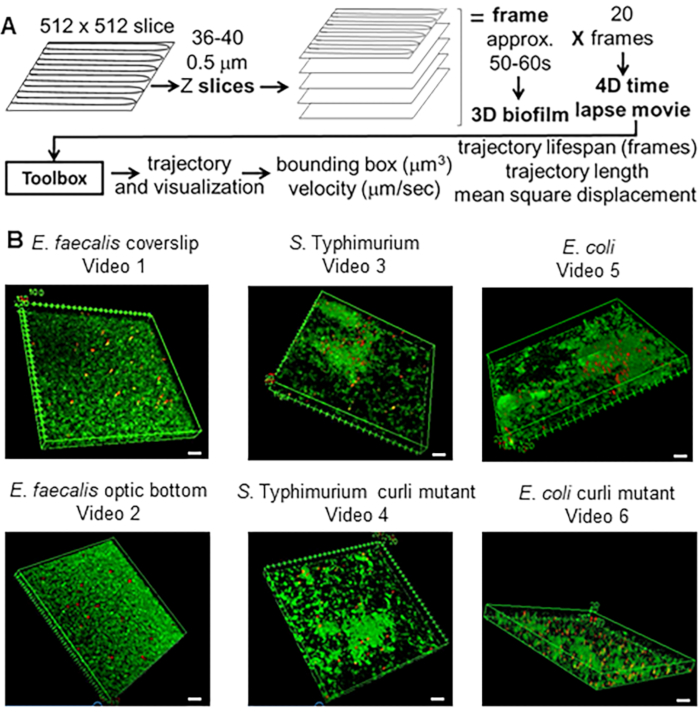
Figura 1. Pipeline de imagem e análise (etapas 2-4) (A) Os biofilmes são visualizados conforme descrito em 2.2. Usando os biofilmes em imagem (ver 3), as trajetórias das contas foram geradas conforme descrito em 4. Usando as trajetórias, os dados relevantes foram calculados com a caixa de ferramentas de análise (ver 5) (B) Os biofilmes foram cultivados conforme descrito na etapa 1 em lamínulas (E. faecalis, Vídeo 1), S. Tifimúrio (Vídeo 3), E. coli (Vídeo 5) e mutantes curli isogênicos (Vídeo 4, Vídeo 6) ou em uma placa de fundo óptico de 96 poços (E. faecalis bottom, Vídeo 2). A barra branca da escala é de 20 mm. Esta figura é reproduzida com permissão de (31). Clique aqui para ver uma versão maior desta figura.
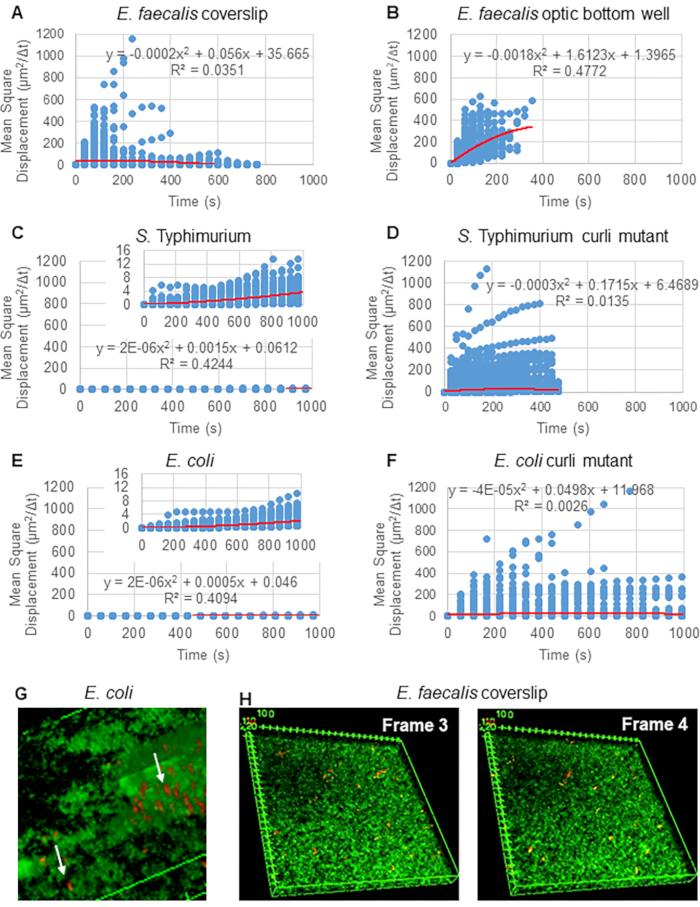
Figura 2. O movimento do grânulo nos biofilmes foi determinado como sendo de natureza difusiva (movimento browniano, consulte a etapa 5) (AF) Os dados MSD mostram comportamento linear (linha vermelha) para validar o movimento browniano. Os biofilmes foram cultivados de acordo com a etapa 1 (G) Exemplo de movimento elíptico do grânulo observado em biofilmes de E. coli e S. Typhimurium retirados de um quadro do ensaio de biofilme 4D de E. coli (H) Exemplo de grandes mudanças nos padrões de grânulos entre os quadros 3 e 4 retirados do poço de fundo óptico de E. faecalis . Observe que o próprio biofilme provoca algum fluxo (Vídeo 1 e 2), o que faz parecer que os quadros 3 e 4 são orientados de forma diferente. Esta figura é reproduzida com permissão de (31). Clique aqui para ver uma versão maior desta figura.
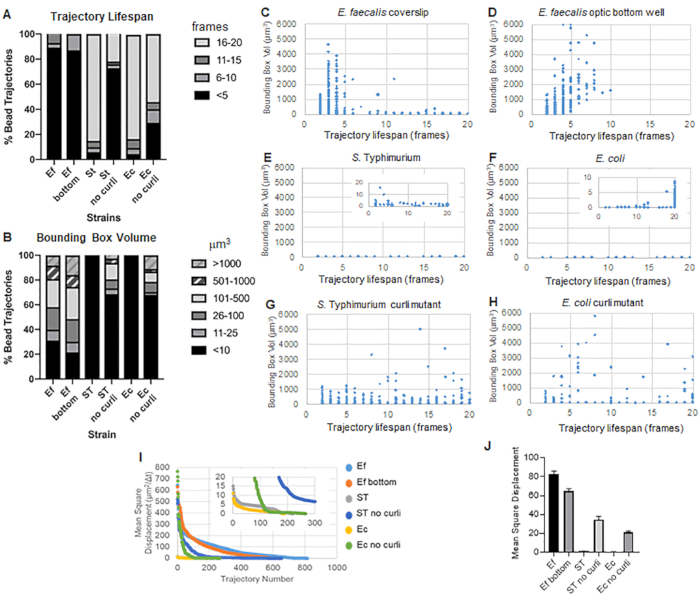
Figura 3. Análise de diferenças de rigidez usando caixas delimitadoras e tempo de vida da trajetória. Os biofilmes foram cultivados conforme descrito na etapa 1 em lamínulas (E. faecalis, Vídeo 1), S. Tifimúrio (Vídeo 3), E. coli (Vídeo 5) e mutantes curli isogênicos (Vídeo 4, Vídeo 6) ou em uma placa de fundo óptico de 96 poços (E. faecalis bottom, Vídeo 2). A vida útil da trajetória é apresentada em % das trajetórias totais do grânulo (A) e gráficos de dispersão (CH), juntamente com os volumes da caixa delimitadora (calculados na etapa 5) (I) Comparação dos MSDs do grânulo nos diferentes biofilmes (H) MSDs médios de cada tipo de biofilme. As barras indicam o intervalo de confiança de 95% dos dados. Esta figura é reproduzida com permissão de (31). Clique aqui para ver uma versão maior desta figura.
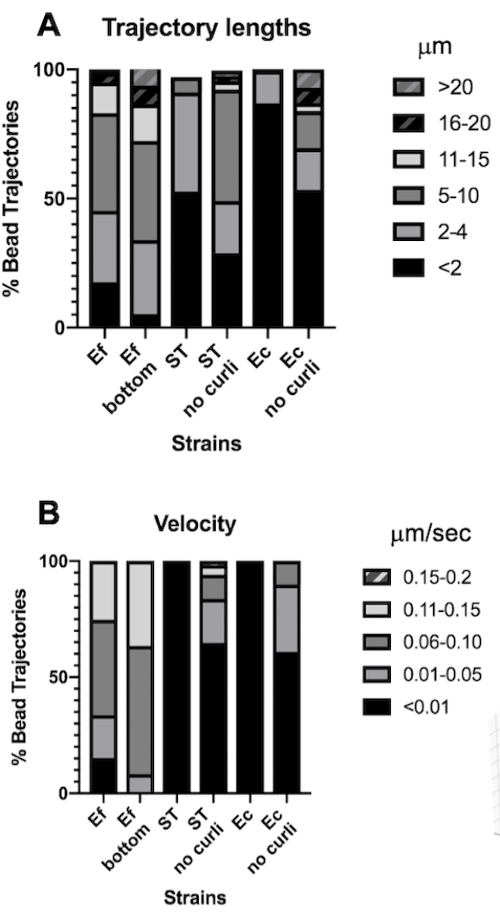
Figura 4. Análise das diferenças no comprimento da trajetória e na velocidade do cordão. Os biofilmes foram cultivados conforme descrito na etapa 1. Os comprimentos das trajetórias são mostrados em μm e são apresentados como % do total de trajetórias do cordão (A). A velocidade é mostrada em μm/s e apresentada como % das trajetórias totais do cordão (B). Esta figura é adaptada com permissão de (31). Clique aqui para ver uma versão maior desta figura.
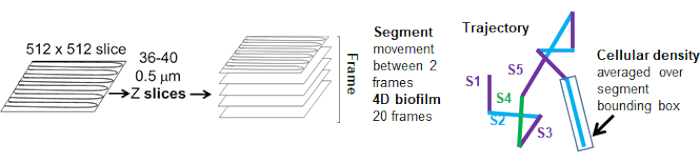
Figura 5. Esboço da análise do segmento de trajetória. Esta figura é adaptada com permissão de (31). Clique aqui para ver uma versão maior desta figura.
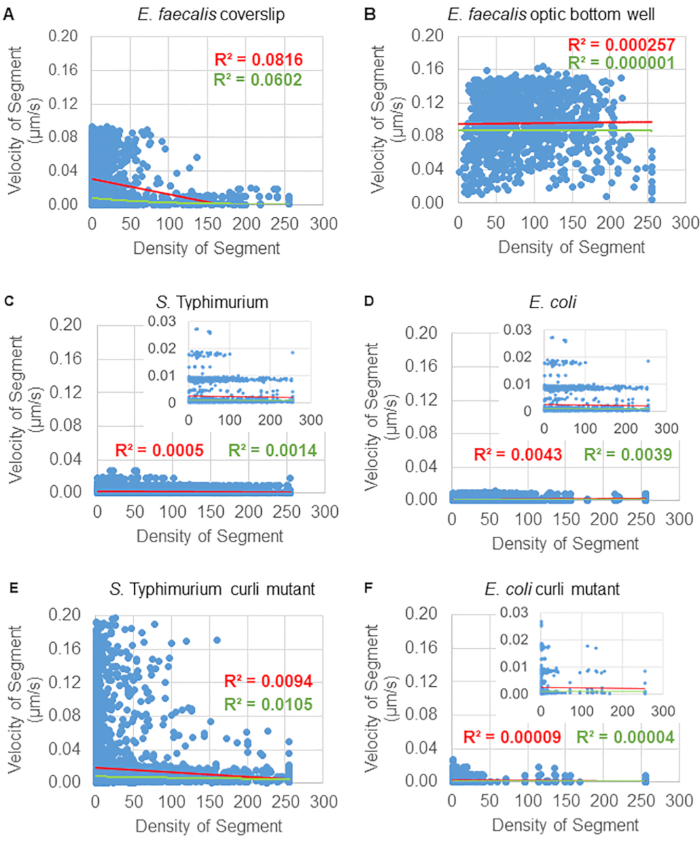
Figura 6. Estudando o efeito da densidade do biofilme na velocidade do grânulo usando o pipeline de análise de biofilme (etapa 5). Os resultados indicaram que a velocidade do grânulo não depende exclusivamente da densidade celular. Os biofilmes foram cultivados conforme descrito na etapa 1. As trajetórias foram analisadas na escala de segmentos de trajetória individuais (Figura 5). Para cada segmento, a velocidade do grânulo em μm / s foi plotada em relação à densidade celular da caixa delimitadora (GFP média por voxel dentro da caixa delimitadora). A linha vermelha mostra a regressão linear e a linha verde o traço de regressão exponencial. Esta figura é reproduzida com permissão de (31). Clique aqui para ver uma versão maior desta figura.
Vídeo suplementar 1. Vídeo 4D de biofilme E. faecalis OG1RF de 24 horas cultivado em uma lamínula de vidro óptico de 1,5 de espessura. O vídeo de lapso de tempo 4D foi gerado usando um microscópio com resolução de 512x512. Uma série Z de 40 imagens foi gerada pela imagem de uma região de aproximadamente 20 μm de espessura de um biofilme em etapas de 0,5 μm. Cada série Z tinha um quadro e exigia de 50 a 60 s para capturar. Uma série de 20 quadros contíguos foram capturados para produzir um vídeo 4D. A reprodução do vídeo é de aproximadamente 120x. O vídeo é representativo de pelo menos 6 experimentos independentes. Clique aqui para baixar este vídeo.
Vídeo suplementar 2. Vídeo 4D de um biofilme de E. faecalis OG1RF de 24 horas cultivado em uma placa de fundo óptico de 96 poços. O vídeo de lapso de tempo 4D foi gerado usando um microscópio com resolução de 512x512. Uma série Z de 40 imagens foi gerada pela imagem de uma região de aproximadamente 20 μm de espessura de um biofilme em etapas de 0,5 μm. Cada série Z tinha um quadro e exigia de 50 a 60 s para capturar. Uma série de 20 quadros contíguos foram capturados para produzir um vídeo 4D. A reprodução do vídeo é de aproximadamente 120x. O vídeo é representativo de 3 experimentos independentes.Clique aqui para fazer o download vídeo.
Vídeo suplementar 3. Vídeo 4D de um biofilme ATCC 14028 de Typhimurium do sorotipo Salmonella enterica cultivado em lamínulas de vidro óptico de 1,5 de espessura por 6-7 dias. O vídeo de lapso de tempo 4D foi gerado usando um microscópio com resolução de 512x512. Uma série Z de 40 imagens foi gerada pela imagem de uma região de aproximadamente 20 μm de espessura de um biofilme em etapas de 0,5 μm. Cada série Z tinha um quadro e exigia de 50 a 60 s para capturar. Uma série de 20 quadros contíguos foram capturados para produzir um vídeo 4D. A reprodução do vídeo é de aproximadamente 120x. O vídeo é representativo de 3 experimentos independentes.Clique aqui para fazer o download vídeo.
Vídeo suplementar 4. Vídeo 4D de biofilmes de Salmonella enterica sorotipo Typhimurium ATCC 14028 curli (csgBA) mutante foram cultivados em lamínulas de vidro óptico de 1,5 espessura por 6-7 dias. O vídeo de lapso de tempo 4D foi gerado usando um microscópio com resolução de 512x512. Uma série Z de 40 imagens foi gerada pela imagem de uma região de aproximadamente 20 μm de espessura de um biofilme em etapas de 0,5 μm. Cada série Z tinha um quadro e exigia de 50 a 60 s para capturar. Uma série de 20 quadros contíguos foram capturados para produzir um vídeo 4D. A reprodução do vídeo é de aproximadamente 120x. O vídeo é representativo de 3 experimentos independentes.Clique aqui para fazer o download vídeo.
Vídeo suplementar 5. Vídeo 4D de E. coli UTI89 cultivada em lamínulas de vidro óptico de 1,5 de espessura por 6-7 dias. O vídeo de lapso de tempo 4D foi gerado usando um microscópio com resolução de 512x512. Uma série Z de 40 imagens foi gerada pela imagem de uma região de aproximadamente 20 μm de espessura de um biofilme em etapas de 0,5 μm. Cada série Z tinha um quadro e exigia de 50 a 60 s para capturar. Uma série de 20 quadros contíguos foram capturados para produzir um vídeo 4D. A reprodução do vídeo é de aproximadamente 120x. O vídeo é representativo de 3 experimentos independentes. Clique aqui para fazer o download vídeo.
Vídeo suplementar 6. Mutante de E. coli UTI89 curli (csgBA) de vídeo 4D cultivado em lamínulas de vidro óptico de 1,5 de espessura por 6-7 dias. O vídeo de lapso de tempo 4D foi gerado usando um microscópio com resolução de 512x512. Uma série Z de 40 imagens foi gerada pela imagem de uma região de aproximadamente 20 μm de espessura de um biofilme em etapas de 0,5 μm. Cada série Z tinha um quadro e exigia de 50 a 60 s para capturar. Uma série de 20 quadros contíguos foram capturados para produzir um vídeo 4D. A reprodução do vídeo é de aproximadamente 120x. O vídeo é representativo de 3 experimentos independentes.Clique aqui para fazer o download vídeo.
Discussão
Etapas críticas e solução de problemas
O maior desafio dessa técnica é usar uma lamínula montada com um biofilme muito viscoso como E. faecalis. A lamínula precisa ser colocada com cuidado e precisão na lâmina de poços múltiplos sem reposicioná-la. Durante a etapa de vedação, é necessário ter cuidado para evitar empurrar a lamínula para baixo ou empurrá-la/deslizá-la acidentalmente pela superfície da corrediça. Qualquer movimento ou pressão pode criar tensão superficial e bloquear o movimento de um biofilme viscoso. Se possível, comparar as propriedades do material de biofilme por meio de imagens de um biofilme em um poço de fundo óptico com uma montagem de lamínula permitirá a avaliação da técnica. Quando executada corretamente, uma montagem de lamínula se assemelhava muito a um biofilme em uma placa inferior óptica para E. faecalis.
Além disso, ao usar uma lamínula montada, deve-se evitar imagens das interfaces do biofilme com a lamínula na parte inferior ou a lâmina na parte superior. Ao usar um endoscópio invertido, com a lamínula na parte inferior, pode haver grânulos presos na base do biofilme contra a lamínula. Essas esferas passam pelo biofilme e ficam presas contra a lamínula, mesmo após uma lavagem suave. Eles têm coordenadas x, y e z de 0 e coordenadas de caixa delimitadora de 0. No entanto, para certas aplicações, como examinar a integridade do biofilme após o tratamento, esses pontos de dados podem ser usados como uma ferramenta. A capacidade dos grânulos de penetrar através de uma região espessa de biofilme até o fundo da lamínula pode ser usada para avaliar a integridade do biofilme após o tratamento (manuscrito em preparação em colaboração com o laboratório Tükel). No topo do biofilme, em um biofilme viscoso como E. faecalis , tínhamos algumas evidências de compactação imposta pela lamínula. Isso limitou o movimento de alguns grânulos na interface da lâmina de vidro e pode ter introduzido alguma dependência de densidade na análise do movimento do grânulo.
As etapas de lavagem foram necessárias para os biofilmes, pois o meio de crescimento possui forte autofluorescência no canal verde. Optamos por usar contas em excesso e remover contas não associadas lavando para maximizar as contas associadas para obter a caracterização mais precisa das regiões observadas.
O número de grânulos e lavagens necessários para obter os conjuntos de dados desejados precisa ser determinado empiricamente. A presença de muitas esferas em um biofilme gera conjuntos de dados impossivelmente grandes que são difíceis de analisar. A presença de poucas esferas não gera uma amostragem completa dos ambientes de biofilme. No entanto, o controle do número de grânulos adicionados (2x107 grânulos em 1 mL de PBS) e o uso de etapas de lavagem resultaram em um número relativamente consistente de grânulos (40-140) associados ao biofilme, dependendo de sua estrutura, arranjo espacial e composição.
Ao estudar biofilmes com regiões viscosas e rígidas mistas, as contas podem ficar presas nas regiões rígidas ao longo do tempo. Nesse caso, a imagem precisa ser iniciada imediatamente após a adição das contas. Isso geralmente não pode ser feito usando lamínulas, mas requer placas de fundo óptico ou células de fluxo, onde a imagem pode ser feita imediatamente após a adição dos grânulos e da(s) etapa(s) de lavagem.
Modificações e aplicações futuras
Uso de dispositivos microfluídicos. Em nossos estudos, as condições ótimas estabelecidas para o estudo dos biofilmes de Enterobacteriaceae exigiram o crescimento do biofilme como uma película na interface ar-líquido. Isso limitou o uso de placas de fundo óptico e dispositivos microfluídicos nos estudos. No entanto, quando as condições de formação de biofilme permitem, os biofilmes podem ser cultivados em câmaras microfluídicas ou células de fluxo. Os biofilmes podem então ser lavados e as esferas introduzidas através do dispositivo microfluídico com o mínimo de interrupção do biofilme.
Adição de grânulos durante o crescimento do biofilme. Optamos por adicionar o excesso de grânulos aos biofilmes e, em seguida, remover os grânulos não associados por lavagem suave para otimizar o número de grânulos presentes durante a análise. Nos biofilmes viscosos de E. faecalis , é possível que as esferas se desassociem e se reassociem durante o tempo de imagem de 20 minutos. Se um pequeno número de grânulos for adicionado em momentos diferentes durante o crescimento do biofilme, pode ser possível prender os grânulos no biofilme, permitindo uma caracterização mais precisa do movimento do biofilme em biofilmes mais viscosos.
Escolha da região a ser fotografada. Para estudos sobre as propriedades do material, é melhor escolher regiões grossas e finas do biofilme. No entanto, ao estudar as mudanças nas propriedades do material de um biofilme tratado, regiões confluentes espessas podem ser visualizadas para determinar mudanças nas propriedades viscoelásticas e penetração de esferas nessas regiões. Nesse caso, procurar contas que penetraram no biofilme e acabaram presas contra a lamínula é uma medida útil de ruptura do biofilme.
Imagem sob fluxo. Usando células de fluxo de vidro óptico ou dispositivos microfluídicos, o movimento de grânulos ou bactérias em um biofilme sob fluxo pode ser visualizado. Isso pode ser feito de diferentes maneiras. Isso pode ser feito por injeção de grânulos em toda a câmara, seguida de breve incubação para permitir a associação dos grânulos com o biofilme. Os grânulos não associados podem ser removidos por lavagem e o biofilme fotografado com ou sem fluxo. Por outro lado, um pequeno número de esferas pode ser introduzido em um lado da câmara e seu movimento através e dentro do biofilme pode ser rastreado sob fluxo. Ao usar o fluxo, será necessário ter cuidado ao escolher as configurações de rastreamento de contas do mosaico (Etapa 4.5). Nos estudos atuais, a configuração da dinâmica foi browniana. Os cálculos do MSD confirmaram que o movimento provavelmente seria difusivo, tornando o browniano a configuração apropriada.
Coloração da matriz. Nos estudos atuais, a coloração com Syto9 examina a densidade celular e não a densidade da estrutura do biofilme. Por exemplo, a presença de amilóides provavelmente aumenta a densidade do material da matriz do biofilme. A dependência do movimento da densidade amilóide pode ser determinada usando colorações de matriz fluorescente em vez de Syto9.
Bactérias marcadas com fluorescência. Bactérias marcadas com fluorescência podem ser usadas para rastrear o movimento de bactérias exógenas através de biofilmes (por exemplo, bactérias contendo plasmídeos). O desafio com bactérias marcadas com fluorescência, como Enterococcus, é que elas formam cadeias únicas, diplococas e curtas, o que complica a capacidade de rastrear com precisão as bactérias. Esse processo seria mais fácil se as bactérias tivessem uma morfologia unicelular.
Limitações
Limitações na visualização e costura da trajetória.
Uma limitação do método é a visualização e costura da trajetória. As trajetórias reconstruídas e analisadas consistem em coordenadas de pontos x, y, z, onde os pontos subsequentes definem o caminho linear entre esses pontos. A visualização de tais trajetórias lineares por partes pode ser alcançada por várias ferramentas. Uma abordagem foi usar notebooks Python e Jupyter junto com os plug-ins Python, Pandas e Matplotlib. Embora tenha sido possível visualizar trajetórias individuais existentes no artigo do Journal of Bacteriology, onde essa técnica foi publicada originalmente34, ainda havia limitações significativas que estão sendo abordadas em pesquisas futuras.
Atualmente, o número de trajetórias reconstruídas é maior do que o número de grânulos no biofilme, o que significa que várias trajetórias podem corresponder a um grânulo. Isso pode ser causado por um sinal confocal fraco em um quadro, onde o Mosaic encerrará uma trajetória e iniciará um segundo. Isso pode ser registrado como várias trajetórias mais curtas para uma conta, especialmente em biofilmes menos viscosos. Outra causa para o grande número de trajetórias é a falta de costura de trajetória. Especialmente em biofilmes de poço de fundo óptico de E. faecalis , as esferas permanecem visualmente associadas ao biofilme durante a imagem (Vídeo Suplementar 2). No entanto, não houve trajetórias maiores que 10 e mais de 90% das trajetórias tiveram uma vida útil de 5 quadros ou menos (Figura 3D). Se o software for usado para analisar apenas trajetórias acima de um comprimento definido (por exemplo, ao rastrear células capazes de transferir plasmídeos), trajetórias mais curtas podem ser removidas automaticamente do conjunto de dados. No entanto, existem outros propósitos para os quais costurar as trajetórias pode ser muito importante. Finalmente, a incapacidade de rastrear o movimento rápido do grânulo como uma única trajetória pode resultar em mais trajetórias nos biofilmes de Enterobacteriaceae devido ao movimento rápido na direção Z, resultando em grânulos de formato elíptico ( Figura 2G ). A possibilidade de trajetórias de sutura interrompidas por movimentos anisotrópicos rápidos será importante para estudar o efeito da matriz amilóide curli em Enterobacteriaceae.
Significado
Um fluxo de trabalho computacional foi desenvolvido para estudar as trajetórias dos grânulos para comparar as propriedades do material de biofilmes 3D. O fluxo de trabalho permite que os pesquisadores identifiquem parâmetros críticos que podem ser usados na modelagem computacional da dinâmica de fluidos em biofilmes heterogêneos. Com a ajuda deste avaliador de esferas de código aberto, o efeito da ondulação amilóide bacteriana nas propriedades do material pode ser estudado, mostrando maior rigidez da matriz de biofilme devido à ondulação. Em um contexto mais geral, o avaliador pode ser usado para estudar mudanças na estrutura do biofilme induzidas pelo tratamento com biofilme ou diferentes condições ambientais, como fluxo. Por exemplo, a ferramenta está sendo usada para analisar o efeito do tratamento com anticorpos monoclonais na ruptura das estruturas do biofilme em colaboração com o laboratório Tükel (LKSOM Temple University). A caixa de ferramentas do avaliador de contas é totalmente adaptável e extensível de forma modular usando o VRL-Studio para aprimorar e estender ainda mais suas funções.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
O trabalho nos laboratórios GQ e BAB não recebeu nenhuma doação específica de qualquer agência de financiamento nos setores público, comercial ou sem fins lucrativos. Os autores agradecem a Isaac Klapper, Ph.D (Departamento de Matemática, Temple University) pela discussão útil e Çagla Tükel (Departamento de Microbiologia e Imunologia, Temple University) pela experiência em Enterobacteriaceae na publicação inicial da contenção desta técnica.
Materiais
| Name | Company | Catalog Number | Comments |
| 96-well plates, No. 1.5 Uncoated Coverslip, 5 mm Glass Diameter | MatTek | P96G1.55F | |
| Fisherbrand Cover Glasses: Circles | Fisher Scientific | 12-293-232P | 1.5 optic glass coverslip |
| Invitrogen Syto 9 Green Fluorescent Nucleic Acid Stain | Invitrogen | S34854 | |
| Molecular Probes FluoSpheres Carboxylate-modified Microspheres, 1 um, crimson fluorescent (625/645) | Molecular Probes | F8816 |
Referências
- Huang, R., Li, M., Gregory, R. L. Bacterial interactions in dental biofilm. Virulence. 2, 435-444 (2011).
- Nadell, C. D., Drescher, K., Wingreen, N. S., Bassler, B. L. Extracellular matrix structure governs invasion resistance in bacterial biofilms. ISME Journal. 9, 1700-1709 (2015).
- Billings, N., Birjiniuk, A., Samad, T. S., Doyle, P. S., Ribbeck, K. Material properties of biofilms - key methods for understanding permeability and mechanics. Reports on Progress in Physics. 78, 036601 (2015).
- Araújo, G. R. d. e. S., Viana, N. B., Gómez, F., Pontes, B., Frases, S. The mechanical properties of microbial surfaces and biofilms. The Cell Surface. 5, 100028 (2019).
- Tallawi, M., Opitz, M., Lieleg, O. Modulation of the mechanical properties of bacterial biofilms in response to environmental challenges. Biomaterials Science. 5, 887-900 (2017).
- Tursi, S. A., Tükel, &. #. 1. 9. 9. ;. Curli-Containing Enteric Biofilms Inside and Out, Matrix Composition, Immune Recognition, and Disease Implications. Microbiology and Molecular Biology Reviews. 82, (2018).
- Dueholm, M. S., Albertsen, M., Otzen, D., Nielsen, P. H. Curli functional amyloid systems are phylogenetically widespread and display large diversity in operon and protein structure. PLoS One. 7 (12), 51274 (2012).
- Hung, C., et al. Escherichia coli biofilms have an organized and complex extracellular matrix structure. mBio. 4, 00645 (2013).
- Kikuchi, T., Mizunoe, Y., Takade, A., Naito, S., Yoshida, S. Curli fibers are required for development of biofilm architecture in Escherichia coli K-12 and enhance bacterial adherence to human uroepithelial cells. Microbiology and Immunology. 49, 875-884 (2005).
- Gallo, P. M., et al. Amyloid-DNA Composites of Bacterial Biofilms Stimulate Autoimmunity. Immunity. 42, 1171-1184 (2015).
- Serra, D. O., Richter, A. M., Hengge, R. Cellulose as an architectural element in spatially structured Escherichia coli biofilms. Journal of Bacteriology. 195, 5540-5554 (2013).
- Oh, Y. J. Characterization of curli A production on living bacterial surfaces by scanning probe microscopy. Biophysical Journal. 103, 1666-1671 (2012).
- Lembré, P., Di Martino, P., Vendrely, C. Amyloid peptides derived from CsgA and FapC modify the viscoelastic properties of biofilm model matrices. Biofouling. 30, 415-426 (2014).
- Oh, Y. J., et al. Curli mediate bacterial adhesion to fibronectin via tensile multiple bonds. Scientific Reports. 6, 33909 (2016).
- Vidakovic, L., Singh, P. K., Hartmann, R., Nadell, C. D., Drescher, K. Dynamic biofilm architecture confers individual and collective mechanisms of viral protection. Nature Microbiology. 3, 26-31 (2018).
- Tursi, S. A., et al. Salmonella Typhimurium biofilm disruption by a human antibody that binds a pan-amyloid epitope on curli. Nature Communications. 11, 1007 (2020).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15 (8), 1007978 (2019).
- Taglialegna, A., et al. The biofilm-associated surface protein Esp of Enterococcus faecalis forms amyloid-like fibers. Npj Biofilms and Microbiomes. 6, 15 (2020).
- Gour, S., Kumar, V., Rana, M., Yadav, J. K. Pheromone peptide cOB1 from native Enterococcus faecalis forms amyloid-like structures, A new paradigm for peptide pheromones. Journal of Peptide Science. 25, 3178 (2019).
- Stoodley, P., Debeer, D., Lewandowski, Z. Liquid flow in biofilm systems. Applied and Environmental Microbiology. 60, 2711-2716 (1994).
- Birjiniuk, A., et al. Single particle tracking reveals spatial and dynamic organization of the E. coli biofilm matrix. New Journal of Physics. 16, 085014 (2014).
- Chew, S. C., et al. Dynamic remodeling of microbial biofilms by functionally distinct exopolysaccharides. mBio. 5, 01536 (2014).
- Cao, H., et al. Revealing region-specific biofilm viscoelastic properties by means of a micro-rheological approach. Npj Biofilms and Microbiomes. 2, 5 (2016).
- Galy, O., et al. Mapping of bacterial biofilm local mechanics by magnetic microparticle actuation. Biophysical Journal. 103, 1400-1408 (2012).
- Rogers, S. S., vander Walle, C., Waigh, T. A. Microrheology of bacterial biofilms in vitro, Staphylococcus aureus and Pseudomonas aeruginosa. Langmuir. 24, 13549-13555 (2008).
- Hart, J. W., Waigh, T. A., Lu, J. R., Roberts, I. S. Microrheology and Spatial Heterogeneity of Staphylococcus aureus Biofilms Modulated by Hydrodynamic Shear and Biofilm-Degrading Enzymes. Langmuir. 35 (9), 3553-3561 (2019).
- van Merode, A. E. J., van der Mei, H. C., Busscher, H. J., Krom, B. P. Influence of culture heterogeneity in cell surface charge on adhesion and biofilm formation by Enterococcus faecalis. Journal of Bacteriology. 188, 2421-2426 (2006).
- Tariq, M., Bruijs, C., Kok, J., Krom, B. P. Link between Culture Zeta Potential Homogeneity and Ebp in Enterococcus faecalis. Applied and Environmental Microbiology. 78, 2282-2288 (2012).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ, 25 years of image analysis. Nature Methods. 9, 671-675 (2012).
- . Oracle.com Available from: https://docs.oracle.com/en/java (2021)
- Hoffer, M., Poliwoda, C., Wittum, G. Visual reflection library, a framework for declarative GUI programming on the Java platform. Computing and Visualization in Science. 16, 181-192 (2013).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15, 1007978 (2019).
- Malhotra, K., et al. Development of a New Bead Movement-Based Computational Framework Shows that Bacterial Amyloid Curli Reduces Bead Mobility in Biofilms. Journal of Bacteriology. 202, 00253 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados