Method Article
Un cadre de calcul basé sur le mouvement des billes pour l’analyse tridimensionnelle de l’hétérogénéité des matériaux de biofilm
Dans cet article
Résumé
Nous décrivons une méthode d’analyse et de quantification du mouvement de billes de carboxylate de 1 μm à travers des biofilms bactériens hétérogènes. La comparaison des modèles de mouvement peut être utilisée pour quantifier les différences dans les propriétés matérielles des biofilms.
Résumé
Des différences dans les propriétés matérielles des biofilms bactériens ont été observées dans les biofilms de différentes espèces bactériennes, au sein d’une même espèce dans différentes conditions de croissance et après traitement avec des molécules modifiant la matrice. Afin de mieux quantifier les propriétés matérielles des biofilms 3D, un flux de travail expérimental et informatique a été développé et appliqué pour examiner les différences entre les biofilms d’Enterococcus faecalis, de Salmonella enterica sérotype Typhimurium et d’Escherichia coli , ainsi que le rôle de la boucle amyloïde dans la confirmation de la rigidité des biofilms d’Enterobacteriaceae. La dynamique spatio-temporelle de billes de carboxylate de 1 μm dans les biofilms a été suivie dans 20 μm de biofilms 3D pendant 20 minutes. Les piles d’images 4D ont été traitées à l’aide du plugin Mosaic dans ImageJ pour produire des données de trajectoire 3D du mouvement des billes. Ces données de trajectoire ont été analysées à l’aide d’une nouvelle boîte à outils Bead Evaluator, où les données de mouvement, y compris la durée de vie des trajectoires, les vitesses des billes, les densités cellulaires le long des trajectoires et les informations de boîte englobante, ont été calculées et stockées dans des fichiers csv. Cet article présente le flux de travail, de la configuration expérimentale et de l’enregistrement d’images au calcul et à l’analyse de la trajectoire des billes. La structure des biofilms contenant des boucles a entraîné des interactions de billes plus stables et moins de mouvement des billes que dans les biofilms mutants et entérocoques. Le mouvement des billes n’est pas apparu fortement dépendant de la densité cellulaire lors de la mesure de la vitesse des billes et du volume de la boîte de délimitation de la trajectoire, ce qui soutient l’hypothèse selon laquelle d’autres propriétés matérielles des biofilms contrôlent la dynamique des billes. Cette technique est largement applicable pour quantifier les différences entre les biofilms de différentes compositions matricielles ainsi que les biofilms avant et après les traitements de modification de la matrice.
Introduction
Les biofilms bactériens sont omniprésents dans le microbiote humain et interagissent en permanence avec les molécules. La taille de ces molécules varie d’antibiotiques de 1 nm et de bactéries de 1 à 3 mm à des particules de fibres plus grosses dans le tractus gastro-intestinal. La composition des biofilms monospécifiques ou multi-espèces affecte les propriétés du matériau et donc le mouvement des particules à travers les biofilms 1,2,3,4,5. Un exemple est celui des amyloïdes bactériennes, qui ont une structure fibrillaire conservée en feuillet croisébêta 6. Le curli amyloïde est exprimé par des bactéries entériques telles que Escherichia coli et Salmonella enterica de sérotype Typhimurium et des gènes ont été détectés dans plusieurs autres embranchements bactériens7. Diverses propriétés matérielles des biofilms sont affectées par les boucles 8,9. Curli interagit directement avec d’autres composants de la matrice tels que l’ADN extracellulaire (ADNe) et la cellulose10,11. Curli entoure les cellules et affecte la rigidité de la membrane cellulaire12 et les propriétés viscoélastiques globales du biofilm13. La boucle médie une résistance à la traction accrue en se liant à la fibronectine, ce qui entraîne une augmentation de la forte fixation de la surface du verre14. Les bactériophages entrants se lient aux boucles et limitent l’invasion des phages dans les biofilms15.
Lors de l’utilisation de lames de puits revêtues de plusieurs tests pour analyser des Enterococcus faecalis, E. coli et S. Biofilms de typhimurium utilisant la microscopie confocale, différences claires entre E. coli, S. Des biofilmsde Typhimurium 10,16 et d’E. faecalis (étude en cours) ont pu être observés. Alors que les biofilms des espèces d’Enterobacteriaceae présentaient un niveau élevé de rigidité et que les zones à faible densité cellulaire étaient faciles à imager, l’obtention d’images claires à haute résolution des biofilms d’E. faecalis à l’aide de la moyenne linéaire et de l’image nécessitait l’application d’une pression sur la lame pour induire une tension superficielle suffisante pour la stabilité cellulaire pendant le processus d’imagerie. Les amyloïdes bactériennes telles que les curli forment des structures très ordonnées, ce qui suggère qu’elles peuvent être relativement rigides17. Cela a motivé l’hypothèse selon laquelle les boucles amyloïdes pourraient induire une rigidité chez E. coli et S. Biofilms de Typhimurium. Il n’y avait pas de preuve claire que E. faecalis exprimait des amyloïdes dans les conditions étudiées. Il a récemment été démontré que la protéine Esp, un gène de la piline associé à des souches plus pathogènes d’E. faecalis, produisait des structures amyloïdes18 ; Cependant, à l’aide des recherches blastn et blastp, ce gène n’a pas été détecté dans la souche OG1RF de type commensal d’E. faecalis utilisée dans ces études. La phéromone cOB1, produite par OG1RF, peut former des structures de type amyloïde19. Cependant, avec les conditions de croissance du biofilm données et les méthodes de détection de l’amyloïde précédemment utilisées pour S. La coloration amyloïdede Typhimurium 10 chez E. faecalis, les amyloïdes OG1RF n’a pas pu être détectée (données non présentées). Une nouvelle technique d’image quadridimensionnelle (4D) a été mise au point pour comparer les propriétés globales des matériaux parmi les viscosques E. faecalis, E. coli et S. Typhimurium, ainsi que pour déterminer la contribution de l’amyloïde aux biofilms d’entérobactéries à l’aide de mutants amyloïdes de S. Typhimurium et E. coli.
Dans le passé, les billes fluorescentes étaient utilisées avec succès pour analyser les propriétés des matériaux des biofilms en deux dimensions (2D) à l’aide de la microrhéologie 20,21,22,23,24,25. Cela peut être appliqué à un biofilm tridimensionnel en étudiant des coupes optiques 2D à différentes profondeurs dans le biofilm26. La technique actuelle a été développée pour suivre des microbilles de 1 μm en 3D au fil du temps pour une utilisation dans la modélisation 4D. Une partie de la justification était le concept global d’utilisation de la modélisation 4D pour comprendre le mouvement des plasmides à travers les communautés de microbiote gastro-intestinal. Des billes de carboxylate chargées par fluorescence d’un diamètre de 1 m ont été utilisées, car elles correspondent bien, en ce qui concerne la taille et la charge, à E. faecalis, l’organisme modèle choisi pour le mouvement et la maintenance des plasmides27, 28. Un test 4D pour quantifier les propriétés physiques des biofilms a été mis au point (Figure 1A). Dans la méthodologie conçue, des billes ont été ajoutées à des biofilms et leurs trajectoires spatio-temporelles ont été enregistrées à travers des biofilms de 10 à 20 μm d’épaisseur pendant 10 à 20 minutes. Les trajectoires des billes en 3D ont ensuite été quantifiées en termes de longueur de trajectoire, de vitesse de bille, de volume de boîte englobante de trajectoire (boîte minimale contenant la trajectoire) et de densité cellulaire de boîte englobante à l’aide d’une boîte à outils nouvellement développée. Le protocole suivant peut être utilisé pour générer des données d’image 4D de bactéries et de biofilms contenant des billes, pour prétraiter les données avec ImageJ29 et le plugin Mosaic, et pour analyser les trajectoires des billes avec une boîte à outils Bead Evaluator.
Cette technique a de multiples applications pour examiner les propriétés des matériaux ainsi que pour suivre les mouvements des particules et des bactéries en trois dimensions. Par exemple, une première version de cette technique a été utilisée pour caractériser l’effet des anticorps monoclonaux dirigés contre les courbettes sur l’intégrité structurelle des biofilms16. La version complète dispose de plusieurs outils pour fournir une analyse plus détaillée des propriétés du matériau du biofilm et continue d’être utilisée pour examiner les effets du traitement par anticorps monoclonaux sur les biofilms. Des particules de charges différentes peuvent être utilisées pour examiner les propriétés de charge matérielle des biofilms et le mouvement des particules à travers des biofilms de différentes compositions matricielles. Cela pourrait être utilisé pour comparer les résultats de la microrhéologie 2D qui révèlent les propriétés matérielles responsables du mouvement des billes que nous avons observées dans des biofilms qui n’étaient pas sous écoulement. Cette technique pourrait également être utilisée sur des biofilms d’espèces mixtes avec des régions de composition de biofilm différente. Les biofilms peuvent être imagés en direct sous écoulement dans des dispositifs microfluidiques et des cellules d’écoulement pour examiner les changements dans les propriétés des matériaux entre les biofilms statiques et d’écoulement ainsi que l’effet de l’écoulement sur le mouvement des particules. Les techniques peuvent également être appliquées à des bactéries marquées par fluorescence pour caractériser le mouvement des bactéries exogènes à travers une communauté de biofilm. À l’aide de trois couleurs, des bactéries donneuses marquées par fluorescence, des bactéries réceptrices marquées par fluorescence et des plasmides marqués par fluorescence pourraient être utilisées pour suivre le mouvement, l’amarrage et le transfert des plasmides.
Protocole
1. Préparation du biofilm
REMARQUE : Les biofilms pour l’analyse peuvent être cultivés en utilisant n’importe quelle méthode qui permet au biofilm de se former sur une surface en verre optique. La structure du biofilm doit adhérer suffisamment à la surface optique pour que la structure ne soit pas perturbée pendant les étapes de lavage et/ou de montage du protocole. Vous trouverez ci-dessous une description de la technique pour les plaques inférieures optiques à 96 puits et les lamelles en verre de 12 mm dans les plaques à 24 puits. D’autres options incluent des plaques inférieures optiques de différentes tailles et des chambres d’écoulement optique avec et sans flux.
- Configuration du biofilm
- Ajouter le milieu de croissance bactérienne dans les puits de la plaque. Pour cette étude, pour E. faecalis, ajouter 2 mL de Todd-Hewitt (TH) dans une plaque à 24 puits et ajouter 0,4 mL de TH dans des plaques optiques à 96 puits. Pour S. Typhimurium, E. coli et les mutants isogéniques csgAB curli ajoutent 0,7 mL de bouillon Luria sans sel (LB) dans les puits. Si vous utilisez des chambres inférieures optiques, passez à l’étape 1.2.
- Placez des lamelles optiques en verre #1.5 de 12 mm dans une boîte de Pétri et couvrez-les d’éthanol.
- À l’aide d’une pince, retirez la lamelle et allumez le feu à l’alcool restant à l’aide d’une flamme. Laissez l’alcool brûler. Utilisez simplement la flamme pour les allumer ; Ne tenez pas la lamelle dans la flamme car elle se fissurerait. Laissez refroidir la lamelle pendant 10 à 20 s avant de la placer dans le puits pour éviter qu’elle ne se fissure.
- Placez la lamelle dans le puits contenant le fluide à un angle pour éviter qu’il ne repose sur le fluide. N’ajoutez pas la lamelle dans un puits sec, puis ajoutez du moyen, car cela ferait coller la lamelle au fond du puits.
- À l’aide d’une pointe de pipette stérile, poussez délicatement la lamelle au fond du puits contenant le fluide.
REMARQUE : N’oubliez pas de faire correspondre l’épaisseur (#1 ou #1.5) de la lamelle en verre optique ou de la plaque inférieure optique à l’épaisseur de l’optique du microscope confocal.
- Incuber le biofilm dans des conditions propices à la croissance du biofilm. Dans ces études, on cultive des biofilms d’E. faecalis sous forme de cultures statiques à 37 °C et des biofilms d’E. coli en aérobie à 30 °C.
- Dans ces études, cultivez des biofilms d’E. faecalis pendant 2 jours avec des changements de milieu le matin et en début de soirée. Pour éviter d’endommager ou de déloger le biofilm, inclinez soigneusement la plaque. Placez la pointe de la pipette près du bord inférieur du puits et retirez lentement le fluide. Ajouter le premier mL de milieu frais de la même façon. Ajouter lentement le deuxième mL près de l’interface milieu/puits.
- Dans ces études, utilisez les conditions de croissance pour une production optimale de courbes. Croissance S. Les biofilms de Typhimurium à 28 °C pendant 6 à 8 jours ont été incubés sur une pente qui a permis au biofilm de se fixer aux 2/3 de la hauteur de la lame, puis de se développer sous forme de pellicule à l’interface air-liquide. Cela a été fait sans changement de moyen. Pour éviter que le fluide ne se dessèche, placez la plaque à 24 puits dans une chambre avec un bac à eau.
2. Imagerie 4D
- Préparation du support de biofilm
- Billes FluoSpheres de carboxylate Crimson 1 μm diluées 1:50 dans du PBS (2 x 107 billes dans 1 mL de PBS). Si vous utilisez des chambres inférieures optiques, ajoutez Syto9 à une dilution de 1 μL à 300 μL de préparation de billes.
- (facultatif) Lavez le biofilm pour éliminer les traces de milieu de croissance si le milieu de croissance a une autofluorescence. Dans ces expériences, laver deux fois avec 1 mL de PBS, en inclinant soigneusement la plaque, en plaçant la pointe de la pipette près du bord inférieur du puits et en retirant lentement le milieu. Ajoutez du PBS en plaçant l’embout près du bord inférieur et remplissez lentement le puits. Utilisez cette technique sur des puits de fond optiques en plus des lamelles dans une plaque à 24 puits.
- Retirez le support ou le PBS. Ajouter les billes de pourpre diluées préparées en 2.1.1 au biofilm. Dans ces études, ajouter 1 mL de billes (2x107) aux lamelles et 0,2 mL de billes (4x106) et de Syto9 aux plaques inférieures optiques à 96 puits.
- Incuber 1 min à température ambiante pour permettre l’association des billes.
- Retirez les billes et lavez doucement le biofilm une fois avec du PBS pour éliminer les billes non associées. Dans ces études, pour la lamelle, laver doucement avec 1 mL de PBS, remplir le puits avec 1 mL de PBS et passer à la section 2.1.6. Pour les plaques inférieures optiques à 96 puits, lavez doucement le biofilm avec 0,2 ml de PBS et remplissez le puits avec 0,2 ml de PBS. La chambre inférieure optique est maintenant prête à recevoir des images, passez donc à l’étape 2.2.
- (En cas d’utilisation d’une lamelle) Ajouter 1 μL de Syto9 (colorant d’ADN fluorescent vert ; dilué selon les instructions du fabricant) au centre d’un puits sur une lame multitest 10 puits enduite. Ces lamelles recouvertes ont des profondeurs de puits de 23 à 25 μm.
- (En cas d’utilisation d’une lamelle) À l’aide d’une pince stérilisée à la flamme d’alcool, retirez délicatement la lamelle du puits et retournez-la sur le puits contenant la goutte Syto9. Le fait de laisser la lamelle dans 1 mL de PBS facilite son retrait du puits et aide à éliminer les billes non associées du biofilm.
- (En cas d’utilisation d’une lamelle) Scellez soigneusement la lamelle avec du vernis à ongles sans glisser ni appuyer sur la lamelle, ce qui peut provoquer une tension superficielle qui arrêtera le mouvement dans les biofilms plus visqueux et moins rigides.
- (En cas d’utilisation d’une lamelle) Laissez sécher le vernis à ongles. Essuyez soigneusement la surface extérieure de la lamelle avec de l’éthanol à 70 %. Essuyez sans appliquer de pression sur la lamelle pour les raisons indiquées ci-dessus.
- Imagerie confocale
REMARQUE : Dans ces études, utilisez un microscope confocal à balayage laser à imagerie spectrale inversée équipé d’un système confocal TCS avec l’objectif 63x. L’oscilloscope sera utilisé pour générer une vidéo 4D (Figure 1). Le biofilm 3D sera composé de tranches Z capturées par pas de 0,5 m à travers des biofilms de 18 à 20 m d’épaisseur, générant 36 à 40 tranches Z. Chaque biofilm 3D prendra 50 à 60 s à capturer. Les tranches forment ensemble une image, qui peut être visualisée comme un biofilm 3D. Ce processus sera répété 20 fois pour générer la vidéo 4D time lapse pour un temps de suivi total de 18 à 20 minutes.- Réglez la lunette pour capturer les fluorophores. Dans ces études, excitez Syto9 (coloration de l’ADN bactérien) avec un laser de 488 nm et mesurez l’émission de 495 à 540 nm (le Leica Sp5 est un microscope d’imagerie spectrale). Excitez des perles cramoisies (rouges) avec un laser de 633 nm et mesurez l’émission de 650 nm à 700 nm. Ces paramètres de fluorophore peuvent être ajustés pour capturer tous les fluorophores souhaités.
- Choisissez le mode d’imagerie xyzt.
- Identifiez une région du biofilm avec un mélange de régions de densité supérieure et inférieure pour capturer les différences de propriétés viscoélastiques dans les régions épaisses et minces du biofilm.
- Réglez une pile Z de 18 à 20 μm d’épaisseur. Dans un biofilm monté sur lamelle, évitez que le haut et le bas du biofilm ne touchent le verre pour éviter les artefacts de piégeage des billes (voir la discussion).
- Ajustez le gain et le décalage pour utiliser toute la plage dynamique de l’intensité dans le point le plus lumineux du biofilm. Cela minimise le chevauchement du signal des couches inférieures du biofilm.
- Réglez l’épaisseur de la tranche sur 0,5 μm. Cela permet une imagerie rapide sans perte d’informations sur le talon.
- Réglez la résolution sur 512 x 512 (0,48 μm). Cela permet une imagerie rapide tout en générant des images avec une résolution suffisante pour voir la structure du biofilm et les détails du mouvement des billes.
- Minimisez le temps d’imagerie.
- Configuré pour capturer 20 piles.
- Enregistrez au format .lif (ou fichier confocal similaire). Le film 4D peut être généré dans ImageJ.
3. Générer la vidéo du biofilm 4D avec ImageJ
- Ouvrez le fichier .lif dans ImageJ avec les paramètres suivants : Vue de la pile avec Hyperstack, Ordre de la pile XYCZT, Mode colorimétrique : colorisé, mise à l’échelle automatique cochée. Appuyez ensuite sur OK.
- Sélectionnez Image > Couleur > Diviser les canaux.
- Sélectionnez Image > Couleur > Fusionner les couches > Créer une > composite OK.
- Sélectionnez Plugins > visionneuse 3D > Chaînes, décochez bleu > OK.
- Sélectionnez Modifier > Afficher le contenu coché > Afficher la boîte englobante cochée.
- Cliquez avec la souris et maintenez-la enfoncée pour sélectionner l’image. Ensuite, faites pivoter l’image tout en maintenant le bouton de clic enfoncé. Faites pivoter l’image de sorte que le bas du biofilm soit sur le bas et que l’angle permette de visualiser les billes. Relâchez ensuite l’image.
- Appuyez sur le bouton rouge Enregistrer en bas de la fenêtre pour enregistrer une vidéo.
- Enregistrez en tant que fichier .avi en utilisant jpeg comme compression.
4. Générer des données de trajectoire
- Installez l’outil open-source ImageJ (https://imagej.net/Fiji) et le plugin de suivi de particules Mosaic (https://imagej.net/MOSAICsuite).
- Importez le fichier .lif contenant les deux canaux pour les billes et les bactéries. Divisez les canaux et enregistrez les fichiers séparément.
- Dans ImageJ, stockez la taille du voxel de l’image (dimensions x, y et z) et la taille du pas temporel dans un fichier texte.
- Dans ImageJ, allez dans Plugins | Mosaic et lancez le Particle Tracker 2D/3D.
- Entrez les paramètres suivants : Rayon : 3, Coupure : 0,003, Per/Abs : 0,12, Plage de liaison : 2, Déplacement : 10,00 avec la dynamique réglée sur Brownien. Veuillez consulter les résultats pour savoir pourquoi le choix du brownien est fait dans ces expériences.
- Générez des trajectoires et exportez la liste des trajectoires sous forme de fichiers .csv.
5. Analyser les trajectoires
- Installer VRL Studio (https://vrl-studio.mihosoft.eu)
- Téléchargez le projet Biofilm (https:// neurobox3d.github.io/Biofilm/) et lancez-le dans VRL Studio
- Charger le fichier de trajectoire dans ImportData
- Spécifiez la taille de pixel x, y et z (utilisez ImageJ pour localiser ces valeurs) dans ProcessTrajectories.
- Spécifiez l’intervalle d’image dans ComputeVelocity (utilisez ImageJ pour localiser cette valeur).
- Chargez la lime bactérienne (voir étape 3.2) dans Comdensity.
- Définissez les chemins de sortie pour les données de vitesse dans SaveVelocityDataToFile et les données de trajectoire dans SaveTrajectoryDataToFile.
- Appelez SaveVelocityDataToFile et SaveTrajectoryDataToFile.
- Importez des données dans Excel pour analyse. Ces données comprendront les longueurs de trajectoire, les durées de vie des trajectoires, les dimensions et les volumes de la boîte de délimitation de trajectoire, les vitesses moyennes des billes et les variances. L’analyse calcule des variables pondérées à l’aide du canal des bactéries marquées par Syto9 pour calculer les densités cellulaires locales (à l’intérieur de boîtes de délimitation de trajectoire données). L’analyse aboutit au calcul des vitesses moyennes pondérées et des variances ainsi que des moyennes pondérées et des variances de la boîte englobante.
Résultats
Cette méthode a été utilisée pour tester l’hypothèse selon laquelle les boucles peuvent conférer une rigidité à E. coli et S. Biofilms de typhimurium, réduisant le mouvement des billes lors d’expériences de microscopie confocale. La boîte à outils actuelle a été utilisée pour comparer les propriétés matérielles de la souche OG1RF de type commensale d’Enterococcus faecalis à celles du sérotype Typhimurium et E. coli de Salmonella enterica et de leurs mutants isogéniques respectifs (figure 1B et vidéo supplémentaire 1, vidéo supplémentaire 2, vidéo supplémentaire 3, vidéo supplémentaire 4, vidéo supplémentaire 5, vidéo supplémentaire 6). Les propriétés des matériaux du biofilm pourraient potentiellement différer en ce qui concerne la rigidité (par exemple, les boucles liées à l’ADNe) ou les interactions électrostatiques et hydrophobes entre les billes chargées négativement et les cellules du biofilm et les matériaux matriciels, ainsi que la densité cellulaire.
Reproductibilité
La boîte à outils Biofilm a été programmée en Groovy30 et Java31 au sein de VRL-Studio32 , permettant une conception modulaire du flux de travail avec génération automatique d’interface utilisateur (UI) de tous les composants de calcul. Cela a permis un flux de travail automatisé, éliminant les biais induits par l’expérimentateur involontaire lors de l’analyse des résultats.
Utilisation de la TMS pour confirmer le type de mouvement dans les biofilms
Pour l’analyse des trajectoires à l’aide de Particle Tracker 2D/3D, différents paramètres de dynamique pour l’analyse de différents types de mouvements de billes sont disponibles. Pour ces études, le réglage du « mouvement brownien » (c’est-à-dire le mouvement induit par la diffusion) a été choisi puisque E. faecalis est une bactérie non mobile, E. coli et Salmonella n’expriment pas de flagelles dans les biofilms, et les expériences ont été réalisées dans un système fermé en l’absence d’écoulement. Ce réglage a pu être validé par les déplacements carrés moyens (MSD) calculés des billes. En utilisant la définition 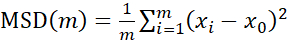 où m est le nombre de segments de trajectoire, la variation de la MSD au cours de chaque trajectoire peut être calculée. Les trajectoires linéaires indiquent un mouvement diffusif du cordon (Figure 2A). À l’aide de l’ajustement des moindres carrés quadratiques, le modèle de mouvement moyen de toutes les billes du biofilm a été calculé, montrant l’ordre linéaire dominant et validant la diffusion passive comme force motrice (Figure 2A-2F).
où m est le nombre de segments de trajectoire, la variation de la MSD au cours de chaque trajectoire peut être calculée. Les trajectoires linéaires indiquent un mouvement diffusif du cordon (Figure 2A). À l’aide de l’ajustement des moindres carrés quadratiques, le modèle de mouvement moyen de toutes les billes du biofilm a été calculé, montrant l’ordre linéaire dominant et validant la diffusion passive comme force motrice (Figure 2A-2F).
Analyse de la boîte englobante.
La boîte à outils utilise ImageJ Mosaic et Particle Tracker 2D/3D pour générer des trajectoires (étape 4), puis à l’aide du pipeline d’analyse automatisé des biofilms, génère des données importantes sur les trajectoires des billes qui peuvent être utilisées pour comparer les propriétés des matériaux du biofilm. Le volume de la boîte englobante en μm3 a été mesuré en construisant la boîte minimale qui contient une trajectoire et en mesurant son volume (Figure 3).
Les biofilms d’E. faecalis ont un mouvement de bille plus important avec des valeurs de boîte englobante de 1 à 6000 μm3 (figures 3B, 3C et 3D). Les résultats confirment que le mouvement observé dans une lamelle de verre montée sur une lame revêtue avec un puits revêtu d’environ 25 μm (Figure 3C) par rapport aux biofilms cultivés au fond des puits de verre optique et directement imagés (Figure 3D) donne des résultats équivalents avec peu de différences. La seule différence était que près du haut de la lamelle de couverture montée des biofilms d’E. faecalis, des trajectoires stables avec une durée de vie supérieure à 10 minutes mais en même temps de petites boîtes englobantes pouvaient être enregistrées, tandis que dans la plaque inférieure optique, un nombre sélectionné de billes avec une mobilité plus élevée pouvait être enregistré. Dans l’ensemble, cela suggère que le montage de la lame de verre peut avoir modifié la tension superficielle du système au sommet du biofilm contre la lame dans le biofilm de lamelle de recouvrement montée, ce qui a finalement diminué la mobilité de certaines billes dans les régions de biofilm moins visqueuses (figures 3B, 3C, 3D et 3I). Il s’est avéré que les trajectoires qui entrent dans cette catégorie représentaient un très faible pourcentage et, même avec ce petit nombre de billes piégées, la TMS moyenne d’E. faecalis sur une lamelle de couverture montée était légèrement plus élevée que la TMS calculée à partir d’un biofilm de plaque inférieure optique (figure 3).
L. Les trajectoires des billes de Typhimurium et d’E. coli avaient des volumes de boîte englobante plus petits de 0 à 10 μm3 (figures 3A, 3B, 3E et 3F), par rapport aux curli mutants isogéniques avec des boîtes englobantes de 1-6000 μm3 pour E. coli et de 1-5000 μm3 pour S. Typhimurium (figures 3A, 3B, 3F et 3H), montrant une plus grande mobilité des billes. Ces résultats suggèrent que la présence de l’amyloïde est corrélée à une rigidité accrue dans les biofilms et est cohérente avec l’absence de mouvement notable du biofilm dans les vidéos. Les volumes de la boîte englobante étaient constamment petits (0-10 μm3), même dans les régions de faible densité du biofilm. Cette observation est cohérente avec les observations précédentes selon lesquelles les boucles peuvent être présentes dans les régions à faible densité cellulaire du biofilm10.
Il n’a pas été possible de comparer le comportement des biofilms d’Enterobacteriaceae sur les plaques inférieures optiques car ils se développent sous forme de pellicules à l’interface air-liquide (étape 1.2.2). Lors de l’utilisation d’une lamelle, la pellicule est fixée à la lamelle au niveau de l’interface et lorsque la lamelle est retirée, la pellicule est posée sur la lamelle, créant ainsi une seule surface d’image. Dans une plaque inférieure optique cultivée en biais, l’imagerie a été réalisée avec du liquide encore dans le puits. Cela signifie que la pellicule flotte toujours au-dessus du fond de l’optique et qu’elle sort la pellicule de la profondeur de travail d’une lunette inversée telle que le Leica Sp5. En enlevant suffisamment de milieu pour amener le biofilm dans la profondeur de travail du microscope, l’échantillon s’est desséché au cours du processus d’imagerie de 20 minutes.
Dans l’ensemble, les graphiques confirment les observations visuelles dans les films supplémentaires et sont cohérents avec les différences de TMS observées (figures 3I et 3J).
Durée de vie des trajectoires
La durée de vie de la trajectoire a été mesurée comme le nombre d’images consécutives dans lesquelles une bille a été enregistrée (figure 3).
Dans les biofilms d’E. faecalis , plus visqueux et fluides, toutes les billes avaient une durée de vie de trajectoire inférieure à 10 minutes et la majorité des trajectoires variaient entre 2 et 5 minutes pour les biofilms d’E. faecalis . Cependant, des billes dont la durée de vie de trajectoire est courte ont pu être localisées par inspection visuelle dans les biofilms d’E. faecalis sur toute la plage horaire d’imagerie (vidéos supplémentaires 1 et 2). Ainsi, il est possible que les billes se déplacent le long d’une trajectoire enregistrée, se dissociant par intermittence du biofilm et terminant une trajectoire, et se réassociant au biofilm, à ce moment-là une nouvelle trajectoire est initiée. Cela conduirait finalement à des durées de vie courtes sous la présence continue de billes dans le biofilm. Il est important de noter qu’en utilisant cette technique, la durée de vie de la trajectoire, en particulier dans un biofilm visqueux, a tendance à sous-estimer le temps total pendant lequel une bille est associée au biofilm.
Dans S. La majorité des billes (environ 80 %) avaient une longue durée de vie de 16 à 20 images, ce qui correspond à environ 15 à 20 minutes en temps réel (figures 3A, 3G et 3H). Contrairement à ceux-ci, les biofilms curlites isogéniques portaient davantage de billes mobiles avec des volumes de boîte englobante allant de 1 à 6000 μm3 (E. coli) et de 1 à 5000 μm3 (S. Typhimurium) (figures 3A, 3B, 3F et 3H). Cependant, contrairement aux biofilms d’E. faecalis dont les trajectoires sont à >70 % et dont le volume de la boîte englobante est supérieur à 10 μm3, les biofilms des espèces d’Enterobacteriaceae n’ont enregistré que 30 % de trajectoires de billes avec des volumes de boîte englobante supérieurs à 10 μm3. Même si la durée de vie globale des trajectoires des billes était plus petite dans les biofilms de mutants courblits, certaines trajectoires reflétaient un mouvement important des billes et des longues durées de vie (Figure 3H). Cette observation pourrait indiquer que cette variabilité peut correspondre à des propriétés variables des matériaux du biofilm, telles que la viscoélasticité, et/ou à des changements chimiques de surface des particules tels que la charge.
Analyse de la longueur et de la vitesse de la trajectoire des talons
La longueur de trajectoire est une mesure de la distance parcourue par les billes en μm. Cette mesure est cohérente avec la vitesse de mouvement du cordon en μm/s. Conformément aux volumes de boîte englobante plus grands, les billes dans les biofilms d’E. faecalis avaient des trajectoires 10 fois plus longues, de 5 à 20 μm, contre <4 μm dans les biofilms contenant des courbes. Cohérent avec les trajectoires plus courtes (figure 4A). Les billes d’E. faecalis ont mesuré des vitesses jusqu’à 15 fois plus élevées, la majorité d’entre elles ayant des vitesses de l’ordre de 0,01 à 0,15 μm/s contre des vitesses de <0,006 μm/s (figure 4B). Néanmoins, les biofilms mutants de curli ont mesuré des vitesses globalement plus faibles et des trajectoires plus courtes par rapport aux biofilms d’E. faecalis , mais des trajectoires plus longues et des vitesses plus élevées que les curli contenant des souches parentales (figures 4A et 4B).
Il convient de noter que la structure en treillis fibrillaire de curli30 peut affecter la mobilité de manière anisotrope, réduisant le mouvement dans le plan xy et permettant une mobilité accrue dans la direction z (Figure 2G). Le grand nombre de trajectoires (environ 800) appartenant à environ 50 billes uniques dans des biofilms contenant des boucles serait cohérent avec les limites du suivi des particules en mosaïque, en comptant chacune de ces billes se déplaçant rapidement comme une seule bille en x, y et z. Des recherches supplémentaires et le développement de logiciels seront nécessaires pour confirmer cette observation.
Analyse de la dépendance du mouvement des billes par rapport à la densité cellulaire
La dépendance du mouvement des billes par rapport à la densité cellulaire a été déterminée à l’aide de vitesses et de variances moyennes pondérées, ainsi que de moyennes/moyennes pondérées et de variances des volumes de la boîte englobante. Le deuxième canal d’imagerie pour les bactéries marquées Syto9 a été utilisé pour calculer les densités cellulaires locales dans le calcul des vitesses pondérées. La densité cellulaire a été calculée en faisant la moyenne des données de voxel Syto9 sur la boîte englobante de chaque bord de trajectoire (Figure 5, à droite). Ainsi, la vitesse du cordon peut être pondérée par les densités cellulaires (locales) sur les bords. Il existe plusieurs types de colorants qui pourraient être utilisés pour visualiser les bactéries, y compris les colorants de la paroi cellulaire, des membranes et du contenu en ADN. Pour déterminer la densité cellulaire, Syto9 a été choisi parce qu’il donne le signal le plus cohérent, quelle que soit la tranche Z optique visualisée. Les taches d’enveloppe (paroi cellulaire et membrane) donneront un signal différent en fonction de la position de la tranche Z. Si la tranche Z comprend le haut ou le bas de la cellule, le signal sera plus fort que si la tranche Z se trouve au milieu de la cellule où seul le contour de la cellule est coloré.
Les trajectoires des billes du canal rouge ont été suivies sur 20 images, où les trajectoires individuelles avaient une durée de vie minimale de 2 images et un maximum de 20 images avec 19 segments de trajectoire reliant les images (figure 5). Pour étudier la dépendance de la mobilité des billes à la densité cellulaire, l’intensité de la GFP par voxel (chacune des mesures individuelles dans l’image de voxel 512x512) a été déterminée. La densité cellulaire autour de chaque segment de la trajectoire du talon a été calculée comme la densité moyenne localement dans la boîte englobante du segment.
Pour certains biofilms, une dépendance statistiquement significative à la densité a pu être documentée (figure 6), plus particulièrement pour les biofilms d’E . faecalis qui ont été cultivés sur une lamelle de verre et inversés sur une lame multipuits (figure 6A). Au contraire, les biofilms d’E. faecalis qui ont été cultivés au fond d’une plaque de 96 puits (figure 6B) n’ont montré aucune dépendance à la densité. En conclusion, cela suggère que les biofilms très fluides d’E. faecalis pourraient potentiellement être légèrement comprimés en raison du montage sur une lame à plusieurs puits, ce qui est cohérent avec une réduction du nombre de billes se déplaçant plus rapidement et de celles avec de petits volumes de boîte englobante qui sont piégées au sommet du biofilm contre la lame de verre (Figure 3C vs Figure 3D). Les biofilms de Salmonella et d’E. coli (figures 6C et 6D) et leurs mutants isogéniques (figures 6E et 6F) ont montré une dépendance mineure ou nulle de la densité cellulaire.
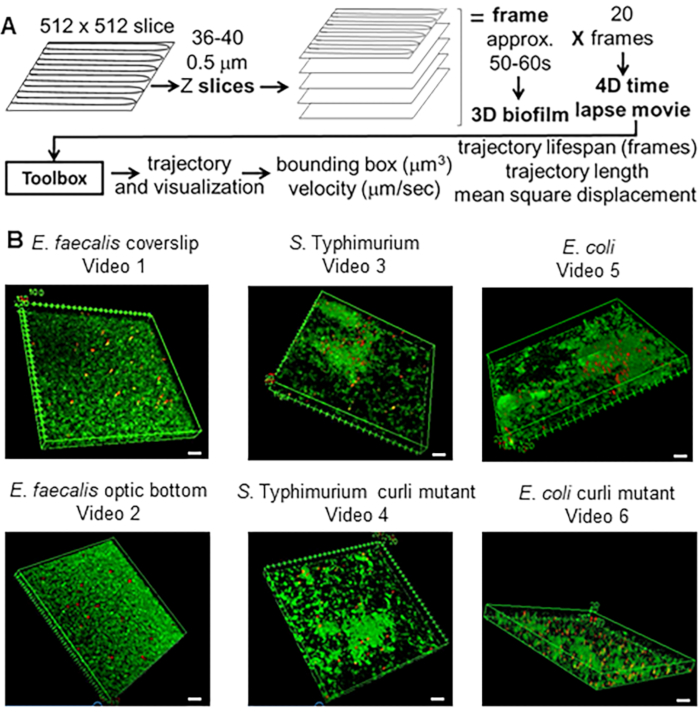
Graphique 1. Pipeline d’imagerie et d’analyse (étapes 2 à 4) (A) Les biofilms sont imagés comme indiqué à la section 2.2. À l’aide des biofilms imagés (voir 3), les trajectoires des billes ont été générées comme décrit en 4. À l’aide des trajectoires, les données pertinentes ont été calculées à l’aide de la boîte à outils d’analyse (voir 5) (B) Des biofilms ont été cultivés comme décrit à l’étape 1 sur des lamelles (E. faecalis, Vidéo 1), S. Typhimurium (Vidéo 3), E. coli (Vidéo 5) et les curli mutants isogéniques (Vidéo 4, Vidéo 6) ou dans une plaque à fond optique à 96 puits (E. faecalis bottom, Vidéo 2). La barre d’échelle blanche est de 20 mm. Cette figure est reproduite avec la permission de (31). Veuillez cliquer ici pour voir une version agrandie de cette figure.
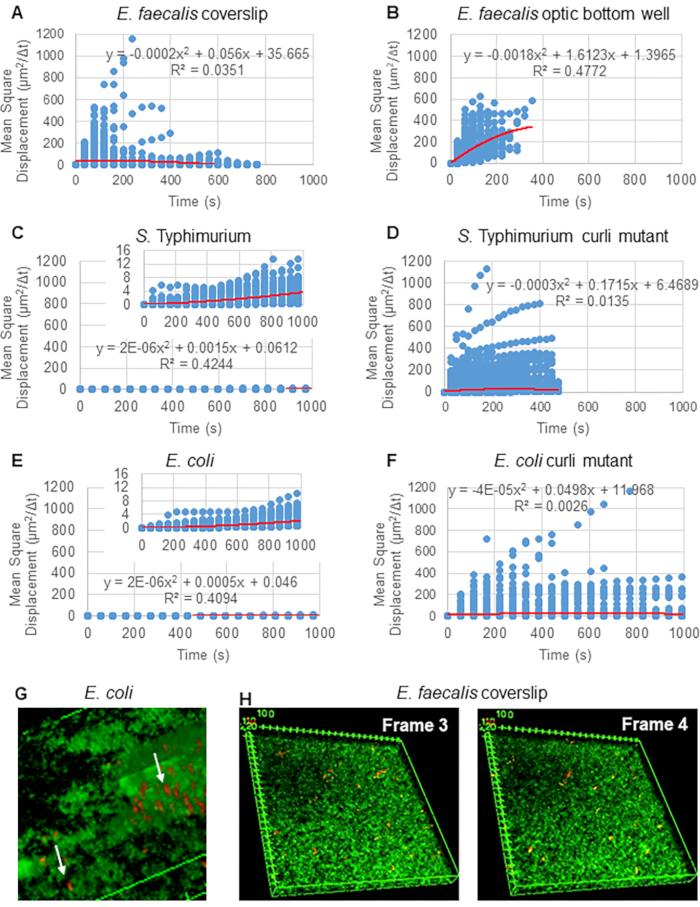
Graphique 2. Il a été déterminé que le mouvement des billes dans les biofilms était de nature diffusive (mouvement brownien, voir étape 5) (A-F) Les données MSD montrent un comportement linéaire (ligne rouge) pour valider le mouvement brownien. Les biofilms ont été cultivés conformément à l’étape 1 (G) Exemple de mouvement elliptique des billes observé dans les biofilms d’E. coli et de S. Typhimurium prélevés dans une image de l’essai de biofilm 4D d’E. coli (H) Exemple de changements importants dans les motifs des billes entre les images 3 et 4 pris dans le puits de fond optique d’E. faecalis. Notez que le biofilm lui-même suscite un certain flux (vidéos 1 et 2), ce qui donne l’impression que les images 3 et 4 sont orientées différemment. Cette figure est reproduite avec la permission de (31). Veuillez cliquer ici pour voir une version agrandie de cette figure.
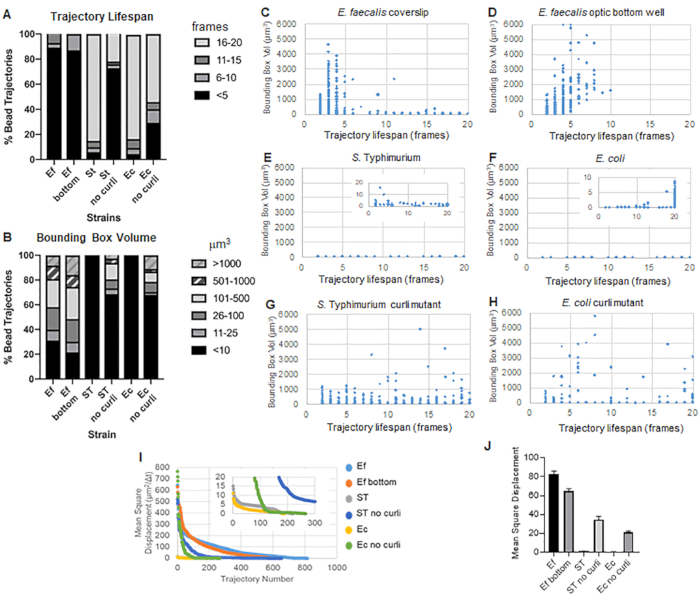
Graphique 3. Analyse des différences de rigidité à l’aide de boîtes englobantes et de durées de vie des trajectoires. Des biofilms ont été cultivés comme décrit à l’étape 1 sur des lamelles (E. faecalis, Vidéo 1), S. Typhimurium (Vidéo 3), E. coli (Vidéo 5) et les curli mutants isogéniques (Vidéo 4, Vidéo 6) ou dans une plaque à fond optique à 96 puits (E. faecalis bottom, Vidéo 2). Les durées de vie des trajectoires sont présentées en % des trajectoires totales des billes (A) et des graphiques de dispersion (C-H), ainsi que des volumes de boîte englobante (calculés à l’étape 5) (I) Comparaison des TMS des billes dans les différents biofilms (H) TMS moyens de chaque type de biofilm. Les barres indiquent l’intervalle de confiance à 95 % des données. Cette figure est reproduite avec la permission de (31). Veuillez cliquer ici pour voir une version agrandie de cette figure.
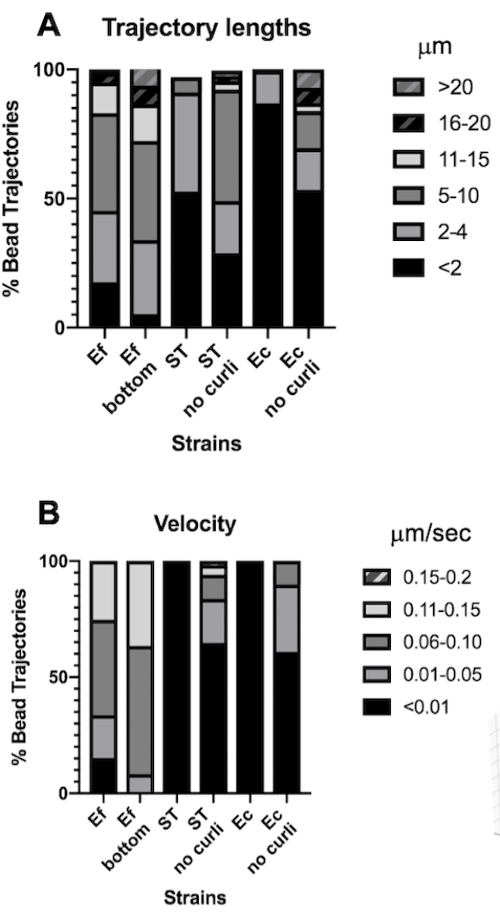
Graphique 4. Analyse des différences de longueur de trajectoire et de vitesse du talon. Les biofilms ont été cultivés comme décrit à l’étape 1. Les longueurs de trajectoire sont indiquées en μm et sont présentées en % des trajectoires totales des billes (A). La vitesse est indiquée en μm/s et présentée en % des trajectoires totales des billes (B). Cette figure est adaptée avec la permission de (31). Veuillez cliquer ici pour voir une version agrandie de cette figure.
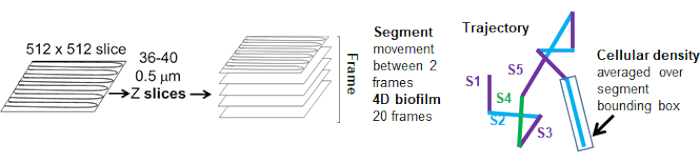
Graphique 5. Aperçu de l’analyse des segments de trajectoire. Cette figure est adaptée avec la permission de (31). Veuillez cliquer ici pour voir une version agrandie de cette figure.
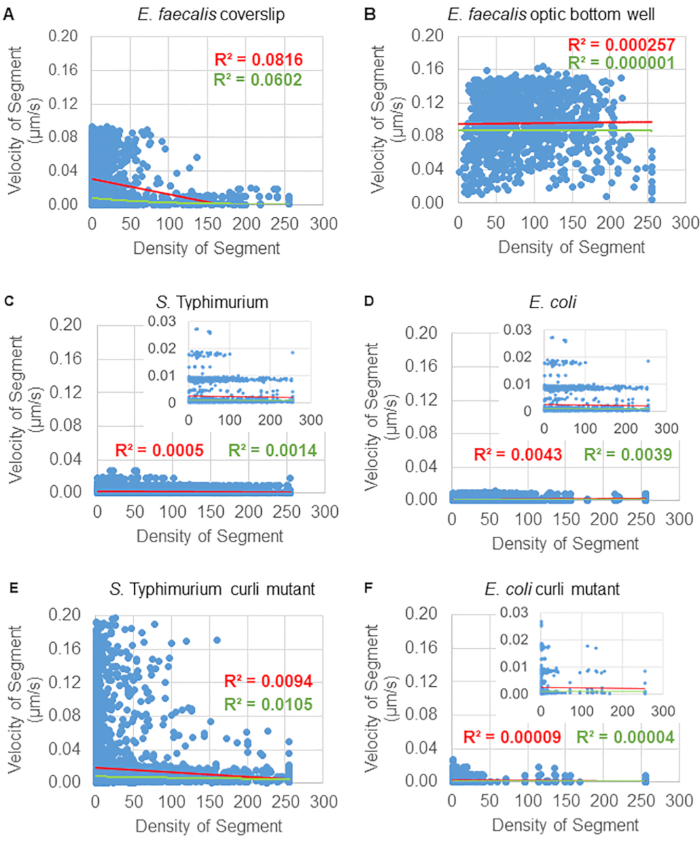
Graphique 6. Étude de l’effet de la densité du biofilm sur la vitesse des billes à l’aide du pipeline d’analyse du biofilm (étape 5). Les résultats ont indiqué que la vitesse des billes n’est pas exclusivement dépendante de la densité cellulaire. Les biofilms ont été cultivés comme décrit à l’étape 1. Les trajectoires ont été analysées à l’échelle de segments de trajectoire individuels (Figure 5). Pour chaque segment, la vitesse de la bille en μm/s a été tracée en fonction de la densité cellulaire de la boîte englobante (GFP moyenne par voxel à l’intérieur de la boîte englobante). La ligne rouge montre la régression linéaire et la ligne verte la trace de régression exponentielle. Cette figure est reproduite avec la permission de (31). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Vidéo supplémentaire 1. Vidéo 4D d’un biofilm OG1RF d’E. faecalis de 24 heures cultivé sur une lamelle en verre optique de 1,5 d’épaisseur. La vidéo 4D en accéléré a été générée à l’aide d’un microscope à une résolution de 512x512. Une série Z de 40 images a été générée en imageant une région d’environ 20 m d’épaisseur d’un biofilm par pas de 0,5 m. Chaque série Z était d’une image et nécessitait 50 à 60 s pour être capturée. Une série de 20 images contiguës a été capturée pour produire une vidéo 4D. La lecture vidéo est d’environ 120x. La vidéo est représentative d’au moins 6 expériences indépendantes. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire 2. Vidéo 4D d’un biofilm OG1RF d’E. faecalis de 24 heures cultivé sur une plaque inférieure optique à 96 puits. La vidéo 4D en accéléré a été générée à l’aide d’un microscope à une résolution de 512x512. Une série Z de 40 images a été générée en imageant une région d’environ 20 m d’épaisseur d’un biofilm par pas de 0,5 m. Chaque série Z était d’une image et nécessitait 50 à 60 s pour être capturée. Une série de 20 images contiguës a été capturée pour produire une vidéo 4D. La lecture vidéo est d’environ 120x. La vidéo est représentative de 3 expériences indépendantes.Veuillez cliquer ici pour télécharger ce vidéo.
Vidéo supplémentaire 3. Vidéo 4D d’un biofilm ATCC 14028 de sérotype de Salmonella enterica cultivé sur des lamelles en verre optique de 1,5 d’épaisseur pendant 6 à 7 jours. La vidéo 4D en accéléré a été générée à l’aide d’un microscope à une résolution de 512x512. Une série Z de 40 images a été générée en imageant une région d’environ 20 m d’épaisseur d’un biofilm par pas de 0,5 m. Chaque série Z était d’une image et nécessitait 50 à 60 s pour être capturée. Une série de 20 images contiguës a été capturée pour produire une vidéo 4D. La lecture vidéo est d’environ 120x. La vidéo est représentative de 3 expériences indépendantes.Veuillez cliquer ici pour télécharger ce vidéo.
Vidéo supplémentaire 4. Vidéo 4D du sérotype de Salmonella enterica Les biofilms ATCC 14028 curli (csgBA) mutants ont été cultivés sur des lamelles en verre optique de 1,5 épaisseur pendant 6 à 7 jours. La vidéo 4D en accéléré a été générée à l’aide d’un microscope à une résolution de 512x512. Une série Z de 40 images a été générée en imageant une région d’environ 20 m d’épaisseur d’un biofilm par pas de 0,5 m. Chaque série Z était d’une image et nécessitait 50 à 60 s pour être capturée. Une série de 20 images contiguës a été capturée pour produire une vidéo 4D. La lecture vidéo est d’environ 120x. La vidéo est représentative de 3 expériences indépendantes.Veuillez cliquer ici pour télécharger ce vidéo.
Vidéo supplémentaire 5. Vidéo 4D d’E. coli UTI89 cultivé sur des lamelles en verre optique de 1,5 d’épaisseur pendant 6 à 7 jours. La vidéo 4D en accéléré a été générée à l’aide d’un microscope à une résolution de 512x512. Une série Z de 40 images a été générée en imageant une région d’environ 20 m d’épaisseur d’un biofilm par pas de 0,5 m. Chaque série Z était d’une image et nécessitait 50 à 60 s pour être capturée. Une série de 20 images contiguës a été capturée pour produire une vidéo 4D. La lecture vidéo est d’environ 120x. La vidéo est représentative de 3 expériences indépendantes.Veuillez cliquer ici pour télécharger ce vidéo.
Vidéo supplémentaire 6. Vidéo 4D E. coli UTI89 curli (csgBA) cultivé sur des lamelles en verre optique de 1,5 épaisseur pendant 6 à 7 jours. La vidéo 4D en accéléré a été générée à l’aide d’un microscope à une résolution de 512x512. Une série Z de 40 images a été générée en imageant une région d’environ 20 m d’épaisseur d’un biofilm par pas de 0,5 m. Chaque série Z était d’une image et nécessitait 50 à 60 s pour être capturée. Une série de 20 images contiguës a été capturée pour produire une vidéo 4D. La lecture vidéo est d’environ 120x. La vidéo est représentative de 3 expériences indépendantes.Veuillez cliquer ici pour télécharger ce vidéo.
Discussion
Étapes critiques et dépannage
Le plus grand défi de cette technique est d’utiliser une lamelle portée avec un biofilm très visqueux comme E. faecalis. La lamelle doit être placée avec soin et précision sur la glissière multipuits sans la repositionner. Pendant l’étape de scellement, il faut veiller à ne pas appuyer sur la lamelle ou à la pousser/glisser accidentellement sur la surface de la glissière. Tout mouvement ou pression peut créer une tension superficielle et bloquer le mouvement d’un biofilm visqueux. Si possible, la comparaison des propriétés d’un matériau de biofilm en imageant un biofilm sur un puits de fond optique à une monture de lamelle permettra d’évaluer la technique. Lorsqu’il était correctement exécuté, un support de lamelle ressemblait beaucoup à un biofilm dans une plaque inférieure optique pour E. faecalis.
De plus, lors de l’utilisation d’une lamelle montée, il faut éviter l’imagerie des interfaces du biofilm avec la lamelle en bas ou la lame tout en haut. Lors de l’utilisation d’une lunette inversée, avec la lamelle en bas, il peut y avoir des billes piégées à la base du biofilm contre la lamelle. Ces billes traversent le biofilm et se coincent contre la lamelle même après un lavage doux. Ils ont des coordonnées x, y et z de 0 et des coordonnées de boîte englobante de 0. Cependant, pour certaines applications, telles que l’examen de l’intégrité du biofilm après traitement, ces points de données peuvent être utilisés comme un outil. La capacité des billes à pénétrer à travers une région épaisse de biofilm jusqu’au fond de la lamelle peut être utilisée pour évaluer l’intégrité du biofilm après traitement (manuscrit en préparation en collaboration avec le laboratoire Tükel). Au sommet du biofilm, dans un biofilm visqueux comme E. faecalis , nous avions des signes de compactage imposés par la lamelle. Cela a limité le mouvement de certaines billes à l’interface de la lame de verre et a peut-être introduit une certaine dépendance de la densité dans l’analyse du mouvement des billes.
Les étapes de lavage étaient nécessaires pour les biofilms car le milieu de croissance présente une forte autofluorescence dans le canal vert. Nous choisissons d’utiliser des billes en excès et d’enlever les billes non associées par lavage afin de maximiser les billes associées afin d’obtenir la caractérisation la plus précise des régions observées.
Le nombre de billes et de lavages nécessaires pour obtenir les ensembles de données souhaités doit être déterminé empiriquement. La présence d’un trop grand nombre de billes dans un biofilm génère des ensembles de données incroyablement volumineux et difficiles à analyser. La présence d’un nombre insuffisant de billes ne génère pas un échantillonnage complet des environnements de biofilm. Cependant, le contrôle du nombre de billes ajoutées (2 x 107 billes dans 1 mL de PBS) et l’utilisation d’étapes de lavage ont permis d’obtenir un nombre relativement constant de billes (40 à 140) associées au biofilm en fonction de sa structure, de sa disposition spatiale et de sa composition.
Lors de l’étude de biofilms avec des régions visqueuses et rigides mixtes, les billes peuvent se coincer dans les régions rigides au fil du temps. Dans ce cas, l’imagerie doit être commencée immédiatement après l’ajout des billes. Cela ne peut souvent pas être accompli à l’aide de lamelles, mais nécessite des plaques inférieures optiques ou des cellules d’écoulement où l’imagerie peut être effectuée immédiatement après l’ajout des billes et la ou les étapes de lavage.
Modifications et applications futures
Utilisation de dispositifs microfluidiques. Dans nos études, les conditions optimales établies pour l’étude des biofilms d’entérobactéries nécessitaient la croissance du biofilm sous forme de pellicule à l’interface air-liquide. Cela a limité l’utilisation de plaques inférieures optiques et de dispositifs microfluidiques dans les études. Cependant, lorsque les conditions de formation du biofilm le permettent, les biofilms peuvent être cultivés dans des chambres microfluidiques ou des cellules d’écoulement. Les biofilms ont ensuite pu être lavés et les billes introduites à travers le dispositif microfluidique avec une perturbation minimale du biofilm.
Ajout de billes lors de la croissance du biofilm. Nous avons choisi d’ajouter des billes en excès aux biofilms, puis d’éliminer les billes non associées par un lavage doux afin d’optimiser le nombre de billes présentes lors de l’analyse. Dans les biofilms visqueux d’E. faecalis , il est possible que les billes se soient dissociées et se soient réassociées pendant le temps d’imagerie de 20 minutes. Si un petit nombre de billes est ajouté à différents moments de la croissance du biofilm, il pourrait être possible de piéger les billes dans le biofilm, ce qui permettrait une caractérisation plus précise du mouvement du biofilm dans les biofilms plus visqueux.
Choix de la région à imager. Pour les études sur les propriétés des matériaux, il est préférable de choisir des régions épaisses et minces du biofilm. Cependant, lors de l’étude des changements dans les propriétés des matériaux d’un biofilm traité, des régions confluentes épaisses peuvent être imagées pour déterminer les changements dans les propriétés viscoélastiques et la pénétration des billes dans ces régions. Dans ce cas, la recherche de billes qui ont pénétré dans le biofilm et qui se sont retrouvées coincées contre la lamelle est une mesure utile de la perturbation du biofilm.
Imagerie sous flux. À l’aide de cellules d’écoulement en verre optique ou de dispositifs microfluidiques, le mouvement des billes ou des bactéries dans un biofilm sous écoulement peut être imagé. Cela peut se faire de différentes manières. Cela peut se faire par injection de billes dans toute la chambre suivie d’une brève incubation pour permettre l’association des billes avec le biofilm. Les billes non associées peuvent être éliminées par lavage et le biofilm peut être imagé avec ou sans flux. À l’inverse, un petit nombre de billes peut être introduit d’un côté de la chambre et leur mouvement à travers et dans le biofilm peut être suivi sous l’effet de l’écoulement. Lors de l’utilisation du flux, il faudra faire preuve de prudence lors du choix des paramètres de suivi des perles de la mosaïque (étape 4.5). Dans les études actuelles, le cadre dynamique était brownien. Les calculs MSD ont confirmé que le mouvement était susceptible d’être diffusif, ce qui fait du brownien le cadre approprié.
Coloration matricielle. Dans les études actuelles, la coloration avec Syto9 examine la densité cellulaire et non la densité de la structure du biofilm. Par exemple, la présence d’amyloïdes augmente probablement la densité du matériau matriciel du biofilm. La dépendance du mouvement à la densité amyloïde a pu être déterminée en utilisant des colorants à matrice fluorescente au lieu de Syto9.
Bactéries marquées par fluorescence. Les bactéries marquées par fluorescence peuvent être utilisées pour suivre le mouvement des bactéries exogènes à travers les biofilms (p. ex., les bactéries contenant des plasmides). Le problème avec les bactéries marquées par fluorescence, telles que les entérocoques, est qu’elles forment des simples, des diplocoques et des chaînes courtes, ce qui complique la capacité de suivre avec précision les bactéries. Ce processus serait plus facile si les bactéries avaient une morphologie unicellulaire.
Limitations
Limitations dans la visualisation de la trajectoire et l’assemblage.
L’une des limites de la méthode est la visualisation de la trajectoire et l’assemblage. Les trajectoires reconstruites et analysées sont constituées de coordonnées de points x, y, z, où les points suivants définissent le chemin linéaire entre ces points. La visualisation de telles trajectoires linéaires par morceaux peut être réalisée à l’aide de divers outils. Une approche consistait à utiliser des notebooks Python et Jupyter avec les plugins Python, Pandas et Matplotlib. Bien qu’il ait été possible de visualiser les trajectoires individuelles existantes dans l’article du Journal of Bacteriology où cette technique a été publiée à l’origine34, il y avait encore des limites importantes qui sont abordées dans les recherches futures.
Actuellement, le nombre de trajectoires reconstruites est supérieur au nombre de billes dans le biofilm, ce qui signifie que plusieurs trajectoires peuvent correspondre à une seule bille. Cela peut être causé par un signal confocal faible dans une image où Mosaic terminera une trajectoire et en initiera une seconde. Cela peut se traduire par plusieurs trajectoires plus courtes pour une seule bille, en particulier dans les biofilms moins visqueux. Une autre cause du grand nombre de trajectoires est l’absence de couture de trajectoire. En particulier dans les biofilms de fond optique d’E. faecalis , les billes restent visuellement associées au biofilm pendant l’imagerie (vidéo supplémentaire 2). Cependant, il n’y avait pas de trajectoires de plus de 10 et plus de 90 % des trajectoires avaient une durée de vie de 5 images ou moins (figure 3D). Si le logiciel est utilisé pour analyser uniquement les trajectoires supérieures à une longueur définie (par exemple, lors du traçage de cellules capables de transférer des plasmides), les trajectoires plus courtes peuvent être automatiquement supprimées de l’ensemble de données. Cependant, il existe d’autres objectifs pour lesquels la couture des trajectoires peut être très importante. Enfin, l’incapacité de suivre le mouvement rapide des billes en une seule trajectoire pourrait entraîner un plus grand nombre de trajectoires dans les biofilms d’Enterobacteriaceae en raison du mouvement rapide dans la direction Z entraînant des billes de forme elliptique (figure 2G). La possibilité de coudre des trajectoires perturbées par un mouvement anisotrope rapide sera importante pour étudier l’effet de la matrice amyloïde curli chez les entérobactéries.
Importance
Un flux de travail informatique a été développé pour étudier les trajectoires des billes afin de comparer les propriétés des matériaux des biofilms 3D. Le flux de travail permet aux chercheurs d’identifier des paramètres critiques qui peuvent être utilisés dans la modélisation computationnelle de la dynamique des fluides dans des biofilms hétérogènes. Avec l’aide de cet évaluateur de billes open-source, l’effet des boucles amyloïdes bactériennes sur les propriétés du matériau a pu être étudié, montrant une rigidité accrue de la matrice du biofilm due aux boucles. Dans un contexte plus général, l’évaluateur peut être utilisé pour étudier les modifications de la structure du biofilm induites par le traitement du biofilm ou différentes conditions environnementales, telles que l’écoulement. Par exemple, l’outil est utilisé pour analyser l’effet du traitement par anticorps monoclonaux sur la perturbation des structures du biofilm en collaboration avec le laboratoire Tükel (LKSOM Temple University). La boîte à outils de l’évaluateur de billes est entièrement adaptable et extensible de manière modulaire à l’aide de VRL-Studio pour améliorer et étendre encore ses fonctions.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Le travail dans les laboratoires GQ et BAB n’a reçu aucune subvention spécifique d’un organisme de financement des secteurs public, commercial ou à but non lucratif. Les auteurs remercient Isaac Klapper, Ph.D (Département de mathématiques, Université Temple) pour la discussion utile et Çagla Tükel (Département de microbiologie et d’immunologie, Université Temple) pour son expertise dans les entérobactéries dans la publication initiale de contenir cette technique.
matériels
| Name | Company | Catalog Number | Comments |
| 96-well plates, No. 1.5 Uncoated Coverslip, 5 mm Glass Diameter | MatTek | P96G1.55F | |
| Fisherbrand Cover Glasses: Circles | Fisher Scientific | 12-293-232P | 1.5 optic glass coverslip |
| Invitrogen Syto 9 Green Fluorescent Nucleic Acid Stain | Invitrogen | S34854 | |
| Molecular Probes FluoSpheres Carboxylate-modified Microspheres, 1 um, crimson fluorescent (625/645) | Molecular Probes | F8816 |
Références
- Huang, R., Li, M., Gregory, R. L. Bacterial interactions in dental biofilm. Virulence. 2, 435-444 (2011).
- Nadell, C. D., Drescher, K., Wingreen, N. S., Bassler, B. L. Extracellular matrix structure governs invasion resistance in bacterial biofilms. ISME Journal. 9, 1700-1709 (2015).
- Billings, N., Birjiniuk, A., Samad, T. S., Doyle, P. S., Ribbeck, K. Material properties of biofilms - key methods for understanding permeability and mechanics. Reports on Progress in Physics. 78, 036601 (2015).
- Araújo, G. R. d. e. S., Viana, N. B., Gómez, F., Pontes, B., Frases, S. The mechanical properties of microbial surfaces and biofilms. The Cell Surface. 5, 100028 (2019).
- Tallawi, M., Opitz, M., Lieleg, O. Modulation of the mechanical properties of bacterial biofilms in response to environmental challenges. Biomaterials Science. 5, 887-900 (2017).
- Tursi, S. A., Tükel, &. #. 1. 9. 9. ;. Curli-Containing Enteric Biofilms Inside and Out, Matrix Composition, Immune Recognition, and Disease Implications. Microbiology and Molecular Biology Reviews. 82, (2018).
- Dueholm, M. S., Albertsen, M., Otzen, D., Nielsen, P. H. Curli functional amyloid systems are phylogenetically widespread and display large diversity in operon and protein structure. PLoS One. 7 (12), 51274 (2012).
- Hung, C., et al. Escherichia coli biofilms have an organized and complex extracellular matrix structure. mBio. 4, 00645 (2013).
- Kikuchi, T., Mizunoe, Y., Takade, A., Naito, S., Yoshida, S. Curli fibers are required for development of biofilm architecture in Escherichia coli K-12 and enhance bacterial adherence to human uroepithelial cells. Microbiology and Immunology. 49, 875-884 (2005).
- Gallo, P. M., et al. Amyloid-DNA Composites of Bacterial Biofilms Stimulate Autoimmunity. Immunity. 42, 1171-1184 (2015).
- Serra, D. O., Richter, A. M., Hengge, R. Cellulose as an architectural element in spatially structured Escherichia coli biofilms. Journal of Bacteriology. 195, 5540-5554 (2013).
- Oh, Y. J. Characterization of curli A production on living bacterial surfaces by scanning probe microscopy. Biophysical Journal. 103, 1666-1671 (2012).
- Lembré, P., Di Martino, P., Vendrely, C. Amyloid peptides derived from CsgA and FapC modify the viscoelastic properties of biofilm model matrices. Biofouling. 30, 415-426 (2014).
- Oh, Y. J., et al. Curli mediate bacterial adhesion to fibronectin via tensile multiple bonds. Scientific Reports. 6, 33909 (2016).
- Vidakovic, L., Singh, P. K., Hartmann, R., Nadell, C. D., Drescher, K. Dynamic biofilm architecture confers individual and collective mechanisms of viral protection. Nature Microbiology. 3, 26-31 (2018).
- Tursi, S. A., et al. Salmonella Typhimurium biofilm disruption by a human antibody that binds a pan-amyloid epitope on curli. Nature Communications. 11, 1007 (2020).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15 (8), 1007978 (2019).
- Taglialegna, A., et al. The biofilm-associated surface protein Esp of Enterococcus faecalis forms amyloid-like fibers. Npj Biofilms and Microbiomes. 6, 15 (2020).
- Gour, S., Kumar, V., Rana, M., Yadav, J. K. Pheromone peptide cOB1 from native Enterococcus faecalis forms amyloid-like structures, A new paradigm for peptide pheromones. Journal of Peptide Science. 25, 3178 (2019).
- Stoodley, P., Debeer, D., Lewandowski, Z. Liquid flow in biofilm systems. Applied and Environmental Microbiology. 60, 2711-2716 (1994).
- Birjiniuk, A., et al. Single particle tracking reveals spatial and dynamic organization of the E. coli biofilm matrix. New Journal of Physics. 16, 085014 (2014).
- Chew, S. C., et al. Dynamic remodeling of microbial biofilms by functionally distinct exopolysaccharides. mBio. 5, 01536 (2014).
- Cao, H., et al. Revealing region-specific biofilm viscoelastic properties by means of a micro-rheological approach. Npj Biofilms and Microbiomes. 2, 5 (2016).
- Galy, O., et al. Mapping of bacterial biofilm local mechanics by magnetic microparticle actuation. Biophysical Journal. 103, 1400-1408 (2012).
- Rogers, S. S., vander Walle, C., Waigh, T. A. Microrheology of bacterial biofilms in vitro, Staphylococcus aureus and Pseudomonas aeruginosa. Langmuir. 24, 13549-13555 (2008).
- Hart, J. W., Waigh, T. A., Lu, J. R., Roberts, I. S. Microrheology and Spatial Heterogeneity of Staphylococcus aureus Biofilms Modulated by Hydrodynamic Shear and Biofilm-Degrading Enzymes. Langmuir. 35 (9), 3553-3561 (2019).
- van Merode, A. E. J., van der Mei, H. C., Busscher, H. J., Krom, B. P. Influence of culture heterogeneity in cell surface charge on adhesion and biofilm formation by Enterococcus faecalis. Journal of Bacteriology. 188, 2421-2426 (2006).
- Tariq, M., Bruijs, C., Kok, J., Krom, B. P. Link between Culture Zeta Potential Homogeneity and Ebp in Enterococcus faecalis. Applied and Environmental Microbiology. 78, 2282-2288 (2012).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ, 25 years of image analysis. Nature Methods. 9, 671-675 (2012).
- . Oracle.com Available from: https://docs.oracle.com/en/java (2021)
- Hoffer, M., Poliwoda, C., Wittum, G. Visual reflection library, a framework for declarative GUI programming on the Java platform. Computing and Visualization in Science. 16, 181-192 (2013).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15, 1007978 (2019).
- Malhotra, K., et al. Development of a New Bead Movement-Based Computational Framework Shows that Bacterial Amyloid Curli Reduces Bead Mobility in Biofilms. Journal of Bacteriology. 202, 00253 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon