Method Article
使用短读和长读测序技术生成尿细菌完整基因组的杂交从 头 基因组组装
摘要
该协议详细介绍了尿细菌培养,测序和 从头 杂交基因组组装的综合方法。它为生成完整的圆形基因组序列提供了一种可重复的程序,可用于研究有助于尿定植、发病机制和抗菌素耐药性传播的染色体和染色体外遗传元件。
摘要
完整的基因组序列为了解泌尿微生物的遗传多样性和独特定植因子提供了有价值的数据。这些数据可能包括移动遗传元件,如质粒和染色体外噬菌体,它们有助于抗菌素耐药性的传播,并使尿路感染(UTI)的治疗进一步复杂化。除了提供基因组结构的精细分辨率外,完整的闭合基因组还允许进行详细的比较基因组学和进化分析。由于现有测序技术的局限性 ,从头开始 生成完整基因组长期以来一直是一项具有挑战性的任务。配对端下一代测序(NGS)产生高质量的短读数,通常导致准确但片段化的基因组组装。相反,纳米孔测序提供质量较低的长读数,通常会导致容易出错的完整组件。这些错误可能会妨碍全基因组关联研究或提供误导性的变异分析结果。因此,结合短读和长读的混合方法已成为实现高度准确的闭合细菌基因组的可靠方法。本文报道的用于培养多种尿菌的综合方法,通过16S rRNA基因测序进行物种鉴定,提取基因组DNA(gDNA),以及分别通过NGS和Nanopore平台生成短读和长读。此外,该方法还描述了用于生成注释的完整基因组序列的质量控制,组装和基因预测算法的生物信息学管道。生物信息学工具的组合可以选择高质量的读取数据,用于杂交基因组组装和下游分析。该协议中描述的用于杂交 从头 基因组组装的简化方法可以适用于任何可培养细菌的使用。
引言
泌尿微生物组是一个新兴的研究领域,它打破了长达数十年的误解,即尿路在健康个体中是无菌的。尿微生物群的成员可用于平衡尿液环境并预防尿路感染(UTI)1,2。泌尿致病细菌侵入尿路,并采用多种毒力机制来取代常驻微生物群,定植尿路上皮,逃避免疫反应并抵消环境压力3,4。尿液是一种营养相对有限的培养基,其特征在于高渗透压,有限的氮和碳水化合物可用性,低氧合和低pH 5,6,7。尿液也被认为是抗菌的,由高浓度的抑制性尿素和抗菌肽如人cathelicidin LL-378组成。调查常驻细菌和泌尿道病原体在尿路定植的机制对于进一步了解尿路健康和制定UTI治疗的新策略至关重要。此外,随着一线抗菌疗法的失败变得越来越普遍,监测携带抗菌素耐药性决定因素的移动遗传元件在尿细菌种群内的传播变得越来越重要9,10。
为了研究尿细菌的基因型和表型,它们的成功培养和随后的全基因组测序(WGS)势在必行。培养依赖性方法对于检测和鉴定尿液样品中的活微生物是必要的11。标准临床尿培养包括将尿液接种到5%绵羊血琼脂(BAP)和麦康基琼脂上,并在35°C下有氧孵育24小时12。然而,由于检测阈值为≥105 CFU / mL13,许多尿微生物群的成员没有通过这种方法报告。改进的培养技术,如增强定量尿培养(EQUC)11, 采用不同尿量,孵育时间,培养基和大气条件的各种组合来鉴定标准尿培养物通常遗漏的微生物。该协议中描述的是EQUC的修改版本,在这里称为改良增强尿培养方案,其能够使用选择性培养基和最佳大气条件培养各种尿细菌和尿路病原体,但不是固有的定量。尿液细菌的成功分离使得能够提取用于下游WGS和基因组组装的基因组DNA(gDNA)。
基因组组装,特别是完整的组装,能够发现可能有助于常驻微生物群和泌尿致病细菌的定植,生态位维持和毒力的遗传因素。基因组组装草案包含大量连续序列(重叠群),这些序列可能包含测序错误并且缺乏方向信息。在完整的基因组组装中,每个碱基对的方向和准确性都得到了验证14.此外,获得完整的基因组序列可以深入了解基因组结构,遗传多样性和移动遗传元件15。仅短读物可以识别重要基因的存在与否,但可能无法确定其基因组背景16。随着牛津纳米孔和PacBio等长读测序技术的实现,生成细菌基因组的闭合从头组装不再需要费力的方法,例如通过多重PCR17,18手动关闭从头组装。下一代短读测序和纳米孔长读测序技术的结合允许以相对较低的成本轻松生成准确,完整和封闭的细菌基因组组装19。短读测序产生准确但碎片化的基因组组装体,通常平均由40-100个重叠群组成,而Nanopore测序产生长度约为5-100 kb的长读数,这些读数不太准确,但可以作为支架加入重叠群并解析基因组合成。利用短读和长读技术的混合方法可以产生准确和完整的细菌基因组19。
这里描述的是一个全面的方案,用于从人尿液中分离和鉴定细菌,基因组DNA提取,测序和使用混合组装方法的完整基因组组装。该协议特别强调了正确修改由短读和长读测序生成的读数所需的步骤,以便准确组装封闭的细菌染色体和染色体外元件(如质粒)。
研究方案
作为机构审查委员会批准的研究19MR0011(UTD)和STU 032016-006(UTSW)的一部分,从同意妇女收集的尿液中培养细菌。
1. 改良增强型尿培养
注意:所有培养步骤必须在无菌条件下进行。对所有器械、溶液和培养基进行灭菌。用70%乙醇清洁工作区域,然后设置本生燃烧器,并在靠近火焰的地方小心地工作,以减少污染的机会。或者,可以使用II类生物安全柜来维持无菌环境。穿戴适当的个人防护装备(PPE),以避免接触潜在的致病微生物。
- 接种甘油储备尿液和集落分离
- 在室温(RT)下解冻储存甘油的尿液。解冻后,涡旋样品5秒以混合。在无菌微量离心管中,在无菌1x磷酸盐缓冲盐水(PBS)中制备1:3和1:30稀释的尿液,最终体积为100μL。
注意:将500μL未稀释的尿液和500μL50%无菌甘油混合在冷冻管中并储存在-80°C下来制备甘油储备尿液。 - 使用前在37°C下预热琼脂平板15分钟。请参阅 图1, 了解适合常见尿细菌属的培养基类型和培养条件。在接种前通过移液将稀释的尿液混合均匀,将稀释的尿液平板100μL放在所需的琼脂平板上,并使用无菌玻璃珠传播样品。将100μL的1x PBS稀释剂的板放在单独的板上作为无生长对照。
注意:如果试图培养常见的泌尿病理性物种(例如, 大肠杆菌,克雷伯氏菌属,粪肠球菌 等),建议使用显色琼脂(材料表),因为它可以轻松识别泌尿病源性细菌物种(图1)。粘菌素纳利迪酸 (CNA) 或 MRS 琼脂有助于从已知含有革兰氏阴性尿路病原体的尿液中分离出挑剔的革兰氏阳性种属(例如 乳酸杆菌属),这些菌株在非选择性琼脂中可能优于挑剔的种类。 - 将板倒置在35°C的所需大气条件下孵育24小时,用于泌尿道病原体,3-5天用于挑剔细菌(图1)。
- 孵育期过后,从培养箱中取出板。从每个板中,选择表现出独特颜色,形态或溶血模式的菌落。
- 使用无菌环将细菌菌落重新划到相应的琼脂上,并将平板倒置在所需气氛中孵育2-5天,以获得良好分离的菌落。
注意:如果使用BAP进行原代培养,则在显色琼脂上修补菌落可能提供有关样品中细菌群异质性的有用信息。
- 在室温(RT)下解冻储存甘油的尿液。解冻后,涡旋样品5秒以混合。在无菌微量离心管中,在无菌1x磷酸盐缓冲盐水(PBS)中制备1:3和1:30稀释的尿液,最终体积为100μL。
- 在液体肉汤和甘油放养细菌分离物中培养
- 一旦获得与母体菌落形态相匹配的分离菌落,选择单个菌落并使用无菌接种回路接种到3mL液体肉汤中。参见 图1, 了解能够支持普通尿液微生物群属生长的肉汤。用石膏膜密封琼脂板,并将其储存在4°C下2-4天。将液体培养物在所需的大气条件下孵育1-5天,直到培养物明显浑浊。
- 观察生长后,涡旋培养物,然后将1mL过夜培养物加入500μL无菌50%甘油在2mL冷冻管中;密封并通过倒置轻轻混合。为每个菌落准备两个甘油储备(一个用作备用)并储存在-80°C。
2. 通过16S rRNA基因桑格测序鉴定细菌种类
注意:微生物身份可以使用基质辅助激光解吸电离飞行时间质谱(MALDI-TOF)20进行确认。
- 菌落-聚合酶链反应 (PCR)
- 通过加入12.5μL的2x Taq聚合酶预混液,0.5μL的10μM 8F引物,0.5μL的10μM 1492R引物(材料表)和11.5μL无核酸酶水21,在PCR管中制备25μLPCR反应。
注意:如果对多个样品进行PCR,请制作Taq聚合酶混合物,引物和无菌无核酸酶水的反应预混液。然后将25μL等分到每个PCR管中。 - 要进行集落-PCR,请使用无菌牙签或移液器吸头从重新条纹中擦拭分离良好的集落。将菌落重悬于步骤2.1.1制备的PCR反应混合物中。轻轻混合。通过以2000 x g的快速旋转收集管底部的液体。
注:确保样品没有气泡。包括单独含有PCR反应混合物的无模板对照(NTC)样品。 - 将样品管放入热循环器中,并运行以下程序:95°C3分钟;40次循环:95°C持续30秒,51°C持续30秒,72°C持续1分钟30秒;72°C持续10分钟;保持在10°C。
- 通过加入12.5μL的2x Taq聚合酶预混液,0.5μL的10μM 8F引物,0.5μL的10μM 1492R引物(材料表)和11.5μL无核酸酶水21,在PCR管中制备25μLPCR反应。
- 凝胶提取和物种鉴定
- 完成PCR运行后,在0.5x Tris-硼酸盐-EDTA(TBE)缓冲液中制备的1%琼脂糖凝胶上检查PCR产物。在浇注凝胶之前,加入溴化乙锭(EtBr)。然后,使用梳子将凝胶浇铸到至少20μL样品体积的孔中。
注意:EtBr是一种怀疑致癌的插层剂。处理时始终戴上手套和PPE,并根据机构的指导方针处理含有EtBr的材料。 - 凝胶凝固后,将凝胶放入充满0.5x TBE缓冲液的电泳槽中,然后取出梳子。将第一孔中的1 kb分子量标准品和10-20μL的PCR反应加载到后续孔中。以 100-140 V 电压运行,直至解决。在紫外光下观察凝胶,并确认在~1.5 kb处存在NTC孔中不存在的明确定义的条带。
注意:紫外线对皮肤和眼睛有害,在观察凝胶时使用适当的防护装置,并穿戴适当的个人防护用品。
注意:对于某些细菌,集落 PCR 可能不成功。从分离的gDNA进行PCR是替代选择22。 - 使用剃须刀切除~1.5 kb的条带,并将凝胶插条转移到干净的微量离心管中。按照制造商的说明(材料表)继续进行凝胶提取方案。通过微量分光光度计测量纯化DNA的浓度。
注:需要>10 ng/μL的浓度,1.7-2.0之间的A260/280是可以接受的。 - 根据任何所选 Sanger 测序服务的指南,为每个样品准备两个 Sanger 测序反应,一个使用 8F,另一个使用 1492R 引物在无核酸酶水中。
- 收到测序数据后,将DNA序列上传到NCBI基本局部比对搜索工具(BLAST)网站(blast.ncbi.nlm.nih.gov/Blast.cgi),选择核苷酸BLAST(blastn),选择rRNA / ITS数据库16S核糖体RNA序列(细菌和古菌),然后运行Megablast程序。隔离点可以通过对数据库中引用的最高质量命中来识别。
注意:一些细菌物种在其16S rRNA序列中表现出高同一性,并且仅通过这种方法可能无法区分。物种形成将需要DNA同源性和生化分析,以自信地区分同一属23的成员。
- 完成PCR运行后,在0.5x Tris-硼酸盐-EDTA(TBE)缓冲液中制备的1%琼脂糖凝胶上检查PCR产物。在浇注凝胶之前,加入溴化乙锭(EtBr)。然后,使用梳子将凝胶浇铸到至少20μL样品体积的孔中。
3. 基因组DNA(gDNA)的提取
注:本节利用 材料表中 引用的gDNA提取试剂盒中提供的试剂和离心柱,从不同细菌物种中高产提取优质基因组DNA。下面提供了建议的修改和说明。
- 根据制造商的说明准备试剂盒试剂盒。
- 将来自良好分离的菌落的细菌接种到培养基中,并在图1中注明的温度和大气压下孵育直到观察到足够的生长,在适当的无菌肉汤(图1)中制备 3-10mL 培养物。
- 孵育后,使用分光光度计24测量培养物在600nm(OD 600)处的光密度。
- 通过以1:10的比例稀释过夜培养物来制备样品以进行定量。包括无菌培养基的空白,以便测量。通过从样品读数中减去空白读数并乘以稀释因子10来计算光密度。
- 使用OD600 测量和预先建立的OD600 与CFU / mL的物种比率,计算获得2 x 109 个细胞所需的培养物数量。
- 将所需的培养体积在5000×g下离心5分钟至沉淀。 吸出上清液并将沉淀重悬于200μL冷TE缓冲液中(在程序开始时在冰上预冷)。
- 将样品在5000×g下离心2分钟。 除去上清液,然后将沉淀重悬于180μL酶解缓冲液(ELB)中,并加入20μL预煮的RNase A(10mg / mL)。为了有效裂解革兰氏阳性细菌,加入18μL突变溶素(25 kU / mL)。涡旋均匀,然后将样品在37°C的旋转器上孵育2小时。
注意:建议将制造商方案中描述的ELB用于革兰氏阳性和革兰氏阴性细菌。 - 按照制造商的说明继续。
注意:如果需要,重复洗脱步骤一次或两次,以获得额外的gDNA产量。 - 按照第4节中的说明评估提取的gDNA的质量,如果将在1周内使用,则将gDNA储存在4°C。或者,将gDNA保持在-20°C以长期储存。
4. 评估提取的gDNA的质量
- 为了通过凝胶电泳评估质量,如第2.2小节所述制备1%琼脂糖凝胶。在干净的管中制备样品:在副膜上混合1-2μL提取的gDNA和3μL的2x上样染料。上样后运行凝胶,然后在紫外光下可视化。
注意:成功的gDNA提取将通过凝胶顶部的离散条带和最小的涂片来证明(图2A)。拖尾表明剪切。如果没有明显的 gDNA 波段和/或大量涂片,则重复 gDNA 提取。考虑减少 RNase A 和 Proteinase K 的孵育时间。如果观察到1.5-3 kb左右的两个条带,这表明RNA污染(图2B)。准备新鲜的RNase A并重复提取。 - 要使用微量分光光度计评估质量,请使用微量分光光度计测量gDNA浓度和吸光度比A260/280。浓度>50 ng/μL,A260/280在1.7-2.0之间是可以接受的。
注意:低gDNA产量可能是由于低输入,高输入,核酸酶污染,裂解不足。吸光度高于该范围表示RNA污染。如果 gDNA 质量较差,则重复提取。 - 要通过荧光计评估质量,请按照制造商的说明使用高灵敏度测定试剂盒和荧光计仪器(材料表)量化gDNA浓度。需要>50纳克/μL的浓度。
5. 配对端的下一代短读测序和文库制备
注:短读测序可以在不同的读取长度和方向上对各种仪器进行。建议对细菌 WGS 进行 150 bp(300 周期)配对测序。文库制备和测序均可外包给核心设施或商业实验室。
- 根据制造商的说明(材料表)准备测序库。遵循制造商推荐的最终加载库浓度;但是,建议的修改是在1.8 pM下加载池化库,以便在NextSeq仪器上生成最佳读取。
- 虽然是可选的,但使用生物分析仪(材料表)来评估合并文库片段分布,并确保片段大小平均为600 bp。
6. 纳米孔 MinION 测序文库制备
- 根据制造商的协议(材料表)准备测序库。使用两个条形码扩展试剂盒,可在单个流通池上多路复用多达 24 个样品。建议分两部分进行文库制备,一次12个样品,同时多路复用24个样品。所有24个样本可以如下所述合并。
注意:样品可以在完成原生条形码连接后在4°C下储存过夜 - 如有必要,这可在方案中提供停止点。在文库制备方案的天然条形码连接部分结束时,建议将每个样品的等摩尔量汇集到最大DNA质量(ng)可能。- 为此,请根据制造商的说明使用荧光计(材料表)在条形码连接后量化所有样品。估计具有最低dsDNA浓度的样品的体积,然后计算该样品中发现的总dsDNA。使用此数字可确定将汇集在一起的所有其他样品的等摩尔量。
注意:由于等摩尔计算将最大化池化dsDNA的量,从而产生高容量池(>65μL),因此需要清理以浓缩池。
- 为此,请根据制造商的说明使用荧光计(材料表)在条形码连接后量化所有样品。估计具有最低dsDNA浓度的样品的体积,然后计算该样品中发现的总dsDNA。使用此数字可确定将汇集在一起的所有其他样品的等摩尔量。
- dsDNA池清理和浓缩
- 将2.5倍体积的顺磁性珠(材料表)加入DNA池中,然后轻轻轻地轻拂管子以混合内容物。将试管置于室温下旋转器5分钟,以2000×g旋转样品并在磁铁上沉淀。
- 加入250μL新鲜制备的70%乙醇(在无核酸酶的水中),注意不要干扰沉淀。吸出乙醇并重复乙醇洗涤一次。
- 第二次吸气后,以2000×g旋转样品并将其放回磁铁上。移走任何残留的乙醇,让样品干燥约30秒。
- 从磁铁中取出管并将沉淀重悬于60-70μL无核酸酶水中。在室温下孵育2分钟。将样品沉淀在磁铁上,直到洗脱物清晰,然后取出洗脱并转移到干净的1.5mL微量离心管中。
- 使用荧光计定量浓缩池,然后制备等分试样以进入适配器连接步骤:在65μL最终体积中制备700ng样品。将池的其余部分保留在4°C,以便在第一次运行完成后完成第二次运行。
- 按照制造商的指示继续进行适配器连接,并将样品加载到流通池上。启动排序运行。
注意:在上样前,从流通池底液端口吸入空气和~200 μL储存缓冲液。这对于流池引物的成功和样品上样至关重要。通过流通池的底漆口抽取和浇注溶液时,使用p1000移液器和吸头。
- 根据制造商的说明对库进行排序。
- 打开用于排序的操作软件,然后单击"开始"。输入实验的名称,建议的命名法包括运行日期和用户名。单击"继续选择试剂盒",选择使用的相应库制备试剂盒和条形码扩展包,然后单击"继续运行选项"。
- 如果计划为第二次运行准备足够的库,请将运行长度调整为 48 小时(否则,请保留默认的 72 小时)。单击"继续基本呼叫"。
- 检查基本调用选项 "配置:快速基本调用 ",并确保将 "条形码" 设置为 "已启用", 以便输出 FASTQ 文件将修剪条形码序列,并根据条形码解复用到单独的目录中。单击 继续 输出。
- 选择保存输出排序数据的位置。如果仅保存 FASTQ 输出,则预计大约需要 30-50 Gb 的数据,如果还保存 FAST5 输出,则>500 Gb 的数据。取消选中筛选选项 Qscore: 7 |Readlength:如果未筛选 ,如果计划继续进行第 7.2 节中描述的过滤,否则请保留选中状态并将 Readlength 调整为 200。
- 单击" 继续运行安装程序"并查看所有设置。如果设置正确,请单击" 开始",否则单击" 上一步 "并进行任何必要的调整。
- 如果需要,可以按照制造商的说明清洗流通池,并重新装入剩余的池中。对剩余的池重复 6.2 中的步骤,一旦第一次运行完成并且流通池已洗涤。
注:设置第二次运行时,根据制造商对先前在48小时内运行中使用的流通池的建议,将偏置电压调整为-250 mV。
7. 评估和准备阅读材料
注意:推荐的目录结构如图 4 所示。在继续下面的计算步骤之前,创建 在桌面中找到的目录,即Long_Reads,Short_Reads和Trimmed_Reads。
- 短读 (图 3)
注意:短读以 FASTQ 格式生成。这些文件包含每个 FASTQ 的最大读取次数 4000 次。这些通常被压缩(.gz存档)并组织成多个文件。根据平台的不同,条形码通常会被修剪。某些程序接受压缩格式的文件,其他程序可能需要在导入之前提取它们。读数必须通过质量控制(QC)步骤,以确保基因组组装过程中的数据准确性。如果CLC基因组学工作台不可用,则可以使用替代程序来修剪和QC短读,例如Trimomatic25 或 Trim Galore (https://www.bioinformatics.babraham.ac.uk/projects/trim_galore/) 用于修整,FastQC (http://www.bioinformatics.babraham.ac.uk/projects/fastqc/) 用于评估读取质量。建议将平均短读覆盖率(通过将读取次数乘以平均读取长度并除以基因组大小来估计)>100x。- 打开基因组学工作台软件(材料表)并导入所有配对端的短读FASTQ文件。配对的文件将自动生成。
- 在CLC_Data下创建一个新文件夹,方法是单击顶部工具栏上的 "新建 ",然后选择 "文件夹..."以存储文件。根据需要命名文件夹,建议的约定是使用示例 ID。将以下步骤的所有输出保存到此文件夹中。
- 在顶部工具栏上,单击" 导入 "按钮,然后选择 "Illumina...导航到与示例对应的所有短读文件并选定。确保选中配对读取选项,然后取消选中 删除失败的读取 选项。单击" 下一步",选择" 保存",然后再次单击" 下一步 "。选择将导入的文件保存在上一步中创建的新文件夹中,然后单击 完成。
- 为隔离创建所有配对文件的序列列表;这会将读取的数据连接到单个文件中,以便于分析。
- 在顶部工具栏上,单击"新建"按钮,然后选择"序列列表..."在左侧的目录列表中,选择要连接的文件,然后使用箭头将这些文件移动到右侧的选定文件列表中。单击"下一步",选择"保存",然后再次单击"下一步"。选择保存序列列表,然后单击"完成"。
- 生成序列列表后,立即使用示例 ID 对其进行重命名。
- 在序列列表上运行 QC 测序读取 工具:此过程将评估短读 NGS 生成的读取的整体质量参数。
- 在工具箱菜单(左下角窗口)中搜索 "读取排序"工具的 QC。 双击该工具,然后选择要分析的序列列表,然后单击" 下一步"。
- 确保选中所有输出选项,然后选择结果处理下保存。单击"下一步"并指定以保存输出文件,然后单击"完成"。
- 在序列列表上运行 "修剪读取" 工具:将根据质量、长度和模糊性进行修剪。此过程假定在此步骤之前已修剪排序中使用的条形码。
- 在工具箱(左下角窗口)中搜索 "修剪读取" 工具。双击 修剪 读取,然后选择要分析的序列列表,然后单击 下一步。
- 质量修剪:将质量评分限制设置为0.01,并将模糊的核苷酸保留在2。单击"下一步"。
注:参数可由用户自行决定调整;这些是建议的设置。 - 取消选中 自动直读适配器修整 (仅当适配器在导入到 CLC 之前已从读取中修整时才执行此操作)。单击" 下一步 "并选中 "放弃长度以下的读取",使用默认值 15。
- 单击"下一步",选中"创建报告",然后选择"保存"。单击"下一步"并指定保存输出文件的位置。单击"完成"。
- 导出修剪的序列列表:后续的混合组装和分析将在 CLC 之外完成,并且需要导出修剪的短读文件。
- 从左上角的目录导航中,选择在步骤 7.1.4 中生成的已修剪文件,然后单击顶部工具栏上的" 导出 "。选择 Fastq 作为导出文件类型,然后单击 "下一步"。选中 将配对序列列表 导出到两个文件。然后,单击" 下一步 "并选择要将文件导出到的Trimmed_Reads目录。单击 "完成"。确保已修整的短读文件已成功导出为扩展名为 .fastq的两个文件(R1 和 R2)。
注意:修剪后的序列列表必须导出为两个文件,CLC 通常将其指定为 R1 和 R2。这一点至关重要,因为下游混合组件需要这样设置短读数据输入。 - 重命名导出的文件,请不要在文件名中使用空格和特殊字符。为简单起见,建议的格式trimmed_short_file。R1.fastq.
- 从左上角的目录导航中,选择在步骤 7.1.4 中生成的已修剪文件,然后单击顶部工具栏上的" 导出 "。选择 Fastq 作为导出文件类型,然后单击 "下一步"。选中 将配对序列列表 导出到两个文件。然后,单击" 下一步 "并选择要将文件导出到的Trimmed_Reads目录。单击 "完成"。确保已修整的短读文件已成功导出为扩展名为 .fastq的两个文件(R1 和 R2)。
- 打开基因组学工作台软件(材料表)并导入所有配对端的短读FASTQ文件。配对的文件将自动生成。
- 长(小黄人)读取(图3)
注意:以下用于准备混合组装的Long(MinION)测序读取的管道利用了由命令行执行的NanoFilt和Nanostat程序26。 在继续之前安装这些工具,并熟悉 UNIX 的基础知识以执行这些命令。建议使用默认终端和 Bash Shell。常见终端命令和用法的课程指南可在 软件木工27中找到。以下说明假定生成的文件将使用条形码命名法(NB01,NB02等)命名,并保存在Long_Reads目录中。或者,可以在设置排序运行时使用 MinKNOW 完成读取筛选。建议平均长读取覆盖率为 >100 倍。建议的平均读取长度为 >2000 bp;因此,所需的长读取数低于短读取数。- 在Long_Reads目录中为运行中使用的每个条形码(条形码01,条形码02等)创建新目录(图4)。将与每个条形码对应的所有 .fastq 文件复制到相应的文件夹中。合并每次运行的每个条形码的所有 .fastq 文件。
- 打开 终端 并使用 cd 命令导航到Long_Reads目录中的条形码目录 :cd Desktop/Long_Reads/barcode01
- 通过执行以下命令,将每个条形码的所有 .fastq 文件连接成一个 .fastq 文件 :cat *.fastq > NB01.fastq
注意:此命令将每个 FASTQ 文件中的所有读取内容合并到一个名为 NB01.fastq 的大型单 FASTQ 中。 - 使用 NanoStat 通过执行以下命令来评估样本的读取质量 :NanoStat --fastq NB01.fastq
- 通过将输出复制到文本或 Word 文件中以供将来参考来记录结果。
- 使用 NanoFilt 通过执行以下命令来过滤 MinION 读取丢弃的读数,Q < 7 且长度< 200:NanoFilt -q 7 -l 200 bp NB01.fastq | gzip > NB01 _trimmed.fastq.gz
- 通过执行命令在步骤 7.2.6 中生成的修剪过的文件上运行NanoStat:NanoStat --fastq NB01 _trimmed.fastq.gz
- 通过将输出复制到文本或Word文件中来记录结果,并与步骤7.2.4中的结果进行比较,以确保筛选成功(表1)。
- 对排序运行中使用的每个条形码重复步骤 7.2.2 到 7.2.8。
注意:在步骤 7.2.6 中生成的 NB01_trimmed.fastq.gz 文件将用于混合程序集。
8. 生成杂交基因组组装
注:以下装配管线利用独轮车19、28、29、30组合了第7.1节和第7.2节中准备的短读和长读(图3)。安装独轮车及其依赖项并执行以下命令。在步骤 7.1.5 中导出的短读文件假定trimmed_short_file命名。R1.fastq和trimmed_short_file。R2.fastq 用于简化。
- 将短读文件和长读文件组织到名为 Trimmed_Reads 的目录中。该目录必须包含以下内容:
- 用于修剪长读取 的 .fastq.gz 文件(在步骤 7.2.6 中生成)。
- 两个 .fastq 文件(R1 和 R2),用于修整的短读(在步骤 7.1.5 中生成)。
- 导航到使用终端中的 cd 命令存储已读文件的目录 Trimmed_Reads:cd 桌面/Trimmed_Reads
- 进入正确的目录后,通过执行以下命令,压缩两个短读取文件,使它们也采用 .fastq.gz 格式 :gzip trimmed_short_file。R1.快餐
- 对 R1 和 R2 重复步骤 8.2。检查所有读取的文件现在是否都采用 .fastq.gz 格式,并验证所有文件是否都与同一隔离点匹配。
- 通过运行以下命令,使用独轮车开始混合程序集:
独轮车-1 trimmed_short_file。 R1.fastq.gz -2 trimmed_short_file。R2.fastq.gz -l NB01 _trimmed.fastq.gz -o unicycler_output_directory
注意:-o 指定将保存独轮车输出的目录,执行命令后,独轮车将创建此目录;不要事先生成目录。运行时间因所用计算机的计算能力以及基因组大小和读取次数而异。这可能需要4小时到1或2天不等。该协议是在具有250 Gb RAM的CentOS Linux 7机器,具有2.5 GHz 12个实用内核和48个虚拟内核的Intel Xeon (R)CPU上执行的。或者,具有 16 Gb RAM 和 2.6 GHz 6 核处理器的个人计算机可以在更长的处理时间内计算这些程序集。 - 运行完成后,查看独轮车.log文件以确保没有错误 - 记录生成的重叠群的数量、大小和状态(完整、不完整)。
- 如果标识了不完整的重叠群(在独轮车日志中表示为不完整),请通过在步骤 8.4 中的命令中添加以下标志,以粗体模式重新运行独轮车:--mode bold。
注意:粗体模式将降低装配过程中接受的长读桥接的质量阈值;这可能会产生完整的装配,但装配质量可能会降低。建议仅在必要时使用粗体模式,并作为重叠群连接的初步证据,以便稍后通过PCR确认。
- 如果标识了不完整的重叠群(在独轮车日志中表示为不完整),请通过在步骤 8.4 中的命令中添加以下标志,以粗体模式重新运行独轮车:--mode bold。
9. 评估装配质量
注意:以下协议使用Bandage31 和QUAST32,这两个程序必须在使用前设置(图2 和 图4)。下载后不需要安装绷带,QUAST需要熟悉基本的命令行用法。还建议使用基准通用单拷贝正交学(BUSCO)33评估基因组完整性。
- 绷带: 点击 文件.然后,选择" 荷载图 "并选择保存到由 Unicycler 在步骤 8.4 中生成的unicycler_output_directory的 assembly.gfa 文件。加载后,单击左侧工具栏上的" 绘制图形 "按钮,然后查看如何连接和组织重叠群(称为节点),以评估程序集是否完成(图 5)。
注:完整的组件由两端连接的单个圆形重叠群表示(图5A,B)。不完整的组件具有多个连接在一起的重叠群或线性的(图5C)。小的线性重叠群可能并不完整,因为它们可能表明线性染色体外元素。覆盖,也称为深度,将在绷带中注明,并代表染色体重叠群的相对丰度,在Unicycler中归一化为1x。 - 夸斯特
- 在终端中,使用 cd 命令导航到存储独轮车输出的文件夹 :cd Desktop/Trimmed_Reads/unicycler_output_directory
注意:程序集所在位置的路径中不允许有空格,即,指向 Unicycler 输出的任何目录的名称中都不能包含空格。或者,将 assembly.fasta 文件复制到桌面以便于访问。 - 通过执行以下命令来运行 QUAST:quast assembly.fasta -o quast_output_directory
- 查看 QUAST 在输出目录中生成的报告quast_output_directory。
- 在终端中,使用 cd 命令导航到存储独轮车输出的文件夹 :cd Desktop/Trimmed_Reads/unicycler_output_directory
10. 基因组注释
注意:下面的注释管道使用 Prokka34,这是一个命令行工具,必须在使用前安装。或者,通过自动GUI K-Base(材料表)使用Prokka,或通过Web服务器RAST35注释基因组。如果将基因组沉积到NCBI中,它们将使用原核基因组注释管道(PGAP)36自动注释。
- 使用 cd 命令在终端内导航到存储独轮车输出的文件夹(请参阅步骤 9.2.1)。然后,通过执行以下命令运行 Prokka:prokka --前缀 sample_ID --outdir prokka_output_directory assembly.fasta
注意:--前缀将根据指定的sample_ID命名所有输出文件。--outdir将创建一个具有指定名称的输出目录,其中将保存所有Prokka输出文件;不要事先为 Prokka 创建输出目录。 - 通过打开.tsv表和/或将生成的.gff文件上传到序列分析软件中以可视化和分析注释来查看注释(图 6)。
- 可以根据感兴趣的遗传因素生成特定类型的注释。建议从基因组流行病学中心(www.genomicepidemiology.org/)Web服务器上的用户友好工具开始进行初步分析37,38,39,40,41。其他用于检测CRISPR-cas系统和前噬菌体的工具是可用的(图3)42,43。
11. 建议的数据民主化做法
- 如果可能,将所有原始读取数据以及组装的基因组存放在公共存储库中,例如NCBI序列读取存档(SRA)和Genbank。在NCBI沉积过程中,基因组通过PGAP管道自动注释。
结果
该方案已针对属于 图1中所列属的尿细菌的培养和测序进行了优化。并非所有的尿细菌都可以通过这种方法培养。培养基和条件由 图1中的属指定。gDNA完整性的示例性凝胶电泳评估如图 2所示。 图3描述了用于测序读取处理,基因组组装和注释的生物信息学管道的概述。 图 4 中提供了计算目录结构指南,以简化协议理解并为成功的组织提供框架。此外,还包括由该协议产生的两种克雷伯菌属的代表性完整基因组 ,肺炎 克雷伯菌和 催产衣原体。 图5 提供了这些组装体的表示形式,还包括一个额外的不完整的例子 肺炎克雷伯菌 基因组。每个完全注释的完整基因组的详细概述如图 6所示。最后, 表1 提供了测序读取统计数据的摘要,以提供对原始和修剪数据的广泛理解,这些数据足以生成高质量的封闭基因组组装。此外,两个代表性的关键参数完全完成克雷伯菌 属。列出了基因组。基因组和原始数据被存放在BioProject PRJNA683049下的Genbank中。
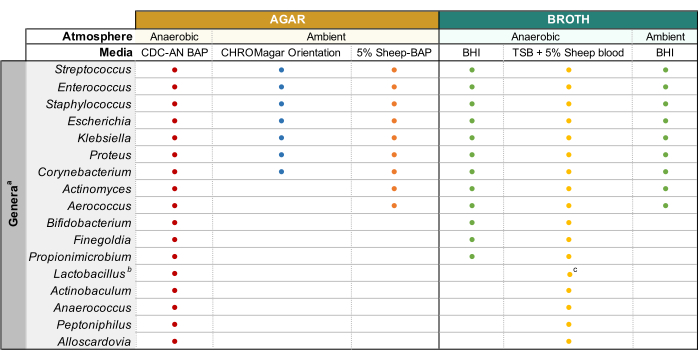
图1:不同尿液属的改良增强尿培养。可用于培养不同尿液属的琼脂和液体肉汤的图表。建议所有培养均在35°C下进行,如第1.1小节所述。圆圈代表适合培养特定属的培养基,任意选择颜色以区分一种培养基类型与另一种培养基类型。CDC-AN BAP(红色),CDC厌氧菌绵羊血琼脂;5%绵羊BAP(橙色),绵羊血琼脂;BHI(绿色),脑心脏输注;TSB(黄色),胰蛋白酸大豆汤;CHROMagar 方向(蓝色)。a阴道加德纳菌应在HBT双层G.阴道培养中培养选择性琼脂在微嗜酸气氛中和特殊肉汤培养要求下培养44。 b乳酸杆菌应在微嗜酸气氛中在5%兔-BAP平板和NYCIII肉汤上培养。c乳酸杆菌属。在微嗜酸性条件下,可在 MRS 上培养。请点击此处查看此图的放大版本。
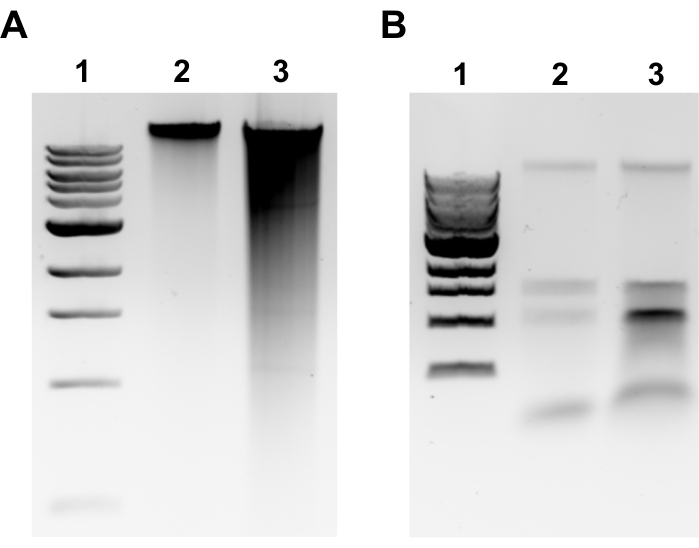
图2:琼脂糖凝胶图像的基因组DNA提取。 描绘gDNA提取结果的代表性凝胶图像。(A) 泳道 1: 1 kb 阶梯, 泳道 2: 完整 gDNA 代表成功提取, 泳道 3: 涂抹表示片段化 gDNA。(B) 泳道 1:1 kb 阶梯,泳道 2 和 3:rRNA 污染由 1.5 kb 至 3 kb 之间的两个条带表示。 请点击此处查看此图的放大版本。
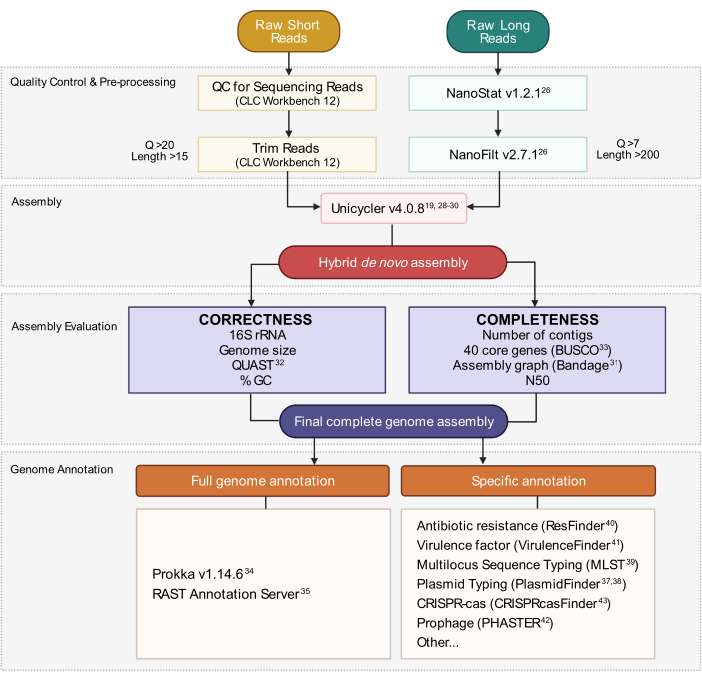
图3:混合基因组组装工作流程。 从读取质量控制和预处理到装配注释的步骤示意图。读取修剪可消除不明确和低质量的读取。指示 Q 评分和长度参数,并表示保留的读数。组装利用短读和长读来生成杂交 从头 基因组组装。使用指定的工具和参数根据完整性和正确性评估装配质量。最终的基因组组装对所有基因和感兴趣的特定位点进行注释。 请点击此处查看此图的放大版本。
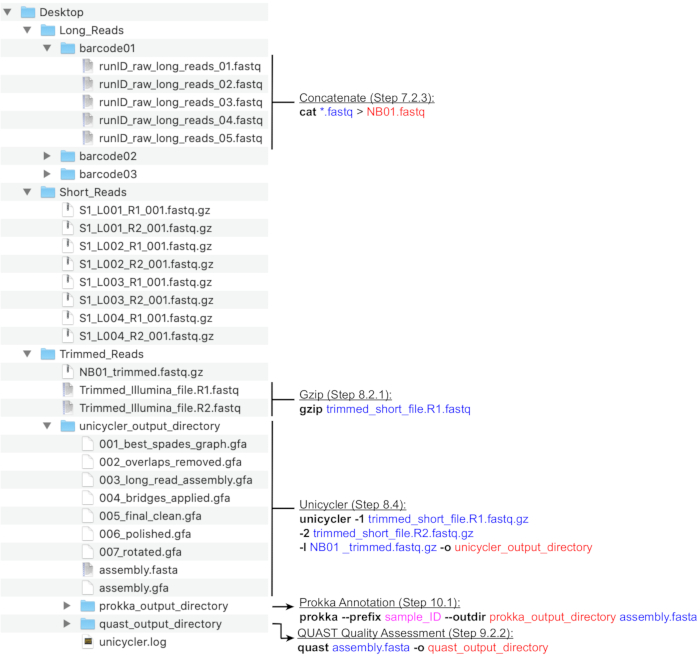
图4:生物信息学目录结构指南。 用于处理短读和长读、混合组装、基因组注释和 QC 的推荐目录和文件组织的示意图。关键命令行数据处理步骤在相应的文件和目录旁边突出显示。引出命令和标志(粗体)、输入文件(蓝色)、输出文件或目录(红色)、用户输入(如文件命名约定(洋红色)。 请点击此处查看此图的放大版本。
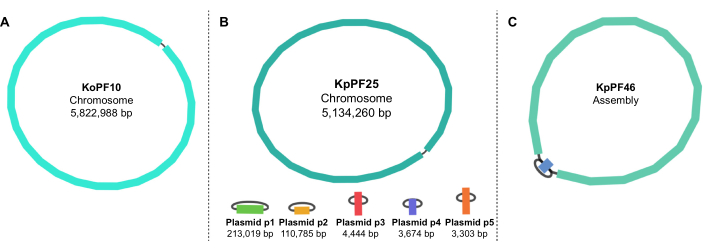
图5:绷带的基因组组装图。(A)催产克雷伯菌KoPF10和(B)肺炎克雷伯菌KpPF25的代表性完整基因组组装图和(C)肺炎克雷伯菌KpPF46的不完全基因组组装图。KoPF10的完整基因组显示了单个闭合染色体,KpPF25的完整基因组由一条闭合染色体和五条闭合质粒组成。KpPF46的不完全染色体由两个相互连接的重叠群组成。独轮车混合从头组件生成由绷带可视化的组件图。组装图提供了基因组的简单示意图,表明通过连接单个重叠群两端的连接子连接闭合的染色体或质粒。存在多个互连的重叠群表示装配不完整。重叠群的大小和深度也可以在绷带中注明。请点击此处查看此图的放大版本。
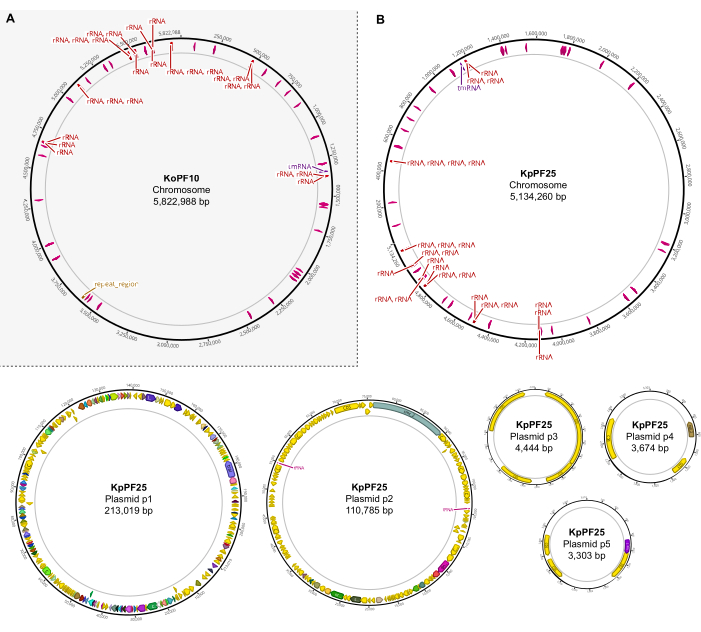
图6:带注释的杂交组件的完整基因组图谱。 Geneious Prime为(A)K. oxytoca KoPF10和(B)K. pneumoniae KpPF25的完整基因组生成的组装图显示了沿质粒骨架的彩色箭头表示的注释基因。为了简单起见,染色体仅显示rRNA和tRNA基因。基因组注释使用Prokka进行,如该协议第10节所示。 请点击此处查看此图的放大版本。
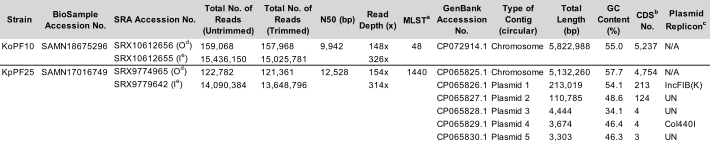
表1:代表性克雷伯菌 属完整组装特性。 提供 催产衣原 体KoPF10和 肺炎克雷伯菌 株KpPF25的组装参数。为两种测序技术指定了修剪前后的读取次数。N50 仅用于长读取,因为短读取的长度是受控的。使用质粒查找器 v2.1 肠杆菌科数据库预测的质粒,参数设置为 80% 身份和 60% 长度。a MLST,多位点序列类型。 b 光盘,编码序列。 c 使用质粒查找器v2.1肠杆菌科数据库预测的质粒复制子,参数设置为80%身份和60%长度。 d 牛津纳米孔技术公司(ONT)存入的读取数据。 e Illumina存入读取数据。请点击此处下载此表格。
讨论
这里描述的综合混合基因组组装方案为成功培养各种尿液微生物群和尿路病原体以及其基因组的完整组装提供了一种简化的方法。细菌基因组的成功WGS始于分离各种有时挑剔的微生物,以提取其基因组DNA。迄今为止,现有的尿培养方案要么缺乏检测许多尿种的必要敏感性,要么涉及需要延长时间和资源的冗长而广泛的方法11。所描述的改良增强型尿培养方法为成功分离属于17种常见尿属的细菌(包括潜在致病性或有益的共生菌种,以及兼性和专性需氧或厌氧菌)提供了简化而全面的方案。这反过来又为细菌基因组的准确测序和组装以及关键的表型实验提供了必要的起始材料,这有助于了解泌尿健康和疾病。此外,这种改进的培养方法为尿液标本中发现的活微生物提供了更明确的临床诊断,并允许其生物库用于未来的基因组研究。但是,此协议并非没有限制。它可能需要较长的孵育时间,具体取决于生物体以及使用资源,例如缺氧室或可能不易获得的受控培养箱。使用厌氧气体包提供了一种替代解决方案,但这些解决方案成本高昂,并且并不总是产生持续和受控的环境。最后,培养偏倚以及样本多样性可能允许特定生物体和泌尿道病原体胜过挑剔的细菌。尽管有这些局限性,但这种方法使多种尿液细菌的培养成为可能。
随着下一代测序技术的进步,基因组测序越来越受欢迎,这大大提高了测序数据的产量和准确性14,15。再加上用于数据处理和从头组装的算法的发展,完整的基因组序列在新手和专家科学家的指尖15,45。全基因组提供的整体基因组组织知识提供了重要的进化和生物学见解,包括基因复制,基因丢失和水平基因转移14。此外,对抗菌素耐药性和毒力很重要的基因通常定位于移动元件上,这些元件通常在基因组组装草案15,16中未得到解决。
本文的方案遵循混合方法,用于组合来自短读和长读平台的测序数据,以产生完整的基因组组装。虽然专注于尿液细菌基因组,但该程序可以适应来自各种分离来源的各种细菌。该方法的关键步骤包括遵循适当的无菌技术,并利用适当的培养基和培养条件分离纯尿细菌。此外,提取完整的高产量gDNA对于生成没有污染读数的测序数据至关重要,这些读数可能会阻碍组装成功。后续的文库制备方案对于生成足够长度和深度的高质量读数至关重要。因此,在库制备长读测序期间小心处理gDNA非常重要,因为该技术的最大好处是生成没有理论长度上限的长读取。还概述了测序读数的适当质量控制(QC)部分,可消除噪声数据并改善组装结果。
尽管DNA分离,文库制备和测序成功,但某些物种的基因组结构的性质仍可能为闭合基因组组装的产生提供障碍45,46。重复序列通常使装配计算复杂化,尽管读取数据很长,但这些区域可能以低置信度解决,或者根本不解决。因此,长读取必须平均长于基因组中最大的重复区域或覆盖率必须高(>100x)19。一些基因组可能仍然不完整,需要手动方法才能完成。然而,杂交组装的不完整基因组通常由比短读草稿基因组更少的重叠群组成。调整装配算法的默认参数或遵循更严格的读取 QC 截止值可能会有所帮助。或者,一种建议的方法是将长读数映射到不完整的区域,以寻找最可能的组装路径的证据,然后利用扩增区域的PCR和Sanger测序确认路径。建议使用Minimap2进行映射读取,并且Bandage提供了一个有用的工具,用于可视化沿组合重叠群的映射读取,为重叠群链接47提供证据。
生成完整基因组的另一个挑战在于对命令行工具的熟悉程度和舒适度。许多生物信息学工具的开发旨在为任何用户提供计算机会;但是,它们的使用依赖于对 UNIX 和编程基础知识的理解。该协议旨在提供足够详细的说明,使没有命令行经验的个体能够生成封闭的基因组组装并对其进行注释。
披露声明
作者没有什么可透露的。
致谢
我们感谢Moutuse Jubaida Islam博士和Luke Joyce博士对该协议的贡献。我们还要感谢德克萨斯大学达拉斯分校基因组中心的反馈和支持。这项工作由韦尔奇基金会资助,奖项编号为AT-2030-20200401给N.J.D.,由美国国立卫生研究院资助,奖项编号R01AI116610给K.P.,以及由Felecia和John Cain妇女健康主席,由P.E.Z.持有。
材料
| Name | Company | Catalog Number | Comments |
| Equipment: | |||
| Bioanalyzer 2100 | Agilent | G29398A | Optional but recommended |
| Centrifuge | Eppendorf | -- | Any centrifuge for spinning conicals and microcentrifuge tubes (e.g. Models 5810R/5424R) |
| Electrophoresis | BioRad Laboratories | 1645070 | |
| Gel Imaging System | BioRad Laboratories | ChemiDoc models | |
| Incubator | ThermoFisher Scientific | -- | Any CO2 Incubator (e.g. Thermo Forma model 3110) |
| Magnetic Rack | New England BioLabs | S15095 | 12-tube rack |
| MinION | Oxford Nanopore Technologies | -- | |
| Nanodrop | ThermoFisher Scientific | ND-ONE-W | |
| NextSeq 500 | Illumina | SY-415-1002 | Other Illumina models are acceptable |
| Plate Reader | BioTek | -- | Synergy H1 |
| Qubit fluorometer | ThermoFisher Scientific | Q33238 | |
| Rotator | Benchmark Scientific | H2024 | |
| Thermocycler | ThermoFisher Scientific | -- | Any thermocycler for PCR reactions (e.g. ProFlex PCR system) |
| Materials: | |||
| 10X Phosphate Buffered Saline (PBS) | Fisher Scientific | BP3991 | |
| 10X TBE buffer | -- | -- | 1M Tris,1M Boric Acid,0.2M EDTA (pH 8.0) |
| 1429R primer | Sigma Aldrich (Custom oligos) | -- | GGTTACCTTGTTACGACTT |
| 1kb Ladder | VWR | 101228-494 | |
| 1M Tris-Cl (pH 7.5) | ThermoFisher Scientific | 15567027 | |
| 6x Loading dye | Fisher Scientific | NC0783588 | |
| 8F primer | Sigma Aldrich (Custom oligos) | -- | AGAGTTTGATCCTGGCTCAG |
| Agar | Fisher Scientific | BP1423-2 | |
| Agarose | BioRad Laboratories | 63001 | |
| AMPure XP Beads | Beckman Coulter | A63880 | |
| Anaerobe Pouch System - GasPak EZ | BD Diagnostic Systems | B260683 | |
| Boric Acid | Fisher Scientific | A73-500 | |
| Brain Heart Infusion Broth | BD Diagnostic Systems | 212304 | |
| CDC Anaerobe 5% Sheep Blood Agar | BD Diagnostic Systems | L007357 | |
| CHROMagar Orientation | BD Diagnostic Systems | PA-257481.04 | |
| DNeasy Blood & Tissue | QIAGEN | 69504 | |
| DreamTaq Master Mix | ThermoFisher Scientific | K1081 | |
| Dry Anaerobic Indicator Strips | BD Diagnostic Systems | 271051 | |
| EDTA | Fisher Scientific | S311-500 | |
| Ethanol 200 Proof | Sigma Aldrich | E7023 | For molecular biology |
| Ethidium Bromide | ThermoFisher Scientific | BP130210 | |
| Flow cell priming kit | Oxford Nanopore Technologies | EXP-FLP002 | |
| Flow cell wash kit | Oxford Nanopore Technologies | EXP-WSH003 | |
| Gel Extraction Miniprep Kit | BioBasic | BS654 | |
| Ligation sequencing kit | Oxford Nanopore Technologies | SQK-LSK109 | |
| Lysozyme | Research Products International Corp | L381005.05 | |
| Mutanolysin | Sigma Aldrich | M9901-5KU | |
| Native barcoding expansion 1-12 | Oxford Nanopore Technologies | EXP-NBD104 | |
| NEB Blunt/TA Ligase Master Mix | New England BioLabs | M0367L | |
| NEBNext FFPE DNA Repair Mix | New England BioLabs | M6630L | |
| NEBNext quick ligation buffer | New England BioLabs | B6058S | |
| NEBNext Ultra II End repair / dA-tailing module | New England BioLabs | E7546L | |
| Nextera DNA CD Indexes | Illumina | 20018708 | |
| Nextera DNA Flex Library Prep - (M) Tagmentation | Illumina | 20018705 | |
| Nuclease-free water | Sigma Aldrich | W4502 | |
| Qubit 1X dsDNA HS Assay Kit | ThermoFisher Scientific | Q33230 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Quick T4 DNA Ligase | New England BioLabs | E6056L | |
| R9 Flow cell | Oxford Nanopore Technologies | FLO-MIN106D | |
| RNase A | ThermoFisher Scientific | EN0531 | |
| Sheep Blood | Hemostat Laboratories | DS13250 | |
| TE buffer | -- | -- | 10mM Tris, 1mM EDTA (pH 8.0) |
| Triton X-100 | Sigma Aldrich | T8787 | |
| Tryptic Soy Broth | BD Diagnostic Systems | 211825 | |
| Software & Bioinformatic Tools: | |||
| Bandage | -- | -- | https://rrwick.github.io/Bandage/ |
| Center for Genomic Epidemiology | -- | -- | http://www.genomicepidemiology.org/ |
| CLC Genomics Workbench 12 | QIAGEN | -- | |
| CRISPRcasFinder | -- | -- | https://crisprcas.i2bc.paris-saclay.fr/ |
| FastQC | -- | -- | https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ |
| Geneious Prime | Geneious | -- | |
| gVolante (BUSCO) | -- | -- | https://gvolante.riken.jp/ |
| Kbase Prokka Wrapper | -- | -- | https://kbase.us/applist/apps/ProkkaAnnotation/annotate_contigs/release |
| Minimap2 | -- | -- | https://github.com/lh3/minimap2 |
| MinKNOW | Oxford Nanopore Technologies | -- | |
| NanoFilt | -- | -- | https://github.com/wdecoster/nanofilt |
| NanoStat | -- | -- | https://github.com/wdecoster/nanostat |
| PHASTER | -- | -- | https://phaster.ca/ |
| Prokka | -- | -- | https://github.com/tseemann/prokka |
| QUAST | -- | -- | http://quast.sourceforge.net/quast |
| Trim Galore | -- | -- | https://www.bioinformatics.babraham.ac.uk/projects/trim_galore/ |
| Trimmomatic | -- | -- | http://www.usadellab.org/cms/?page=trimmomatic |
| Unicycler | -- | -- | https://github.com/rrwick/Unicycler#necessary-read-length |
参考文献
- Brubaker, L., Wolfe, A. The urinary microbiota: a paradigm shift for bladder disorders. Current Opinion in Obstetrics & Gynecology. 28 (5), 407-412 (2016).
- Neugent, M. L., Hulyalkar, N. V., Nguyen, V. H., Zimmern, P. E., De Nisco, N. J. Advances in understanding the human urinary microbiome and its potential role in urinary tract infection. mBio. 11 (2), (2020).
- Klein, R. D., Hultgren, S. J. Urinary tract infections: microbial pathogenesis, host-pathogen interactions and new treatment strategies. Nature Reviews. Microbiology. 18 (4), 211-226 (2020).
- Horsley, H., et al. Enterococcus faecalis subverts and invades the host urothelium in patients with chronic urinary tract infection. PLoS One. 8 (12), 83637 (2013).
- Reitzer, L., Zimmern, P. Rapid growth and metabolism of uropathogenic Escherichia coli in relation to urine composition. Clinical Microbiology Reviews. 33 (1), 00101-00119 (2019).
- Snyder, J. A., et al. Transcriptome of uropathogenic Escherichia coli during urinary tract infection. Infection and Immunity. 72 (11), 6373-6381 (2004).
- Ipe, D. S., Horton, E., Ulett, G. C. The basics of bacteriuria: Strategies of microbes for persistence in urine. Frontiers in Cellular and Infection Microbiology. 6, 14 (2016).
- Babikir, I. H., et al. The impact of cathelicidin, the human antimicrobial peptide LL-37 in urinary tract infections. BMC Infectious Diseases. 18 (1), 17 (2018).
- Jancel, T., Dudas, V. Management of uncomplicated urinary tract infections. The Western Journal of Medicine. 176 (1), 51-55 (2002).
- Ventola, C. L. The antibiotic resistance crisis: part 1: causes and threats. P & T. 40 (4), 277-283 (2015).
- Price, T. K., et al. The clinical urine culture: Enhanced techniques improve detection of clinically relevant microorganisms. Journal of Clinical Microbiology. 54 (5), 1216-1222 (2016).
- Kass, E. H. Asymptomatic infections of the urinary tract. Transactions of the Association of American Physicians. 69, 56-64 (1956).
- Garcia, L. S. . Clinical microbiology procedures handbook. 3rd edn. , (2010).
- Fraser, C. M., Eisen, J. A., Nelson, K. E., Paulsen, I. T., Salzberg, S. L. The value of complete microbial genome sequencing (you get what you pay for). Journal of Bacteriology. 184 (23), 6403-6405 (2002).
- Chen, Z., Erickson, D. L., Meng, J. Benchmarking hybrid assembly approaches for genomic analyses of bacterial pathogens using Illumina and Oxford Nanopore sequencing. BMC Genomics. 21 (1), 631 (2020).
- Greig, D. R., Dallman, T. J., Hopkins, K. L., Jenkins, C. MinION nanopore sequencing identifies the position and structure of bacterial antibiotic resistance determinants in a multidrug-resistant strain of enteroaggregative Escherichia coli. Microbial Genomics. 4 (10), 000213 (2018).
- Carraro, D. M., et al. PCR-assisted contig extension: stepwise strategy for bacterial genome closure. Biotechniques. 34 (3), 626-628 (2003).
- Tettelin, H., Radune, D., Kasif, S., Khouri, H., Salzberg, S. L. Optimized multiplex PCR: efficiently closing a whole-genome shotgun sequencing project. Genomics. 62 (3), 500-507 (1999).
- Wick, R. R., Judd, L. M., Gorrie, C. L., Holt, K. E. Unicycler: Resolving bacterial genome assemblies from short and long sequencing reads. PLoS Computational Biology. 13 (6), 1005595 (2017).
- Singhal, N., Kumar, M., Kanaujia, P. K., Virdi, J. S. MALDI-TOF mass spectrometry: an emerging technology for microbial identification and diagnosis. Frontiers in Microbiology. 6, 791 (2015).
- Turner, S., Pryer, K. M., Miao, V. P., Palmer, J. D. Investigating deep phylogenetic relationships among cyanobacteria and plastids by small subunit rRNA sequence analysis. The Journal of Eukaryotic Microbiology. 46 (4), 327-338 (1999).
- Weisburg, W. G., Barns, S. M., Pelletier, D. A., Lane, D. J. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology. 173 (2), 697-703 (1991).
- Janda, J. M., Abbott, S. L. 16S rRNA gene sequencing for bacterial identification in the diagnostic laboratory: pluses, perils, and pitfalls. Journal of Clinical Microbiology. 45 (9), 2761-2764 (2007).
- Stevenson, K., McVey, A. F., Clark, I. B. N., Swain, P. S., Pilizota, T. General calibration of microbial growth in microplate readers. Science Reports. 6, 38828 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- De Coster, W., D'Hert, S., Schultz, D. T., Cruts, M., Van Broeckhoven, C. NanoPack: visualizing and processing long-read sequencing data. Bioinformatics. 34 (15), 2666-2669 (2018).
- Wilson, G., et al. The UNIX Shell. Zenodo. , (2019).
- Bankevich, A., et al. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing. Journal of Computational Biology. 19 (5), 455-477 (2012).
- Vaser, R., Sovic, I., Nagarajan, N., Sikic, M. Fast and accurate de novo genome assembly from long uncorrected reads. Genome Research. 27 (5), 737-746 (2017).
- Walker, B. J., et al. Pilon: an integrated tool for comprehensive microbial variant detection and genome assembly improvement. PLoS One. 9 (11), 112963 (2014).
- Wick, R. R., Schultz, M. B., Zobel, J., Holt, K. E. Bandage: interactive visualization of de novo genome assemblies. Bioinformatics. 31 (20), 3350-3352 (2015).
- Gurevich, A., Saveliev, V., Vyahhi, N., Tesler, G. QUAST: quality assessment tool for genome assemblies. Bioinformatics. 29 (8), 1072-1075 (2013).
- Simao, F. A., Waterhouse, R. M., Ioannidis, P., Kriventseva, E. V., Zdobnov, E. M. BUSCO: assessing genome assembly and annotation completeness with single-copy orthologs. Bioinformatics. 31 (19), 3210-3212 (2015).
- Seemann, T. Prokka: rapid prokaryotic genome annotation. Bioinformatics. 30 (14), 2068-2069 (2014).
- Aziz, R. K., et al. The RAST server: rapid annotations using subsystems technology. BMC Genomics. 9, 75 (2008).
- Tatusova, T., et al. NCBI prokaryotic genome annotation pipeline. Nucleic Acids Research. 44 (14), 6614-6624 (2016).
- Carattoli, A., Hasman, H. PlasmidFinder and In Silico pMLST: Identification and Typing of Plasmid Replicons in Whole-Genome Sequencing (WGS). Methods in Molecular Biology. 2075, 285-294 (2020).
- Carattoli, A., et al. In silico detection and typing of plasmids using PlasmidFinder and plasmid multilocus sequence typing. Antimicrobial Agents and Chemotherapy. 58 (7), 3895-3903 (2014).
- Larsen, M. V., et al. Multilocus sequence typing of total-genome-sequenced bacteria. Journal of Clinical Microbiology. 50 (4), 1355-1361 (2012).
- Bortolaia, V., et al. ResFinder 4.0 for predictions of phenotypes from genotypes. The Journal of Antimicrobial Chemotherapy. 75 (12), 3491-3500 (2020).
- Joensen, K. G., et al. Real-time whole-genome sequencing for routine typing, surveillance, and outbreak detection of verotoxigenic Escherichia coli. Journal of Clinical Microbiology. 52 (5), 1501-1510 (2014).
- Arndt, D., et al. PHASTER: a better, faster version of the PHAST phage search tool. Nucleic Acids Research. 44 (1), 16-21 (2016).
- Couvin, D., et al. CRISPRCasFinder, an update of CRISRFinder, includes a portable version, enhanced performance and integrates search for Cas proteins. Nucleic Acids Research. 46 (1), 246-251 (2018).
- Totten, P. A., Amsel, R., Hale, J., Piot, P., Holmes, K. K. Selective differential human blood bilayer media for isolation of Gardnerella (Haemophilus) vaginalis. Journal of Clinical Microbiology. 15 (1), 141-147 (1982).
- Nagarajan, N., Pop, M. Sequence assembly demystified. Nat Reviews. Genetics. 14 (3), 157-167 (2013).
- Phillippy, A. M., Schatz, M. C., Pop, M. Genome assembly forensics: finding the elusive mis-assembly. Genome Biology. 9 (3), 55 (2008).
- . Unicycler Wiki Available from: https://github.com/rrwick/Unicycler/wiki (2017)
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。