Method Article
짧고 긴 읽기 시퀀싱 기술을 사용하여 요로나박테리아의 완전한 게놈 생성을 위한 하이브리드 드 노보 게놈 조립
요약
이 프로토콜은 오줌 박테리아의 배양, 시퀀싱 및 드 노보 하이브리드 게놈 조립에 대한 포괄적 인 접근 방식을 자세히 설명합니다. 그것은 오줌 식민지, 병기 발생 및 항균 저항 보급에 기여하는 염색체 및 외염색체 유전 요소 둘 다 공부에 유용한 완전하고 원형 게놈 서열의 생성을 위한 재현 가능한 절차를 제공합니다.
초록
완전한 게놈 서열은 유전적 다양성과 오줌 미생물의 독특한 식민지 요인에 대한 이해를 위한 귀중한 데이터를 제공합니다. 이러한 데이터는 플라스미드 및 외염색체 파지와 같은 이동식 유전 적 원소를 포함할 수 있으며, 이는 항균 저항의 보급에 기여하고 요로 감염 (UTI)의 치료를 더욱 복잡하게 합니다. 게놈 구조의 미세한 분해능을 제공하는 것 외에도, 완전하고 폐쇄된 게놈은 상세한 비교 유전체학 및 진화 분석을 가능하게 합니다. 완전한 게놈 드 노보의 생성은 사용 가능한 시퀀싱 기술의 한계로 인해 오랫동안 도전적인 작업이었습니다. 페어링 엔드 차세대 염기서열 분석(NGS)은 고품질의 짧은 판독을 생성하여 정확하지만 단편화된 게놈 어셈블리를 생성합니다. 반대로, 나노포어 시퀀싱은 일반적으로 오류가 발생하기 쉬운 완전한 어셈블리로 이어지는 낮은 품질의 긴 판독을 제공합니다. 이러한 오류는 게놈 전체 협회 연구를 방해하거나 오해의 소지가 있는 변형 분석 결과를 제공할 수 있습니다. 따라서 짧고 긴 판독을 모두 결합한 하이브리드 접근법은 매우 정확한 폐쇄세균 게놈을 달성하기 위한 신뢰할 수 있는 방법으로 부상했습니다. 본 명세서에서 보고된 다양한 오줌 박테리아의 배양, 종 식별에 의한 16S rRNA 유전자 시퀀싱, 게놈 DNA(gDNA)의 추출, NGS 및 나노포어 플랫폼에 의한 짧고 긴 판독의 생성이 각각 이다. 또한,이 방법은 추가로, 추가된 완전한 게놈 서열의 생성을 위한 품질 관리, 조립 및 유전자 예측 알고리즘의 생물포계 파이프라인을 기술합니다. 생물 정보 도구의 조합은 하이브리드 게놈 조립 및 다운스트림 분석을위한 고품질 읽기 데이터의 선택을 가능하게합니다. 이 프로토콜에 기재된 하이브리드 드 노보 게놈 어셈블리에 대한 간소화된 접근법은 임의의 컬투성 박테리아에서의 사용을 위해 적응될 수 있다.
서문
요로 미생물군유전체는 요로가 건강한 개인에게 멸균된다는 수십 년 의 긴 오해를 산산조각 난 연구의 신흥 영역입니다. 요로 미생물의 구성원은 요로 환경의 균형을 맞추고 요로 감염 (UTI)1,2를예방하는 역할을 할 수 있습니다. 비뇨기증 균은 요로를 침범하고 상주 미생물을 대체하고, 비뇨기과를 식민지화하고, 면역 반응을 회피하고, 환경 압력에 중화하기 위해 다양한 독성 메커니즘을사용한다 3,4. 소변은 높은 진동, 제한된 질소 및 탄수화물 가용성, 낮은 산소, 낮은 pH5,6,7을특징으로 상대적으로 영양 제한 매체이다. 소변은 또한 항균제로 간주되며, 인체 카플리시딘 LL-378과같은 억제 우레아 및 항균 펩티드의 고농도로 구성된다. 요로를 식민지화하기 위해 상주 박테리아와 비뇨기과 물질 모두에 의해 채택된 기계장치를 조사하는 것은 요로 건강을 더 이해하고 UTI 처리를 위한 새로운 전략을 개발하는 것이 중요합니다. 더욱이, 일선 항균요법의 실패가 보편화됨에 따라, 오줌박테리아의 집단 내에서 항균저항 결정제를 운반하는 이동식 유전적 원소의 보급을 감시하는 것이 점점 더 중요해지고 있다9,10.
요로 박테리아의 유전자형 과 표현형을 조사하기 위해 성공적인 배양과 후속 전체 게놈 시퀀싱 (WGS)이 필수적입니다. 배양의존적 방법은 소변샘플(11)에서가능한 미생물을 검출하고 식별하는 데 필요하다. 표준 임상 소변 배양은 소변을 5% 양 혈액 한천(BAP)과 맥콩키 한천에 도금하고 24h12에대해 35°C에서 항공적으로 배양하는 것을 포함한다. 그러나, ≥105 CFU/mL13의검출 임계값을 가진, 오줌 microbiota의 많은 구성원은 이 방법에 의해 보고되지 않습니다. 향상된 정량소변 배양(EQUC)(11)와 같은 향상된 배양 기술은 표준 소변 문화에 의해 일반적으로 놓친 미생물을 확인하기 위하여 다른 소변 볼륨, 잠복기, 배양 매체 및 대기 조건의 각종 조합을 채택합니다. 본 프로토콜에 기재된 EQUC의 수정된 버전은, 여기에 변형된 향상된 소변 배양 프로토콜이라고 불리는, 선택적 배지와 최적의 대기 조건을 사용하여 다양한 오줌 박테리아와 비뇨기병균을 배양할 수 있지만 본질적으로 정량적이지는 않습니다. 오줌 박테리아의 성공적인 격리는 하류 WGS 및 게놈 조립을 위한 게놈 DNA (gDNA)의 추출을 가능하게 합니다.
게놈 어셈블리, 특히 완전한 어셈블리는, 상주 미생물과 비뇨기과 박테리아 둘 다 중 식민지화, 틈새 유지 보수 및 독성에 기여할 수 있는 유전 적 요인의 발견을 가능하게 합니다. 초안 게놈 어셈블리에는 시퀀싱 오류및 방향 정보가 부족할 수 있는 다양한 수의 연속 서열(연속)이 포함되어 있습니다. 완전한 게놈 어셈블리에서 모든 베이스 쌍의 방향과 정확도가모두 14로확인되었습니다. 더욱이, 완전한 게놈 서열을 얻는 것은 게놈 구조, 유전적 다양성 및 이동성 유전원15에대한 통찰력을 제공한다. 짧은 읽기만으로는 중요한 유전자의 존재 또는 부재를 식별할 수 있지만 그들의 게놈컨텍스트(16)를정확히 찾아낼 수는 없다. 옥스포드 나노포어 및 PacBio와 같은 장시간 읽은 시퀀싱 기술을 가능하게 함으로써, 세균 게놈의 폐쇄 드 노보 어셈블리를 생성하면 더 이상 멀티플렉스 PCR17,18에의한 드 노보 어셈블리의 수동 폐쇄와 같은 격렬한 방법이 필요하지 않다. 차세대 단기 판독 시퀀싱 과 나노포어 장기 읽기 시퀀싱 기술의 조합은 상대적으로 저렴한비용으로정확하고 완전하며 폐쇄된 세균 게놈 어셈블리의 촉진 생성을 허용한다. 짧은 읽기 시퀀싱은 일반적으로 평균 40-100 개의 연속으로 구성된 정확하면서도 단편화된 게놈 어셈블리를 생성하며, Nanopore 시퀀싱은 정확도가 낮지만 연통에 가입하고 유전체 시니를 해결하는 스캐폴드로 작용할 수 있는 길이약 5-100 kb의 긴 판독을 생성합니다. 짧은 읽기 및 긴 읽기 기술을 모두 활용하는 하이브리드 접근 방식은 정확하고 완전한 세균 게놈19를생성할 수 있습니다.
여기에 설명된 것은 하이브리드 어셈블리 접근법을 이용한 인간 소변, 게놈 추출, 시퀀싱 및 완전한 게놈 조립으로부터 박테리아의 격리 및 식별을 위한 포괄적인 프로토콜이다. 이 프로토콜은 플라스미드와 같은 폐쇄된 세균 염색체 및 초염색체 원소의 정확한 조립을 위해 짧은 읽기 및 긴 읽기 시퀀싱으로 생성된 읽기를 올바르게 수정하는 데 필요한 단계에 중점을 둡니다.
프로토콜
박테리아는 기관 검토 위원회 승인 연구의 일환으로 동의 여성에서 수집 소변에서 배양 되었다 19MR0011 (UTD) 및 STU 032016-006 (UTSW).
1. 수정 된 강화 된 소변 문화
참고: 모든 배양 단계는 멸균 조건하에서 수행해야 합니다. 모든 기기, 솔루션 및 미디어를 소독합니다. 70%의 에탄올로 작업 영역을 청소한 다음 분젠 버너를 설치하고 화염에 조심스럽게 가까이 가서 오염 가능성을 줄입니다. 또는, 클래스 II 생물 안전 캐비닛멸균 환경을 유지하기 위해 사용될 수있다. 잠재적으로 병원성 미생물에 노출되지 않도록 적절한 개인 보호 장비 (PPE)를 착용하십시오.
- 글리세롤을 비축한 소변과 식민지 격리도 금식
- 실온 (RT)에서 글리세롤 이산 소변을 해동하십시오. 일단 해동되면, 5 s가 섞일 샘플을 소용돌이. 멸균 미세 원심 분리기 튜브에서 멸균 1x 인산염 완충식식염수(PBS)에서 소변의 희석을 1:3 및 1:30 희석하여 100 μL의 최종 부피로 준비한다.
참고: 글리세롤 재고 소변은 희석되지 않은 소변 500 μL과 500 μL의 500 μL을 극저온에 혼합하고 -80°C에 보관하여 제조됩니다. - 사용하기 전에 37 °C에서 미리 따뜻하게 한 천 접시를 15 분 동안. 일반적인 오줌 세균 제네라에 적합한 미디어 유형 및 배양 조건에 대해서는 도 1을 참조하십시오. 희석된 소변을 도금 전에 배관하여 잘 섞고, 희석된 소변의 100 μL을 원하는 한천 판에 접시에 넣고 멸균 유리 구슬을 사용하여 샘플을 확산시다. 1x PBS의 플레이트 100 μL은 성장 제어가 없는 별도의 플레이트에 희석됩니다.
참고: 일반적인 비뇨생식기 종(예: 대장균, 클레브시엘라 스PP., 엔테로코커스 파레칼리스 등)을 배양하려고 시도하는 경우, 비뇨기과 세균종의 쉽게 식별할 수 있으므로 염색체 식아갈루기(재료표)를 사용하는 것이 좋습니다(그림 1). Colistin Nalidixic acid (CNA) 또는 MRS agar는 비 선택적 한고에서 까다로운 종을 능가 할 수있는 그람 음성 비뇨기물질을 함유하는 것으로 알려진 소변에서 까다로운 그램 양성 종 (예를 들어, 유산 균 spp.)을분리하는 데 유용합니다. - 위증균제의 경우 24시간 동안 35°C의 원하는 대기 상태에서 반전된 플레이트를 배양하고 까다로운 박테리아를 위해 3-5일(도1).
- 인큐베이션 기간 이후에인 배양기에서 플레이트를 제거합니다. 각 플레이트에서 독특한 색상, 형태 또는 혈연 패턴을 나타내는 식민지를 선택합니다.
- 세균 식민지를 상응하는 한천에 멸균 루프를 사용하여 세균 식민지를 재줄무늬로 하고, 원하는 분위기에서 2-5일 동안 반전된 플레이트를 배양하여 잘 고립된 식민지를 얻습니다.
참고: 1차 배양에 BAP를 활용하는 경우, 염색체 한천에 콜로니를 패치하면 샘플내세균 집단의 이질성에 대한 유용한 정보를 제공할 수 있다.
- 실온 (RT)에서 글리세롤 이산 소변을 해동하십시오. 일단 해동되면, 5 s가 섞일 샘플을 소용돌이. 멸균 미세 원심 분리기 튜브에서 멸균 1x 인산염 완충식식염수(PBS)에서 소변의 희석을 1:3 및 1:30 희석하여 100 μL의 최종 부피로 준비한다.
- 액체 국물 및 글리세롤 스타킹 세균 분리에서 배양
- 부모 식민지의 형태와 일치하는 고립 된 식민지가 얻어지면, 단일 식민지를 선택하고 멸균 접종 루프를 사용하여 액체 국물의 3 mL로 접종. 일반적인 오줌 microbiota genera의 성장을 지원할 수 있는 국물에 대한 그림 1을 참조하십시오. 파라필름으로 한천판을 밀봉하여 2-4일 동안 4°C로 보관하십시오. 배양이 눈에 띄게 탁상될 때까지 원하는 대기 조건에서 1-5일 동안 액체 배양을 배양합니다.
- 성장이 관찰된 후, 배양, 다음 2 mL 극저온에서 멸균 500 μL50%의 글리세롤에 하룻밤 문화의 1mL을 추가; 인감과 부드럽게 반전으로 혼합. 각 콜로니에 대해 두 개의 글리세롤 스톡을 준비하고 (하나는 백업 역할을) -80 °C에 보관하십시오.
2. 16S rRNA 유전자 Sanger 시퀀싱에 의한 세균성 종 식별
참고: 미생물 정체성은 매트릭스 보조 레이저 탈광 이온화 시간 비행 질량 분석법(MALDI-TOF)20을사용하여 확인될 수 있다.
- 콜로니-폴리머라제 연쇄 반응(PCR)
- PCR 튜브에서 PCR 반응의 25 μL을 2x Taq 폴리머라제 마스터 믹스의 12.5 μL, 10 μM 8F 프라이머의 0.5 μL, 10μM 1492R 프라이머의 0.5 μL(재료표),핵11.5μL을 추가하여 PCR 튜브에서 PCR반응의25 μL을 준비하십시오.
참고: 여러 시료에 대한 PCR을 수행하는 경우 Taq 폴리머라제 믹스, 프라이머 및 멸균 뉴클레아제 가 없는 물의 반응 마스터 믹스를 만듭니다. 그런 다음 각 PCR 튜브에 25 μL을 알리쿼트합니다. - 콜로니-PCR을 수행하려면 멸균 이쑤시개 또는 파이펫 팁을 사용하여 리스트씬으로부터 잘 분리된 콜로니를 스 와이프합니다. 2.1.1 단계에서 제조된 PCR 반응 혼합물에서 콜로니를 다시 중단한다. 부드럽게 섞습니다. 2000 x g에서 빠른 스핀으로 튜브 의 바닥에액체를 수집합니다.
참고: 샘플에 기포가 없는지 확인합니다. PCR 반응 혼합만을 포함하는 비템플릿 제어(NTC) 샘플을 포함한다. - 시료 튜브를 써모사이클러에 놓고 다음 프로그램을 실행하십시오: 3분 동안 95°C; 40 사이클: 30s용 95°C, 30초용 51°C, 1분 30초동안 72°C; 72 °C 10 분; 10 °C에서 유지하십시오.
- PCR 튜브에서 PCR 반응의 25 μL을 2x Taq 폴리머라제 마스터 믹스의 12.5 μL, 10 μM 8F 프라이머의 0.5 μL, 10μM 1492R 프라이머의 0.5 μL(재료표),핵11.5μL을 추가하여 PCR 튜브에서 PCR반응의25 μL을 준비하십시오.
- 젤 추출 및 종 식별
- PCR 실행이 완료되면 PCR 제품을 0.5배 Tris-Borate-EDTA(TBE) 버퍼로 제조한 1% 아가로즈 젤로 확인하십시오. 젤을 주조하기 전에 에디듐 브로마이드 (EtBr)를 추가하십시오. 그런 다음 적어도 20 μL 샘플 볼륨을 보유하는 우물에 빗을 사용하여 젤을 캐스팅합니다.
주의: EtBr은 발암성으로 의심되는 인터컬링 에이전트입니다. 항상 장갑과 PPE를 취급할 때 착용하고 기관의 지침에 따라 EtBr이 포함된 자재를 폐기하십시오. - 젤이 설정되면 0.5배 TBE 버퍼로 채워진 전기 전광 탱크에 젤을 놓고 빗을 제거합니다. 첫 번째 우물에 1 kb 사다리를 로드하고 PCR 반응의 10-20 μL을 후속 우물에 로드합니다. 해결 될 때까지 100-140 V에서 실행합니다. UV 광 하에서 젤을 시각화하고 NTC에 잘 없는 ~1.5 kb에서 명확하게 정의된 대역의 존재를 확인합니다.
주의: 자외선은 피부와 눈에 유해하며, 젤을 시각화할 때 적절한 가드를 사용하고 적절한 PPE를 착용하십시오.
참고: 식민지 PCR 일부 박테리아에 대 한 실패 할 수 있습니다.; 격리 된 gDNA에서 PCR을 진행하는 것은 대체 옵션(22)입니다. - 면도기를 사용하여 ~ 1.5 kb 밴드를 소비하고 젤 절단을 깨끗한 미세 원심 분리 튜브로 옮기십시오. 제조업체의지침(재료 표)에따라 젤 추출 프로토콜을 진행합니다. 마이크로볼륨 분광광계에 의한 정제된 DNA의 농도를 측정한다.
참고: 농도 >10 ng/μL이 바람직하며, 1.7-2.0 사이의 A260/280이 허용된다. - 선택된 Sanger 시퀀싱 서비스의 지침에 따라 뉴클레아제 없는 물에 1492R 프라이머를 사용하여 각 샘플에 대한 2개의 Sanger 시퀀싱 반응을 준비한다.
- 시퀀싱 데이터가 수신되면, NCBI 기본 로컬 정렬 검색 도구 blast.ncbi.nlm.nih.gov/Blast.cgi (BLAST) 웹 사이트 (blast)에 DNA 서열을 업로드, 뉴클레오티드 블라스트 (blastn)를 선택, rRNA / ITS 데이터베이스를 선택 16S 리보소말 RNA 서열 (박테리아와 고고학), 메가 블라스트 프로그램을 실행. 격리는 데이터베이스에서 참조에 대한 최고 품질 히트에 의해 식별될 수 있다.
참고: 일부 세균종은 16S rRNA 서열에서 높은 정체성을 나타내며 이 방법만으로는 구별할 수 없습니다. 스펙션은 DNA 상동성 및 생화학적 분석이 필요하며 동일한 속23의구성원을 자신있게 구별할 수 있습니다.
- PCR 실행이 완료되면 PCR 제품을 0.5배 Tris-Borate-EDTA(TBE) 버퍼로 제조한 1% 아가로즈 젤로 확인하십시오. 젤을 주조하기 전에 에디듐 브로마이드 (EtBr)를 추가하십시오. 그런 다음 적어도 20 μL 샘플 볼륨을 보유하는 우물에 빗을 사용하여 젤을 캐스팅합니다.
3. 게놈 DNA (gDNA) 추출
참고: 이 섹션에서는 다양한 세균 성 종에서 고품질 게놈 DNA의 높은 수율 추출을 위해 재료 표에 참조 된 gDNA 추출 키트에 제공 된 시약 및 스핀 컬럼을 활용합니다. 아래는 권장 수정 및 지침입니다.
- 제조업체의 지침에 따라 키트 시약을 준비합니다.
- 3-10mL 배양을 적절한 멸균 국물에준비(도 1)충분한 성장이 관찰될 때까지 잘 분리된 식민지로부터 배지로 박테리아를 접종하고 도 1에 주목된 온도 및 대기압에서 배양함으로써 적절한 멸균 국물에 접종한다.
- 인큐베이션 후,분광광계(24)를이용하여 배양의 600nm(OD600)에서광학 밀도를 측정한다.
- 1:10 비율로 하룻밤 문화를 희석하여 정량화를 위해 샘플을 준비합니다. 또한 측정을위한 멸균 배양 매체의 공백을 포함한다. 샘플 판독값에서 빈 판독값을 빼고 10분의 희석계수를 곱하여 광학 밀도를 계산합니다.
- 종에 대한 OD600 측정 및 사전 설정된 OD600 ~ CFU/mL 비율을 사용하여 2 x 109 세포를 얻기 위해 배양소의 밀리리터 수를 계산합니다.
- 원심 분리기5000 x g에서 5 분 동안 필요한 배양 볼륨을 펠릿에 원심 분리합니다. 200 μL 콜드 TE 버퍼에서 슈퍼네티를 흡인하고 펠릿을 재놓습니다(시술 초기에 얼음에 미리 식힙니다).
- 원심 분리는 5000 x g에서 2 분 동안 샘플을 원심 분리합니다. 상체를 제거한 다음 엔지틱 리시스 버퍼(ELB)의 180 μL에서 펠릿을 다시 한 번 제거하고 삶은 RNase A(10 mg/mL)의 20 μL을 추가합니다. 그람 양성 박테리아의 효율적인 용해를 위해, 뮤타놀리신 (25 kU /mL)의 18 μL을 추가합니다. 소용돌이잘, 그리고 2 시간 동안 회전에 37 °C에서 샘플을 배양.
참고: 그람 양성 박테리아와 그람 음성 박테리아에 대 한 제조 업체의 프로토콜에 설명 된 ELB를 활용 하는 것이 좋습니다. - 제조업체의 지시에 따라 진행합니다.
참고: 원하는 경우 추가 gDNA 수율을 얻기 위해 1~2회 이상 용출 단계를 반복합니다. - 제4항에 지시된 바와 같이 추출된 gDNA의 품질을 평가하고 1주일 이내에 사용될 경우 4°C에서 gDNA를 저장한다. 또는 gDNA를 장기 저장을 위해 -20°C로 유지하십시오.
4. 추출 된 gDNA의 품질 평가
- 젤 전기포고증에 의해 품질을 평가하기 위해, 2.2 절제부에 설명된 바와 같이 1% 아가로즈 젤을 준비한다. 깨끗한 튜브에서 샘플을 준비 : 추출 된 gDNA의 1-2 μL과 파라 필름에 2 배 로딩 염료의 3 μL을 혼합합니다. 로드된 젤을 한 번 실행한 다음 UV 빛 아래에서 시각화합니다.
참고 : 성공적인 gDNA 추출은 젤의 상단에있는 이산 밴드와 최소 번짐(그림 2A)에의해 분명합니다. 번짐은 전단을 나타냅니다. gDNA 대역이 분명하지 않거나 얼룩이 상당하지 않으면 gDNA 추출을 반복하십시오. RNase A 및 Proteinase K에서 잠복기 시간을 줄이는 것이 좋습니다. 약 1.5-3 kb 의 두 밴드가 관찰되는 경우, 이것은 RNA 오염(도 2B)을시사한다. 신선한 RNase A를 준비하고 추출을 반복합니다. - 마이크로 볼륨 분광광미터별 품질을 평가하기 위해, 마이크로볼륨 분광광계에 의한 gDNA 농도 및 흡광도 비 A260/280을 측정합니다. 농도 >50 ng/μL 및 1.7-2.0 사이의 A260/280은 허용됩니다.
참고: gDNA 수율이 낮기 때문에 입력이 적고 입력이 많고, 핵염의 오염이 불충분하기 때문일 수 있다. 범위 위의 흡광도 비율은 RNA 오염을 나타냅니다. gDNA 품질이 좋지 않으면 반복 추출하십시오. - 불소계에 의한 품질을 평가하기 위해 제조업체의 지시에 따라 고감도 분석 키트 및 불소계 계측기(재료표)를사용하여 gDNA 농도를 정량화하십시오. 농도 >50 ng/μL이 바람직하다.
5. 페어링 엔드 차세대 단기 읽기 시퀀싱 및 라이브러리 준비
참고: 짧은 읽기 시퀀싱은 뚜렷한 읽기 길이와 방향으로 다양한 계측기에서 수행될 수 있습니다. 세균WGS에 150 bp (300 사이클) 페어링 엔드 시퀀싱을 권장합니다. 도서관 준비와 시퀀싱은 모두 핵심 시설 이나 상업 실험실에 아웃소싱 될 수 있습니다.
- 제조업체의 지침(재료 표)에 따라 시퀀싱라이브러리를준비합니다. 제조업체가 권장하는 최종 적재 라이브러리 농도를 따르십시오. 그러나 권장 사항은 NextSeq 계측기에서 최적의 읽기 생성을 위해 풀이 있는 라이브러리를 1.8pM로 로드하는 것이 좋습니다.
- 선택 사항이지만, 생체 분석기(재료표)를사용하여 풀이 된 라이브러리 조각 분포를 평가하고 조각 크기가 평균 600bp인지 확인합니다.
6. 나노포레 미니온 시퀀싱 라이브러리 준비
- 제조업체의 프로토콜(재료 표)에 따라 시퀀싱라이브러리를 준비합니다. 두 개의 바코드 확장 키트를 사용하면 단일 흐름 셀에서 최대 24개의 샘플을 멀티플렉싱할 수 있습니다. 24개의 샘플을 멀티플렉스할 때 한 번에 12개의 샘플, 두 부분으로 라이브러리 준비를 수행하는 것이 좋습니다. 24개의 모든 샘플은 아래에 설명된 대로 풀로 풀화될 수 있다.
참고: 샘플은 네이티브 바코드 리선전을 마무리할 때 하룻밤 사이에 4°C로 저장될 수 있습니다 - 필요한 경우 프로토콜의 정지 지점을 제공합니다. 라이브러리 준비 프로토콜의 네이티브 바코드 결찰 섹션의 끝에서, 가능한 최대 DNA 질량(ng)까지 각 샘플의 평형 양을 풀하는 것이 좋습니다.- 이렇게 하려면 제조업체의 지침에 따라 불소계(재료표)를사용하여 바코드 결찰에 따른 모든 샘플을 정량화합니다. 가장 낮은 dsDNA 농도로 샘플의 부피를 추정한 다음 이 샘플에서 발견되는 총 dsDNA를 계산합니다. 이 번호를 사용하여 함께 풀로 풀로 풀려질 다른 모든 샘플의 평수 양을 확인합니다.
참고: 평년 계산은 풀드드DNA의 양을 최대화하여 대용량 풀(>65 μL)을 산출하기 때문에 풀을 집중하려면 정리가 필요합니다.
- 이렇게 하려면 제조업체의 지침에 따라 불소계(재료표)를사용하여 바코드 결찰에 따른 모든 샘플을 정량화합니다. 가장 낮은 dsDNA 농도로 샘플의 부피를 추정한 다음 이 샘플에서 발견되는 총 dsDNA를 계산합니다. 이 번호를 사용하여 함께 풀로 풀로 풀려질 다른 모든 샘플의 평수 양을 확인합니다.
- dsDNA 풀 정리 및 농도
- DNA 풀에 2.5배 의 파라자성 구슬(재료표)을추가한 다음 튜브를 부드럽게 쓸어서 내용물의 혼합을 혼합합니다. 튜브를 회전기에 5분 동안 배치하십시오.
- 250 μL을 새로 준비한 70% 에탄올(뉴클레아제 프리 워터)을 추가하여 펠릿을 방해하지 않도록 주의하십시오. 에탄올을 흡입하고 에탄올 세척을 한 번 반복합니다.
- 두 번째 포부 후 2000 x g의 샘플을 회전하고 자석에 다시 놓습니다. 파이펫은 잔류 에탄올을 벗겨내고 샘플을 약 30s동안 건조시킬 수 있도록 합니다.
- 자석에서 튜브를 제거하고 뉴클레아제 자유 물의 60-70 μL에서 펠릿을 다시 분리합니다. RT에서 2분 동안 인큐베이션을 합니다. 엘ute가 명확해질 때까지 자석에 샘플을 펠렛한 다음 엘루트를 제거하고 깨끗한 1.5 mL 마이크로 센심 분리기 튜브로 옮김합니다.
- 플루오로미터를 사용하여 농축 된 풀을 정량화 한 다음 어댑터 결찰 단계로 진행하기 위해 알리쿼트를 준비하십시오 : 65 μL 최종 부피로 샘플의 700 ng를 준비합니다. 첫 번째 실행이 완료되면 완료될 두 번째 실행을 위해 풀의 나머지 부분을 4°C로 유지합니다.
- 제조업체의 지시에 따라 어댑터 결찰을 진행하고 흐름 셀에 샘플을 로드합니다. 시퀀싱 실행을 시작합니다.
참고: 시료 하중 전에 유동 셀 프라이밍 포트로부터 공기 및 ~200 μL의 저장 버퍼를 흡인한다. 이는 성공적인 유동 셀 프라이밍 및 샘플 로딩에 매우 중요합니다. 플로우 셀의 프라이밍 포트를 통해 솔루션을 도면하고 예치할 때 p1000 파이펫과 팁을 사용합니다.
- 제조업체의 지침에 따라 라이브러리를 시퀀스합니다.
- 시퀀싱을 위한 운영 소프트웨어를 열고 시작을 클릭합니다. 실험에 대한 이름을 입력하면 권장 명명법에는 실행 날짜와 사용자의 이름이 포함됩니다. 키트 선택 계속을클릭하고 적절한 라이브러리 준비 키트 및 바코드 확장 팩을 선택한 다음 계속 실행 옵션을 클릭합니다.
- 두 번째 실행을 위해 충분한 라이브러리를 준비하려는 경우 실행 길이를 48h로 조정합니다(그렇지 않으면 기본값 72h로 둡니다). 베이스 콜링 계속을 클릭합니다.
- 기본 호출 옵션 Config: Fast Basecalling을 확인하고 바코딩이 활성화되도록 설정되어 있는지 확인하여 FASTQ 파일을 바코드 시퀀스를 트리밍하고 바코드를 기반으로 별도의 디렉터리로 디멀티플렉션되도록 합니다. 출력 계속을 클릭합니다.
- 출력 시퀀싱 데이터를 저장할 위치를 선택합니다. FASTQ 출력을 저장하고 FAST5 출력을 저장하는 경우 >500Gb의 데이터만 절약하면 약 30-50Gb의 데이터를 기대할 수 있습니다. 필터링 옵션 Qscore 선택 취소: 7 | 읽기 길이: 섹션 7.2에 설명된 필터링을 진행할 계획이라면 필터링되지 않은 상태로 선택된 상태로 두고 읽기 길이를 200으로 조정합니다.
- 설치 를 계속실행하고 모든 설정을 검토하려면 클릭합니다. 설정이 올바른 경우 시작을클릭하고 뒤로 클릭하고 필요한 조정을 합니다.
- 원하는 경우, 흐름 셀은 제조업체의 지침에 따라 세척하고 나머지 풀로 다시 로드될 수 있습니다. 첫 번째 실행이 완료되고 흐름 셀이 세척된 후 나머지 풀에 대해 6.2의 단계를 반복합니다.
참고: 두 번째 실행을 설정할 때 이전에 48h 이상의 런에 사용되었던 흐름 셀에 대한 제조업체의 권장 사항에 따라 바이어스 전압을 -250 mV로 조정합니다.
7. 읽기 평가 및 준비
참고: 권장 디렉토리 구조가 그림 4에묘사됩니다. 아래 계산 단계를 진행하기 전에 데스크톱(즉Long_Reads, Short_Reads 및 Trimmed_Reads)에 있는 디렉터리를 만듭니다.
- 짧은 읽기 (그림 3)
참고: 짧은 읽기는 FASTQ 형식으로 생성됩니다. 파일에는 FASTQ당 최대 읽기 4000개가 포함되어 있습니다. 이들은 종종 지퍼 (.gz 아카이브) 여러 파일로 구성됩니다. 플랫폼에 따라 바코드는 일반적으로 트리밍됩니다. 일부 프로그램은 지퍼 형식으로 파일을 수락하고 다른 프로그램은 가져오기 전에 추출이 필요할 수 있습니다. 판독은 게놈 조립 중 데이터 정확도를 보장하기 위해 품질 관리(QC) 단계를 통과해야 합니다. CLC 유전체학 워크벤치를 사용할 수 없는 경우, 트림 및 TRIMmomatic과 같은 QC 짧은 읽기를 트림하는 데 대체 프로그램을 사용할 수 있습니다.25 또는 읽기 품질을 평가하기 위한 트리밍 및 FastQC(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)를 위한 풍부한 맛(https://www.bioinformatics.babraham.ac.uk/projects/trim_galore/)을 트리밍합니다. 평균 읽기 길이와 게놈 크기로 나누어 읽기 수를 곱하여 추정평균 짧은 읽기 범위는 >100x로 하는 것이 좋습니다.- 유전체학 워크벤치소프트웨어(자료 테이블)를열고 모든 페어링형 단축 읽기 FASTQ 파일을 가져옵니다. 페어링된 파일이 자동으로 생성됩니다.
- 상단 도구 모음의 새를 클릭하고 폴더를선택하여 CLC_Data 아래 새 폴더를 만듭니다... 파일을 저장합니다. 원하는 대로 폴더의 이름을 지정하면 권장되는 규칙이 샘플 ID를 사용하고 있습니다.
- 상단 도구 모음에서 가져오기 버튼을 클릭하고 일루미나를선택합니다... 샘플에 해당하는 모든 짧은 읽기 파일을 탐색하고 선택합니다. 페어링된 읽기 옵션이 선택되어 있는지 확인하고 실패한 읽기 제거 옵션을 선택 취소합니다. 다음을클릭하고 저장을선택하고 다음을 다시 클릭합니다. 이전 단계에서 만든 새 폴더에 가져온 파일을 저장하고 완료를 클릭하도록 선택합니다.
- 격리를 위해 페어링된 모든 파일의 시퀀스 목록을 만듭니다. 이렇게 하면 읽기 데이터를 단일 파일에 연결하여 분석의 단순성으로 전환됩니다.
- 상단 도구 모음에서 새 단추를 클릭하고 시퀀스 목록을선택합니다... 왼쪽의 디렉터리 목록에서 연결할 파일을 선택하고 화살표를 사용하여 오른쪽에 있는 선택한 파일 목록으로 이동합니다. 다음을클릭하고 저장을선택하고 다음을 다시 클릭합니다. 시퀀스 목록을 저장하고 완료를 클릭하도록 선택합니다.
- 시퀀스 목록이 생성되면 즉시 샘플 ID로 이름을 바꿉니다.
- 시퀀스 목록에서 읽기 시퀀싱에 대한 QC 실행: 이 절차는 짧은 읽기 NGS에 의해 생성된 읽기의 전반적인 품질 매개 변수를 평가합니다.
- 도구 상자 메뉴(왼쪽 아래 창)에서 시퀀싱 읽기 도구에 대한 QC를 검색합니다. 도구를 두 번 클릭한 다음 분석할 시퀀스 목록을 선택하고 다음을 클릭합니다.
- 모든 출력 옵션을 확인하고 결과 처리 에서 저장을 선택합니다. 다음을 클릭하고 출력 파일을 저장하도록 지정한 다음 완료를 클릭합니다.
- 시퀀스 목록에서 트림 읽기 도구를 실행: 트리밍은 품질, 길이 및 모호성에 따라 수행됩니다. 이 프로세스는 시퀀싱에 사용된 바코드가 이 단계 이전에 트리밍되었다고 가정합니다.
- 도구 상자(왼쪽 아래 창)에서 트림 읽기 도구를 검색합니다. 읽기트림을 두 번 클릭한 다음 분석할 시퀀스 목록을 선택하고 다음을 클릭합니다.
- 품질 트리밍: 품질 점수 제한을 0.01로 설정하고 모호한 뉴클레오티드를 2에 둡니다. 다음을클릭합니다.
참고: 매개 변수는 사용자 재량에 따라 조정될 수 있습니다. 이러한 설정은 권장되는 설정입니다. - 자동 읽기-스루 어댑터 트리밍의 선택을 취소합니다(CLC로 가져오기 전에 어댑터가 읽기에서 트리밍된 경우에만 이 작업을 수행합니다). 다음을 클릭하고 길이 아래 읽기 를 취소확인, 기본 15를 사용합니다.
- 다음을클릭하고 보고서 만들기를확인한 다음 저장을 선택합니다. 다음을 클릭하고 출력 파일을 저장할 위치를 지정합니다. 마무리를클릭합니다.
- 트리밍된 시퀀스 목록 내보내기: 후속 하이브리드 어셈블리 및 분석은 CLC 외부에서 완료되며 잘라서 짧은 읽기 파일을 내보내야 합니다.
- 왼쪽 상단의 디렉터리 탐색에서 7.1.4 단계에서 생성된 트리밍된 파일을 선택한 다음 상단 도구 모음에서 내보내기를 클릭합니다. 내보내기 파일 유형에 대한 Fastq를 선택하고 다음을 클릭합니다. 두 파일로 쌍시퀀스 목록을 내보내기를 확인합니다. 그런 다음 다음을 클릭하고 Trimmed_Reads 디렉토리를 선택하여 파일을 내보냅니다. 마무리를클릭합니다. 확장 .fastq를사용하여 트리밍된 짧은 읽기 파일이 두 개의 파일(R1 및 R2)으로 성공적으로 내보내도록 합니다.
참고: 트리밍된 시퀀스 목록은 일반적으로 CLC에서 R1 및 R2로 지정한 두 개의 파일로 내보내야 합니다. 이는 다운스트림 하이브리드 어셈블리에서 이와 같이 짧은 읽기 데이터 입력을 설정해야 하기 때문에 매우 중요합니다. - 내보낸 파일의 이름을 바꾸면 파일 이름에 공백과 특수 문자의 사용을 삼가하십시오. 단순성으로 권장되는 형식은 trimmed_short_file. R1.fastq.
- 왼쪽 상단의 디렉터리 탐색에서 7.1.4 단계에서 생성된 트리밍된 파일을 선택한 다음 상단 도구 모음에서 내보내기를 클릭합니다. 내보내기 파일 유형에 대한 Fastq를 선택하고 다음을 클릭합니다. 두 파일로 쌍시퀀스 목록을 내보내기를 확인합니다. 그런 다음 다음을 클릭하고 Trimmed_Reads 디렉토리를 선택하여 파일을 내보냅니다. 마무리를클릭합니다. 확장 .fastq를사용하여 트리밍된 짧은 읽기 파일이 두 개의 파일(R1 및 R2)으로 성공적으로 내보내도록 합니다.
- 유전체학 워크벤치소프트웨어(자료 테이블)를열고 모든 페어링형 단축 읽기 FASTQ 파일을 가져옵니다. 페어링된 파일이 자동으로 생성됩니다.
- 긴 (미니온) 읽기(그림 3)
참고: 하이브리드 어셈블리에 대한 롱(MinION) 시퀀싱 읽기를 준비하기 위한 다음 파이프라인은 명령줄에 의해 실행되는 NanoFilt 및 Nanostat프로그램(26)을 활용합니다. 진행 하기 전에 도구를 설치 하 고 이러한 명령을 실행 하기 위해 UNIX의 기본사항에 익숙해. 기본 단자와 Bash 셸을 권장합니다. 소프트웨어 목공27에서공통 터미널 명령 및 사용에 대한 수업 가이드가 있습니다. 아래 지침은 생성된 파일이 바코드 명명법(NB01, NB02 등)으로 명명되고 Long_Reads 디렉터리에 저장된다고 가정합니다. 또는 시퀀싱 실행을 설정할 때 MinKNOW를 사용하여 읽기 필터링을 수행할 수 있습니다. 평균 긴 읽기 범위는 > 100x로 권장됩니다. 권장평균 판독 길이는 >2000 bp; 따라서 필요한 긴 읽기 수는 짧은 읽기 수보다 낮습니다.- Long_Reads 디렉토리(그림4)에서실행에 사용되는 각 바코드(barcode01, barcode02 등)에 대한 새 디렉토리를 만듭니다. 각 바코드에 해당하는 .fastq 파일을 모두 적절한 폴더에 복사합니다. 모든 실행에서 각 바코드에 대한 모든 .fastq 파일을 결합합니다.
- 터미널을 열고 cd 명령을 사용하여 Long_Reads 디렉터리 내의 바코드 디렉토리로 이동: cd 데스크톱/Long_Reads/바코드01
- 다음 명령을 실행하여 바코드당 모든 .fastq 파일을 단일 .fastq 파일로 연결합니다: cat *.fastq > NB01.fastq
참고: 이 명령은 FASTQ 파일의 모든 읽기를 NB01.fastq라는 하나의 큰 단일 FASTQ로 결합합니다. - NanoStat를 사용하여 다음 명령을 실행하여 샘플의 판독 품질을 평가합니다: NanoStat --fastq NB01.fastq
- 출력을 텍스트 또는 Word 파일로 복사하여 결과를 기록하여 향후 참조를 기록합니다.
- 나노필렛을 사용하여 미니언읽기를 필터로 사용하여 Q < 7및 길이 < 200을 사용하여 읽기를 삭제하는 경우 명령을 실행합니다: NanoFilt -q 7 -l 200 bp NB01.fastq | gzip > NB01 _trimmed.fastq.gz
- 명령을 실행하여 7.2.6 단계에서 생성된 트리밍 된 파일에 NanoStat를 실행하십시오: NanoStat --fastq NB01 _trimmed.fastq.gz
- 출력을 텍스트 또는 Word 파일로 복사하여 결과를 기록하고 필터링이 성공되었는지 확인하기 위해 7.2.4 단계의 결과와 비교하여 필터링이 성공하도록합니다(표 1).
- 시퀀싱 실행에 사용되는 각 바코드에 대해 7.2.2 ~ 7.2.8 단계를 반복합니다.
참고: 7.2.6 단계에서 생성된 NB01_trimmed.fastq.gz 파일은 하이브리드 어셈블리에 사용됩니다.
8. 하이브리드 게놈 조립 생성
참고: 다음 조립 파이프라인은 유니사이클러19,28,29,30을 사용하여 7.1 과 7.2(그림3)에서준비된 짧고 긴 읽기를 결합합니다. 외발 자전거 및 종속성을 설치하고 아래 명령을 실행합니다. 7.1.5 단계에서 내보낸 짧은 읽기 파일은 trimmed_short_file 명명된 것으로 가정합니다. R1.fastq 및 trimmed_short_file. 단순성 R2.fastq.
- 짧은 읽기 파일과 긴 읽기 파일을 Trimmed_Reads 라는 단일 디렉토리로 구성합니다. 디렉터리에는 다음이 포함되어야 합니다.
- [7.2.6단계에서 생성된] 긴 읽기를 트리밍한 .fastq.gz 파일입니다.
- 잘라낸 짧은 읽기에 대한 두 개의 .fastq 파일(R1 및 R2)(7.1.5 단계에서 생성).
- 터미널에서 CD 명령을 사용하여 읽기 파일을 저장하는 디렉토리 Trimmed_Reads 이동: cd 데스크톱/Trimmed_Reads
- 올바른 디렉터리에서 두 개의 짧은 읽기 파일을 지퍼로 압축하여 다음 명령을 실행하여 .fastq.gz 형식에 trimmed_short_file. R1.fastq
- R1과 R2 모두에 대해 8.2 단계를 반복합니다. 이제 모든 읽기 파일이 .fastq.gz 형식인지 확인하고 모든 파일이 동일한 격리와 일치하는지 확인합니다.
- 다음 명령을 실행하여 유니사이클러를 사용하여 하이브리드 어셈블리를 시작합니다.
외발 자전거 -1 trimmed_short_file. R1.fastq.gz -2 trimmed_short_file. R2.fastq.gz -l NB01 _trimmed.fastq.gz -o unicycler_output_directory
참고: -o는 외발 자전거 출력이 저장되는 디렉토리를 지정하고, 단발자전거는 명령이 실행되면 이 디렉터리를 만듭니다. 미리 디렉터리를 생성하지 마십시오. 런타임은 게놈 크기및 읽기 수뿐만 아니라 사용되는 컴퓨터의 계산 성능에 따라 다릅니다. 이 4 시간에서 1 또는 2 일 정도 걸릴 수 있습니다. 이 프로토콜은 CentOS Linux 7 기계에서 250Gb RAM, 인텔 제온(R) CPU2.5GHz 12개의 실용적인 코어 및 48개의 가상 코어에서 수행되었습니다. 또는 16Gb RAM과 2.6GHz 6코어 프로세서가 장착된 개인용 컴퓨터는 더 긴 처리 시간에 이러한 어셈블리를 계산할 수 있습니다. - 실행이 완료되면 외발 자전거 .log 파일을 검토하여 생성된 연속의 수, 크기 및 상태(완전, 불완전)를 기록합니다.
- 불완전한 연속화가 식별되면(외발자전거 로그에서 불완전한 것으로 표시됨) 8.4 단계의 명령에 다음 플래그를 추가하여 굵은 모드에서 유니사이클러를 다시 실행하십시오.
참고: 굵은 모드는 조립 중에 긴 읽기 브리지에 대해 허용되는 품질 임계값을 낮춥습니다. 이렇게 하면 완전한 어셈블리가 생성될 수 있지만 어셈블리 품질은 저하될 수 있습니다. 필요할 때만 굵은 모드를 활용하고 나중에 PCR에 의해 확인될 연속 가입에 대한 예비 증거로 사용하는 것이 좋습니다.
- 불완전한 연속화가 식별되면(외발자전거 로그에서 불완전한 것으로 표시됨) 8.4 단계의 명령에 다음 플래그를 추가하여 굵은 모드에서 유니사이클러를 다시 실행하십시오.
9. 조립 품질 평가
참고: 다음 프로토콜은 붕대31 및 QUAST32를사용하며, 사용하기 전에 설정해야 하는 두 가지 프로그램(그림2 및 그림 4)을사용합니다. 붕대는 일단 다운로드하면 설치가 필요하지 않으며 QUAST는 기본 명령줄 사용에 익숙해야합니다. 또한 벤치마킹 범용 단일 복사 정형(BUSCO)33을사용하여 게놈 완성도를 평가하는 것이 좋습니다.
- 붕대 : 파일을클릭합니다. 그런 다음 로드 그래프를 선택하고 8.4 단계에서 유니사이클러에서 생성된 unicycler_output_directory 저장한 assembly.gfa 파일을 선택합니다. 로드되면 왼쪽 도구 모음의 그리기 그래프 버튼을 클릭하고 연속체(노드라고 함)가 어떻게 연결되고 구성되어 어셈블리가 완료되었는지(그림5)를평가합니다.
참고: 전체 어셈블리는 양쪽 끝에 연결된 단일 원형 연속체로표시됩니다(그림 5A,B). 불완전한 어셈블리에는 여러 개의 연속체가 함께 연결되거나선형(그림 5C)입니다. 작은 선형 연속체는 선형 초염색체 요소를 나타낼 수 있으므로 불완전하지 않을 수 있습니다. 깊이라고도 하는 커버리지는 붕대에 지적되며 염색체에 대한 연속체의 상대적 풍부를 나타내며, 외발자전거에서 1x로 정규화된다. - 쿼스트
- 터미널 내에서 CD 명령:cd 데스크톱/Trimmed_Reads/unicycler_output_directory 사용하여 유니사이클러 출력을 저장하는 폴더로 이동합니다.
참고: 어셈블리가 있는 경로,즉 유니사이클러 출력으로 이어지는 디렉토리가 이름에 공백을 가질 수 없습니다. 또는 쉽게 액세스할 수 있도록 assembly.fasta 파일을 데스크톱에 복사합니다. - 다음 명령을 실행하여 QUAST 실행: quast assembly.fasta -o quast_output_directory
- 출력 디렉터리 quast_output_directory QUAST에서 생성된 보고서를 검토합니다.
- 터미널 내에서 CD 명령:cd 데스크톱/Trimmed_Reads/unicycler_output_directory 사용하여 유니사이클러 출력을 저장하는 폴더로 이동합니다.
10. 게놈 성서
참고: 아래 음표 파이프라인은 사용 전에 설치해야 하는 명령줄 도구인 Prokka34를사용합니다. 또는, 자동 GUIK-베이스(재료표)를통해 Prokka를 사용하거나 웹 서버RAST(35)를통해 게놈에 부가한다. 게놈을 NCBI에 증착하는 경우, 프로카요틱 게놈 노팅 파이프라인(PGAP)36을사용하여 자동으로 인장됩니다.
- 터미널 내에서 CD 명령을 사용하여 유니사이클러 출력을 저장하는 폴더로 이동합니다(9.2.1 단계 참조). 그런 다음 prokka를 실행하여 prokka를 실행하십시오: prokka --접두사 sample_ID -prokka_output_directory assembly.fasta
참고: --접두사는 지정된 sample_ID 기반으로 모든 출력 파일의 이름을 지정합니다. - outdir는 모든 Prokka 출력 파일이 저장되는 지정된 이름으로 출력 디렉토리를 만듭니다. Prokka에 대한 출력 디렉토리를 미리 만들지 마십시오. - .tsv 테이블을 열거나 시퀀스 분석 소프트웨어에 생성된 .gff 파일을 업로드하여 주석을 시각화하고 분석하여 주석을 검토합니다(그림6).
- 주석의 특정 유형은 관심의 유전 적 요인에 따라 생성 될 수있다. 예비 분석37,38,39,40,41에대한 유전체 역학 센터 (www.genomicepidemiology.org/) 웹 서버에서 사용자 친화적 인 도구로 시작하는 것이 좋습니다. CRISPR-cas 시스템 및 프로파지 감지를 위한 추가 도구를 사용할 수있습니다(그림 3)42,43.
11. 데이터 민주화를 위한 제안 된 관행
- 가능하면 NCBI 시퀀스 읽기 아카이브(SRA) 및 Genbank와 같은 공용 저장소에 모든 원시 판독 데이터와 조립된 게놈을 입금합니다. 게놈은 NCBI 증착 프로세스 중에 PGAP 파이프라인을 통해 자동으로 인장됩니다.
결과
이 프로토콜은 도 1에나열된 제네라에 속하는 요로균의 배양 및 시퀀싱에 최적화되어 있다. 모든 오줌 박테리아는이 방법에 의해 컬터. 문화권 미디어 및 조건은 그림 1의속에 의해 지정됩니다. gDNA 무결성의 예시겔 전기전도 평가는 도 2에서묘사된다. 판독 처리, 게놈 어셈블리 및 별표 시퀀싱을 위한 생물정보학 파이프라인의 개요는 도 3에설명되어 있다. 프로토콜 이해를 단순화하고 성공적인 조직을 위한 프레임워크를 제공하기 위해 그림 4에 계산 디렉터리 구조에 대한 가이드가 제공됩니다. 더욱이, 이 프로토콜에 의해 생성된 2개의 Klebsiella spp., K. pneumoniae 및 K. oxytoca의대표적인 완전 게놈이 포함됩니다. 이러한 어셈블리의 표현은 도 5에 제공되며 또한 추가 불완전한 예 K. 폐렴 게놈을 포함한다. 완전히 인가된 각 전체 게놈에 대한 자세한 개요는 도 6에나와 있다. 마지막으로, 표 1에 시퀀싱 읽기 통계의 요약이 제공되어 고품질 폐쇄 게놈 어셈블리생성에 충분한 원시 및 트리밍 된 데이터에 대한 광범위한 이해를 제공합니다. 또한 두 대표의 주요 매개 변수는 Klebsiella spp를 완료합니다. 게놈이 나열됩니다. 게놈과 원시 데이터는 BioProject PRJNA683049에 따라 Genbank에 입금되었습니다.
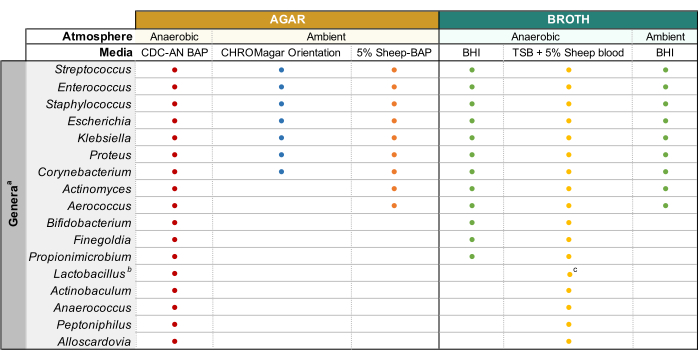
그림 1: 다양한 요로 게네라의 소변 배양을 수정했습니다. 다양한 오줌 제네라를 배양하는 데 사용될 수 있는 한천 및 액체 국물에 대한 차트. 모든 배양은 1.1절에 기재된 바와 같이 35°C에서 수행될 것을 제안된다. 원은 특정 속을 배양하는 데 적합한 미디어를 나타내며, 색상은 임의로 선택되어 한 미디어 유형을 다른 미디어 유형과 구별합니다. CDC-AN BAP (빨간색), CDC 혐기성 양 혈액 천; 5% 양-BAP (오렌지), 양 피 한천; BHI (녹색), 뇌 심장 주입; TSB (노란색), 트립틱 콩 국물; 크로마가르 오리엔테이션(파란색). 가드너엘라질은 HBT 바이레이어 G. 질경 선택적 천에서 미세 열과소및 특별한 국물 배양 요건44하에서배양되어야 한다. b유산균 이너는 5% 토끼-BAP 플레이트와 NYCIII 국물에 미세 비행대기에서 배양되어야 합니다. c유산균 스프. 미세 에어로필 조건에서 MRS에 배양 될 수있다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
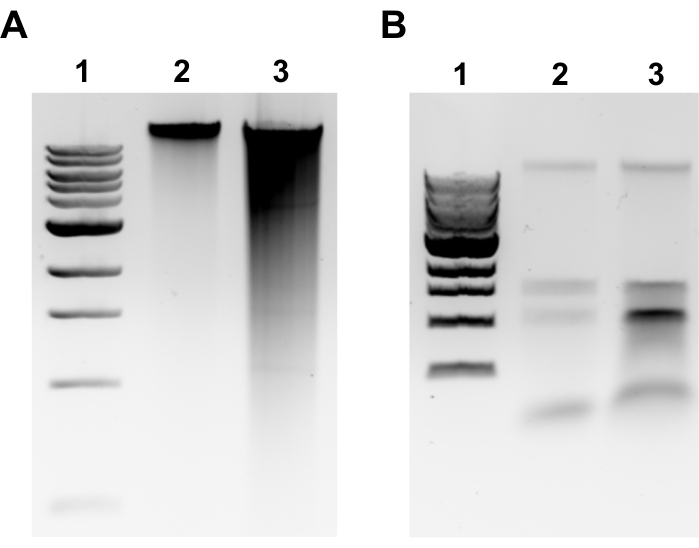
그림 2: 유전체 DNA 추출 아가로즈 젤 이미지. gDNA 추출 결과를 묘사한 대표적인 젤 이미지. (A)레인 1: 1 kb 사다리, 레인 2: 손상되지 않은 gDNA는 성공적인 추출, 레인 3: 단편화된 gDNA를 나타내는 얼룩을 나타낸다. (B)레인 1: 1 kb 사다리, 차선 2 및 3 : rRNA 오염은 1.5 kb와 3 kb 사이의 두 밴드에 의해 표시된다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
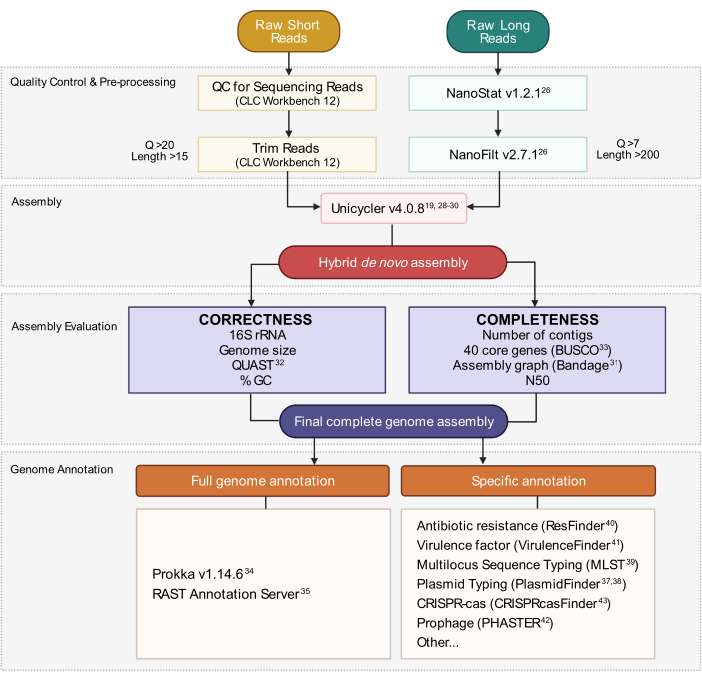
그림 3: 하이브리드 게놈 조립 워크플로우. 읽기 품질 관리 및 사전 처리에서 어셈블리 별표에 이르는 단계의 회로도. 읽기 트리밍은 모호하고 품질이 낮은 읽기를 제거합니다. Q 점수 및 길이 매개 변수가 표시되고 유지되는 읽기를 나타냅니다. 어셈블리는 짧고 긴 읽기를 모두 활용하여 하이브리드 드 노보 게놈 어셈블리를 생성합니다. 조립 품질은 지정된 도구및 매개 변수를 사용하여 완전성과 정확성에 따라 평가됩니다. 최종 게놈 어셈블리는 모든 유전자와 관심의 특정 loci에 대해 추가됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
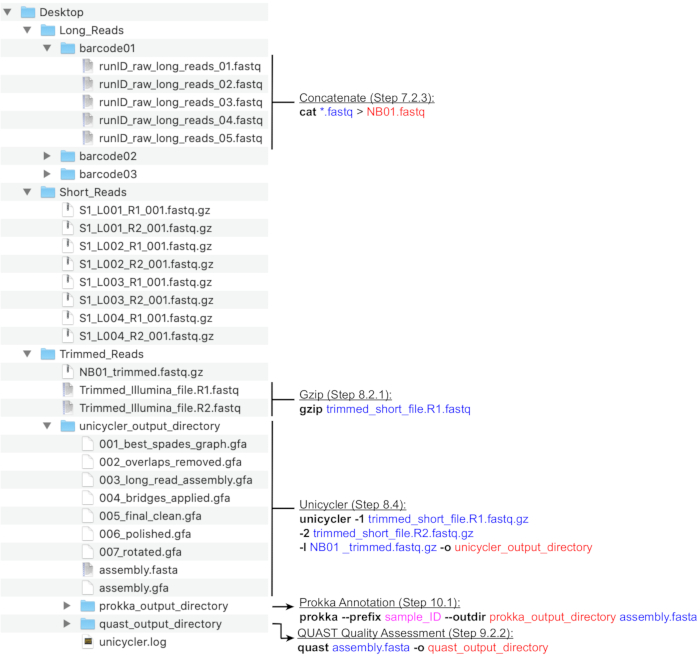
그림 4: 생물 정보학 디렉토리 구조 가이드. 짧고 긴 읽기, 하이브리드 어셈블리 및 게놈 별표 및 QC의 처리를 위한 권장 디렉토리 및 파일 조직의 회로도. 주요 명령줄 데이터 처리 단계는 해당 파일 및 디렉터리 옆에 강조 표시됩니다. 명령 및 플래그(굵게), 입력 파일(파란색), 출력 파일 또는 디렉토리(빨간색), 파일 명명 규칙(마젠타)과 같은 사용자 입력을 유도합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
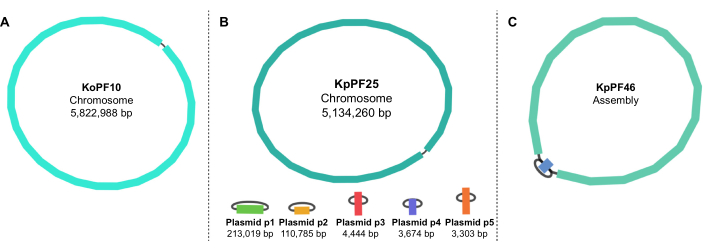
그림 5: 붕대에 의한 게놈 어셈블리 그래프. 대표적인 완전한 게놈 조립 그래프(A) Klebsiella oxytoca KoPF10 및(B) 클렙시엘라 폐렴 KpPF25 및 불완전한 게놈 조립의(C) Klebsiella pneumoniae KpPF46. KoPF10의 완전한 게놈은 단일 폐쇄 염색체를 보여주고 KpPF25의 완전한 게놈은 닫힌 염색체 및 5개의 닫힌 플라스미드로 이루어져 있습니다. KpPF46의 불완전한 염색체는 두 개의 상호 연결된 연속체로 구성됩니다. 외발 자전거 하이브리드 드 노버 어셈블리는 붕대에 의해 시각화되는 어셈블리 그래프를 생성합니다. 어셈블리 그래프는 단일 연속의 두 끝을 연결하는 링커에 의해 닫힌 염색체 또는 플라스미드를 나타내는 게놈의 단순 한 회로도를 제공합니다. 상호 연결된 연속체가 하나 이상 존재하면 불완전한 어셈블리가 있음을 나타냅니다. 붕대에서도 연속적인 크기와 깊이를 지적할 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
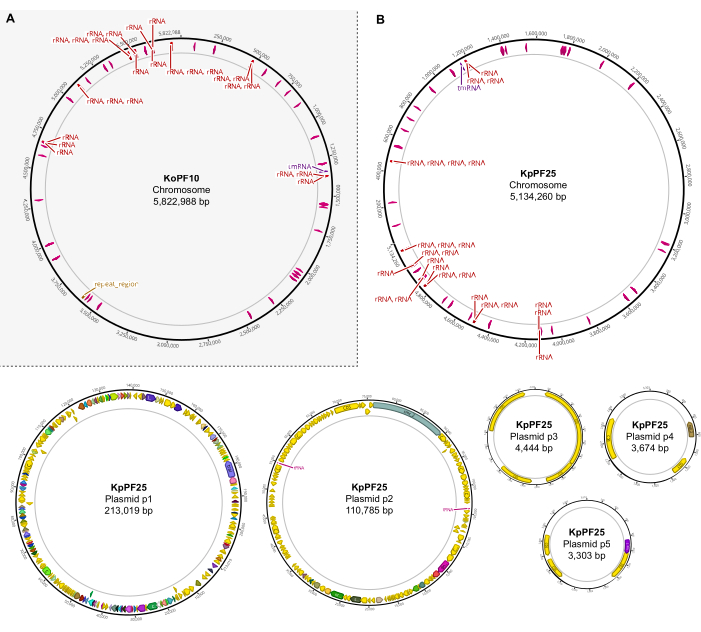
그림 6: 추가 된 하이브리드 어셈블리의 완전한 게놈지도. (A) K. 옥시토카 KoPF10 및(B) K. 폐렴 KpPF25의 완전한 게놈에 대한 제네어스 프라임에 의해 생성된 조립 지도는 플라스미드 백본을 따라 유색 화살표로 표시된 불음 유전자를 보여주는 것이다. 염색체는 단순성만을 위한 rRNA 및 tRNA 유전자를 보여줍니다. 게놈 주석은 이 프로토콜의 섹션 10에 표시된 바와 같이 Prokka를 사용하여 수행되었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
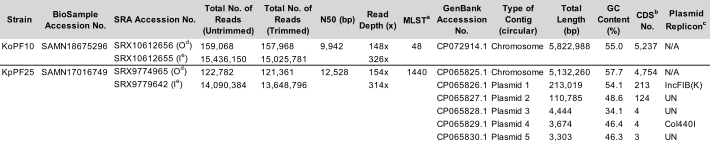
표 1: 대표 클레브시엘라 spp. K. 옥시토카 균주 KoPF10 및 K. 폐렴 균주 KpPF25. NCBI에 대한 증착된 데이터에 대한 수탁 번호가 제공된다. 트리밍 이전과 트리밍 후 의 읽기 수는 시퀀싱 기술 모두에 대해 지정됩니다. N50은 짧은 읽기가 제어 된 길이이기 때문에 긴 읽기에 대해 제공됩니다. 플라스미드 레플리시온은 플라스미드파인더 v2.1 엔테로박테리아세이 데이터베이스를 사용하여 80%의 정체성과 60%길이로 설정된 파라미터를 사용할 것으로 예측했다. MLST, 멀티로커스 시퀀스 유형. b CDS, 코딩 시퀀스. c 플라스미드 레플리콘은 플라스미드파인더 v2.1 엔테로박테리아세이 데이터베이스를 사용하여 80%의 정체성과 60%길이로 설정된 파라미터를 사용할 것으로 예측하였다. d 옥스포드 나노 포어 기술 (ONT) 읽기 데이터를 입금. e 일루미나예금 읽기 데이터. 이 테이블을 다운로드하려면 여기를 클릭하십시오.
토론
여기에서 기술된 포괄적인 하이브리드 게놈 조립 프로토콜은 다양한 오줌 microbiota 및 비뇨기요법의 성공적인 배양및 그들의 게놈의 완전한 조립을 위한 능률적인 접근을 제공합니다. 세균성 게놈의 성공적인 WGS는 그들의 게놈 DNA를 추출하기 위하여 다양하고 때때로 까다로운 세균의 격리로 시작합니다. 현재까지, 기존의 소변 배양 프로토콜은 많은 오줌 종을 검출하는 데 필요한 감도가 부족하거나 연장 된 시간과 자원을 필요로하는 길고 광범위한 접근 방식을 포함(11). 설명된 수정된 향상된 소변 문화 접근은 잠재적으로 병원성 또는 유익한 성공종을 포함하여 17개의 일반적인 오줌 제네라에 속하는 박테리아의 성공적인 격리를 위한 단순화되면서도 포괄적인 프로토콜을 제안하며, 촉진및 의무유산소 또는 혐기성 박테리아. 이것은 차례차례로 세균게놈의 정확한 순서 및 조립및 오줌 건강과 질병의 이해에 기여하는 중요한 현상제 실험을 위한 필요한 시작 물질을 제공합니다. 더욱이, 이 수정된 배양 접근법은 소변 견본에서 찾아낸 실행 가능한 미생물의 보다 정의된 임상 진단을 제공하고 미래 게놈 연구를 위한 그들의 biobanking를 허용합니다. 그러나 이 프로토콜은 제한 없이 는 아닙니다. 그것은 유기체에 따라서 긴 잠복기뿐만 아니라 쉽게 사용할 수 없을 수 있습니다 저산소실 또는 제어 인큐베이터와 같은 자원의 사용을 필요로 할 수있다. 혐기성 GasPaks의 사용은 대체 솔루션을 제공하지만 비용이 많이 들고 항상 지속적이고 통제 된 환경을 생성하지는 않습니다. 마지막으로, 배양 편향뿐만 아니라 샘플 다양성은 특정 유기체와 비뇨기과균이 까다로운 박테리아를 능가하는 것을 허용할 수 있습니다. 이러한 제한에도 불구 하 고, 다양 한 오줌 박테리아의 문화는이 접근에 의해 가능.
유전체 시퀀싱은 차세대 시퀀싱 기술의 발전으로 인기를 얻고 있으며, 이는 시퀀싱 데이터14,15의수율과 정확도를 크게 증가시켰습니다. 데이터 처리 및 드 노보 어셈블리를 위한 알고리즘의 개발과 함께 완전한 게놈 서열은 초보자와 전문 과학자 모두15,45의손끝에 있습니다. 완전한 게놈에 의해 제공된 전반적인 게놈 조직의 지식은 유전자 중복, 유전자 손실 및 수평 유전자 전송14를포함하여 중요한 진화적이고 생물학 통찰력을 제공합니다. 추가적으로, 항균 저항 및 독성에 중요한 유전자는 수시로 초안 게놈 어셈블리에서 전형적으로 해결되지 않는 모바일 요소에 국한됩니다15,16.
본 명세서에서 프로토콜은 완전한 게놈 어셈블리를 생성하기 위해 짧은 읽기 및 긴 읽기 플랫폼에서 데이터를 시퀀싱하는 조합을 위한 하이브리드 접근 방식을 따릅니다. 오줌 세균게놈에 집중하는 동안, 이 절차는 각종 격리 근원에서 다양한 박테리아에 적응될 수 있습니다. 이 접근법의 중요한 단계는 적당한 살균 기술을 따르고 순수한 오줌 박테리아의 격리를 위한 적당한 매체 및 문화 조건을 이용하는 것을 포함합니다. 또한, 고수율 gDNA의 추출은 조립 성공을 저해할 수 있는 오염 판독이 없는 시퀀싱 데이터를 생성하는 데 필수적입니다. 후속 라이브러리 준비 프로토콜은 충분한 길이와 깊이의 품질 읽기 생성에 매우 중요합니다. 따라서, 이 기술의 가장 큰 장점은 이론적 상반수 제한없이 긴 읽기의 생성이기 때문에, 특히 긴 읽기 시퀀싱에 대한 라이브러리 준비 중에 주의gDNA를 처리하는 것이 매우 중요합니다. 또한 시끄러운 데이터를 제거하고 어셈블리 결과를 향상시키는 시퀀싱 읽기의 적절한 품질 관리(QC)에 대한 섹션도 설명되어 있습니다.
성공적인 DNA 격리, 라이브러리 준비 및 시퀀싱에도 불구하고, 일부 종의 게놈 아키텍처의 특성은 여전히 폐쇄 게놈 어셈블리45,46의생성에 대한 장애물을 제공 할 수 있습니다. 반복적인 시퀀스는 종종 어셈블리 계산을 복잡하게 하며 긴 읽기 데이터에도 불구하고 이러한 영역은 낮은 신뢰도로 해결되거나 전혀 해결되지 않을 수 있습니다. 따라서 긴 읽기는 게놈 또는 커버리지에서 가장 큰 반복 영역보다 평균적으로 더 길어야 한다 (>100x)19. 일부 게놈은 불완전한 상태로 남아 있으며 완료를 위해 수동 접근이 필요할 수 있습니다. 그럼에도 불구하고, 하이브리드 조립 불완전한 게놈은 일반적으로 짧은 읽기 초안 게놈 보다는 더 적은 연속으로 이루어지고 있습니다. 어셈블리 알고리즘의 기본 매개 변수를 조정하거나 읽기 QC에 대한 보다 엄격한 차단을 따르는 것이 도움이 될 수 있습니다. 또는 가장 가능성이 높은 어셈블리 경로에 대한 증거를 찾기 위해 불완전한 영역에 긴 판독을 매핑한 다음 증폭 된 영역의 PCR 및 Sanger 시퀀싱을 사용하여 경로를 확인하는 것이 좋습니다. Minimap2를 사용하여 읽기 매핑이 제안되고 붕대는 연속연결(47)에대한 증거를 제공하는 조립된 연속화를 따라 매핑된 읽기의 시각화를 위한 유용한 도구를 제공합니다.
완전한 게놈을 생성하는 추가적인 과제는 명령줄 도구를 사용하여 친숙함과 편안함에 있습니다. 많은 생물 정보 도구는 모든 사용자에게 계산 기회를 제공하기 위해 개발되었습니다. 그러나, 그들의 활용은 UNIX 및 프로그래밍의 기초와 이해에 의존. 이 프로토콜은 사전 명령줄 경험이 없는 개인이 폐쇄된 게놈 어셈블리를 생성하고 이에 대해 알수 할 수 있도록 충분히 상세한 지침을 제공하는 것을 목표로 합니다.
공개
저자는 공개 할 것이 없습니다.
감사의 말
우리는 이 프로토콜에 기여한 무두세 주바이다 이슬람 박사와 루크 조이스 박사에게 감사드립니다. 우리는 또한 그들의 의견과 지원을 위해 달라스 게놈 센터에 있는 텍사스 대학을 인식하고 싶습니다. 이 작품은 웰치 재단에 의해 지원되었다, 수상 번호 AT-2030-20200401 N.J.D.에, 건강의 국립 연구소에 의해, K.P.에 수여 번호 R01AI116610, 그리고 펠레시아와 존 케인 의자에 의해, P.E.Z.에 의해 개최.
자료
| Name | Company | Catalog Number | Comments |
| Equipment: | |||
| Bioanalyzer 2100 | Agilent | G29398A | Optional but recommended |
| Centrifuge | Eppendorf | -- | Any centrifuge for spinning conicals and microcentrifuge tubes (e.g. Models 5810R/5424R) |
| Electrophoresis | BioRad Laboratories | 1645070 | |
| Gel Imaging System | BioRad Laboratories | ChemiDoc models | |
| Incubator | ThermoFisher Scientific | -- | Any CO2 Incubator (e.g. Thermo Forma model 3110) |
| Magnetic Rack | New England BioLabs | S15095 | 12-tube rack |
| MinION | Oxford Nanopore Technologies | -- | |
| Nanodrop | ThermoFisher Scientific | ND-ONE-W | |
| NextSeq 500 | Illumina | SY-415-1002 | Other Illumina models are acceptable |
| Plate Reader | BioTek | -- | Synergy H1 |
| Qubit fluorometer | ThermoFisher Scientific | Q33238 | |
| Rotator | Benchmark Scientific | H2024 | |
| Thermocycler | ThermoFisher Scientific | -- | Any thermocycler for PCR reactions (e.g. ProFlex PCR system) |
| Materials: | |||
| 10X Phosphate Buffered Saline (PBS) | Fisher Scientific | BP3991 | |
| 10X TBE buffer | -- | -- | 1M Tris,1M Boric Acid,0.2M EDTA (pH 8.0) |
| 1429R primer | Sigma Aldrich (Custom oligos) | -- | GGTTACCTTGTTACGACTT |
| 1kb Ladder | VWR | 101228-494 | |
| 1M Tris-Cl (pH 7.5) | ThermoFisher Scientific | 15567027 | |
| 6x Loading dye | Fisher Scientific | NC0783588 | |
| 8F primer | Sigma Aldrich (Custom oligos) | -- | AGAGTTTGATCCTGGCTCAG |
| Agar | Fisher Scientific | BP1423-2 | |
| Agarose | BioRad Laboratories | 63001 | |
| AMPure XP Beads | Beckman Coulter | A63880 | |
| Anaerobe Pouch System - GasPak EZ | BD Diagnostic Systems | B260683 | |
| Boric Acid | Fisher Scientific | A73-500 | |
| Brain Heart Infusion Broth | BD Diagnostic Systems | 212304 | |
| CDC Anaerobe 5% Sheep Blood Agar | BD Diagnostic Systems | L007357 | |
| CHROMagar Orientation | BD Diagnostic Systems | PA-257481.04 | |
| DNeasy Blood & Tissue | QIAGEN | 69504 | |
| DreamTaq Master Mix | ThermoFisher Scientific | K1081 | |
| Dry Anaerobic Indicator Strips | BD Diagnostic Systems | 271051 | |
| EDTA | Fisher Scientific | S311-500 | |
| Ethanol 200 Proof | Sigma Aldrich | E7023 | For molecular biology |
| Ethidium Bromide | ThermoFisher Scientific | BP130210 | |
| Flow cell priming kit | Oxford Nanopore Technologies | EXP-FLP002 | |
| Flow cell wash kit | Oxford Nanopore Technologies | EXP-WSH003 | |
| Gel Extraction Miniprep Kit | BioBasic | BS654 | |
| Ligation sequencing kit | Oxford Nanopore Technologies | SQK-LSK109 | |
| Lysozyme | Research Products International Corp | L381005.05 | |
| Mutanolysin | Sigma Aldrich | M9901-5KU | |
| Native barcoding expansion 1-12 | Oxford Nanopore Technologies | EXP-NBD104 | |
| NEB Blunt/TA Ligase Master Mix | New England BioLabs | M0367L | |
| NEBNext FFPE DNA Repair Mix | New England BioLabs | M6630L | |
| NEBNext quick ligation buffer | New England BioLabs | B6058S | |
| NEBNext Ultra II End repair / dA-tailing module | New England BioLabs | E7546L | |
| Nextera DNA CD Indexes | Illumina | 20018708 | |
| Nextera DNA Flex Library Prep - (M) Tagmentation | Illumina | 20018705 | |
| Nuclease-free water | Sigma Aldrich | W4502 | |
| Qubit 1X dsDNA HS Assay Kit | ThermoFisher Scientific | Q33230 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Quick T4 DNA Ligase | New England BioLabs | E6056L | |
| R9 Flow cell | Oxford Nanopore Technologies | FLO-MIN106D | |
| RNase A | ThermoFisher Scientific | EN0531 | |
| Sheep Blood | Hemostat Laboratories | DS13250 | |
| TE buffer | -- | -- | 10mM Tris, 1mM EDTA (pH 8.0) |
| Triton X-100 | Sigma Aldrich | T8787 | |
| Tryptic Soy Broth | BD Diagnostic Systems | 211825 | |
| Software & Bioinformatic Tools: | |||
| Bandage | -- | -- | https://rrwick.github.io/Bandage/ |
| Center for Genomic Epidemiology | -- | -- | http://www.genomicepidemiology.org/ |
| CLC Genomics Workbench 12 | QIAGEN | -- | |
| CRISPRcasFinder | -- | -- | https://crisprcas.i2bc.paris-saclay.fr/ |
| FastQC | -- | -- | https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ |
| Geneious Prime | Geneious | -- | |
| gVolante (BUSCO) | -- | -- | https://gvolante.riken.jp/ |
| Kbase Prokka Wrapper | -- | -- | https://kbase.us/applist/apps/ProkkaAnnotation/annotate_contigs/release |
| Minimap2 | -- | -- | https://github.com/lh3/minimap2 |
| MinKNOW | Oxford Nanopore Technologies | -- | |
| NanoFilt | -- | -- | https://github.com/wdecoster/nanofilt |
| NanoStat | -- | -- | https://github.com/wdecoster/nanostat |
| PHASTER | -- | -- | https://phaster.ca/ |
| Prokka | -- | -- | https://github.com/tseemann/prokka |
| QUAST | -- | -- | http://quast.sourceforge.net/quast |
| Trim Galore | -- | -- | https://www.bioinformatics.babraham.ac.uk/projects/trim_galore/ |
| Trimmomatic | -- | -- | http://www.usadellab.org/cms/?page=trimmomatic |
| Unicycler | -- | -- | https://github.com/rrwick/Unicycler#necessary-read-length |
참고문헌
- Brubaker, L., Wolfe, A. The urinary microbiota: a paradigm shift for bladder disorders. Current Opinion in Obstetrics & Gynecology. 28 (5), 407-412 (2016).
- Neugent, M. L., Hulyalkar, N. V., Nguyen, V. H., Zimmern, P. E., De Nisco, N. J. Advances in understanding the human urinary microbiome and its potential role in urinary tract infection. mBio. 11 (2), (2020).
- Klein, R. D., Hultgren, S. J. Urinary tract infections: microbial pathogenesis, host-pathogen interactions and new treatment strategies. Nature Reviews. Microbiology. 18 (4), 211-226 (2020).
- Horsley, H., et al. Enterococcus faecalis subverts and invades the host urothelium in patients with chronic urinary tract infection. PLoS One. 8 (12), 83637 (2013).
- Reitzer, L., Zimmern, P. Rapid growth and metabolism of uropathogenic Escherichia coli in relation to urine composition. Clinical Microbiology Reviews. 33 (1), 00101-00119 (2019).
- Snyder, J. A., et al. Transcriptome of uropathogenic Escherichia coli during urinary tract infection. Infection and Immunity. 72 (11), 6373-6381 (2004).
- Ipe, D. S., Horton, E., Ulett, G. C. The basics of bacteriuria: Strategies of microbes for persistence in urine. Frontiers in Cellular and Infection Microbiology. 6, 14 (2016).
- Babikir, I. H., et al. The impact of cathelicidin, the human antimicrobial peptide LL-37 in urinary tract infections. BMC Infectious Diseases. 18 (1), 17 (2018).
- Jancel, T., Dudas, V. Management of uncomplicated urinary tract infections. The Western Journal of Medicine. 176 (1), 51-55 (2002).
- Ventola, C. L. The antibiotic resistance crisis: part 1: causes and threats. P & T. 40 (4), 277-283 (2015).
- Price, T. K., et al. The clinical urine culture: Enhanced techniques improve detection of clinically relevant microorganisms. Journal of Clinical Microbiology. 54 (5), 1216-1222 (2016).
- Kass, E. H. Asymptomatic infections of the urinary tract. Transactions of the Association of American Physicians. 69, 56-64 (1956).
- Garcia, L. S. . Clinical microbiology procedures handbook. 3rd edn. , (2010).
- Fraser, C. M., Eisen, J. A., Nelson, K. E., Paulsen, I. T., Salzberg, S. L. The value of complete microbial genome sequencing (you get what you pay for). Journal of Bacteriology. 184 (23), 6403-6405 (2002).
- Chen, Z., Erickson, D. L., Meng, J. Benchmarking hybrid assembly approaches for genomic analyses of bacterial pathogens using Illumina and Oxford Nanopore sequencing. BMC Genomics. 21 (1), 631 (2020).
- Greig, D. R., Dallman, T. J., Hopkins, K. L., Jenkins, C. MinION nanopore sequencing identifies the position and structure of bacterial antibiotic resistance determinants in a multidrug-resistant strain of enteroaggregative Escherichia coli. Microbial Genomics. 4 (10), 000213 (2018).
- Carraro, D. M., et al. PCR-assisted contig extension: stepwise strategy for bacterial genome closure. Biotechniques. 34 (3), 626-628 (2003).
- Tettelin, H., Radune, D., Kasif, S., Khouri, H., Salzberg, S. L. Optimized multiplex PCR: efficiently closing a whole-genome shotgun sequencing project. Genomics. 62 (3), 500-507 (1999).
- Wick, R. R., Judd, L. M., Gorrie, C. L., Holt, K. E. Unicycler: Resolving bacterial genome assemblies from short and long sequencing reads. PLoS Computational Biology. 13 (6), 1005595 (2017).
- Singhal, N., Kumar, M., Kanaujia, P. K., Virdi, J. S. MALDI-TOF mass spectrometry: an emerging technology for microbial identification and diagnosis. Frontiers in Microbiology. 6, 791 (2015).
- Turner, S., Pryer, K. M., Miao, V. P., Palmer, J. D. Investigating deep phylogenetic relationships among cyanobacteria and plastids by small subunit rRNA sequence analysis. The Journal of Eukaryotic Microbiology. 46 (4), 327-338 (1999).
- Weisburg, W. G., Barns, S. M., Pelletier, D. A., Lane, D. J. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology. 173 (2), 697-703 (1991).
- Janda, J. M., Abbott, S. L. 16S rRNA gene sequencing for bacterial identification in the diagnostic laboratory: pluses, perils, and pitfalls. Journal of Clinical Microbiology. 45 (9), 2761-2764 (2007).
- Stevenson, K., McVey, A. F., Clark, I. B. N., Swain, P. S., Pilizota, T. General calibration of microbial growth in microplate readers. Science Reports. 6, 38828 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- De Coster, W., D'Hert, S., Schultz, D. T., Cruts, M., Van Broeckhoven, C. NanoPack: visualizing and processing long-read sequencing data. Bioinformatics. 34 (15), 2666-2669 (2018).
- Wilson, G., et al. The UNIX Shell. Zenodo. , (2019).
- Bankevich, A., et al. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing. Journal of Computational Biology. 19 (5), 455-477 (2012).
- Vaser, R., Sovic, I., Nagarajan, N., Sikic, M. Fast and accurate de novo genome assembly from long uncorrected reads. Genome Research. 27 (5), 737-746 (2017).
- Walker, B. J., et al. Pilon: an integrated tool for comprehensive microbial variant detection and genome assembly improvement. PLoS One. 9 (11), 112963 (2014).
- Wick, R. R., Schultz, M. B., Zobel, J., Holt, K. E. Bandage: interactive visualization of de novo genome assemblies. Bioinformatics. 31 (20), 3350-3352 (2015).
- Gurevich, A., Saveliev, V., Vyahhi, N., Tesler, G. QUAST: quality assessment tool for genome assemblies. Bioinformatics. 29 (8), 1072-1075 (2013).
- Simao, F. A., Waterhouse, R. M., Ioannidis, P., Kriventseva, E. V., Zdobnov, E. M. BUSCO: assessing genome assembly and annotation completeness with single-copy orthologs. Bioinformatics. 31 (19), 3210-3212 (2015).
- Seemann, T. Prokka: rapid prokaryotic genome annotation. Bioinformatics. 30 (14), 2068-2069 (2014).
- Aziz, R. K., et al. The RAST server: rapid annotations using subsystems technology. BMC Genomics. 9, 75 (2008).
- Tatusova, T., et al. NCBI prokaryotic genome annotation pipeline. Nucleic Acids Research. 44 (14), 6614-6624 (2016).
- Carattoli, A., Hasman, H. PlasmidFinder and In Silico pMLST: Identification and Typing of Plasmid Replicons in Whole-Genome Sequencing (WGS). Methods in Molecular Biology. 2075, 285-294 (2020).
- Carattoli, A., et al. In silico detection and typing of plasmids using PlasmidFinder and plasmid multilocus sequence typing. Antimicrobial Agents and Chemotherapy. 58 (7), 3895-3903 (2014).
- Larsen, M. V., et al. Multilocus sequence typing of total-genome-sequenced bacteria. Journal of Clinical Microbiology. 50 (4), 1355-1361 (2012).
- Bortolaia, V., et al. ResFinder 4.0 for predictions of phenotypes from genotypes. The Journal of Antimicrobial Chemotherapy. 75 (12), 3491-3500 (2020).
- Joensen, K. G., et al. Real-time whole-genome sequencing for routine typing, surveillance, and outbreak detection of verotoxigenic Escherichia coli. Journal of Clinical Microbiology. 52 (5), 1501-1510 (2014).
- Arndt, D., et al. PHASTER: a better, faster version of the PHAST phage search tool. Nucleic Acids Research. 44 (1), 16-21 (2016).
- Couvin, D., et al. CRISPRCasFinder, an update of CRISRFinder, includes a portable version, enhanced performance and integrates search for Cas proteins. Nucleic Acids Research. 46 (1), 246-251 (2018).
- Totten, P. A., Amsel, R., Hale, J., Piot, P., Holmes, K. K. Selective differential human blood bilayer media for isolation of Gardnerella (Haemophilus) vaginalis. Journal of Clinical Microbiology. 15 (1), 141-147 (1982).
- Nagarajan, N., Pop, M. Sequence assembly demystified. Nat Reviews. Genetics. 14 (3), 157-167 (2013).
- Phillippy, A. M., Schatz, M. C., Pop, M. Genome assembly forensics: finding the elusive mis-assembly. Genome Biology. 9 (3), 55 (2008).
- . Unicycler Wiki Available from: https://github.com/rrwick/Unicycler/wiki (2017)
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유