需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
使用二分法减少牛卵母细胞中的线粒体DNA
* 这些作者具有相同的贡献
摘要
在这里,我们提出了一种方案,以显着减少牛卵母细胞中的线粒体DNA拷贝数(P <0.0001)。该方法利用离心和二分切来大大减少卵母细胞线粒体,并且可以增加在重建的种间体细胞核移植胚胎中发育的机会。
摘要
种间体细胞核转移(iSCNT)可用于拯救濒危物种,但重建的胚胎中存在两个不同的线粒体DNA(mtDNA)群体:一个在受体卵质内,一个在供体体细胞内。这种线粒体异质体可导致胚胎和胎儿的发育问题。手工克隆方案包括卵母细胞二裂,其可用于减少mtDNA拷贝数,降低重建胚胎中线粒体异质体的程度。离心剥脱的成熟牛卵母细胞在卵母细胞的一极产生可见的线粒体致密部分。卵母细胞的透明带通过暴露于原胶酶溶液中去除。使用微刀片进行一对剖除以除去可见的线粒体部分。qPCR用于量化从整个卵母细胞和双裂卵母细胞中提取的DNA样品中存在的mtDNA,从而提供了切片前后mtDNA拷贝数的比较。使用周期阈值、标准曲线的回归线公式以及包括 mtDNA PCR 产物和基因组 PCR 产物各自大小的比率来计算拷贝数。一头牛卵母细胞的平均mtDNA拷贝数(±标准差)为137,904±94,768(n = 38)。一个线粒体耗尽的ooplast的平均mtDNA拷贝数为8,442±13,806(n = 33)。存在于富含线粒体的卵巢中的平均mtDNA拷贝数为79,390±58,526个mtDNA拷贝(n = 28)。这些计算平均值之间的差异表明,与原始卵母细胞相比,离心和随后的平分可以显着降低线粒体耗尽的ooplast中存在的mtDNA拷贝数(P <0.0001,由单向方差分析确定)。mtDNA的减少应降低重建胚胎中线粒体异质体的程度,可能促进标准胚胎和胎儿发育。补充来自体细胞供体的线粒体提取物对于实现成功的胚胎发育也可能是必不可少的。
引言
体细胞核转移(SCNT)包括来自一只动物的去核卵母细胞和来自同一物种动物的体细胞的融合。在大多数情况下,卵母细胞和体细胞起源于同一物种,活产率低于6%1。一些研究涉及使用种间SCNT(iSCNT),其中包括来自两个不同物种的体细胞和卵母细胞的融合。在这些研究中,活产率甚至低于SCNT,通常低于1%1。然而,iSCNT有能力用作拯救濒危物种的方法,因为这些动物的体细胞比它们的生殖细胞更容易获得1。iSCNT中使用的受体卵母细胞通常是家用或常见的实验室物种,如牛,猪和小鼠。到目前为止,一些尝试已经成功地生产出活的幼崽,尽管产生的后代是属内动物(受体卵母细胞物种和供体细胞物种是同一属的成员)2,3,4。属间模型(利用来自不同属动物的卵母细胞和体细胞)尚未产生活体动物,大多数重建的胚胎在体外发育的8-16细胞阶段停止5,6,7,8。这种胚胎发育停滞的一种可能的解释是胚胎中线粒体异质体的发生 - 单个细胞中存在一种以上的线粒体DNA(mtDNA)类型。异型增生术可导致胚胎或活体动物发育效率低下或失败等问题1。发病机制也可能发生在动物生命的后期9.虽然这个问题也存在于SCNT后代中,但iSCNT胚胎中的种间成分加剧了这个问题。
当胚胎mtDNA来自两个不同的物种时,代表大多数的受体卵母细胞线粒体不能有效地与供体细胞的细胞核1,10一起工作。iSCNT中使用的两个物种之间更大的分类学差距可能加剧了这一问题;产生属内活的后代(使用金牛座卵母细胞的博斯高鲁斯和博斯印度支原体后代)以及通过传统SCNT产生的后代(例如使用奥维斯白羊卵母细胞的奥维斯白羊座后代)被证明是嵌合体(来自两个个体的mtDNA存在于这些动物11,12,13中)。然而,它们比代间SCNT胚胎14,15发育得更远。卵母细胞线粒体和供体细胞核之间的信息交换在属内胚胎中可能比在代间胚胎中更成功16。
成熟牛卵母细胞中mtDNA的量大约是一个体细胞12中发现的量的100倍。降低该比例可以鼓励体细胞线粒体在重建的胚胎内增殖,从而允许存在更多的生产性线粒体群体16。这反过来又可以提供更多的能量来满足发育中的胚胎15的要求。先前为减少卵母细胞或胚胎的mtDNA拷贝数所做的尝试包括化学应用、显微操作、以及用来自供体细胞种16、17、18、19、20的附加线粒体补充卵母细胞或胚胎。然而,化学应用(如2',3'-二脱氧胞苷)对于胚胎发育并不理想,并且已将卵母细胞mtDNA拷贝数减少了约18的一半。先前通过显微操作减少卵母细胞mtDNA仅平均去除了卵母细胞mtDNA17的64%。虽然补充供体细胞线粒体可能是一个可行的选择,但在iSCNT研究21中,其使用尚未产生活的代间动物。
使用二分法减少卵母细胞mtDNA拷贝数尚未在已发表的研究中使用。将卵母细胞一分为二,目的是将卵母细胞与体细胞融合在一起是手工克隆(HMC)的前提,它通常使用二分切作为从中期II(MII)卵母细胞中去除极体和中期板的方法。HMC已经成功地在几个物种中产生了后代,包括山羊,牛,猪,绵羊和马22,23,24,25,26,但通常不包括在一对分离之前的离心步骤。整合卵母细胞的高速离心允许在卵母细胞的一极分离线粒体(因此mtDNA),然后可以使用微刀片将其一分为二以除去那些线粒体致密的组分。然后,两个线粒体耗尽的ooplast可以与体细胞融合,就像HMC中的情况一样,形成一个重建的胚胎,其含有来自卵母细胞物种的mtDNA相当少。
我们试图用这个方案来回答的问题是如何减少牛卵母细胞中的mtDNA,以产生含有较少异质mtDNA的活的重建胚胎。在该协议中,卵母细胞被离心并一分为二。计算卵母细胞和完整的卵母细胞mtDNA拷贝数,以确定该技术在减少牛卵母细胞的mtDNA拷贝数方面的有效性。
Access restricted. Please log in or start a trial to view this content.
研究方案
以下协议遵循犹他州立大学提供的动物护理和伦理指南。
1. 培养基准备
- 在处理卵母细胞之前,准备以下溶液,如 表1所述:400μL透明质酸酶溶液,500μL T2培养基,1,020μL T20培养基和800μL T10培养基。
- 将T10培养基分成四孔板的两个孔,每孔400μL。用"M"标记一个井,用"MR"标记第二个孔。放入5%CO2 培养箱中,直到切片后。
- 用HEPES(HSOF)制备500μL细胞查拉菌素B(CB)/合成输卵管液。将50μLCB / HSOF溶液等分到单独的1.5 mL离心管中。
- 准备40μL原酶溶液。以2,680 x g 离心至少30 秒。将20μL上清液与20μLT2混合到新的离心管中;形成稀释的5mg / mL蛋白酶溶液。
- 准备 3 毫升高纯营养物质。在搜索板上,分别沉积四个400μL滴剂;将此板放在立体显微镜下。
- 制备500μLCB / T20溶液。
2. 牛卵母细胞的 体外 成熟(IVM)
- IVM:在含有10%FBS,0.26 IU / mL FSH和100 U / mL青霉素/链霉素的四孔成熟培养基培养基中,在38.5°C下在5%CO2 培养箱中培养吸出的卵丘 - 卵母细胞复合物(COCs)21小时。
- 执行以下步骤以去除卵母细胞。
- 使用200μL移液管收集所需数量的COC,并将其沉积在1.5μL离心管的底部。
- 向装有卵母细胞的离心管中加入相同体积的0.6mg / mL透明质酸酶(即,如果卵母细胞和IVM培养基的体积为100μL,则加入100μL透明质酸酶)。
- 上下移液溶液而不产生气泡,直到除去所有积云细胞。
- 检查卵母细胞的成熟度。
- 使用移液管将来自搜索板上四种HSOF滴剂之一的200μL HSOF加入到卵母细胞/透明质酸酶溶液中。
- 从透明质酸酶/ HSOF滴剂中转移卵母细胞,并将其置于未使用的400μL HSOF滴剂中。用另外两滴HSOF液滴剂洗涤它们,以帮助去除透明质酸酶和积云细胞残留物。
- 使用口移液器(或10μL移液器和吸头)和高显微放大倍率,根据极体的存在滚动和选择卵母细胞。
- 对卵母细胞进行分选后,收集所需数量的MII卵母细胞进行一分为二,并将它们置于400μLHSOF滴剂中。
注意:如果卵母细胞在培养箱外放置超过30分钟,则可以将卵母细胞放回成熟培养基孔中并置于CO2控制的培养箱中以静置至少30分钟。将要一分为二的卵母细胞数量限制在大约30分钟内完成一分为二的可行量,以尽量减少孵育以外的时间。如果卵母与体细胞融合,激活并培养为胚胎,则应在此时对卵母细胞进行去核。
3. 卵母细胞离心
注意:如果将卵母细胞放置在成熟培养基的培养箱中,请将其移至最近收集它们的HSOF液滴中。
- 使用口移液管,从HSOF液滴中收集选定的成熟卵母细胞,并将其放入含有50μL HSOF / CB溶液的1.5 mL管中。将卵母细胞以15,000× g 离心12分钟。
注意:离心管中存在气泡并不有害。 - 在离心卵母细胞时,准备二分切板。
- 在60毫米培养皿(每滴20μL)的盖子上制作 如图1 所示的图案,并用矿物油完全覆盖滴剂。
- 使用细尖标记,标记盘子下方的线条,如图 2所示。
- 在原酶液滴周围画一个框,并在CB / T20液滴和T20滴底行之间画一条线。
- 用不透明的盖子盖住培养皿,直到离心完成,以防止微滴的渗透压变化。
- 离心完成后,使用200μL移液管收集溶液中的卵母细胞,并将它们移动到带有四个400μL HSOF滴剂的新搜索板的空部分。
- 通过四个HSOF滴剂收集并洗涤卵母细胞。
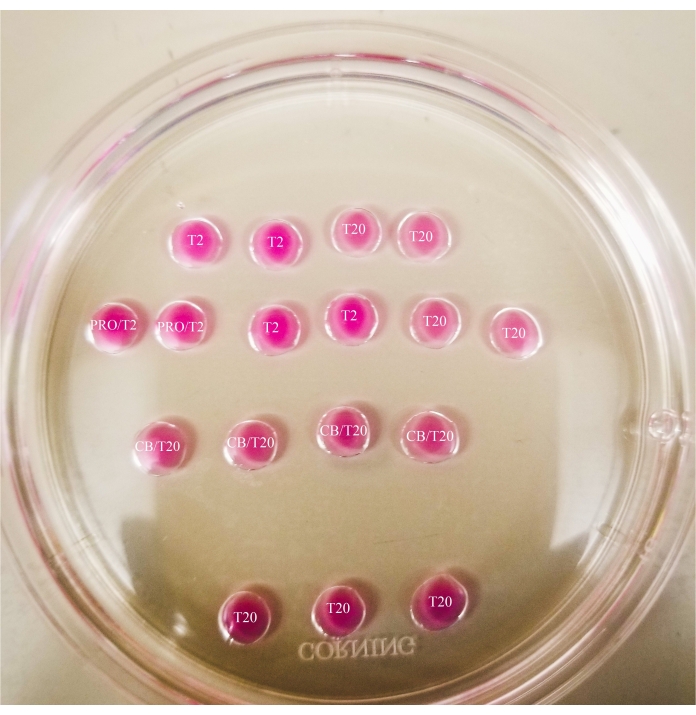
图1:二分板。 显示的所有滴剂的体积均为20 μL。该板的直径为60毫米。滴剂已被矿物油完全覆盖。卵母细胞将首先放置在最上部和最左侧的T2液滴中(此处用星号表示)。PRO/T2:10 μL 丙氨酸酶和 10 μL T2,在产生微滴之前合并。CB/T20:每 1 mL T20 含 1 μL 细胞查拉菌素 B,在产生微量滴剂之前合并。 请点击此处查看此图的大图。
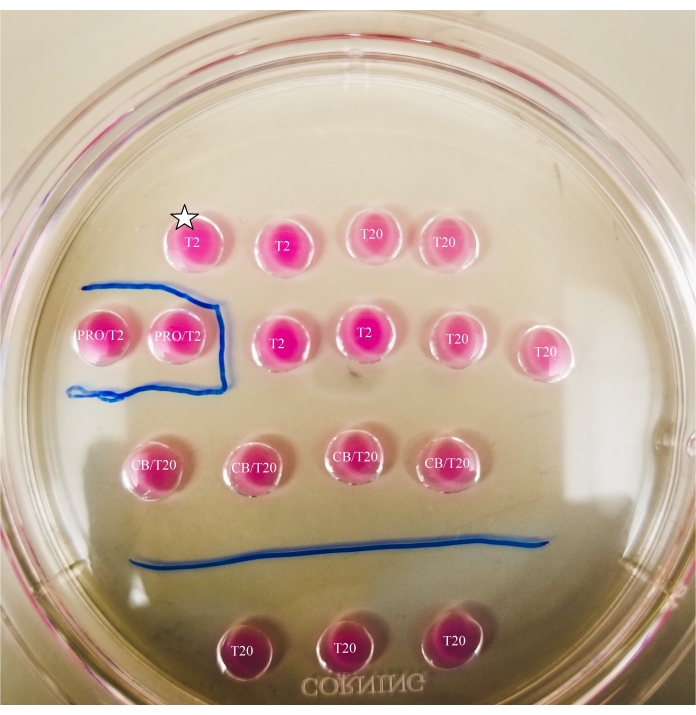
图2:标记的二分板。 在板的底部用一个细尖的标记画线,为观察和在显微镜下进行的卵母细胞和卵母细胞转移提供位置参考。卵母细胞将首先放置在最上部和最左侧的T2液滴中(此处用星号表示)。 请点击此处查看此图的大图。
4. 制备用于二分切片的卵母细胞
注意:以下过程涉及制备用于二分切片的卵母细胞。
- 将加热台打开至37°C。
- 使用口移液管将离心的卵母细胞从HSOF滴剂移动到平分板的左上角T2滴,然后通过顶排(T2,T20,T20)上的下三滴洗涤卵母细胞。
- 将卵母细胞沉积在其中一个原酶滴剂中,确保它们之间几乎没有接触。
注意:去除透明带可能需要不同的时间,但通常发生在30-120秒之间,使用加热阶段。没有加热阶段将延长此时间。 - 观察卵母细胞,直到透明带有明显的变形(见 图3)。
- 一旦单个卵母细胞透明带变形,将该卵母细胞移动到相邻的T2滴。当其他卵母细胞变形时重复上述步骤,直到所有卵母细胞均已从原酶中取出并置于T2滴剂中。
- 观察卵母细胞,直到只剩下一层薄薄的卵泡区域。
- 通过行内接下来的三滴(T2,T20,T20)洗涤卵母细胞,然后将其沉积在CB / T20滴剂内的垂直线上(见 图4)。
- 从垂直线中较少的卵母细胞开始,并随着平分技能的进展增加每滴卵母细胞的数量。
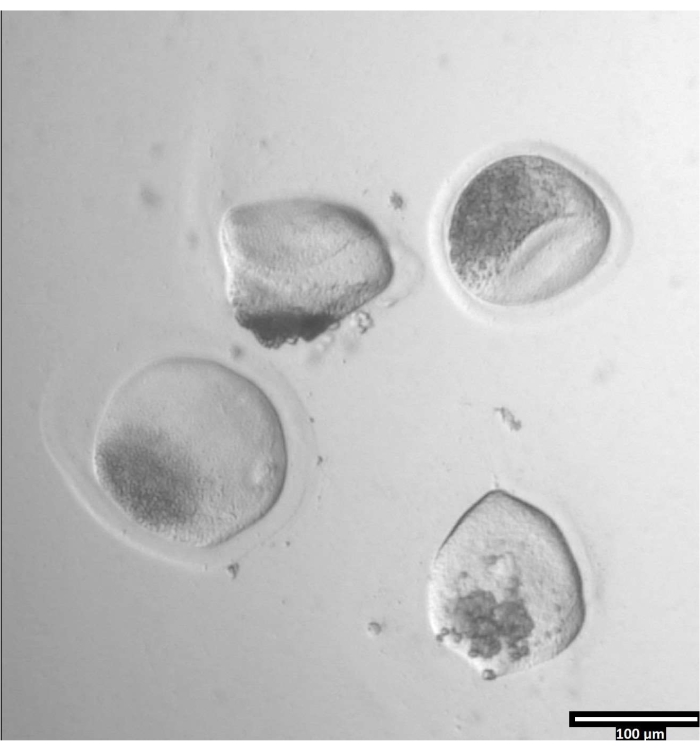
(80x)当卵泡酶对透明带的影响足以使卵母细胞移动到相邻的T2液滴时,卵母细胞透明带将开始变形。请点击此处查看此图的大图。

(80x)无透明带卵母细胞在对等分滴之前以近乎垂直的方向沉积在每个CB / T20滴落中。请点击此处查看此图的大图。
5. 卵母细胞一分为二
- 将焦点放在第一个包含卵母细胞的最左侧的CB / T20(二分)滴剂上,并使用嘴移液器旋转卵母细胞,使每个卵母细胞的线粒体致密部分朝向或远离显微镜的手臂。
注意:线粒体密集的部分将不含脂质,脂质在离心后显示为卵母细胞最暗的部分。 - 将微叶片的尖端放在最上面的卵母细胞的左侧,与线粒体致密部分的正上方的空间一致。将刀片的尖端保持在同一位置,小心地放下刀片以一直穿过卵母细胞。
- 确保线粒体密集的ooplast和线粒体减少的ooplast的大小相似;二分切应将每个卵母细胞切成两半,以产生两个卵母细胞。
注意:单个卵母细胞产生的卵母细胞的比较大小可能因研究目标而异;如果要将线粒体减少的ooplast与体细胞融合,则其体积应大于线粒体致密的ooplasts。 - 确保刀片将卵母细胞一分为二,高于线粒体致密部分和脂质致密部分,在离心卵母细胞最清晰的部分。
- 确保线粒体密集的ooplast和线粒体减少的ooplast的大小相似;二分切应将每个卵母细胞切成两半,以产生两个卵母细胞。
- 抬起刀片时,请确保保持相同的线,并且刀片的尖端保持在同一位置,然后轻轻地将尖端从板上抬起。
- 对第一滴二分滴中的所有卵母细胞重复二分法步骤。如果卵母细胞存在于额外的滴剂中,则将剩余的卵母细胞定向并一分为二。
- 使用口移液器,从第一个二分滴中收集线粒体减少的ooplasts。将它们放在位于板底部一排的左侧T20液滴中。对所有剩余的二分滴液重复上述步骤。
注意:线粒体减少的卵巢将含有脂质,其颜色比卵母细胞的其余部分更深。 - 使用口移液器,从第一个二分滴中收集线粒体密集的ooplasts。将它们放在位于板底部一排的右侧T20液滴中。对所有剩余的二分滴液重复上述步骤。
注意:线粒体密集区域将具有可见的细胞器聚集,其外观将为浅灰色。 - 从培养箱中取出T10四孔培养皿。使用口移液器,将所有线粒体减少的ooplast移动到标记为"MR"的孔中,并将线粒体致密的ooplast移动到标记为"M"的孔中。
- 将四孔板放回CO2控制的培养箱中。在对mtDNA进行定量之前,让卵子休息至少30分钟。
6. 甲基靹病毒的定量
- 使用设计用于从小样品中提取材料的DNA提取试剂盒,从单个卵母细胞、单个线粒体致密的ooplasts、单个线粒体耗尽的ooplasts)中提取DNA。
- 使用所选的DNA定量方法,以确保从每个样品中成功提取DNA。如果当时无法完成定量聚合酶链反应(qPCR),请在-80°C冰箱中将提取的DNA冷冻在标记的管中。
- 执行 qPCR
- 请参阅 表2 以确定要添加到每个qPCR管中的每种试剂的体积以及引物序列。这些引物旨在扩增牛mtDNA的12S区域。
- 将初始变性时间设置为10分钟,然后进行35个循环:在94°C下变性30秒,在60°C下退火15秒,在72°C下延伸15秒。 反应完成后,记录循环阈值(Ct)值。
注意:为了获得相对的mtDNA拷贝数值,必须使用具有已知mtDNA拷贝数的样品生成标准曲线,这些拷贝数呈指数级增长。然后需要使用以下公式操作周期阈值,以确定相对mtDNA拷贝数。 - 使用以下等式获得DNA的浓度:
DNA 浓度 = (10(克拉截距/斜率)) x 测试体积 - 使用以下等式获取拷贝数:
拷贝数 =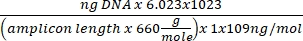
- 使用以下等式获取拷贝数:
每个单元格的拷贝数 = 2 x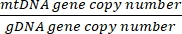
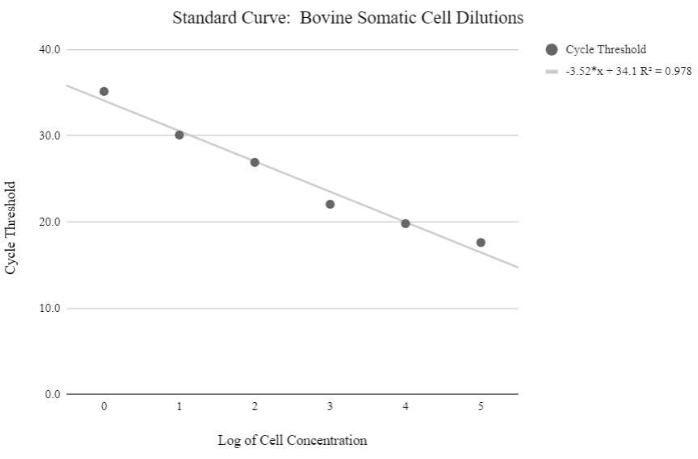
图5:体细胞mtDNA标准曲线。 该标准曲线是通过使用qPCR试剂和程序对牛体细胞的对数浓度进行mtDNA定量而创建的,如方案步骤6.3中所述。 请点击此处查看此图的大图。
Access restricted. Please log in or start a trial to view this content.
结果
定量 PCR (qPCR) 结果用于确定每个卵巢中存在的 mtDNA 的相对数量。所描述的反应旨在扩增牛mtDNA的12S区域。
如果二分切成功,来自整个卵母细胞和线粒体致密卵巢的样品将具有相似的Ct 值。与其他两组的样品相比,来自线粒体减少的ooplast的样品将具有更高的Ct 值。下图显示了显示成功对等的结果的Ct 图。这些结果表明,二分切确实有效地降低了线粒...
Access restricted. Please log in or start a trial to view this content.
讨论
以前用于减少卵母细胞中mtDNA拷贝数的方法有其各自的缺点。基于显微操作的卵母细胞线粒体去除使mtDNA拷贝数平均降低64%27。以前用于去核的独特方法涉及使用小直径巴斯德移液器和在微滴介质和周围矿物油之间的边界处分裂无透明带卵母细胞。随着卵母细胞离心的利用,这种方法可以产生可行的线粒体减少的ooplasts28。然而,离心和这种卵母细胞分裂方法的组...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有什么可透露的。
致谢
作者希望感谢犹他州立大学的同事,圣地亚哥动物园的生殖科学研究人员以及Genes PLC的Rebecca Krisher博士。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| 1.5 mL centrifuge tubes | Fisher Scientific | 5408129 | |
| 60 mm dish | Sigma-Aldrich | D8054 | |
| Centrifuge | Eppendorf | 5424 | |
| Cytochalasin B | Sigma-Aldrich | C6762 | |
| Fetal Bovine Serum | Sigma-Aldrich | F2442 | |
| M199 Media | Sigma-Aldrich | M4530 | |
| Mineral Oil | Sigma-Aldrich | M8410 | |
| Mini Centrifuge | SCILOGEX | D1008 | |
| mtDNA Primer: Forward (12S) | GGGCTACATTCTCTACACCAAG | ||
| mtDNA Primer: Reverse (12S) | GTGCTTCATGGCCTAATTCAAC | ||
| NanoDrop Spectrophotometer | Thermo Scientific | ND2000 | |
| Opthalmic Scalpel with Aluminum Handle | PFM Medical | 207300633 | Microblade for bisection |
| Protease/pronase | Sigma-Aldrich | P5147 | |
| QIAamp DNA Micro Kit | Qiagen | 56304 | |
| QuantStudio™ 3 - 96-Well 0.2-mL | ThermoFisher | A28567 | |
| Search plate | Fisher Scientific | FB0875711A | |
| SYBR Green qPCR Master Mix | ThermoFisher | K0221 | qPCR master mix |
| Synthetic Oviductal Fluid with HEPES (HSOF) | |||
| ThermoPlate | Tokai Hit | TPi-SMZSSX | Heating stage |
参考文献
- Loi, P., Modlinski, J. A., Ptak, G. Interspecies somatic cell nuclear transfer: A salvage tool seeking first aid. Theriogenology. 76 (2), 217-228 (2011).
- Wani, N. A., Vettical, B. S., Hong, S. B. First cloned Bactrian camel (camelus bactrianus) calf produced by interspecies somatic cell nuclear transfer: A step towards preserving the critically endangered wild Bactrian camels. PLOS ONE. 12 (5), 0177800(2017).
- Oh, H. J., et al. Cloning endangered gray wolves (canis lupus) from somatic cells collected postmortem. Theriogenology. 70 (4), 638-647 (2008).
- Srirattana, K., et al. Full-term development of gaur-bovine interspecies somatic cell nuclear transfer embryos: Effect of trichostatin a treatment. Cellular Reprogramming. 14 (3), 248-257 (2012).
- Kwon, D. K., et al. Blastocysts derived from adult fibroblasts of a rhesus monkey (macaca mulatta) using interspecies somatic cell nuclear transfer. Zygote. 19 (3), 199-204 (2011).
- Lee, E., et al. Production of cloned sei whale (Balaenoptera borealis) embryos by interspecies somatic cell nuclear transfer using enucleated pig oocytes. Journal of Veterinary Science. 10 (4), 285(2009).
- Lorthongpanich, C., Laowtammathron, C., Chan, A. W., Kedutat-Cairns, M., Parnpai, R. Development of interspecies cloned monkey embryos reconstructed with bovine enucleated oocytes. Journal of Reproduction and Development. 54 (5), 306-313 (2008).
- Hong, S. G., et al. Production of transgenic canine embryos using interspecies somatic cell nuclear transfer. Zygote. 20 (1), 67-72 (2011).
- Stewart, J. B., Chinnery, P. F. The dynamics of mitochondrial DNA heteroplasmy: Implications for human health and disease. Nature Reviews Genetics. 16 (9), 530-542 (2015).
- Takeda, K. Mitochondrial DNA transmission and confounding mitochondrial influences in cloned cattle and pigs. Reproductive Medicine and Biology. 12 (2), 47-55 (2013).
- Lanza, R. P., et al. Cloning of an endangered species (Bos Gaurus) using interspecies nuclear transfer. Cloning. 2 (2), 79-90 (2000).
- Evans, M. J., et al. Mitochondrial DNA genotypes in nuclear transfer-derived cloned sheep. Nature Genetics. 23 (1), 90-93 (1999).
- Meirelles, F. V., et al. Complete replacement of the mitochondrial genotype in a Bos indicus calf reconstructed by nuclear transfer to a Bos taurus oocyte. Genetics. 158 (1), 351-356 (2001).
- Beyhan, Z., Iager, A. E., Cibelli, J. B. Interspecies nuclear transfer: Implications for embryonic stem cell biology. Cell Stem Cell. 1 (5), 502-512 (2007).
- Lagutina, I., Fulka, H., Lazzari, G., Galli, C. Interspecies somatic cell nuclear transfer: advancements and problems. Cellular Reprogramming. 15 (5), 374-384 (2013).
- Jiang, Y., et al. Interspecies somatic cell nuclear transfer is dependent on compatible mitochondrial DNA and reprogramming factors. PLoS ONE. 6 (4), 14805(2011).
- Chiaratti, M. R., et al. Embryo mitochondrial DNA depletion is reversed during early embryogenesis in cattle. Biology of Reproduction. 82 (1), 76-85 (2010).
- Spikings, E. C., Alderson, J., John, J. C. Regulated mitochondrial DNA replication during oocyte maturation is essential for successful porcine embryonic development. Biology of Reproduction. 76 (2), 327-335 (2007).
- Cagnone, G. L., et al. Restoration of normal embryogenesis by mitochondrial supplementation in pig oocytes exhibiting mitochondrial DNA deficiency. Scientific Reports. 6 (1), 1-15 (2016).
- Spikings, E. C., Alderson, J., John, J. C. Regulated mitochondrial DNA replication during oocyte maturation is essential for successful porcine embryonic development. Biology of Reproduction. 76 (2), 327-335 (2007).
- Ferreira, A. F., et al. Does supplementation with mitochondria improve oocyte competence? A systematic review. Reproduction. 161 (3), 269-287 (2021).
- Bhat, M. H., et al. Live birth of a pashmina goat kid after transfer of handmade cloned embryos. Journal of Reproduction and Development. , (2019).
- Tecirlioglu, R. T., et al. Birth of a cloned calf derived from a vitrified hand-made cloned embryo. Reproduction, Fertility and Development. 15 (7), 361(2003).
- Zhang, P., et al. Handmade cloned transgenic piglets expressing the nematode fat-1 gene. Cellular Reprogramming. 14 (3), 258-266 (2012).
- Zhang, P., et al. Handmade cloned transgenic sheep rich in omega-3 fatty acids. PLOS ONE. 8 (2), 55941(2013).
- Lagutina, I., et al. Somatic cell nuclear transfer in horses: Effect of oocyte morphology, embryo reconstruction method and donor cell type. Reproduction. 130 (4), 559-567 (2005).
- Chiaratti, M. R., et al. Embryo mitochondrial DNA depletion is reversed during early embryogenesis in cattle. Biology of Reproduction. 82 (1), 76-85 (2010).
- Hosseini, S. M., et al. and efficient method of manual oocyte enucleation using a pulled pasteur pipette. In Vitro Cellular and Developmental Biology - Animal. 49 (8), 569-575 (2013).
- Zampolla, T., Spikings, E., Rawson, D., Zhang, T. Cytoskeleton proteins F-actin and tubulin distribution and interaction with mitochondria in the granulosa cells surrounding stage III zebrafish (danio rerio) oocytes. Theriogenology. 76 (6), 1110-1119 (2011).
- International Union for Conservation of Nature. The IUCN Red List of Threatened Species. International Union for Conservation of Nature. , (2021).
- Berg, D. K., Li, C., Asher, G., Wells, D. N., Oback, B. Red deer cloned from antler stem cells and their differentiated progeny. Biology of Reproduction. 77 (3), 384-394 (2007).
- Gómez, M. C., et al. Birth of African wildcat cloned kittens born from domestic cats. Cloning and Stem Cells. 6 (3), 247-258 (2004).
- Lanza, R. P., et al. Cloning of an endangered species (Bos gaurus) using interspecies nuclear transfer. Cloning. 2 (2), 79-90 (2000).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。