Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Verwendung von Bisektion zur Reduzierung der mitochondrialen DNA in der Rinderzelle
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll vor, um die mitochondrialen DNA-Kopienzahlen in einer Rinderzelle signifikant zu reduzieren (P < 0,0001). Diese Methode nutzt Zentrifugation und Bisektion, um die Mitochondrien der Eizellen erheblich zu reduzieren und kann eine erhöhte Entwicklungschance in den rekonstruierten interspeziesen somatischen Zellkerntransferembryonen ermöglichen.
Zusammenfassung
Interspezies-somatischer Zellkerntransfer (iSCNT) kann verwendet werden, um gefährdete Arten zu retten, aber zwei verschiedene Populationen mitochondrialer DNA (mtDNA) existieren innerhalb des rekonstruierten Embryos: eine innerhalb des Empfängerooplasmas und eine innerhalb der somatischen Spenderzelle. Diese mitochondriale Heteroplasmie kann zu Entwicklungsproblemen im Embryo und im Fötus führen. Handgefertigte Klonprotokolle umfassen die Eizellbisektion, die verwendet werden kann, um die mtDNA-Kopienzahl zu verringern und den Grad der mitochondrialen Heteroplasmie in einem rekonstruierten Embryo zu reduzieren. Die Zentrifugation von entblößten, reifen Rindereizellen erzeugte eine sichtbare mitochondriendichte Fraktion an einem Pol der Eizelle. Die Zonae pellucidae der Eizellen wurden durch Exposition gegenüber einer Pronasenlösung entfernt. Die Bisektion wurde mit einer Mikroklinge durchgeführt, um die sichtbare Mitochondrienfraktion zu entfernen. qPCR wurde verwendet, um die mtDNA zu quantifizieren, die in DNA-Proben aus ganzen Eizellen und halbierten Ooplasten vorhanden ist, und lieferte einen Vergleich der mtDNA-Kopienzahlen vor und nach der Bisektion. Die Kopienzahlen wurden unter Verwendung von Zyklusschwellenwerten, der Regressionslinienformel einer Standardkurve und einem Verhältnis berechnet, das die jeweiligen Größen von mtDNA-PCR-Produkten und genomischen PCR-Produkten umfasste. Eine Rinderzelle hatte eine durchschnittliche mtDNA-Kopienzahl (± Standardabweichung) von 137.904 ± 94.768 (n = 38). Ein Mitochondrien-erschöpfter Ooplast hatte eine durchschnittliche mtDNA-Kopienzahl von 8.442 ± 13.806 (n = 33). Die durchschnittlichen mtDNA-Kopien in einem mitochondrienreichen Ooplast betrugen 79.390 ± 58.526 mtDNA-Kopien (n = 28). Die Unterschiede zwischen diesen berechneten Mittelwerten deuten darauf hin, dass die Zentrifugation und die anschließende Bisektion die in der Mitochondrien-erschöpften Ooplast vorhandenen mtDNA-Kopienzahlen im Vergleich zur ursprünglichen Eizelle signifikant verringern können (P < 0,0001, bestimmt durch Einweg-ANOVA). Die Reduktion der mtDNA sollte den Grad der mitochondrialen Heteroplasmie in einem rekonstruierten Embryo verringern und möglicherweise die Standardentwicklung von Embryonen und Föten fördern. Die Ergänzung mit mitochondrialem Extrakt aus der somatischen Spenderzelle kann ebenfalls für eine erfolgreiche Embryonalentwicklung unerlässlich sein.
Einleitung
Der somatische Zellkerntransfer (SCNT) umfasst die Fusion einer enukleierten Eizelle aus einem Tier und einer somatischen Zelle aus einem Tier derselben Art. In den meisten Fällen stammen die Eizelle und die somatische Zelle von derselben Art, und die Lebendgeburtenraten liegen unter 6%1. Einige Forschungen beinhalten die Verwendung von Interspezies-SCNT (iSCNT), die die Fusion einer somatischen Zelle und einer Eizelle beinhaltet, die von zwei verschiedenen Arten stammen. In diesen Studien sind die Lebendgeburtenraten sogar niedriger als in SCNT - typischerweise weniger als 1%1. iSCNT hat jedoch die Fähigkeit, als Methode zur Rettung gefährdeter Arten verwendet zu werden, da somatische Zellen dieser Tiere zugänglicher sind als ihre Keimzellen1. Empfängeroozyten, die in iSCNT verwendet werden, sind oft heimische oder häufige Laborarten wie Kühe, Schweine und Mäuse. Einige bisher unternommene Versuche haben erfolgreich lebende Junge hervorgebracht, obwohl die produzierten Nachkommen intragenerische Tiere waren (die Empfänger-Eizellenarten und die Spenderzellarten waren Mitglieder derselben Gattung)2,3,4. Intergenerische Modelle (die eine Eizelle und eine somatische Zelle von Tieren verschiedener Gattungen verwenden) haben noch keine lebenden Tiere hervorgebracht, und die Mehrheit der rekonstruierten Embryonen stoppt im 8-16-Zellstadium der In-vitro-Entwicklung 5,6,7,8. Eine mögliche Erklärung für diesen embryonalen Entwicklungsstillstand ist das Auftreten von mitochondrialer Heteroplasmie in den Embryonen - das Vorhandensein von mehr als einem Mitochondrien-DNA-Typ (mtDNA) in einer einzelnen Zelle. Heteroplasmie kann zu Problemen wie Entwicklungsineffizienz oder Versagen im Embryo oder im lebenden Tierführen 1. Die Pathogenese kann auch später im Leben des Tieres auftreten9. Obwohl dieses Problem auch bei SCNT-Nachkommen auftritt, verschärft die interspezifische Komponente in iSCNT-Embryonen das Problem.
Wenn die embryonale mtDNA von zwei verschiedenen Spezies stammt, arbeiten die Mitochondrien der Empfängerzelle, die die Mehrheit darstellen, nicht effizient oder effektiv mit dem Kern der Spenderzelle 1,10. Größere taxonomische Lücken zwischen den beiden in iSCNT verwendeten Arten verstärken dieses Problem wahrscheinlich; Intragenerische lebende Nachkommen, die produziert wurden (Bos gaurus und Bos indicus Nachkommen mit Bos taurus Eizellen), sowie Nachkommen, die über traditionelle SCNT produziert wurden (z. B. Ovis aries Nachkommen mit Ovis aries Eizellen) wurden als Chimären gezeigt (mtDNA von zwei Individuen war in diesen Tierenvorhanden 11,12,13). Sie entwickelten sich jedoch viel weiter als die intergenerischen SCNT-Embryonen14,15. Der Informationsaustausch zwischen den Mitochondrien der Eizellen und dem Zellkern der Spenderzelle könnte im intragenerischen Embryo erfolgreicher sein als im intergenerischenEmbryo 16.
Die Menge an mtDNA in einer reifen Rinderzelle ist etwa 100-mal größer als die Menge, die in einer somatischen Zellegefunden wird 12. Eine Verringerung dieses Verhältnisses könnte die Mitochondrien der somatischen Zellen dazu ermutigen, sich innerhalb des rekonstruierten Embryos zu vermehren, so dass eine größere Population produktiver Mitochondrien vorhanden seinkann 16. Dies könnte wiederum mehr Energie liefern, um den Bedarf des sich entwickelnden Embryoszu decken 15. Frühere Versuche, die mtDNA-Kopienzahl der Eizelle oder des Embryos zu reduzieren, umfassen die chemische Anwendung, Mikromanipulation und die Ergänzung der Eizelle oder des Embryos mit zusätzlichen Mitochondrien aus den Spenderzellspezies 16,17,18,19,20. Die chemische Anwendung (wie 2',3'-Dideoxycytidin) ist jedoch nicht ideal für die Embryonalentwicklung und hat die MTDNA-Kopienzahlen der Eizellen um etwadie Hälfte von 18 reduziert. Frühere mtDNA-Reduktionen der Eizellen durch Mikromanipulation haben nur durchschnittlich 64% der mtDNA17 der Eizelle entfernt. Obwohl die Supplementierung von Spenderzellmitochondrien eine praktikable Option sein könnte, hat ihre Verwendung in den iSCNT-Studien noch kein lebendes intergenerisches Tier hervorgebracht21.
Die Verwendung von Bisektion zur Reduzierung der mtDNA-Kopienzahl der Eizellen wurde in veröffentlichten Studien noch nicht verwendet. Das Bisektieren von Eizellen mit der Absicht, die Ooplasten mit einer somatischen Zelle zu verschmelzen, ist die Prämisse des handgemachten Klonens (HMC), bei dem typischerweise Bisektion als Methode zur Entfernung des Polkörpers und der Metaphasenplatte aus der Metaphase II (MII) -Eizelle verwendet wird. HMC hat erfolgreich Nachkommen in mehreren Arten produziert, darunter Ziegen, Rinder, Schweine, Schafe und Pferde 22,23,24,25,26, beinhaltet aber normalerweise keinen Zentrifugationsschritt vor der Bisektion. Die Integration der Hochgeschwindigkeitszentrifugation der Eizelle ermöglicht die Isolierung von Mitochondrien (und damit mtDNA) an einem Pol der Eizelle, die dann mit einer Mikroklinge halbiert werden kann, um diese mitochondriendichten Fraktionen zu entfernen. Zwei Mitochondrien-erschöpfte Ooplasten können dann, wie es bei HMC der Fall ist, mit einer somatischen Zelle zu einem rekonstruierten Embryo verschmolzen werden, der wesentlich weniger mtDNA von den Eizellenarten enthält.
Die Frage, die wir mit diesem Protokoll zu beantworten versuchen, ist, wie mtDNA in der Rinderzelle reduziert werden kann, um einen lebensfähigen rekonstruierten Embryo zu erzeugen, der weniger heteroplasmatische mtDNA enthält. In diesem Protokoll wurden Eizellen zentrifugiert und halbiert. Ooplast und intakte Eizelle mtDNA-Kopienzahlen wurden berechnet, um die Wirksamkeit dieser Technik bei der Reduzierung der mtDNA-Kopienzahl der Rinderzelle zu bestimmen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Das folgende Protokoll folgt den Tierpflege- und Ethikrichtlinien der Utah State University.
1. Medienaufbereitung
- Bereiten Sie vor der Behandlung mit Eizellen die folgenden Lösungen vor, wie in Tabelle 1 beschrieben: 400 μL Hyaluronidase-Lösung, 500 μL T2-Medien, 1.020 μL T20-Medien und 800 μL T10-Medien.
- Teilen Sie das T10-Medium in zwei Vertiefungen einer Vier-Well-Platte auf, 400 μL pro Well. Beschriften Sie einen Brunnen mit "M" und den zweiten Brunnen mit "MR". Bis nach der Bisektion in einen 5% CO2 -Inkubator geben.
- Bereiten Sie 500 μL Cytochalasin B (CB) / synthetische oviduktale Flüssigkeit mit HEPES (HSOF) vor. Aliquot 50 μL CB/HSOF-Lösung in ein separates 1,5-ml-Zentrifugenröhrchen.
- Bereiten Sie 40 μL Pronasenlösung vor. Zentrifuge für mindestens 30 s bei 2.680 x g. Mischen Sie 20 μL des Überstands mit 20 μL T2 zu einem neuen Zentrifugenröhrchen; Dies bildet eine verdünnte 5 mg/ml Pronasenlösung.
- Bereiten Sie 3 ml HSOF vor. Auf einem Suchschild vier 400-μL-Tropfen separat ablegen; Legen Sie diese Platte unter ein Stereomikroskop.
- Bereiten Sie 500 μL CB/T20-Lösung vor.
2. In-vitro-Reifung (IVM) von Rindereizellen
- IVM: Kulturaspirierte Kumulus-Eizell-Komplexe (COCs) bei 38,5 °C in einem 5% CO 2-Inkubator für 21 h in einer Vier-Well-Schale von Reifungsmedien mit 10% FBS, 0,26 IE/ml FSH und 100 U/ml Penicillin/Streptomycin.
- Führen Sie die folgenden Schritte aus, um die Eizellen zu entleeren.
- Sammeln Sie die gewünschte Anzahl der COCs mit einer 200-μL-Pipette und legen Sie sie am Boden eines 1,5-μL-Zentrifugenröhrchens ab.
- Das gleiche Volumen von 0,6 mg/ml Hyaluronidase in das Zentrifugenröhrchen mit den Eizellen geben (d. h. wenn das Volumen der Eizellen und IVM-Medien 100 μL beträgt, fügen Sie 100 μL Hyaluronidase hinzu).
- Pippieren Sie die Lösung auf und ab, ohne Blasen zu erzeugen, bis alle Kumuluszellen entfernt wurden.
- Überprüfen Sie die Reifung der Eizellen.
- Verwenden Sie die Pipette, um 200 μL HSOF aus einem der vier HSOF-Tropfen auf dem Suchschild in die Eizell-/Hyaluronidaselösung zu geben.
- Übertragen Sie die Eizellen aus dem Hyaluronidase/HSOF-Tropfen und legen Sie sie in unbenutzte 400 μL HSOF-Tropfen. Waschen Sie sie mit zwei zusätzlichen HSOF-Tropfen, um die Entfernung von Hyaluronidase und Cumuluszellresten zu unterstützen.
- Mit einer Mundpipette (oder einer 10 μL Pipette und Spitze) und hoher mikroskopischer Vergrößerung rollen und selektieren Sie die Eizellen basierend auf der Anwesenheit des Polkörpers.
- Nachdem Sie die Eizellen sortiert haben, sammeln Sie die gewünschte Anzahl von MII-Eizellen, die halbiert werden sollen, und legen Sie sie in 400 μL HSOF-Tropfen.
HINWEIS: Wenn sich die Eizellen länger als 30 min außerhalb des Inkubators befunden haben, können die Eizellen wieder in einen Brunnen aus Reifungsmedien gegeben und in einen CO 2-kontrollierten Inkubator gelegt werden, um mindestens 30 min zu ruhen. Begrenzen Sie die Anzahl der zu halbierenden Eizellen auf eine Menge, die in etwa 30 Minuten durchführbar ist, um die Zeit außerhalb der Inkubation zu minimieren. Wenn Ooplasten mit einer somatischen Zelle verschmolzen, aktiviert und als Embryonen kultiviert werden sollen, sollten die Eizellen zu diesem Zeitpunkt enukleiert werden.
3. Zentrifugation der Eizellen
HINWEIS: Wenn Eizellen in Reifemedien in den Inkubator gegeben wurden, bringen Sie sie in den HSOF-Tropfen, aus dem sie zuletzt entnommen wurden.
- Sammeln Sie mit einer Mundpipette die ausgewählten reifen Eizellen aus dem HSOF-Tropfen und geben Sie sie in das 1,5-ml-Röhrchen, das 50 μL der HSOF/CB-Lösung enthält. Die Eizellen bei 15.000 x g für 12 min zentrifugieren.
HINWEIS: Es ist nicht schädlich, wenn Blasen im Zentrifugenröhrchen vorhanden sind. - Während die Eizellen zentrifugiert werden, bereiten Sie die Bisektionsplatte vor.
- Machen Sie das Muster wie in Abbildung 1 auf dem Deckel einer 60-mm-Petrischale (20 μL pro Tropfen) und bedecken Sie die Tropfen vollständig mit Mineralöl.
- Markieren Sie mit einem Marker mit dünner Spitze die Linien unter der Schale, wie in Abbildung 2 dargestellt.
- Zeichne eine Box um den Pronase-Drop und eine Linie zwischen den CB/T20-Drops und der unteren Reihe der T20-Drops.
- Decken Sie die Schale mit einem undurchsichtigen Deckel ab, bis die Zentrifugation abgeschlossen ist, um Osmolaritätsänderungen von Mikrotropfen zu verhindern.
- Sobald die Zentrifugation abgeschlossen ist, verwenden Sie eine 200-μL-Pipette, um die Eizellen in der Lösung zu sammeln, und bewegen Sie sie in einen leeren Teil eines neuen Suchfeldes mit vier 400 μL HSOF-Tropfen.
- Sammeln und waschen Sie die Eizellen durch die vier HSOF-Tropfen.
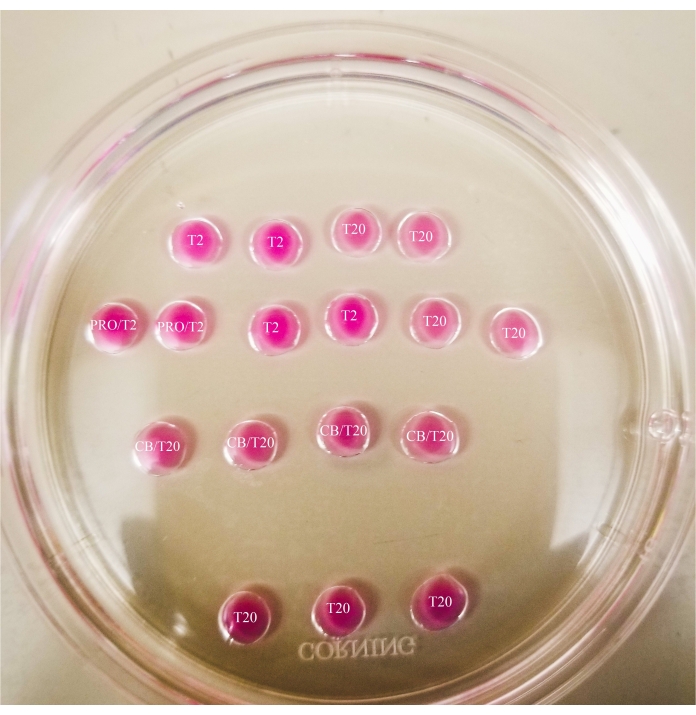
Abbildung 1: Bisektionsplatte Alle gezeigten Tropfen haben ein Volumen von 20 μL. Die Platte hat einen Durchmesser von 60 mm. Die Tropfen wurden vollständig mit Mineralöl bedeckt. Die Eizellen werden zuerst in den obersten und linken T2-Tropfen gelegt (hier mit einem Stern gekennzeichnet). PRO/T2: 10 μL Pronase und 10 μL T2, kombiniert vor der Bildung von Mikrotropfen. CB/T20: 1 μL Cytochalasin B pro 1 ml T20, kombiniert vor der Bildung von Mikrotropfen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
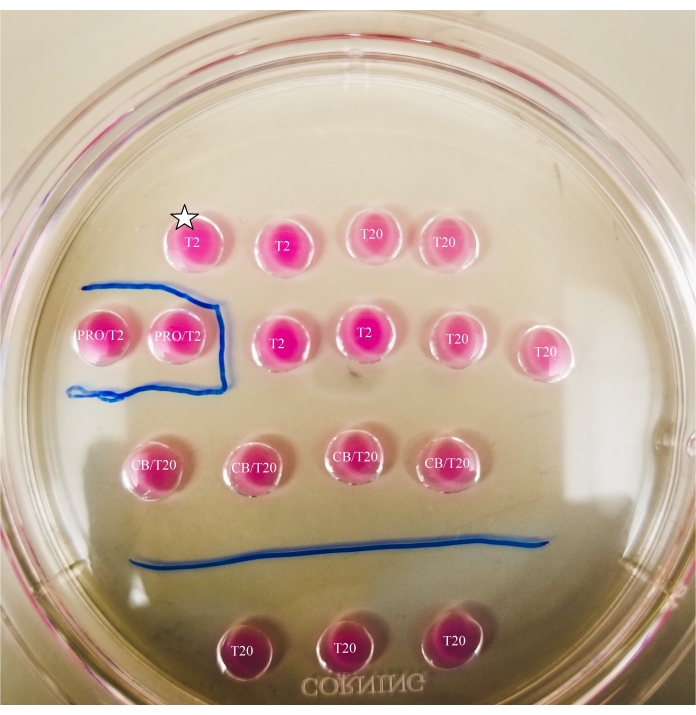
Abbildung 2: Markiertes Zweiteilungsschild. Die Linien werden mit einem dünnspitzen Marker auf der Unterseite der Platte gezeichnet, um Standortreferenzen für Beobachtungen und Eizell- und Ooplastentransfers unter dem Mikroskop bereitzustellen. Die Eizellen werden zuerst in den obersten und linken T2-Tropfen gelegt (hier mit einem Stern gekennzeichnet). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
4. Vorbereitung der Eizelle für die Bisektion
HINWEIS: Der folgende Prozess beinhaltet die Vorbereitung der Eizellen für die Bisektion.
- Schalten Sie die Heizstufe auf 37 °C.
- Verwenden Sie eine Mundpipette, um die zentrifugierten Eizellen vom HSOF-Tropfen zum oberen linken T2-Tropfen der Bisektionsplatte zu bewegen, und waschen Sie die Eizellen dann durch die nächsten drei Tropfen in der oberen Reihe (T2, T20, T20).
- Deponieren Sie die Eizellen in einem der Nasentropfen und stellen Sie sicher, dass es wenig bis gar keinen Kontakt zwischen ihnen gibt.
HINWEIS: Die Entfernung der Zona pellucida kann unterschiedlich lange dauern, erfolgt jedoch typischerweise zwischen 30-120 s unter Verwendung einer Heizstufe. Das Fehlen einer Heizstufe wird sich diesmal verlängern. - Beobachten Sie die Eizellen, bis es zu einer deutlichen Verformung der Zonae pellucidae kommt (siehe Abbildung 3).
- Sobald eine einzelne Eizelle zona pellucida deformiert ist, bewegen Sie diese Eizelle in den benachbarten T2-Tropfen. Wiederholen Sie den Vorgang, wenn sich zusätzliche Eizellen verformen, bis alle Eizellen aus der Pronase entfernt und in den T2-Tropfen gelegt wurden.
- Beobachten Sie die Eizellen, bis nur noch eine dünne Schicht der Zone pellucida übrig bleibt.
- Waschen Sie die Eizellen durch die nächsten drei Tropfen innerhalb der Reihe (T2, T20, T20) und legen Sie sie dann in vertikalen Linien innerhalb der CB/T20-Tropfen ab (siehe Abbildung 4).
- Beginnen Sie mit weniger Eizellen in den vertikalen Linien und erhöhen Sie die Anzahl der Eizellen pro Tropfen, wenn die Bisektionsfähigkeiten fortschreiten.
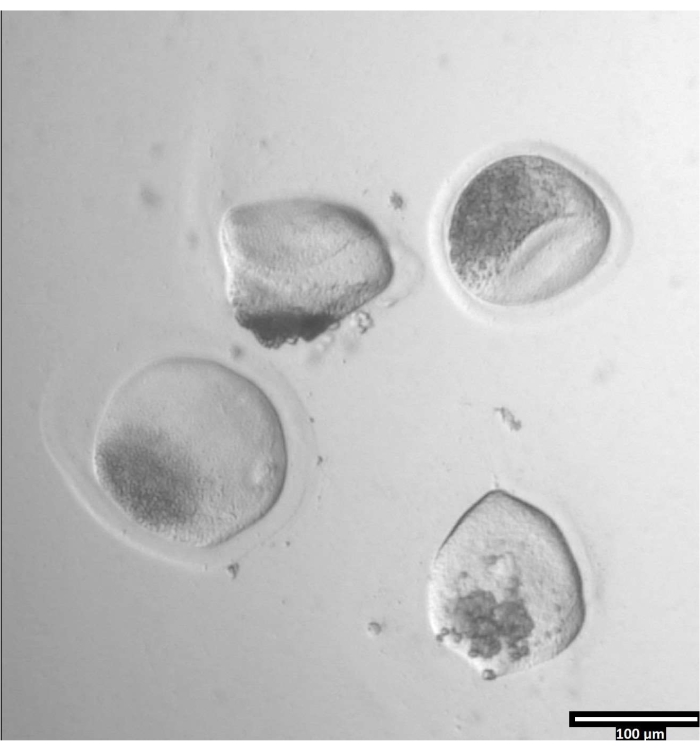
Abbildung 3: Entfernung der Zona pellucida mittels Pronase. (80x) Eine Eizelle Zona pellucida beginnt deformiert zu erscheinen, wenn die Pronase die Zona pellucida so stark beeinflusst hat, dass die Eizelle in den benachbarten T2-Tropfen bewegt werden kann. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 4: Oozytenorientierung in Bisektionstropfen . (80x) Zonafreie Eizellen werden in einer nahezu vertikalen Ausrichtung innerhalb jedes CB/T20-Tropfens vor der Bisektion abgeschieden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
5. Bisektion der Eizellen
- Konzentrieren Sie sich auf den ersten, ganz links stehenden CB / T20 (Bisektions-) Tropfen, der Eizellen enthält, und verwenden Sie eine Mundpipette, um die Eizellen zu drehen, so dass der mitochondriendichte Teil jeder Eizelle entweder zum Arm des Mikroskops zeigt oder vom Arm des Mikroskops weg ist.
HINWEIS: Der mitochondriendichte Teil enthält keine Lipide, die nach der Zentrifugation als der dunkelste Teil der Eizelle erscheinen. - Legen Sie die Spitze der Mikroklinge links von der obersten Eizelle ab, in Übereinstimmung mit dem Raum direkt über dem mitochondriendichten Teil. Halten Sie die Spitze der Klinge an der gleichen Stelle und senken Sie die Klinge vorsichtig ab, um den ganzen Weg durch die Eizelle zu schneiden.
- Stellen Sie sicher, dass der mitochondriendichte Ooplast und der mitochondrienreduzierte Ooplast eine ähnliche Größe haben; Bisektion sollte jede Eizelle in zwei Hälften schneiden, um zwei Ooplasten zu produzieren.
HINWEIS: Vergleichende Größen von Ooplasten, die aus einer einzelnen Eizelle hergestellt werden, können je nach Forschungszielen variieren. Wenn Mitochondrien-reduzierte Ooplasten mit einer somatischen Zelle verschmolzen werden sollen, sollte ihr Volumen größer sein als das der mitochondriendichten Ooplasten. - Stellen Sie sicher, dass die Klinge die Eizelle oberhalbiert den mitochondriendichten Teil und unterhalb des lipiddichten Anteils im klarsten Segment der zentrifugierten Eizelle.
- Stellen Sie sicher, dass der mitochondriendichte Ooplast und der mitochondrienreduzierte Ooplast eine ähnliche Größe haben; Bisektion sollte jede Eizelle in zwei Hälften schneiden, um zwei Ooplasten zu produzieren.
- Stellen Sie beim Anheben der Klinge sicher, dass die gleiche Linie beibehalten wird und dass die Spitze der Klinge an derselben Stelle verbleibt, und heben Sie dann die Spitze vorsichtig von der Platte an.
- Wiederholen Sie die Bisektionsschritte für alle Eizellen innerhalb des ersten Bisektionstropfens. Wenn Eizellen in zusätzlichen Tropfen vorhanden sind, orientieren und halbieren Sie die verbleibenden Eizellen.
- Sammeln Sie mit einer Mundpipette mitochondrienreduzierte Ooplasten aus dem ersten Bisektionstropfen. Legen Sie sie in den linken T20-Tropfen in der unteren Reihe der Platte. Wiederholen Sie dies für alle verbleibenden Bisektionstropfen.
HINWEIS: Mitochondrien-reduzierte Ooplasten enthalten Lipide, die dunkler sind als der Rest der Eizelle. - Sammeln Sie mit einer Mundpipette mitochondriendichte Ooplasten aus dem ersten Bisektionstropfen. Legen Sie sie in den rechten T20-Tropfen in der unteren Reihe der Platte. Wiederholen Sie dies für alle verbleibenden Bisektionstropfen.
HINWEIS: Mitochondrien-dichte Regionen haben ein sichtbares Konglomerat von Organellen, die hellgrau aussehen. - Holen Sie sich die T10-Viererschale aus dem Inkubator. Bewegen Sie mit einer Mundpipette alle mitochondrienreduzierten Ooplasten in die gut beschriftete "MR" und die mitochondriendichten Ooplasten in das gut beschriftete "M".
- Legen Sie die Vier-Well-Platte wieder in den CO 2-gesteuerten Inkubator. Lassen Sie die Ooplasten mindestens 30 Minuten ruhen, bevor Sie mtDNA quantifizieren.
6. Quantifizierung von mtDNA
- Verwenden Sie ein DNA-Extraktionskit, das entwickelt wurde, um Material aus einer kleinen Probe zu extrahieren, um DNA aus einzelnen Proben zu extrahieren (einzelne Eizellen, einzelne Mitochondrien-dichte Ooplasten, einzelne Mitochondrien-erschöpfte Ooplasten).
- Verwenden Sie eine DNA-Quantifizierungsmethode der Wahl, um sicherzustellen, dass DNA aus jeder Probe erfolgreich extrahiert wurde. Wenn die quantitative Polymerase-Kettenreaktion (qPCR) zu diesem Zeitpunkt nicht abgeschlossen ist, frieren Sie die extrahierte DNA in einem markierten Röhrchen in einem -80 ° C-Gefrierschrank ein.
- qPCR durchführen
- In Tabelle 2 können Sie das Volumen jedes Reagenzes bestimmen, das jedem qPCR-Röhrchen hinzugefügt werden soll, zusammen mit den Primersequenzen. Diese Primer wurden entwickelt, um die 12S-Region der Rinder-mtDNA zu verstärken.
- Stellen Sie die anfängliche Denaturierungszeit auf 10 min ein, gefolgt von 35 Zyklen von: 30 s Denaturierung bei 94 °C, 15 s Glühen bei 60 °C und 15 s Verlängerung bei 72 °C. Wenn die Reaktion abgeschlossen ist, notieren Sie sich die Zyklusschwellenwerte (Ct).
HINWEIS: Um relative mtDNA-Kopienzahlwerte zu erhalten, muss eine Standardkurve mit Proben mit bekannten Mengen an mtDNA-Kopienzahlen erstellt werden, die exponentiell ansteigen. Die Zyklusschwellenwerte müssen dann mit den folgenden Formeln manipuliert werden, um die relativen mtDNA-Kopienzahlen zu bestimmen. - Erhalten Sie die Konzentration der DNA mit der folgenden Gleichung:
DNA-Konzentration = (10(Ct-Intercept/Slope)) x getestetes Volumen - Erhalten Sie die Kopiennummer mit der folgenden Gleichung:
Kopiennummer =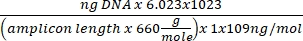
- Erhalten Sie die Kopiennummer mit der folgenden Gleichung:
Kopienzahl pro Zelle = 2 x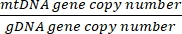
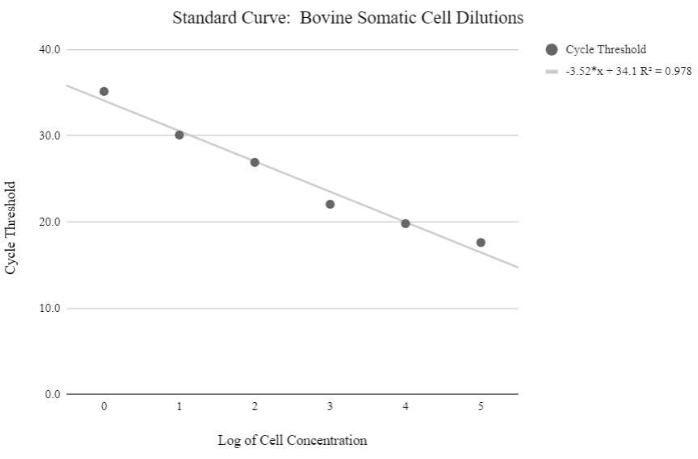
Abbildung 5: Somatische Zell-mtDNA-Standardkurve. Diese Standardkurve wurde durch die mtDNA-Quantifizierung logarithmischer Konzentrationen von somatischen Rinderzellen unter Verwendung der qPCR-Reagenzien und des Programms erstellt, wie in Protokollschritt 6.3 beschrieben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Quantitative PCR (qPCR) Ergebnisse werden verwendet, um die relativen Mengen an mtDNA zu bestimmen, die in jedem Ooplast vorhanden sind. Die beschriebene Reaktion soll die 12S-Region der bovinen mtDNA amplifizieren.
Wenn die Bisektion erfolgreich war, haben die Proben von ganzen Eizellen und mitochondriendichten Ooplasten ähnlicheCt-Werte. Die Proben von mitochondrienreduzierten Ooplasten haben im Vergleich zu den Proben aus den anderen beiden Gruppenhöhere KT-Werte. Ei...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Methoden, die bisher zur Verringerung der mtDNA-Kopienzahlen in Eizellen verwendet wurden, haben ihre jeweiligen Nachteile. Die auf Mikromanipulation basierende Entfernung von Mitochondrien aus Eizellen verringert die mtDNA-Kopienzahlen um durchschnittlich 64%27. Eine einzigartige Methode, die zuvor für die Enukleation verwendet wurde, beinhaltet die Verwendung von Pasteur-Pipetten mit kleinem Durchmesser und die Spaltung einer zona-pellucida-freien Eizelle an der Grenze zwischen einem Mikrotropf...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Die Autoren danken ihren Kollegen an der Utah State University, den Forschern der Reproduktionswissenschaften im San Diego Zoo und Dr. Rebecca Krisher von Genus PLC.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 1.5 mL centrifuge tubes | Fisher Scientific | 5408129 | |
| 60 mm dish | Sigma-Aldrich | D8054 | |
| Centrifuge | Eppendorf | 5424 | |
| Cytochalasin B | Sigma-Aldrich | C6762 | |
| Fetal Bovine Serum | Sigma-Aldrich | F2442 | |
| M199 Media | Sigma-Aldrich | M4530 | |
| Mineral Oil | Sigma-Aldrich | M8410 | |
| Mini Centrifuge | SCILOGEX | D1008 | |
| mtDNA Primer: Forward (12S) | GGGCTACATTCTCTACACCAAG | ||
| mtDNA Primer: Reverse (12S) | GTGCTTCATGGCCTAATTCAAC | ||
| NanoDrop Spectrophotometer | Thermo Scientific | ND2000 | |
| Opthalmic Scalpel with Aluminum Handle | PFM Medical | 207300633 | Microblade for bisection |
| Protease/pronase | Sigma-Aldrich | P5147 | |
| QIAamp DNA Micro Kit | Qiagen | 56304 | |
| QuantStudio™ 3 - 96-Well 0.2-mL | ThermoFisher | A28567 | |
| Search plate | Fisher Scientific | FB0875711A | |
| SYBR Green qPCR Master Mix | ThermoFisher | K0221 | qPCR master mix |
| Synthetic Oviductal Fluid with HEPES (HSOF) | |||
| ThermoPlate | Tokai Hit | TPi-SMZSSX | Heating stage |
Referenzen
- Loi, P., Modlinski, J. A., Ptak, G. Interspecies somatic cell nuclear transfer: A salvage tool seeking first aid. Theriogenology. 76 (2), 217-228 (2011).
- Wani, N. A., Vettical, B. S., Hong, S. B. First cloned Bactrian camel (camelus bactrianus) calf produced by interspecies somatic cell nuclear transfer: A step towards preserving the critically endangered wild Bactrian camels. PLOS ONE. 12 (5), 0177800(2017).
- Oh, H. J., et al. Cloning endangered gray wolves (canis lupus) from somatic cells collected postmortem. Theriogenology. 70 (4), 638-647 (2008).
- Srirattana, K., et al. Full-term development of gaur-bovine interspecies somatic cell nuclear transfer embryos: Effect of trichostatin a treatment. Cellular Reprogramming. 14 (3), 248-257 (2012).
- Kwon, D. K., et al. Blastocysts derived from adult fibroblasts of a rhesus monkey (macaca mulatta) using interspecies somatic cell nuclear transfer. Zygote. 19 (3), 199-204 (2011).
- Lee, E., et al. Production of cloned sei whale (Balaenoptera borealis) embryos by interspecies somatic cell nuclear transfer using enucleated pig oocytes. Journal of Veterinary Science. 10 (4), 285(2009).
- Lorthongpanich, C., Laowtammathron, C., Chan, A. W., Kedutat-Cairns, M., Parnpai, R. Development of interspecies cloned monkey embryos reconstructed with bovine enucleated oocytes. Journal of Reproduction and Development. 54 (5), 306-313 (2008).
- Hong, S. G., et al. Production of transgenic canine embryos using interspecies somatic cell nuclear transfer. Zygote. 20 (1), 67-72 (2011).
- Stewart, J. B., Chinnery, P. F. The dynamics of mitochondrial DNA heteroplasmy: Implications for human health and disease. Nature Reviews Genetics. 16 (9), 530-542 (2015).
- Takeda, K. Mitochondrial DNA transmission and confounding mitochondrial influences in cloned cattle and pigs. Reproductive Medicine and Biology. 12 (2), 47-55 (2013).
- Lanza, R. P., et al. Cloning of an endangered species (Bos Gaurus) using interspecies nuclear transfer. Cloning. 2 (2), 79-90 (2000).
- Evans, M. J., et al. Mitochondrial DNA genotypes in nuclear transfer-derived cloned sheep. Nature Genetics. 23 (1), 90-93 (1999).
- Meirelles, F. V., et al. Complete replacement of the mitochondrial genotype in a Bos indicus calf reconstructed by nuclear transfer to a Bos taurus oocyte. Genetics. 158 (1), 351-356 (2001).
- Beyhan, Z., Iager, A. E., Cibelli, J. B. Interspecies nuclear transfer: Implications for embryonic stem cell biology. Cell Stem Cell. 1 (5), 502-512 (2007).
- Lagutina, I., Fulka, H., Lazzari, G., Galli, C. Interspecies somatic cell nuclear transfer: advancements and problems. Cellular Reprogramming. 15 (5), 374-384 (2013).
- Jiang, Y., et al. Interspecies somatic cell nuclear transfer is dependent on compatible mitochondrial DNA and reprogramming factors. PLoS ONE. 6 (4), 14805(2011).
- Chiaratti, M. R., et al. Embryo mitochondrial DNA depletion is reversed during early embryogenesis in cattle. Biology of Reproduction. 82 (1), 76-85 (2010).
- Spikings, E. C., Alderson, J., John, J. C. Regulated mitochondrial DNA replication during oocyte maturation is essential for successful porcine embryonic development. Biology of Reproduction. 76 (2), 327-335 (2007).
- Cagnone, G. L., et al. Restoration of normal embryogenesis by mitochondrial supplementation in pig oocytes exhibiting mitochondrial DNA deficiency. Scientific Reports. 6 (1), 1-15 (2016).
- Spikings, E. C., Alderson, J., John, J. C. Regulated mitochondrial DNA replication during oocyte maturation is essential for successful porcine embryonic development. Biology of Reproduction. 76 (2), 327-335 (2007).
- Ferreira, A. F., et al. Does supplementation with mitochondria improve oocyte competence? A systematic review. Reproduction. 161 (3), 269-287 (2021).
- Bhat, M. H., et al. Live birth of a pashmina goat kid after transfer of handmade cloned embryos. Journal of Reproduction and Development. , (2019).
- Tecirlioglu, R. T., et al. Birth of a cloned calf derived from a vitrified hand-made cloned embryo. Reproduction, Fertility and Development. 15 (7), 361(2003).
- Zhang, P., et al. Handmade cloned transgenic piglets expressing the nematode fat-1 gene. Cellular Reprogramming. 14 (3), 258-266 (2012).
- Zhang, P., et al. Handmade cloned transgenic sheep rich in omega-3 fatty acids. PLOS ONE. 8 (2), 55941(2013).
- Lagutina, I., et al. Somatic cell nuclear transfer in horses: Effect of oocyte morphology, embryo reconstruction method and donor cell type. Reproduction. 130 (4), 559-567 (2005).
- Chiaratti, M. R., et al. Embryo mitochondrial DNA depletion is reversed during early embryogenesis in cattle. Biology of Reproduction. 82 (1), 76-85 (2010).
- Hosseini, S. M., et al. and efficient method of manual oocyte enucleation using a pulled pasteur pipette. In Vitro Cellular and Developmental Biology - Animal. 49 (8), 569-575 (2013).
- Zampolla, T., Spikings, E., Rawson, D., Zhang, T. Cytoskeleton proteins F-actin and tubulin distribution and interaction with mitochondria in the granulosa cells surrounding stage III zebrafish (danio rerio) oocytes. Theriogenology. 76 (6), 1110-1119 (2011).
- International Union for Conservation of Nature. The IUCN Red List of Threatened Species. International Union for Conservation of Nature. , (2021).
- Berg, D. K., Li, C., Asher, G., Wells, D. N., Oback, B. Red deer cloned from antler stem cells and their differentiated progeny. Biology of Reproduction. 77 (3), 384-394 (2007).
- Gómez, M. C., et al. Birth of African wildcat cloned kittens born from domestic cats. Cloning and Stem Cells. 6 (3), 247-258 (2004).
- Lanza, R. P., et al. Cloning of an endangered species (Bos gaurus) using interspecies nuclear transfer. Cloning. 2 (2), 79-90 (2000).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten