É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Uso de Bisection para reduzir DNA mitocondrial no Oócito Bovino
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para reduzir significativamente os números de cópia de DNA mitocondrial em um oócito bovino (P < 0,0001). Este método utiliza centrifugação e biseção para reduzir substancialmente mitocôndrias oócitos e pode permitir uma maior chance de desenvolvimento nos embriões de transferência nuclear de células somáticas de células somáticas reconstruídas.
Resumo
A transferência nuclear de células somáticas interespécies (iSCNT) pode ser usada para resgatar espécies ameaçadas de extinção, mas existem duas populações distintas de DNA mitocondrial (mtDNA) dentro do embrião reconstruído: uma dentro do ooplasma receptor e outra dentro da célula somática do doador. Essa heteroplasma mitocondrial pode levar a problemas de desenvolvimento no embrião e no feto. Protocolos de clonagem artesanal incluem biseção de oócito, que pode ser usada para diminuir o número de cópia de MTDNA, reduzindo o grau de heteroplasmia mitocondrial em um embrião reconstruído. Centrifugação de oócitos bovinos desnudos e maduros produziu uma fração visível mitocôndria-densa em um polo do oócito. A zonae pellucidae dos oócitos foi removida pela exposição a uma solução de pronase. A biseção foi realizada usando um microdílo para remover a fração de mitocôndrias visíveis. qPCR foi utilizado para quantificar o mtDNA presente em amostras de DNA extraídos de oócitos inteiros e ooplastos bisseccionados, proporcionando uma comparação dos números de cópias de MTDNA antes e depois da biseção. Os números de cópia foram calculados utilizando-se valores de limiar de ciclo, fórmula de linha de regressão de uma curva padrão e uma razão que incluía os respectivos tamanhos de produtos DE PCR mtDNA e produtos PCR genômicos. Um oócito bovino tinha um número médio de cópia mtDNA (± desvio padrão) de 137.904 ± 94.768 (n = 38). Um ooplasto empobrecido por mitocôndrias tinha um número médio de cópia mtDNA de 8.442 ± 13.806 (n = 33). As cópias médias de mtDNA presentes em um ooplasto rico em mitocôndrias foram de 79.390 ± 58.526 cópias mtDNA (n = 28). As diferenças entre essas médias calculadas indicam que a centrifugação e a biseção subsequente podem diminuir significativamente os números de cópias de MTDNA presentes no ooplasto empobrecido por mitocôndrias quando comparado com o oócito original (P < 0,0001, determinado pela ANOVA unidirecional). A redução do dNH deve diminuir o grau de heteroplasmia mitocondrial em um embrião reconstruído, possivelmente promovendo o desenvolvimento embrionário e fetal padrão. A suplementação com extrato mitocondrial da célula doador somática também pode ser essencial para alcançar o desenvolvimento embrionário bem sucedido.
Introdução
A transferência nuclear de células somáticas (SCNT) inclui a fusão de um oócito enucleado de um animal e uma célula somática de um animal da mesma espécie. Na maioria dos casos, o oócito e a célula somática são originários da mesma espécie, e as taxas de natalidade viva estão abaixo de 6%1. Algumas pesquisas envolvem o uso de interespécies SCNT (iSCNT), que inclui a fusão de uma célula somática e oócito que se originam de duas espécies diferentes. Nesses estudos, as taxas de natalidade ao vivo são ainda menores do que no SCNT- tipicamente inferior a 1%1. No entanto, o iSCNT tem a capacidade de ser usado como método de resgate de espécies ameaçadas de extinção, uma vez que as células somáticas desses animais são mais acessíveis do que suas células germinativas1. Os oócitos receptores usados no iSCNT são frequentemente espécies domésticas ou comuns de laboratório, como vacas, suínos e camundongos. Algumas tentativas feitas até agora produziram com sucesso jovens vivos, embora os filhotes produzidos tenham sido animais intrageníricos (as espécies de oócitos receptores e espécies de células doadoras eram membros do mesmo gênero)2,3,4. Os modelos intergênicos (que utilizam uma célula oótil e somática de animais em diferentes gêneros) ainda não produziram animais vivos, e a maioria dos embriões reconstruídos prende no estágio 8-16 de desenvolvimento in vitro 5,6,7,8. Uma possível explicação para essa parada embrionária de desenvolvimento é a ocorrência de heteroplasmia mitocondrial nos embriões - a presença de mais de um tipo de DNA mitocôndria (mtDNA) em uma única célula. A heteroplasma pode levar a questões como ineficiência do desenvolvimento ou falha no embrião ou no animal vivo1. A patogênese também pode ocorrer mais tarde na vidado animal 9. Embora esta questão também esteja presente na prole do SCNT, o componente interespecífico dentro dos embriões iSCNT agrava a questão.
Quando o mtDNA embrionário vem de duas espécies diferentes, as mitocôndrias oócitos receptoras, que representam a maioria, não funcionam de forma eficiente ou eficaz com o núcleo 1,10 da célula doadora. Maiores lacunas taxonômicas entre as duas espécies utilizadas no iSCNT provavelmente intensificam esse problema; descendentes vivos intragênicos produzidos (bos gaurus e bos indicus prole usando oócitos de Bos taurus), bem como descendentes produzidos através do TRADICIONAL SCNT (por exemplo, ovis aries prole usando oócitos Ovis aries) foram mostrados como quimeras (mtDNA de dois indivíduos estava presente nestes animais 11,12,13). No entanto, eles se desenvolveram muito mais do que os embriões SCNT intergênicos14,15. A troca de informações entre as mitocôndrias oócitos e o núcleo da célula doadora poderia ser mais bem sucedida no embrião intragênico do que no embrião intergênico16.
A quantidade de dN em um oócito bovino maduro é aproximadamente 100 vezes maior do que a quantidade encontrada em uma célula somática12. A redução dessa proporção poderia incentivar a proliferação das mitocôndrias celulares somáticas dentro do embrião reconstruído, permitindo que uma maior população de mitocôndrias produtivas esteja presente16. Isso poderia, por sua vez, fornecer mais energia para atender aos requisitos do embriãoem desenvolvimento 15. Tentativas anteriores feitas para reduzir o número de cópia de mtDNA do oócito ou embrião incluem aplicação química, micromanipulação e suplementação do oócito ou embrião com mitocôndrias adicionais da espécie celular doadora 16,17,18,19,20. No entanto, a aplicação química (como 2',3'-dideoxycytidine) não é ideal para o desenvolvimento embrionário, e reduziu os números de cópias de oócito mtDNA em aproximadamente metadede 18. A redução de oócitos anteriores por micromanipulação só removeu uma média de 64% do mtDNA17 do oócito. Embora a suplementação de mitocôndrias de células doadoras possa ser uma opção viável, seu uso ainda não produziu um animal intergenérico vivo dentro dos estudos do iSCNT21.
O uso de biseção para reduzir o número de cópia oócito mtDNA ainda não foi utilizado em estudos publicados. Bissectar oócitos com a intenção de fundir os ooplastos com uma célula somática é a premissa da clonagem artesanal (HMC), que normalmente utiliza biseção como método de remoção do corpo polar e placa de metafase do oócito metafase II (MII). O HMC produziu com sucesso descendentes em várias espécies, incluindo cabras, bovinos, suínos, ovinos e cavalos 22,23,24,25,26, mas normalmente não inclui um passo de centrifugação antes da biseção. A integração da centrífuga de alta velocidade do oócito permite o isolamento das mitocôndrias (e, portanto, mtDNA) em um polo do oócito, que pode então ser bissecto usando um microblade para remover essas frações mitocondria-densas. Dois ooplastos empobrecidos mitocôndrias podem então ser fundidos com uma célula somática, como é o caso do HMC, para formar um embrião reconstruído que contém consideravelmente menos mtDNA da espécie oócteto.
A pergunta que tentamos responder com este protocolo é como reduzir o dNM no oócito bovino, a fim de produzir um embrião reconstruído viável que contenha mtDNA menos heteroplasmica. Neste protocolo, os oócitos foram centrifudos e bisseccionados. Os números de cópias ooplastia e ooplasta ooplasta mtDNA foram calculados para determinar a eficácia desta técnica na redução do número de cópia mtDNA do oocito bovino.
Protocolo
O seguinte protocolo segue as diretrizes de cuidado e ética dos animais fornecidas pela Universidade Estadual de Utah.
1. Preparação da mídia
- Antes do manuseio de oócitos, prepare as seguintes soluções, conforme descrito na Tabela 1: 400 μL da Solução Hyaluronidase, 500 μL de mídia T2, 1.020 μL de mídia T20 e 800 μL de mídia T10.
- Divida a mídia T10 em dois poços de uma placa de quatro poços, 400 μL por poço. Rotule um bem com "M" e o segundo bem com "MR". Coloque em uma incubadora de CO2 de 5% até depois da biseção.
- Prepare 500 μL de Citochalasina B (CB)/Fluido Oviduto sintético com HEPES (HSOF). Aliquot 50 μL de solução CB/HSOF em um tubo de centrífuga separado de 1,5 mL.
- Prepare 40 μL de Solução Pronase. Centrifugar por pelo menos 30 s a 2.680 x g. Misture 20 μL do supernasal com 20 μL de T2 a um novo tubo de centrífuga; isso forma uma solução de pronase diluída de 5 mg/mL.
- Prepare 3 mL de HSOF. Em uma placa de pesquisa, deposite separadamente quatro gotas de 400 μL; coloque esta placa sob um estereótipo.
- Prepare 500 μL de solução CB/T20.
2. Maturação in vitro (IVM) de oócitos bovinos
- IVM: Complexos de cumulus-oócito aspirados de cultura (COCs) a 38,5 °C em uma incubadora de CO2 de 5% por 21 h em um prato de quatro poços de mídia de maturação contendo 10% FBS, 0,26 UI/mL FSH e 100 penicilina/estreptomicina U/mL de 100 U/mL.
- Realize as seguintes etapas para denude os oócitos.
- Colete o número desejado dos COCs usando uma pipeta de 200 μL e deposite-os na parte inferior de um tubo de centrífuga de 1,5 μL.
- Adicione o mesmo volume de 0,6 mg/mL de hialuronidase ao tubo centrífuga com os oócitos (ou seja, se o volume de oócitos e mídia IVM for de 100 μL, adicione 100 μL de hialuronidase).
- Pipeta a solução para cima e para baixo sem criar bolhas, até que todas as células cumulus tenham sido removidas.
- Verifique o amadurecimento dos oócitos.
- Use a pipeta para adicionar 200 μL de HSOF de uma das quatro gotas de HSOF na placa de pesquisa para a solução oótida/hialuronidase.
- Transfira os oócitos da gota hyaluronidase/HSOF e coloque-os em 400 μL nãoused de gota HSOF. Lave-os com duas gotas adicionais de HSOF para ajudar na remoção de resquícios de células de hialuronidase e cumulus.
- Utilizando uma pipeta bucal (ou uma pipeta e ponta de 10 μL) e alta ampliação microscópica, role e selecione os oócitos com base na presença polar do corpo.
- Depois de classificar os oócitos, colete o número desejado de oócitos MII a serem bissectos e coloque-os em 400 μL de gota HSOF.
NOTA: Se os oócitos estiverem fora da incubadora por mais de 30 minutos, os oócitos podem ser colocados de volta em um poço de mídia de maturação e colocados em uma incubadora controlada por CO2 para descansar por pelo menos 30 minutos. Limitar o número de oócitos a serem bisseccionados a uma quantidade que é viável para terminar a bissecção em aproximadamente 30 minutos para minimizar o tempo fora da incubação. Se os ooplastos devem ser fundidos com uma célula somática, ativada e cultivada como embriões, os oócitos devem ser enucleados neste momento.
3. Centrifugação dos oócitos
NOTA: Se os oócitos foram colocados na incubadora em meios de maturação, mova-os para a gota HSOF da qual foram coletados mais recentemente.
- Utilizando uma pipeta bucal, colete os oócitos maduros selecionados da gota HSOF e coloque-os no tubo de 1,5 mL contendo 50 μL da solução HSOF/CB. Centrifugar os oócitos a 15.000 x g por 12 min.
NOTA: Não é prejudicial que as bolhas estejam presentes no tubo de centrífuga. - Enquanto os oócitos estão sendo centrifugados, prepare a placa de biseção.
- Faça o padrão como mostrado na Figura 1 na tampa de uma placa de petri de 60 mm (20 μL por gota), e cubra totalmente as gotas com óleo mineral.
- Usando um marcador de ponta fina, marque as linhas por baixo do prato, como mostrado na Figura 2.
- Desenhe uma caixa em torno da queda de pronase e uma linha entre as gotas CB/T20 e a linha inferior do T20 cai.
- Cubra o prato com uma tampa opaca até que a centrifugação esteja completa para evitar alterações de osmolaridade de microdrops.
- Assim que a centrifugação estiver completa, use uma pipeta de 200 μL para coletar os oócitos dentro da solução e mova-os para uma porção vazia de uma nova placa de pesquisa com quatro gotas HSOF de 400 μL.
- Coletar e lavar os oócitos através das quatro gotas de HSOF.
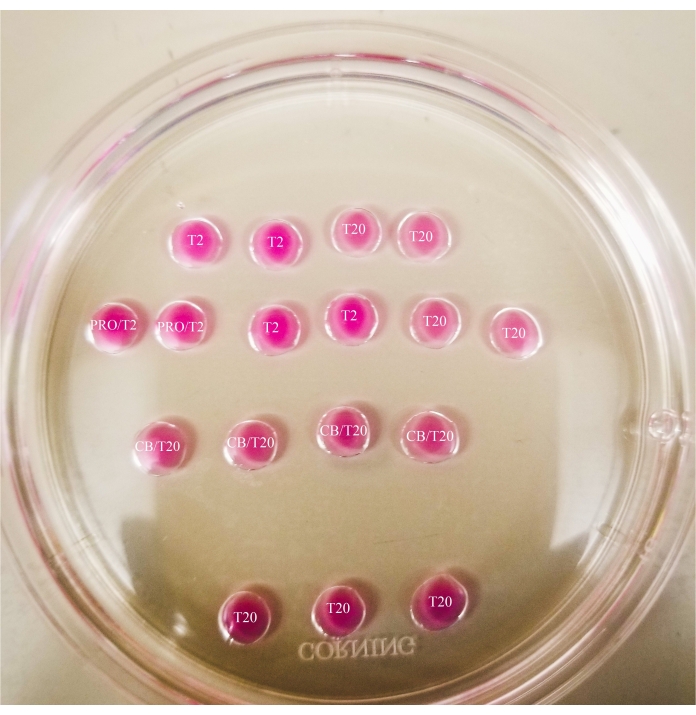
Figura 1: Placa de biseção. Todas as gotas mostradas têm um volume de 20 μL. A placa tem um diâmetro de 60 mm. As gotas foram completamente cobertas com óleo mineral. Os oócitos serão colocados primeiro na queda mais alta e mais à esquerda do T2 (indicado aqui com uma estrela). PRO/T2: 10 μL de pronase e 10 μL de T2, combinados antes da criação de microdrops. CB/T20: 1 μL de citochalasina B por 1 mL de T20, combinado antes da criação de microdrops. Clique aqui para ver uma versão maior desta figura.
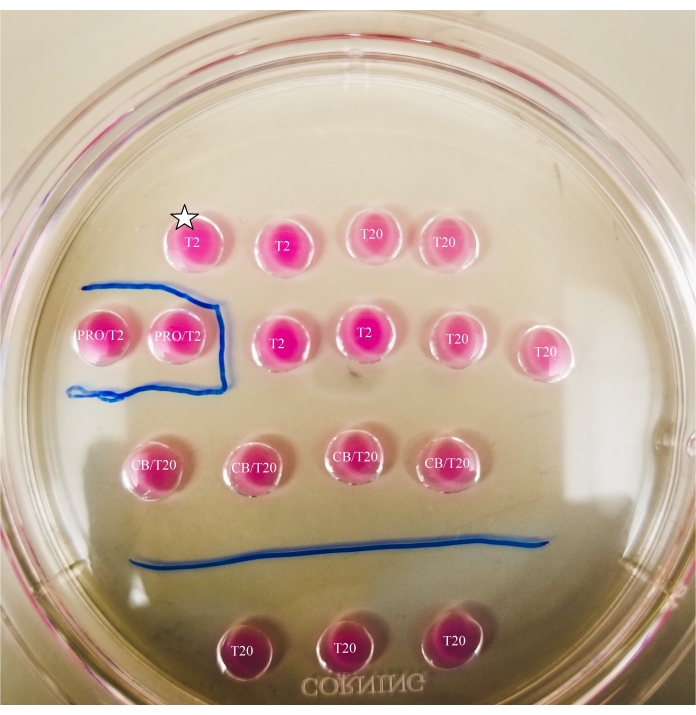
Figura 2: Placa de biseção marcada. As linhas são desenhadas com um marcador de ponta fina na parte inferior da placa, para fornecer referências de localização para observações e transferências de oóplastes e ooplastos feitas sob o microscópio. Os oócitos serão colocados primeiro na queda mais alta e mais à esquerda do T2 (indicado aqui com uma estrela). Clique aqui para ver uma versão maior desta figura.
4. Preparação do oócito para biseção
NOTA: O seguinte processo envolve a preparação dos oócitos para biseção.
- Ligue o estágio de aquecimento para 37 °C.
- Use uma pipeta bucal para mover os oócitos centrifugados da gota HSOF para a gota T2 superior esquerda da placa de biseção, em seguida, lave os oócitos através das próximas três gotas na linha superior (T2, T20, T20).
- Deposite os oócitos em uma das gotas de pronase, assegurando que há pouco ou nenhum contato entre eles.
NOTA: A remoção da zona pellucida pode levar quantidades variadas de tempo, mas normalmente ocorre entre 30-120 s com o uso de um estágio de aquecimento. A ausência de uma etapa de aquecimento se estenderá desta vez. - Observe os oócitos até que haja deformação clara da zonae pellucidae (ver Figura 3).
- Uma vez que uma única zona oocyte pellucida se tornou deformada, mova esse oócito para a queda T2 vizinha. Repita à medida que os oócitos adicionais se deformam, até que todos os oócitos tenham sido removidos do pronase e colocados na queda do T2.
- Observe os oócitos até que apenas uma fina camada da zona pellucida permaneça.
- Lave os oócitos através das próximas três gotas dentro da linha (T2, T20, T20), depois deposite-os em linhas verticais dentro das gotas CB/T20 (ver Figura 4).
- Comece com menos oócitos nas linhas verticais e aumente o número de oócitos por gota à medida que as habilidades de biseção progredirem.
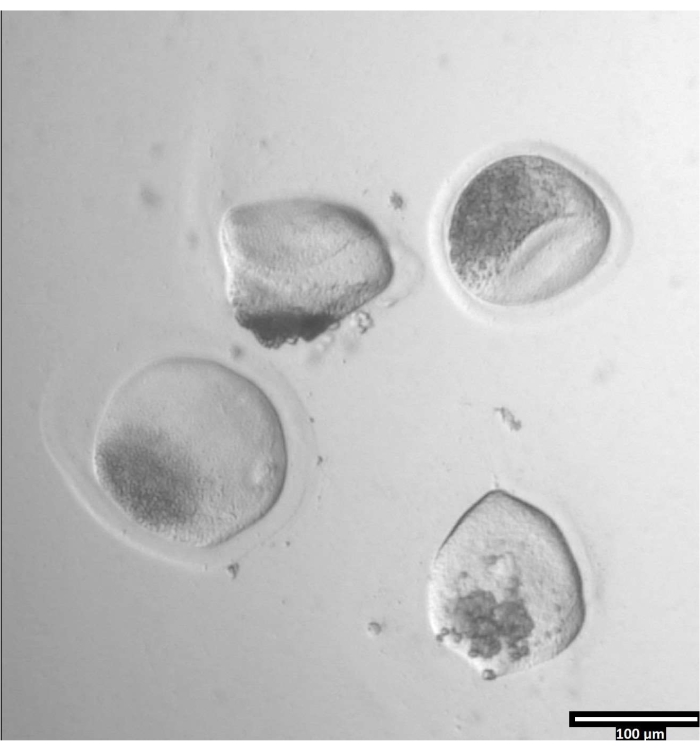
Figura 3: Remoção da zona pellucida utilizando pronase. (80x) Uma zona oócica pellucida começará a parecer deformada quando a pronase tiver afetado a zona pellucida o suficiente para que o oócito seja movido para a queda t2 adjacente. Clique aqui para ver uma versão maior desta figura.

Figura 4: Orientação oócida em gotas de biseção. (80x) Oócitos livres de zona são depositados em uma orientação quase vertical dentro de cada gota CB/T20 antes da biseção. Clique aqui para ver uma versão maior desta figura.
5. Biseção dos oócitos
- Concentre-se na primeira gota cb/t20 (biseção) que contém oócitos, e use uma pipeta bucal para girar os ocitos, de modo que a porção mitocôndria-densa de cada oócito esteja voltada para ou para longe do braço do microscópio.
NOTA: A porção mitocôndria-densa não conterá lipídios, que aparecem como a porção mais escura do oócito após centrifugação. - Descanse a ponta do microplade à esquerda do oócito mais alto, em linha com o espaço diretamente acima da porção mitocôndria-densa. Mantendo a ponta da lâmina no mesmo lugar, abaixe cuidadosamente a lâmina para cortar todo o caminho através do oócito.
- Certifique-se de que o ooplasto denso-denso mitocôndria e mitocôndria-reduzido são semelhantes em tamanho; biseção deve cortar cada oócito ao meio para produzir dois ooplastos.
NOTA: Os tamanhos comparativos de ooplastos produzidos a partir de um único oócito podem variar de acordo com as metas de pesquisa; se os ooplastos reduzidos à mitocôndria devem ser fundidos com uma célula somática, seu volume deve ser maior do que os ooplastos mitocondria-densos. - Certifique-se de que a lâmina está bisseccionando o oócito acima da porção densa mitocôndria e abaixo da porção lipídica-densa, no segmento mais claro do oócito centrifutado.
- Certifique-se de que o ooplasto denso-denso mitocôndria e mitocôndria-reduzido são semelhantes em tamanho; biseção deve cortar cada oócito ao meio para produzir dois ooplastos.
- Ao levantar a lâmina, certifique-se de que a mesma linha está mantida, e que a ponta da lâmina permaneça no mesmo lugar, em seguida, levante suavemente a ponta da placa.
- Repita os passos de biseção para todos os oócitos dentro da primeira queda de biseção. Se os oócitos estiverem presentes em gotas adicionais, oriente e bissecto os oócitos restantes.
- Usando uma pipeta bucal, colete ooplastos reduzidos por mitocôndrias da primeira queda de biseção. Coloque-os na gota T20 à esquerda localizada na linha inferior da placa. Repita todas as gotas de biseção restantes.
NOTA: Os ooplastos reduzidos à mitocôndria conterão lipídios, que são mais escuros em cores do que o resto do oócito. - Usando uma pipeta bucal, colete ooplastos mitocôndrias-densas da primeira queda de biseção. Coloque-os na gota T20 da direita localizada na linha inferior da placa. Repita todas as gotas de biseção restantes.
NOTA: As regiões densas mitocôndrias terão um conglomerado visível de organelas que serão cinza-claro na aparência. - Recupere o prato T10 de quatro poços da incubadora. Usando uma pipeta bucal, mova todos os ooplastos reduzidos para mitocôndrias para o bem rotulado "MR", e mova os ooplastos mitocondria-densos para o bem rotulado "M".
- Coloque a placa de quatro poços de volta na incubadora controlada por CO2. Deixe os ooplastos descansarem por pelo menos 30 minutos antes da quantificação do mtDNA.
6. Quantificação do mtDNA
- Use um kit de extração de DNA projetado para extrair material de uma pequena amostra para extrair DNA de amostras individuais (oócitos únicos, ooplastos densos mitocôndrias únicos, ooplastos empobrecidos de mitocôndrias únicas).
- Use um método de quantificação de DNA da escolha para garantir que o DNA foi extraído com sucesso de cada amostra. Se a reação quantitativa da cadeia de polimerase (qPCR) não for concluída nesse momento, congele o DNA extraído em um tubo rotulado em um congelador de -80 °C.
- Executar qPCR
- Consulte a Tabela 2 para determinar o volume de cada reagente a ser adicionado a cada tubo qPCR, juntamente com as sequências de primer. Estes primers são projetados para amplificar a região 12S do mtDNA bovino.
- Defina o tempo inicial de desnaturação para 10 min, seguido por 35 ciclos de: 30 s de desnaturação a 94 °C, 15 s de ressarem a 60 °C e 15 s de extensão a 72 °C. Quando a reação estiver completa, regissão os valores do limiar de ciclo (Ct).
NOTA: Para obter valores relativos do número de cópias do MTDNA, uma curva padrão deve ser produzida utilizando amostras com quantidades conhecidas de números de cópias de mtDNA, que aumentam exponencialmente. Os valores do limiar do ciclo precisam então ser manipulados usando as seguintes fórmulas para determinar números relativos de cópias do MTDNA. - Obtenha a concentração do DNA usando a equação abaixo:
Concentração de DNA = (10(Ct-intercept/slope)) x volume testado - Obtenha o número da cópia usando a equação abaixo:
Número da cópia =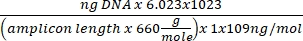
- Obtenha o número da cópia usando a equação abaixo:
Número da cópia por célula = 2 x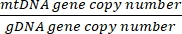
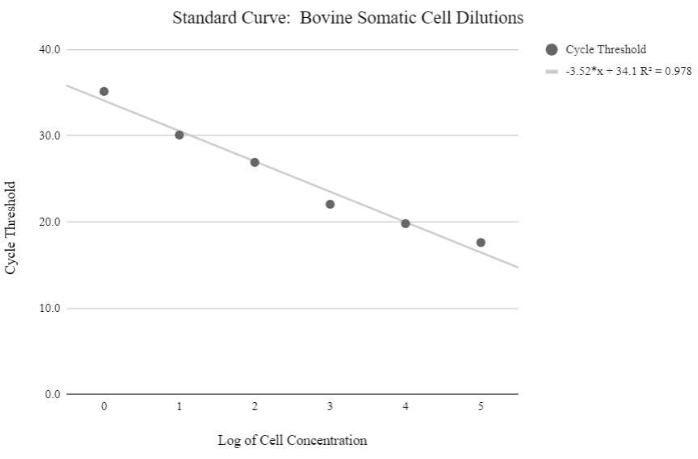
Figura 5: Curva padrão mtDNA de célula somática. Esta curva padrão foi criada através da quantificação mtDNA de concentrações logarítmicas de células somáticas bovinas utilizando os reagentes qPCR e programa conforme descrito na etapa 6.3 do protocolo. Clique aqui para ver uma versão maior desta figura.
Resultados
Os resultados quantitativos de PCR (qPCR) são utilizados para determinar as quantidades relativas de mtDNA presentes em cada ooplasto. A reação descrita foi projetada para amplificar a região 12S do mtDNA bovino.
Se a biseção foi bem sucedida, as amostras de oócitos inteiros e ooplastos mitocôndrias-densas terão valores Ct semelhantes. As amostras de ooplastos reduzidos por mitocôndrias terão valores Ct mais elevados quando comparadas com as amostras dos outros...
Discussão
Métodos anteriormente utilizados para diminuir o número de cópias de MTDNA em oócitos têm suas respectivas desvantagens. A remoção baseada em micromanipulação de mitocôndrias de oócitos diminui os números de cópias de MTDNA em uma média de 64%27. Um método único, anteriormente utilizado para a enucleação, envolve o uso de pipetas Pasteur de pequeno diâmetro e a divisão de um oócito zona pellucida na fronteira entre uma microdrop de mídia e o óleo mineral circundante. Juntame...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores desejam agradecer aos seus colegas da Universidade Estadual de Utah, aos pesquisadores de Ciência Reprodutiva do Zoológico de San Diego e à Dra.
Materiais
| Name | Company | Catalog Number | Comments |
| 1.5 mL centrifuge tubes | Fisher Scientific | 5408129 | |
| 60 mm dish | Sigma-Aldrich | D8054 | |
| Centrifuge | Eppendorf | 5424 | |
| Cytochalasin B | Sigma-Aldrich | C6762 | |
| Fetal Bovine Serum | Sigma-Aldrich | F2442 | |
| M199 Media | Sigma-Aldrich | M4530 | |
| Mineral Oil | Sigma-Aldrich | M8410 | |
| Mini Centrifuge | SCILOGEX | D1008 | |
| mtDNA Primer: Forward (12S) | GGGCTACATTCTCTACACCAAG | ||
| mtDNA Primer: Reverse (12S) | GTGCTTCATGGCCTAATTCAAC | ||
| NanoDrop Spectrophotometer | Thermo Scientific | ND2000 | |
| Opthalmic Scalpel with Aluminum Handle | PFM Medical | 207300633 | Microblade for bisection |
| Protease/pronase | Sigma-Aldrich | P5147 | |
| QIAamp DNA Micro Kit | Qiagen | 56304 | |
| QuantStudio™ 3 - 96-Well 0.2-mL | ThermoFisher | A28567 | |
| Search plate | Fisher Scientific | FB0875711A | |
| SYBR Green qPCR Master Mix | ThermoFisher | K0221 | qPCR master mix |
| Synthetic Oviductal Fluid with HEPES (HSOF) | |||
| ThermoPlate | Tokai Hit | TPi-SMZSSX | Heating stage |
Referências
- Loi, P., Modlinski, J. A., Ptak, G. Interspecies somatic cell nuclear transfer: A salvage tool seeking first aid. Theriogenology. 76 (2), 217-228 (2011).
- Wani, N. A., Vettical, B. S., Hong, S. B. First cloned Bactrian camel (camelus bactrianus) calf produced by interspecies somatic cell nuclear transfer: A step towards preserving the critically endangered wild Bactrian camels. PLOS ONE. 12 (5), 0177800 (2017).
- Oh, H. J., et al. Cloning endangered gray wolves (canis lupus) from somatic cells collected postmortem. Theriogenology. 70 (4), 638-647 (2008).
- Srirattana, K., et al. Full-term development of gaur-bovine interspecies somatic cell nuclear transfer embryos: Effect of trichostatin a treatment. Cellular Reprogramming. 14 (3), 248-257 (2012).
- Kwon, D. K., et al. Blastocysts derived from adult fibroblasts of a rhesus monkey (macaca mulatta) using interspecies somatic cell nuclear transfer. Zygote. 19 (3), 199-204 (2011).
- Lee, E., et al. Production of cloned sei whale (Balaenoptera borealis) embryos by interspecies somatic cell nuclear transfer using enucleated pig oocytes. Journal of Veterinary Science. 10 (4), 285 (2009).
- Lorthongpanich, C., Laowtammathron, C., Chan, A. W., Kedutat-Cairns, M., Parnpai, R. Development of interspecies cloned monkey embryos reconstructed with bovine enucleated oocytes. Journal of Reproduction and Development. 54 (5), 306-313 (2008).
- Hong, S. G., et al. Production of transgenic canine embryos using interspecies somatic cell nuclear transfer. Zygote. 20 (1), 67-72 (2011).
- Stewart, J. B., Chinnery, P. F. The dynamics of mitochondrial DNA heteroplasmy: Implications for human health and disease. Nature Reviews Genetics. 16 (9), 530-542 (2015).
- Takeda, K. Mitochondrial DNA transmission and confounding mitochondrial influences in cloned cattle and pigs. Reproductive Medicine and Biology. 12 (2), 47-55 (2013).
- Lanza, R. P., et al. Cloning of an endangered species (Bos Gaurus) using interspecies nuclear transfer. Cloning. 2 (2), 79-90 (2000).
- Evans, M. J., et al. Mitochondrial DNA genotypes in nuclear transfer-derived cloned sheep. Nature Genetics. 23 (1), 90-93 (1999).
- Meirelles, F. V., et al. Complete replacement of the mitochondrial genotype in a Bos indicus calf reconstructed by nuclear transfer to a Bos taurus oocyte. Genetics. 158 (1), 351-356 (2001).
- Beyhan, Z., Iager, A. E., Cibelli, J. B. Interspecies nuclear transfer: Implications for embryonic stem cell biology. Cell Stem Cell. 1 (5), 502-512 (2007).
- Lagutina, I., Fulka, H., Lazzari, G., Galli, C. Interspecies somatic cell nuclear transfer: advancements and problems. Cellular Reprogramming. 15 (5), 374-384 (2013).
- Jiang, Y., et al. Interspecies somatic cell nuclear transfer is dependent on compatible mitochondrial DNA and reprogramming factors. PLoS ONE. 6 (4), 14805 (2011).
- Chiaratti, M. R., et al. Embryo mitochondrial DNA depletion is reversed during early embryogenesis in cattle. Biology of Reproduction. 82 (1), 76-85 (2010).
- Spikings, E. C., Alderson, J., John, J. C. Regulated mitochondrial DNA replication during oocyte maturation is essential for successful porcine embryonic development. Biology of Reproduction. 76 (2), 327-335 (2007).
- Cagnone, G. L., et al. Restoration of normal embryogenesis by mitochondrial supplementation in pig oocytes exhibiting mitochondrial DNA deficiency. Scientific Reports. 6 (1), 1-15 (2016).
- Spikings, E. C., Alderson, J., John, J. C. Regulated mitochondrial DNA replication during oocyte maturation is essential for successful porcine embryonic development. Biology of Reproduction. 76 (2), 327-335 (2007).
- Ferreira, A. F., et al. Does supplementation with mitochondria improve oocyte competence? A systematic review. Reproduction. 161 (3), 269-287 (2021).
- Bhat, M. H., et al. Live birth of a pashmina goat kid after transfer of handmade cloned embryos. Journal of Reproduction and Development. , (2019).
- Tecirlioglu, R. T., et al. Birth of a cloned calf derived from a vitrified hand-made cloned embryo. Reproduction, Fertility and Development. 15 (7), 361 (2003).
- Zhang, P., et al. Handmade cloned transgenic piglets expressing the nematode fat-1 gene. Cellular Reprogramming. 14 (3), 258-266 (2012).
- Zhang, P., et al. Handmade cloned transgenic sheep rich in omega-3 fatty acids. PLOS ONE. 8 (2), 55941 (2013).
- Lagutina, I., et al. Somatic cell nuclear transfer in horses: Effect of oocyte morphology, embryo reconstruction method and donor cell type. Reproduction. 130 (4), 559-567 (2005).
- Chiaratti, M. R., et al. Embryo mitochondrial DNA depletion is reversed during early embryogenesis in cattle. Biology of Reproduction. 82 (1), 76-85 (2010).
- Hosseini, S. M., et al. and efficient method of manual oocyte enucleation using a pulled pasteur pipette. In Vitro Cellular and Developmental Biology - Animal. 49 (8), 569-575 (2013).
- Zampolla, T., Spikings, E., Rawson, D., Zhang, T. Cytoskeleton proteins F-actin and tubulin distribution and interaction with mitochondria in the granulosa cells surrounding stage III zebrafish (danio rerio) oocytes. Theriogenology. 76 (6), 1110-1119 (2011).
- International Union for Conservation of Nature. The IUCN Red List of Threatened Species. International Union for Conservation of Nature. , (2021).
- Berg, D. K., Li, C., Asher, G., Wells, D. N., Oback, B. Red deer cloned from antler stem cells and their differentiated progeny. Biology of Reproduction. 77 (3), 384-394 (2007).
- Gómez, M. C., et al. Birth of African wildcat cloned kittens born from domestic cats. Cloning and Stem Cells. 6 (3), 247-258 (2004).
- Lanza, R. P., et al. Cloning of an endangered species (Bos gaurus) using interspecies nuclear transfer. Cloning. 2 (2), 79-90 (2000).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados