このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
牛卵母細胞のミトコンドリアDNAを減少させるための二分法の使用
* これらの著者は同等に貢献しました
要約
ここでは、ウシ卵母細胞におけるミトコンドリアDNAコピー数を有意に減少させるプロトコールを提示する(P < 0.0001)。この方法は、遠心分離および二分法を利用して卵母細胞ミトコンドリアを実質的に減少させ、再構成された種間体細胞核移植胚における発生の増大の機会を可能にし得る。
要約
種間体細胞核移植(iSCNT)は絶滅危惧種を救うために使用することができるが、ミトコンドリアDNA(mtDNA)の2つの異なる集団が再構築された胚内に存在する:1つはレシピエント卵形質内および1つはドナー体細胞内である。このミトコンドリアヘテロプラスミーは、胚および胎児の発達上の問題につながる可能性がある。手作りのクローニングプロトコルには卵母細胞二切除が含まれ、mtDNAコピー数を減らし、再構築された胚におけるミトコンドリアヘテロプラズムの程度を低下させるために使用できる。脱蹄した成熟したウシ卵母細胞の遠心分離は、卵母細胞の一極に目に見えるミトコンドリア緻密画分を生じた。卵母細胞の透明帯は、プロナーゼ溶液への曝露によって除去した。可視ミトコンドリア画分を除去するためにマイクロブレードを用いて二分断を行った。qPCRを使用して、卵母細胞全体および二等分した卵子から抽出したDNAサンプル中に存在するmtDNAを定量し、二分切除前後のmtDNAコピー数を比較した。コピー数は、サイクル閾値、標準曲線の回帰直線式、およびmtDNA PCR産物およびゲノムPCR産物のそれぞれのサイズを含む比率を用いて計算した。1つのウシ卵母細胞は、137,904±94,768(n=38)の平均mtDNAコピー数(標準偏差±)を有していた。1つのミトコンドリア枯渇卵母細胞は、8,442±13,806(n = 33)の平均mtDNAコピー数を有していた。ミトコンドリアに富む卵母細胞中に存在する平均mtDNAコピーは、79,390±58,526 mtDNAコピーであった(n = 28)。これらの計算された平均の差は、遠心分離とその後の二分割が、元の卵母細胞(P<0.0001、一元配置分散分析によって決定される)と比較して、ミトコンドリア枯渇した卵細胞中に存在するmtDNAコピー数を有意に減少させることができることを示している。mtDNAの減少は、再構築された胚におけるミトコンドリアヘテロプラミーの程度を低下させ、おそらく標準的な胚および胎児の発達を促進するはずである。体細胞ドナー細胞からのミトコンドリア抽出物の補充もまた、胚発生を成功させるために不可欠であり得る。
概要
体細胞核移植(SCNT)は、1匹の動物からの除核卵母細胞と、同じ種の動物からの体細胞との融合を含む。ほとんどの場合、卵母細胞と体細胞は同じ種に由来し、出生率は6%1未満です。いくつかの研究は、2つの異なる種に由来する体細胞と卵母細胞の融合を含む種間SCNT(iSCNT)の使用を含む。これらの研究では、出生率はSCNTよりもさらに低く、通常は1%未満です1。しかしながら、iSCNTは、これらの動物由来の体細胞が生殖細胞1よりもアクセスしやすいため、絶滅危惧種を救出する方法として使用できる能力を有する。iSCNTで使用されるレシピエント卵母細胞は、多くの場合、ウシ、ブタ、およびマウスなどの国内または一般的な実験室種である。これまでになされたいくつかの試みは、生産された子孫がジェネリック内動物であったが(レシピエント卵母細胞種およびドナー細胞種は同じ属のメンバーであった)2、3、4。ジェネリック間モデル(異なる属の動物由来の卵母細胞および体細胞を利用する)はまだ生きた動物を産生しておらず、再構成された胚の大部分は、in vitro発生の8〜16細胞段階で停止する5、6、7、8。この胚発生停止の1つの可能な説明は、胚におけるミトコンドリアヘテロプラスミーの発生、すなわち単一の細胞における複数のミトコンドリアDNA(mtDNA)型の存在である。ヘテロプラスミーは、胚または生きた動物1における発生の非効率性または障害などの問題をもたらし得る。病因は、動物の生涯の後半にも起こり得る9。この問題はSCNTの子孫にも存在するが、iSCNT胚内の種間成分が問題を悪化させる。
胚性mtDNAが2つの異なる種に由来する場合、大多数を占めるレシピエント卵母細胞ミトコンドリアは、ドナー細胞の核1,10と効率的または効果的に機能しない。iSCNTで使用される2つの種の間の分類学的ギャップが大きいほど、この問題が激化する可能性が高い。内ジェネリック内で産生された生きた子孫(ボス・ガウルスおよびボス・インディカス子孫はボス・トーラス・卵母細胞を使用)、ならびに伝統的なSCNTを介して産生された子孫(例えばオービス・アリエス・卵母細胞を用いたオビス・アリエス子孫)はキメラであることが示された(2個体由来のmtDNAがこれらの動物に存在していた11、12、13)。しかし、彼らはジェネリック間SCNT胚よりもはるかに発展しました14,15。卵母細胞ミトコンドリアとドナー細胞の核との間の情報交換は、ジェネリック間胚よりもジェネリック内胚においてより成功する可能性がある16。
成熟ウシ卵母細胞中のmtDNAの量は、1つの体細胞12中に見出される量の約100倍である。この比率を低くすると、体細胞ミトコンドリアが再構築された胚内で増殖し、生産的なミトコンドリアのより大きな集団が存在することを可能にする可能性がある16。これは、順番に、発達胚15の要件を満たすためにより多くのエネルギーを提供することができる。卵母細胞または胚のmtDNAコピー数を減少させるためになされた以前の試みには、化学的適用、微小操作、およびドナー細胞種からの追加のミトコンドリアで卵母細胞または胚を補うことが含まれる16、17、18、19、20。しかし、化学的適用(2',3'-ジデオキシシチジンなど)は胚発生には理想的ではなく、卵母細胞mtDNAコピー数を約半分に18個減少させている。マイクロマニピュレーションによる以前の卵母細胞mtDNA減少は、卵母細胞のmtDNAの平均64%しか除去していない17。ドナー細胞ミトコンドリアの補充は実行可能な選択肢であるかもしれないが、その使用はまだiSCNT研究21内で生きたジェネリック間動物を産生していない。
卵母細胞mtDNAコピー数を減らすための二分法の使用は、公表された研究においてまだ使用されていない。卵子細胞と体細胞を融合させる目的で卵母細胞を二等分することは、手作りクローニング(HMC)の前提であり、これは典型的には、中相II(MII)卵母細胞から極性体および中期プレートを除去する方法として二分割を利用する。HMCは、ヤギ、ウシ、ブタ、ヒツジ、ウマ22、23、24、25、26を含むいくつかの種で子孫を首尾よく産出してきたが、典型的には二分分離前の遠心分離工程を含まない。卵母細胞の高速遠心分離を統合することで、卵母細胞の1極でミトコンドリア(したがってmtDNA)を単離することができ、マイクロブレードを使用して二等分してミトコンドリア密度の高い画分を除去することができます。その後、2つのミトコンドリア枯渇した卵胞体を、HMCの場合のように体細胞と融合させて、卵母細胞種からのmtDNAをかなり少なく含む再構成胚を形成することができる。
このプロトコルで答えようとする質問は、ヘテロプラズムmtDNAの含有量が少ない生存可能な再構築胚を生成するために、ウシ卵母細胞中のmtDNAをどのように低減するかということです。このプロトコールでは、卵母細胞を遠心分離し、二等分した。ウープラストおよびインタクトな卵母細胞のmtDNAコピー数を計算し、ウシ卵母細胞のmtDNAコピー数を減少させる上でのこの技術の有効性を決定した。
プロトコル
以下のプロトコルは、ユタ州立大学が提供する動物ケアと倫理のガイドラインに従っています。
1. メディアの準備
- 卵母細胞の取り扱いに先立って、 表1に記載されるように、以下の溶液を調製する:400 μLのヒアルロニダーゼ溶液、500 μLのT2培地、1,020 μLのT20培地、および800 μLのT10培地。
- T10培地を4ウェルプレートの2つのウェル(1ウェルあたり400μL)に分割します。1 つのウェルに "M" のラベルを付け、2 番目のウェルに "MR" のラベルを付けます。二分後まで5%CO2 インキュベーターに入れる。
- サイトカラシンB(CB)/合成卵管液500μLをHEPES(HSOF)で調製する。50 μL の CB/HSOF 溶液を別の 1.5 mL 遠沈管にアリコートします。
- 40 μLのプロナーゼ溶液を調製する。2,680 x gで少なくとも30秒間遠心分離機。20 μLの上清と20 μLのT2を新しい遠沈管に混合する。これは、希釈された5mg/mLプロナーゼ溶液を形成する。
- 3mLのHSOFを調製する。検索プレート上に、400μL滴4滴を別々に堆積させる。このプレートを実体顕微鏡の下に置きます。
- 500 μL の CB/T20 溶液を調製します。
2. ウシ卵母細胞の インビトロ 成熟(IVM)
- IVM:吸引した積雲-卵母細胞複合体(COC)を、10%FBS、0.26 IU/mL FSH、および100 U/mLペニシリン/ストレプトマイシンを含む成熟培地の4ウェルディッシュ中で、5%CO2インキュベーター内で38.5°Cで21時間培養した。
- 卵母細胞をデヌードするには、次の手順を実行します。
- 200 μL のピペットを使用して必要な数の COC を収集し、1.5 μL の遠沈管の底に堆積させます。
- 同じ容量の0.6 mg/mLヒアルロニダーゼを卵母細胞とともに遠沈管に加えます(すなわち、卵母細胞およびIVM培地の体積が100 μLの場合は、100 μLのヒアルロニダーゼを加えます)。
- すべての積雲細胞が除去されるまで、泡を作らずに溶液を上下にピペットで送ります。
- 卵母細胞の成熟を確認してください。
- ピペットを使用して、検索プレート上の4つのHSOF滴のうちの1つから200μLのHSOFを卵母細胞/ヒアルロニダーゼ溶液に加えます。
- ヒアルロニダーゼ/HSOF滴から卵母細胞を移し、未使用の400μLのHSOF滴に入れます。ヒアルロニダーゼと積雲細胞の残骸を除去するのを助けるために、2つの追加のHSOF滴でそれらを洗ってください。
- 口内ピペット(または10 μLピペットおよびチップ)および高顕微鏡倍率を用いて、ロールし、極性体存在に基づいて卵母細胞を選択する。
- 卵母細胞を選別した後、二等分されるMII卵母細胞の所望の数を収集し、400μLのHSOF滴に入れる。
注:卵母細胞が30分を超えてインキュベーターの外側にいた場合、卵母細胞を成熟培地の井戸に戻してCO2制御インキュベーターに入れ、少なくとも30分間休ませることができます。二等分される卵母細胞の数を、インキュベーション以外の時間を最小限に抑えるために、約30分で二等分を終えることが実現可能な量に制限する。卵子を体細胞と融合させ、活性化し、胚として培養する場合、卵母細胞はこの時点で除核されるべきである。
3. 卵母細胞の遠心分離
注:卵母細胞を成熟培地中のインキュベーターに入れた場合は、最後に収集されたHSOFドロップに移動します。
- マウスピペットを使用して、HSOF滴から選択した成熟卵母細胞を回収し、50μLのHSOF/CB溶液を含む1.5mLチューブに入れます。卵母細胞を15,000 x g で12分間遠心分離する。
注:気泡が遠沈管に存在することは有害ではありません。 - 卵母細胞が遠心分離されている間に、二分割プレートを準備する。
- 60mmシャーレの蓋に 図1 のようなパターンを作り(1滴あたり20μL)、鉱油で滴を完全に覆います。
- 図 2 に示すように、先端の細いマーカーを使用して、皿の下の線に印を付けます。
- プロナーゼ滴の周囲にボックスを描き、CB/T20 滴と T20 滴の一番下の行の間に線を引きます。
- マイクロドロップの浸透圧変化を防ぐために、遠心分離が完了するまで皿を不透明な蓋で覆います。
- 遠心分離が完了したらすぐに、200 μLのピペットを使用して溶液中の卵母細胞を収集し、4つの400 μL HSOF滴を含む新しい探索プレートの空部分に移動します。
- 4滴のHSOF滴を通して卵母細胞を集めて洗い流します。
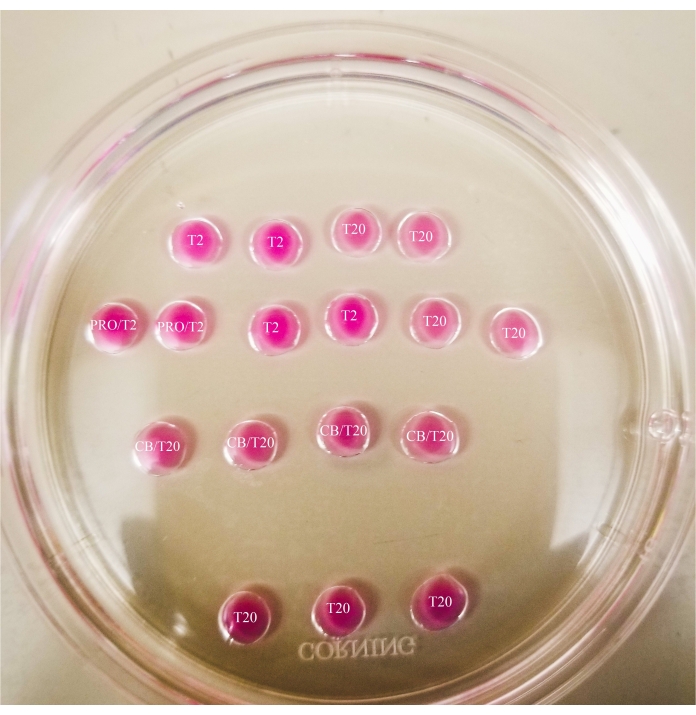
図1:二等分プレート 示されているすべての滴は、20μLの容量を有する。プレートの直径は60mmです。滴は鉱物油で完全に覆われています。卵母細胞は、まず最上部と左端のT2ドロップに配置されます(ここでは星印で示されています)。PRO/T2:10 μL のプロナーゼと 10 μL の T2 をマイクロドロップを作成する前に組み合わせた。CB/T20:T20の1mLあたり1μLのサイトカラシンBを、マイクロドロップを作成する前に組み合わせた。 この図の拡大版を表示するには、ここをクリックしてください。
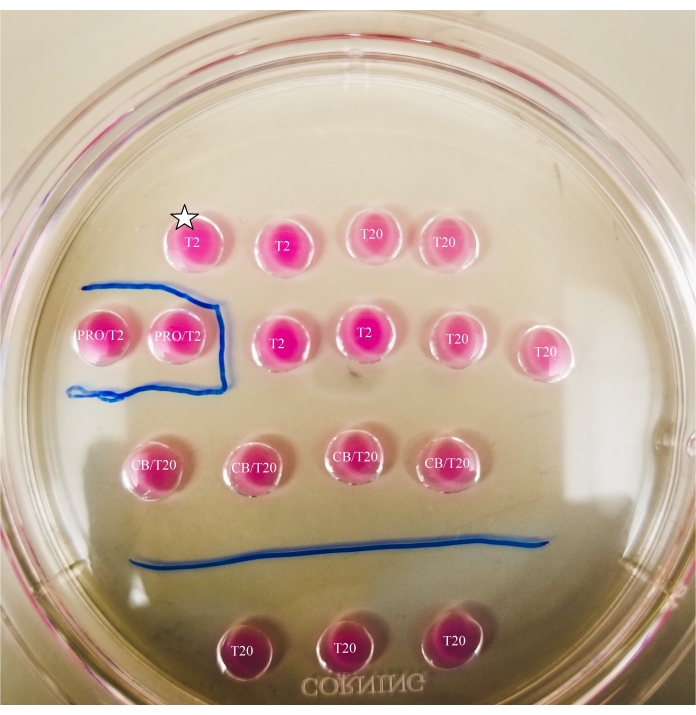
図2:マークされた二等分プレート プレートの下部に細い先端のマーカーで線が描かれ、顕微鏡の下で行われた観察および卵母細胞および卵芽細胞の移動のための位置参照を提供します。卵母細胞は、まず最上部と左端のT2ドロップに配置されます(ここでは星印で示されています)。 この図の拡大版を表示するには、ここをクリックしてください。
4.二分切除のための卵母細胞の調製
注:以下のプロセスは、二分切除のための卵母細胞の調製を含む。
- 加熱ステージを37°Cにオンにします。
- マウスピペットを使用して、遠心分離した卵母細胞をHSOF滴から二分割プレートの左上のT2滴に移動し、次に卵母細胞を上段の次の3滴(T2、T20、T20)を通して洗浄する。
- 卵母細胞をプロナーゼ滴の1つに沈着させ、それらの間にほとんどまたはまったく接触がないことを保証する。
注:透明帯の除去にはさまざまな時間がかかることがありますが、通常、加熱段階を使用すると30〜120秒の間に発生します。加熱段階の不在は、この時間を延長する。 - 透明帯の明確な変形があるまで卵母細胞を観察します( 図3参照)。
- 単一の卵母細胞透明帯が変形したら、その卵母細胞を隣接するT2ドロップに移動します。追加の卵母細胞が変形するにつれて、すべての卵母細胞がプロナーゼから除去され、T2ドロップに入れられるまで繰り返す。
- ゾーン透明体の薄い層だけが残るまで卵母細胞を観察します。
- 卵母細胞を列内の次の3滴(T2、T20、T20)を通して洗浄し、CB/T20滴内の垂直線に沈着させます( 図4参照)。
- 縦線の卵母細胞の数を減らすことから始め、二分法スキルが進歩するにつれて、1滴あたりの卵母細胞の数を増やします。
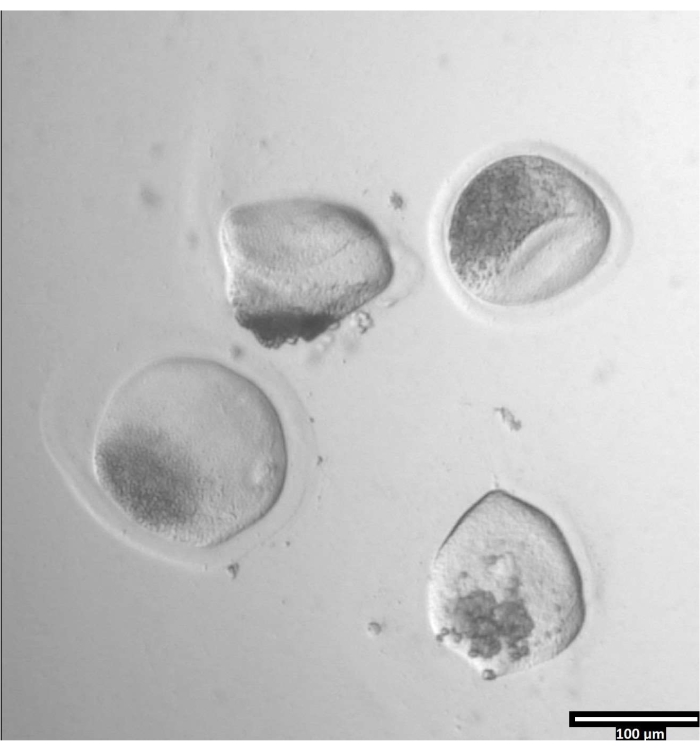
図3:プロナーゼを使用した透明帯の除去(80x)プロナーゼが透明帯に影響を与え、卵母細胞が隣接するT2滴に移動するのに十分になると、透明帯が変形して見え始めます。この図の拡大版を表示するには、ここをクリックしてください。

図4:二分割滴における卵母細胞の向き (80x) Zonaフリーの卵母細胞は、二分切除前に各CB/T20液滴内で垂直に近い向きで沈着する。 この図の拡大版を表示するには、ここをクリックしてください。
5.卵母細胞の二分割
- 卵母細胞を含む最初の左端のCB/T20(二分割)滴に焦点を合わせ、口のピペットを使用して卵母細胞を回転させ、各卵母細胞のミトコンドリア密度部分が顕微鏡の腕に向かっているか、または顕微鏡の腕から離れるようにします。
注:ミトコンドリアの緻密な部分には脂質は含まれず、遠心分離後に卵母細胞の最も暗い部分として表示されます。 - マイクロブレードの先端を、ミトコンドリア緻密な部分の真上の空間に沿って、最上部の卵母細胞の左側に置きます。刃の先端を同じ場所に保ち、卵母細胞を最後まで切断するために慎重に刃を下げます。
- ミトコンドリア密度の高い卵子とミトコンドリア縮小卵子の大きさが類似していることを確認する。二分割は、2つの卵胞体を生成するために各卵母細胞を半分に切断する必要があります。
注:単一の卵母細胞から産生される卵胞体の比較サイズは、研究目標に基づいて異なる可能性がある。ミトコンドリア還元型卵胞体を体細胞と融合させる場合、その体積はミトコンドリア緻密性卵胞体よりも大きくなければならない。 - ブレードが、遠心分離された卵母細胞の最も透明な部分において、ミトコンドリア緻密部の上および脂質緻密部の下方の卵母細胞を二等分していることを確認する。
- ミトコンドリア密度の高い卵子とミトコンドリア縮小卵子の大きさが類似していることを確認する。二分割は、2つの卵胞体を生成するために各卵母細胞を半分に切断する必要があります。
- ブレードを持ち上げるときは、同じラインが維持されていること、およびブレードの先端が同じ場所に残っていることを確認してから、プレートから先端を慎重に持ち上げます。
- 最初の二分切除滴内のすべての卵母細胞について二分切開ステップを繰り返します。卵母細胞が追加の滴で存在する場合は、残りの卵母細胞を配向させ、二等分する。
- 口のピペットを使用して、最初の二分割滴からミトコンドリア還元型卵子を採取する。プレートの一番下の列にある左側のT20ドロップに置きます。残りのすべての二等分滴について繰り返します。
注:ミトコンドリア還元卵子は脂質を含みます, それは卵母細胞の残りの部分よりも色が濃いです. - 口のピペットを使用して、最初の二分割滴からミトコンドリア密度のオプラストを収集する。プレートの一番下の列にある右側のT20ドロップに置きます。残りのすべての二等分滴について繰り返します。
注:ミトコンドリア密集領域は、外観が薄い灰色になる細胞小器官の目に見える集合体を有する。 - インキュベーターからT10 4ウェルディッシュを取り出します。マウスピペットを使用して、すべてのミトコンドリア還元型卵子を「MR」とラベル付けされたウェルに移動し、ミトコンドリア密度のウープラストを「M」とラベル付けされたウェルに移動します。
- 4ウェルプレートをCO2制御インキュベーターに戻します。mtDNAの定量を行う前に、ウープラストを少なくとも30分間休ませてください。
6. mtDNAの定量
- 個々のサンプル(単一卵母細胞、単一ミトコンドリア高密度卵細胞、単一ミトコンドリア枯渇卵胞)からDNAを抽出するために、小さなサンプルから材料を抽出するように設計されたDNA抽出キットを使用します。
- 選択した DNA 定量方法を使用して、各サンプルから DNA が正常に抽出されたことを確認します。その時点で定量的ポリメラーゼ連鎖反応(qPCR)が完了しない場合は、-80°Cの冷凍庫で標識チューブ内の抽出DNAを凍結する。
- qPCR の実行
- 表 2 を参照して、プライマー配列とともに各 qPCR チューブに追加する各試薬の容量を決定します。これらのプライマーは、ウシmtDNAの12S領域を増幅するように設計されている。
- 初期変性時間を10分間に設定し、続いて94°Cで30秒の変性、60°Cで15秒のアニーリング、72°Cで15秒の伸長を35サイクル行った。 反応が完了したら、サイクルしきい値 (Ct) の値を記録します。
注: 相対的な mtDNA コピー数値を取得するには、指数関数的に増加する既知の量の mtDNA コピー数を持つサンプルを使用して標準曲線を作成する必要があります。サイクルしきい値は、以下の式を使用して操作し、相対的なmtDNAコピー数を決定する必要があります。 - 以下の式を使用してDNAの濃度を求める:
DNA濃度 = (10(Ct-切片/傾き)) x 試験量 - コピー番号は、次の式を使用して求めます。
コピー番号 =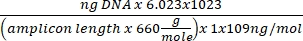
- コピー番号は、次の式を使用して求めます。
セルあたりのコピー数 = 2 x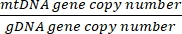
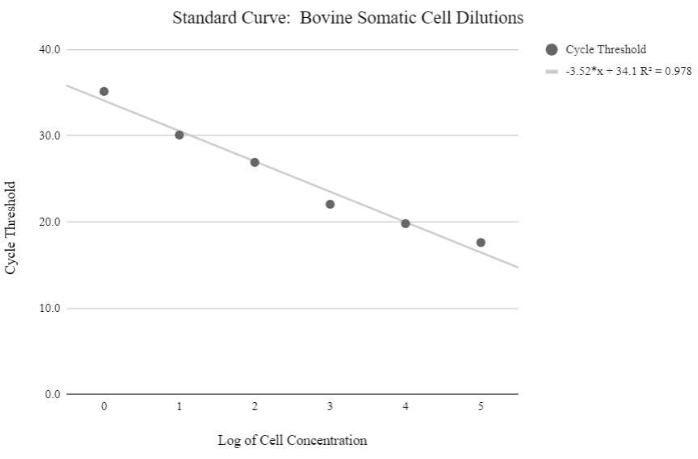
図5:体細胞mtDNA標準曲線。 この標準曲線は、プロトコルステップ6.3に記載されているqPCR試薬およびプログラムを使用したウシ体細胞の対数濃度のmtDNA定量によって作成されました。 この図の拡大版を表示するには、ここをクリックしてください。
結果
定量PCR(qPCR)結果は、各ウープラスト中に存在するmtDNAの相対量を決定するために使用されます。記載された反応は、ウシmtDNAの12S領域を増幅するように設計されている。
二分割が成功した場合、卵母細胞全体およびミトコンドリア密度の卵子からのサンプルは、同様のCt 値を有するであろう。ミトコンドリア還元型卵子からのサンプルは、他の2つの群からのサン?...
ディスカッション
卵母細胞におけるmtDNAコピー数を減少させるために以前に用いられた方法は、それぞれの欠点を有する。卵母細胞からのミトコンドリアのマイクロマニピュレーションベースの除去は、mtDNAコピー数を平均64%減少させる27。以前は除核に使用されていたユニークな方法は、小径パスツールピペットの使用と、媒体のマイクロドロップと周囲の鉱物油との境界で透明帯を含まな?...
開示事項
著者らには開示するものは何もありません。
謝辞
著者らは、ユタ州立大学の同僚、サンディエゴ動物園の生殖科学研究者、およびPLC属のレベッカ・クリッシャー博士に感謝したい。
資料
| Name | Company | Catalog Number | Comments |
| 1.5 mL centrifuge tubes | Fisher Scientific | 5408129 | |
| 60 mm dish | Sigma-Aldrich | D8054 | |
| Centrifuge | Eppendorf | 5424 | |
| Cytochalasin B | Sigma-Aldrich | C6762 | |
| Fetal Bovine Serum | Sigma-Aldrich | F2442 | |
| M199 Media | Sigma-Aldrich | M4530 | |
| Mineral Oil | Sigma-Aldrich | M8410 | |
| Mini Centrifuge | SCILOGEX | D1008 | |
| mtDNA Primer: Forward (12S) | GGGCTACATTCTCTACACCAAG | ||
| mtDNA Primer: Reverse (12S) | GTGCTTCATGGCCTAATTCAAC | ||
| NanoDrop Spectrophotometer | Thermo Scientific | ND2000 | |
| Opthalmic Scalpel with Aluminum Handle | PFM Medical | 207300633 | Microblade for bisection |
| Protease/pronase | Sigma-Aldrich | P5147 | |
| QIAamp DNA Micro Kit | Qiagen | 56304 | |
| QuantStudio™ 3 - 96-Well 0.2-mL | ThermoFisher | A28567 | |
| Search plate | Fisher Scientific | FB0875711A | |
| SYBR Green qPCR Master Mix | ThermoFisher | K0221 | qPCR master mix |
| Synthetic Oviductal Fluid with HEPES (HSOF) | |||
| ThermoPlate | Tokai Hit | TPi-SMZSSX | Heating stage |
参考文献
- Loi, P., Modlinski, J. A., Ptak, G. Interspecies somatic cell nuclear transfer: A salvage tool seeking first aid. Theriogenology. 76 (2), 217-228 (2011).
- Wani, N. A., Vettical, B. S., Hong, S. B. First cloned Bactrian camel (camelus bactrianus) calf produced by interspecies somatic cell nuclear transfer: A step towards preserving the critically endangered wild Bactrian camels. PLOS ONE. 12 (5), 0177800 (2017).
- Oh, H. J., et al. Cloning endangered gray wolves (canis lupus) from somatic cells collected postmortem. Theriogenology. 70 (4), 638-647 (2008).
- Srirattana, K., et al. Full-term development of gaur-bovine interspecies somatic cell nuclear transfer embryos: Effect of trichostatin a treatment. Cellular Reprogramming. 14 (3), 248-257 (2012).
- Kwon, D. K., et al. Blastocysts derived from adult fibroblasts of a rhesus monkey (macaca mulatta) using interspecies somatic cell nuclear transfer. Zygote. 19 (3), 199-204 (2011).
- Lee, E., et al. Production of cloned sei whale (Balaenoptera borealis) embryos by interspecies somatic cell nuclear transfer using enucleated pig oocytes. Journal of Veterinary Science. 10 (4), 285 (2009).
- Lorthongpanich, C., Laowtammathron, C., Chan, A. W., Kedutat-Cairns, M., Parnpai, R. Development of interspecies cloned monkey embryos reconstructed with bovine enucleated oocytes. Journal of Reproduction and Development. 54 (5), 306-313 (2008).
- Hong, S. G., et al. Production of transgenic canine embryos using interspecies somatic cell nuclear transfer. Zygote. 20 (1), 67-72 (2011).
- Stewart, J. B., Chinnery, P. F. The dynamics of mitochondrial DNA heteroplasmy: Implications for human health and disease. Nature Reviews Genetics. 16 (9), 530-542 (2015).
- Takeda, K. Mitochondrial DNA transmission and confounding mitochondrial influences in cloned cattle and pigs. Reproductive Medicine and Biology. 12 (2), 47-55 (2013).
- Lanza, R. P., et al. Cloning of an endangered species (Bos Gaurus) using interspecies nuclear transfer. Cloning. 2 (2), 79-90 (2000).
- Evans, M. J., et al. Mitochondrial DNA genotypes in nuclear transfer-derived cloned sheep. Nature Genetics. 23 (1), 90-93 (1999).
- Meirelles, F. V., et al. Complete replacement of the mitochondrial genotype in a Bos indicus calf reconstructed by nuclear transfer to a Bos taurus oocyte. Genetics. 158 (1), 351-356 (2001).
- Beyhan, Z., Iager, A. E., Cibelli, J. B. Interspecies nuclear transfer: Implications for embryonic stem cell biology. Cell Stem Cell. 1 (5), 502-512 (2007).
- Lagutina, I., Fulka, H., Lazzari, G., Galli, C. Interspecies somatic cell nuclear transfer: advancements and problems. Cellular Reprogramming. 15 (5), 374-384 (2013).
- Jiang, Y., et al. Interspecies somatic cell nuclear transfer is dependent on compatible mitochondrial DNA and reprogramming factors. PLoS ONE. 6 (4), 14805 (2011).
- Chiaratti, M. R., et al. Embryo mitochondrial DNA depletion is reversed during early embryogenesis in cattle. Biology of Reproduction. 82 (1), 76-85 (2010).
- Spikings, E. C., Alderson, J., John, J. C. Regulated mitochondrial DNA replication during oocyte maturation is essential for successful porcine embryonic development. Biology of Reproduction. 76 (2), 327-335 (2007).
- Cagnone, G. L., et al. Restoration of normal embryogenesis by mitochondrial supplementation in pig oocytes exhibiting mitochondrial DNA deficiency. Scientific Reports. 6 (1), 1-15 (2016).
- Spikings, E. C., Alderson, J., John, J. C. Regulated mitochondrial DNA replication during oocyte maturation is essential for successful porcine embryonic development. Biology of Reproduction. 76 (2), 327-335 (2007).
- Ferreira, A. F., et al. Does supplementation with mitochondria improve oocyte competence? A systematic review. Reproduction. 161 (3), 269-287 (2021).
- Bhat, M. H., et al. Live birth of a pashmina goat kid after transfer of handmade cloned embryos. Journal of Reproduction and Development. , (2019).
- Tecirlioglu, R. T., et al. Birth of a cloned calf derived from a vitrified hand-made cloned embryo. Reproduction, Fertility and Development. 15 (7), 361 (2003).
- Zhang, P., et al. Handmade cloned transgenic piglets expressing the nematode fat-1 gene. Cellular Reprogramming. 14 (3), 258-266 (2012).
- Zhang, P., et al. Handmade cloned transgenic sheep rich in omega-3 fatty acids. PLOS ONE. 8 (2), 55941 (2013).
- Lagutina, I., et al. Somatic cell nuclear transfer in horses: Effect of oocyte morphology, embryo reconstruction method and donor cell type. Reproduction. 130 (4), 559-567 (2005).
- Chiaratti, M. R., et al. Embryo mitochondrial DNA depletion is reversed during early embryogenesis in cattle. Biology of Reproduction. 82 (1), 76-85 (2010).
- Hosseini, S. M., et al. and efficient method of manual oocyte enucleation using a pulled pasteur pipette. In Vitro Cellular and Developmental Biology - Animal. 49 (8), 569-575 (2013).
- Zampolla, T., Spikings, E., Rawson, D., Zhang, T. Cytoskeleton proteins F-actin and tubulin distribution and interaction with mitochondria in the granulosa cells surrounding stage III zebrafish (danio rerio) oocytes. Theriogenology. 76 (6), 1110-1119 (2011).
- International Union for Conservation of Nature. The IUCN Red List of Threatened Species. International Union for Conservation of Nature. , (2021).
- Berg, D. K., Li, C., Asher, G., Wells, D. N., Oback, B. Red deer cloned from antler stem cells and their differentiated progeny. Biology of Reproduction. 77 (3), 384-394 (2007).
- Gómez, M. C., et al. Birth of African wildcat cloned kittens born from domestic cats. Cloning and Stem Cells. 6 (3), 247-258 (2004).
- Lanza, R. P., et al. Cloning of an endangered species (Bos gaurus) using interspecies nuclear transfer. Cloning. 2 (2), 79-90 (2000).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved