需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
基于CRISPR-Cas系统和抗CRISPR蛋白的基因数字电路
摘要
CRISPR-Cas系统和抗CRISPR蛋白被整合到 酿酒酵母的布尔门方案中。新的小逻辑电路表现出良好的性能,加深了对基于dCas9/dCas12a的转录因子和抗CRISPR蛋白性质的理解。
摘要
合成基因布尔门和数字电路具有广泛的应用,从医疗诊断到环境保护。CRISPR-Cas系统及其天然抑制剂——抗CRISPR蛋白(Acrs)的发现为设计和实施 体内 基因数字电路提供了一种新工具。在这里,我们描述了一种协议,该协议遵循"设计-构建-测试-学习"生物工程循环的思想,并利用dCas9 / dCas12a及其相应的Acrs来建立小型转录网络,其中一些行为类似于 酿酒酵母中的布尔门。这些结果指出了dCas9/dCas12a作为转录因子的性质。特别是,为了实现基因表达的最大激活,dSpCas9需要与收集VP64激活域(AD)多个拷贝的工程支架RNA相互作用。相比之下,dCas12a应在C末端与强VP64-p65-RTA (VPR) AD融合。此外,两种Cas蛋白的活性不会通过增加细胞中sgRNA / crRNA的量来增强。本文还解释了如何基于CRISPR-dCas-Acr相互作用构建布尔门。人雌激素受体的 AcrIIA4 融合激素结合结构域是对 β-雌二醇有反应的 NOT 门的核心,而由诱导性 GAL1 启动子合成的 AcrVA 允许以半乳糖作为输入来模拟 YES 和 NOT 门。在后一个电路中,AcrVA5与dLbCas12a一起表现出最佳的逻辑行为。
引言
2011年,研究人员提出了一种计算方法,并开发了相应的软件,用于数字合成基因电路的自动设计1。用户必须指定输入数量(三个或四个)并填写电路真值表;这提供了使用电子技术推导电路结构的所有必要信息。真值表通过 Karnaugh 映射方法2 转换为两个布尔公式。每个布尔公式都由描述电路输入(部分)之间的逻辑运算(和或乘法)及其否定(文字)的子句组成。子句依次相加(OR)或乘法(AND)以计算电路输出。每个电路都可以根据其两个相应的公式中的任何一个来实现:一个以POS(总和乘积)形式编写,另一个以SOP(产品总和)表示形式编写。前者由包含文字逻辑和的子句(即布尔门)的乘法组成。相比之下,后者是文字相乘的子句的总和。
电路可以在面包板上通过将不同的门物理连接在一起来实现。电流允许门之间交换信号,从而计算输出。
在生物学中,情况更为复杂。布尔门可以实现为转录单元(TU;即真核细胞内的序列"启动子编码区域终止子"),其中转录或翻译(或两者)受到调节。因此,至少有两种分子建立了生物线路:转录因子蛋白和非编码、反义RNA。
基因数字电路被组织成两层或三层门,即:1)输入层,由YES(缓冲器)和NOT门组成,并将输入化学物质转换为布线分子;2) 内层,它由与相应布尔公式中的子句一样多的 TU 组成。如果电路按照SOP公式设计,则内层中的每个子句都将在所谓的分布式输出架构中产生电路输出(例如荧光)。如果使用和积(POS)公式,则需要3)最后一层,它将包含一个从内层收集布线分子的乘法门。
总体而言,在合成生物学中,可以为同一电路设计许多不同的方案。它们在TU和连接分子的数量和种类上有所不同。为了选择在酵母细胞中实现的最简单解决方案,每个电路设计都与复杂性评分S相关联,定义为
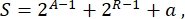
其中 A 代表激活剂的数量, R 代表阻遏剂的数量, a 是反义RNA分子的数量。如果电路中没有激活剂或抑制因子,则它们对S的贡献为零。因此,当需要大量正交转录因子时,在实验室中实现电路方案(高S)更加困难。这意味着新的活化器和抑制器应 重新 设计,以实现数字电路内部的完整布线。原则上,可以使用锌指蛋白3 和TAL效应子4 作为模板来组装新型DNA结合蛋白。但是,此选项似乎过于艰巨和耗时;因此,人们应该主要依靠小RNA和翻译调控来完成复杂的基因回路。
最初,这种方法是为了在细菌中制造数字电路而开发的。事实上,在真核细胞中,比起反义RNA,更适合谈论microRNA(miRNA)或小干扰RNA(siRNA)5。然而,RNAi途径不存在于 酿酒酵母中。因此,应该选择完全转录网络。假设一个电路需要五个激活器和五个抑制器;其复杂性分数为 S = 32。通过将10个转录因子替换为融合到激活域(AD)的单个dCas96 (核酸酶缺陷型Cas9),可以降低电路复杂性。如7所示,当在TATA盒和TSS(转录起始位点)之间结合启动子时,dCas9-AD在酵母中充当阻遏剂,当在TATA盒上游结合时,作为激活剂起作用。因此,可以用单个dCas9-AD融合蛋白和10个sgRNA(单个向导RNA)替换10个转录因子,总复杂性得分为S = 11。合成十种sgRNA既快速又容易,而如前所述,组装10种蛋白质需要更长、更复杂的工作。
或者,可以使用两种正交的dCas蛋白(例如dCas9和dCas12a):一种融合到AD,另一种裸露或与抑制结构域组合。复杂度分数只会增加一个单位 (S = 12)。因此,CRISPR-dCas系统是 酿酒酵母中构建非常复杂的基因数字电路的关键。
本文深入表征了酵母中基于dCas9和dCas12a的阻遏剂和激活剂的效率。结果表明,它们不需要大量的sgRNA来优化其活性,因此优先避免使用游离体质粒。此外,当使用募集VP64 AD拷贝的支架RNA(scRNA)时,基于dCas9的激活剂更有效。相比之下,dCas12a在直接融合到强VPR AD时效果很好。此外,根据激活剂的配置,合成活化启动子需要可变数量的靶位点(例如,使用 dCas12a-VPR 时为 3 个,dCas9-VP64 为 6 个,dCas9 和 scRNA 时只有一个)。作为阻遏因子,dCas12a在结合编码区而不是启动子时显得更敏锐。
然而,作为一个缺点,CRISPR-dCas9 / dCas12a不直接与化学物质相互作用。因此,它们在输入图层中可能没有用处。出于这个原因,已经研究了含有抗CRISPR蛋白(Acrs)的替代布尔门设计。Acrs作用于(d)Cas蛋白并抑制其工作8。因此,它们是调节CRISPR-(d)Cas系统活性的一种手段。本文深入分析了酿酒酵母II型Acrs与(d)Cas9以及V型Acrs和(d)Cas12a之间的相互作用。由于Acrs比Cas蛋白小得多,因此通过将人雌激素受体9-HBD(hER)的激素结合结构域融合到AcrIIA4,构建了对雌激素β-雌二醇有反应的NOT门。此外,在用半乳糖诱导时实现了少数表达dCas12a(-AD)的YES和NOT门和AcrVAs。目前,这些门仅用作概念证明。然而,它们也代表了深入重新思考算法的第一步,以执行酵母细胞中合成基因数字电路的计算自动设计。
研究方案
1. sgRNA/crRNA表达盒的设计与构建
注意:有两种sgRNA / crRNA表达盒:一种称为SNR5210-由RNA聚合酶III依赖性SNR52启动子,sgRNA / crRNA序列和SUP4终止子组成;另一个缩写为RGR11由RNA聚合酶II依赖性ADH1启动子,RGR(核酶引导RNA-核酶)结构组成,其中包含两个核酶(锤头核酶-HH和肝炎δ病毒核酶-HDV)和介于两者之间的sgRNA/crRNA序列,以及ADH1终止子。引导Cas9同系物的sgRNA由间隔序列和特征性直接重复12组成,而Cas12a蛋白的crRNA包括直接重复序列,然后是间隔序列13,14(本研究中使用的所有DNA序列见补充表1)。
- 设计用于Cas9 / Cas12a介导的转录激活的间隔序列。
- 利用细菌 lex 运算符序列(名为 lexOp)作为靶位点15,16 ,并将其插入驱动酵母增强绿色荧光蛋白 (yEGFP) 表达的 CYC1 启动子中17。因此,间隔序列由插入的 lexOp 定义并互补。
- 通过CRISPRDIRECT工具18检查间隔序列的正交性。
- 将 lexOp 序列粘贴到文本字段中,将 PAM 序列定义为 dCas9 的 NRG 和 dCas12a 的 TTTV,并从下拉列表中指定物种为 芽酵母(酿酒酵母)S288C 基因组。单击 "设计"。确保在 20mer+PAM 或 12mer+PAM 搜索中没有匹配的目标站点。
- 构建sgRNA/crRNA表达盒。
- 使用触地 PCR 扩增标准生物部分的 DNA 序列,如启动子、编码序列和终止子。
- 制备含有以下物质的反应混合物:20-40 ng DNA 模板、1 μL 10 μM 正向引物(即 ot25、sgRNA/crRNA 表达质粒构建)、1 μL 10 μM 反向引物(即 ot26、sgRNA/crRNA 表达质粒构建)、5 μL 2.5 mM dNTP 混合物、0.5 μL DNA 聚合酶、10 μL 5x DNA 聚合酶反应缓冲液、 和双蒸水 (ddH2O),总体积可达 50 μL。
注意:有关本研究中使用的引物列表,请参阅 补充表2 。 - 在热循环仪上运行触地PCR程序:
第 1 阶段:98 °C 持续 30 秒。
具有10个循环的第2阶段:98°C持续10秒,68°C持续20秒,72°C持续15秒。
具有25个循环的第3阶段:98°C持续10秒,59°C持续20秒,72°C持续15秒。
第 4 阶段:72 °C 2 分钟。
最后,保持在4°C直到开始后续实验。
注意:第2阶段的68°C和第3阶段的59°C取决于正向和反向引物的熔解温度,因不同的引物对而异。在第2阶段和第3阶段中,72°C下的时间由PCR产物的长度和DNA聚合酶的速度决定。
- 制备含有以下物质的反应混合物:20-40 ng DNA 模板、1 μL 10 μM 正向引物(即 ot25、sgRNA/crRNA 表达质粒构建)、1 μL 10 μM 反向引物(即 ot26、sgRNA/crRNA 表达质粒构建)、5 μL 2.5 mM dNTP 混合物、0.5 μL DNA 聚合酶、10 μL 5x DNA 聚合酶反应缓冲液、 和双蒸水 (ddH2O),总体积可达 50 μL。
- 通过凝胶电泳(100 V,30分钟) 分离 PCR产物。通过DNA凝胶提取试剂盒 从 琼脂糖凝胶中洗脱DNA序列(参见 材料表)。
注意:长度超过500 nt的片段需要0.8%琼脂糖凝胶,100 nt和500 nt之间的片段需要1.5%琼脂糖凝胶,短于100 nt的片段需要2%琼脂糖凝胶。 - 将表达 sgRNA/crRNA 的 TU 插入 pRSII404/424 穿梭载体19 中,该载体包含氨苄青霉素抗性基因和酵母选择性的营养不足标记基因-TRP1。
- 用两种限制性内切酶SacI和Acc65I在37°C下消化穿梭载体1小时。通过加入 5 μg 穿梭载体、推荐量的酶、消化缓冲液(根据酶说明)和 ddH2O 制备反应混合物,总体积可达 30 μL。
- 通过凝胶电泳 验证 梭式载体消解(参见子步骤1.3.2)。然后,在65°C下灭活两种酶20分钟。
- 使用Gibson组装方法20 通过将等摩尔DNA混合物在50°C下放入1小时,将纯化的PCR产物插入切开的穿梭载体中。
- 通过热冲击转化方法21,用上述吉布森反应混合物转化大肠杆菌DH5α感受态细胞。将转化的大肠杆菌细胞转移到含有氨苄西林(0.1g / L)的Luria-Bertani(LB)琼脂平板上。将板放入37°C的培养箱中,让细胞生长过夜。
- 从LB琼脂平板中挑选四个菌落,并在含有氨苄西林(0.1g / L)的LB溶液中分别在37°C下培养过夜。然后,使用微型制备程序从 大肠杆菌 细胞中提取质粒22。
- 使用引物ot18和ot19(参见寡核苷酸序列的补充表2)通过Sanger方法23对插入的转录单元进行测序和确认。
注意:在以后的实验中,构建和确认的质粒将通过PEG / LiAc协议24插入酵母基因组中。
- 使用触地 PCR 扩增标准生物部分的 DNA 序列,如启动子、编码序列和终止子。
2. 支架RNA表达盒的设计与构建
注意:支架引导RNA(scRNA)由sgRNA序列和MS2发夹结构25组成。本工作使用了两种MS2发夹结构:野生型MS2发夹-wt和f6 MS2外壳蛋白(MCP)适配体-f6。
- 合成能够适应不同间隔序列(即pSNR52-间隔_DR(SpCas9)-2×MS2(wt+f6)-SUP4t)的scRNA模板。
注意:在本研究中,scRNA模板由基因合成公司合成(见 材料表)。 - 设计适当的引物(见 补充表2)以对必要的间隔序列进行PCR。
- 按照步骤1.3中的步骤构建scRNA表达盒。
3. dSpCas9工程与表达质粒构建
- 获得质粒pTPGI_dSpCas9_VP64(见 材料表)。
- 基于pRSII406穿梭载体,通过触地PCR和Gibson组装方法 构建 受体载体pRSII406-pGPD-ATG-XbaI-XhoI-CYC1t(参见步骤1.3)。质粒提供强组成型启动子-pGPD和终止子-CYC1t。
- 在37°C下用XbaI和XhoI消化pTPGI_dCas9_VP64质粒和新构建的受体载体(5μg过夜1小时或10μg过夜 - 参见步骤1.3.3.1作为参考)。 分离并纯化插入片段和受体载体,如步骤1.3.2所示。
- 将纯化的插入片段片段和受体载体与T4 DNA连接酶在16°C下连接8小时。通过加入 50-100 ng 纯化的受体载体、等摩尔量的纯化靶片段、1.5 μL T4 缓冲液、0.5 μL T4 连接酶和 ddH2O 制备连接溶液,总体积高达 15 μL。
- 按照步骤 1.3.3.3 和 1.3.3.4 进行操作。然后,通过用XbaI和Xhol(37°C,1小时)和凝胶电泳消化(参见步骤1.3.2)确认新构建的质粒是正确的。
4. dCas12a工程与质粒构建
- 构建表达dCas12a-AD的质粒。
- 合成两种酵母密码子优化的dCas12a蛋白(denAsCas12a和dLbCas12a),两侧是BamHI和Xhol限制性内切酶位点。
注意:在这项研究中,两种酵母密码子优化的dCas12a蛋白由一家基因合成公司合成(见 材料表)。 - 通过触地PCR和Gibson组装方法 构建 受体载体pRSII406-启动子-ATG-NLS-GS-HIStag-GS-BamHI-sp-Xhoi-ad-nls-taa-mTGUO1(参见步骤1.3),其中"启动子"是pGPD或pGAL1,"sp"代表一个短的随机序列,"AD"是VPR或VP64。
- 通过BamHI和XhoI消化以及T4 DNA连接酶连接 ,将 每个dCas12a蛋白插入两个新构建的受体载体中(参见步骤3.3和3.4)。
- 合成两种酵母密码子优化的dCas12a蛋白(denAsCas12a和dLbCas12a),两侧是BamHI和Xhol限制性内切酶位点。
- 构建表达裸dCas12a的质粒。
- 使用触地PCR和Gibson组装方法构建dCas12a半乳糖诱导表达盒的受体载体pRSII406-Acc651-pGAL1-ATG-NLS-GS-HIStag-GS-BamHI-sp-XhoI-GS-NLS-TAA-CYC1t(参见步骤1.3)。
- 用BamHI和Xhol消化含有dCas12a蛋白和上述受体载体的质粒,然后用T4 DNA连接酶连接它们以获得质粒pRSII406-pGAL1-Acc651-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1t(参见步骤3.3和3.4)。
- 用Acc65I和BamHI消化步骤4.2.2中获得的质粒,然后通过触地PCR和Gibson组装方法 用 pGPD替换pGAL1,以构建pRSII406-pGPD-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1t。
5. 抗CRISPR蛋白工程与质粒构建
注意:已使用三种启动子来驱动Acrs表达:诱导启动子-pGAL1,四种酵母组成型启动子-pGPD,pACT1,pTEF1和pTEF2,以及合成组成型启动子-genCYC1t_pCYC1noTATA26。
- 从基因合成公司获得含有II型Acr(AcrIIA2,AcrIIA427和AcrIIA5 28)和V-A型Acr(AcrVA1,AcrVA4和AcrVA529)序列的质粒。
- 基于pRSII403穿梭载体构建质粒以表达Acrs。
- 构建 AcrIIA 表达盒。
注意:使用触地PCR扩增四种不同的启动子(即pGPD,pACT1,pTEF2和genCYC1t_pCYC1noTATA),三种AcrIIA和两种终止子(ADH1t和CYC1t)。通过Gibson组装方法 ,在不同的 启动子下构建一系列表达AcrIIA的TU(参见步骤1.3)。 - 构建 AcrVA 表达盒。
- 合成受体序列:ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6,其中"sp"是一个随机序列,稍后将被AcrVA取代。
注意:在本研究中,受体序列由基因合成公司合成(材料表)。 - 组装受体载体pRSII403-promoter-ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6,其中"启动子"是:pGAL1,pTEF1和genCYC1t_pCYC1noTATA。使用 Gibson 汇编方法(请参阅步骤 1.3)。
- 将三种AcrVA中的每一个插入步骤5.2.2.2中描述的受体载体中(通过 触地PCR和Gibson组装方法[见步骤1.3]),以构建一组产生AcrVA的质粒。
- 合成受体序列:ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6,其中"sp"是一个随机序列,稍后将被AcrVA取代。
- 构建 AcrIIA 表达盒。
- 通过用GS接头和HBD(hER)的序列扩展其序列来进一步设计AcrIIA4。这允许建立β雌二醇响应回路。
- 使用触地PCR分别获得GS-HBD和AcrIIA4部分(见步骤1.3.1)。
- 将AcrIIA4,GS-HBD和穿梭载体放入Gibson混合物中,并通过Gibson方法 构建 完整的质粒(参见步骤1.3.3)。
6. crRNA检测:RT-qPCR和引物设计
注意:crRNA检测是通过RT-qPCR 实现 的,RT-qPCR分为三个步骤。
- 通过RNA试剂盒 从 酵母细胞中提取和纯化RNA。
- 使用 24 孔板(240 rpm,30 °C)在 2 mL 合成定义的完全培养基(SDC、1 L 体积:20 g 葡萄糖、2 g AA 混合物、6.7 g YNB、396 mg 亮氨酸、79.2 mg 色氨酸、79.2 mg 组氨酸和 79.2 mg 尿嘧啶)中培养酵母细胞过夜。
- 早上,将细胞培养物(1:100)稀释到2mL新鲜SDC中,并在30°C,240rpm下继续生长酵母细胞4小时。
- 收获整个 2 mL 细胞溶液并以 20,238 x g 离心 2 分钟。小心地除去上清液,因为细胞沉淀很小。
- 使用 RNA 试剂盒从酵母细胞中提取 RNA。
- 检查RNA质量。
- 准备1%琼脂糖凝胶。将 5 μL 的每个 RNA 样品与 1 μL DNA 上样染料混合。然后将混合物加载到凝胶上并运行。
- 确保凝胶上存在~4,000 nt和~2,000 nt处的两个透明条带,对应于核糖体RNA (25S / 18S)。在 tRNA 的 ~80 nt 处可以看到进一步模糊的条带。
- 立即使用RNA样品进行cDNA合成或将其储存在-80°C以备将来使用。
- 逆转录:使用茎环方法30 形成对应于crRNA的第一链cDNA(40nt)。对于sgRNA的逆转录(近100 nt),该过程与参考基因 ACT1的cDNA合成程序相同。
注意:crRNA的逆转录方法与sgRNA和 ACT1 mRNA使用的方法不同。由于crRNA很短,因此将其视为microRNA,并使用microRNA逆转录方法(stem-loop方法)获得相应的cDNA。两种cDNA合成试剂盒(用于crRNA的茎环试剂盒和用于 ACT1 基因的常用逆转录试剂盒)用于crRNA定量。在两个试剂盒中使用相同量的RNA(参见 材料表),以使实验结果具有可比性。与茎环试剂盒一起使用的引物是根据茎环序列和crRNA3'末端的最后六个核苷酸设计的(有关茎环和引物序列,请参见 补充表2)。- crRNA逆转录的茎环法
- 从冰箱中取出RNA模板,引物和缓冲液,让它们在冰上融化。
- 基因组 DNA 去除:首先,根据试剂盒说明制备 10 μL 的反应混合物。将混合物置于42°C的热循环仪中2分钟。最后,将其转移到冰上。
- 第一条 cDNA 链的合成:通过加入步骤 6.2.1.2 中的 10 μL 混合物、1 μL 茎环引物(2 μM 浓度)、2 μL 10x RT 反应缓冲液、2 μL 逆转录酶混合物(含逆转录酶)和 5 μL 无 RNase H2O 制备 20 μL 反应混合物。
- 将反应混合物放入热循环仪中并运行以下程序:25°C5分钟,50°C15分钟,85°C5分钟。立即使用本品进行qPCR反应或将其储存在-80°C。
- sgRNA和 ACT1 mRNA逆转录
- 第一次反应:根据试剂盒说明制备由引物混合物、dNTP 混合物、RNA 模板 (50 pg-5 μg) 和无 RNase 的水(除 RNA 模板外,均由试剂盒提供)组成的 13 μL 混合物。使用 1 μg RNA 模板。将混合物放入70°C的热循环仪中10分钟。
- 第二次反应(cDNA合成):通过将试剂(如试剂盒说明中所述)添加到13μL的第一反应溶液中来制备反应混合物,直至最终体积为20μL。 将混合物置于50°C的热循环仪中15分钟,然后在85°C下再放置5分钟。 立即使用本品进行qPCR反应或将其储存在-80°C。
- crRNA逆转录的茎环法
- 用于 qPCR 的 SYBR 试剂盒:Ct 值检测
注意:crRNA的qPCR中使用的反向引物是固定的,因为它对应于茎环序列的反向补体(见 补充表2)。相反,正向引物是可变的,取决于crRNA的序列。sgRNA和 ACT1 mRNA qPCR的正向和反向引物设计为 https://www.ncbi.nlm.nih.gov/tools/primer-blast/。当两种引物的熔解温度差不大于2°C时,选择两种引物(见 补充表2)。每个样品分三次重复进行测量。- 根据制造商对SYBR试剂盒的说明制备qPCR反应混合物。
- 在实时荧光定量 PCR 机中设置以下 qPCR 程序。
预孵育:在95°C下孵育10分钟。
PCR阶段:在95°C下15秒,然后在55°C下34秒。 将PCR阶段的循环设置为45次。熔化阶段:95°C下10秒,65°C下60秒,97°C下1秒。 - 通过 Pfaffl 公式31 计算相对 mRNA 表达值。
7. 免疫荧光检测Cas蛋白
注意:Cas蛋白(CasP)融合到His_tag上。
- 酵母细胞制备
- 使用无菌环挑选一些酵母细胞,并在5mL富含YPD的培养基中以240rpm在30°C下培养过夜。 早上,将 500 μL 细胞溶液加入 20 mL 新鲜 YPD 中,并在 30 °C 下以 240 rpm 的速度生长,直到 OD600 达到 0.6。
- 取 5 mL 培养物,与 500 μL 37% 甲醛混合。让混合物在室温(RT)下保持10分钟。通过以1,500× g 离心5分钟来收获细胞。
- 除去上清液,用1mL固定缓冲液(0.1M KH 2 PO4,0.5M MgCl2,3.7%甲醛,pH = 6.5)重悬细胞。将细胞溶液在室温下保持20分钟。
- 将细胞溶液以1,500×g离心5分钟。弃去上清液并将细胞重悬于1 mL洗涤缓冲液(0.1 M KH 2 PO4,1.2M山梨糖醇,pH = 6.5)中,补充有4μLβ-巯基乙醇和4μL裂解液(5mg / mL)。将细胞溶液放入37°C的培养箱中20分钟。
- 将细胞溶液以1,500× g 离心5分钟并弃去上清液。通过离心(1,500 x g 5分钟)用1mL PBS洗涤细胞沉淀两次。
- 将细胞重悬于 100 μL PBS 加 0.05% 吐温 20 中,并加入 4 μL BSA 溶液 (10 mg/mL)。将细胞溶液在室温下保持20分钟。
- 与一抗一起孵育
- 将Anti-He标签一抗以1:400稀释度加入步骤7.1.6中的混合物中。将溶液在室温下保持2小时。
- 将步骤7.2.1中的混合物以1,500× g 离心5分钟并除去上清液。加入 1 mL PBST 并离心 (1,500 x g) 5 分钟以洗涤细胞沉淀。重复此操作两次。最后,弃去上清液并将细胞悬浮在 100 μL 的 1x PBST 中。
- 显微镜细胞检测
- 将细胞安装在载玻片上;从步骤7.2.2中取2μL细胞溶液,并将其放在载玻片上。用盖玻片盖住它。
- 在荧光显微镜下观察细胞。打开荧光光源、显微镜和计算机。记下荧光光源编号,打开电脑上的显微镜软件。
- 将载玻片放在显微镜载物台上。选择40倍物镜并在绿光(550nm)下观察细胞。移动粗焦旋钮,直到出现酵母细胞的轮廓。移动精细对焦旋钮以聚焦细胞。
- 使用显微镜软件检测细胞。关闭显微镜视野并切换到计算机屏幕。单击 实时,等待 3-5 秒,然后单击 捕获 以拍照。保存图片。
- 关闭电脑、显微镜和荧光光源。
8. 数据采集:FACS
注意:绿色荧光通过流式细胞术(即荧光激活细胞分选[FACS]测量) 检测 。通常,酵母细胞在30°C和240rpm下培养以运行FACS实验。然而,细胞可能需要一些预防措施,这取决于它们的遗传内容。含有 dCas12a-VPR 基因(由 GPD 组成型启动子控制)的细胞必须在SDC溶液中生长24小时。之后,将细胞在新鲜SDC中以1:100的比例稀释并在测量荧光强度之前再生长12小时。用 AcrIIA4-HBD(hER) 基因修饰的细胞也需要稀释。此外,OD600 需要控制。首先,允许细胞在SDC中生长过夜(超过14小时)。早上,测量OD600 。然后将培养物在SDC中稀释,提供不同浓度的β-雌二醇,低至OD600 = 0.1。在FACS实验之前,将细胞再生长7小时,使得OD600 达到0.8-1.0。表达dCas9-VP64或dCas12a-VP64的细胞在SDC中生长20-24小时,无需稀释,在FACS机器测量之前进一步生长。
- 在测量前 20 分钟打开 FACS 机器以预热激光。
- 制备样品(稀释):将 20 μL 细胞培养物与 300 μL ddH2O 混合。
- 在连接到 FACS 计算机的计算机上运行 FACS 软件并创建新实验。设置测量参数(即 FSC(SSC)-A/H/W 和直方图)。
- 根据样品的激发和发射波长选择滤光片。例如,这里的目标蛋白是yEGFP,其激发和发射波长分别为488nm和507nm。因此,选择 FITC 或 GFP 滤光片(激发波长:488 nm;发射波长:527/32 nm)。将采集单元号设置为 10,000。
- 通过测量荧光珠的强度来调节FITC滤波电压。确保两个连续实验之间磁珠强度的相对差异不超过5%。
- 用ddH2O清洗机器几秒钟,以去除任何可能的多余珠子。
- 测量样品荧光强度。单击 预览 并等待 3-5 秒以获得样品进样稳定性。最后,单击 获取。
- 在实验结束时再次测量珠子。电压是初始磁珠测量期间使用的电压(参见步骤8.4,438-441 V)。检查两个磁珠的测量值之间的相对差异是否超过5%。
- 将 FACS 数据导出为 FCS 文件。
- 使用 R Studio 软件分析 FCS 文件。
9. 数据分析
注意:在 R studio 中使用 Flowcore R Bioconductor 包32 。使用用R语言编写的脚本分析FCS文件。
- 打开 R 工作室并加载脚本 Bdverse 分析。R 来分析 FCS 文件。
- 设置实验名称(ename),FCS文件所在的目录(路径)
存储 (dir_d),以及创建结果文件的位置 (dir_r)。 - 设置荧光通道。例如,如果测量绿色荧光,select_ch = "GPA-A"。
- 设置已测量的样本数 (s_num)。
- 设置每个点图的尺寸(sxlim, sylim)。设置条形图和箱形图的 x 轴和 y 轴的最大长度(xlimit、 ylimit)。极限必须大于或等于s_num。
- 通过从相应的行中删除 # 来选择门控方法。
注意:morphGate是由脚本执行的自动门控方法(即,程序识别并选择细胞密度较大的点图区域)。polygonGate 和 RectangleGate 要求查看点图并定义多边形的顶点或包含大多数像元所在区域的矩形的边。 - 选择与所选门控方法对应的流 集对象门控。选择点阵图分辨率(res;应至少等于 256)。
- 取消注释 flt_low <- filter_low(sp) 以删除荧光为负的测量值。取消注释 flt_sp <- filter(flt_lw) 以删除由于其他实验引起的异常值。
- 按 源 并运行脚本。包含分析结果的所有文件都是在 dir_r中创建的。
结果
通过RNA聚合酶III型启动子表达sgRNA/crRNA
首先,这项工作解决了 图1A所示的转录激活电路(电路1)的工程设计。它包含三个基本组成部分:1)编码yEGFP的基因(报告基因),其之前是一系列不同的合成启动子,为dCas9 / dCas12a-AD提供靶位点;2)酵母密码子优化版本的dCas9或dCas12a融合到激活域(分别为VP64和VPR),并包含一个或两个核定位序列(NLS)。两种dCas蛋白均?...
讨论
该协议显示了合成基因数字电路可能的完整工作流程,遵循"设计-构建-测试-学习"(DBTL)生物工程周期,涉及干实验室和湿实验室实验。在这里,我们专注于CRISPR-Cas系统,主要是dSpCas9,denAsCas12a,dLbCas12a和相应的抗CRISPR蛋白,通过在 酿酒酵母 中设计和构建小转录网络。其中一些模仿布尔门,布尔门是数字电路的基本组件。这里描述的所有回路都允许我们描述酿 酒酵母中CRISPR相关和...
披露声明
作者声明没有竞争性经济利益。
致谢
我们要感谢TJU合成生物学实验室-SPST的所有学生的帮助,以及李智和张向阳在FACS实验中的帮助。
材料
| Name | Company | Catalog Number | Comments |
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | - | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | - | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | - | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | - | - | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
参考文献
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。