Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Цифровые схемы генов на основе систем CRISPR-Cas и белков анти-CRISPR
* Эти авторы внесли равный вклад
В этой статье
Резюме
Системы CRISPR-Cas и анти-CRISPR белки были интегрированы в схему булевых ворот у Saccharomyces cerevisiae. Новые небольшие логические схемы показали хорошую производительность и углубили понимание как факторов транскрипции на основе dCas9/dCas12a, так и свойств белков анти-CRISPR.
Аннотация
Логические вентили синтетических генов и цифровые схемы имеют широкий спектр применения, от медицинской диагностики до заботы об окружающей среде. Открытие систем CRISPR-Cas и их естественных ингибиторов — белков анти-CRISPR (Acrs) — предоставляет новый инструмент для разработки и реализации цифровых схем генов in vivo . Здесь мы описываем протокол, который следует идее цикла биологической инженерии «Проектирование-Строительство-Тестирование-Обучение» и использует dCas9/dCas12a вместе с соответствующими Acrs для создания небольших транскрипционных сетей, некоторые из которых ведут себя как логические ворота, в Saccharomyces cerevisiae. Эти результаты указывают на свойства dCas9/dCas12a в качестве факторов транскрипции. В частности, для достижения максимальной активации экспрессии генов dSpCas9 должен взаимодействовать с сконструированной каркасной РНК, которая собирает несколько копий домена активации VP64 (AD). Напротив, dCas12a должен быть сплавлен на конце C с сильным VP64-p65-Rta (VPR) AD. Кроме того, активность обоих белков Cas не усиливается за счет увеличения количества сгРНК/крРНК в клетке. В этой статье также объясняется, как построить логические вентили на основе взаимодействия CRISPR-dCas-Acr. Слитый гормон-связывающий домен AcrIIA4 рецептора эстрогена человека является ядром ворот NOT, реагирующих на β-эстрадиол, тогда как AcrVA, синтезированные индуцируемым промотором GAL1 , позволяют имитировать как YES, так и NOT gates с галактозой в качестве входа. В последних схемах AcrVA5 вместе с dLbCas12a показали наилучшее логическое поведение.
Введение
В 2011 году исследователи предложили вычислительный метод и разработали соответствующее программное обеспечение для автоматического проектирования цифровых синтетических генных схем1. Пользователь должен был указать количество входов (три или четыре) и заполнить таблицу истинности схемы; Это предоставило всю необходимую информацию для получения структуры схемы с использованием методов электроники. Таблица истинности была переведена в две логические формулы с помощью методакарты Карно 2. Каждая булева формула состоит из предложений, описывающих логические операции (сумма или умножение) между (частью) входов схемы и их отрицаний (литералов). Предложения, в свою очередь, либо суммируются (ИЛИ), либо умножаются (И) для вычисления выходного сигнала схемы. Каждая схема может быть реализована в соответствии с любой из двух соответствующих формул: одна записана в форме POS (произведение сумм), а другая - в представлении SOP (сумма произведений). Первый состоит из умножения предложений (т. е. булевых ворот), которые содержат логическую сумму литералов. Последний, напротив, представляет собой сумму предложений, в которых перемножаются литералы.
Электрические цепи могут быть реализованы на макетной плате путем физического соединения различных ворот вместе. Электрический ток позволяет обмениваться сигналами между вентилями, что приводит к вычислению выхода.
В биологии ситуация сложнее. Булевы ворота могут быть реализованы как транскрипционная единица (TU; т.е. последовательность «промотор-кодирующая область-терминатор» внутри эукариотических клеток), где транскрипция или трансляция (или и то, и другое) регулируются. Таким образом, по крайней мере, два вида молекул устанавливают биологическую связь: белки фактора транскрипции и некодирующие, антисмысловые РНК1.
Цифровая схема гена организована в два или три слоя вентилей, а именно: 1) входной слой, который состоит из затворов YES (буфер) и NOT и преобразует входные химические вещества в молекулы проводки; 2) внутренний слой, который состоит из стольких ТУ, сколько предложений в соответствующей булевой формуле. Если схема спроектирована в соответствии с формулой СОП, каждый пункт во внутреннем слое будет производить выходной сигнал схемы (например, флуоресценцию) в так называемой распределенной выходной архитектуре. Если используется формула произведения суммы (POS), то требуется 3) конечный слой, который будет содержать один мультипликативный затвор, собирающий молекулы проводки из внутреннего слоя.
В целом, в синтетической биологии для одной и той же схемы может быть разработано множество различных схем. Они различаются по количеству и виду как TU, так и молекул проводки. Чтобы выбрать самое простое решение, которое будет реализовано в дрожжевых клетках, каждая схема связана с оценкой сложности S, определяемой как
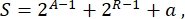
где A — количество активаторов, R — количество репрессоров, a — количество молекул антисмысловой РНК. Если в цепи отсутствуют активаторы или репрессоры, их вклад в S равен нулю. Следовательно, сложнее реализовать схему в лаборатории (высокий S), когда требуется большое количество ортогональных факторов транскрипции. Это означает, что новые активаторы и репрессоры должны быть спроектированы заново, чтобы реализовать полную проводку внутри цифровых цепей. В принципе, новые ДНК-связывающие белки могут быть собраны с использованием белков цинкового пальца3 и эффекторовTAL 4 в качестве шаблонов. Однако этот вариант представляется слишком трудным и трудоемким; поэтому следует полагаться в основном на малые РНК и регуляцию трансляции для завершения сложных генных цепей.
Первоначально этот метод был разработан для изготовления цифровых схем у бактерий. Действительно, в эукариотических клетках вместо антисмысловых РНК более уместно говорить о микроРНК (миРНК) или малых интерферирующих РНК (миРНК)5. Однако путь РНКи отсутствует у дрожжей S. cerevisiae. Следовательно, следует выбирать полностью транскрипционные сети. Предположим, что для цепи требуется пять активаторов и пять репрессоров; его оценка сложности будет S = 32. Сложность схемы может быть уменьшена путем замены 10 факторов транскрипции одним dCas96 (Cas9 с дефицитом нуклеазы), слитым с активационным доменом (AD). Как показано нарисунке 7, dCas9-AD работает как репрессор у дрожжей при связывании промотора между коробкой TATA и TSS (начальным сайтом транскрипции) и как активатор при связывании значительно выше по течению от коробки TATA. Таким образом, можно заменить 10 факторов транскрипции одним слитым белком dCas9-AD и 10 сгРНК (одиночными направляющими РНК) для общей оценки сложности S = 11. Синтезировать десять сгРНК можно быстро и легко, в то время как, как отмечалось ранее, сборка 10 белков потребовала бы гораздо более длительной и сложной работы.
В качестве альтернативы можно использовать два ортогональных белка dCas (например, dCas9 и dCas12a): один для слияния с AD, а другой голый или в сочетании с доменом репрессии. Оценка сложности увеличится всего на одну единицу (S = 12). Следовательно, системы CRISPR-dCas являются ключом к построению очень сложных цифровых схем генов у S. cerevisiae.
В данной работе подробно охарактеризована эффективность репрессоров и активаторов на основе dCas9 и dCas12a у дрожжей. Результаты показывают, что они не требуют большого количества сгРНК для оптимизации своей активности, поэтому эписомальные плазмиды предпочтительно избегают. Более того, активаторы на основе dCas9 гораздо более эффективны при использовании каркасной РНК (скРНК), которая рекрутирует копии VP64 AD. Напротив, dCas12a хорошо работает при непосредственном слиянии с сильным AD VPR. Кроме того, синтетический активированный промотор требует переменного числа целевых сайтов, в зависимости от конфигурации активатора (например, три при использовании dCas12a-VPR, шесть для dCas9-VP64 и только один с dCas9 и скРНК). В качестве репрессора dCas12a кажется более острым при связывании кодирующей области, а не промотора.
Однако недостатком является то, что CRISPR-dCas9/dCas12a не взаимодействуют с химическими веществами напрямую. Поэтому они могут быть бесполезны на входном уровне. По этой причине были исследованы альтернативные конструкции булевых затворов, содержащие белки анти-CRISPR (Acrs). Acrs действуют на (d)Cas белки и ингибируют их работу8. Следовательно, они являются средством модуляции активности систем CRISPR-(d)Cas. В данной работе подробно анализируются взаимодействия между типом II Acrs и (d)Cas9, а также типом V Acrs и (d)Cas12a у S. cerevisiae. Поскольку Acrs намного меньше, чем белки Cas, ворота NOT, реагирующие на эстроген β-эстрадиол, были построены путем слияния гормонсвязывающего домена рецептора эстрогеначеловека 9-HBD (hER) - с AcrIIA4. Кроме того, было реализовано несколько вентилей ДА и НЕ, которые конститутивно экспрессировали dCas12a(-AD) и AcrVA при индукции с галактозой. В настоящее время эти ворота служат только доказательством концепции. Тем не менее, они также представляют собой первый шаг к глубокому переосмыслению алгоритма для выполнения вычислительного автоматического проектирования цифровых схем синтетических генов в дрожжевых клетках.
протокол
1. Проектирование и конструирование кассеты экспрессии сгРНК/крРНК
ПРИМЕЧАНИЕ: Существует два типа кассет для экспрессии сгРНК/крРНК: одночленный SNR5210 - состоит из РНК-полимеразы III-зависимого промотора SNR52, последовательности сгРНК/крРНК и терминатора SUP4; другой, сокращенно RGR11, состоит из РНК-полимеразы II-зависимого промотора ADH1, структуры RGR (рибозим-направляющая РНК-рибозим), которая содержит два рибозима (молотоголовый рибозим-HH и рибозим-HDV вируса гепатита дельта) и последовательность sgRNA / crRNA между ними, а также терминатор ADH1. Гомологи Cas9, направляющие сгРНК, состоят из спейсерной последовательности и характерного прямого повтора12, тогда как крРНК для белков Cas12a содержит прямой повтор, за которым следует спейсерная последовательность13,14 (см. Дополнительную таблицу 1 для всех последовательностей ДНК, использованных в этом исследовании).
- Разработайте спейсерную последовательность для Cas9/Cas12a-опосредованной активации транскрипции.
- Используйте бактериальную последовательность оператора lex (называемую lexOp) в качестве целевого сайта 15,16 и вставьте ее в промотор CYC1, который управляет экспрессией зеленого флуоресцентного белка, усиленного дрожжами (yEGFP)17. Следовательно, последовательность спейсеров определяется вставленным lexOp и дополняет его.
- Проверьте ортогональность спейсерной последовательности с помощью инструментаCRISPRDIRECT 18.
- Вставьте последовательность lexOp в окружении последовательности PAM в текстовое поле, определите последовательность PAM как NRG для dCas9 и TTTV для dCas12a и укажите вид из выпадающего списка как геном S288C почковающихся дрожжей (Saccharomyces cerevisiae). Нажмите на Дизайн. Убедитесь, что ни в поиске 20mer+PAM, ни в поиске 12mer+PAM нет соответствующего целевого сайта.
- Постройте кассету экспрессии sgRNA/crRNA.
- Используйте ПЦР для амплификации последовательностей ДНК стандартных биологических частей, таких как промоторы, кодирующие последовательности и терминаторы.
- Приготовьте реакционную смесь, содержащую: 20-40 нг матрицы ДНК, 1 мкл прямого праймера 10 мкМ (т.е. ot25, плазмидную конструкцию экспрессии sgRNA/crRNA), 1 мкл обратного праймера 10 мкМ (т.е. ot26, плазмидную конструкцию экспрессии sgRNA/crRNA), 5 мкл смеси 2,5 мМ dNTP, 0,5 мкл ДНК-полимеразы, 10 мкл 5-кратного реакционного буфера ДНК-полимеразы, и двойная дистиллированная вода (ddH2O) общим объемом до 50 мкл.
ПРИМЕЧАНИЕ: См. Дополнительную таблицу 2 для получения списка праймеров, использованных в этом исследовании. - Запустите программу приземления ПЦР на термоциклере:
Стадия 1: 98 °C в течение 30 с.
Этап 2 с 10 циклами: 98 °C в течение 10 с, 68 °C в течение 20 с и 72 °C в течение 15 с.
Этап 3 с 25 циклами: 98 °C в течение 10 с, 59 °C в течение 20 с и 72 °C в течение 15 с.
Стадия 4: 72 °C в течение 2 мин.
Наконец, держите при температуре 4 ° C до начала последующих экспериментов.
ПРИМЕЧАНИЕ: 68 °C на стадии 2 и 59 °C на стадии 3 зависят от температур плавления как прямых, так и обратных грунтовок, варьирующихся от разных пар грунтовок. Время при 72 °C на стадиях 2 и 3 определяется длиной продукта ПЦР и скоростью ДНК-полимеразы.
- Приготовьте реакционную смесь, содержащую: 20-40 нг матрицы ДНК, 1 мкл прямого праймера 10 мкМ (т.е. ot25, плазмидную конструкцию экспрессии sgRNA/crRNA), 1 мкл обратного праймера 10 мкМ (т.е. ot26, плазмидную конструкцию экспрессии sgRNA/crRNA), 5 мкл смеси 2,5 мМ dNTP, 0,5 мкл ДНК-полимеразы, 10 мкл 5-кратного реакционного буфера ДНК-полимеразы, и двойная дистиллированная вода (ddH2O) общим объемом до 50 мкл.
- Изолируют продукты ПЦР с помощью гель-электрофореза (100 В, 30 мин). Выхолащивайте последовательности ДНК из агарозного геля с помощью набора для экстракции геля ДНК (см. Таблицу материалов).
ПРИМЕЧАНИЕ: 0,8% агарозный гель требуется для фрагментов длиннее 500 нт, 1,5% агарозный гель для фрагментов от 100 до 500 нт и 2% агарозный гель для фрагментов короче 100 нт. - Вставьте TU, экспрессирующий sgRNA/crRNA, в челночный вектор pRSII404/42419, который содержит ген резистентности к ампициллину и ген ауксотрофного маркера, выбираемый дрожжами-TRP1.
- Переваривают челночный вектор при 37 °C в течение 1 ч с двумя рестрикционными ферментами SacI и Acc65I. Приготовьте реакционную смесь, добавив 5 мкг челночного вектора, рекомендуемые количества ферментов, буфер для разложения (в соответствии с инструкциями по ферменту) иddH2Oдо общего объема 30 мкл.
- Проверьте челночное векторное расщепление с помощью гель-электрофореза (см. подэтап 1.3.2). Затем инактивируйте два фермента при 65 ° C в течение 20 минут.
- Используйте методсборки Гибсона 20 для вставки очищенных продуктов ПЦР в разрезанный челночный вектор, впуская эквимолярную смесь ДНК при 50 ° C в течение 1 часа.
- Трансформируют компетентные клетки Escherichia coli DH5α с помощью вышеуказанной реакционной смеси Гибсона с помощью методатрансформации теплового шока 21. Перенесите трансформированные клетки E. coli на агаровые пластины Лурия-Бертани (LB), содержащие ампициллин (0,1 г/л). Поместите пластины в инкубатор при температуре 37 ° C и дайте клеткам вырасти в течение ночи.
- Выберите четыре колонии из агаровой пластины LB и культивируйте их отдельно в течение ночи при 37 ° C в растворе LB, содержащем ампициллин (0,1 г / л). Затем используйте процедуру мини-подготовки для извлечения плазмид из клеток E. coli 22.
- Используйте праймеры ot18 и ot19 (см. Дополнительную таблицу 2 для олигопоследовательностей) для секвенирования и подтверждения вставленной транскрипционной единицы с помощью метода Сэнгера23.
ПРИМЕЧАНИЕ: В более поздних экспериментах сконструированные и подтвержденные плазмиды будут вставлены в геном дрожжей по протоколу PEG/LiAc24.
- Используйте ПЦР для амплификации последовательностей ДНК стандартных биологических частей, таких как промоторы, кодирующие последовательности и терминаторы.
2. Проектирование и конструирование каркасной кассеты для экспрессии РНК
ПРИМЕЧАНИЕ: Каркасная направляющая РНК (скРНК) состоит из последовательности сгРНК и шпилечных структурMS2 25. В этой работе используются два вида структур шпилек MS2: шпилька MS2 дикого типа и аптамер f6 белка оболочки MS2 (MCP) aptamer-f6.
- Синтезируйте шаблон скРНК, способный вмещать различные спейсерные последовательности (например, pSNR52-спейсер_DR(SpCas9)-2×MS2(wt+f6)-SUP4t).
ПРИМЕЧАНИЕ: В этом исследовании матрица скРНК была синтезирована компанией по синтезу генов (см. Таблицу материалов). - Разработайте подходящие праймеры (см. Дополнительную таблицу 2) для проведения ПЦР на необходимых спейсерных последовательностях.
- Следуйте процедурам, описанным на шаге 1.3, чтобы создать кассету экспрессии скРНК.
3. Инженерное и экспрессионное построение плазмиды dSpCas9
- Получают плазмиду pTPGI_dSpCas9_VP64 (см. Таблицу материалов).
- Постройте вектор-акцептор pRSII406-pGPD-ATG-XbaI-XhoI-CYC1t на основе челночного вектора pRSII406 с помощью ПЦР приземления и метода сборки Гибсона (см. шаг 1.3). Плазмида обеспечивает сильный конститутивный промотор - pGPD и терминатор - CYC1t.
- Переваривают плазмиду pTPGI_dCas9_VP64 и вновь сконструированный акцепторный вектор (5 мкг в течение 1 ч или 10 мкг в течение ночи - см. шаг 1.3.3.1 в качестве эталона) с помощью XbaI и XhoI при 37 °C. Разделите и очистите вставной фрагмент и вектор-акцептор, как показано на шаге 1.3.2.
- Очищенный фрагмент вставки и акцепторный вектор связывают с ДНК-лигазой Т4 при 16 °С в течение 8 ч. Готовят раствор лигирования, добавляя 50-100 нг очищенного акцепторного вектора, очищенные фрагменты-мишени в эквимолярном количестве с акцепторным вектором, 1,5 мкл буфера Т4, 0,5 мкл Т4-лигазы иddH2Oдо общего объема 15 мкл.
- Выполните шаги 1.3.3.3 и 1.3.3.4. Затем подтвердите правильность вновь сконструированной плазмиды путем разложения с помощью XbaI и Xhol (37 °C, 1 ч) и гель-электрофореза (см. этап 1.3.2).
4. Инженерия dCas12a и плазмидное строительство
- Построить плазмиды, экспрессирующие dCas12a-AD.
- Синтезируйте два дрожжевых кодон-оптимизированных белка dCas12a (denAsCas12a и dLbCas12a), окруженных сайтами рестрикционных ферментов BamHI и Xhol.
ПРИМЕЧАНИЕ: В этом исследовании два белка dCas12a, оптимизированные для кодонов дрожжей, были синтезированы компанией по синтезу генов (см. Таблицу материалов). - Постройте акцепторный вектор pRSII406-promoter-ATG-NLS-GS-HIStag-GS-BamHI-sp-XhoI-AD-NLS-TAA-mTGUO1 с помощью ПЦР с приземлением и метода сборки Гибсона (см. шаг 1.3), где «промотором» является либо pGPD, либо pGAL1, «sp» представляет собой короткую случайную последовательность, а «AD» — либо VPR, либо VP64.
- Вставьте каждый белок dCas12a в два вновь сконструированных акцепторных вектора путем разложения с помощью BamHI и XhoI и лигирования ДНК-лигазой T4 (см. шаги 3.3 и 3.4).
- Синтезируйте два дрожжевых кодон-оптимизированных белка dCas12a (denAsCas12a и dLbCas12a), окруженных сайтами рестрикционных ферментов BamHI и Xhol.
- Постройте плазмиды, экспрессирующие голый dCas12a.
- Постройте акцепторный вектор для кассет экспрессии, индуцируемой галактозой, dCas12a как pRSII406-Acc651-pGAL1-ATG-NLS-GS-HIStag-GS-BamHI-sp-XhoI-GS-NLS-TAA-CYC1t, используя ПЦР с приземлением и метод сборки Гибсона (см. шаг 1.3).
- Переваривают плазмиды, содержащие белки dCas12a и вышеуказанный акцепторный вектор, с помощью BamHI и Xhol, затем лигируют их ДНК-лигазой T4, чтобы получить плазмиду pRSII406-pGAL1-Acc651-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1t (см. шаги 3.3 и 3.4).
- Переварите плазмиду, полученную на этапе 4.2.2, с помощью Acc65I и BamHI, а затем замените pGAL1 на pGPD с помощью ПЦР с приземлением и метода сборки Гибсона для построения pRSII406-pGPD-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1t.
5. Анти-CRISPR-белковая инженерия и построение плазмиды
ПРИМЕЧАНИЕ: Для стимулирования экспрессии Acrs использовались три вида промоторов: индуцируемый промотор - pGAL1, четыре промотора - pGPD, pACT1, pTEF1 и pTEF2 и синтетический конститутивный промотор - genCYC1t_pCYC1noTATA26.
- Получить плазмиды, содержащие последовательности Acrs типа II (AcrIIA2, AcrIIA427 и AcrIIA5 28) и типа V-A Acrs (AcrVA1, AcrVA4 и AcrVA529), от компании по синтезу генов.
- Построение плазмид на основе челночного вектора pRSII403 для экспрессии Acrs.
- Создание кассет экспрессии AcrIIA.
ПРИМЕЧАНИЕ: Используйте тачдаун-ПЦР для амплификации четырех различных промоторов (т.е. pGPD, pACT1, pTEF2 и genCYC1t_pCYC1noTATA), трех видов AcrIIA и двух терминаторов (ADH1t и CYC1t). Постройте серию TU, экспрессирующих AcrIIA, под разными промоторами, с помощью метода сборки Гибсона (см. шаг 1.3). - Создание кассет экспрессии AcrVA.
- Синтезируем акцепторную последовательность: ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6, где «sp» — случайная последовательность, которая позже будет заменена на AcrVA.
ПРИМЕЧАНИЕ: В этом исследовании акцепторные последовательности были синтезированы компанией по синтезу генов (таблица материалов). - Соберите векторы-акцепторы pRSII403-promoter-ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6, где «промотором» является: pGAL1, pTEF1 и genCYC1t_pCYC1noTATA. Используйте метод сборки Gibson (см. шаг 1.3).
- Вставьте каждый из трех AcrVA в акцепторный вектор, описанный на шаге 5.2.2.2 (с помощью ПЦР-приземления и метода сборки Гибсона [см. этап 1.3]), чтобы построить набор плазмид, продуцирующих AcrVA.
- Синтезируем акцепторную последовательность: ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6, где «sp» — случайная последовательность, которая позже будет заменена на AcrVA.
- Создание кассет экспрессии AcrIIA.
- Далее инженер AcrIIA4, расширив его последовательность последовательностями линкера GS и HBD (hER). Это позволяет построить схему, реагирующую на β-эстрадиол.
- Используйте сенсорную ПЦР для получения деталей GS-HBD и AcrIIA4 по отдельности (см. этап 1.3.1).
- Поместите AcrIIA4, GS-HBD и челночный вектор в смесь Гибсона и сконструируйте полную плазмиду с помощью метода Гибсона (см. этап 1.3.3).
6. Обнаружение крРНК: ОТ-кПЦР и дизайн праймеров
ПРИМЕЧАНИЕ: обнаружение крРНК было достигнуто с помощью ОТ-кПЦР, которая организована в три этапа.
- Выполните экстракцию и очистку РНК из дрожжевых клеток с помощью набора РНК.
- Культивируют дрожжевые клетки в течение ночи в 2 мл синтетической определенной полной среды (SDC, объем 1 л: 20 г глюкозы, 2 г смеси АА, 6,7 г YNB, 396 мг лейцина, 79,2 мг триптофана, 79,2 мг гистидина и 79,2 мг урацила) с использованием 24-луночной пластины (240 об/мин, 30 °C).
- Утром разбавьте клеточную культуру (1:100) в 2 мл свежего SDC и продолжайте выращивать дрожжевые клетки при 30 ° C, 240 об/мин, еще 4 часа.
- Соберите целые 2 мл клеточного раствора и центрифугу в дозе 20,238 x g в течение 2 мин. Осторожно удалите надосадочную жидкость, так как клеточная гранула небольшая.
- Извлеките РНК из дрожжевых клеток с помощью набора РНК.
- Проверьте качество РНК.
- Приготовьте 1% агарозный гель. Смешайте 5 мкл каждого образца РНК с 1 мкл красителя, загружающего ДНК. Затем загрузите смесь на гель и запустите его.
- Убедитесь, что на геле присутствуют две прозрачные полосы с ~ 4,000 нт и ~2,000 нт, соответствующие рибосомной РНК (25S / 18S). Еще одна размытая полоса может быть видна на ~ 80 нт для тРНК.
- Немедленно используйте образцы РНК для синтеза кДНК или храните их при -80 ° C для использования в будущем.
- Обратная транскрипция: Используйте методстволовой петли 30 для формирования первой цепи кДНК, соответствующей крРНК (40 нт). Для обратной транскрипции сгРНК (почти 100 нт) процедура такая же, как и для синтеза кДНК эталонного гена ACT1.
ПРИМЕЧАНИЕ: Метод обратной транскрипции крРНК отличается от метода, используемого с sgРНК и мРНК ACT1 . Поскольку крРНК очень короткая, ее рассматривали как микроРНК и использовали метод обратной транскрипции микроРНК (подход стволовой петли) для получения соответствующей кДНК. Два набора для синтеза кДНК (набор стволовой петли для крРНК и обычный набор обратной транскрипции для гена ACT1 ) использовались для количественного определения крРНК. Одинаковое количество РНК было использовано в двух наборах (см. Таблицу материалов), чтобы сделать результаты эксперимента сопоставимыми. Праймер, используемый с набором стволовых петель, был разработан в соответствии с последовательностью стволовой петли и последними шестью нуклеотидами на 3'-конце крРНК (для стволовой петли и последовательности праймера см. Дополнительную таблицу 2).- Метод стволовой петли для обратной транскрипции крРНК
- Достаньте шаблон РНК, праймер и буферы из морозильной камеры и дайте им растаять на льду.
- Удаление геномной ДНК: Сначала приготовьте 10 мкл реакционной смеси в соответствии с инструкциями набора. Поместите смесь в термоциклер при температуре 42 °C на 2 мин. Наконец, перенесите его на лед.
- Синтез первой цепи кДНК: Приготовьте 20 мкл реакционной смеси, добавив 10 мкл смеси из этапа 6.2.1.2, 1 мкл праймера со стволовой петлей (концентрация 2 мкМ), 2 мкл 10-кратного реакционного буфера RT, 2 мкл смеси ферментов обратной транскрипции (содержащей обратную транскриптазу) и 5 мкл H2O, не содержащей РНКазы.
- Поместите реакционную смесь в термоциклер и запустите следующую программу: 25 °C в течение 5 мин, 50 °C в течение 15 мин и 85 °C в течение 5 мин. Немедленно используйте продукт для реакции кПЦР или храните его при температуре -80 °C.
- Обратная транскрипция мРНК sgRNA и ACT1
- Первая реакция: Приготовьте смесь объемом 13 мкл, состоящую из смеси праймера, смеси dNTP, матрицы РНК (50 пг-5 мкг) и воды, не содержащей РНКазы (кроме матрицы РНК, все они входят в комплект), в соответствии с инструкциями набора. Используйте 1 мкг матрицы РНК. Поместите смесь в термоциклер при температуре 70 °C на 10 минут.
- Вторая реакция (синтез кДНК): Приготовьте реакционную смесь, добавив реагенты (как описано в инструкциях к набору) в 13 мкл первого реакционного раствора до конечного объема 20 мкл. Поместите смесь в термоциклер на 15 мин при 50 °C, а затем еще на 5 мин при 85 °C. Немедленно используйте продукт для реакции кПЦР или храните его при температуре -80 °C.
- Метод стволовой петли для обратной транскрипции крРНК
- Набор SYBR для кПЦР: определение значения Ct
ПРИМЕЧАНИЕ: Обратный праймер, используемый в кПЦР крРНК, является фиксированным, поскольку он соответствует обратному дополнению последовательности стволовой петли (см. Дополнительную таблицу 2). Прямой праймер, напротив, вариабельен и зависит от последовательности крРНК. Прямой и обратный праймеры для кПЦР сгРНК и мРНК ACT1 разработаны на https://www.ncbi.nlm.nih.gov/tools/primer-blast/. Две грунтовки выбираются в том случае, если разница между их температурами плавления не превышает 2 °C (см. дополнительную таблицу 2). Каждый образец измеряется в трех повторениях.- Приготовьте реакционную смесь для кПЦР в соответствии с инструкциями производителя для набора SYBR.
- Установите следующую программу qPCR на ПЦР-аппарате в реальном времени.
Предварительная инкубация: 10 мин при 95 °C.
Стадия ПЦР: 15 с при 95 °C, затем 34 с при 55 °C. Установите цикл этапа ПЦР на 45 раз. Стадия плавления: 10 с при 95 °C, затем 60 с при 65 °C и, наконец, 1 с при 97 °C. - Рассчитайте относительные значения экспрессии мРНК с помощью формулыPfaffl 31.
7. Иммунофлюоресценция для обнаружения белков Cas
ПРИМЕЧАНИЕ: Белки Cas (CasP) сливаются с His_tag.
- Подготовка дрожжевых клеток
- Соберите несколько дрожжевых клеток с помощью стерильной петли и культивируйте их в 5 мл среды, богатой YPD, в течение ночи при 240 об/мин при 30 ° C. Утром добавьте 500 мкл клеточного раствора к 20 мл свежего YPD и выращивайте их при 240 об/мин при 30 ° C, пока OD600 не достигнет 0,6.
- Возьмите 5 мл культуры и смешайте ее с 500 мкл 37% формальдегида. Оставьте смесь при комнатной температуре (RT) на 10 минут. Собирают клетки центрифугированием при 1,500 x g в течение 5 мин.
- Удалить надосадочную жидкость и ресуспендировать клетки с помощью 1 мл фиксирующего буфера (0,1 МKH2PO4, 0,5 М MgCl2, 3,7% формальдегида, pH =6,5). Держите клеточный раствор при РТ в течение 20 мин.
- Центрифугируют клеточный раствор в дозе 1 500 x g в течение 5 мин. Выбросьте надосадочную жидкость и ресуспендируйте клетки в 1 мл промывочного буфера (0,1 М KH 2 PO 4,1,2М сорбита, pH = 6,5) с добавлением 4 мкл бета-меркаптоэтанола и4 мкл раствора лизата (5 мг / мл). Поместите клеточный раствор в инкубатор при температуре 37 °C на 20 мин.
- Центрифугируйте клеточный раствор при 1,500 x g в течение 5 мин и выбросьте надосадочную жидкость. Дважды промойте гранулы клетки 1 мл PBS центрифугированием (1 500 x г в течение 5 мин).
- Ресуспендируют клетки в 100 мкл PBS плюс 0,05% Tween 20 и добавляют 4 мкл раствора BSA (10 мг / мл). Держите клеточный раствор при РТ в течение 20 мин.
- Инкубация с первичным антителом
- Добавьте первичное антитело метки Anti-His в смесь на шаге 7.1.6 в разведении 1:400. Выдерживают раствор при РТ в течение 2 ч.
- Центрифугируют смесь на этапе 7.2.1 при 1 500 x g в течение 5 мин и удаляют надосадочную жидкость. Добавьте 1 мл PBST и центрифугу (1,500 x г) в течение 5 минут, чтобы промыть гранулы клетки. Повторите эту операцию дважды. Наконец, откажитесь от надосадочной жидкости и суспендируйте клетки в 100 мкл 1x PBST.
- Обнаружение клеток с помощью микроскопии
- Закрепите ячейки на слайде; возьмите 2 мкл клеточного раствора из шага 7.2.2 и поместите его на предметное стекло. Накройте его покровным стеклом.
- Наблюдайте за клетками под флуоресцентным микроскопом. Включите флуоресцентный источник света, микроскоп и компьютер. Запишите номер источника флуоресцентного света и откройте программное обеспечение микроскопа на компьютере.
- Положите предметное стекло на предметный столик микроскопа. Выберите объектив с 40-кратным увеличением и наблюдайте за ячейками под зеленым светом (550 нм). Перемещайте грубую ручку фокусировки до тех пор, пока не появится контур дрожжевых клеток. Переместите ручку точного фокуса, чтобы сфокусировать ячейки.
- Обнаружение клеток с помощью программного обеспечения микроскопа. Закройте поле зрения микроскопа и переключитесь на экран компьютера. Нажмите « Live», подождите 3-5 секунд и нажмите « Захват», чтобы сделать снимок. Сохраните рисунок.
- Выключите компьютер, микроскоп и флуоресцентный источник света.
8. Сбор данных: FACS
ПРИМЕЧАНИЕ: Зеленая флуоресценция обнаруживается с помощью проточной цитометрии (т.е. измерений сортировки клеток, активированных флуоресценцией [FACS]). Дрожжевые клетки культивируют, как правило, при 30 ° C и 240 об/мин для проведения экспериментов FACS. Тем не менее, клетки могут потребовать некоторых мер предосторожности в зависимости от их генетического содержания. Клетки, содержащие ген dCas12a-VPR (контролируемый конститутивным промотором GPD ), необходимо выращивать в течение 24 ч в растворе SDC. После этого клетки разбавляют в соотношении 1:100 в свежем SDC и выращивают еще 12 часов перед измерением интенсивности флуоресценции. Клетки, модифицированные геном AcrIIA4-HBD (hER), также требуют разведения. Кроме того, OD600 необходимо контролировать. Во-первых, клеткам дают расти в SDC в течение ночи (более 14 часов). Утром измеряется OD600 . Затем культуру разводят в SDC, снабжают разнообразной концентрацией β-эстрадиола, вплоть до OD600 = 0,1. Перед экспериментами FACS клетки выращивают еще 7 ч так, чтобы OD600 достигал 0,8-1,0. Клетки, экспрессирующие dCas9-VP64 или dCas12a-VP64, выращивают в SDC в течение 20-24 ч без разбавления и дальнейшего роста перед измерениями на машине FACS.
- Включите аппарат FACS за 20 минут до измерений, чтобы разогреть лазер.
- Подготовьте образцы (разведение): смешайте 20 мкл клеточной культуры с 300 мкл ddH2O.
- Запустите программное обеспечение FACS на компьютере, подключенном к компьютеру FACS, и создайте новый эксперимент. Установите параметры измерения (например, FSC(SSC)-A/H/W и гистограмму).
- Выберите фильтр в соответствии с длинами волн возбуждения и излучения образцов. Например, белком-мишенью здесь является yEGFP, длины волн возбуждения и излучения которого составляют 488 нм и 507 нм соответственно. Итак, выбираем фильтр FITC или GFP (длина волны возбуждения: 488 нм; длина волны излучения: 527/32 нм). Установите номер ячейки сбора данных на 10 000.
- Отрегулируйте напряжение фильтра FITC, измерив интенсивность флуоресцентных шариков. Следите за тем, чтобы относительная разница в интенсивности бусин между двумя последовательными экспериментами не превышала 5%.
- Вымойте машину с ddH2O в течение нескольких секунд, чтобы удалить все возможные лишние шарики.
- Измерьте интенсивность флуоресценции образца. Нажмите « Предварительный просмотр » и подождите 3-5 секунд для стабильности впрыска образца. Наконец, нажмите « Приобрести».
- Измерьте бусины еще раз в конце эксперимента. Напряжение - это то, которое использовалось при первоначальном измерении шариков (см. шаг 8.4, 438-441 В). Проверьте, не превышает ли относительная разница между размерами двух бусин 5%.
- Экспортируйте данные FACS в виде файлов FCS.
- Проанализируйте файлы FCS с помощью программного обеспечения R Studio.
9. Анализ данных
ПРИМЕЧАНИЕ: Используйте пакет Flowcore R Bioconductor 32 в студии R. Файлы FCS были проанализированы с помощью скрипта, написанного на языке R.
- Откройте R studio и загрузите скрипт Bdverse analysis. R для анализа файлов FCS.
- Задайте имя эксперимента (ename), каталог (путь), в котором находятся файлы FCS
Сохранено (dir_d) и где будут созданы файлы результатов (dir_r). - Установите флуоресцентный канал. Например, select_ch = «GPA-A», если измерялась зеленая флуоресценция.
- Установите количество измеряемых образцов (s_num).
- Задайте размеры каждого точечного графика (sxlim, sylim). Установите максимальную длину осей x и y для гистограмм и блочных диаграмм (xlimit, ylimit). xlimit должен быть больше или равен s_num.
- Выберите метод стробирования, удалив # из соответствующих строк.
ПРИМЕЧАНИЕ: morphGate - это автоматический метод стробирования, выполняемый скриптом (т.е. область точечных графиков, где ячейки плотнее, распознается и выбирается программой). polygonGate и rectangleGate требуют взглянуть на точечные графики и определить либо вершины многоугольника, либо сторону прямоугольника, охватывающую зону, где лежит большинство ячеек. - Выберите объект flowSet gateated, соответствующий выбранному методу стробирования. Выберите разрешение точечного графика (res; должно быть не менее 256).
- Раскомментируйте flt_low <-filter_low(sp), чтобы удалить измерения, в которых флуоресценция отрицательна. Раскомментируйте flt_sp <- filter(flt_lw), чтобы удалить выбросы, вызванные другими экспериментами.
- Нажмите Source и запустите сценарий. Все файлы, содержащие результаты анализа, создаются в dir_r.
Результаты
Экспрессия сгРНК/крРНК промотором РНК-полимеразы III типа
Во-первых, эта работа была посвящена проектированию схемы активации транскрипции (схема 1), показанной на рисунке 1A. Он содержал три основных компонента: 1) ген, кодирующий yEGFP (репортер), которому предшест...
Обсуждение
Протокол показал возможный полный рабочий процесс для цифровых схем синтетических генов в соответствии с циклом биологической инженерии «Проектирование-Строительство-Тестирование-Обучение» (DBTL) и в отношении экспериментов как в сухой, так и в мокрой лаборатории. Здесь мы сосредоточи...
Раскрытие информации
Авторы заявляют об отсутствии конкурирующих финансовых интересов.
Благодарности
Мы хотим поблагодарить всех студентов лаборатории синтетической биологии SPST, TJU за их общую помощь, а также Чжи Ли и Сянъян Чжан за помощь в экспериментах FACS.
Материалы
| Name | Company | Catalog Number | Comments |
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | - | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | - | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | - | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | - | - | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
Ссылки
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены