Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Circuits numériques géniques basés sur des systèmes CRISPR-CAS et des protéines anti-CRISPR
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Les systèmes CRISPR-Cas et les protéines anti-CRISPR ont été intégrés dans le schéma des portes booléennes chez Saccharomyces cerevisiae. Les nouveaux petits circuits logiques ont montré de bonnes performances et approfondi la compréhension des facteurs de transcription basés sur dCas9 / dCas12a et des propriétés des protéines anti-CRISPR.
Résumé
Les portes booléennes géniques synthétiques et les circuits numériques ont un large éventail d’applications, du diagnostic médical à la protection de l’environnement. La découverte des systèmes CRISPR-Cas et de leurs inhibiteurs naturels, les protéines anti-CRISPR (Acrs), fournit un nouvel outil pour concevoir et mettre en œuvre des circuits numériques géniques in vivo . Ici, nous décrivons un protocole qui suit l’idée du cycle de génie biologique « Design-Build-Test-Learn » et utilise dCas9 / dCas12a avec leurs Acrs correspondants pour établir de petits réseaux transcriptionnels, dont certains se comportent comme des portes booléennes, chez Saccharomyces cerevisiae. Ces résultats soulignent les propriétés de dCas9/dCas12a comme facteurs de transcription. En particulier, pour obtenir une activation maximale de l’expression génique, dSpCas9 doit interagir avec un ARN d’échafaudage modifié qui collecte plusieurs copies du domaine d’activation (AD) VP64. En revanche, dCas12a doit être fusionné, à l’extrémité C, avec le puissant VP64-p65-Rta (VPR) AD. De plus, l’activité des deux protéines Cas n’est pas améliorée par l’augmentation de la quantité d’ARNg / ARNcr dans la cellule. Cet article explique également comment construire des portes booléennes basées sur l’interaction CRISPR-dCas-Acr. Le domaine de liaison hormonale fusionné AcrIIA4 du récepteur humain des œstrogènes est le cœur d’une porte NOT sensible au β-estradiol, tandis que les AcrVAs synthétisés par le promoteur GAL1 inductible permettent d’imiter les portes YES et NOT avec le galactose comme entrée. Dans ces derniers circuits, AcrVA5, avec dLbCas12a, a montré le meilleur comportement logique.
Introduction
En 2011, des chercheurs ont proposé une méthode de calcul et développé un logiciel correspondant pour la conception automatique de circuits génétiques synthétiques numériques1. Un utilisateur devait spécifier le nombre d’entrées (trois ou quatre) et remplir la table de vérité du circuit; Cela a fourni toutes les informations nécessaires pour dériver la structure du circuit en utilisant des techniques de l’électronique. La table de vérité a été traduite en deux formules booléennes via la méthode2 de la carte de Karnaugh. Chaque formule booléenne est constituée de clauses qui décrivent des opérations logiques (somme ou multiplication) entre (partie de) les entrées du circuit et leurs négations (les littéraux). Les clauses, à leur tour, sont soit additionnées (OR) ou multipliées (ET) pour calculer la sortie du circuit. Chaque circuit peut être réalisé selon l’une de ses deux formules correspondantes : l’une écrite sous forme POS (produit des sommes) et l’autre en SOP (somme des produits). Le premier consiste en une multiplication de clauses (c’est-à-dire des portes booléennes) qui contiennent une somme logique des littéraux. Ce dernier, en revanche, est une somme de clauses où les littéraux sont multipliés.
Les circuits électriques peuvent être réalisés, sur une planche à pain, en câblant physiquement différentes portes ensemble. Le courant électrique permet l’échange de signaux entre les portes, ce qui conduit au calcul de la sortie.
En biologie, la situation est plus complexe. Une porte booléenne peut être réalisée comme une unité de transcription (TU; c’est-à-dire la séquence « promoteur-codant région-terminateur » à l’intérieur des cellules eucaryotes), où la transcription ou la traduction (ou les deux) sont régulées. Ainsi, au moins deux sortes de molécules établissent un câblage biologique : les protéines du facteur de transcription et les ARN antisens non codants1.
Un circuit numérique génétique est organisé en deux ou trois couches de portes, à savoir: 1) la couche d’entrée, qui est faite de portes YES (tampon) et NOT et convertit les produits chimiques d’entrée en molécules de câblage; 2) la couche interne, qui se compose d’autant de TU qu’il y a de clauses dans la formule booléenne correspondante. Si le circuit est conçu selon la formule SOP, chaque clause de la couche interne produira la sortie du circuit (par exemple, la fluorescence) dans une architecture dite de sortie distribuée. Si la formule du produit de somme (POS) est utilisée, alors une couche finale 3) est nécessaire, qui contiendra une seule porte multiplicative collectant les molécules de câblage de la couche interne.
Dans l’ensemble, en biologie synthétique, de nombreux schémas différents peuvent être conçus pour le même circuit. Ils diffèrent par le nombre et le type de TU et de molécules de câblage. Afin de choisir la solution la plus simple à mettre en œuvre dans les cellules de levure, chaque conception de circuit est associée à un score de complexité S, défini par
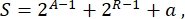
où A représente le nombre d’activateurs, R représente le nombre de répresseurs et a est la quantité de molécules d’ARN antisens. Si les activateurs ou les répresseurs sont absents du circuit, leur contribution à S est nulle. Par conséquent, il est plus difficile de réaliser un schéma de circuit en laboratoire (S élevé) lorsqu’il nécessite un nombre élevé de facteurs de transcription orthogonaux. Cela signifie que de nouveaux activateurs et répresseurs doivent être conçus de novo afin de réaliser le câblage complet à l’intérieur des circuits numériques. En principe, de nouvelles protéines de liaison à l’ADN peuvent être assemblées en utilisant les protéines Zinc Finger3 et les effecteurs TAL4 comme modèles. Cependant, cette option semble trop ardue et prend trop de temps; par conséquent, il faut s’appuyer principalement sur les petits ARN et la régulation de la traduction pour finaliser des circuits génétiques complexes.
À l’origine, cette méthode a été développée pour fabriquer des circuits numériques dans des bactéries. En effet, dans les cellules eucaryotes, au lieu d’ARN antisens, il est plus approprié de parler de microARN (miARN) ou de petits ARN interférents (siRNAs)5. Cependant, la voie ARNi n’est pas présente dans la levure S. cerevisiae. Par conséquent, il faut opter pour des réseaux entièrement transcriptionnels. Supposons qu’un circuit ait besoin de cinq activateurs et de cinq répresseurs; son score de complexité serait S = 32. La complexité du circuit peut être réduite en remplaçant les 10 facteurs de transcription par un seul dCas96 (Cas9 déficient en nucléase) fusionné à un domaine d’activation (AD). Comme le montrele point 7, dCas9-AD fonctionne comme un répresseur dans la levure lors de la liaison d’un promoteur entre la boîte TATA et le TSS (site de début de transcription) et comme activateur lors de la liaison bien en amont de la boîte TATA. Ainsi, on peut remplacer 10 facteurs de transcription par une seule protéine de fusion dCas9-AD et 10 sgRNA (ARN guides uniques) pour un score de complexité totale de S = 11. Il est rapide et facile de synthétiser dix ARNg, alors que, comme indiqué précédemment, l’assemblage de 10 protéines nécessiterait un travail beaucoup plus long et plus compliqué.
Alternativement, on peut utiliser deux protéines orthogonales dCas (par exemple, dCas9 et dCas12a): l’une pour fusionner avec une AD, et l’autre nue ou en combinaison avec un domaine de répression. Le score de complexité n’augmenterait que d’une unité (S = 12). Par conséquent, les systèmes CRISPR-dCas sont la clé de la construction de circuits numériques de gènes très complexes chez S. cerevisiae.
Cet article caractérise en profondeur l’efficacité des répresseurs et des activateurs à base de dCas9 et dCas12a chez la levure. Les résultats montrent qu’ils ne nécessitent pas une grande quantité d’ARNg pour optimiser leur activité, de sorte que les plasmides épisomiques sont préférentiellement évités. De plus, les activateurs basés sur dCas9 sont beaucoup plus efficaces lorsqu’ils utilisent un ARN d’échafaudage (scRNA) qui recrute des copies du VP64 AD. En revanche, dCas12a fonctionne bien lorsqu’il est fusionné directement avec le puissant VPR AD. De plus, un promoteur activé synthétique exige un nombre variable de sites cibles, en fonction de la configuration de l’activateur (par exemple, trois lors de l’utilisation de dCas12a-VPR, six pour dCas9-VP64 et un seul avec dCas9 et un scRNA). En tant que répresseur, dCas12a apparaît plus incisif lors de la liaison de la région de codage plutôt que du promoteur.
Cependant, CRISPR-dCas9 / dCas12a n’interagissent pas directement avec les produits chimiques. Par conséquent, ils peuvent ne pas être utiles dans la couche d’entrée. Pour cette raison, d’autres conceptions de portes booléennes contenant des protéines anti-CRISPR (Acrs) ont été étudiées. Les ACRS agissent sur les protéines (d)Cas et inhibent leur fonctionnement8. Par conséquent, ils sont un moyen de moduler l’activité des systèmes CRISPR-(d)Cas. Cet article analyse en profondeur les interactions entre les Acrs de type II et (d)Cas9, ainsi que les Acrs de type V et (d)Cas12a chez S. cerevisiae. Étant donné que les Acr sont beaucoup plus petits que les protéines Cas, une porte NOT sensible à l’œstrogène β-estradiol a été construite en fusionnant le domaine de liaison hormonale du récepteur humaindes œstrogènes 9-HBD(hER)-à AcrIIA4. En outre, une poignée de portes YES et NOT qui exprimaient dCas12a (-AD) de manière constitutive et AcrVAs lors de l’induction avec du galactose ont été réalisées. À l’heure actuelle, ces portes ne servent que de preuve de concept. Cependant, ils représentent également la première étape vers une refonte profonde de l’algorithme pour réaliser la conception automatique computationnelle de circuits numériques de gènes synthétiques dans les cellules de levure.
Protocole
1. Conception et construction de la cassette d’expression sgRNA/ARNcr
NOTE: Il existe deux types de cassettes d’expression sgRNA/ARNcr : SNR5210-est composé du promoteur SNR52 dépendant de l’ARN polymérase III, de la séquence sgRNA/ARNcr et du terminateur SUP4 ; un autre, abrégé en RGR11, se compose du promoteur ADH1 dépendant de l’ARN polymérase II, de la structure RGR (ribozyme-guide RNA-ribozyme) qui contient deux ribozymes (un ribozyme-HH à tête de marteau et un ribozyme-HDV du virus de l’hépatite delta) et la séquence de l’ARNg / ARNcr entre les deux, et le terminateur ADH1. Les homologues d’ARNg guidant Cas9 sont constitués d’une séquence d’espacement et de la répétition directe caractéristique12, tandis que l’ARNcr pour les protéines Cas12a comprend la répétition directe suivie de la séquence d’espacement13,14 (voir le tableau supplémentaire 1 pour toutes les séquences d’ADN utilisées dans cette étude).
- Concevoir la séquence d’espacement pour l’activation transcriptionnelle médiée par Cas9/Cas12a.
- Exploitez la séquence bactérienne de l’opérateur lex (appelée lexOp) pour en faire le site cible 15,16 et insérez-la dans le promoteur CYC1 qui entraîne l’expression de la protéine fluorescente verte améliorée par la levure (yEGFP)17. Par conséquent, la séquence d’espacement est définie par et complémentaire du lexOp inséré.
- Vérifiez l’orthogonalité de la séquence d’espacement via l’outil CRISPRDIRECT18.
- Collez la séquence lexOp flanquée de la séquence PAM dans le champ de texte, définissez la séquence PAM comme NRG pour dCas9 et TTTV pour dCas12a, et spécifiez les espèces de la liste déroulante comme Levure bourgeonnante (Saccharomyces cerevisiae) S288C Genome. Cliquez sur Conception. Assurez-vous qu’il n’y a pas de site cible correspondant dans 20mer + PAM ni dans la recherche 12mer + PAM.
- Construisez la cassette d’expression sgRNA/ARNcr.
- Utilisez la PCR d’atterrissage pour amplifier les séquences d’ADN de parties biologiques standard, telles que les promoteurs, les séquences codantes et les terminateurs.
- Préparer un mélange réactionnel contenant : 20 à 40 ng de matrice d’ADN, 1 μL d’amorce directe de 10 μM (c.-à-d. ot25, construction plasmidique d’expression sgRNA/ARNcr), 1 μL d’amorce inverse de 10 μM (c.-à-d. ot26, construction plasmidique d’expression sgRNA/ARNcr), 5 μL de mélange de dNTP 2,5 mM, 0,5 μL d’ADN polymérase, 10 μL de tampon réactionnel ADN polymérase 5x, et double eau distillée (ddH2O) jusqu’à un volume total de 50 μL.
REMARQUE : Veuillez consulter le tableau supplémentaire 2 pour obtenir la liste des amorces utilisées dans la présente étude. - Exécutez le programme de PCR d’atterrissage sur un thermocycleur :
Étape 1: 98 °C pendant 30 s.
Étape 2 avec 10 cycles : 98 °C pendant 10 s, 68 °C pendant 20 s et 72 °C pendant 15 s.
Étape 3 avec 25 cycles : 98 °C pendant 10 s, 59 °C pendant 20 s et 72 °C pendant 15 s.
Étape 4 : 72 °C pendant 2 min.
Enfin, maintenir à 4 °C jusqu’au début des expériences de suivi.
REMARQUE : Les 68 °C à l’étape 2 et les 59 °C à l’étape 3 dépendent des températures de fusion des amorces avant et inverse, variant d’une paire d’amorces différentes. Le temps à 72 °C dans les stades 2 et 3 est déterminé par la longueur du produit PCR et la vitesse de l’ADN polymérase.
- Préparer un mélange réactionnel contenant : 20 à 40 ng de matrice d’ADN, 1 μL d’amorce directe de 10 μM (c.-à-d. ot25, construction plasmidique d’expression sgRNA/ARNcr), 1 μL d’amorce inverse de 10 μM (c.-à-d. ot26, construction plasmidique d’expression sgRNA/ARNcr), 5 μL de mélange de dNTP 2,5 mM, 0,5 μL d’ADN polymérase, 10 μL de tampon réactionnel ADN polymérase 5x, et double eau distillée (ddH2O) jusqu’à un volume total de 50 μL.
- Isoler les produits PCR par électrophorèse sur gel (100 V, 30 min). Éluer les séquences d’ADN du gel d’agarose à l’aide d’un kit d’extraction de gel d’ADN (voir Tableau des matériaux).
REMARQUE : Un gel d’agarose à 0,8 % est requis pour les fragments de plus de 500 nt, un gel d’agarose à 1,5 % pour les fragments entre 100 nt et 500 nt, et un gel d’agarose à 2 % pour les fragments de moins de 100 nt. - Insérez le TU exprimant l’ARNg/ARNcr dans un vecteur navette19 pRSII404/424, qui contient le gène de résistance à l’ampicilline et le gène marqueur auxotrophe sélectionnable par levure-TRP1.
- Digérer le vecteur navette à 37 °C pendant 1 h avec les deux enzymes de restriction SacI et Acc65I. Préparer le mélange réactionnel en ajoutant 5 μg du vecteur navette, les quantités recommandées d’enzymes, un tampon de digestion (selon les instructions enzymatiques) et duddH2Ojusqu’à un volume total de 30 μL.
- Vérifier la digestion du vecteur navette par électrophorèse sur gel (voir sous-étape 1.3.2). Ensuite, inactivez les deux enzymes à 65 °C pendant 20 min.
- Utiliser la méthode d’assemblageGibson 20 pour insérer les produits PCR purifiés dans le vecteur navette coupé-ouvert en laissant entrer le mélange d’ADN équimolaire à 50 °C pendant 1 h.
- Transformer les cellules compétentes d’Escherichia coli DH5α avec le mélange réactionnel Gibson ci-dessus par la méthode de transformation par choc thermique21. Transférer les cellules E . coli transformées sur des plaques de gélose Luria-Bertani (LB) contenant de l’ampicilline (0,1 g/L). Mettez les plaques dans l’incubateur à 37 °C et laissez les cellules se développer pendant la nuit.
- Prélever quatre colonies dans la plaque de gélose LB et les cultiver séparément pendant la nuit à 37 °C dans une solution LB contenant de l’ampicilline (0,1 g/L). Ensuite, utilisez la procédure de mini-préparation pour extraire les plasmides des cellules E. coli22.
- Utilisez les amorces ot18 et ot19 (voir le tableau supplémentaire 2 pour les séquences oligo) pour séquencer et confirmer l’unité de transcription insérée via la méthode de Sanger23.
REMARQUE: Dans des expériences ultérieures, les plasmides construits et confirmés seront insérés dans le génome de la levure via le protocole PEG / LiAc24.
- Utilisez la PCR d’atterrissage pour amplifier les séquences d’ADN de parties biologiques standard, telles que les promoteurs, les séquences codantes et les terminateurs.
2. Conception et construction de la cassette d’expression de l’ARN de l’échafaudage
NOTE: L’ARN guide de l’échafaudage (scRNA) est composé de la séquence sgRNA et des structures en épingle à cheveux MS225. Deux types de structures en épingle à cheveux MS2 sont utilisés dans ce travail : l’épingle à cheveux MS2 de type sauvage et l’aptamère f6 MS2 coat protein (MCP) f6.
- Synthétiser un modèle scRNA capable de s’adapter à différentes séquences d’espacement (c.-à-d. espaceur pSNR52_DR(SpCas9)-2×MS2(wt+f6)-SUP4t).
REMARQUE : Dans cette étude, le modèle d’ARNsc a été synthétisé par une société de synthèse génique (voir le tableau des matériaux). - Concevoir des amorces appropriées (voir le tableau supplémentaire 2) pour exécuter la PCR sur les séquences d’espacement nécessaires.
- Suivez les procédures de l’étape 1.3 pour construire une cassette d’expression scRNA.
3. Ingénierie dSpCas9 et construction plasmidique d’expression
- Procurez-vous le plasmide pTPGI_dSpCas9_VP64 (voir Tableau des matériaux).
- Construire le vecteur accepteur pRSII406-pGPD-ATG-XbaI-XhoI-CYC1t, basé sur le vecteur navette pRSII406, via la PCR tactile et la méthode d’assemblage Gibson (voir étape 1.3). Le plasmide fournit un puissant promoteur constitutif-pGPD, et un terminateur-CYC1t.
- Digérer le plasmide pTPGI_dCas9_VP64 et le vecteur accepteur nouvellement construit (5 μg pendant 1 h ou 10 μg pendant la nuit - voir l’étape 1.3.3.1 comme référence) avec XbaI et XhoI à 37 °C. Séparer et purifier le fragment d’insertion et le vecteur accepteur comme indiqué à l’étape 1.3.2.
- Litruer le fragment d’insert purifié et le vecteur accepteur avec l’ADN ligase T4 à 16 °C pendant 8 h. Préparer la solution de ligature en ajoutant 50-100 ng du vecteur accepteur purifié, des fragments cibles purifiés en quantité équimolaire avec le vecteur accepteur, 1,5 μL de tampon T4, 0,5 μL de T4 ligase etddH2Ojusqu’à un volume total de 15 μL.
- Suivez les étapes 1.3.3.3 et 1.3.3.4. Ensuite, confirmez que le plasmide nouvellement construit est correct par digestion avec XbaI et Xhol (37 °C, 1 h) et électrophorèse sur gel (voir étape 1.3.2).
4. dCas12a ingénierie et construction plasmidique
- Construire les plasmides exprimant dCas12a-AD.
- Synthétiser deux protéines dCas12a optimisées pour les codons de levure (denAsCas12a et dLbCas12a) flanquées de sites enzymatiques de restriction BamHI et Xhol.
REMARQUE : Dans cette étude, les deux protéines dCas12a optimisées pour les codons de levure ont été synthétisées par une société de synthèse génique (voir le tableau des matériaux). - Construisez le vecteur accepteur pRSII406-promoter-ATG-NLS-GS-HIStag-GS-BamHI-sp-XhoI-AD-NLS-TAA-mTGUO1 via la PCR tactile et la méthode d’assemblage de Gibson (voir étape 1.3), où le « promoteur » est pGPD ou pGAL1, « sp » représente une courte séquence aléatoire et « AD » est VPR ou VP64.
- Insérez chaque protéine dCas12a dans les deux vecteurs accepteurs nouvellement construits par digestion avec BamHI et XhoI et ligature avec T4 DNA ligase (voir étapes 3.3 et 3.4).
- Synthétiser deux protéines dCas12a optimisées pour les codons de levure (denAsCas12a et dLbCas12a) flanquées de sites enzymatiques de restriction BamHI et Xhol.
- Construire les plasmides exprimant un dCas12a nu.
- Construire un vecteur accepteur pour les cassettes d’expression inductibles par le galactose de dCas12a en tant que pRSII406-Acc651-pGAL1-ATG-NLS-GS-HIStag-GS-BamHI-sp-XhoI-GS-NLS-TAA-CYC1t en utilisant la PCR tactile et la méthode d’assemblage Gibson (voir étape 1.3).
- Digérer les plasmides contenant les protéines dCas12a et le vecteur accepteur ci-dessus avec BamHI et Xhol, puis les ligaturer avec l’ADN ligase T4 pour obtenir le plasmide pRSII406-pGAL1-Acc651-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1t (voir les étapes 3.3 et 3.4).
- Digérer le plasmide obtenu à l’étape 4.2.2 avec Acc65I et BamHI, puis remplacer pGAL1 par pGPD via la PCR tactile et la méthode d’assemblage Gibson pour construire pRSII406-pGPD-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1t.
5. Ingénierie des protéines anti-CRISPR et construction plasmidique
REMARQUE: Trois types de promoteurs ont été utilisés pour stimuler l’expression Acrs: un promoteur inductible-pGAL1, quatre promoteurs constitutifs de levure-pGPD, pACT1, pTEF1 et pTEF2, et un promoteur constitutif synthétique-genCYC1t_pCYC1noTATA26.
- Obtenir les plasmides contenant les séquences des Acrs de type II (AcrIIA2, AcrIIA427 et AcrIIA5 28) et des Acrs de type V-A (AcrVA1, AcrVA4 et AcrVA529) auprès d’une société de synthèse de gènes.
- Construisez les plasmides en fonction du vecteur navette pRSII403 pour exprimer Acrs.
- Construire des cassettes d’expression AcrIIA.
REMARQUE: Utilisez la PCR d’atterrissage pour amplifier quatre promoteurs différents (c’est-à-dire pGPD, pACT1, pTEF2 et genCYC1t_pCYC1noTATA), les trois types d’AcrIIA et deux terminateurs (ADH1t et CYC1t). Construire une série d’UT exprimant des AcrIIA, sous différents promoteurs, via la méthode d’assemblage Gibson (voir étape 1.3). - Construire des cassettes d’expression AcrVA.
- Synthétisez la séquence acceptrice : ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6, où « sp » est une séquence aléatoire qui sera remplacée par AcrVAs plus tard.
NOTE: Dans cette étude, les séquences acceptrices ont été synthétisées par une société de synthèse de gènes (Table of Materials). - Assemblez les vecteurs accepteurs pRSII403-promoter-ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6, où le « promoteur » est : pGAL1, pTEF1 et genCYC1t_pCYC1noTATA. Utilisez la méthode d’assemblage Gibson (reportez-vous à l’étape 1.3).
- Insérer chacune des trois AcrVA dans le vecteur accepteur décrit à l’étape 5.2.2.2 (via la PCR au toucher des roues et la méthode d’assemblage de Gibson [voir étape 1.3]) pour construire un ensemble de plasmides produisant des AcrVA.
- Synthétisez la séquence acceptrice : ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6, où « sp » est une séquence aléatoire qui sera remplacée par AcrVAs plus tard.
- Construire des cassettes d’expression AcrIIA.
- Poursuite de l’ingénierie AcrIIA4 en étendant sa séquence avec les séquences du linker GS et HBD(hER). Cela permet la construction d’un circuit sensible au β-estradiol.
- Utilisez la PCR tactile pour obtenir séparément les pièces GS-HBD et AcrIIA4 (voir étape 1.3.1).
- Introduire AcrIIA4, GS-HBD et le vecteur navette dans le mélange de Gibson et construire le plasmide complet par la méthode de Gibson (voir étape 1.3.3).
6. Détection de l’ARNcr : RT-qPCR et conception des amorces
REMARQUE: La détection de l’ARNcr a été réalisée via RT-qPCR, qui est organisée en trois étapes.
- Effectuer l’extraction et la purification de l’ARN des cellules de levure via un kit d’ARN.
- Culture de cellules de levure pendant une nuit dans 2 mL de milieu complet défini synthétique (SDC, volume de 1 L : 20 g de glucose, 2 g de mélange AA, 6,7 g de YNB, 396 mg de leucine, 79,2 mg de tryptophane, 79,2 mg d’histidine et 79,2 mg d’uracile) en utilisant une plaque de 24 puits (240 rpm, 30 °C).
- Le matin, diluez la culture cellulaire (1:100) dans 2 ml de SDC frais et continuez à faire pousser les cellules de levure à 30 °C, 240 rpm, pendant encore 4 h.
- Prélever la totalité des 2 mL de la solution cellulaire et centrifuger à 20 238 x g pendant 2 min. Retirez le surnageant avec précaution car la pastille de la cellule est petite.
- Extraire l’ARN des cellules de levure à l’aide d’un kit d’ARN.
- Vérifiez la qualité de l’ARN.
- Préparez un gel d’agarose à 1%. Mélanger 5 μL de chaque échantillon d’ARN avec 1 μL de colorant de chargement d’ADN. Ensuite, chargez le mélange sur le gel et exécutez-le.
- Assurez-vous que deux bandes claires à ~4 000 nt et ~2 000 nt, correspondant à l’ARN ribosomique (25S/18S), sont présentes sur le gel. Une autre bande floue peut être vue à ~80 nt pour l’ARNt.
- Utilisez immédiatement les échantillons d’ARN pour la synthèse de l’ADNc ou stockez-les à -80 °C pour une utilisation ultérieure.
- Transcription inverse: Utilisez la méthode de la boucle souche30 pour former le premier brin d’ADNc correspondant à l’ARNcr (40 nt). Pour la transcription inverse de l’ARNg (près de 100 nt), la procédure est la même que pour la synthèse de l’ADNc du gène de référence ACT1.
NOTE: La méthode de transcription inverse de l’ARNcr est différente de celle utilisée avec l’ARNg et l’ARNm ACT1 . Comme l’ARNcr est très court, il a été traité comme un microARN et a utilisé la méthode de transcription inverse du microARN (approche de la boucle tige) pour obtenir l’ADNc correspondant. Deux kits de synthèse d’ADNc (un kit de boucle souche pour l’ARNcr et un kit de transcription inverse habituel pour le gène ACT1 ) ont été utilisés dans la quantification de l’ARNcr. La même quantité d’ARN a été utilisée dans les deux trousses (voir le tableau des matériaux) pour rendre les résultats de l’expérience comparables. L’amorce utilisée avec le kit tige-boucle a été conçue en fonction de la séquence tige-boucle et des six derniers nucléotides à l’extrémité 3' de l’ARNcr (pour la séquence de la boucle tige et de l’amorce, voir le tableau supplémentaire 2).- Méthode de boucle souche pour la transcription inverse de l’ARNcr
- Sortez le gabarit d’ARN, l’apprêt et les tampons du congélateur et laissez-les fondre sur la glace.
- Élimination de l’ADN génomique: Tout d’abord, préparez 10 μL du mélange réactionnel selon les instructions du kit. Mettre le mélange dans un thermocycleur à 42 °C pendant 2 min. Enfin, transférez-le sur la glace.
- Synthèse du premier brin d’ADNc : Préparer 20 μL du mélange réactionnel en ajoutant 10 μL du mélange de l’étape 6.2.1.2, 1 μL d’amorce tige-boucle (concentration de 2 μM), 2 μL de tampon réactionnel RT 10x, 2 μL de mélange d’enzymes de transcription inverse (contenant la transcriptase inverse) et 5 μL deH2Osans RNase.
- Placer le mélange réactionnel dans un thermocycleur et exécuter le programme suivant : 25 °C pendant 5 min, 50 °C pendant 15 min et 85 °C pendant 5 min. Utilisez immédiatement le produit pour la réaction qPCR ou conservez-le à -80 °C.
- Transcription inverse de l’ARNg et de l’ARNm ACT1
- Première réaction : Préparez un mélange de 13 μL composé du mélange d’amorce, du mélange de dNTP, de la matrice d’ARN (50 pg-5 μg) et de l’eau sans RNase (à l’exception de la matrice d’ARN, tous fournis par le kit), conformément aux instructions du kit. Utilisez 1 μg de matrice d’ARN. Mettre le mélange dans un thermocycleur à 70 °C pendant 10 min.
- Deuxième réaction (synthèse de l’ADNc): Préparer le mélange réactionnel en ajoutant les réactifs (comme décrit dans les instructions du kit) aux 13 μL de la première solution réactionnelle jusqu’à un volume final de 20 μL. Placer le mélange dans un thermocycleur pendant 15 min à 50 °C, puis pendant 5 minutes supplémentaires à 85 °C. Utilisez immédiatement le produit pour la réaction qPCR ou conservez-le à -80 °C.
- Méthode de boucle souche pour la transcription inverse de l’ARNcr
- Kit SYBR pour qPCR : détection de la valeur Ct
NOTE: L’amorce inverse utilisée dans la qPCR de l’ARNcr est fixe car elle correspond au complément inverse de la séquence tige-boucle (voir tableau supplémentaire 2). L’amorce avant, en revanche, est variable et dépend de la séquence de l’ARNcr. Les amorces avant et arrière pour la qPCR de l’ARNg et de l’ARNm ACT1 sont conçues à https://www.ncbi.nlm.nih.gov/tools/primer-blast/. Deux amorces sont choisies lorsque la différence entre leurs températures de fusion n’est pas supérieure à 2 °C (voir tableau supplémentaire 2). Chaque échantillon est mesuré en trois répétitions.- Préparez le mélange réactionnel qPCR conformément aux instructions du fabricant pour le kit SYBR.
- Définissez le programme qPCR suivant dans une machine de PCR en temps réel.
Préincubation : 10 min à 95 °C.
Stade PCR : 15 s à 95 °C, suivi de 34 s à 55 °C. Réglez le cycle de l’étape PCR à 45 fois. Phase de fusion : 10 s à 95 °C, puis 60 s à 65 °C, et enfin 1 s à 97 °C. - Calculer les valeurs relatives d’expression de l’ARNm via la formule de Pfaffl31.
7. Immunofluorescence pour détecter les protéines Cas
REMARQUE: Les protéines Cas (CasP) sont fusionnées à une His_tag.
- Préparation de cellules de levure
- Prélever des cellules de levure à l’aide d’une boucle stérile et les cultiver dans 5 mL de milieu riche en YPD pendant une nuit à 240 rpm à 30 °C. Le matin, ajouter 500 μL de la solution cellulaire à 20 mL de YPD frais et les cultiver à 240 rpm à 30 °C jusqu’à ce que le diamètre DO600 atteigne 0,6.
- Prenez 5 mL de la culture et mélangez-la avec 500 μL de formaldéhyde à 37%. Laisser le mélange à température ambiante (RT) pendant 10 min. Récolter les cellules par centrifugation à 1 500 x g pendant 5 min.
- Retirer le surnageant et remettre les cellules en suspension avec 1 mL de tampon de fixation (0,1 M KH2PO4, 0,5 M MgCl2, 3,7 % de formaldéhyde, pH =6,5). Conserver la solution cellulaire à TA pendant 20 min.
- Centrifuger la solution cellulaire à 1 500 x g pendant 5 min. Jeter le surnageant et remettre les cellules en suspension dans 1 mL de tampon de lavage (0,1 M KH 2 PO 4,1,2M de sorbitol, pH = 6,5) complété par 4 μL de bêta-mercaptoéthanol et4 μL de solution de lysat (5 mg/mL). Placer la solution cellulaire dans un incubateur à 37 °C pendant 20 min.
- Centrifuger la solution cellulaire à 1 500 x g pendant 5 min et jeter le surnageant. Laver la pastille de cellule deux fois avec 1 mL de PBS par centrifugation (1 500 x g pendant 5 min).
- Remettez les cellules en suspension dans 100 μL de PBS plus 0,05% de Tween 20 et ajoutez 4 μL de solution BSA (10 mg/mL). Conserver la solution cellulaire à TA pendant 20 min.
- Incubation avec anticorps primaire
- Ajouter l’anticorps primaire anti-His au mélange à l’étape 7.1.6 à une dilution de 1:400. Conserver la solution à TA pendant 2 h.
- Centrifuger le mélange à l’étape 7.2.1 à 1 500 x g pendant 5 min et retirer le surnageant. Ajouter 1 mL de PBST et centrifuger (1 500 x g) pendant 5 min pour laver la pastille de cellule. Répétez cette opération deux fois. Enfin, jetez le surnageant et suspendez les cellules dans 100 μL de 1x PBST.
- Détection de cellules de microscopie
- Montez les cellules sur une diapositive; prélever 2 μL de la solution cellulaire de l’étape 7.2.2 et la placer sur une lame de verre. Couvrez-le d’un bordereau.
- Observez les cellules au microscope à fluorescence. Allumez la source de lumière fluorescente, le microscope et l’ordinateur. Notez le numéro de la source de lumière fluorescente et ouvrez le logiciel du microscope sur l’ordinateur.
- Placez la lame sur la scène du microscope. Choisissez l’objectif 40x et observez les cellules sous la lumière verte (550 nm). Déplacez le bouton de mise au point grossière jusqu’à ce que le contour des cellules de levure apparaisse. Déplacez le bouton de mise au point fine pour concentrer les cellules.
- Détectez les cellules avec le logiciel de microscope. Fermez le champ de vision du microscope et passez à l’écran de l’ordinateur. Cliquez sur Live, attendez 3-5 s et cliquez sur Capturer pour prendre une photo. Enregistrez l’image.
- Éteignez l’ordinateur, le microscope et la source lumineuse fluorescente.
8. Acquisition de données : FACS
REMARQUE : La fluorescence verte est détectée par cytométrie en flux (c.-à-d. mesures de tri cellulaire activées par fluorescence [FACS]). Les cellules de levure sont cultivées, en général, à 30 °C et 240 rpm pour exécuter des expériences FACS. Cependant, les cellules peuvent exiger certaines précautions en fonction de leur contenu génétique. Les cellules qui contiennent le gène dCas12a-VPR (contrôlé par le promoteur constitutif GPD ) doivent être cultivées pendant 24 h dans la solution SDC. Ensuite, les cellules sont diluées dans un rapport de 1:100 dans du SDC frais et cultivées pendant 12 heures supplémentaires avant de mesurer l’intensité de fluorescence. Les cellules modifiées avec le gène AcrIIA4-HBD(hER) exigent également une dilution. En outre, la DO600 doit être contrôlée. Tout d’abord, les cellules sont autorisées à se développer en SDC pendant la nuit (plus de 14 h). Le matin, OD600 est mesuré. Ensuite, la culture est diluée dans du SDC, fourni avec une concentration variée de β-estradiol, jusqu’àOD 600 = 0,1. Avant les expériences FACS, les cellules sont cultivées pendant encore 7 heures de sorte que OD600 atteigne 0,8-1,0. Les cellules exprimant dCas9-VP64 ou dCas12a-VP64 sont cultivées en SDC pendant 20-24 h sans dilution ni croissance supplémentaire avant les mesures à la machine FACS.
- Allumez la machine FACS 20 min avant les mesures pour réchauffer le laser.
- Préparer les échantillons (dilution) : mélanger 20 μL de la culture cellulaire avec 300 μL deddH2O.
- Exécutez le logiciel FACS sur l’ordinateur connecté à la machine FACS et créez une nouvelle expérience. Réglez les paramètres de mesure (c.-à-d. FSC(SSC)-A/H/W et histogramme).
- Sélectionnez le filtre en fonction des longueurs d’onde d’excitation et d’émission des échantillons. Par exemple, la protéine cible ici est yEGFP, dont les longueurs d’onde d’excitation et d’émission sont respectivement de 488 nm et 507 nm. Sélectionnez donc le filtre FITC ou GFP (longueur d’onde d’excitation : 488 nm ; longueur d’onde d’émission : 527/32 nm). Définissez le numéro de cellule d’acquisition sur 10 000.
- Ajustez la tension du filtre FITC en mesurant l’intensité des billes fluorescentes. S’assurer que la différence relative d’intensité des billes entre deux expériences consécutives ne dépasse pas 5 %.
- Lavez la machine avec ddH2O pendant quelques secondes pour éliminer tout excès de billes éventuel.
- Mesurer l’intensité de fluorescence de l’échantillon. Cliquez sur Aperçu et attendez 3-5 s pour la stabilité de l’injection de l’échantillon. Enfin, cliquez sur Acquérir.
- Mesurez à nouveau les perles à la fin de l’expérience. La tension est celle qui a été utilisée lors de la mesure initiale des billes (voir étape 8.4, 438-441 V). Vérifiez si la différence relative entre les mesures des deux perles dépasse 5%.
- Exportez les données FACS sous forme de fichiers FCS.
- Analysez les fichiers FCS avec le logiciel R Studio.
9. Analyse des données
REMARQUE: Utilisez le package Flowcore R Bioconductor 32 dans R studio. Les fichiers FCS ont été analysés à l’aide d’un script écrit en langage R.
- Ouvrez R studio et chargez le script d’analyse Bdverse. R pour analyser les fichiers FCS.
- Définissez le nom de l’expérience (ename), le répertoire (chemin) dans lequel se trouvent les fichiers FCS
Stocké (dir_d) et où les fichiers de résultats seront créés (dir_r). - Réglez le canal de fluorescence. Par exemple, select_ch = « GPA-A » si la fluorescence verte a été mesurée.
- Définissez le nombre d’échantillons mesurés (s_num).
- Définissez les dimensions de chaque diagramme à points (sxlim, sylim). Définissez la longueur maximale des axes x et y pour les diagrammes à barres et les diagrammes en boîte (xlimit, ylimit). xlimit doit être supérieur ou égal à s_num.
- Choisissez la méthode de contrôle en supprimant le # des lignes correspondantes.
REMARQUE: morphGate est une méthode de contrôle automatique effectuée par le script (c’est-à-dire que la région des diagrammes de points où les cellules sont plus denses est reconnue et sélectionnée par le programme). polygonGate et rectangleGate demandent d’examiner les diagrammes de points et de définir les sommets d’un polygone ou le côté d’un rectangle qui embrasse la zone où se trouvent la plupart des cellules. - Sélectionnez l’objet flowSet gated, correspondant à la méthode de contrôle choisie. Sélectionnez la résolution du graphique à points (res ; doit être au moins égale à 256).
- Décommenter flt_low <- filter_low(sp) pour supprimer les mesures où la fluorescence est négative. Décommentez flt_sp < filtre(flt_lw s) pour supprimer les valeurs aberrantes dues à d’autres expériences.
- Appuyez sur Source et exécutez le script. Tous les fichiers contenant les résultats de l’analyse sont créés dans dir_r.
Résultats
Expression d’ARNg/ARNcr par un promoteur de type ARN polymérase III
Tout d’abord, ces travaux ont porté sur l’ingénierie du circuit d’activation transcriptionnelle (circuit 1) illustré à la figure 1A. Il contenait trois composants de base: 1) le gène codant pour yEGFP (le rapporteur), qui a été précédé par une série de promoteurs synthétiques différents qui ont fourni des sites cibles pour dCas9 / dCas12a-AD; 2) une version optimisée pour les codons...
Discussion
Le protocole a montré un flux de travail complet possible pour les circuits numériques de gènes synthétiques, suivant le cycle de génie biologique « Design-Build-Test-Learn » (DBTL) et concernant à la fois des expériences de laboratoire sec et de laboratoire humide. Ici, nous nous sommes concentrés sur le système CRISPR-Cas, principalement dSpCas9, denAsCas12a, dLbCas12a et les protéines anti-CRISPR correspondantes, en concevant et en construisant dans S. cerevisiae de petits réseaux transcription...
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt financier concurrent.
Remerciements
Nous tenons à remercier tous les étudiants du laboratoire de biologie synthétique SPST, TJU pour leur aide générale, ainsi que Zhi Li et Xiangyang Zhang pour leur aide dans les expériences FACS.
matériels
| Name | Company | Catalog Number | Comments |
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | - | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | - | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | - | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | - | - | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
Références
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon