Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
CRISPR-Cas Sistemlerine ve Anti-CRISPR Proteinlerine Dayalı Gen Dijital Devreleri
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
CRISPR-Cas sistemleri ve anti-CRISPR proteinleri, Saccharomyces cerevisiae'deki Boole kapılarının şemasına entegre edildi. Yeni küçük mantık devreleri iyi performans gösterdi ve hem dCas9 / dCas12a tabanlı transkripsiyon faktörlerinin hem de anti-CRISPR proteinlerinin özelliklerinin anlaşılmasını derinleştirdi.
Özet
Sentetik gen Boole kapıları ve dijital devreler, tıbbi teşhisten çevresel bakıma kadar geniş bir uygulama alanına sahiptir. CRISPR-Cas sistemlerinin ve doğal inhibitörlerinin (anti-CRISPR proteinleri (Acrs)) keşfi, in vivo gen dijital devrelerini tasarlamak ve uygulamak için yeni bir araç sağlar. Burada, "Tasarla-İnşa Et-Test Et-Öğren" biyolojik mühendislik döngüsü fikrini takip eden ve Saccharomyces cerevisiae'de bazıları Boole kapıları gibi davranan küçük transkripsiyonel ağlar oluşturmak için dCas9 / dCas12a'yı karşılık gelen Acrs ile birlikte kullanan bir protokolü açıklıyoruz. Bu sonuçlar, transkripsiyon faktörleri olarak dCas9 / dCas12a'nın özelliklerine işaret etmektedir. Özellikle, gen ekspresyonunun maksimum aktivasyonunu elde etmek için, dSpCas9'un VP64 aktivasyon alanının (AD) çoklu kopyalarını toplayan tasarlanmış bir iskele RNA'sı ile etkileşime girmesi gerekir. Buna karşılık, dCas12a, C terminusta, güçlü VP64-p65-Rta (VPR) AD ile kaynaştırılmalıdır. Ayrıca, her iki Cas proteininin aktivitesi, hücredeki sgRNA / crRNA miktarını artırarak arttırılmaz. Bu makalede ayrıca CRISPR-dCas-Acr etkileşimine dayalı Boole kapılarının nasıl oluşturulacağı açıklanmaktadır. İnsan östrojen reseptörünün AcrIIA4 kaynaşmış hormon bağlama alanı, β-östradiol'e yanıt veren bir NOT kapısının çekirdeğidir, oysa indüklenebilir GAL1 promotörü tarafından sentezlenen AcrVA'lar, hem EVET hem de NOT kapılarını bir girdi olarak galaktoz ile taklit etmeye izin verir. İkinci devrelerde, AcrVA5, dLbCas12a ile birlikte en iyi mantık davranışını gösterdi.
Giriş
2011 yılında, araştırmacılar hesaplamalı bir yöntem önerdiler ve dijital sentetik gen devrelerinin otomatik tasarımı için karşılık gelen bir yazılım parçası geliştirdiler1. Bir kullanıcının giriş sayısını (üç veya dört) belirtmesi ve devre doğruluk tablosunu doldurması gerekiyordu; Bu, elektronikten teknikler kullanarak devre yapısını türetmek için gerekli tüm bilgileri sağladı. Doğruluk tablosu, Karnaugh harita yöntemi2 aracılığıyla iki Boole formülüne çevrildi. Her Boole formülü, devre girişleri ve bunların olumsuzlamaları (değişmez değerler) arasındaki (bir kısmı) mantık işlemlerini (toplama veya çarpma) tanımlayan yan tümcelerden oluşur. Yan tümceler, sırasıyla, devre çıkışını hesaplamak için ya toplanır (OR) ya da çarpılır (AND). Her devre, karşılık gelen iki formülden herhangi birine göre gerçekleştirilebilir: biri POS (toplamların ürünü) formunda, diğeri SÇP (ürünlerin toplamı) temsilinde yazılmıştır. İlki, değişmez değerlerin mantıksal bir toplamını içeren cümleciklerin (yani, Boole kapıları) çarpımından oluşur. İkincisi, aksine, değişmez değerlerin çarpıldığı bir cümle toplamıdır.
Elektrik devreleri, bir breadboard üzerinde, farklı kapıları fiziksel olarak birbirine bağlayarak gerçekleştirilebilir. Elektrik akımı, kapılar arasında sinyal alışverişine izin verir ve bu da çıkışın hesaplanmasına yol açar.
Biyolojide durum daha karmaşıktır. Bir Boole kapısı, transkripsiyon veya translasyonun (veya her ikisinin) düzenlendiği bir transkripsiyon birimi (TU; yani, ökaryotik hücreler içindeki "promotör-kodlayıcı bölge-sonlandırıcı" dizisi) olarak gerçekleştirilebilir. Böylece, en az iki tür molekül biyolojik bir kablolama kurar: transkripsiyon faktörü proteinleri ve kodlamayan, antisens RNA'lar1.
Bir gen dijital devresi, iki veya üç kapı katmanı halinde düzenlenir, yani: 1) EVET (tampon) ve NOT kapılarından oluşan ve giriş kimyasallarını kablolama moleküllerine dönüştüren giriş katmanı; 2) karşılık gelen Boole formülünde yan tümceler olduğu kadar TU'dan oluşan iç katman. Devre SÇP formülüne göre tasarlanmışsa, iç katmandaki her cümle, dağıtılmış çıkış mimarisi olarak adlandırılan bir devrede devre çıkışını (örneğin, floresan) üretecektir. Toplam (POS) formülünün ürünü kullanılırsa, kablolama moleküllerini iç katmandan toplayan tek bir çarpma kapısı içerecek olan 3) son katman gereklidir.
Genel olarak, sentetik biyolojide, aynı devre için birçok farklı şema tasarlanabilir. Hem TU'ların hem de kablolama moleküllerinin sayısı ve türü bakımından farklılık gösterirler. Maya hücrelerinde uygulanacak en kolay çözümü seçmek için, her devre tasarımı, şu şekilde tanımlanan bir karmaşıklık skoru S ile ilişkilendirilir:
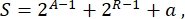
burada A, aktivatörlerin sayısını, R, baskılayıcıların sayısını ve A, antisens RNA moleküllerinin miktarını temsil eder. Aktivatörler veya baskılayıcılar devrede yoksa, S'ye katkıları sıfırdır. Bu nedenle, çok sayıda ortogonal transkripsiyon faktörü gerektirdiğinde laboratuarda bir devre şeması (yüksek S) gerçekleştirmek daha zordur. Bu, dijital devrelerin içindeki tüm kablolamayı gerçekleştirmek için yeni aktivatörlerin ve baskılayıcıların de novo olarak tasarlanacağı anlamına gelir. Prensip olarak, yeni DNA bağlayıcı proteinler, şablon olarak Çinko Parmak proteinleri3 ve TAL efektörleri4 kullanılarak birleştirilebilir. Ancak, bu seçenek çok zorlu ve zaman alıcı görünüyor; bu nedenle, karmaşık gen devrelerini sonlandırmak için çoğunlukla küçük RNA'lara ve çeviri düzenlemesine güvenilmelidir.
Başlangıçta, bu yöntem bakterilerde dijital devreler üretmek için geliştirilmiştir. Gerçekten de, ökaryotik hücrelerde, antisens RNA'lar yerine, mikroRNA'lardan (miRNA'lar) veya küçük müdahale eden RNA'lardan (siRNA'lar)5 bahsetmek daha uygundur. Bununla birlikte, RNAi yolu maya S. cerevisiae'de mevcut değildir. Bu nedenle, kişi tamamen transkripsiyonel ağları tercih etmelidir. Bir devrenin beş aktivatöre ve beş baskıcıya ihtiyacı olduğunu varsayalım; karmaşıklık puanı S = 32 olacaktır. Devre karmaşıklığı, 10 transkripsiyon faktörünün bir aktivasyon alanına (AD) kaynaşmış tek bir dCas96 (nükleaz eksikliği Cas9) ile değiştirilmesiyle azaltılabilir. 7'de gösterildiği gibi, dCas9-AD, TATA kutusu ile TSS (transkripsiyon başlangıç bölgesi) arasında bir promotör bağlarken mayada bir baskılayıcı olarak ve TATA kutusunun yukarı akışını iyi bağlarken bir aktivatör olarak çalışır. Böylece, 10 transkripsiyon faktörünü, tek bir dCas9-AD füzyon proteini ve 10 sgRNA (tek kılavuz RNA'lar) ile S = 11'lik toplam karmaşıklık skoru için değiştirebiliriz. On sgRNA'yı sentezlemek hızlı ve kolaydır, oysa daha önce yorumlandığı gibi, 10 proteinin montajı çok daha uzun ve daha karmaşık bir çalışma gerektirecektir.
Alternatif olarak, biri iki ortogonal dCas proteini (örneğin, dCas9 ve dCas12a) kullanabilir: biri bir AD ile kaynaşmak için, diğeri çıplak veya bir baskı alanı ile kombinasyon halinde. Karmaşıklık puanı yalnızca bir birim artacaktır (S = 12). Bu nedenle, CRISPR-dCas sistemleri, S. cerevisiae'de çok karmaşık gen dijital devrelerinin inşasının anahtarıdır.
Bu makale, mayadaki hem dCas9- hem de dCas12a bazlı baskılayıcıların ve aktivatörlerin verimliliğini derinden karakterize etmektedir. Sonuçlar, aktivitelerini optimize etmek için yüksek miktarda sgRNA talep etmediklerini göstermektedir, bu nedenle epizomal plazmidlerden tercihen kaçınılmaktadır. Dahası, dCas9 tabanlı aktivatörler, VP64 AD'nin kopyalarını toplayan bir iskele RNA'sı (scRNA) kullanıldığında çok daha etkilidir. Buna karşılık, dCas12a doğrudan güçlü VPR AD ile kaynaştırıldığında iyi çalışır. Ayrıca, sentetik olarak aktive edilmiş bir promotör, aktivatörün konfigürasyonuna bağlı olarak değişken sayıda hedef bölge gerektirir (örneğin, dCas12a-VPR kullanırken üç, dCas9-VP64 için altı ve dCas9 ve scRNA ile sadece bir tane). Bir baskılayıcı olarak, dCas12a, promotörden ziyade kodlama bölgesini bağlarken daha keskin görünür.
Bununla birlikte, bir dezavantaj olarak, CRISPR-dCas9 / dCas12a kimyasallarla doğrudan etkileşime girmez. Bu nedenle, giriş katmanında hiçbir işe yaramayabilirler. Bu nedenle, anti-CRISPR proteinleri (Acrs) içeren alternatif Boole kapısı tasarımları araştırılmıştır. Akr'ler (d)Cas proteinleri üzerinde etkili olur ve çalışmalarını engeller8. Bu nedenle, CRISPR-(d)Cas sistemlerinin aktivitesini modüle etmenin bir yoludur. Bu yazıda S. cerevisiae'de tip II Acrs ve (d)Cas9 ile tip V Acrs ve (d)Cas12a arasındaki etkileşimler kapsamlı bir şekilde analiz edilmiştir. Acrs, Cas proteinlerinden çok daha küçük olduğundan, östrojen β-östradiol'e yanıt veren bir NOT kapısı, insan östrojen reseptörü 9-HBD'nin (hER) hormon bağlama alanını AcrIIA4'e kaynaştırarak inşa edilmiştir. Ayrıca, dCas12a(-AD)'yi yapısal olarak ifade eden bir avuç EVET ve DEĞİL kapısı ve galaktoz ile indüksiyon üzerine AcrVA'lar gerçekleştirildi. Şu anda, bu kapılar sadece bir kavram kanıtı olarak hizmet vermektedir. Bununla birlikte, aynı zamanda, maya hücrelerindeki sentetik gen dijital devrelerinin hesaplamalı otomatik tasarımını gerçekleştirmek için algoritmanın derinlemesine yeniden düşünülmesine yönelik ilk adımı temsil ederler.
Protokol
1. sgRNA/crRNA ekspresyon kasetinin tasarımı ve yapımı
NOT: İki tür sgRNA/crRNA ekspresyon kaseti vardır: bir terimli SNR5210-, RNA polimeraz III'e bağımlı SNR52 promotörü, sgRNA/crRNA dizisi ve SUP4 sonlandırıcısından oluşur; RGR11 olarak kısaltılan bir diğeri, RNA polimeraz II'ye bağımlı ADH1 promotörü, iki ribozim (bir çekiç kafalı ribozim-HH ve bir hepatit delta virüsü ribozim-HDV) içeren RGR (ribozim-kılavuz RNA-ribozim) yapısından ve aradaki sgRNA / crRNA dizisinden ve ADH1 sonlandırıcısından oluşur. Cas9 homologlarını yönlendiren sgRNA, bir ara parça dizisinden ve karakteristik doğrudan tekrar12'den oluşurken, Cas12a proteinleri için crRNA, doğrudan tekrarı ve ardından ara parça dizisi13,14'ü içerir (bu çalışmada kullanılan tüm DNA dizileri için Ek Tablo 1'e bakınız).
- Cas9/Cas12a aracılı transkripsiyonel aktivasyon için ara parça dizisini tasarlayın.
- Bakteriyel lex operatör dizisini (lexOp olarak adlandırılır) hedef bölge15,16 olacak şekilde kullanın ve mayayla güçlendirilmiş yeşil floresan proteininin (yEGFP) ekspresyonunu yönlendiren CYC1 promotörüne yerleştirin17. Bu nedenle, ara parça dizisi eklenen lexOp tarafından tanımlanır ve tamamlayıcı niteliktedir.
- Ara parça dizisinin ortogonalliğini CRISPRDIRECT aracı18 ile kontrol edin.
- PAM dizisinin yanında bulunan lexOp dizisini metin alanına yapıştırın, PAM dizisini dCas9 için NRG ve dCas12a için TTTV olarak tanımlayın ve açılır listedeki türleri Tomurcuklanan Maya (Saccharomyces cerevisiae) S288C Genomu olarak belirtin. Tasarım'a tıklayın. 20mer+PAM veya 12mer+PAM aramasında eşleşen hedef site olmadığından emin olun.
- SgRNA/crRNA ekspresyon kasetini oluşturun.
- Promotörler, kodlama dizileri ve sonlandırıcılar gibi standart biyolojik parçaların DNA dizilerini yükseltmek için touchdown PCR'yi kullanın.
- Aşağıdakileri içeren bir reaksiyon karışımı hazırlayın: 20-40 ng DNA şablonu, 1 μL 10 μM ileri astar (yani, ot25, sgRNA / crRNA ekspresyon plazmid yapısı), 1 μL 10 μM ters astar (yani, ot26, sgRNA / crRNA ekspresyon plazmid yapısı), 5 μL 2.5 mM dNTP karışımı, 0.5 μL DNA polimeraz, 10 μL 5x DNA polimeraz reaksiyon tamponu, ve toplam 50 μL hacme kadar çift damıtılmış su (ddH2O).
NOT: Bu çalışmada kullanılan primerlerin bir listesi için Ek Tablo 2'ye bakınız. - Touchdown PCR programını bir termosikletçi üzerinde çalıştırın:
1. Aşama: 30 sn için 98 °C.
10 döngülü Aşama 2: 10 s için 98 °C, 20 s için 68 °C ve 15 s için 72 °C.
25 döngülü Aşama 3: 10 s için 98 °C, 20 s için 59 °C ve 15 s için 72 °C.
4. Aşama: 2 dakika boyunca 72 °C.
Son olarak, takip deneylerine başlayana kadar 4 ° C'de tutun.
NOT: Aşama 2'deki 68 °C ve aşama 3'teki 59 °C, farklı astar çiftlerinden değişen hem ileri hem de geri astarların erime sıcaklıklarına bağlıdır. Aşama 2 ve 3'te 72 ° C'deki zaman, PCR ürününün uzunluğu ve DNA polimerazın hızı ile belirlenir.
- Aşağıdakileri içeren bir reaksiyon karışımı hazırlayın: 20-40 ng DNA şablonu, 1 μL 10 μM ileri astar (yani, ot25, sgRNA / crRNA ekspresyon plazmid yapısı), 1 μL 10 μM ters astar (yani, ot26, sgRNA / crRNA ekspresyon plazmid yapısı), 5 μL 2.5 mM dNTP karışımı, 0.5 μL DNA polimeraz, 10 μL 5x DNA polimeraz reaksiyon tamponu, ve toplam 50 μL hacme kadar çift damıtılmış su (ddH2O).
- PCR ürünlerini jel elektroforezi ile izole edin (100 V, 30 dk). Agaroz jelinden DNA dizilerini bir DNA jeli ekstraksiyon kiti ile çıkarın (bakınız Malzeme Tablosu).
NOT: 500 nt'den uzun fragmanlar için %0.8 agaroz jeli, 100 nt ile 500 nt arasındaki fragmanlar için %1.5 agaroz jeli ve 100 nt'den kısa fragmanlar için %2 agaroz jeli gereklidir. - TU eksprese eden sgRNA/crRNA'yı, ampisilin direnç genini ve mayayla seçilebilen oksirofik belirteç geni-TRP1'i içeren bir pRSII404/424 mekik vektörü19'a yerleştirin.
- Mekik vektörünü 37 ° C'de iki kısıtlama enzimi SacI ve Acc65I ile 1 saat boyunca sindirin. 5 μg mekik vektörü, önerilen enzim miktarları, sindirim tamponu (enzim talimatlarına göre) ve toplam 30 μL hacme kadar ddH2O ekleyerek reaksiyon karışımını hazırlayın.
- Mekik vektör sindirimini jel elektroforezi ile doğrulayın (bkz. alt adım 1.3.2). Daha sonra, iki enzimi 65 ° C'de 20 dakika boyunca inaktive edin.
- Saflaştırılmış PCR ürünlerini, ekomolar DNA karışımını 1 saat boyunca 50 ° C'de bırakarak kesik açık mekik vektörüne yerleştirmek için Gibson montaj yöntemi20'yi kullanın.
- Escherichia coli DH5α yetkin hücreleri, ısı şoku dönüşüm yöntemi21 ile yukarıdaki Gibson reaksiyon karışımı ile dönüştürün. Dönüştürülmüş E. coli hücrelerini ampisilin (0.1 g / L) içeren Luria-Bertani (LB) agar plakalarına aktarın. Plakaları 37 ° C'de inkübatöre koyun ve hücrelerin gece boyunca büyümesine izin verin.
- LB agar plakasından dört koloni seçin ve bunları ampisilin (0.1 g / L) içeren LB çözeltisinde 37 ° C'de gece boyunca ayrı ayrı kültürleyin. Daha sonra, E. coli hücrelerinden plazmidleri çıkarmak için mini hazırlama prosedürünü kullanın22.
- Eklenen transkripsiyon ünitesini Sanger yöntemi23 aracılığıyla sıralamak ve onaylamak için ot18 ve ot19 primerlerini kullanın (oligo dizileri için Ek Tablo 2'ye bakınız).
NOT: Daha sonraki deneylerde, inşa edilmiş ve doğrulanmış plazmidler, PEG / LiAc protokolü24 aracılığıyla maya genomuna yerleştirilecektir.
- Promotörler, kodlama dizileri ve sonlandırıcılar gibi standart biyolojik parçaların DNA dizilerini yükseltmek için touchdown PCR'yi kullanın.
2. İskele RNA ekspresyon kasetinin tasarımı ve yapımı
NOT: İskele kılavuz RNA (scRNA), sgRNA dizisi ve MS2 saç tokası yapıları25'ten oluşur. Bu çalışmada iki tür MS2 saç tokası yapısı kullanılmıştır: vahşi tip MS2 saç tokası-wt ve f6 MS2 kaplama proteini (MCP) aptamer-f6.
- Farklı ara parça dizilerini (yani, pSNR52-spacer_DR(SpCas9)-2×MS2(wt+f6)-SUP4t) barındırabilen bir scRNA şablonu sentezleyin.
NOT: Bu çalışmada, scRNA şablonu bir gen sentez şirketi tarafından sentezlenmiştir (bakınız Malzeme Tablosu). - PCR'yi gerekli ara parça dizilerinde çalıştırmak için uygun astarlar tasarlayın ( bakınız Ek Tablo 2).
- Bir scRNA ekspresyon kaseti oluşturmak için adım 1.3'teki yordamları izleyin.
3. dSpCas9 mühendislik ve ekspresyon plazmid yapımı
- Plazmid pTPGI_dSpCas9_VP64 elde edin (bkz.
- Alıcı vektör pRSII406-pGPD-ATG-XbaI-XhoI-CYC1t'yi, pRSII406 mekik vektörünü temel alarak, touchdown PCR ve Gibson montaj yöntemiyle oluşturun (adım 1.3'e bakın). Plazmid, güçlü bir kurucu promotör-pGPD ve bir sonlandırıcı-CYC1t sağlar.
- pTPGI_dCas9_VP64 plazmidini ve yeni inşa edilen alıcı vektörü (1 saat için 5 μg veya bir gecede 10 μg - referans olarak adım 1.3.3.1'e bakınız) XbaI ve XhoI ile 37 ° C'de sindirin. Kesici uç parçasını ve alıcı vektörünü adım 1.3.2'deki gibi ayırın ve saflaştırın.
- Saflaştırılmış kesici uç parçasını ve alıcı vektörü T4 DNA ligaz ile 16 °C'de 8 saat boyunca bağla. Saflaştırılmış alıcı vektörün 50-100 ng'sini, alıcı vektör ile eşamolar miktarda saflaştırılmış hedef parçaları, 1.5 μL T4 tamponu, 0.5 μL T4 ligaz veddH2O ekleyerek toplam 15 μL hacme kadar ligasyon çözeltisini hazırlayın.
- 1.3.3.3 ve 1.3.3.4 adımlarını izleyin. Ardından, yeni oluşturulan plazmidin XbaI ve Xhol (37 °C, 1 saat) ve jel elektroforezi (bkz. adım 1.3.2) ile sindirimi ile doğru olduğunu onaylayın.
4. dCas12a mühendislik ve plazmid yapımı
- dCas12a-AD'yi ifade eden plazmidleri oluşturun.
- BamHI ve Xhol kısıtlama enzim bölgeleri tarafından kuşatılmış iki maya kodonu için optimize edilmiş dCas12a proteinini (denAsCas12a ve dLbCas12a) sentezleyin.
NOT: Bu çalışmada, iki maya kodonu için optimize edilmiş dCas12a proteini bir gen sentez şirketi tarafından sentezlenmiştir (bakınız Malzeme Tablosu). - Alıcı vektör pRSII406-promoter-ATG-NLS-GS-HIStag-GS-BamHI-sp-XhoI-AD-NLS-TAA-mTGUO1'i touchdown PCR ve Gibson montaj yöntemi (bkz. adım 1.3) aracılığıyla oluşturun, burada "promotör" pGPD veya pGAL1, "sp" kısa bir rastgele diziyi temsil eder ve "AD" VPR veya VP64'tür.
- Her bir dCas12a proteinini, BamHI ve XhoI ile sindirim ve T4 DNA ligaz ile ligasyon yoluyla yeni oluşturulan iki alıcı vektöre yerleştirin (adım 3.3 ve 3.4'e bakın).
- BamHI ve Xhol kısıtlama enzim bölgeleri tarafından kuşatılmış iki maya kodonu için optimize edilmiş dCas12a proteinini (denAsCas12a ve dLbCas12a) sentezleyin.
- Çıplak bir dCas12a ifade eden plazmidleri oluşturun.
- dCas12a'nın galaktozla indüklenebilir ifade kasetleri için pRSII406-Acc651-pGAL1-ATG-NLS-GS-HIStag-GS-BamHI-sp-XhoI-GS-NLS-TAA-CYC1t olarak touchdown PCR ve Gibson montaj yöntemini kullanarak bir alıcı vektör oluşturun (bkz. adım 1.3).
- dCas12a proteinlerini ve yukarıdaki alıcı vektörü içeren plazmidleri BamHI ve Xhol ile sindirin, ardından plazmid pRSII406-pGAL1-Acc651-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1t'yi almak için bunları T4 DNA ligaz ile birleştirin (adım 3.3 ve 3.4'e bakın).
- Adım 4.2.2'de elde edilen plazmidi Acc65I ve BamHI ile sindirin ve ardından pRSII406-pGPD-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1t'yi oluşturmak için touchdown PCR ve Gibson montaj yöntemi ile pGAL1'i pGPD ile değiştirin.
5. Anti-CRISPR protein mühendisliği ve plazmid yapımı
NOT: Acrs ekspresyonunu yönlendirmek için üç tür promotör kullanılmıştır: indüklenebilir bir promotör-pGAL1, dört maya kurucu promotör-pGPD, pACT1, pTEF1 ve pTEF2 ve sentetik bir kurucu promotör-genCYC1t_pCYC1noTATA26.
- Tip II Acrs (AcrIIA2, AcrIIA427 ve AcrIIA5 28) ve tip V-A Acrs (AcrVA1, AcrVA4 ve AcrVA529) dizilerini içeren plazmidleri bir gen sentez şirketinden elde edin.
- Acrs'yi ifade etmek için pRSII403 mekik vektörünü temel alan plazmidleri oluşturun.
- AcrIIA ifade kasetleri oluşturun.
NOT: Dört farklı promotörü (yani pGPD, pACT1, pTEF2 ve genCYC1t_pCYC1noTATA), üç tür AcrIIA'yı ve iki sonlandırıcıyı (ADH1t ve CYC1t) yükseltmek için touchdown PCR'yi kullanın. Gibson derleme yöntemiyle farklı destekleyiciler altında AcrIIA'ları ifade eden bir dizi TU oluşturun (adım 1.3'e bakın). - AcrVA ifade kasetleri oluşturun.
- Alıcı dizisini sentezleyin: ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6, burada "sp" daha sonra AcrVA'larla değiştirilecek rastgele bir dizidir.
NOT: Bu çalışmada, alıcı diziler bir gen sentez şirketi tarafından sentezlenmiştir (Malzeme Tablosu). - pRSII403-promoter-ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6 alıcı vektörlerini bir araya getirin, burada "promotör" şu şekildedir: pGAL1, pTEF1 ve genCYC1t_pCYC1noTATA. Gibson derleme yöntemini kullanın (adım 1.3'e bakın).
- AcrVA'lar üreten bir dizi plazmid oluşturmak için üç AcrVA'nın her birini adım 5.2.2.2'de açıklanan alıcı vektöre (touchdown PCR ve Gibson montaj yöntemi aracılığıyla [bkz. adım 1.3]) ekleyin.
- Alıcı dizisini sentezleyin: ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6, burada "sp" daha sonra AcrVA'larla değiştirilecek rastgele bir dizidir.
- AcrIIA ifade kasetleri oluşturun.
- AcrIIA4'ü GS bağlayıcı ve HBD (hER) dizileri ile genişleterek daha fazla mühendislik yapın. Bu, β-estradiol-duyarlı bir devrenin oluşturulmasına izin verir.
- GS-HBD ve AcrIIA4 parçalarını ayrı ayrı elde etmek için touchdown PCR kullanın (bkz. adım 1.3.1).
- AcrIIA4, GS-HBD ve mekik vektörünü Gibson karışımına koyun ve Gibson yöntemiyle tam plazmidi oluşturun (bkz. adım 1.3.3).
6. crRNA tespiti: RT-qPCR ve primerlerin tasarımı
NOT: crRNA tespiti, üç adımda düzenlenen RT-qPCR ile sağlanmıştır.
- RNA ekstraksiyonunu ve maya hücrelerinden saflaştırmayı bir RNA kiti aracılığıyla gerçekleştirin.
- Kültür mayası hücreleri, 24 delikli bir plaka (240 rpm, 30 °C) kullanılarak 2 mL sentetik tanımlı tam ortamda (SDC, 1 L hacim: 20 g Glikoz, 2 g AA karışımı, 6.7 g YNB, 396 mg Lösin, 79.2 mg Triptofan, 79.2 mg Histidin ve 79.2 mg Urasil) gece boyunca toplanır.
- Sabahları, hücre kültürünü (1:100) 2 mL taze SDC'ye seyreltin ve maya hücrelerini 30 ° C, 240 rpm'de 4 saat daha büyütmeye devam edin.
- Hücre çözeltisinin 2 mL'sinin tamamını hasat edin ve 2 dakika boyunca 20.238 x g'de santrifüjleyin. Hücre peleti küçük olduğu için süpernatantı dikkatlice çıkarın.
- RNA'yı bir RNA kiti kullanarak maya hücrelerinden çıkarın.
- RNA kalitesini kontrol edin.
- % 1'lik bir agaroz jeli hazırlayın. Her RNA örneğinin 5 μL'sini 1 μL DNA yükleme boyası ile karıştırın. Sonra karışımı jelin üzerine yükleyin ve çalıştırın.
- Jel üzerinde ribozomal RNA'ya (25S / 18S) karşılık gelen ~ 4.000 nt ve ~ 2.000 nt'de iki net bandın bulunduğundan emin olun. tRNA için ~ 80 nt'de daha bulanık bir bant görülebilir.
- RNA örneklerini cDNA sentezi için hemen kullanın veya ileride kullanmak üzere -80 ° C'de saklayın.
- Ters transkripsiyon: crRNA'ya (40 nt) karşılık gelen ilk cDNA ipliğini oluşturmak için kök döngü yöntemi30'u kullanın. SgRNA'nın ters transkripsiyonu için (neredeyse 100 nt), prosedür ACT1 referans geninin cDNA sentezi ile aynıdır.
NOT: crRNA'nın ters transkripsiyonu için kullanılan yöntem, sgRNA ve ACT1 mRNA ile kullanılandan farklıdır. crRNA çok kısa olduğu için, bir mikroRNA olarak muamele edildi ve karşılık gelen cDNA'yı elde etmek için mikroRNA ters transkripsiyon yöntemini (kök döngü yaklaşımı) kullandı. crRNA nicelleştirmesinde iki cDNA sentez kiti (crRNA için bir kök döngü kiti ve ACT1 geni için normal bir ters transkripsiyon kiti) kullanılmıştır. Deney sonuçlarını karşılaştırılabilir hale getirmek için iki kitte ( Malzeme Tablosuna bakınız) aynı miktarda RNA kullanılmıştır. Sap-döngü kiti ile kullanılan astar, sap-döngü dizisine ve crRNA'nın 3' ucundaki son altı nükleotite göre tasarlanmıştır (sap-döngü ve primer dizisi için Ek Tablo 2'ye bakınız).- crRNA ters transkripsiyonu için kök döngü yöntemi
- RNA şablonunu, astarı ve tamponları dondurucudan çıkarın ve buz üzerinde erimelerine izin verin.
- Genomik DNA çıkarılması: İlk olarak, kit talimatlarına göre reaksiyon karışımının 10 μL'sini hazırlayın. Karışımı 2 dakika boyunca 42 ° C'de bir termal döngüleyiciye koyun. Son olarak, buza aktarın.
- İlk cDNA ipliğinin sentezi: Adım 6.2.1.2'den 10 μL karışım, 1 μL kök döngü astarı (2 μM konsantrasyonu), 2 μL 10x RT reaksiyon tamponu, 2 μL ters transkripsiyon enzim karışımı (ters transkriptaz içeren) ve 5 μL RNaz içermeyenH2O ekleyerek reaksiyon karışımının 20 μL'sini hazırlayın.
- Reaksiyon karışımını bir termal döngüleyiciye yerleştirin ve aşağıdaki programı çalıştırın: 5 dakika boyunca 25 °C, 15 dakika boyunca 50 °C ve 5 dakika boyunca 85 °C. Ürünü qPCR reaksiyonu için hemen kullanın veya -80 ° C'de saklayın.
- sgRNA ve ACT1 mRNA ters transkripsiyon
- İlk reaksiyon: Kit talimatlarına göre astar karışımı, dNTP karışımı, RNA şablonu (50 pg-5 μg) ve RNaz içermeyen sudan (hepsi kit tarafından sağlanan RNA şablonu dışında) oluşan 13 μL'lik bir karışım hazırlayın. 1 μg RNA şablonu kullanın. Karışımı 10 dakika boyunca 70 ° C'de bir termal döngüleyiciye koyun.
- İkinci reaksiyon (cDNA sentezi): Reaktifleri (kit talimatlarında açıklandığı gibi) ilk reaksiyon çözeltisinin 13 μL'sine 20 μL'lik son bir hacme kadar ekleyerek reaksiyon karışımını hazırlayın. karışımı 50 ° C'de 15 dakika boyunca bir termal döngüleyiciye yerleştirin ve daha sonra 85 ° C'de 5 dakika daha bekleyin. Ürünü qPCR reaksiyonu için hemen kullanın veya -80 ° C'de saklayın.
- crRNA ters transkripsiyonu için kök döngü yöntemi
- qPCR için SYBR kiti: Ct değeri algılama
NOT: crRNA'nın qPCR'sinde kullanılan ters astar, sap-döngü dizisinin ters tamamlayıcısına karşılık geldiği için sabittir (bakınız Ek Tablo 2). İleri astar, aksine, değişkendir ve crRNA'nın dizisine bağlıdır. SgRNA ve ACT1 mRNA qPCR için ileri ve geri primerler https://www.ncbi.nlm.nih.gov/tools/primer-blast/ olarak tasarlanmıştır. İki astar, erime sıcaklıkları arasındaki fark 2 ° C'den büyük olmadığında seçilir (bakınız Ek Tablo 2). Her numune üç kopya halinde ölçülür.- qPCR reaksiyon karışımını üreticinin SYBR kiti talimatlarına göre hazırlayın.
- Gerçek zamanlı bir PCR makinesinde aşağıdaki qPCR programını ayarlayın.
Ön inkübasyon: 95 ° C'de 10 dakika.
PCR aşaması: 95 °C'de 15 sn, ardından 55 °C'de 34 sn. PCR aşamasının döngüsünü 45 kez olarak ayarlayın. Erime aşaması: 95 ° C'de 10 sn, ardından 65 ° C'de 60 s ve son olarak 97 ° C'de 1 sn. - Pfaffl formülü31 ile göreceli mRNA ekspresyon değerlerini hesaplayın.
7. Cas proteinlerini tespit etmek için immünofloresan
NOT: Cas proteinleri (CasP) bir His_tag kaynaştırılır.
- Maya hücresi hazırlığı
- Steril bir döngü kullanarak bazı maya hücrelerini seçin ve bunları 30 ° C'de 240 rpm'de bir gecede 5 mL YPD bakımından zengin ortamda kültürleyin. Sabahları, 20 mL taze YPD'ye 500 μL hücre çözeltisi ekleyin ve OD600 0.6'ya ulaşana kadar 30 ° C'de 240 rpm'de büyütün.
- 5 mL kültür alın ve 500 μL% 37 formaldehit ile karıştırın. Karışımın oda sıcaklığında (RT) 10 dakika kalmasına izin verin. Hücreleri 5 dakika boyunca 1.500 x g'de santrifüjleme ile toplayın.
- Süpernatantı çıkarın ve hücreleri 1 mL fiksasyon tamponu ile yeniden askıya alın (0.1 M KH 2 PO4, 0.5 M MgCl2,% 3.7 formaldehit, pH = 6.5). Hücre solüsyonunu RT'de 20 dakika tutun.
- Hücre çözeltisini 5 dakika boyunca 1.500 x g'de santrifüj edin. Süpernatantı atın ve hücreleri 4 μL beta-merkaptoetanol ve 4 μL lizat çözeltisi (5 mg / mL) ile desteklenmiş 1 mL yıkama tamponunda (0.1 M KH 2 PO4,1.2M sorbitol, pH = 6.5) yeniden askıya alın. Hücre solüsyonunu 20 dakika boyunca 37 ° C'de bir inkübatöre koyun.
- Hücre çözeltisini 5 dakika boyunca 1.500 x g'de santrifüj edin ve süpernatanı atın. Hücre peletini santrifüjleme ile 1 mL PBS ile iki kez yıkayın (5 dakika boyunca 1.500 x g ).
- Hücreleri 100 μL PBS artı % 0.05 Tween 20 içinde yeniden askıya alın ve 4 μL BSA çözeltisi (10 mg / mL) ekleyin. Hücre solüsyonunu RT'de 20 dakika tutun.
- Primer antikor ile inkübasyon
- Anti-His etiketi birincil antikoru karışıma adım 7.1.6'da 1:400 seyreltmede ekleyin. Çözeltiyi RT'de 2 saat tutun.
- Karışımı adım 7.2.1'de 1.500 x g'de 5 dakika boyunca santrifüj edin ve süpernatanı çıkarın. Hücre peletini yıkamak için 1 mL PBST ve santrifüj (1.500 x g) 5 dakika ekleyin. Bu işlemi iki kez tekrarlayın. Son olarak, süpernatantı atın ve hücreleri 100 μL 1x PBST'de askıya alın.
- Mikroskopi hücre tespiti
- Hücreleri bir slayda monte edin; adım 7.2.2'den itibaren hücre çözeltisinin 2 μL'sini alın ve bir cam slayt üzerine yerleştirin. Bir kapak fişi ile örtün.
- Hücreleri floresan mikroskobu altında gözlemleyin. Floresan ışık kaynağını, mikroskobu ve bilgisayarı açın. Floresan ışık kaynağı numarasını yazın ve bilgisayardaki mikroskop yazılımını açın.
- Slaytı mikroskop aşamasına getirin. 40x objektif lensi seçin ve hücreleri yeşil ışık altında (550 nm) gözlemleyin. Maya hücrelerinin konturu görünene kadar kaba odak düğmesini hareket ettirin. Hücreleri odaklamak için ince netleme düğmesini hareket ettirin.
- Mikroskop yazılımı ile hücreleri tespit edin. Mikroskop görüş alanını kapatın ve bilgisayar ekranına geçin. Canlı'ya tıklayın, 3-5 saniye bekleyin ve fotoğraf çekmek için Yakala'ya tıklayın. Resmi kaydedin.
- Bilgisayarı, mikroskobu ve floresan ışık kaynağını kapatın.
8. Veri toplama: FACS
NOT: Yeşil floresan, akış sitometrisi (yani, floresan ile aktive edilmiş hücre sıralama [FACS] ölçümleri) ile tespit edilir. Maya hücreleri, FACS deneylerini yürütmek için genel olarak 30 ° C ve 240 rpm'de kültürlenir. Bununla birlikte, hücreler genetik içeriklerine bağlı olarak bazı önlemler talep edebilirler. dCas12a-VPR genini içeren hücreler ( GPD kurucu promotörü tarafından kontrol edilir) SDC çözeltisinde 24 saat boyunca büyütülmelidir. Daha sonra, hücreler taze SDC'de 1:100 oranında seyreltilir ve floresan yoğunluğunu ölçmeden önce 12 saat daha büyütülür. AcrIIA4-HBD (hER) geni ile modifiye edilmiş hücreler de seyreltme gerektirir. Ayrıca, OD600'ün kontrol edilmesi gerekir. İlk olarak, hücrelerin bir gecede SDC'de büyümesine izin verilir (14 saatten fazla). Sabahları, OD600 ölçülür. Daha sonra kültür, OD600 = 0.1'e kadar çeşitli bir β-östradiol konsantrasyonu ile beslenen SDC'de seyreltilir. FACS deneylerinden önce, hücreler OD600'ün 0.8-1.0'a ulaşacağı şekilde 7 saat daha büyütülür. dCas9-VP64 veya dCas12a-VP64 eksprese eden hücreler, FACS makinesindeki ölçümlerden önce seyreltme ve daha fazla büyüme olmadan SDC'de 20-24 saat boyunca yetiştirilir.
- Lazeri ısıtmak için ölçümlerden 20 dakika önce FACS makinesini açın.
- Numuneleri hazırlayın (seyreltme): 20 μL hücre kültürünü 300 μLddH2O ile karıştırın.
- FACS makinesine bağlı bilgisayarda FACS yazılımını çalıştırın ve yeni bir deneme oluşturun. Ölçüm parametrelerini ayarlayın (örneğin, FSC(SSC)-A/H/W ve histogram).
- Numunelerin uyarma ve emisyon dalga boylarına göre filtreyi seçin. Örneğin, buradaki hedef protein, uyarma ve emisyon dalga boyları sırasıyla 488 nm ve 507 nm olan yEGFP'dir. Bu nedenle, FITC veya GFP filtresini seçin (uyarma dalga boyu: 488 nm; emisyon dalga boyu: 527/32 nm). Alma hücresi numarasını 10.000 olarak ayarlayın.
- Floresan boncukların yoğunluğunu ölçerek FITC filtre voltajını ayarlayın. İki ardışık deney arasındaki boncukların yoğunluğundaki nispi farkın% 5'i geçmediğinden emin olun.
- Olası fazla boncukları çıkarmak için makineyi ddH2O ile birkaç saniye yıkayın.
- Numune floresan yoğunluğunu ölçün. Önizleme'ye tıklayın ve numune enjeksiyon stabilitesi için 3-5 saniye bekleyin. Son olarak, Al'a tıklayın.
- Deneyin sonunda boncukları tekrar ölçün. Gerilim, ilk boncukların ölçümü sırasında kullanılan voltajdır (bkz. adım 8.4, 438-441 V). İki boncuğun ölçümleri arasındaki nispi farkın% 5'i aşıp aşmadığını kontrol edin.
- FACS verilerini FCS dosyaları olarak dışa aktarın.
- FCS dosyalarını R Studio yazılımı ile analiz edin.
9. Veri analizi
NOT: R stüdyosu içinde Flowcore R Bioconductor paket32'yi kullanın. FCS dosyaları R dilinde yazılmış bir komut dosyası kullanılarak analiz edildi.
- R studio'yu açın ve Bdverse analizi betiğini yükleyin. FCS dosyalarını analiz etmek için R.
- Deneme adını (ename), FCS dosyalarının bulunduğu dizini (yol) ayarlayın
Depolanan (dir_d) ve sonuç dosyalarının oluşturulacağı yer (dir_r). - Floresan kanalını ayarlayın. Örneğin, yeşil floresan ölçülmüşse select_ch = "GPA-A".
- Ölçülen numune sayısını ayarlayın (s_num).
- Her nokta grafiğinin boyutlarını ayarlayın (sxlim, sylim). Çubuk grafikler ve kutu grafikleri (xlimit, ylimit) için x ve y ekseninin maksimum uzunluğunu ayarlayın. xLimit s_num'den büyük veya ona eşit olmalıdır.
- İlgili satırlardan # işaretini kaldırarak geçit yöntemini seçin.
NOT: morphGate, komut dosyası tarafından gerçekleştirilen otomatik bir geçit yöntemidir (yani, hücrelerin daha yoğun olduğu nokta grafiklerinin bölgesi program tarafından tanınır ve seçilir). polygonGate ve rectangleGate, nokta grafiklerine bakmayı ve bir çokgenin köşelerini veya hücrelerin çoğunun bulunduğu bölgeyi kucaklayan bir dikdörtgenin kenarını tanımlamayı gerektirir. - Seçilen geçit yöntemine karşılık gelen, geçitli flowSet nesnesini seçin. Nokta grafiği çözünürlüğünü seçin (res; en az 256'ya eşit olmalıdır).
- Floresanın negatif olduğu ölçümleri kaldırmak için flt_low <- filter_low(sp) açıklamasını kaldırın. Diğer denemelerden kaynaklanan aykırı değerleri kaldırmak için flt_sp <- filtresi(flt_lw) açıklamasını kaldırın.
- Kaynak tuşuna basın ve komut dosyasını çalıştırın. Analiz sonuçlarını içeren tüm dosyalar dir_r oluşturulur.
Sonuçlar
RNA polimeraz III-tipi promotör tarafından sgRNA/crRNA ekspresyonu
İlk olarak, bu çalışma Şekil 1A'da gösterilen transkripsiyonel aktivasyon devresinin (devre 1) mühendisliğini ele almıştır. Üç temel bileşen içeriyordu: 1) dCas9 / dCas12a-AD için hedef bölgeler sağlayan bir dizi farklı sentetik promotörden önce gelen yEGFP (raporlayıcı) için kodlayan gen; 2) dCas9 veya dCas12a'nın maya kodonu için optimize edilmiş bir versiyonu, bir aktivasyon...
Tartışmalar
Protokol, "Tasarla-Yap-Test Et-Öğren" (DBTL) biyolojik mühendislik döngüsünü takiben ve hem kuru laboratuvar hem de ıslak laboratuvar deneyleriyle ilgili sentetik gen dijital devreleri için olası bir tam iş akışı gösterdi. Burada, CRISPR-Cas sistemine, özellikle dSpCas9, denAsCas12a, dLbCas12a ve buna karşılık gelen anti-CRISPR proteinlerine, S. cerevisiae küçük transkripsiyonel ağlarını tasarlayarak ve inşa ederek odaklandık. Bazıları dijital devrelerin temel bileşenleri olan Boole...
Açıklamalar
Yazarlar rakip finansal çıkar beyan etmemektedir.
Teşekkürler
Sentetik Biyoloji laboratuvarı SPST, TJU'nun tüm öğrencilerine genel yardımları için, Zhi Li ve Xiangyang Zhang ile birlikte FACS deneylerindeki yardımları için teşekkür etmek istiyoruz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | - | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | - | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | - | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | - | - | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
Referanslar
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır