Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Circuitos digitales genéticos basados en sistemas CRISPR-Cas y proteínas anti-CRISPR
* Estos autores han contribuido por igual
En este artículo
Resumen
Los sistemas CRISPR-Cas y las proteínas anti-CRISPR se integraron en el esquema de puertas booleanas en Saccharomyces cerevisiae. Los nuevos circuitos lógicos pequeños mostraron un buen rendimiento y profundizaron la comprensión de los factores de transcripción basados en dCas9 / dCas12a y las propiedades de las proteínas anti-CRISPR.
Resumen
Las puertas booleanas de genes sintéticos y los circuitos digitales tienen una amplia gama de aplicaciones, desde el diagnóstico médico hasta el cuidado del medio ambiente. El descubrimiento de los sistemas CRISPR-Cas y sus inhibidores naturales, las proteínas anti-CRISPR (Acrs), proporciona una nueva herramienta para diseñar e implementar circuitos digitales de genes in vivo . Aquí, describimos un protocolo que sigue la idea del ciclo de ingeniería biológica "Diseñar-Construir-Probar-Aprender" y hace uso de dCas9/dCas12a junto con sus correspondientes Acrs para establecer pequeñas redes transcripcionales, algunas de las cuales se comportan como puertas booleanas, en Saccharomyces cerevisiae. Estos resultados señalan las propiedades de dCas9/dCas12a como factores de transcripción. En particular, para lograr la activación máxima de la expresión génica, dSpCas9 necesita interactuar con un ARN de andamio diseñado que recolecta múltiples copias del dominio de activación VP64 (AD). Por el contrario, dCas12a se fusionará, en el extremo C, con el fuerte VP64-p65-Rta (VPR) AD. Además, la actividad de ambas proteínas Cas no se ve reforzada por el aumento de la cantidad de sgRNA/crRNA en la célula. Este artículo también explica cómo construir puertas booleanas basadas en la interacción CRISPR-dCas-Acr. El dominio de unión a hormonas fusionado AcrIIA4 del receptor de estrógeno humano es el núcleo de una puerta NOT que responde al β-estradiol, mientras que los AcrVA sintetizados por el promotor GAL1 inducible permiten imitar las puertas SÍ y NO con galactosa como entrada. En estos últimos circuitos, AcrVA5, junto con dLbCas12a, mostró el mejor comportamiento lógico.
Introducción
En 2011, los investigadores propusieron un método computacional y desarrollaron una pieza correspondiente de software para el diseño automático de circuitos genéticos sintéticos digitales1. Un usuario tenía que especificar el número de entradas (tres o cuatro) y rellenar la tabla de verdad del circuito; Esto proporcionó toda la información necesaria para derivar la estructura del circuito utilizando técnicas de la electrónica. La tabla de verdad se tradujo a dos fórmulas booleanas a través del método 2 del mapa de Karnaugh. Cada fórmula booleana está hecha de cláusulas que describen operaciones lógicas (suma o multiplicación) entre (parte de) las entradas del circuito y sus negaciones (los literales). Las cláusulas, a su vez, se suman (OR) o se multiplican (AND) para calcular la salida del circuito. Cada circuito se puede realizar de acuerdo con cualquiera de sus dos fórmulas correspondientes: una escrita en forma POS (producto de sumas) y la otra en representación SOP (suma de productos). El primero consiste en una multiplicación de cláusulas (es decir, puertas booleanas) que contienen una suma lógica de los literales. Este último, en cambio, es una suma de cláusulas donde los literales se multiplican.
Los circuitos eléctricos se pueden realizar, en una placa de pruebas, conectando físicamente diferentes puertas juntas. La corriente eléctrica permite el intercambio de señales entre puertas, lo que conduce al cálculo de la salida.
En biología, la situación es más compleja. Una puerta booleana se puede realizar como una unidad de transcripción (TU; es decir, la secuencia "promotor-codificante de la región-terminador" dentro de las células eucariotas), donde la transcripción o la traducción (o ambas) están reguladas. Así, al menos dos tipos de moléculas establecen un cableado biológico: las proteínas del factor de transcripción y los ARN antisentido no codificantes1.
Un circuito digital de genes está organizado en dos o tres capas de puertas, a saber: 1) la capa de entrada, que está hecha de puertas SÍ (tampón) y NO y convierte los productos químicos de entrada en moléculas de cableado; 2) la capa interna, que consiste en tantas TU como cláusulas hay en la fórmula booleana correspondiente. Si el circuito está diseñado de acuerdo con la fórmula SOP, cada cláusula en la capa interna producirá la salida del circuito (por ejemplo, fluorescencia) en una llamada arquitectura de salida distribuida. Si se utiliza la fórmula del producto de suma (POS), entonces se requiere una capa final 3), que contendrá una sola puerta multiplicativa que recoge las moléculas de cableado de la capa interna.
En general, en biología sintética, se pueden diseñar muchos esquemas diferentes para el mismo circuito. Difieren en el número y el tipo de TU y moléculas de cableado. Para elegir la solución más fácil de implementar en celdas de levadura, cada diseño de circuito se asocia con una puntuación de complejidad S, definida como
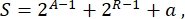
donde A representa el número de activadores, R representa el número de represores y a es la cantidad de moléculas de ARN antisentido. Si los activadores o represores están ausentes del circuito, su contribución a S es cero. Por lo tanto, es más difícil realizar un esquema de circuito en el laboratorio (S alta) cuando requiere un alto número de factores de transcripción ortogonales. Esto significa que los nuevos activadores y represores deben diseñarse de novo para realizar el cableado completo dentro de los circuitos digitales. En principio, las nuevas proteínas de unión al ADN se pueden ensamblar utilizando las proteínas Zinc Finger3 y los efectores TAL4 como plantillas. Sin embargo, esta opción parece demasiado ardua y lenta; por lo tanto, uno debe confiar principalmente en pequeños ARN y regulación de traducción para finalizar circuitos genéticos complejos.
Originalmente, este método fue desarrollado para fabricar circuitos digitales en bacterias. De hecho, en las células eucariotas, en lugar de ARN antisentido, es más adecuado hablar de microARN (miARN) o pequeños ARN interferentes (siRNAs)5. Sin embargo, la vía del ARNi no está presente en la levadura S. cerevisiae. Por lo tanto, uno debe optar por redes completamente transcripcionales. Supongamos que un circuito necesita cinco activadores y cinco represores; su puntuación de complejidad sería S = 32. La complejidad del circuito puede reducirse reemplazando los 10 factores de transcripción con un solo dCas96 (Cas9 deficiente en nucleasa) fusionado a un dominio de activación (AD). Como se muestra en7, dCas9-AD funciona como un represor en la levadura cuando se une a un promotor entre la caja TATA y el TSS (sitio de inicio de la transcripción) y como un activador cuando se une bien aguas arriba de la caja TATA. Por lo tanto, se pueden reemplazar 10 factores de transcripción con una sola proteína de fusión dCas9-AD y 10 sgRNAs (ARN guía únicos) para una puntuación de complejidad total de S = 11. Es rápido y fácil sintetizar diez sgRNAs, mientras que, como se comentó anteriormente, el ensamblaje de 10 proteínas exigiría un trabajo mucho más largo y complicado.
Alternativamente, se podrían usar dos proteínas dCas ortogonales (por ejemplo, dCas9 y dCas12a): una para fusionarse con un AD y la otra desnuda o en combinación con un dominio de represión. La puntuación de complejidad aumentaría en una sola unidad (S = 12). Por lo tanto, los sistemas CRISPR-dCas son la clave para la construcción de circuitos digitales de genes muy intrincados en S. cerevisiae.
Este documento caracteriza profundamente la eficiencia de los represores y activadores basados en dCas9 y dCas12a en levadura. Los resultados muestran que no demandan una gran cantidad de sgRNA para optimizar su actividad, por lo que los plásmidos episomales se evitan preferentemente. Además, los activadores basados en dCas9 son mucho más efectivos cuando se usa un ARN de andamio (scRNA) que recluta copias del VP64 AD. Por el contrario, dCas12a funciona bien cuando se fusiona directamente con el fuerte VPR AD. Además, un promotor activado sintético exige un número variable de sitios diana, dependiendo de la configuración del activador (por ejemplo, tres cuando se utiliza dCas12a-VPR, seis para dCas9-VP64 y solo uno con dCas9 y un scRNA). Como represor, dCas12a parece más incisivo cuando se une a la región codificante que al promotor.
Sin embargo, como inconveniente, CRISPR-dCas9 / dCas12a no interactúan directamente con los productos químicos. Por lo tanto, es posible que no sean útiles en la capa de entrada. Por esta razón, se han investigado diseños alternativos de puertas booleanas que contienen proteínas anti-CRISPR (Acrs). Acrs actúan sobre las proteínas (d)Cas e inhiben su funcionamiento8. Por lo tanto, son un medio para modular la actividad de los sistemas CRISPR-(d)Cas. Este artículo analiza a fondo las interacciones entre Acrs tipo II y (d)Cas9, así como Acrs tipo V y (d)Cas12a en S. cerevisiae. Dado que los Acr son mucho más pequeños que las proteínas Cas, se construyó una puerta NOT que responde al estrógeno β-estradiol fusionando el dominio de unión hormonal del receptor de estrógeno humano9-HBD (hER) con AcrIIA4. Además, se realizaron un puñado de puertas SÍ y NO que expresaban dCas12a (-AD) constitutivamente y AcrVA en la inducción con galactosa. En la actualidad, estas puertas sirven solo como prueba de concepto. Sin embargo, también representan el primer paso hacia un replanteamiento profundo del algoritmo para llevar a cabo el diseño automático computacional de circuitos digitales de genes sintéticos en células de levadura.
Protocolo
1. Diseño y construcción del casete de expresión sgRNA/crRNA
NOTA: Hay dos tipos de casetes de expresión de sgRNA/crRNA: SNR5210-está compuesto por el promotor SNR52 dependiente de la ARN polimerasa III, la secuencia sgRNA/crRNA y el terminador SUP4; otro, abreviado como RGR11, consiste en el promotor ADH1 dependiente de la ARN polimerasa II, la estructura RGR (ribozima-ribozima de ARN guía de ribozima) que contiene dos ribozimas (una ribozima de cabeza de martillo-HH y una ribozima del virus delta de la hepatitis HDV) y la secuencia del sgRNA / crRNA intermedio, y el terminador ADH1. Los homólogos de Cas9 que guían el sgRNA están formados por una secuencia espaciadora y la característica repetición directa12, mientras que el crRNA para las proteínas Cas12a comprende la repetición directa seguida de la secuencia espaciadora13,14 (véase la Tabla suplementaria 1 para todas las secuencias de ADN utilizadas en este estudio).
- Diseñar la secuencia espaciadora para la activación transcripcional mediada por Cas9/Cas12a.
- Aproveche la secuencia del operador lex bacteriano (llamada lexOp) para que sea el sitio objetivo 15,16 e insértela en el promotor CYC1 que impulsa la expresión de la proteína fluorescente verde mejorada con levadura (yEGFP)17. Por lo tanto, la secuencia espaciadora está definida y es complementaria al lexOp insertado.
- Compruebe la ortogonalidad de la secuencia espaciadora mediante la herramienta CRISPRDIRECT18.
- Pegue la secuencia lexOp flanqueada por la secuencia PAM en el campo de texto, defina la secuencia PAM como NRG para dCas9 y TTTV para dCas12a, y especifique las especies de la lista desplegable como Levadura en ciernes (Saccharomyces cerevisiae) Genoma S288C. Haga clic en Diseño. Asegúrese de que no haya un sitio de destino coincidente en 20mer + PAM ni en la búsqueda de 12mer + PAM.
- Construir el casete de expresión sgRNA/crRNA.
- Utilice PCR de toma para amplificar las secuencias de ADN de las partes biológicas estándar, como promotores, secuencias codificantes y terminadores.
- Preparar una mezcla de reacción que contenga: 20-40 ng de plantilla de ADN, 1 μL de cebador directo de 10 μM (es decir, ot25, construcción de plásmido de expresión de sgRNA/crRNA), 1 μL de cebador inverso de 10 μM (es decir, ot26, construcción de plásmido de expresión de sgRNA/crRNA), 5 μL de mezcla de 2,5 mM dNTP, 0,5 μL de ADN polimerasa, 10 μL de 5x tampón de reacción de ADN polimerasa, y agua destilada doble (ddH2O) hasta un volumen total de 50 μL.
NOTA: Consulte la Tabla suplementaria 2 para obtener una lista de cebadores utilizados en este estudio. - Ejecute el programa de PCR de toma de contacto en un termociclador:
Etapa 1: 98 °C durante 30 s.
Etapa 2 con 10 ciclos: 98 °C durante 10 s, 68 °C durante 20 s y 72 °C durante 15 s.
Etapa 3 con 25 ciclos: 98 °C durante 10 s, 59 °C durante 20 s y 72 °C durante 15 s.
Etapa 4: 72 °C durante 2 min.
Por último, mantener a 4 °C hasta el inicio de los experimentos de seguimiento.
NOTA: Los 68 °C en la etapa 2 y los 59 °C en la etapa 3 dependen de las temperaturas de fusión de los cebadores directos e inversos, que varían de diferentes pares de cebadores. El tiempo a 72 °C en las etapas 2 y 3 está determinado por la longitud del producto de PCR y la velocidad de la ADN polimerasa.
- Preparar una mezcla de reacción que contenga: 20-40 ng de plantilla de ADN, 1 μL de cebador directo de 10 μM (es decir, ot25, construcción de plásmido de expresión de sgRNA/crRNA), 1 μL de cebador inverso de 10 μM (es decir, ot26, construcción de plásmido de expresión de sgRNA/crRNA), 5 μL de mezcla de 2,5 mM dNTP, 0,5 μL de ADN polimerasa, 10 μL de 5x tampón de reacción de ADN polimerasa, y agua destilada doble (ddH2O) hasta un volumen total de 50 μL.
- Aislar los productos de PCR mediante electroforesis en gel (100 V, 30 min). Eluya las secuencias de ADN del gel de agarosa a través de un kit de extracción de gel de ADN (consulte la Tabla de materiales).
NOTA: Se requiere un gel de agarosa al 0,8% para fragmentos de más de 500 nt, un gel de agarosa al 1,5% para fragmentos entre 100 nt y 500 nt, y un gel de agarosa al 2% para fragmentos de menos de 100 nt. - Insertar la TU que expresa sgRNA/crRNA en un vector lanzadera pRSII404/42419, que contiene el gen de resistencia a la ampicilina y el gen marcador auxotrofo seleccionable por levadura-TRP1.
- Digerir el vector lanzadera a 37 °C durante 1 h con las dos enzimas de restricción SacI y Acc65I. Preparar la mezcla de reacción añadiendo 5 μg del vector lanzadera, las cantidades recomendadas de enzimas, tampón de digestión (según las instrucciones de la enzima) yddH2Ohasta un volumen total de 30 μL.
- Verificar la digestión del vector lanzadera mediante electroforesis en gel (ver subpaso 1.3.2). A continuación, inactivar las dos enzimas a 65 °C durante 20 min.
- Utilice el método de ensamblaje 20 de Gibson para insertar los productos de PCR purificados en el vector lanzadera abierto de corte dejando entrar la mezcla de ADN equimolar a50 °C durante 1 h.
- Transforme células competentes de Escherichia coli DH5α con la mezcla de reacción de Gibson anterior mediante el método de transformación de choque térmico21. Transfiera las células transformadas de E. coli a placas de agar Luria-Bertani (LB) que contienen ampicilina (0,1 g / L). Coloque las placas en la incubadora a 37 ° C y deje que las células crezcan durante la noche.
- Recoger cuatro colonias de la placa de agar LB y cultivarlas por separado durante la noche a 37 °C en una solución de LB que contenga ampicilina (0,1 g/L). Luego, use el procedimiento de mini preparación para extraer plásmidos de las células de E. coli 22.
- Utilice los cebadores ot18 y ot19 (véase la Tabla complementaria 2 para las secuencias oligo) para secuenciar y confirmar la unidad de transcripción insertada mediante el método de Sanger23.
NOTA: En experimentos posteriores, los plásmidos construidos y confirmados se insertarán en el genoma de la levadura a través del protocolo PEG / LiAc24.
- Utilice PCR de toma para amplificar las secuencias de ADN de las partes biológicas estándar, como promotores, secuencias codificantes y terminadores.
2. Diseño y construcción del cassette de expresión de ARN del andamio
NOTA: El ARN guía del andamio (scRNA) está compuesto por la secuencia sgRNA y las estructuras de horquilla MS225. En este trabajo se utilizan dos tipos de estructuras de horquilla MS2: horquilla MS2 de tipo salvaje y aptámero-f6 de proteína de recubrimiento MS2 f6 (MCP).
- Sintetice una plantilla de scRNA que pueda acomodar diferentes secuencias espaciadoras (es decir, pSNR52-spacer_DR(SpCas9)-2×MS2(wt+f6)-SUP4t).
NOTA: En este estudio, la plantilla de scRNA fue sintetizada por una compañía de síntesis de genes (ver Tabla de materiales). - Diseñar cebadores adecuados (ver Tabla Suplementaria 2) para ejecutar PCR en las secuencias espaciadoras necesarias.
- Siga los procedimientos del paso 1.3 para construir un casete de expresión de scRNA.
3. Ingeniería de dSpCas9 y construcción de plásmidos de expresión
- Obtenga el plásmido pTPGI_dSpCas9_VP64 (consulte la Tabla de materiales).
- Construya el vector aceptor pRSII406-pGPD-ATG-XbaI-XhoI-CYC1t, basado en el vector lanzadera pRSII406, mediante PCR de toma de contacto y el método de ensamblaje de Gibson (consulte el paso 1.3). El plásmido proporciona un fuerte promotor constitutivo-pGPD, y un terminador-CYC1t.
- Digerir el plásmido pTPGI_dCas9_VP64 y el vector aceptor recién construido (5 μg durante 1 h o 10 μg durante la noche; véase el paso 1.3.3.1 como referencia) con XbaI y XhoI a 37 °C. Separar y purificar el fragmento de inserción y el vector aceptor como en el paso 1.3.2.
- Ligar el fragmento de inserto purificado y el vector aceptor con ADN ligasa T4 a 16 °C durante 8 h. Preparar la solución de ligadura añadiendo 50-100 ng del vector aceptor purificado, fragmentos diana purificados en cantidad equimolar con el vector aceptor, 1,5 μL de tampón T4, 0,5 μL de ligasa T4 yddH2Ohasta un volumen total de 15 μL.
- Siga los pasos 1.3.3.3 y 1.3.3.4. A continuación, confirmar que el plásmido recién construido es correcto mediante digestión con XbaI y Xhol (37 °C, 1 h) y electroforesis en gel (ver paso 1.3.2).
4. Ingeniería dCas12a y construcción de plásmidos
- Construir los plásmidos que expresan dCas12a-AD.
- Sintetizar dos proteínas dCas12a optimizadas para codones de levadura (denAsCas12a y dLbCas12a) flanqueadas por sitios de enzimas de restricción BamHI y Xhol.
NOTA: En este estudio, las dos proteínas dCas12a optimizadas para codones de levadura fueron sintetizadas por una compañía de síntesis de genes (ver Tabla de materiales). - Construya el vector aceptor pRSII406-promoter-ATG-NLS-GS-HIStag-GS-BamHI-sp-XhoI-AD-NLS-TAA-mTGUO1 mediante PCR de toma de contacto y el método de ensamblaje de Gibson (consulte el paso 1.3), donde el "promotor" es pGPD o pGAL1, "sp" representa una secuencia aleatoria corta y "AD" es VPR o VP64.
- Insertar cada proteína dCas12a en los dos vectores aceptores recién construidos mediante digestión con BamHI y XhoI y ligadura con ADN ligasa T4 (consultar los pasos 3.3 y 3.4).
- Sintetizar dos proteínas dCas12a optimizadas para codones de levadura (denAsCas12a y dLbCas12a) flanqueadas por sitios de enzimas de restricción BamHI y Xhol.
- Construir los plásmidos expresando un dCas12a desnudo.
- Construya un vector aceptor para los casetes de expresión inducibles por galactosa de dCas12a como pRSII406-Acc651-pGAL1-ATG-NLS-GS-HIStag-GS-BamHI-sp-XhoI-GS-NLS-TAA-CYC1t utilizando PCR de toma de contacto y el método de ensamblaje de Gibson (consulte el paso 1.3).
- Digiera los plásmidos que contienen las proteínas dCas12a y el vector aceptor anterior con BamHI y Xhol, luego ligarlos con ADN ligasa T4 para obtener el plásmido pRSII406-pGAL1-Acc651-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1t (consulte los pasos 3.3 y 3.4).
- Digerir el plásmido obtenido en el paso 4.2.2 con Acc65I y BamHI, y luego reemplazar pGAL1 con pGPD mediante PCR de toma de contacto y el método de ensamblaje de Gibson para construir pRSII406-pGPD-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1t.
5. Ingeniería de proteínas anti-CRISPR y construcción de plásmidos
NOTA: Se han empleado tres tipos de promotores para impulsar la expresión de Acrs: un promotor inducible-pGAL1, cuatro promotores constitutivos de levadura-pGPD, pACT1, pTEF1 y pTEF2, y un promotor constitutivo sintético-genCYC1t_pCYC1noTATA26.
- Obtener los plásmidos que contienen las secuencias de Acrs tipo II (AcrIIA2, AcrIIA427 y AcrIIA5 28) y Acrs tipo V-A (AcrVA1, AcrVA4 y AcrVA529) de una compañía de síntesis génica.
- Construir los plásmidos basados en el vector lanzadera pRSII403 para expresar Acrs.
- Construir casetes de expresión AcrIIA.
NOTA: Utilice la PCR de toma de contacto para amplificar cuatro promotores diferentes (es decir, pGPD, pACT1, pTEF2 y genCYC1t_pCYC1noTATA), los tres tipos de AcrIIA y dos terminadores (ADH1t y CYC1t). Construya una serie de TU que expresen AcrIIAs, bajo diferentes promotores, a través del método de ensamblaje de Gibson (consulte el paso 1.3). - Construir casetes de expresión AcrVA.
- Sintetizar la secuencia aceptora: ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6, donde "sp" es una secuencia aleatoria que será reemplazada por AcrVAs más adelante.
NOTA: En este estudio, las secuencias aceptoras fueron sintetizadas por una compañía de síntesis de genes (Tabla de Materiales). - Ensamble los vectores aceptores pRSII403-promoter-ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6, donde el "promotor" es: pGAL1, pTEF1 y genCYC1t_pCYC1noTATA. Utilice el método de ensamblaje Gibson (consulte el paso 1.3).
- Inserte cada uno de los tres AcrVA en el vector aceptor descrito en el paso 5.2.2.2 (mediante PCR de toma de contacto y el método de ensamblaje de Gibson [consulte el paso 1.3]) para construir un conjunto de plásmidos que produzcan AcrVA.
- Sintetizar la secuencia aceptora: ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6, donde "sp" es una secuencia aleatoria que será reemplazada por AcrVAs más adelante.
- Construir casetes de expresión AcrIIA.
- Diseñe aún más AcrIIA4 extendiendo su secuencia con las secuencias del enlazador GS y HBD(hER). Esto permite la construcción de un circuito sensible al β-estradiol.
- Utilice PCR de toma de contacto para obtener piezas GS-HBD y AcrIIA4 por separado (consulte el paso 1.3.1).
- Coloque AcrIIA4, GS-HBD y el vector lanzadera en la mezcla de Gibson y construya el plásmido completo mediante el método de Gibson (ver paso 1.3.3).
6. detección de crRNA: RT-qPCR y diseño de cebadores
NOTA: la detección de crRNA se logró a través de RT-qPCR, que se organiza en tres pasos.
- Realice la extracción y purificación de ARN de las células de levadura a través de un kit de ARN.
- Cultive células de levadura durante la noche en 2 ml de medio completo sintético definido (SDC, volumen de 1 L: 20 g de glucosa, 2 g de mezcla AA, 6,7 g de YNB, 396 mg de leucina, 79,2 mg de triptófano, 79,2 mg de histidina, y 79,2 mg de uracilo) utilizando una placa de 24 pocillos (240 rpm, 30 °C).
- Por la mañana, diluir el cultivo celular (1:100) en 2 ml de SDC fresco y seguir cultivando las células de levadura a 30 °C, 240 rpm, durante otras 4 h.
- Cosechar los 2 ml enteros de la solución celular y centrifugar a 20,238 x g durante 2 min. Retire el sobrenadante con cuidado ya que el pellet celular es pequeño.
- Extraiga el ARN de las células de levadura utilizando un kit de ARN.
- Compruebe la calidad del ARN.
- Prepara un gel de agarosa al 1%. Mezclar 5 μL de cada muestra de ARN con 1 μL de colorante de carga de ADN. Luego cargue la mezcla en el gel y ejecútelo.
- Asegúrese de que dos bandas claras a ~4,000 nt y ~2,000 nt, correspondientes al ARN ribosómico (25S / 18S), estén presentes en el gel. Se puede ver una banda borrosa adicional a ~ 80 nt para el ARNt.
- Utilice las muestras de ARN inmediatamente para la síntesis de ADNc o almacénelas a -80 °C para su uso futuro.
- Transcripción inversa: Utilice el método stem-loop 30 para formar la primera cadena de ADNc correspondiente al crRNA (40 nt). Para la transcripción inversa del sgRNA (casi 100 nt), el procedimiento es el mismo que para la síntesis de ADNc del gen de referencia ACT1.
NOTA: El método para la transcripción inversa del ARNcr es diferente del utilizado con el ARNg y el ARNm ACT1 . Dado que el ARNc es muy corto, se trató como un microARN y se utilizó el método de transcripción inversa de microARN (enfoque tallo-bucle) para obtener el ADNc correspondiente. Se utilizaron dos kits de síntesis de ADNc (un kit de bucle madre para el ARNc y un kit de transcripción inversa habitual para el gen ACT1 ) en la cuantificación del ARNcr. La misma cantidad de ARN se utilizó en los dos kits (ver Tabla de materiales) para hacer que los resultados del experimento fueran comparables. El cebador utilizado con el kit tallo-bucle se diseñó de acuerdo con la secuencia tallo-bucle y los últimos seis nucleótidos en el extremo 3' del crRNA (para el bucle madre y la secuencia de cebadores, ver Tabla complementaria 2).- Método de bucle madre para la transcripción inversa de crRNA
- Saque la plantilla de ARN, la imprimación y los amortiguadores del congelador y déjelos derretir en hielo.
- Eliminación del ADN genómico: Primero, prepare 10 μL de la mezcla de reacción de acuerdo con las instrucciones del kit. Poner la mezcla en un termociclador a 42 °C durante 2 min. Finalmente, transfiéralo al hielo.
- Síntesis de la primera cadena de ADNc: Preparar 20 μL de la mezcla de reacción añadiendo 10 μL de la mezcla del paso 6.2.1.2, 1 μL de cebador tallo-bucle (concentración de 2 μM), 2 μL de tampón de reacción RT 10x, 2 μL de mezcla enzimática de transcripción inversa (que contiene la transcriptasa inversa) y 5 μL deH2Olibre de RNasa.
- Coloque la mezcla de reacción en un termociclador y ejecute el siguiente programa: 25 °C durante 5 min, 50 °C durante 15 min y 85 °C durante 5 min. Utilice el producto para la reacción de qPCR inmediatamente o guárdelo a -80 °C.
- Transcripción inversa de ARNm de sgRNA y ACT1
- Primera reacción: Prepare una mezcla de 13 μL que consista en la mezcla de cebador, la mezcla de dNTP, la plantilla de ARN (50 pg-5 μg) y agua sin RNasa (aparte de la plantilla de ARN, todas proporcionadas por el kit), de acuerdo con las instrucciones del kit. Utilice 1 μg de plantilla de ARN. Poner la mezcla en un termociclador a 70 °C durante 10 min.
- Segunda reacción (síntesis de ADNc): Prepare la mezcla de reacción añadiendo los reactivos (como se describe en las instrucciones del kit) a los 13 μL de la primera solución de reacción hasta un volumen final de 20 μL. Coloque la mezcla en un termociclador durante 15 min a 50 °C y luego durante 5 min adicionales a 85 °C. Utilice el producto para la reacción de qPCR inmediatamente o guárdelo a -80 °C.
- Método de bucle madre para la transcripción inversa de crRNA
- Kit SYBR para qPCR: detección de valores de Ct
NOTA: El cebador inverso utilizado en la qPCR del ARNcr es fijo porque corresponde al complemento inverso de la secuencia tallo-asa (ver Tabla complementaria 2). El cebador directo, por el contrario, es variable y depende de la secuencia del crRNA. Los cebadores directos e inversos para la qPCR de ARNm de sgRNA y ACT1 están diseñados a https://www.ncbi.nlm.nih.gov/tools/primer-blast/. Se seleccionan dos imprimaciones cuando la diferencia entre sus temperaturas de fusión no es superior a 2 °C (véase el cuadro complementario 2). Cada muestra se mide en tres réplicas.- Prepare la mezcla de reacción qPCR de acuerdo con las instrucciones del fabricante para el kit SYBR.
- Establezca el siguiente programa de qPCR en una máquina de PCR en tiempo real.
Preincubación: 10 min a 95 °C.
Etapa de PCR: 15 s a 95 °C, seguida de 34 s a 55 °C. Ajuste el ciclo de la etapa de PCR a 45 veces. Etapa de fusión: 10 s a 95 °C, seguida de 60 s a 65 °C, y finalmente 1 s a 97 °C. - Calcule los valores relativos de expresión de ARNm mediante la fórmula31 de Pfaffl.
7. Inmunofluorescencia para detectar proteínas Cas
NOTA: Las proteínas Cas (CasP) se fusionan a un His_tag.
- Preparación de células de levadura
- Recoger algunas células de levadura utilizando un bucle estéril y cultivarlas en 5 ml de medio rico en YPD durante la noche a 240 rpm a 30 °C. Por la mañana, añadir 500 μL de la solución celular a 20 ml de YPD fresco y cultivarlos a 240 rpm a 30 °C hasta que el OD600 alcance 0,6.
- Tomar 5 mL del cultivo y mezclarlo con 500 μL de formaldehído al 37%. Deje que la mezcla permanezca a temperatura ambiente (RT) durante 10 min. Cosechar las células por centrifugación a 1.500 x g durante 5 min.
- Retirar el sobrenadante y resuspender las células con 1 mL de tampón de fijación (0,1 M KH 2 PO4, 0,5 M MgCl2, formaldehído al 3,7%, pH = 6,5). Mantenga la solución celular en RT durante 20 min.
- Centrifugar la solución celular a 1.500 x g durante 5 min. Desechar el sobrenadante y resuspender las células en 1 ml de tampón de lavado (0,1 M KH 2 PO 4,1,2M de sorbitol, pH = 6,5) suplementado con 4 μL de beta-mercaptoetanol y4 μL de solución de lisado (5 mg/ml). Colocar la solución celular en una incubadora a 37 °C durante 20 min.
- Centrifugar la solución celular a 1.500 x g durante 5 min y desechar el sobrenadante. Lavar el pellet celular dos veces con 1 ml de PBS por centrifugación (1.500 x g durante 5 min).
- Resuspender las células en 100 μL de PBS más 0,05% Tween 20 y añadir 4 μL de solución BSA (10 mg/ml). Mantenga la solución celular en RT durante 20 min.
- Incubación con anticuerpos primarios
- Añadir el anticuerpo primario Anti-His tag a la mezcla en el paso 7.1.6 con una dilución de 1:400. Mantener la solución en RT durante 2 h.
- Centrifugar la mezcla en el paso 7.2.1 a 1.500 x g durante 5 min y retirar el sobrenadante. Añadir 1 ml de PBST y centrifugar (1.500 x g) durante 5 min para lavar el pellet celular. Repita esta operación dos veces. Finalmente, deseche el sobrenadante y suspenda las células en 100 μL de 1x PBST.
- Detección de células de microscopía
- Monte las celdas en una diapositiva; Tomar 2 μL de la solución celular del paso 7.2.2 y colocarla sobre un portaobjetos de vidrio. Cúbralo con un cubreobjetos.
- Observe las células bajo un microscopio de fluorescencia. Encienda la fuente de luz fluorescente, el microscopio y la computadora. Anote el número de fuente de luz fluorescente y abra el software del microscopio en la computadora.
- Coloque el portaobjetos en la plataforma del microscopio. Elija la lente del objetivo 40x y observe las celdas bajo la luz verde (550 nm). Mueva la perilla de enfoque grueso hasta que aparezca el contorno de las células de levadura. Mueva la perilla de enfoque fino para enfocar las celdas.
- Detecte las células con el software del microscopio. Cierre el campo de visión del microscopio y cambie a la pantalla del ordenador. Haga clic en En vivo, espere 3-5 s y haga clic en Capturar para tomar una foto. Guarde la imagen.
- Apague la computadora, el microscopio y la fuente de luz fluorescente.
8. Adquisición de datos: FACS
NOTA: La fluorescencia verde se detecta a través de la citometría de flujo (es decir, mediciones de clasificación celular activada por fluorescencia [FACS]). Las células de levadura se cultivan, en general, a 30 °C y 240 rpm para realizar experimentos FACS. Sin embargo, las células pueden exigir algunas precauciones dependiendo de su contenido genético. Las células que contienen el gen dCas12a-VPR (controlado por el promotor constitutivo GPD ) deben cultivarse durante 24 h en la solución SDC. Después, las células se diluyen en una proporción de 1:100 en SDC fresco y se cultivan durante otras 12 h antes de medir la intensidad de fluorescencia. Las células modificadas con el gen AcrIIA4-HBD (hER) también requieren dilución. Además, OD600 necesita ser controlado. Primero, se permite que las células crezcan en SDC durante la noche (más de 14 h). Por la mañana, se mide OD600 . Luego, el cultivo se diluye en SDC, suministrado con una concentración diversa de β-estradiol, hasta OD600 = 0.1. Antes de los experimentos FACS, las células se cultivan durante otras 7 h de modo que OD600 alcanza 0.8-1.0. Las células que expresan dCas9-VP64 o dCas12a-VP64 se cultivan en SDC durante 20-24 h sin dilución y crecimiento adicional antes de las mediciones en la máquina FACS.
- Encienda la máquina FACS 20 minutos antes de las mediciones para calentar el láser.
- Preparar las muestras (dilución): mezclar 20 μL del cultivo celular con 300 μL deddH2O.
- Ejecute el software FACS en el ordenador conectado a la máquina FACS y cree un nuevo experimento. Establezca los parámetros de medición (es decir, FSC (SSC) - A / H / W e histograma).
- Seleccione el filtro de acuerdo con las longitudes de onda de excitación y emisión de las muestras. Por ejemplo, la proteína objetivo aquí es yEGFP, cuyas longitudes de onda de excitación y emisión son 488 nm y 507 nm, respectivamente. Por lo tanto, seleccione el filtro FITC o GFP (longitud de onda de excitación: 488 nm; longitud de onda de emisión: 527/32 nm). Establezca el número de celda de adquisición en 10.000.
- Ajuste el voltaje del filtro FITC midiendo la intensidad de las perlas fluorescentes. Asegúrese de que la diferencia relativa en la intensidad de las perlas entre dos experimentos consecutivos no supere el 5%.
- Lave la máquina conddH2Odurante unos segundos para eliminar cualquier posible exceso de perlas.
- Mida la intensidad de fluorescencia de la muestra. Haga clic en Vista previa y espere 3-5 s para la estabilidad de la inyección de la muestra. Finalmente, haga clic en Adquirir.
- Mide las cuentas de nuevo al final del experimento. El voltaje es el que se utilizó durante la medición inicial de las perlas (ver paso 8.4, 438-441 V). Compruebe si la diferencia relativa entre las medidas de las dos perlas supera el 5%.
- Exporte los datos FACS como archivos FCS.
- Analice los archivos FCS con el software R Studio.
9. Análisis de datos
NOTA: Utilice el paquete Flowcore R Bioconductor 32 en R studio. Los archivos FCS se analizaron utilizando un script escrito en lenguaje R.
- Abra R studio y cargue el script Bdverse analysis. R para analizar los archivos FCS.
- Establezca el nombre del experimento (ename), el directorio (ruta) donde se encuentran los archivos FCS
Almacenado (dir_d) y donde se crearán los archivos de resultados (dir_r). - Ajuste el canal de fluorescencia. Por ejemplo, select_ch = "GPA-A" si se midió la fluorescencia verde.
- Establezca el número de muestras que se midieron (s_num).
- Establezca las dimensiones de cada diagrama de puntos (sxlim, sylim). Establezca la longitud máxima de los ejes x e y para gráficos de barras y diagramas de caja (xlimit, ylimit). xlimit debe ser mayor o igual que s_num.
- Elija el método de acceso eliminando el # de las líneas correspondientes.
NOTA: morphGate es un método de activación automática llevado a cabo por el script (es decir, la región de los diagramas de puntos donde las celdas son más densas es reconocida y seleccionada por el programa). polygonGate y rectangleGate exigen observar los diagramas de puntos y definir los vértices de un polígono o el lado de un rectángulo que abarca la zona donde se encuentran la mayoría de las celdas. - Seleccione el objeto flowSet gated, correspondiente al método de gating elegido. Seleccione la resolución del diagrama de puntos (res; debe ser al menos igual a 256).
- Quite el comentario flt_low <- filter_low(sp) para eliminar las mediciones en las que la fluorescencia es negativa. Uncomment flt_sp <- filter(flt_lw) para eliminar valores atípicos debido a otros experimentos.
- Presione Source y ejecute el script. Todos los archivos que contienen los resultados del análisis se crean en dir_r.
Resultados
Expresión de sgRNA/crRNA por un promotor de tipo ARN polimerasa III
Primero, este trabajo abordó la ingeniería del circuito de activación transcripcional (circuito 1) que se muestra en la Figura 1A. Contenía tres componentes básicos: 1) el gen que codifica para yEGFP (el informador), que fue precedido por una serie de diferentes promotores sintéticos que proporcionaron sitios objetivo para dCas9 / dCas12a-AD; 2) una versión optimizada para codones de levadura de d...
Discusión
El protocolo mostró un posible flujo de trabajo completo para los circuitos digitales de genes sintéticos, siguiendo el ciclo de ingeniería biológica "Diseñar-Construir-Probar-Aprender" (DBTL) y con respecto a los experimentos de laboratorio seco y laboratorio húmedo. Aquí, nos centramos en el sistema CRISPR-Cas, principalmente dSpCas9, denAsCas12a, dLbCas12a y las proteínas anti-CRISPR correspondientes, diseñando y construyendo en S. cerevisiae pequeñas redes transcripcionales. Algunos de ellos imitab...
Divulgaciones
Los autores declaran que no hay intereses financieros contrapuestos.
Agradecimientos
Queremos agradecer a todos los estudiantes del laboratorio de Biología Sintética -SPST, TJU- por su ayuda general, junto con Zhi Li y Xiangyang Zhang por su asistencia en los experimentos de FACS.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | - | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | - | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | - | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | - | - | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
Referencias
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados