É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Circuitos Digitais Gênicos Baseados em Sistemas CRISPR-Cas e Proteínas Anti-CRISPR
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Sistemas CRISPR-Cas e proteínas anti-CRISPR foram integrados ao esquema de portões booleanos em Saccharomyces cerevisiae. Os novos circuitos lógicos pequenos mostraram bom desempenho e aprofundaram a compreensão dos fatores de transcrição baseados em dCas9/dCas12a e das propriedades das proteínas anti-CRISPR.
Resumo
Os portões booleanos de genes sintéticos e os circuitos digitais têm uma ampla gama de aplicações, desde diagnósticos médicos até cuidados ambientais. A descoberta dos sistemas CRISPR-Cas e seus inibidores naturais - as proteínas anti-CRISPR (Acrs) - fornece uma nova ferramenta para projetar e implementar circuitos digitais gênicos in vivo . Aqui, descrevemos um protocolo que segue a ideia do ciclo de engenharia biológica "Design-Build-Test-Learn" e faz uso de dCas9/dCas12a juntamente com seus Acrs correspondentes para estabelecer pequenas redes transcricionais, algumas das quais se comportam como portões booleanos, em Saccharomyces cerevisiae. Estes resultados apontam as propriedades de dCas9/dCas12a como fatores de transcrição. Em particular, para alcançar a ativação máxima da expressão gênica, dSpCas9 precisa interagir com um RNA de andaime projetado que coleta múltiplas cópias do domínio de ativação VP64 (AD). Em contraste, dCas12a deve ser fundido, no terminal C, com o forte VP64-p65-Rta (VPR) AD. Além disso, a atividade de ambas as proteínas Cas não é aumentada pelo aumento da quantidade de sgRNA/crRNA na célula. Este artigo também explica como criar portas booleanas com base na interação CRISPR-dCas-Acr. O domínio de ligação hormonal fundido AcrIIA4 do receptor de estrogênio humano é o núcleo de uma porta NOT responsiva ao β-estradiol, enquanto AcrVAs sintetizados pelo promotor induzível GAL1 permite imitar ambas as portas SIM e NÃO com galactose como entrada. Nos últimos circuitos, AcrVA5, juntamente com dLbCas12a, apresentou o melhor comportamento lógico.
Introdução
Em 2011, pesquisadores propuseram um método computacional e desenvolveram um software correspondente para o projeto automático de circuitos gênicos sintéticos digitais1. Um usuário tinha que especificar o número de entradas (três ou quatro) e preencher a tabela verdade do circuito; Isso forneceu todas as informações necessárias para derivar a estrutura do circuito usando técnicas da eletrônica. A tabela verdade foi traduzida em duas fórmulas booleanas através do método do mapa de Karnaugh2. Cada fórmula booleana é feita de cláusulas que descrevem operações lógicas (soma ou multiplicação) entre (parte de) as entradas do circuito e suas negações (os literais). As cláusulas, por sua vez, são somadas (OR) ou multiplicadas (AND) para calcular a saída do circuito. Cada circuito pode ser realizado de acordo com qualquer uma de suas duas fórmulas correspondentes: uma escrita em forma de POS (produto de somas) e a outra em representação de SOP (soma de produtos). O primeiro consiste em uma multiplicação de orações (isto é, portas booleanas) que contêm uma soma lógica dos literais. Esta última, ao contrário, é uma soma de orações onde os literais são multiplicados.
Os circuitos elétricos podem ser realizados, em uma protoboard, conectando fisicamente diferentes portões juntos. A corrente elétrica permite a troca de sinais entre as comportas, o que leva ao cálculo da saída.
Na biologia, a situação é mais complexa. Uma porta booleana pode ser realizada como uma unidade de transcrição (TU; ou seja, a sequência "promotor-codificador de região-terminador" dentro de células eucarióticas), onde a transcrição ou tradução (ou ambas) são reguladas. Assim, pelo menos dois tipos de moléculas estabelecem uma fiação biológica: as proteínas do fator de transcrição e os RNAs antisenso não codificadores1.
Um circuito digital de genes é organizado em duas ou três camadas de portas, a saber: 1) a camada de entrada, que é feita de portas SIM (tampão) e NÃO e converte os produtos químicos de entrada em moléculas de fiação; 2) a camada interna, que consiste em tantas TUs quantas forem as cláusulas na fórmula booleana correspondente. Se o circuito for projetado de acordo com a fórmula SOP, cada cláusula na camada interna produzirá a saída do circuito (por exemplo, fluorescência) em uma chamada arquitetura de saída distribuída. Se o produto da fórmula da soma (POS) for usado, então uma camada final 3) é necessária, que conterá uma única porta multiplicativa coletando as moléculas de fiação da camada interna.
No geral, em biologia sintética, muitos esquemas diferentes podem ser projetados para o mesmo circuito. Eles diferem no número e no tipo de TUs e moléculas de fiação. A fim de escolher a solução mais fácil de ser implementada em células de levedura, cada projeto de circuito é associado a um escore de complexidade S, definido como
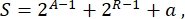
onde A representa o número de ativadores, R representa o número de repressores e a é a quantidade de moléculas de RNA antisense. Se ativadores ou repressores estiverem ausentes do circuito, sua contribuição para S é zero. Portanto, é mais difícil realizar um esquema de circuito em laboratório (S alto) quando ele requer um alto número de fatores de transcrição ortogonais. Isso significa que novos ativadores e repressores devem ser projetados de novo para realizar a fiação completa dentro dos circuitos digitais. Em princípio, novas proteínas ligadoras de DNA podem ser montadas usando as proteínas Zinc Finger3 e TAL efetores4 como modelos. No entanto, essa opção parece muito árdua e demorada; portanto, deve-se confiar principalmente em pequenos RNAs e regulação da tradução para finalizar circuitos gênicos complexos.
Originalmente, este método foi desenvolvido para fabricar circuitos digitais em bactérias. De fato, em células eucarióticas, em vez de RNAs antisense, é mais adequado falar em microRNAs (miRNAs) ou pequenos RNAs interferentes (siRNAs)5. Entretanto, a via RNAi não está presente na levedura S. cerevisiae. Assim, deve-se optar por redes totalmente transcricionais. Suponha que um circuito precise de cinco ativadores e cinco repressores; seu escore de complexidade seria S = 32. A complexidade do circuito pode ser reduzida substituindo os 10 fatores de transcrição por um único dCas96 (Cas9 deficiente em nuclease) fundido a um domínio de ativação (AD). Como mostrado em7, dCas9-AD funciona como um repressor em levedura ao ligar um promotor entre a caixa TATA e o TSS (local de início da transcrição) e como um ativador ao ligar bem a montante da caixa TATA. Assim, pode-se substituir 10 fatores de transcrição por uma única proteína de fusão dCas9-AD e 10 sgRNAs (RNAs guia únicos) por um escore de complexidade total de S = 11. É rápido e fácil sintetizar dez sgRNAs, enquanto, como comentado anteriormente, a montagem de 10 proteínas demandaria um trabalho muito mais longo e complicado.
Alternativamente, pode-se usar duas proteínas dCas ortogonais (por exemplo, dCas9 e dCas12a): uma para fundir-se a um AD e a outra nua ou em combinação com um domínio de repressão. O escore de complexidade aumentaria em apenas uma unidade (S = 12). Assim, os sistemas CRISPR-dCas são a chave para a construção de circuitos digitais gênicos muito intrincados em S. cerevisiae.
Este trabalho caracteriza profundamente a eficiência de repressores e ativadores à base de dCas9 e dCas12a em leveduras. Os resultados mostram que eles não demandam uma grande quantidade de sgRNA para otimizar sua atividade, de modo que plasmídeos epissomais são preferencialmente evitados. Além disso, ativadores baseados em dCas9 são muito mais eficazes quando se usa um RNA de andaime (scRNA) que recruta cópias do AD VP64. Em contraste, o dCas12a funciona bem quando fundido diretamente ao forte VPR AD. Além disso, um promotor ativado sintético demanda um número variável de locais alvo, dependendo da configuração do ativador (por exemplo, três quando se usa dCas12a-VPR, seis em dCas9-VP64 e apenas um com dCas9 e um scRNA). Como repressor, dCas12a parece mais incisivo ao ligar a região codificadora do que o promotor.
Como desvantagem, no entanto, CRISPR-dCas9/dCas12a não interagem diretamente com produtos químicos. Portanto, eles podem não ser úteis na camada de entrada. Por esta razão, projetos alternativos de portas booleanas contendo proteínas anti-CRISPR (Acrs) têm sido investigados. Os acrs atuam sobre as proteínas (d)Cas e inibem seu funcionamento8. Portanto, eles são um meio para modular a atividade dos sistemas CRISPR-(d)Cas. Este trabalho analisa minuciosamente as interações entre Acrs tipo II e (d)Cas9, bem como acrs tipo V e (d)Cas12a em S. cerevisiae. Uma vez que os Acrs são muito menores do que as proteínas Cas, uma porta NOT responsiva ao estrogênio β-estradiol foi construída pela fusão do domínio de ligação hormonal do receptor de estrogênio humano 9-HBD(hER)-ao AcrIIA4. Além disso, um punhado de portas SIM e NÃO que expressavam dCas12a(-AD) constitutivamente e AcrVAs na indução com galactose foram realizados. Atualmente, esses portões servem apenas como uma prova de conceito. No entanto, eles também representam o primeiro passo para um profundo repensar do algoritmo para realizar o projeto automático computacional de circuitos digitais de genes sintéticos em células de levedura.
Protocolo
1. Projeto e construção do de expressão de sgRNA/crRNA
NOTA: Existem dois tipos de de expressão de sgRNA/crRNA: um denominado SNR5210-é composto pelo promotor SNR52 dependente de RNA polimerase III, a sequência sgRNA/crRNA e o terminador SUP4; outra, abreviada como RGR11, consiste no promotor ADH1 dependente de RNA polimerase II, a estrutura RGR (ribozima-guia RNA-ribozima) que contém duas ribozimas (uma ribozima-HH e uma ribozima-HDV do vírus delta da hepatite) e a sequência do sgRNA/crRNA intermediário e o terminador ADH1. Os homólogos Cas9 que guiam o sgRNA são compostos por uma sequência espaçadora e a repetição direta característica12, enquanto o crRNA para proteínas Cas12a compreende a repetição direta seguida da sequência espaçadora13,14 (ver Tabela Suplementar 1 para todas as sequências de DNA usadas neste estudo).
- Projetar a sequência do espaçador para ativação transcricional mediada por Cas9/Cas12a.
- Aproveitar a sequência lex operator bacteriana (denominada lexOp) para ser o sítio alvo 15,16 e inseri-la no promotor CYC1 que impulsiona a expressão da proteína verde fluorescente reforçada por levedura (yEGFP)17. Assim, a sequência do espaçador é definida e complementar ao lexOp inserido.
- Verificar a ortogonalidade da sequência do espaçador através da ferramenta CRISPRDIRECT18.
- Cole a sequência lexOp flanqueada pela sequência PAM no campo de texto, defina a sequência PAM como NRG para dCas9 e TTTV para dCas12a e especifique a espécie da lista suspensa como Genoma S288C de levedura brotante (Saccharomyces cerevisiae). Clique em Design. Verifique se não há nenhum site de destino correspondente no 20mer+PAM nem na pesquisa 12mer+PAM.
- Construa o de expressão de sgRNA/crRNA.
- Use o touchdown PCR para amplificar as sequências de DNA de partes biológicas padrão, como promotores, sequências codificadoras e terminadores.
- Preparar uma mistura de reação contendo: 20-40 ng de molde de DNA, 1 μL de primer de 10 μM (isto é, ot25, construção de plasmídeo de expressão de sgRNA/crRNA), 1 μL de primer reverso de 10 μM (ou seja, ot26, construção de plasmídeo de expressão de sgRNA/crRNA), 5 μL de mistura de 2,5 mM dNTP, 0,5 μL de DNA polimerase, 10 μL de tampão de reação de DNA polimerase 5x, e água bidestilada (ddH2O) até um volume total de 50 μL.
NOTA: Consulte a Tabela Suplementar 2 para obter uma lista dos primers utilizados neste estudo. - Execute o programa PCR de touchdown em um termociclador:
Estágio 1: 98 °C por 30 s.
Estágio 2 com 10 ciclos: 98 °C por 10 s, 68 °C por 20 s e 72 °C por 15 s.
Estágio 3 com 25 ciclos: 98 °C por 10 s, 59 °C por 20 s e 72 °C por 15 s.
Estágio 4: 72 °C por 2 min.
Finalmente, manter a 4 °C até iniciar os experimentos de acompanhamento.
NOTA: Os 68 °C no estágio 2 e 59 °C no estágio 3 dependem das temperaturas de fusão dos primers forward e reverse, variando de diferentes pares de primers. O tempo a 72 °C nos estágios 2 e 3 é determinado pelo comprimento do produto da PCR e pela velocidade da DNA polimerase.
- Preparar uma mistura de reação contendo: 20-40 ng de molde de DNA, 1 μL de primer de 10 μM (isto é, ot25, construção de plasmídeo de expressão de sgRNA/crRNA), 1 μL de primer reverso de 10 μM (ou seja, ot26, construção de plasmídeo de expressão de sgRNA/crRNA), 5 μL de mistura de 2,5 mM dNTP, 0,5 μL de DNA polimerase, 10 μL de tampão de reação de DNA polimerase 5x, e água bidestilada (ddH2O) até um volume total de 50 μL.
- Isole os produtos da PCR por eletroforese em gel (100 V, 30 min). Eluir as sequências de ADN do gel de agarose através de um kit de extracção de gel de ADN (ver Tabela de Materiais).
NOTA: Um gel de agarose a 0,8% é necessário para fragmentos com mais de 500 nt, um gel de agarose a 1,5% para fragmentos entre 100 nt e 500 nt e um gel de agarose a 2% para fragmentos com menos de 100 nt. - Inserir o TU que expressa sgRNA/crRNA em um vetor de vaivém pRSII404/42419, que contém o gene de resistência à ampicilina e o gene do marcador auxotrófico selecionável por levedura - TRP1.
- Digerir o vetor shuttle a 37 °C por 1 h com as duas enzimas de restrição SacI e Acc65I. Preparar a mistura de reacção adicionando 5 μg do vector vaivém, as quantidades recomendadas de enzimas, tampão de digestão (de acordo com as instruções enzimáticas) e ddH2O até um volume total de 30 μL.
- Verificar a digestão do vector por electroforese em gel (ver subpasso 1.3.2). Em seguida, inative as duas enzimas a 65 °C por 20 min.
- Use o método de montagem de Gibson 20 para inserir os produtos purificados de PCR no vetor de vaivém aberto cortado, deixando entrar a mistura de DNA equimolar a50 °C por 1 h.
- Transformar células competentes de Escherichia coli DH5α com a mistura de reação de Gibson acima através do método de transformação de choque térmico21. Transferir as células de E. coli transformadas para placas de ágar Luria-Bertani (LB) contendo ampicilina (0,1 g/L). Coloque as placas na incubadora a 37 °C e deixe as células crescerem durante a noite.
- Colher quatro colónias da placa de ágar LB e cultivá-las separadamente durante a noite a 37 °C em solução LB contendo ampicilina (0,1 g/L). Em seguida, utilizar o procedimento de mini-preparo para extração de plasmídeos das células de E. coli 22.
- Utilizar os primers ot18 e ot19 (ver Tabela Suplementar 2 para sequências oligo) para sequenciar e confirmar a unidade de transcrição inserida através do método de Sanger23.
NOTA: Em experimentos posteriores, os plasmídeos construídos e confirmados serão inseridos no genoma da levedura através do protocolo PEG/LiAc24.
- Use o touchdown PCR para amplificar as sequências de DNA de partes biológicas padrão, como promotores, sequências codificadoras e terminadores.
2. Projeto e construção do de expressão de RNA do andaime
NOTA: O RNA guia do andaime (scRNA) é composto pela sequência de sgRNA e pelas estruturas hairpin MS225. Dois tipos de estruturas hairpin MS2 são usados neste trabalho: wild-type MS2 hairpin-wt, e f6 MS2 capat protein (MCP) aptamer-f6.
- Sintetize um modelo de scRNA que seja capaz de acomodar diferentes sequências de espaçadores (ou seja, pSNR52-espaçador_DR(SpCas9)-2×MS2(wt+f6)-SUP4t).
NOTA: Neste estudo, o molde de scRNA foi sintetizado por uma empresa de síntese de genes (ver Tabela de Materiais). - Projetar primers adequados (ver Tabela Suplementar 2) para executar a PCR nas sequências de espaçadores necessárias.
- Siga os procedimentos na etapa 1.3 para construir um de expressão de scRNA.
3. Engenharia dSpCas9 e construção de plasmídeos de expressão
- Obter o pTPGI_dSpCas9_VP64 plasmidial (ver Tabela de Materiais).
- Construa o vetor aceitador pRSII406-pGPD-ATG-XbaI-XhoI-CYC1t, baseado no vetor de vaivém pRSII406, via PCR touchdown e o método de montagem Gibson (consulte a etapa 1.3). O plasmídeo fornece um forte promotor constitutivo-pGPD, e um terminador-CYC1t.
- Digerir o plasmídeo pTPGI_dCas9_VP64 e o vector aceitador recém-construído (5 μg durante 1 h ou 10 μg para a noite, ver passo 1.3.3.1 como referência) com XbaI e XhoI a 37 °C. Separe e purifique o fragmento de inserção e o vetor aceitador como na etapa 1.3.2.
- Ligar o fragmento de inserção purificado e o vetor aceitador com T4 DNA ligase a 16 °C por 8 h. Preparar a solução de ligadura adicionando 50-100 ng do vetor aceptor purificado, fragmentos alvo purificados em quantidade equimolar com o vetor aceptor, 1,5 μL de tampão T4, 0,5 μL de T4 ligase e ddH2O até um volume total de 15 μL.
- Siga as etapas 1.3.3.3 e 1.3.3.4. Em seguida, confirme se o plasmídeo recém-construído está correto por digestão com XbaI e Xhol (37 °C, 1 h) e eletroforese em gel (ver passo 1.3.2).
4. dCas12a engenharia e construção de plasmídeos
- Construir os plasmídeos que expressam dCas12a-AD.
- Sintetizar duas proteínas dCas12a otimizadas para códons de levedura (denAsCas12a e dLbCas12a) flanqueadas por sítios da enzima de restrição BamHI e Xhol.
NOTA: Neste estudo, as duas proteínas dCas12a otimizadas para códons de levedura foram sintetizadas por uma empresa de síntese gênica (ver Tabela de Materiais). - Construa o vetor aceitador pRSII406-promoter-ATG-NLS-GS-HIStag-GS-BamHI-sp-XhoI-AD-NLS-TAA-mTGUO1 via touchdown PCR e o método de montagem Gibson (veja a etapa 1.3), onde o "promotor" é pGPD ou pGAL1, "sp" representa uma sequência aleatória curta e "AD" é VPR ou VP64.
- Inserir cada proteína dCas12a nos dois vetores aceptores recém-construídos via digestão com BamHI e XhoI e ligadura com T4 DNA ligase (ver etapas 3.3 e 3.4).
- Sintetizar duas proteínas dCas12a otimizadas para códons de levedura (denAsCas12a e dLbCas12a) flanqueadas por sítios da enzima de restrição BamHI e Xhol.
- Construir os plasmídeos expressando um dCas12a nu.
- Construa um vetor aceitador para os de expressão induzíveis por galactose de dCas12a como pRSII406-Acc651-pGAL1-ATG-NLS-GS-HIStag-GS-BamHI-sp-XhoI-GS-NLS-TAA-CYC1t usando PCR touchdown e o método de montagem Gibson (veja a etapa 1.3).
- Digerir os plasmídeos contendo as proteínas dCas12a e o vetor aceitador acima com BamHI e Xhol, em seguida, ligá-los com T4 DNA ligase para obter o plasmídeo pRSII406-pGAL1-Acc651-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1t (consulte as etapas 3.3 e 3.4).
- Digerir o plasmídeo obtido na etapa 4.2.2 com Acc65I e BamHI e, em seguida, substituir pGAL1 por pGPD via touchdown PCR e o método de montagem Gibson para construir pRSII406-pGPD-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1t.
5. Engenharia de proteínas anti-CRISPR e construção de plasmídeos
NOTA: Três tipos de promotores têm sido empregados para conduzir a expressão de Acrs: um promotor induzível-pGAL1, quatro promotores constitutivos de levedura-pGPD, pACT1, pTEF1 e pTEF2, e um promotor constitutivo sintético-genCYC1t_pCYC1noTATA26.
- Obter os plasmídeos contendo as sequências de Acrs tipo II (AcrIIA2, AcrIIA427 e AcrIIA5 28) e tipo V-A Acrs (AcrVA1, AcrVA4 e AcrVA529) de uma empresa de síntese gênica.
- Construa os plasmídeos com base no vetor de vaivém pRSII403 para expressar Acrs.
- Construa de expressão AcrIIA.
NOTA: Use o PCR touchdown para amplificar quatro promotores diferentes (ou seja, pGPD, pACT1, pTEF2 e genCYC1t_pCYC1noTATA), os três tipos de AcrIIA e dois terminadores (ADH1t e CYC1t). Crie uma série de TUs expressando AcrIIAs, sob diferentes promotores, por meio do método de montagem Gibson (consulte a etapa 1.3). - Construa de expressão AcrVA.
- Sintetize a sequência aceitadora: ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6, onde "sp" é uma sequência aleatória que será substituída por AcrVAs posteriormente.
NOTA: Neste estudo, as sequências aceptoras foram sintetizadas por uma empresa de síntese gênica (Tabela de Materiais). - Monte os vetores aceitadores pRSII403-promoter-ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6, onde o "promotor" é: pGAL1, pTEF1 e genCYC1t_pCYC1noTATA. Use o método de montagem Gibson (consulte a etapa 1.3).
- Insira cada um dos três AcrVAs no vetor aceitador descrito na etapa 5.2.2.2 (via PCR touchdown e o método de montagem Gibson [veja a etapa 1.3]) para construir um conjunto de plasmídeos produzindo AcrVAs.
- Sintetize a sequência aceitadora: ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6, onde "sp" é uma sequência aleatória que será substituída por AcrVAs posteriormente.
- Construa de expressão AcrIIA.
- Projete ainda mais o AcrIIA4 estendendo sua sequência com as sequências do ligador GS e HBD(hER). Isso permite a construção de um circuito responsivo ao β-estradiol.
- Use o PCR touchdown para obter partes GS-HBD e AcrIIA4 separadamente (consulte a etapa 1.3.1).
- Coloque AcrIIA4, GS-HBD e o vetor shuttle na mistura de Gibson e construa o plasmídeo completo através do método de Gibson (ver etapa 1.3.3).
6. Detecção de crRNA: RT-qPCR e desenho de primers
OBS: A detecção do crRNA foi realizada via RT-qPCR, que está organizado em três etapas.
- Realizar a extração e purificação de RNA de células de levedura através de um kit de RNA.
- Cultivar células de levedura durante a noite em 2 mL de meio completo definido sintético (SDC, 1 L volume: 20 g de glicose, 2 g de mistura AA, 6,7 g de YNB, 396 mg de leucina, 79,2 mg de triptofano, 79,2 mg de histidina e 79,2 mg de uracila) usando uma placa de 24 poços (240 rpm, 30 °C).
- Pela manhã, diluir a cultura celular (1:100) em 2 mL de SDC fresco e continuar crescendo as células de levedura a 30 °C, 240 rpm, por mais 4 h.
- Colher os 2 ml inteiros da solução celular e centrifugar a 20,238 x g durante 2 min. Retire o sobrenadante com cuidado, pois o pellet de células é pequeno.
- Extraia o RNA de células de levedura usando um kit de RNA.
- Verifique a qualidade do RNA.
- Prepare um gel de agarose a 1%. Misturar 5 μL de cada amostra de RNA com 1 μL de corante de carga de DNA. Em seguida, carregue a mistura no gel e execute-o.
- Certifique-se de que duas bandas claras em ~4.000 nt e ~2.000 nt, correspondentes ao RNA ribossomal (25S/18S), estejam presentes no gel. Uma outra banda borrada pode ser vista em ~80 nt para tRNA.
- Use as amostras de RNA imediatamente para síntese de cDNA ou armazene-as a -80 °C para uso futuro.
- Transcrição reversa: Use o método stem-loop 30 para formar a primeira fita de cDNA correspondente ao crRNA (40 nt). Para a transcrição reversa do sgRNA (quase 100 nt), o procedimento é o mesmo da síntese de cDNA do gene de referência ACT1.
NOTA: O método para a transcrição reversa do crRNA é diferente daquele usado com o sgRNA e o mRNA ACT1 . Como o crRNA é muito curto, ele foi tratado como um microRNA e usado o método de transcrição reversa de microRNA (abordagem stem-loop) para obter o cDNA correspondente. Dois kits de síntese de cDNA (um kit stem-loop para o crRNA e um kit usual de transcrição reversa para o gene ACT1 ) foram usados na quantificação do crRNA. A mesma quantidade de RNA foi usada nos dois kits (ver Tabela de Materiais) para tornar os resultados do experimento comparáveis. O primer usado com o kit stem-loop foi projetado de acordo com a sequência stem-loop e os últimos seis nucleotídeos na extremidade 3' do crRNA (para a sequência stem-loop e primer, ver Tabela Suplementar 2).- Método stem-loop para transcrição reversa de crRNA
- Tire o modelo de RNA, o primer e os tampões do congelador e deixe-os derreter no gelo.
- Remoção de DNA genômico: Primeiro, prepare 10 μL da mistura de reação de acordo com as instruções do kit. Colocar a mistura num termociclador a 42 °C durante 2 min. Por fim, transfira-o para o gelo.
- Síntese da primeira fita de cDNA: Preparar 20 μL da mistura de reação adicionando 10 μL da mistura da etapa 6.2.1.2, 1 μL de primer stem-loop (concentração de 2 μM), 2 μL de tampão de reação 10x RT, 2 μL de mistura de enzimas de transcrição reversa (contendo a transcriptase reversa) e 5 μL de H2O livre de RNase.
- Coloque a mistura de reação em um termociclador e execute o seguinte programa: 25 °C por 5 min, 50 °C por 15 min e 85 °C por 5 min. Use o produto para a reação de qPCR imediatamente ou armazene-o a -80 °C.
- transcrição reversa de sgRNA e ACT1 mRNA
- Primeira reação: Preparar uma mistura de 13 μL composta pela mistura de primer, mistura de dNTP, molde de RNA (50 pg-5 μg) e água livre de RNase (além do molde de RNA, todos fornecidos pelo kit), de acordo com as instruções do kit. Use 1 μg de molde de RNA. Colocar a mistura num termociclador a 70 °C durante 10 minutos.
- Segunda reação (síntese de cDNA): Preparar a mistura de reação adicionando os reagentes (conforme descrito nas instruções do kit) aos 13 μL da primeira solução de reação até um volume final de 20 μL. Colocar a mistura em um termociclador por 15 min a 50 °C e, em seguida, por mais 5 min a 85 °C. Use o produto para a reação de qPCR imediatamente ou armazene-o a -80 °C.
- Método stem-loop para transcrição reversa de crRNA
- Kit SYBR para qPCR: Detecção de valor Ct
NOTA: O primer reverso usado na qPCR do crRNA é fixo porque corresponde ao complemento reverso da sequência stem-loop (ver Tabela Suplementar 2). O primer forward, ao contrário, é variável e depende da sequência do crRNA. Os primers forward e reverse para o qPCR de mRNA e ACT1 mRNA são projetados em https://www.ncbi.nlm.nih.gov/tools/primer-blast/. Dois primers são seleccionados quando a diferença entre as suas temperaturas de fusão não for superior a 2 °C (ver quadro suplementar 2). Cada amostra é medida em três repetições.- Prepare a mistura de reação qPCR de acordo com as instruções do fabricante para o kit SYBR.
- Defina o seguinte programa qPCR em uma máquina de PCR em tempo real.
Pré-incubação: 10 min a 95 °C.
Estágio de PCR: 15 s a 95 °C, seguido por 34 s a 55 °C. Defina o ciclo do estágio de PCR para 45 vezes. Estágio de derretimento: 10 s a 95 °C, seguido de 60 s a 65 °C e, finalmente, 1 s a 97 °C. - Calcular os valores relativos de expressão de mRNA através da fórmula de Pfaffl31.
7. Imunofluorescência para detecção de proteínas Cas
NOTA: As proteínas Cas (CasP) são fundidas a um His_tag.
- Preparação de células de levedura
- Pegue algumas células de levedura usando um laço estéril e cultive-as em 5 mL de meio rico em YPD durante a noite a 240 rpm a 30 °C. De manhã, adicionar 500 μL da solução celular a 20 ml de YPD fresco e cultivá-los a 240 rpm a 30 °C até que OD600 atinja 0,6.
- Tomar 5 mL da cultura e misturá-la com 500 μL de formaldeído a 37%. Deixe a mistura permanecer em temperatura ambiente (TR) por 10 min. Colher as células por centrifugação a 1.500 x g por 5 min.
- Retirar o sobrenadante e ressuspender as células com 1 mL de tampão de fixação (0,1 M KH 2 PO4, 0,5 M MgCl2, 3,7% formaldeído, pH = 6,5). Manter a solução celular em RT por 20 min.
- Centrifugar a solução celular a 1.500 x g por 5 min. Eliminar o sobrenadante e ressuspender as células em 1 mL de tampão de lavagem (0,1 M KH 2 PO 4,1,2M sorbitol, pH = 6,5) suplementado com 4 μL de beta-mercaptoetanol e4 μL de solução de lisado (5 mg/mL). Colocar a solução celular numa incubadora a 37 °C durante 20 minutos.
- Centrifugar a solução celular a 1.500 x g por 5 min e descartar o sobrenadante. Lavar o pellet de células duas vezes com 1 mL de PBS por centrifugação (1.500 x g por 5 min).
- Ressuspender as células em 100 μL de PBS mais Tween 20 a 0,05% e adicionar 4 μL de solução de BSA (10 mg/mL). Manter a solução celular em RT por 20 min.
- Incubação com anticorpo primário
- Adicione o anticorpo primário Anti-His à mistura no passo 7.1.6 a uma diluição de 1:400. Manter a solução em TR por 2 h.
- Centrifugar a mistura no passo 7.2.1 a 1.500 x g durante 5 min e retirar o sobrenadante. Adicionar 1 mL de PBST e centrifugar (1.500 x g) por 5 min para lavar o pellet de células. Repita esta operação duas vezes. Por fim, descartar o sobrenadante e suspender as células em 100 μL de 1x PBST.
- Detecção de células por microscopia
- Monte as células em um slide; Tomar 2 μL da solução celular do passo 7.2.2 e colocá-la numa lâmina de vidro. Cubra-o com uma lamínula.
- Observe as células sob um microscópio de fluorescência. Ligue a fonte de luz fluorescente, o microscópio e o computador. Anote o número da fonte de luz fluorescente e abra o software do microscópio no computador.
- Coloque a lâmina no palco do microscópio. Escolha a lente objetiva de 40x e observe as células sob a luz verde (550 nm). Mova o botão de foco grosso até que o contorno das células de levedura apareça. Mova o botão de foco fino para focar as células.
- Detecte as células com o software do microscópio. Feche o campo de visão do microscópio e alterne para a tela do computador. Clique em Live, aguarde 3-5 s e clique em Capture para tirar uma foto. Salve a imagem.
- Desligue o computador, o microscópio e a fonte de luz fluorescente.
8. Aquisição de dados: FACS
NOTA: A fluorescência verde é detectada por citometria de fluxo (ou seja, medições de classificação de células ativadas por fluorescência [FACS]). Células de levedura são cultivadas, em geral, a 30 °C e 240 rpm para execução de experimentos FACS. No entanto, as células podem exigir alguns cuidados, dependendo do seu conteúdo genético. As células que contêm o gene dCas12a-VPR (controlado pelo promotor constitutivo GPD ) devem ser cultivadas por 24 h na solução SDC. Depois, as células são diluídas na proporção de 1:100 em SDC fresco e cultivadas por mais 12 h antes de medir a intensidade da fluorescência. Células modificadas com o gene AcrIIA4-HBD(hER) também demandam diluição. Além disso, o OD600 precisa ser controlado. Primeiro, as células são deixadas crescer em SDC durante a noite (mais de 14 h). Pela manhã, o OD600 é medido. Em seguida, a cultura é diluída em SDC, fornecida com uma concentração diversa de β-estradiol, até OD600 = 0,1. Antes dos experimentos FACS, as células são cultivadas por mais 7 h de tal forma que OD600 atinja 0,8-1,0. As células que expressam dCas9-VP64 ou dCas12a-VP64 são cultivadas em SDC por 20-24 h sem diluição e crescimento adicional antes das medições na máquina FACS.
- Ligue a máquina FACS 20 min antes das medições para aquecer o laser.
- Preparar as amostras (diluição): misturar 20 μL da cultura celular com 300 μL de ddH2O.
- Execute o software FACS no computador conectado à máquina FACS e crie um novo experimento. Defina os parâmetros de medição (ou seja, FSC(SSC)-A/H/W e histograma).
- Selecione o filtro de acordo com os comprimentos de onda de excitação e emissão das amostras. Por exemplo, a proteína alvo aqui é yEGFP, cujos comprimentos de onda de excitação e emissão são de 488 nm e 507 nm, respectivamente. Então, selecione o filtro FITC ou GFP (comprimento de onda de excitação: 488 nm; comprimento de onda de emissão: 527/32 nm). Defina o número da célula de aquisição como 10.000.
- Ajuste a tensão do filtro FITC medindo a intensidade das esferas fluorescentes. Certifique-se de que a diferença relativa na intensidade das contas entre dois experimentos consecutivos não exceda 5%.
- Lave a máquina com ddH2O por alguns segundos para remover qualquer possível excesso de contas.
- Medir a intensidade de fluorescência da amostra. Clique em Visualizar e aguarde 3-5 s para estabilidade da injeção da amostra. Por fim, clique em Adquirir.
- Meça as contas novamente no final do experimento. A tensão é a que foi usada durante a medição inicial das contas (ver passo 8.4, 438-441 V). Verifique se a diferença relativa entre as medidas das duas contas é superior a 5%.
- Exporte os dados FACS como arquivos FCS.
- Analise os arquivos FCS com o software R Studio.
9. Análise dos dados
NOTA: Use o pacote Flowcore R Bioconductor 32 dentro do R studio. Os arquivos FCS foram analisados por meio de um script escrito em linguagem R.
- Abra o estúdio R e carregue o script de análise Bdverse. R para analisar os arquivos FCS.
- Defina o nome do experimento (ename), o diretório (caminho) onde os arquivos FCS estão
Armazenado (dir_d) e onde os arquivos de resultado serão criados (dir_r). - Defina o canal de fluorescência. Por exemplo, select_ch = "GPA-A" se a fluorescência verde foi medida.
- Defina o número de amostras que foram medidas (s_num).
- Defina as dimensões de cada gráfico de pontos (sxlim, sylim). Defina o comprimento máximo dos eixos x e y para gráficos de barras e gráficos de caixa (xlimit, ylimit). xlimit deve ser maior ou igual a s_num.
- Escolha o método de gating removendo o # das linhas correspondentes.
NOTA: morphGate é um método de gating automático realizado pelo script (ou seja, a região dos gráficos de pontos onde as células são mais densas é reconhecida e selecionada pelo programa). polygonGate e rectangleGate exigem olhar para os gráficos de pontos e definir os vértices de um polígono ou o lado de um retângulo que abraça a zona onde a maioria das células se encontra. - Selecione o objeto flowSet gated, correspondente ao método de gating escolhido. Selecione a resolução do gráfico de pontos (res; deve ser pelo menos igual a 256).
- Uncomment flt_low <- filter_low(sp) para remover medidas onde a fluorescência é negativa. Uncomment flt_sp <- filter(flt_lw) para remover outliers devido a outros experimentos.
- Pressione Source e execute o script. Todos os arquivos contendo os resultados da análise são criados em dir_r.
Resultados
Expressão de sgRNA/crRNA por um promotor do tipo RNA polimerase III
Primeiramente, este trabalho abordou a engenharia do circuito de ativação transcricional (circuito 1) mostrado na Figura 1A. Ele continha três componentes básicos: 1) o gene que codifica para yEGFP (o repórter), que foi precedido por uma série de diferentes promotores sintéticos que forneceram locais-alvo para dCas9/dCas12a-AD; 2) uma versão otimizada para códons de levedura de dCas9 ou dCas12a ...
Discussão
O protocolo mostrou um possível fluxo de trabalho completo para circuitos digitais de genes sintéticos, seguindo o ciclo de engenharia biológica "Design-Build-Test-Learn" (DBTL) e em experimentos de laboratório seco e úmido. Aqui, nós nos concentramos no sistema CRISPR-Cas, principalmente dSpCas9, denAsCas12a, dLbCas12a, e as proteínas anti-CRISPR correspondentes, projetando e construindo em S. cerevisiae pequenas redes transcricionais. Alguns deles imitavam portões booleanos, que são os componentes bá...
Divulgações
Os autores declaram não haver interesse financeiro concorrente.
Agradecimentos
Queremos agradecer a todos os alunos do laboratório de Biologia Sintética - SPST, TJU - por sua ajuda geral, juntamente com Zhi Li e Xiangyang Zhang por sua assistência em experimentos FACS.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | - | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | - | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | - | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | - | - | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
Referências
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados