このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
CRISPR-Casシステムと抗CRISPRタンパク質に基づく遺伝子デジタル回路
要約
CRISPR-Cas系および抗CRISPRタンパク質は、 出芽酵母のブールゲートのスキームに統合されました。新しい小型論理回路は良好な性能を示し、dCas9/dCas12aベースの転写因子と抗CRISPRタンパク質の特性の両方の理解を深めました。
要約
合成遺伝子ブールゲートとデジタル回路は、医療診断から環境ケアまで、幅広い用途があります。CRISPR-Casシステムとその天然阻害剤である抗CRISPRタンパク質(Acrs)の発見は、 in vivo 遺伝子デジタル回路を設計および実装するための新しいツールを提供します。ここでは、「設計-構築-テスト-学習」生物工学サイクルの考え方に従い、dCas9 / dCas12aとそれに対応するAcrを利用して、 サッカロミセス・セレビシエでブールゲートのように振る舞う小さな転写ネットワークを確立するプロトコルについて説明します。これらの結果は、転写因子としてのdCas9/dCas12aの性質を指摘しています。特に、遺伝子発現の最大活性化を達成するために、dSpCas9はVP64活性化ドメイン(AD)の複数のコピーを収集する操作された足場RNAと相互作用する必要があります。対照的に、dCas12aはC末端で強力なVP64-p65-Rta(VPR)ADと融合する必要があります。さらに、両方のCasタンパク質の活性は、細胞内のsgRNA/crRNAの量を増加させることによって増強されない。この記事では、CRISPR-dCas-Acr の相互作用に基づいてブール型ゲートを構築する方法についても説明します。ヒトエストロゲン受容体のAcrIIA4融合ホルモン結合ドメインは、β-エストラジオールに応答するNOTゲートのコアですが、誘導性 GAL1 プロモーターによって合成されたAcrVAは、ガラクトースを入力としてYESゲートとNOTゲートの両方を模倣することができます。後者の回路では、AcrVA5とdLbCas12aが最良のロジック動作を示しました。
概要
2011年、研究者は計算方法を提案し、デジタル合成遺伝子回路の自動設計に対応するソフトウェアを開発しました1。ユーザーは入力数(3つまたは4つ)を指定し、回路真理値表に記入する必要がありました。これにより、電子機器の技術を使用して回路構造を導き出すために必要なすべての情報が提供されました。真理値表は、Karnaughマップ法2を介して2つのブール式に変換されました。各ブール式は、回路入力(の一部)とその否定(リテラル)間の論理演算(合計または乗算)を記述する句で構成されています。句は、順番に合計(OR)または乗算(AND)されて回路出力を計算します。すべての回路は、POS(合計の積)形式で記述された式とSOP(製品の合計)表現で記述された2つの対応する式のいずれかに従って実現できます。前者は、リテラルの論理和を含む句(つまり、ブールゲート)の乗算で構成されます。対照的に、後者は、リテラルが乗算される句の合計です。
電気回路は、ブレッドボード上で、異なるゲートを物理的に配線することによって実現できます。電流はゲート間の信号交換を可能にし、出力の計算につながります。
生物学では、状況はより複雑です。ブールゲートは、転写または翻訳(またはその両方)が調節される転写単位(TU、すなわち、真核細胞内の配列「プロモーター-コード領域-ターミネーター」)として実現することができる。したがって、少なくとも2種類の分子、すなわち転写因子タンパク質および非コードアンチセンスRNA1の生物学的配線を確立する。
遺伝子デジタル回路は、2層または3層のゲート、すなわち1)YES(バッファー)およびNOTゲートで構成された入力層で構成され、入力化学物質を配線分子に変換する。2)対応するブール式に句があるのと同じ数のTUで構成される内部層。回路がSOP式に従って設計されている場合、内部層のすべての句は、いわゆる分散出力アーキテクチャで回路出力(蛍光など)を生成します。和の積(POS)式が使用される場合、3)最終層が必要であり、これには内部層から配線分子を収集する単一の乗法ゲートが含まれます。
全体として、合成生物学では、同じ回路に対して多くの異なるスキームを設計することができます。それらはTUと配線分子の両方の数と種類が異なります。酵母細胞に実装する最も簡単なソリューションを選択するために、各回路設計は、次のように定義される複雑さスコアSに関連付けられています。
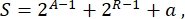
ここで、 A は活性化剤の数を表し、 R はリプレッサーの数を表し、 a はアンチセンスRNA分子の量です。アクティベータまたはリプレッサーのいずれかが回路にない場合、Sへの寄与はゼロです。そのため、ラボでの回路スキーム(高S)の実現は、多数の直交転写因子を必要とする場合により困難である。これは、デジタル回路内の完全な配線を実現するために、新しいアクティベータとリプレッサーを de novo で設計する必要があることを意味します。原理的には、ジンクフィンガータンパク質3 およびTALエフェクター4 を鋳型として使用することにより、新規DNA結合タンパク質を組み立てることができる。ただし、このオプションは非常に困難で時間がかかるように見えます。したがって、複雑な遺伝子回路を完成させるために、主に小さなRNAと翻訳調節に頼る必要があります。
もともと、この方法は細菌のデジタル回路を作製するために開発されました。実際、真核細胞では、アンチセンスRNAの代わりに、マイクロRNA(miRNA)または低分子干渉RNA(siRNA)について話す方が適しています5。しかしながら、RNAi経路は酵母 S.セレビシエには存在しない。したがって、完全に転写ネットワークを選択する必要があります。回路に5つのアクティベータと5つのリプレッサーが必要であるとします。その複雑さのスコアはS = 32になります。10個の転写因子を活性化ドメイン(AD)に融合した単一のdCas96 (ヌクレアーゼ欠損Cas9)に置き換えることで、回路の複雑さを軽減することができます。7に示すように、dCas9-ADは、TATAボックスとTSS(転写開始部位)の間でプロモーターを結合させる際の酵母のリプレッサーとして、またTATAボックスのかなり上流に結合するときの活性化剤として働きます。したがって、10個の転写因子を単一のdCas9-AD融合タンパク質と10個のsgRNA(シングルガイドRNA)に置き換えることができ、合計複雑さスコアはS = 11になります。10個のsgRNAを合成するのは迅速かつ簡単ですが、前述のように、10個のタンパク質の組み立てにははるかに長く複雑な作業が必要になります。
あるいは、2つの直交するdCasタンパク質(例えば、dCas9およびdCas12a)を使用することができ、1つはADに融合し、もう1つは裸または抑制ドメインと組み合わせて使用する。複雑さのスコアは 1 単位だけ増加します (S = 12)。したがって、CRISPR-dCasシステムは、 出芽酵母における非常に複雑な遺伝子デジタル回路の構築の鍵となります。
この論文は、酵母におけるdCas9およびdCas12aベースのリプレッサーおよびアクチベーターの両方の効率を深く特徴付けています。結果は、それらがそれらの活性を最適化するために大量のsgRNAを必要としないことを示したので、エピソームプラスミドは優先的に避けられる。さらに、dCas9ベースのアクチベーターは、VP64 ADのコピーをリクルートするスキャフォールドRNA(scRNA)を使用する場合にはるかに効果的です。対照的に、dCas12aは、強力なVPR ADに直接融合するとうまく機能します。さらに、合成活性化プロモーターは、活性化因子の構成に応じて、可変数の標的部位を必要とします(例えば、dCas12a-VPRを使用する場合は3つ、dCas9-VP64を使用する場合は6つ、dCas9とscRNAを使用する場合は1つだけ)。リプレッサーとして、dCas12aはプロモーターよりもコード領域に結合するとより鋭敏に見えます。
ただし、欠点として、CRISPR-dCas9 / dCas12aは化学物質と直接相互作用しません。したがって、入力層では役に立たない可能性があります。このため、抗CRISPRタンパク質(ACRS)を含む代替ブールゲート設計が検討されています。ACRは(d)Casタンパク質に作用し、その働きを阻害します8.したがって、それらはCRISPR-(d)Casシステムの活性を調節する手段です。この論文では、出芽酵母のII型ACRSと(d)Cas9、およびV型Acrと(d)Cas12aの相互作用を徹底的に分析しています。AcrsはCasタンパク質よりもはるかに小さいため、ヒトエストロゲン受容体9-HBD(hER)-のホルモン結合ドメインをAcrIIA4に融合させることによって、エストロゲンβ-エストラジオールに応答するNOTゲートを構築しました。さらに、dCas12a(-AD)を構成的に発現する少数のYESゲートとNOTゲート、およびガラクトースによる誘導時にAcrVAが実現されました。現在、これらのゲートは概念実証としてのみ機能します。しかし、それらはまた、酵母細胞における合成遺伝子デジタル回路の計算自動設計を実行するためのアルゴリズムの深い再考に向けた第一歩を表しています。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. sgRNA/crRNA発現カセットの設計と構築
注:sgRNA/crRNA発現カセットには2種類あります:1つの用語のSNR5210-は、RNAポリメラーゼIII依存性SNR52プロモーター、sgRNA / crRNA配列、およびSUP4ターミネーターで構成されています。RGR11と略される別のものは、RNAポリメラーゼII依存性ADH1プロモーター、2つのリボザイム(シュモクザイム-HH、および肝炎デルタウイルスリボザイム-HDV)を含むRGR(リボザイムガイドRNA-リボザイム)構造、およびその間のsgRNA/crRNAの配列、およびADH1ターミネーターで構成されています。Cas9ホモログを誘導するsgRNAはスペーサー配列と特徴的なダイレクトリピート12で構成されていますが、Cas12aタンパク質のcrRNAはダイレクトリピートとそれに続くスペーサー配列13,14で構成されています(この研究で使用されたすべてのDNA配列については補足表1を参照)。
- Cas9/Cas12aを介した転写活性化のためのスペーサー配列を設計します。
- 細菌のlexオペレーター配列(lexOpと名付けられた)を標的部位15,16として利用し、酵母増強緑色蛍光タンパク質(yEGFP)17の発現を促進するCYC1プロモーターに挿入します。したがって、スペーサー配列は、挿入されたlexOpによって定義され、相補的である。
- スペーサ配列の直交性は、CRISPRDIRECTツール18を介して確認する。
- PAM 配列に隣接する lexOp 配列をテキストフィールドに貼り付け、PAM 配列を dCas9 の場合は NRG、dCas12a の場合は TTTV として定義し、ドロップダウンリストから種を Budding 酵母 (Saccharomyces cerevisiae) S288C ゲノムとして指定します。 [デザイン]をクリックします。20mer + PAMおよび12mer + PAM検索に一致するターゲットサイトがないことを確認してください。
- sgRNA/crRNA発現カセットを構築します。
- タッチダウンPCRを使用して、プロモーター、コード配列、ターミネーターなどの標準的な生物学的部分のDNA配列を増幅します。
- 以下を含む反応混合物を調製します:20-40 ngのDNAテンプレート、1 μLの10 μMフォワードプライマー(すなわち、ot25、sgRNA/crRNA発現プラスミド構築)、1 μLの10 μMリバースプライマー(すなわち、ot26、sgRNA/crRNA発現プラスミド構築)、5 μLの2.5 mM dNTPミックス、0.5 μLのDNAポリメラーゼ、10 μLの5x DNAポリメラーゼ反応バッファー、 総容量50μLまでの二重蒸留水(ddH2O)。
注:この研究で使用されたプライマーのリストについては、 補足表2 を参照してください。 - サーモサイクラーでタッチダウンPCRプログラムを実行します。
ステージ1:98°Cで30秒
10サイクルのステージ2:10秒間98°C、20秒間68°C、15秒間72°C。
25サイクルのステージ3:98°Cで10秒、59°Cで20秒、72°Cで15秒。
ステージ4:72°Cで2分間。
最後に、フォローアップ実験を開始するまで4°Cで保持する。
注:ステージ2の68°Cとステージ3の59°Cは、フォワードプライマーとリバースプライマーの両方の融解温度に依存し、プライマーのペアによって異なります。ステージ2および3の72°Cでの時間は、PCR産物の長さとDNAポリメラーゼの速度によって決まります。
- 以下を含む反応混合物を調製します:20-40 ngのDNAテンプレート、1 μLの10 μMフォワードプライマー(すなわち、ot25、sgRNA/crRNA発現プラスミド構築)、1 μLの10 μMリバースプライマー(すなわち、ot26、sgRNA/crRNA発現プラスミド構築)、5 μLの2.5 mM dNTPミックス、0.5 μLのDNAポリメラーゼ、10 μLの5x DNAポリメラーゼ反応バッファー、 総容量50μLまでの二重蒸留水(ddH2O)。
- PCR産物をゲル電気泳動(100 V、30分) で 単離します。DNAゲル抽出キット を介して アガロースゲルからDNA配列を溶出します( 材料表を参照)。
注:500 nt より長いフラグメントには 0.8% アガロースゲル、100 nt から 500 nt までのフラグメントには 1.5% アガロースゲル、100 nt より短いフラグメントには 2% アガロースゲルが必要です。 - アンピシリン耐性遺伝子と酵母選択性栄養要求性マーカー遺伝子-TRP1を含むpRSII404/424シャトルベクター19に、sgRNA/crRNAを発現するTUを挿入します。
- シャトルベクターを37°Cで1時間、2つの制限酵素SacIおよびAcc65Iで消化します。5 μgのシャトルベクター、推奨量の酵素、消化バッファー(酵素の説明書による)、およびddH2Oを総容量30 μLまで加えて、反応混合物を調製します。
- ゲル電気泳動 による シャトルベクター消化の検証(サブステップ1.3.2参照)。その後、2つの酵素を65°Cで20分間失活させます。
- ギブソンアセンブリ法20 を使用して、等モルのDNA混合物を50°Cで1時間入れることにより、精製PCR産物をカットオープンシャトルベクターに挿入します。
- 熱ショック形質転換法21を介して上記のギブソン反応混合物を用いて大腸菌DH5αコンピテントセルを形質転換する。形質転換した大腸菌細胞を、アンピシリン(0.1 g / L)を含むルリアベルターニ(LB)寒天プレートに移します。プレートを37°Cのインキュベーターに入れ、細胞を一晩成長させます。
- LB寒天プレートから4つのコロニーを選び、アンピシリンを含むLB溶液(0.1 g/L)中で37°Cで一晩別々に培養します。次いで、ミニ調製手順を用いて、大腸菌細胞22からプラスミドを抽出する。
- プライマーot18およびot19(オリゴ配列については補足表2を参照)を使用して、サンガー法23を介して挿入された転写ユニットを配列決定および確認します。
注:後の実験では、構築および確認されたプラスミドは、PEG / LiAcプロトコル24を介して酵母ゲノムに挿入されます。
- タッチダウンPCRを使用して、プロモーター、コード配列、ターミネーターなどの標準的な生物学的部分のDNA配列を増幅します。
2. 足場RNA発現カセットの設計と構築
注:スキャフォールドガイドRNA(scRNA)は、sgRNA配列とMS2ヘアピン構造25で構成されています。この研究では、野生型MS2ヘアピンwtとf6 MS2コートタンパク質(MCP)アプタマー-f6の2種類のMS2ヘアピン構造が使用されています。
- 異なるスペーサー配列(すなわち、pSNR52スペーサー_DR(SpCas9)-2×MS2(wt+f6)-SUP4t)に対応できるscRNAテンプレートを合成します。
注:この研究では、scRNAテンプレートは遺伝子合成会社によって合成されました( 材料表を参照)。 - 適切なプライマーを設計し( 補足表2を参照)、必要なスペーサー配列でPCRを実行します。
- ステップ 1.3 の手順に従って、scRNA 発現カセットを構築します。
3. dSpCas9のエンジニアリングと発現プラスミドの構築
- プラスミドpTPGI_dSpCas9_VP64を入手します( 材料表参照)。
- タッチダウンPCRとギブソンアセンブリ法 を介して 、pRSII406シャトルベクターに基づいてアクセプターベクターpRSII406-pGPD-ATG-XbaI-XhoI-CYC1tを構築します(ステップ1.3を参照)。プラスミドは、強力な構成的プロモーター-pGPD、およびターミネーター-CYC1tを提供します。
- pTPGI_dCas9_VP64プラスミドと新しく構築したアクセプターベクター(1時間で5 μg、一晩で10 μg-ステップ1.3.3.1を参照)をXbaIおよびXhoIで37°Cで消化します。 ステップ1.3.2と同様に、インサートフラグメントとアクセプターベクターを分離して精製します。
- 精製したインサートフラグメントとアクセプターベクターをT4 DNAリガーゼで16°Cで8時間ライゲートします。精製アクセプターベクター50〜100 ng、アクセプターベクターと等モル量の精製標的断片、1.5 μLのT4バッファー、0.5 μLのT4リガーゼ、およびddH2Oを総容量15 μLまで添加してライゲーション溶液を調製します。
- ステップ 1.3.3.3 および 1.3.3.4 に従います。次に、XbaIおよびXholによる消化(37°C、1時間)およびゲル電気泳動(ステップ1.3.2を参照)により、新しく構築したプラスミドが正しいことを確認します。
4. dCas12aエンジニアリングとプラスミド構築
- dCas12a-ADを発現するプラスミドを構築します。
- BamHIおよびXhol制限酵素部位に隣接する2つの酵母コドン最適化dCas12aタンパク質(denAsCas12aおよびdLbCas12a)を合成します。
注:この研究では、2つの酵母コドン最適化dCas12aタンパク質を遺伝子合成会社によって合成しました( 材料表を参照)。 - タッチダウンPCRおよびギブソンアセンブリ法(ステップ1.3を参照) を介して アクセプターベクターpRSII406-プロモーター-ATG-NLS-GS-HISタグ-GS-BamHI-sp-XhoI-AD-NLS-TAA-mTGUO1を構築し、「プロモーター」はpGPDまたはpGAL1のいずれかであり、「sp」は短いランダム配列を表し、「AD」はVPRまたはVP64のいずれかです。
- 各dCas12aタンパク質を、BamHIおよびXhoIによる消化およびT4 DNAリガーゼによるライゲーション を介して 、新しく構築された2つのアクセプターベクターに挿入します(ステップ3.3および3.4を参照)。
- BamHIおよびXhol制限酵素部位に隣接する2つの酵母コドン最適化dCas12aタンパク質(denAsCas12aおよびdLbCas12a)を合成します。
- 裸のdCas12aを発現するプラスミドを構築します。
- タッチダウンPCRとギブソンアセンブリ法を使用して、dCas12aのガラクトース誘導性発現カセットのアクセプターベクターをpRSII406-Acc651-pGAL1-ATG-NLS-GS-HIStag-GS-BamHI-sp-XhoI-GS-NLS-TAA-CYC1tとして構築します(ステップ1.3を参照)。
- dCas12aタンパク質を含むプラスミドと上記のアクセプターベクターをBamHIとXholで消化し、T4 DNAリガーゼでライゲーションしてプラスミドpRSII406-pGAL1-Acc651-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1tを取得します(ステップ3.3および3.4を参照)。
- ステップ4.2.2で得られたプラスミドをAcc65IとBamHIで消化した後、タッチダウンPCRとギブソンアセンブリ法 を介して pGAL1をpGPDに置換して、pRSII406-pGPD-ATG-NLS-GS-HIStag-GS-BamHI-dCas12a-XhoI-GS-NLS-TAA-CYC1tを構築します。
5. 抗CRISPRタンパク質工学とプラスミド構築
注:Acrs発現を促進するために、誘導性プロモーター-pGAL1、4つの酵母構成プロモーター-pGPD、pACT1、pTEF1、およびpTEF2、および合成構成プロモーター-genCYC1t_pCYC1noTATA26の3種類のプロモーターが使用されています。
- II型ACRS(AcrIIA2、AcrIIA427、AcrIIA5 28)およびV-A型ACRS(AcrVA1、AcrVA4、およびAcrVA529)の配列を含むプラスミドを遺伝子合成会社から入手する。
- acrsを発現させるためのpRSII403シャトルベクターに基づいてプラスミドを構築します。
- AcrIIA発現カセットを構築します。
注:タッチダウンPCRを使用して、4つの異なるプロモーター(pGPD、pACT1、pTEF2、およびgenCYC1t_pCYC1noTATA)、3種類のAcrIIA、および2つのターミネーター(ADH1tおよびCYC1t)を増幅します。ギブソンアセンブリ法 を介して 、異なるプロモーターの下でAcrIIAを発現する一連のTUを構築します(ステップ1.3を参照)。 - AcrVA発現カセットを構築します。
- アクセプター配列を合成します: ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6 ここで、"sp" はランダムな配列で、後で AcrVA に置き換えられます。
注:この研究では、アクセプター配列は遺伝子合成会社によって合成されました(材料表)。 - アクセプターベクターpRSII403-プロモーター-ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6をアセンブルし、「プロモーター」はpGAL1、pTEF1、およびgenCYC1t_pCYC1noTATAです。ギブソンアセンブリ方法を使用します(手順1.3を参照)。
- 3つのAcrVAのそれぞれをステップ5.2.2.2で説明したアクセプターベクターに挿入し(タッチダウンPCRおよびギブソンアセンブリ法[ステップ1.3を参照]を介して )、AcrVAを産生するプラスミドのセットを構築します。
- アクセプター配列を合成します: ATG-FLAGtag-GS-BamHI-sp-XhoI-NLS-GS-NLS-TAA-Tsynth6 ここで、"sp" はランダムな配列で、後で AcrVA に置き換えられます。
- AcrIIA発現カセットを構築します。
- さらに、AcrIIA4の配列をGSリンカーとHBD(hER)の配列で拡張することにより、その配列を設計しました。これにより、βエストラジオール応答回路の構築が可能になります。
- タッチダウンPCRを使用して、GS-HBDおよびAcrIIA4パーツを別々に取得します(ステップ1.3.1を参照)。
- AcrIIA4、GS-HBD、およびシャトルベクターをギブソン混合物に入れ、ギブソン法 を介して 完全なプラスミドを構築します(ステップ1.3.3を参照)。
6. crRNA検出:RT-qPCRおよびプライマーの設計
注:crRNAの検出は、3つのステップで構成されるRT-qPCR を介して 達成されました。
- RNAキット を介して 酵母細胞からRNA抽出および精製を行う。
- 24ウェルプレート(240 rpm、30°C)を用いて、2 mLの合成規定完全培地(SDC、1 L容量:グルコース20 g、AAミックス2 g、YNB6.7 g、ロイシン396 mg、トリプトファン79.2 mg、ヒスチジン79.2 mg、およびウラシル79.2 mg)中で酵母細胞を一晩培養します(240 rpm、30°C)。
- 午前中に、細胞培養(1:100)を2 mLの新鮮なSDCに希釈し、酵母細胞を30°C、240 rpmでさらに4時間増殖させ続けます。
- 細胞溶液2 mL全体を回収し、20,238 x g で2分間遠心分離します。細胞ペレットが小さいため、上清を慎重に除去します。
- RNAキットを用いて酵母細胞からRNAを抽出する。
- RNAの品質を確認してください。
- 1%アガロースゲルを調製します。5 μLの各RNAサンプルを1 μLのDNAローディング色素と混合します。次に、混合物をゲルにロードして実行します。
- リボソームRNA(25S/18S)に対応する~4,000 ntと~2,000 ntの2つの明瞭なバンドがゲル上に存在することを確認します。tRNAの~80 ntでさらにぼやけたバンドが見られます。
- RNAサンプルは、cDNA合成のためにすぐに使用するか、将来の使用のために-80°Cで保存してください。
- 逆転写:ステムループ法30を使用して、crRNA(40 nt)に対応するcDNAの最初の鎖を形成します。sgRNAの逆転写(ほぼ100 nt)については、参照遺伝子 ACT1のcDNA合成の場合と同じ手順である。
注:crRNAの逆転写の方法は、sgRNAおよび ACT1 mRNAで使用される方法とは異なります。crRNAは非常に短いため、マイクロRNAとして扱い、マイクロRNA逆転写法(ステムループアプローチ)を使用して対応するcDNAを取得しました。crRNAの定量には、2つのcDNA合成キット(crRNA用のステムループキットと ACT1 遺伝子用の通常の逆転写キット)を使用しました。実験結果を比較できるように、2つのキット( 材料表を参照)で同じ量のRNAを使用しました。ステムループキットで使用するプライマーは、ステムループ配列とcrRNAの3'末端にある最後の6ヌクレオチドに従って設計されました(ステムループおよびプライマー配列については、 補足表2を参照)。- crRNA逆転写のためのステムループ法
- RNAテンプレート、プライマー、バッファーを冷凍庫から取り出し、氷の上で溶かします。
- ゲノムDNA除去:まず、キットの指示に従って10 μLの反応混合物を調製します。混合物を42°Cのサーマルサイクラーに2分間入れます。最後に、それを氷の上に移します。
- 最初のcDNA鎖の合成:ステップ6.2.1.2の混合物10 μL、ステムループプライマー(2 μM濃度)1 μL、10x RT反応バッファー2 μL、逆転写酵素ミックス(逆転写酵素を含む)2 μL、およびRNaseフリーH2O5 μLを加えて、20 μLの反応混合物を調製します。
- 反応混合物をサーマルサイクラーに入れ、次のプログラムを実行します:25°Cで5分間、50°Cで15分間、および85°Cで5分間。qPCR反応用の生成物をすぐに使用するか、-80°Cで保存してください。
- sgRNA および ACT1 mRNA 逆転写
- 最初の反応:キットの指示に従って、プライマーミックス、dNTPミックス、RNAテンプレート(50 pg-5 μg)、およびRNaseフリー水(RNAテンプレートとは別に、すべてキットに付属)からなる13 μL混合物を調製します。1 μgのRNAテンプレートを使用してください。混合物を70°Cのサーマルサイクラーに10分間入れます。
- 2回目の反応(cDNA合成):試薬(キットの説明書に記載されている)を13 μLの第1反応溶液に最終容量20 μLまで加えて、反応混合物を調製します。 混合物をサーマルサイクラーに50°Cで15分間入れ、次に85°Cでさらに5分間置きます。 qPCR反応用の生成物をすぐに使用するか、-80°Cで保存してください。
- crRNA逆転写のためのステムループ法
- qPCR 用 SYBR キット: Ct 値検出
注:crRNAのqPCRに使用されるリバースプライマーは、ステムループ配列の逆相補に対応するため固定されています( 補足表2を参照)。対照的に、フォワードプライマーは可変であり、crRNAの配列に依存します。sgRNA および ACT1 mRNA qPCR のフォワードプライマーおよびリバースプライマーは、https://www.ncbi.nlm.nih.gov/tools/primer-blast/ で設計されています。2つのプライマーは、それらの融解温度の差が2°C以下の場合に選択される( 補足表2を参照)。各サンプルは3回の反復で測定されます。- SYBR キットの製造元の指示に従って qPCR 反応混合物を調製します。
- リアルタイムPCR装置で以下のqPCRプログラムを設定します。
プレインキュベーション:95°Cで10分。
PCRステージ:95°Cで15秒、続いて55°Cで34秒。 PCRステージのサイクルを45回に設定します。溶融段階:95°Cで10秒、続いて65°Cで60秒、最後に97°Cで1秒。 - Pfaffl式31を介して相対的なmRNA発現値を計算する。
7. Casタンパク質を検出するための免疫蛍光法
注:Casタンパク質(CasP)はHis_tagに融合されています。
- 酵母細胞の準備
- 滅菌ループを使用して酵母細胞をピックし、5 mLのYPDリッチ培地で240 rpm、30°Cで一晩培養します。 午前中に、500 μLの細胞溶液を20 mLの新鮮なYPDに加え、OD 600が0.6に達するまで30 °Cで240 rpmで増殖させます。
- 培養液5 mLを取り、500 μLの37%ホルムアルデヒドと混合します。混合物を室温(RT)で10分間保持します。1,500 x g で5分間遠心分離して細胞を回収します。
- 上清を除去し、1 mLの固定バッファー(0.1 M KH 2 PO4、0.5 M MgCl2、3.7%ホルムアルデヒド、pH = 6.5)で細胞を再懸濁します。細胞溶液をRTで20分間保持します。
- 細胞溶液を1,500 x gで5分間遠心分離します。上清を廃棄し、4 μLのβ-メルカプトエタノールと4 μLのライセート溶液(5 mg/mL)を添加した1 mLの洗浄バッファー(0.1 M KH 2 PO4、1.2Mソルビトール、pH = 6.5)に細胞を再懸濁します。細胞溶液を37°Cのインキュベーターに20分間入れます。
- 細胞溶液を1,500 x g で5分間遠心分離し、上清を廃棄します。遠心分離により細胞ペレットを1 mLのPBSで2回洗浄します(1,500 x g で5分間)。
- 細胞を100 μLのPBSと0.05%トゥイーン20に再懸濁し、4 μLのBSA溶液(10 mg/mL)を加えます。細胞溶液をRTで20分間保持します。
- 一次抗体とのインキュベーション
- ステップ7.1.6で抗Hisタグ一次抗体を1:400希釈で混合物に加えます。溶液をRTで2時間保持します。
- ステップ7.2.1の混合物を1,500 x g で5分間遠心分離し、上清を除去します。1 mLのPBSTを加え、遠心分離機(1,500 x g)で5分間遠心分離して細胞ペレットを洗浄します。この操作を 2 回繰り返します。最後に、上清を廃棄し、細胞を100 μLの1x PBSTに懸濁します。
- 顕微鏡細胞検出
- セルをスライドにマウントします。ステップ7.2.2の細胞溶液2 μLを取り、スライドガラスの上に置きます。カバーガラスで覆います。
- 蛍光顕微鏡で細胞を観察します。蛍光灯、顕微鏡、コンピューターの電源を入れます。蛍光光源番号を書き留め、コンピューターで顕微鏡ソフトウェアを開きます。
- スライドを顕微鏡ステージに置きます。40倍の対物レンズを選択し、緑色光(550 nm)の下で細胞を観察します。酵母細胞の輪郭が現れるまで粗いフォーカスノブを動かします。細かいフォーカスノブを動かして、セルに焦点を合わせます。
- 顕微鏡ソフトウェアで細胞を検出します。顕微鏡の視野を閉じて、コンピューター画面に切り替えます。 ライブをクリックし、3〜5秒待ってから、[ キャプチャ ]をクリックして写真を撮ります。画像を保存します。
- コンピューター、顕微鏡、蛍光灯の電源を切ります。
8. データ収集: FACS
注:緑色蛍光は、フローサイトメトリー(すなわち、蛍光活性化セルソーティング[FACS]測定) によって 検出されます。酵母細胞は、一般に、FACS実験を実行するために30°Cおよび240rpmで培養される。ただし、細胞は遺伝的内容に応じていくつかの予防措置を必要とする場合があります。 dCas12a-VPR 遺伝子( GPD 構成的プロモーターによって制御される)を含む細胞は、SDC溶液中で24時間増殖させなければならない。その後、細胞を新鮮なSDCで1:100の比率で希釈し、蛍光強度を測定する前にさらに12時間増殖させます。 AcrIIA4-HBD(hER) 遺伝子で改変された細胞も同様に希釈を必要とします。さらに、OD600 を制御する必要があります。まず、細胞をSDC中で一晩(14時間以上)増殖させる。午前中はOD600 を測定します。次いで、培養物をSDCで希釈し、OD600 = 0.1まで多様な濃度のβ-エストラジオールを供給する。FACS実験の前に、OD600 が0.8〜1.0に達するように細胞をさらに7時間増殖させる。dCas9-VP64またはdCas12a-VP64を発現する細胞は、FACSマシンでの測定前に、希釈およびさらなる増殖なしにSDCで20〜24時間増殖します。
- 測定の20分前にFACSマシンの電源を入れて、レーザーをウォームアップします。
- サンプルを調製する(希釈):20 μLの細胞培養物を300 μLのddH2Oと混合する。
- FACS コンピューターに接続されているコンピューターで FACS ソフトウェアを実行し、新しい実験を作成します。測定パラメータ(FSC(SSC)-A/H/Wおよびヒストグラム)を設定します。
- サンプルの励起波長と発光波長に応じてフィルターを選択します。例えば、ここでの標的タンパク質はyEGFPであり、その励起波長と発光波長はそれぞれ488nmと507nmです。そこで、FITCまたはGFPフィルター(励起波長:488 nm、発光波長:527/32 nm)を選択します。取得セル番号を 10,000 に設定します。
- 蛍光ビーズの強度を測定してFITCフィルタ電圧を調整します。2つの連続する実験間のビーズの強度の相対差が5%を超えないようにしてください。
- 機械をddH2Oで数秒間洗浄して、余分なビーズを取り除きます。
- サンプルの蛍光強度を測定します。 プレビュー をクリックし、サンプル注入の安定性のために3〜5秒待ちます。最後に、[ 取得]をクリックします。
- 実験の最後にビーズを再度測定します。電圧は、最初のビーズの測定中に使用された電圧です(ステップ8.4、438-441 Vを参照)。2つのビーズの測定値の相対差が5%を超えているかどうかを確認します。
- FACS データを FCS ファイルとしてエクスポートします。
- R Studio ソフトウェアで FCS ファイルを分析します。
9.データ分析
注:Rスタジオ内の フローコアRバイオコンダクター パッケージ32 を使用してください。FCSファイルは、R言語で記述されたスクリプトを使用して分析されました。
- R スタジオを開き、スクリプト Bdverse 分析を読み込みます。R を使用して FCS ファイルを分析します。
- 実験名(ename)、FCSファイルがあるディレクトリ(パス)を設定します
格納 (dir_d)、および結果ファイルが作成される場所 (dir_r)。 - 蛍光チャンネルを設定します。例えば、緑色蛍光を測定した場合はselect_ch = "GPA-A"となります。
- 測定したサンプル数(s_num)を設定します。
- 各ドットプロットの寸法(sxlim、 sylim)を設定します。棒グラフと箱ひげ図(xlimit、ylimit)のx軸とy 軸の最大長を設定します。xlimit は s_num 以上である必要があります。
- 対応する行から # を削除して、ゲーティング方法を選択します。
注:morphGateは、スクリプトによって実行される自動ゲーティング方法です(つまり、セルが密集しているドットプロットの領域がプログラムによって認識および選択されます)。polygonGate と rectangleGate では、ドット プロットを確認し、多角形の頂点、またはほとんどのセルが存在するゾーンを囲む四角形の辺を定義する必要があります。 - 選択したゲーティング方法に対応する flowSet オブジェクト gated を選択します。ドットプロットの解像度を選択します(解像度;少なくとも256に等しい必要があります)。
- flt_low <- filter_low(sp)のコメントを解除して、蛍光が陰性である測定値を削除します。flt_sp <- filter(flt_lw) のコメントを解除して、他の実験による外れ値を削除します。
- [ソース] を押して、スクリプトを実行します。解析結果を含むすべてのファイルがdir_rで作成されます。
Access restricted. Please log in or start a trial to view this content.
結果
RNAポリメラーゼIII型プロモーターによるsgRNA/crRNA発現
まず、この作業では、 図1Aに示す転写活性化回路(回路1)のエンジニアリングについて説明しました。それは3つの基本的な成分を含んでいました:1)yEGFPをコードする遺伝子(レポーター)、dCas9 / dCas12a-ADの標的部位を提供する一連の異なる合成プロモーターが先行しました。2)活性化ドメイン(それぞれVP64?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
このプロトコルは、「設計-構築-テスト-学習」(DBTL)生物工学サイクルに従い、ドライラボ実験とウェットラボ実験の両方に関する合成遺伝子デジタル回路の完全なワークフローの可能性を示しました。ここでは、出 芽酵母 の低分子転写ネットワークを設計・構築することにより、主にdSpCas9、denAsCas12a、dLbCas12a、および対応する抗CRISPRタンパク質であるCRISPR-Casシステムに焦点を当てま?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者は、競合する金銭的利益を宣言しません。
謝辞
合成生物学ラボ(SPST、TJU)のすべての学生に、FACS実験に協力してくれたZhi LiとXiangyang Zhangに感謝したいと思います。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | - | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | - | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | - | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | - | - | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
参考文献
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083(2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459(2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194(2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253(2007).
- Green, M. R., Sambrook, J. Molecular Cloning. Fourth edition. , Cold Spring Harbor Laboratory Press. NY. (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19(2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179(2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45(2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106(2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11(2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved